
中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘良叙, 李朝风, 王嘉伟, 倪俊, 许平
- Liu Liangxu, Li Chaofeng, Wang Jiawei, Ni Jun, Xu Ping
- 芳香类天然产物的合成生物学研究进展
- Synthetic biology for the synthesis of aromatic natural products: a review
- 生物工程学报, 2021, 37(6): 2010-2025
- Chinese Journal of Biotechnology, 2021, 37(6): 2010-2025
- 10.13345/j.cjb.210074
-
文章历史
- Received: January 23, 2021
- Accepted: March 28, 2021
- Published: April 30, 2021
芳香类天然产物是一类来自生物界具有苯环结构的有机化合物,包括对羟基肉桂酸及其衍生物、黄酮类、芪类、香豆素类和芳香类生物碱等,其种类繁多,数量庞大,已知的有数万种以上[1]。根据化学结构的不同,主要分为C6·C1、C6·C2、C6·C3、C6·C2·C6和C6·C3·C6五类(图 1)。C6代表一个芳香环,C1代表一个碳原子,C2、C3代表碳链原子数[2]。C6·C1是带有一个芳香环的化合物,如香草酸、原儿茶酸、没食子酸和苯甲酸等。它们大多数具有抗菌消炎的作用,被广泛的应用于医药行业。同时,部分酚类化合物是性能优良的食品抗氧化剂和调味剂,是食品行业中不可缺少的一部分;没食子酸作为半导体光致抗蚀原料也应用于高分子材料领域[3]。C6·C2型化合物如羟基苯乙酮类化合物(2-羟基苯乙酮、3-羟基苯乙酮、4-羟基苯乙酮等),可作为生化试剂、医药原料、有机化工的原料及食品添加剂。C6·C3型化合物包含对羟基肉桂酸及其衍生物,如阿魏酸、咖啡酸、对香豆酸、芥子酸和香豆素等,具有抗氧化、抗肿瘤、抗癌的功效而被广泛应用于医药行业。其中,木质素是C6·C3型化合物的聚合物,含有丰富的芳环结构、脂肪族和芳香族羟基以及醌基等活性基团,被应用于农业和化工(树脂、感光元件) 等行业。C6·C2·C6型化合物多是芪类化合物,如白藜芦醇、银松素、紫檀芪、松芪和银松素甲醚等,多数具有抗菌、降血脂血压、护肝、抗肿瘤的作用,主要应用于医药行业[4]。C6·C3·C6型化合物有黄酮及其苷类(黄酮、异黄酮、新黄酮等)、原花青素和综合性单宁等,具有抗氧化、消炎、降低血糖的功效。
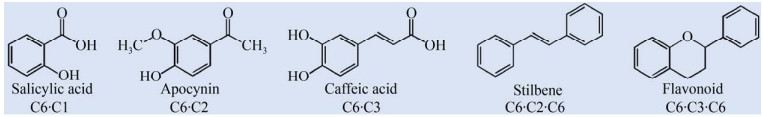
|
| 图 1 芳香类天然产物的分类 Fig. 1 Classification of aromatic natural products. According to the different chemical structures of aromatic compounds, they are divided into five categories: C6·C1, C6·C2, C6·C3, C6·C2·C6 and C6·C3·C6. C6 represents an aromatic ring, C1 represents a carbon atom, C2 and C3 represent the number of carbon chain atoms. |
| |
芳香类天然产物广泛存在于植物中,这些天然产物的存在,关系到植物的生长调节及对病虫害侵袭的防御作用[5]。但这类物质在植物中的含量较低,通常受到来源的限制而无法进行大规模生产。工业上常用蒸馏法、压榨法、溶剂浸出法、组分分馏法和化学分馏法等方法来提取芳香类天然产物,但稳定性差及纯度低阻碍了大多数芳香类天然产物的开发与应用。近年来,研究人员在合成生物学理论的指导下,使用代谢工程技术开发出了生物法制备芳香类天然产物的新策略[6]。生物法制备芳香类天然产物具有周期快、产量高、纯度高、易于分离纯化、可再生和对环境无污染的优点,已成为天然产物研究的热潮。本文重点综述了芳香类天然产物的合成生物学研究进展,并探讨了当前研究所面临的挑战,以期为芳香类天然产物生物合成的进一步研究提供参考。
1 芳香类天然产物的合成途径及关键酶芳香类天然产物的生物合成途径源于莽草酸途径,又称为分支酸途径,主要存在于植物、真菌和细菌中。莽草酸途径始于磷酸烯醇式丙酮酸(Phosphoenolpyruvate,PEP) 和赤藓糖-4-磷酸(Erythrose 4-phosphate,E4P) 缩合形成3-脱氧-D-阿拉伯庚酮糖酸-7-磷酸(3-deoxy-D-arabino- heptulosonate 7-phosphate,DAHP),之后经3-脱氢奎尼酸合成酶(3-dehydroquinate synthase,AroB) 去磷酸化反应,羟醛缩合生成3-脱氢奎尼酸(3-dehydroquinate,DHQ),经3-脱氢奎尼酸脱水酶(3-dehydroquinate dehydratase, AroD) 作用生成3-脱氢莽草酸(3-dehydroshikimate,DHS),后经莽草酸脱氢酶(Shikimate dehydrogenase,AroE) 催化生成莽草酸(Shikimic acid,SA),再由莽草酸生成芳香氨基酸和其他芳香族化合物[7]。
芳香类天然产物合成次级代谢途径(图 2) 和莽草酸途径之间存在着一些关键酶[8-9]:苯丙氨酸解氨酶(Phenylalanine ammonia-lyase,PAL)、酪氨酸解氨酶(Tyrosine ammonia-lyase,TAL)、肉桂酸-4-羟化酶(Cinnamate-4-hydroxylase,C4H)和4-香豆酸辅酶A连接酶(4-coumarate: coenzyme A ligase,4CL) 等。PAL和TAL通过非氧化脱氨作用将苯丙氨酸和酪氨酸分别转化为肉桂酸和对香豆酸,C4H能将肉桂酸对位羟化为对香豆酸,而4CL可以催化肉桂酸及其羟基或甲氧基衍生物生成相应的辅酶A酯[10],这些中间产物随后进入苯丙烷代谢途径。在苯丙烷途径中,查尔酮合酶(Chalcone synthase,CHS) 是黄酮类化合物合成的关键酶,将苯丙烷类代谢中心途径引向黄酮类合成支路[11],二羟基黄烷酮还原酶(Dihydroflavonol reductase,DFR)、无色花青素双加氧酶(Leucoanthocyanidin dioxygenase,LDOX) 和UDPG-黄酮葡萄糖基转移酶(UDPG-flavonoid glucosyl transferase,UFGT) 等酶在花青素和单宁的合成过程中起到关键作用[12]。芪合酶(Stilbene synthase,STS) 是芪类合成途径的关键酶,将苯丙烷类代谢中心途径引向芪类合成支路,STS分成2种类型,一种催化1分子对香豆酰辅酶A和3分子丙二酰辅酶A聚合成白藜芦醇,另一种合成为银松素[13]。
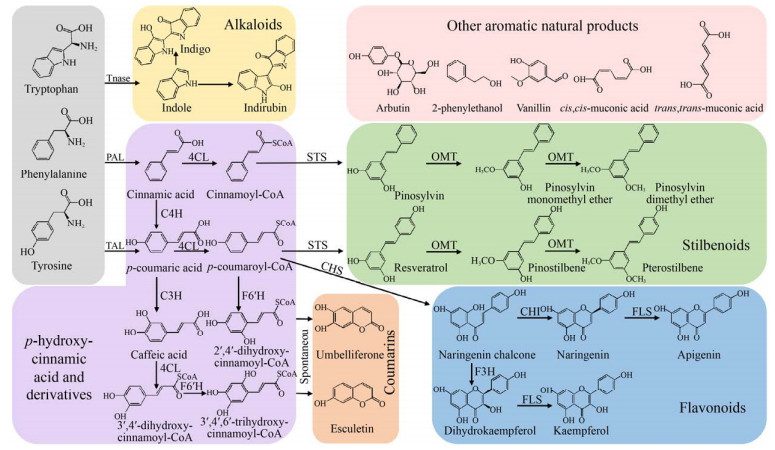
|
| 图 2 芳香类天然产物的合成途径及关键酶 Fig. 2 Synthetic pathways and key enzymes of aromatic natural products. The synthetic pathway of aromatic natural products is mainly the shikimate pathway. Tryptophan, phenylalanine, and tyrosine were converted to hydroxycinnamic acid and derivatives, alkaloids, stilbene, coumarins, flavonoids, and other aromatic compounds the action of different enzymes. Tnase: tryptophanase; PAL: phenylalanine ammonia lyase; TAL: tyrosine ammonia lyase; C4H: cinnamic acid-4-hydroxylase; 4CL: 4-coumarate coenzyme A ligase; STS: stilbene synthase; OMT: methyltransferase; C3H: 4-coumarate-3-hydroxylase; CHS: chalcone synthase; CHI: chalcone isomerase; FLS: flavone synthase; F3H: flavanone-3-hydroxylase; F6′H: flavone-6′-hydroxylase. |
| |
羟基肉桂酸(p-hydroxy cinnamic acid,p-HCA) 及衍生物类是由苯丙氨酸衍生来的参与木质素生物合成途径的酚类化合物,按化学结构可分为羟基和甲氧基数目和取代度不同的羧酸和苯甲酸衍生物,以及性质特殊的酚酸。对香豆酸、咖啡酸、阿魏酸、芥子酸等羟基肉桂酸在植物中几乎是普遍存在的。对香豆酸是生产香料、药品、化妆品、食品和保健品等高值化合物的原料[14]。Ro等在酿酒酵母中成功表达了白杨中编码PAL和C4H的基因,重组菌株可以将苯丙氨酸转化为对香豆酸[15]。Liu等重新设计构建了酵母的中心碳代谢途径,将糖酵解途径的碳流量导向磷酸戊糖途径用于生产更多的赤藓糖-4-磷酸,赤藓糖-4-磷酸是合成对香豆酸的前体,改造后使得对香豆酸的产量提高到12.5 g/L[16]。咖啡酸具有多种药理活性,被用作抗氧化剂、抗炎剂、抗微生物剂和细胞抑制剂。Kawaguchi等在酪氨酸高产的大肠杆菌中表达了铜绿假单胞菌来源的对香豆酸-3-羟化酶和链霉菌WK-5344的酪氨酸解氨酶,利用葡萄糖合成了37.9 mg/L的咖啡酸[17]。Rodrigues等利用热激蛋白启动子dnaK和ibpA来表达酪氨酸解氨酶、对香豆酸-3-羟化酶和细胞色素P450 CYP199A2,并对核糖体结合位点(RBS) 进行了优化和测试,利用大肠杆菌合成了2.5 mmol/L的对香豆酸和370.0 mmol/L的咖啡酸[18]。阿魏酸具有抗炎、抗菌、抗癌、降血糖和抑制黑色素生成等多种生理功能,被广泛用于制药、食品和化妆品行业。An等在大肠杆菌中对酪氨酸解氨酶、苯丙氨酸解氨酶、对香豆酸-3-羟化酶和甲基转移酶进行了不同的组合表达,分别构建了可以生产不同羟基肉桂酸的重组菌株,直接利用葡萄糖生产了100.1 mg/L对香豆酸、138.2 mg/L咖啡酸、64.0 mg/L阿魏酸和1 072.3 mg/L肉桂酸[19]。
2.2 黄酮类黄酮类化合物在自然界广泛存在,属于酚类化合物,主要分为黄酮类和黄烷酮类,泛指2个苯环通过3个碳原子相互结合形成的化合物。黄酮类化合物具有许多生理活性,包括抗氧化、消炎抗菌、抗癌抗肿瘤、抗心血管疾病等作用[20]。松属素(Pinocembrin)、柚皮素(Naringenin) 和圣草酚(Eriodictyol) 是3种最重要的黄酮类化合物,是其他黄酮类化合物衍生物的前体。松属素是从各种植物中分离出的主要类黄酮之一,具有抗菌消炎、抗氧化和抗癌的功能。目前,开发高效的微生物法生产松属素已经成为研究的热点,然而,由于大肠杆菌中丙二酰辅酶A的利用率较低,松属素的生物合成效率受到很大限制。脂肪酸生物合成是大肠杆菌中唯一消耗丙二酰辅酶A的代谢过程,Cao等在大肠杆菌中单独或联合过表达了脂肪酸生物合成途径酶β-酮酰基ACP合成酶Ⅲ (FabH) 和β-酮酰基ACP合成酶Ⅱ(FabF),并研究了其对丙二酰辅酶A的影响。Cao发现在大肠杆菌BL21(DE3) 中过表达FabH和FabF两种酶,增加了丙二酰辅酶A的利用率,减少了脂肪酸的合成。在过表达FabH、FabF、PAL、4CL、CHS和查尔酮异构酶(CHI) 的重组菌株中,松属素的产量最高增加了31.8倍,产量达到29.9 mg/L[21]。2016年,Wu等通过CRISPRi介导的丙二酰辅酶A消耗途径的下调和培养条件的优化,在3 L发酵罐中,将松属素的产量进一步提高到0.53 g/L[22]。柚皮素是二氢黄酮类化合物,具有抗菌消炎、降血脂、抗肿瘤的作用。柚皮素在几种植物中自然产生,它的生物合成已经在苜蓿、欧芹和其他植物中进行了研究。Santos等将一条由CHS、TAL、4CL和CHI组成的途径转入高产酪氨酸的大肠杆菌中,该重组菌株可以将葡萄糖转化为29.0 mg/L的柚皮素,当抑制工程菌中的脂肪酸合成途径时,柚皮素的最高产量可达84.0 mg/L[23]。Xu等分析基因组后,通过代谢工程的调控提高了丙二酰辅酶A的积累,主要上调糖酵解反应,并且下调TCA循环以减少碳的流失,从而将代谢流引入丙二酰辅酶A的合成,基于此可以产生474.0 mg/L柚皮素[24]。除了丙二酰辅酶A,胞内NADPH的供应也是许多苯丙烷类物质合成的一个限制性因素。Chemler等通过化学计量模型确定基因敲除组合来提高胞内NADPH量,从而提高黄酮类的产量。敲除了磷酸葡萄糖异构酶、磷酸烯醇式丙酮酸羧化酶和磷脂酶的重组菌株可以将原花青素和儿茶素的产量分别提高到817.0 mg/L和39.0 mg/L[25]。圣草酚是广泛存在于水果和蔬菜中的多酚黄酮类化合物,具有抗氧化、抗炎、镇痛、改善糖尿病及糖尿病并发症的作用。它的合成受到细胞色素P450的表达和细胞内丙二酰辅酶A浓度的影响。Zhu等在大肠杆菌中将黄酮-3′-羟化酶和P450还原酶(CPR) 进行融合表达,同时过表达酪氨酸解氨酶、4-香豆酸酯-CoA连接酶、查尔酮合酶和查尔酮异构酶,使工程化的大肠杆菌从L-酪氨酸中生产出雌黄醇。此外,采用代谢工程技术来提高丙二酰辅酶A的利用率,从而达到新的代谢平衡并重新平衡基因的相对表达,从而增强雌三醇的积累。这种方法使圣草酚的产量比对照菌株高203%,由L-酪氨酸产生的圣草酚达到107.0 mg/L[26]。灯盏花素可以从植物灯盏花中提取, 它具有多种多样的医疗和药用价值,具有扩张脑血管和抗血凝的作用[27]。Liu等通过对灯盏花基因组数据库进行了深度研究和发掘,成功定位到在灯盏花素合成途径中的两个限速酶(类黄酮-7-O-葡萄糖醛酸转移酶和黄酮-6-羟化酶),之后借助酿酒酵母作为表达系统,合成出了灯盏花素并将其产量提高至108 mg/L[27]。紫云英苷能够提升机体免疫力、保护生物对抗外界病毒的能力,还可以抗应激、促进生长以及改善心肺功能。但由于其在天然产物中含量低,提取和纯化困难,因此可以通过微生物法来生产[28]。Pei等通过筛选关键酶基因,设计合成融合酶,增加基因拷贝数,构建了具有山奈酚高效生物合成途径的重组菌株。同时,通过分批补料发酵条件,使山奈酚产量达到(1 184.2±16.5) mg/L。在此基础上,将糖基转移酶(AtUGT78D2) 和高效的UDP葡萄糖合成途径引入到重组菌株中生产紫云英苷,使其产量达到(1 738.5±24.8) mg/L[28]。
2.3 芪类芪类化合物是指具有均二苯乙烯母核或其聚合物的一类物质的总称。这类物质在植物的正常组织中含量较低,单和二酚羟基芪类化合物主要存在于植物组织木质部的薄壁细胞中,如云杉、松柏、花生等[29]。白藜芦醇是三羟基芪类,存在于大黄、葡萄、花生、蓝莓和覆盆子等植物中[30]。白藜芦醇具有降血脂、抗癌抗肿瘤和预防心脏病的功效。目前市场上的白藜芦醇通常是从植物中提取的,但是这种方法存在不稳定、纯度低、产量低和受季节变化的问题。研究人员利用不同微生物合成了白藜芦醇,包括大肠杆菌、谷氨酸棒杆菌、乳酸乳球菌等[30]。Lim等通过基因工程技术,重新设计构建了高产白藜芦醇的大肠杆菌菌株,提高了细胞内白藜芦醇前体丙二酰辅酶A的含量,最终使白藜芦醇滴度提高到2.3 g/L[31]。Zhao等通过对PcTAL、At4CL和VvSTS三个基因的表达模式进行调控,增加了丙二酰辅酶A的浓度,增加了白藜芦醇的产量,使白藜芦醇的滴度达到238.7 mg/L[32]。Shrestha等采用模块化构建代谢途径,在大肠杆菌中合成白藜芦醇及其邻羟基化衍生物,发现当同时添加乙酸钠和丙二酸二钠时,模块Ⅰ和两个不同的模块Ⅱ基因组合生成白藜芦醇可达137.0 mg/L[33]。
2.4 香豆素类香豆素是顺式邻羟基肉桂酸的内酯,香豆素是植物的次生代谢产物,它的衍生物广泛分布于植物界,如豆科、兰科、芸香科、茄科、菊科等高等植物中,少数也存在于低等植物和微生物中[3]。香豆素具有抗菌消炎、安神镇静、抗癌和抗艾滋病等作用。根据结构的复杂程度,分为简单香豆素、吡喃香豆素、呋喃香豆素及其他异香豆素4大类[34]。Lin等通过在大肠杆菌中建立了一条不同于苯丙烷类物质天然合成途径的新途径,首次实现了4-羟基香豆素的异源合成。这个途径由分支酸作为前体,丙二酰辅酶A作为碳单位,利用异分支酸合成酶(ICS)、异分支酸丙酮酸裂解酶(IPL)、水杨酰辅酶A连接酶(SCL) 和喹诺酮合成酶(BIS) 在大肠杆菌中成功生产了约500 mg/L的4-羟基香豆素[35]。Yang等构建了含有阿魏酰辅酶A羟化酶和4CL的重组菌株,在大肠杆菌中生物合成了香豆素类衍生物伞形酮(82.9 mg/L)、东莨菪素(79.5 mg/L) 和七叶皂素(52.3 mg/L)[36]。
2.5 芳香类生物碱芳香类生物碱广泛分布于植物和微生物中,异喹啉生物碱又称苄基异喹啉生物碱,是最大的一类生物碱[37]。Fossati等在酿酒酵母中重建了一个由10个基因组成的植物通路,该通路允许从(R, S)-norlaudanosoline中生产二氢血根碱及其氧化衍生物血根碱,这项工作是迄今为止酵母中最长的重组生物碱途径,并证明了在微生物系统中生产高价值生物碱的可行性[38]。Galanie等将酶工程、代谢途径和菌株优化相结合,实现了酵母生物合成阿片类药物,为其他异喹啉生物碱的合成提供了思路[39]。Trenchard等通过对酵母中心代谢途径进行修饰,提高酵母的代谢能力,增加苄基异喹啉生物碱前体酪氨酸的供应,从而能够在不供应外源酪氨酸的情况下,使苄基异喹啉的产量增加60倍,进一步通过引入突变的酪氨酸羟化酶,使产量提高了160倍[40]。但目前来讲,利用合成生物学方法生产异喹啉类生物碱仍存在许多问题,如合成途径复杂、操作困难、产量普遍偏低等,因此如何更高效地生产这类生物碱亟待解决。
2.6 其他芳香类天然产物香兰素是从香荚兰中提取的一种芳香醛(3-甲氧基-4-羟基苯甲醛),属于酚类化合物,是苯丙烷类的衍生物。香兰素被认为是世界上最重要的调味品之一,广泛应用于食品、饮料和制药行业[41]。许多微生物可以将阿魏酸、丁香酚、异丁香酚、木质素、香草胺和松柏醇等化合物转化为香兰素。比如Hua等利用链霉菌V-1进行补料发酵,并使用树脂DM11对产物进行吸附,最终可以将45 g/L阿魏酸转化为19.2 g/L香兰素[42]。Yoon等构建了包含阿魏酰CoA合酶和烯酰CoA水合酶的基因工程大肠杆菌,可以将阿魏酸以78%的摩尔转化率转化为1.12 g/L香兰素[43]。Plaggenborg等在红球菌PD630中表达了香草醇氧化酶、松柏醇脱氢酶和松柏醛脱氢酶,从而构建了将丁香酚转化为香兰素的合成途径[44]。从头合成香兰素的优势是可以利用价格低廉、来源广泛的单糖为底物,Kunjapur等通过对芳香醛还原酶的敲除构建了一个芳香醛生产的工程菌株,当引入香兰素从头合成的途径后,重组菌株可以利用葡萄糖产生119.0 mg/L香兰素[45]。Ni等在酪氨酸高产的大肠杆菌中表达了TAL、C3H、甲基转移酶、阿魏酰CoA合酶和烯酰CoA水合酶,利用葡萄糖和甘油等多种碳源合成了香兰素[46]。熊果苷是一种存在于多种植物中的对苯二酚苷类化合物,又名熊果素,可以由杜鹃花科植物熊果叶中萃取而来。它能够通过抑制体内酪氨酸酶的活性,阻止黑色素的生成,从而减少皮肤色素沉积,祛除色斑和雀斑[47]。目前熊果苷的生产多采用植物提取法或酶法,产品收率低,加工成本高[47]。Shen等在大肠杆菌中建立了一条从简单碳源中高效生产熊果苷的人工途径,进一步将碳通量定向到熊果苷生物合成途径,并通过优化培养基条件,使摇瓶实验中熊果苷的产量进一步提高到4.19 g/L[48]。Wang等把染色体整合在天然启动子Pphz上,构建了一条新的熊果苷生物合成途径。并通过葡萄糖与4-羟基苯甲酸混合补料分批发酵的基础上,利用代谢工程方法显著提高了熊果苷的产量,产量可达6.79 g/L[49]。
3 芳香族化合物合成生物学使能策略芳香族化合物传统上来源于石油原料,随着合成生物学和代谢组学的发展,近半个世纪以来,生物法制备芳香类天然产物已经取得了一些突破(图 3)。目前,研究人员对生物法生产芳香类产物采用了多种方法(图 4):对天然和非天然途径的构建;模块化设计途径,例如莽草酸途径的解反馈抑制;开发新型代谢调控工具(如CRISPRi、Small RNA);基于生物传感器的产量优化;使用廉价碳源高值化生产芳香类天然产物等新型策略[74]。
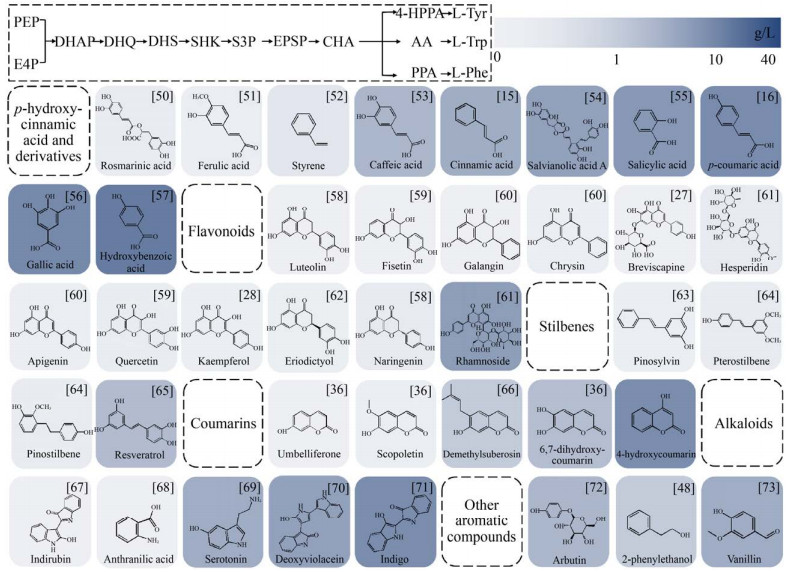
|
| 图 3 常见芳香类天然产物生物合成现状 Fig. 3 Current status of biosynthesis of aromatic natural products. PEP: phosphoenolpyruvate; E4P: erythrose 4-phosphate; DHAP: dihydroxyacetone phosphate; DHQ: dehydroquianate; DHS: dehydroshikimate; SHK: shikimate; S3P: shikimate-3-phosphate; EPSP: 5-enolpyruvyl-shikimate 3-phosphate; CHA: chorismate; 4-HPPA: 4-hydroxyphenylpyruvate; AA: anthranilic acid; PPA: phenylpyruvate. Different shades of color indicate the yield of aromatic compounds synthesized by microorganisms (g/L). |
| |

|
| 图 4 合成生物学策略 Fig. 4 Synthetic biology strategies for production of aromatic natural products. Synthetic biology strategies: construction of natural and unnatural pathways (A), modular design pathways (B), development of new metabolic regulation tools (CRISPRi, sRNA) (C), production optimization based on biosensors (D), and high-value utilization of cheap carbon sources (E). |
| |
莽草酸途径是微生物合成芳香类天然产物的主要途径,增强莽草酸途径的代谢通量,能提高芳香类天然产物的合成产量。目前采取的策略主要有转运系统的工程化[75]、竞争途径的弱化[76]和优化关键酶[77]。研究人员对糖酵解途径和磷酸戊糖途径进行了一定的优化,包括过表达PEP生物合成的关键基因或者下调PEP代谢基因,以此促进PEP的生成量从而增强其向芳香类天然产物生物途径方向的分流,以及通过限制糖酵解途径将更多碳流量导向磷酸戊糖途径用于赤藓糖-4- 磷酸的合成。如Dell等通过在大肠杆菌中过表达芳香酸合成途径中的限速酶AroB,去反馈抑制DAHP合成酶,敲除限速酶AroK和AroL改造莽草酸合成途径,使重组菌株产分支酸(Chorismic acid) 产量达27.2 g/L[78]。
3.2 芳香类天然产物合成的模块化设计模块化设计是指将多步骤的代谢通路分为离散的几个模块,便于组合筛选相关表达参数,以达到最佳表达水平和最大通路流量的目的,可分为模块化合成途径和细胞共培养技术[79]。模块化设计具有分担代谢负担、简易调控以及优化每个模块的代谢通量的优势,能够方便快捷地生产目的芳香类天然产物。Juminaga等将酪氨酸的生物合成途径分解为莽草酸模块和酪氨酸模块,对每个基因的拷贝数、启动子的强度、基因在每个操纵子中的相对位置进行系统优化,在大肠杆菌中高效生产酪氨酸,产量达到2 g/L以上[80]。Wu等采用相似的策略在大肠杆菌中分3个模块构建柚皮素生物合成途径,通过同时改变3个模块的质粒拷贝数和启动子强度,柚皮素的最终产量提高了近3倍,产量达100 mg/L[81]。模块化细胞的发展也涉及将生物合成路径划分为不同的模块,只是这些模块被进一步分配到单个细胞之中,这种方法将实现更好的工作分配,减少单个细胞中过表达多种酶的不良影响,促进目的产物的生成[82]。Zhang等最近开发了大肠杆菌共培养系统来研究从葡萄糖到3-氨基苯甲酸的生产,将代谢途径分为“上游”和“下游”,相较于纯培养,共培养条件下3-氨基苯甲酸产量提高了15倍[83]。Zhang等构建了大肠杆菌与酿酒酵母的共培养体系实现从木糖中高效生产柚皮素,其中大肠杆菌作为一个良好的宿主来生产前体酪氨酸,其余的真核途径基因在酿酒酵母中得到表达,最终产量达(21.16±0.41) mg/L[84]。
3.3 新型代谢调控工具近年来,在常规基因敲除不兼容的情况下,多种代谢调控工具可以用来控制代谢通路,使个体生长所必需的基因被靶向下调,并且在多个途径中可重复使用。目前为止,最具前景的策略包括反义RNA (asRNAs)、小RNA (sRNA)、成簇的规律间隔的短回文重复序列干扰(CRISPRi) 及利用合成电路进行条件基因表达[79]。例如CRISPRi、sRNAs类新型代谢调控工具已被用在芳香族化合物生物生产,并发挥着巨大的潜力。CRISPRi是一种基于Cas9的基因表达控制方法,Cas9是来自Ⅱ型CRISPR系统的RNA引导的DNA核酸内切酶,可以特异性地干扰转录,同时抑制多个靶基因的表达[85]。2016年,Wu等构建了一个规则的、相互间隔的短回文重复干扰,以有效地引导碳通量流向丙二酰辅酶A,使得松属素产量增加至525.8 mg/L[22]。2018年,DeLorenzo等基于CRISPRi技术,利用红球菌Rhodococcus opacus PD630实现了木质素的高值化[86]。另外Cress等报道了CRISPR替代系统CRISPathBrick,它是利用Ⅱ-A型CRISPR来实现基因的抑制,并通过提高柚皮素的产量来验证该替代体系的有效性[87]。合成sRNAs可以靶向特定的mRNA序列,抑制翻译,由于与RNA伴侣的关联和稳定,sRNA同时在提高沉默效率和延长半衰期方面也发挥着作用。Na等报道sRNAs用于生物合成化合物,通过构建sRNAs同时抑制大肠杆菌中TyrR和CsrA的产生,最终实现酪氨酸滴度为2 g/L[88]。同样的系统被用来生产苯酚,最终滴度达1.69 g/L[89]。2015年,Yang等合成RNAs以调节丙二酰辅酶A的代谢,使丙二酰辅酶A的产量增加了4.5倍,4-羟基香豆素、白藜芦醇和橙皮苷的产量分别提高了2.53倍、1.70倍和1.53倍[90]。
3.4 基于生物传感器的产量优化微生物已经进化出各种机制来感知和响应细胞内外的化学变化,包括通过使用许多小分子应答转录因子及其同源启动子[91]。当与一个合适的读出器例如荧光报告基因或抗生素抗性基因相连接时,即在细胞内构建成一个生物传感器,可用于检测和响应细胞内不同产品或中间体的存在。这种生物传感器通常具有高选择性和敏感性,并以剂量依赖的方式存在[92-93]。2015年,Gayathri等开发了基于DNA纳米结构的生物传感器,用于测定芳香族化合物[94]。2017年,John等对生物传感器定向进化,改善了酿酒酵母中粘康酸的产量,使粘康酸的产量提高了近3倍,在分批补料的生物反应器中产生2.1 g/L的粘康酸[95]。2018年,Ray等使用高度稳定的基于酶的体外生物传感器来检测苯酚,准确度达到95%,后经过进一步优化,开发了2, 3-二甲基苯酚传感器,可用于其他芳香类化合物的传感器,为筛选芳香类天然产物提供了可行的策略[96]。
能响应各种芳香化合物的转录调节因子已被报道,以此开发生物传感器来促进微生物体内的芳香族化合物的生产是一种极具潜力的生物合成策略[97]。例如Liu等开发了一种由谷氨酸棒状杆菌的LysR型转录调节子(shiR) 构成的莽草酸生物传感器。使用天然的莽草酸转录调控因子shiR和绿色荧光蛋白(GFP) 作为报告因子用于筛选RBS文库以优化用于莽草酸生产的基因表达,并通过荧光激活细胞分选检测来促进选择,最终莽草酸的产量提高了2.4倍[98]。通过利用大肠杆菌的天然TyrR (一种控制莽草酸途径多个基因表达的转录抑制因子) 来检测苯丙氨酸水平的提高,并从其同源TyrP启动子中驱动黄色荧光蛋白的表达,建立了一个筛选平台来改善苯丙氨酸的生物合成[99]。
3.5 利用廉价碳源生产芳香类天然产物木质素和木质素来源的芳香化合物是自然界中仅次于纤维素的第二丰富的有机化合物[100],它是一种结构不均一的酚类大分子,其重量占植物细胞壁的10%–40%[101]。考虑到木质素是一个名副其实的芳香前体宝库,一旦解聚,无论是直接输入微生物生物转化途径使其增值为芳香化学品,或是为芳香化合物生物合成提供前体的芳烃或衍生产品方面都极具研究前景。目前而言大多数研究聚焦于木质素解聚后的木质素衍生单体的应用上[102]。Ni等开发了一种温度导向的全细胞催化剂,它是通过两种来源于嗜热放线菌的新型酶实现的,在30 ℃的反应温度下,木质素衍生物阿魏酸有效地转化为香草醇,当温度升高至50 ℃时,阿魏酸主要转化为香兰素,产率为1.1 g/(L·h),这是由于内源性醇脱氢酶在这个温度下是不活跃的,而来自嗜热放线菌的新型酶仍然维持着较高的活力[103]。2018年,该团队设计了一种不依赖辅酶的生物催化剂,可以将木质素衍生的芳烃转化为各种具有吸引力的高附加值产物,主要是利用新型的酚酸脱羧酶和芳香双加氧酶极高的催化效率,以不依赖辅酶的方式实现木质素的高值化利用,并且提出一种新的温度和pH导向策略来进一步消除内源性醇脱氢酶的高活性[72]。
鉴于气候变化的挑战,利用太阳能和二氧化碳生产芳香类天然产物使蓝细菌作为代谢工程的宿主逐渐成为研究的热点。光能自养原核微生物蓝藻作为地球碳循环的重要驱动因子之一[104],具有遗传背景简单、营养需求低和CO2固定效率高等优势。同时,蓝藻可以功能性地组装细胞色素P450单加氧酶,以及胞内充足的NADPH和ATP使其成为生产植物天然产物的理想平台[105]。Ni等基于光合微生物蓝藻PCC7942和合成生物学技术,构建了一个光自养合成平台,可直接将温室气体CO2转化为白藜芦醇、柚皮素、双去甲氧基姜黄素、香豆酸、咖啡酸和阿魏酸,这6种天然高值产物可以进一步衍生出其他高值的天然产物[105]。该团队还通过重塑光合链促进CO2转化为有价值的芳香族化合物,通过在蓝藻中引入2-苯乙醇途径和人工解反馈抑制模块,超过30%的固定碳被重定向到莽草酸途径进行苯乙醇的合成,并且碳固定和O2的产生显著增加[106]。Brey等在蓝藻Synechocystis sp. PCC6803中异源表达大肠杆菌中解反馈抑制的AroG和TyrA酶以实现从CO2中高效生产苯丙氨酸和酪氨酸,在光照培养10 d后,苯丙氨酸的滴度为(580±34) mg/L,酪氨酸的生产滴度为(41±2.3) mg/L,由于这两种氨基酸是苯丙烷的合成前体,该团队继续在蓝藻中测试了几种苯丙烷生物合成酶的功能,在光照培养6 d后分别得到对香豆酸(207.0 mg/L)、肉桂酸(114.0 mg/L) 和咖啡酸(12.6 mg/L)[107]。
4 总结与展望能源短缺、资源匮乏和环境污染已成为世界性难题,基于可再生生物质资源生产天然产物成为有效的解决途径之一。近年来,人们对天然芳香族产物的重大工业意义和天然合成途径的理解不断深入,伴随着合成生物学正在以前所未有的速度发展,并且与计算机科学的交叉,开辟了合成生物学用于生产天然产物的新时代,以合适的微生物作为底盘合成芳香类天然产物及其衍生物成为合理、经济、绿色的选择。然而该领域仍然面临着一些挑战:1) 大多数芳香族生物化学品的市场应用潜力仍受到石油衍生产品低成本的挑战,解决这一问题的关键是从廉价和可持续的原料中实现高产芳香烃生物合成。2) 微生物底盘代谢网络的复杂性,使得合成途径的理性设计和优化成为芳香类天然产物合成生物学的主要难点之一。3) 芳香毒性仍然是一个持续性的挑战,进一步优化来提高重组菌株的耐受性对于工业化生物生产芳香类天然产物无疑十分重要。未来芳香类天然产物的合成需要以经济低碳为理念,针对上述问题,提出未来潜在的研究方向:1) 生物炼制会产生大量的木质素废弃物,燃烧这些废弃物会造成环境污染。而木质纤维素主要由纤维素、半纤维素和木质素组成,是地球上最丰富的可再生资源之一。使木质素高效解聚生产高价值的芳香产物是一种有利的木质素高值化策略。另一方面,全球变暖将引发海平面上升、高寒灌丛生存空间减少、高海拔特有物种消失以及两极地区冰封的超级细菌和远古病毒“复出”等问题,CO2是最主要的温室气体,是造成温室效应的主要原因。中国和欧盟分别计划在2060年和2050年实现“碳中和”的目标。光合微生物能够以光能为能量来源,将CO2转化为有机物,提供了一条CO2资源化利用的途径。基于合成生物学和代谢工程的理念,在蓝藻中设计和重构芳香类天然产物的合成途径,可将CO2直接转化为高附加值的芳香族化合物。2) 进一步了解代谢相关通路及调控机制,开发新型代谢调控工具以及对天然和非天然途径的重塑,改变碳通量的流速和流向,突破本领域技术壁垒,以期获得更高的产量。3) 通过基因工程手段,定向筛选出具有芳香类化合物耐受性的工业化高产菌株,并对高密度发酵技术进行放大优化。总而言之,随着合成生物学的发展,先进技术和工具的出现势必会加速高效细胞工厂的开发,必将实现芳香类化合物的低成本工业化生产。
| [1] |
Cao MF, Gao MR, Suástegui M, et al. Building microbial factories for the production of aromatic amino acid pathway derivatives: from commodity chemicals to plant-sourced natural products. Metab Eng, 2020, 58: 94-132. DOI:10.1016/j.ymben.2019.08.008
|
| [2] |
Weisshaar B, Jenkins GI. Phenylpropanoid biosynthesis and its regulation. Curr Opin Plant Biol, 1998, 1(3): 251-257. DOI:10.1016/S1369-5266(98)80113-1
|
| [3] |
Ul Amin FU, Ali Shah S, Kim MO. Vanillic acid attenuates Aβ1-42-induced oxidative stress and cognitive impairment in mice. Sci Rep, 2017, 7: 40753. DOI:10.1038/srep40753
|
| [4] |
Salehi B, Mishra AP, Nigam M, et al. Resveratrol: a double-edged sword in health benefits. Biomedicines, 2018, 6(3): 91. DOI:10.3390/biomedicines6030091
|
| [5] |
Deng YX, Lu SF. Biosynthesis and regulation of phenylpropanoids in plants. Crit Rev Plant Sci, 2017, 36(4): 257-290. DOI:10.1080/07352689.2017.1402852
|
| [6] |
Yang D, Park SY, Park YS, et al. Metabolic engineering of Escherichia coli for natural product biosynthesis. Trends Biotechnol, 2020, 38(7): 745-765. DOI:10.1016/j.tibtech.2019.11.007
|
| [7] |
Averesch NJH, Krömer JO. Metabolic engineering of the shikimate pathway for production of aromatics and derived compounds—present and future strain construction strategies. Front Bioeng Biotechnol, 2018, 6: 32. DOI:10.3389/fbioe.2018.00032
|
| [8] |
Li Z, Wang HY, Ding DQ, et al. Metabolic engineering of Escherichia coli for production of chemicals derived from the shikimate pathway. J Ind Microbiol Biotechnol, 2020, 47(6/7): 525-535. DOI:10.1007/s10295-020-02288-2
|
| [9] |
Barros J, Dixon RA. Plant phenylalanine/tyrosine ammonia-lyases. Trends Plant Sci, 2020, 25(1): 66-79. DOI:10.1016/j.tplants.2019.09.011
|
| [10] |
Lavhale SG, Kalunke RM, Giri AP. Structural, functional and evolutionary diversity of 4-coumarate-CoA ligase in plants. Planta, 2018, 248(5): 1063-1078. DOI:10.1007/s00425-018-2965-z
|
| [11] |
Wang Y, Dou Y, Wang R, et al. Molecular characterization and functional analysis of chalcone synthase from Syringa oblata Lindl. in the flavonoid biosynthetic pathway. Gene, 2017, 635: 16-23. DOI:10.1016/j.gene.2017.09.002
|
| [12] |
Zhu Y, Peng QZ, Li KG, et al. Molecular cloning and functional characterization of a dihydroflavonol 4-reductase from Vitis bellula. Molecules, 2018, 23(4): 861. DOI:10.3390/molecules23040861
|
| [13] |
Tyunin AP, Nityagovsky NN, Grigorchuk VP, et al. Stilbene content and expression of stilbene synthase genes in cell cultures of Vitis amurensis treated with cinnamic and caffeic acids. Biotechnol Appl Biochem, 2018, 65(2): 150-155. DOI:10.1002/bab.1564
|
| [14] |
Adisakwattana S. Cinnamic acid and its derivatives: mechanisms for prevention and management of diabetes and its complications. Nutrients, 2017, 9(2): 163. DOI:10.3390/nu9020163
|
| [15] |
Ro DK, Douglas CJ. Reconstitution of the entry point of plant phenylpropanoid metabolism in yeast (Saccharomyces cerevisiae): implications for control of metabolic flux into the phenylpropanoid pathway. J Biol Chem, 2004, 279(4): 2600-2607. DOI:10.1074/jbc.M309951200
|
| [16] |
Liu QL, Yu T, Li XW, et al. Rewiring carbon metabolism in yeast for high level production of aromatic chemicals. Nat Commun, 2019, 10: 4976. DOI:10.1038/s41467-019-12961-5
|
| [17] |
Kawaguchi H, Katsuyama Y, Du DY, et al. Caffeic acid production by simultaneous saccharification and fermentation of kraft pulp using recombinant Escherichia coli. Appl Microbiol Biotechnol, 2017, 101(13): 5279-5290. DOI:10.1007/s00253-017-8270-0
|
| [18] |
Rodrigues JL, Couto MR, Araújo RG, et al. Hydroxycinnamic acids and curcumin production in engineered Escherichia coli using heat shock promoters. Biochem Eng J, 2017, 125: 41-49. DOI:10.1016/j.bej.2017.05.015
|
| [19] |
An DG, Cha MN, Nadarajan SP, et al. Bacterial synthesis of four hydroxycinnamic acids. Appl Biol Chem, 2016, 59(2): 173-179. DOI:10.1007/s13765-015-0137-4
|
| [20] |
Perez-Vizcaino F, Fraga CG. Research trends in flavonoids and health. Arch Biochem Biophys, 2018, 646: 107-112. DOI:10.1016/j.abb.2018.03.022
|
| [21] |
Cao WJ, Ma WC, Zhang BW, et al. Improved pinocembrin production in Escherichia coli by engineering fatty acid synthesis. J Ind Microbiol Biotechnol, 2016, 43(4): 557-566. DOI:10.1007/s10295-015-1725-3
|
| [22] |
Wu JJ, Zhang X, Zhou JW, et al. Efficient biosynthesis of (2S)-pinocembrin from D-glucose by integrating engineering central metabolic pathways with a pH-shift control strategy. Bioresour Technol, 2016, 218: 999-1007. DOI:10.1016/j.biortech.2016.07.066
|
| [23] |
Santos CNS, Koffas M, Stephanopoulos G. Optimization of a heterologous pathway for the production of flavonoids from glucose. Metab Eng, 2011, 13(4): 392-400. DOI:10.1016/j.ymben.2011.02.002
|
| [24] |
Xu P, Ranganathan S, Fowler ZL, et al. Genome-scale metabolic network modeling results in minimal interventions that cooperatively force carbon flux towards malonyl-CoA. Metab Eng, 2011, 13(5): 578-587. DOI:10.1016/j.ymben.2011.06.008
|
| [25] |
Chemler JA, Fowler ZL, McHugh KP, et al. Improving NADPH availability for natural product biosynthesis in Escherichia coli by metabolic engineering. Metab Eng, 2010, 12(2): 96-104. DOI:10.1016/j.ymben.2009.07.003
|
| [26] |
Zhu SJ, Wu JJ, Du GC, et al. Efficient synthesis of eriodictyol from L-tyrosine in Escherichia coli. Appl Environ Microbiol, 2014, 80(10): 3072-3080. DOI:10.1128/AEM.03986-13
|
| [27] |
Liu XN, Cheng J, Zhang GH, et al. Engineering yeast for the production of breviscapine by genomic analysis and synthetic biology approaches. Nat Commun, 2018, 9: 448. DOI:10.1038/s41467-018-02883-z
|
| [28] |
Pei JJ, Chen AN, Dong P, et al. Modulating heterologous pathways and optimizing fermentation conditions for biosynthesis of kaempferol and astragalin from naringenin in Escherichia coli. J Ind Microbiol Biotechnol, 2019, 46(2): 171-186. DOI:10.1007/s10295-018-02134-6
|
| [29] |
De Filippis B, Ammazzalorso A, Fantacuzzi M, et al. Anticancer activity of stilbene-based derivatives. Chem Med Chem, 2017, 12(8): 558-570. DOI:10.1002/cmdc.201700045
|
| [30] |
Thapa SB, Pandey RP, Park YI, et al. Biotechnological advances in resveratrol production and its chemical diversity. Molecules, 2019, 24(14): 2571. DOI:10.3390/molecules24142571
|
| [31] |
Lim CG, Fowler ZL, Hueller T, et al. High-yield resveratrol production in engineered Escherichia coli. Appl Environ Microbiol, 2011, 77(10): 3451-3460. DOI:10.1128/AEM.02186-10
|
| [32] |
Zhao Y, Wu BH, Liu ZN, et al. Combinatorial optimization of resveratrol production in engineered E. coli. J Agric Food Chem, 2018, 66(51): 13444-13453. DOI:10.1021/acs.jafc.8b05014
|
| [33] |
Shrestha A, Pandey RP, Pokhrel AR, et al. Modular pathway engineering for resveratrol and piceatannol production in engineered Escherichia coli. Appl Microbiol Biotechnol, 2018, 102(22): 9691-9706. DOI:10.1007/s00253-018-9323-8
|
| [34] |
Ren QC, Gao C, Xu Z, et al. Bis-coumarin derivatives and their biological activities. Curr Top Med Chem, 2018, 18(2): 101-113. DOI:10.2174/1568026618666180221114515
|
| [35] |
Lin YH, Shen XL, Yuan QP, et al. Microbial biosynthesis of the anticoagulant precursor 4-hydroxycoumarin. Nat Commun, 2013, 4: 2603. DOI:10.1038/ncomms3603
|
| [36] |
Yang SM, Shim GY, Kim BG, et al. Biological synthesis of coumarins in Escherichia coli. Microb Cell Fact, 2015, 14: 65. DOI:10.1186/s12934-015-0248-y
|
| [37] |
Liu D, Meng X, Wu DL, et al. A natural isoquinoline alkaloid with antitumor activity: studies of the biological activities of berberine. Front Pharmacol, 2019, 10: 9. DOI:10.3389/fphar.2019.00009
|
| [38] |
Fossati E, Ekins A, Narcross L, et al. Reconstitution of a 10-gene pathway for synthesis of the plant alkaloid dihydrosanguinarine in Saccharomyces cerevisiae. Nat Commun, 2014, 5: 3283. DOI:10.1038/ncomms4283
|
| [39] |
Galanie S, Thodey K, Trenchard IJ, et al. Complete biosynthesis of opioids in yeast. Science, 2015, 349(6252): 1095-1100. DOI:10.1126/science.aac9373
|
| [40] |
Trenchard IJ, Siddiqui MS, Thodey K, et al. De novo production of the key branch point benzylisoquinoline alkaloid reticuline in yeast. Metab Eng, 2015, 31: 74-83. DOI:10.1016/j.ymben.2015.06.010
|
| [41] |
Banerjee G, Chattopadhyay P. Vanillin biotechnology: the perspectives and future. J Sci Food Agric, 2019, 99(2): 499-506. DOI:10.1002/jsfa.9303
|
| [42] |
Hua DL, Ma CQ, Song LF, et al. Enhanced vanillin production from ferulic acid using adsorbent resin. Appl Microbiol Biotechnol, 2007, 74(4): 783-790. DOI:10.1007/s00253-006-0735-5
|
| [43] |
Yoon SH, Li C, Kim JE, et al. production of vanillin by metabolically engineered Escherichia coli. Biotechnol Lett, 2005, 27(22): 1829-1832. DOI:10.1007/s10529-005-3561-4
|
| [44] |
Plaggenborg R, Overhage J, Loos A, et al. Potential of Rhodococcus strains for biotechnological vanillin production from ferulic acid and eugenol. Appl Microbiol Biotechnol, 2006, 72(4): 745-755. DOI:10.1007/s00253-005-0302-5
|
| [45] |
Kunjapur AM, Tarasova Y, Prather KLJ. Synthesis and accumulation of aromatic aldehydes in an engineered strain of Escherichia coli. J Am Chem Soc, 2014, 136(33): 11644-11654. DOI:10.1021/ja506664a
|
| [46] |
Ni J, Tao F, Du HQ, et al. Mimicking a natural pathway for de novo biosynthesis: natural vanillin production from accessible carbon sources. Sci Rep, 2015, 5: 13670. DOI:10.1038/srep13670
|
| [47] |
Zhu XT, Tian YQ, Zhang WL, et al. Recent progress on biological production of α-arbutin. Appl Microbiol Biotechnol, 2018, 102(19): 8145-8152. DOI:10.1007/s00253-018-9241-9
|
| [48] |
Shen XL, Wang J, Wang J, et al. High-level de novo biosynthesis of arbutin in engineered Escherichia coli. Metab Eng, 2017, 42: 52-58. DOI:10.1016/j.ymben.2017.06.001
|
| [49] |
Wang SW, Fu C, Bilal M, et al. Enhanced biosynthesis of arbutin by engineering shikimate pathway in Pseudomonas chlororaphis P3. Microb Cell Fact, 2018, 17: 174. DOI:10.1186/s12934-018-1022-8
|
| [50] |
Jiang JJ, Bi HP, Zhuang YB, et al. Engineered synthesis of rosmarinic acid in Escherichia coli resulting production of a new intermediate, caffeoyl-phenyllactate. Biotechnol Lett, 2016, 38(1): 81-88. DOI:10.1007/s10529-015-1945-7
|
| [51] |
Kang SY, Choi O, Lee JK, et al. Artificial biosynthesis of phenylpropanoic acids in a tyrosine overproducing Escherichia coli strain. Microb Cell Fact, 2012, 11: 153. DOI:10.1186/1475-2859-11-153
|
| [52] |
McKenna R, Nielsen DR. Styrene biosynthesis from glucose by engineered E. coli. Metab Eng, 2011, 13(5): 544-554. DOI:10.1016/j.ymben.2011.06.005
|
| [53] |
Furuya T, Arai Y, Kino K. Biotechnological production of caffeic acid by bacterial cytochrome P450 CYP199A2. Appl Environ Microbiol, 2012, 78(17): 6087-6094. DOI:10.1128/AEM.01103-12
|
| [54] |
Zhou L, Ding Q, Jiang GZ, et al. Chromosome engineering of Escherichia coli for constitutive production of salvianic acid A. Microb Cell Fact, 2017, 16(1): 84. DOI:10.1186/s12934-017-0700-2
|
| [55] |
Noda S, Shirai T, Oyama S, et al. Metabolic design of a platform Escherichia coli strain producing various chorismate derivatives. Metab Eng, 2016, 33: 119-129. DOI:10.1016/j.ymben.2015.11.007
|
| [56] |
Kambourakis S, Draths KM, Frost JW. Synthesis of gallic acid and pyrogallol from glucose: replacing natural product isolation with microbial catalysis. J Am Chem Soc, 2000, 122(37): 9042-9043. DOI:10.1021/ja000853r
|
| [57] |
Kitade Y, Hashimoto R, Suda M, et al. Production of 4-hydroxybenzoic acid by an aerobic growth-arrested bioprocess using metabolically engineered Corynebacterium glutamicum. Appl Environ Microbiol, 2018, 84(6): e02587-17.
|
| [58] |
Leonard E, Yan YJ, Lim KH, et al. Investigation of two distinct flavone synthases for plant-specific flavone biosynthesis in Saccharomyces cerevisiae. Appl Environ Microbiol, 2005, 71(12): 8241-8248. DOI:10.1128/AEM.71.12.8241-8248.2005
|
| [59] |
Rodriguez A, Strucko T, Stahlhut SG, et al. Metabolic engineering of yeast for fermentative production of flavonoids. Biores Technol, 2017, 245: 1645-1654. DOI:10.1016/j.biortech.2017.06.043
|
| [60] |
Miyahisa I, Funa N, Ohnishi Y, et al. Combinatorial biosynthesis of flavones and flavonols in Escherichia coli. Appl Microbiol Biotechnol, 2006, 71(1): 53-58. DOI:10.1007/s00253-005-0116-5
|
| [61] |
Ruprecht C, Bönisch F, Ilmberger N, et al. High level production of flavonoid rhamnosides by metagenome-derived glycosyltransferase C in Escherichia coli utilizing dextrins of starch as a single carbon source. Metab Eng, 2019, 55: 212-219. DOI:10.1016/j.ymben.2019.07.002
|
| [62] |
Leonard E, Yan YJ, Fowler ZL, et al. Strain improvement of recombinant Escherichia coli for efficient production of plant flavonoids. Mol Pharmaceutics, 2008, 5(2): 257-265. DOI:10.1021/mp7001472
|
| [63] |
Liang JL, Guo LQ, Lin JF, et al. A novel process for obtaining pinosylvin using combinatorial bioengineering in Escherichia coli. World J Microbiol Biotechnol, 2016, 32(6): 102. DOI:10.1007/s11274-016-2062-z
|
| [64] |
Jeong YJ, An CH, Woo SG, et al. Production of pinostilbene compounds by the expression of resveratrol O-methyltransferase genes in Escherichia coli. Enzyme Microb Technol, 2014, 54: 8-14. DOI:10.1016/j.enzmictec.2013.09.005
|
| [65] |
Li ML, Kildegaard KR, Chen Y, et al. De novo production of resveratrol from glucose or ethanol by engineered Saccharomyces cerevisiae. Metab Eng, 2015, 32: 1-11. DOI:10.1016/j.ymben.2015.08.007
|
| [66] |
Bu XL, He BB, Weng JY, et al. Constructing microbial hosts for the production of benzoheterocyclic derivatives. ACS Synth Biol, 2020, 9(9): 2282-2290. DOI:10.1021/acssynbio.9b00405
|
| [67] |
Du JK, Yang D, Luo ZW, et al. Metabolic engineering of Escherichia coli for the production of indirubin from glucose. J Biotechnol, 2018, 267: 19-28. DOI:10.1016/j.jbiotec.2017.12.026
|
| [68] |
Li ZH, Lu YX, Wang XN, et al. Enhancing anthranilic acid biosynthesis using biosensor-assisted cell selection and in situ product removal. Biochem Eng J, 2020, 162: 107722. DOI:10.1016/j.bej.2020.107722
|
| [69] |
Park S, Kang K, Lee SW, et al. Production of serotonin by dual expression of tryptophan decarboxylase and tryptamine 5-hydroxylase in Escherichia coli. Appl Microbiol Biotechnol, 2011, 89(5): 1387-1394. DOI:10.1007/s00253-010-2994-4
|
| [70] |
Fang MY, Wang TM, Zhang C, et al. Intermediate-sensor assisted push-pull strategy and its application in heterologous deoxyviolacein production in Escherichia coli. Metab Eng, 2016, 33: 41-51. DOI:10.1016/j.ymben.2015.10.006
|
| [71] |
Berry A, Dodge TC, Pepsin M, et al. Application of metabolic engineering to improve both the production and use of biotech indigo. J Ind Microbiol Biotechnol, 2002, 28(3): 127-133. DOI:10.1038/sj.jim.7000228
|
| [72] |
Ni J, Wu YT, Tao F, et al. A Coenzyme-free biocatalyst for the value-added utilization of lignin-derived aromatics. J Am Chem Soc, 2018, 140(47): 16001-16005. DOI:10.1021/jacs.8b08177
|
| [73] |
Wang ZY, Jiang MY, Guo XN, et al. Reconstruction of metabolic module with improved promoter strength increases the productivity of 2-phenylethanol in Saccharomyces cerevisiae. Microb Cell Fact, 2018, 17: 60. DOI:10.1186/s12934-018-0907-x
|
| [74] |
Wang J, Shen XL, Rey J, et al. Recent advances in microbial production of aromatic natural products and their derivatives. Appl Microbiol Biotechnol, 2018, 102(1): 47-61. DOI:10.1007/s00253-017-8599-4
|
| [75] |
Ahn J, Chung BKS, Lee DY, et al. NADPH- dependent pgi-gene knockout Escherichia coli metabolism producing shikimate on different carbon sources. FEMS Microbiol Lett, 2011, 324(1): 10-16. DOI:10.1111/j.1574-6968.2011.02378.x
|
| [76] |
Gottlieb K, Albermann C, Sprenger GA. Improvement of L-phenylalanine production from glycerol by recombinant Escherichia coli strains: the role of extra copies of glpK, glpX, and tktA genes. Microb Cell Fact, 2014, 13: 96. DOI:10.1186/s12934-014-0096-1
|
| [77] |
Gu PF, Fan XY, Liang QF, et al. Novel technologies combined with traditional metabolic engineering strategies facilitate the construction of shikimate-producing Escherichia coli. Microb Cell Fact, 2017, 16: 167. DOI:10.1186/s12934-017-0773-y
|
| [78] |
Dell KA, Frost JW. Identification and removal of impediments to biocatalytic synthesis of aromatics from D-glucose: rate-limiting enzymes in the common pathway of aromatic amino acid biosynthesis. J Am Chem Soc, 1993, 115(24): 11581-11589. DOI:10.1021/ja00077a065
|
| [79] |
Machas M, Kurgan G, Jha AK, et al. Emerging tools, enabling technologies, and future opportunities for the bioproduction of aromatic chemicals. J Chem Technol Biotechnol, 2019, 94(1): 38-52. DOI:10.1002/jctb.5762
|
| [80] |
Juminaga D, Baidoo EEK, Redding-Johanson AM, et al. Modular engineering of L-tyrosine production in Escherichia coli. Appl Environ Microbiol, 2012, 78(1): 89-98. DOI:10.1128/AEM.06017-11
|
| [81] |
Wu JJ, Zhou TT, Du GC, et al. Modular optimization of heterologous pathways for de novo synthesis of (2S)-naringenin in Escherichia coli. PLoS ONE, 2014, 9(7): e101492. DOI:10.1371/journal.pone.0101492
|
| [82] |
Jones JA, Wang X. Use of bacterial co-cultures for the efficient production of chemicals. Curr Opin Biotechnol, 2018, 53: 33-38. DOI:10.1016/j.copbio.2017.11.012
|
| [83] |
Zhang HR, Stephanopoulos G. Co-culture engineering for microbial biosynthesis of 3-amino-benzoic acid in Escherichia coli. J Biotechnol, 2016, 11(7): 981-987. DOI:10.1002/biot.201600013
|
| [84] |
Zhang W, Liu H, Li X, et al. Production of naringenin from D-xylose with co-culture of E. coli and S. cerevisiae. Eng Life Sci, 2017, 17(9): 1021-1029. DOI:10.1002/elsc.201700039
|
| [85] |
Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022
|
| [86] |
DeLorenzo DM, Rottinghaus AG, Henson WR, et al. Molecular toolkit for gene expression control and genome modification in Rhodococcus opacus PD630. ACS Synth Biol, 2018, 7(2): 727-738. DOI:10.1021/acssynbio.7b00416
|
| [87] |
Cress BF, Toparlak D, Guleria S, et al. CRISPathBrick: modular combinatorial assembly of type Ⅱ-A CRISPR arrays for dCas9-mediated multiplex transcriptional repression in E. coli. ACS Synth Biol, 2015, 4(9): 987-1000. DOI:10.1021/acssynbio.5b00012
|
| [88] |
Na D, Yoo SM, Chung H, et al. Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs. Nat Biotechnol, 2013, 31(2): 170-174. DOI:10.1038/nbt.2461
|
| [89] |
Kim B, Park H, Na D, et al. Metabolic engineering of Escherichia coli for the production of phenol from glucose. J Biotechnol, 2014, 9(5): 621-629. DOI:10.1002/biot.201300263
|
| [90] |
Yang YP, Lin YH, Li LY, et al. Regulating malonyl-CoA metabolism via synthetic antisense RNAs for enhanced biosynthesis of natural products. Metab Eng, 2015, 29: 217-226. DOI:10.1016/j.ymben.2015.03.018
|
| [91] |
Stanton BC, Nielsen AAK, Tamsir A, et al. Genomic mining of prokaryotic repressors for orthogonal logic gates. J Biotechnol, 2014, 10(2): 99-105.
|
| [92] |
Fiet SVS, Van Beilen JB, Witholt B. Selection of biocatalysts for chemical synthesis. Proc Natl Acad Sci USA, 2006, 103(6): 1693-1698. DOI:10.1073/pnas.0504733102
|
| [93] |
Li H, Chen W, Jin RN, et al. Biosensor-aided high-throughput screening of hyper-producing cells for malonyl-CoA-derived products. Microb Cell Fact, 2017, 16: 187. DOI:10.1186/s12934-017-0794-6
|
| [94] |
Gayathri SB, Kamaraj P, Arthanareeswari M, et al. DNA nanostructures based biosensor for the determination of aromatic compounds. Microb Cell Fact, 2015, 72: 191-196.
|
| [95] |
Leavitt JM, Wagner JM, Tu CC, et al. Biosensor-enabled directed evolution to improve muconic acid production in Saccharomyces cerevisiae. Biotechnol J, 2017, 12(10): 1600687. DOI:10.1002/biot.201600687
|
| [96] |
Ray S, Senapati T, Sahu S, et al. Design of ultrasensitive protein biosensor strips for selective detection of aromatic contaminants in environmental wastewater. Anal Chem, 2018, 90(15): 8960-8968. DOI:10.1021/acs.analchem.8b01130
|
| [97] |
Morgan SA, Nadler DC, Yokoo R, et al. Biofuel metabolic engineering with biosensors. Curr Opin Chem Biol, 2016, 35: 150-158. DOI:10.1016/j.cbpa.2016.09.020
|
| [98] |
Liu C, Zhang B, Liu YM, et al. New intracellular shikimic acid biosensor for monitoring shikimate synthesis in Corynebacterium glutamicum. ACS Synth Biol, 2018, 7(2): 591-601. DOI:10.1021/acssynbio.7b00339
|
| [99] |
Liu YF, Zhuang YY, Ding DQ, et al. Biosensor-based evolution and elucidation of a biosynthetic pathway in Escherichia coli. ACS Synth Biol, 2017, 6(5): 837-848. DOI:10.1021/acssynbio.6b00328
|
| [100] |
Mnich E, Vanholme R, Oyarce P, et al. Degradation of lignin β-aryl ether units in Arabidopsis thaliana expressing LigD, LigF and LigG from Sphingomonas paucimobilis SYK-6. Plant Biotechnol J, 2016, 15(5): 581-593.
|
| [101] |
Vanholme R, Demedts BB, Morreel K, et al. Lignin biosynthesis and structure. Plant Physiol, 2010, 153(3): 895-905. DOI:10.1104/pp.110.155119
|
| [102] |
Varanasi P, Singh P, Auer M, et al. Survey of renewable chemicals produced from lignocellulosic biomass during ionic liquid pretreatment. Biotechnol Biofuels, 2013, 6: 14. DOI:10.1186/1754-6834-6-14
|
| [103] |
Ni J, Gao YY, Tao F, et al. Temperature-directed biocatalysis for the sustainable production of aromatic aldehydes or alcohols. Angew Chem Int Ed, 2018, 57(5): 1214-1217. DOI:10.1002/anie.201710793
|
| [104] |
Lee MD, Ahlgren NA, Kling JD, et al. Marine Synechococcus isolates representing globally abundant genomic lineages demonstrate a unique evolutionary path of genome reduction without a decrease in GC content. Environ Microbiol, 2019, 21(5): 1677-1686. DOI:10.1111/1462-2920.14552
|
| [105] |
Ni J, Tao F, Wang Y, et al. A photoautotrophic platform for the sustainable production of valuable plant natural products from CO2. Green Chem, 2016, 18(12): 3537-3548. DOI:10.1039/C6GC00317F
|
| [106] |
Ni J, Liu HY, Tao F, et al. Remodeling of the photosynthetic chain promotes direct CO2 conversion into valuable aromatic compounds. Angew Chem Int Ed, 2018, 57(49): 15990-15994. DOI:10.1002/anie.201808402
|
| [107] |
Brey LF, Wlodarczyk AJ, Thøfner JFB, et al. Metabolic engineering of Synechocystis sp. PCC 6803 for the production of aromatic amino acids and derived phenylpropanoids. Metab Eng, 2019, 57: 129-139.
|
 2021, Vol. 37
2021, Vol. 37




