北京中医药大学中药现代研究中心,北京 100029
收稿日期:2020-12-28;接收日期:2021-03-18;网络出版时间:2021-04-02
基金项目:国家自然科学基金(No. 81773842) 资助
作者简介:史社坡 北京中医药大学研究员、博士生导师。2006年7月于北京大学获博士学位;2006年10月–2011年1月,先后在香港科技大学、东京大学、美国华盛顿州立大学等地从事天然产物生物合成与调控相关研究,目前研究方向为中药活性成分发现及合成生物学。主持国家重点研发计划(子课题)、国家自然科学基金、北京市自然科学基金、教育部新世纪优秀人才项目等课题多项;作为骨干人员获国家科技进步二等奖(排名6)、教育部科技进步二等奖(排名3)、李时珍医药创新奖(排名2) 等奖励.
Modern Research Center for Traditional Chinese Medicine, Beijing University of Chinese Medicine, Beijing 100029, China
Received: December 28, 2020; Accepted: March 18, 2021; Published: April 2, 2021
Supported by: National Natural Science Foundation of China (No. 81773842)
姜黄素是姜黄、郁金、生姜等姜科植物的特征成分,具有多种药理活性,如抗炎、抗HIV、抗肿瘤、清除自由基等[1-3]。此外,姜黄素还可以作为食用色素应用于食品工业[4]。大量文献报道姜黄素在预防和治疗疾病方面有巨大潜力[5-8],但目前姜黄素主要依靠从植物中提取分离或者有机合成的方法进行制备。由于植物生长周期长,单纯依靠植物进行提取制备,土地成本及分离制备成本高,自然资源消耗大;有机合成制备姜黄素的方法也存在试剂环境不友好等问题。
近几年来,合成生物学作为新兴学科,在天然产物的绿色合成与制造方面引起了高度关注[9-10],一系列活性天然产物,如青蒿素[11]、紫杉醇[12]、吗啡[13]、人参皂苷[14]、红景天苷[15]、秋水仙碱[16]、灯盏花乙素[17]等的合成生物学研究都取得了突破性的进展。我国当前也把合成生物技术列入战略性、前瞻性重点研究方向。在植物体内,姜黄素是由苯丙素途径合成,酪氨酸解氨酶(Tyrosine aminonialase,TAL)、4-香豆酰辅酶A连接酶(4-coumarate coenzyme A ligase,4CL)、β-酮酰辅酶A合酶(Diketide-CoA synthase,DCS) 和姜黄素合酶(Curcumin synthase,CURS) 参与姜黄素的生物合成,其中DCS和CURS是合成姜黄素的关键限速酶[18]。Rodrigues等将拟南芥中的4CL、姜黄中的DCS和CURS1引入大肠杆菌,成功以阿魏酸为底物合成姜黄素(70 mg/L)[19],经过对一系列发酵培养条件如诱导剂浓度、诱导时间、培养基组成等多个因素的考察优化,姜黄素的产率达到353 mg/L[20]。同时,该课题组研究人员还尝试将来自黏红酵母Rhodotorula glutinis的TAL、西班牙糖丝菌Saccharothrix espanaensis的4-香豆酸3-羟化酶(C3H)、紫花苜蓿Medicago sativa的咖啡酰辅酶A-3-O-甲基转移酶(CCoAOMT) 引入大肠杆菌,以酪氨酸为底物合成姜黄素,但产率仅为0.2 mg/L[19]。与姜黄中的DCS和CURS分步催化合成姜黄素相比,Katsuyama等从水稻Oryza sativa中克隆鉴定的姜黄素合酶(CUS) 能够直接催化阿魏酰辅酶A生成姜黄素及其类似物[21],进一步将深红酵母Rhodotorula rubra中的PAL、紫草Lithospermum erythrorhizon中的4CL与CUS一起引入大肠杆菌,可从酪氨酸或阿魏酸合成姜黄素及其类似物,其中以阿魏酸为底物时,姜黄素产率为100 mg/L左右[22]。此外,也有将CUS与其他来源的TAL、4CL、C3H、CoMT一起引入大肠杆菌[23],或者尝试将CUS在米曲霉Aspergillus oryzae中表达等方法合成姜黄素,但姜黄素产率均较低[24]。
前期研究中,我们成功从鲜姜中克隆鉴定了一个新的姜黄素合酶(ZoCURS),ZoCURS能够接受阿魏酰辅酶A为底物,与从姜黄中克隆的DCS一起两步催化合成姜黄素。进一步构建非天然融合蛋白DCS: : ZoCURS发现,新构建的融合蛋白不仅能够行使DCS和ZoCURS两步合成姜黄素的功能,而且DCS: : ZoCURS在体外酶反应中能够明显提高姜黄素的产率[25]。因此,我们尝试将来自拟南芥中编码4-香豆酰辅酶A连接酶(4CL) 和乙酰辅酶A羧化酶(ACC) 的基因与非天然融合基因DCS: : ZoCURS引入大肠杆菌,构建的工程菌成功以阿魏酸为底物合成姜黄素,结合在培养液中加入大孔吸附树脂等简单方法,使姜黄素在所构建的工程大肠杆菌(1 L摇瓶) 中的合成产量达到386.8 mg/L。
1 材料与方法
1.1 实验材料 感受态细胞Trans1-T1购自北京全式金生物技术有限公司;载体pRSF-duet-1、pCDF-duet-1购自Novagen公司。LA Taq DNA聚合酶、T4 DNA连接酶、dNTP mix、BamHⅠ、Hind Ⅲ、EcoRⅠ及其他限制性内切酶均购自TaKaRa公司。质粒提取试剂盒和PCR纯化试剂盒购自Axygen公司。IPTG、氨苄青霉素、卡那霉素、氯霉素购自Sigma公司。PCR引物由生工生物工程(上海) 股份有限公司合成,其他相关载体及试剂均由实验室自备。
LB培养基:10 g胰蛋白胨,5 g酵母提取物,10 g NaCl,加蒸馏水至1 L,102.9 kPa高压蒸汽灭菌15 min。
TB培养基:12 g胰蛋白胨,24 g酵母提取物,4 mL甘油,17 mmol KH2PO4,72 mmol K2HPO4,pH 7.0,加蒸馏水定容至1 L,102.9 kPa高压蒸汽灭菌15 min。
M9培养基:Na2PO4·7H2O 12.8 g,KH2PO4 3.0 g,NaCl 0.5 g,NH4Cl 1.0 g,加蒸馏水200 mL溶解,102.9 kPa高压蒸汽灭菌15 min。再加入1 mol/L的MgSO4 2 mL、20%的葡萄糖溶液20 mL、1 mol/L的CaCl2 0.1 mL,加灭菌双蒸水至1 000 mL。
改良M9培养基:M9培养基中加入FeCl3 54 mg/L、ZnCl2 4 mg/L、CoCl2 4 mg/L、Na2MoO4 4 mg/L、CuCl2 2 mg/L、H2BO3 1 mg/L、VB1 340 mg/L、VB5 10.8 mg/L、VB6 2.8 mg/L、VB2 0.84 mg/L、烟酸12.2 mg/L、生物素0.12 mg/L。
1.2 实验方法
1.2.1 基因克隆、质粒及工程菌构建 根据NCBI中拟南芥的4-香豆酰辅酶A连接酶(4CL,GenBank登录号:NM_104046.3)、乙酰辅酶A羧化酶(ACC,GenBank登录号:AY142630.1) 设计特异性异物(引物序列见表 1),从拟南芥的新鲜叶片中克隆相关基因全长序列。扩增获得的DNA全长片段用相应的限制性内切酶酶切后,用T4 DNA连接酶分别插入载体pET-28a、pRSF-duet-1、pCDF-duet-1和pET-22b中。将获得的重组质粒转化到大肠杆菌BL21(DE3)中,菌落PCR并测序验证。非天然融合蛋白DCS: : ZoCURS的基因序列及质粒构建方法见前期报道[25]。将不同的质粒组合,转入大肠杆菌BL21(DE3) 中,于对应抗性平板上培养,利用各基因特异性引物进行菌落PCR,筛选出含有所有目标基因的阳性克隆,测序验证后获得工程菌菌株如下:① pET-28a-4CL+ pET-28a-DCS: : ZoCURS;② pET-28a-4CL+pET-28a-
DCS: : ZoCURS+pET-28a-ACC;③ pRSF-duet-1- ACC+pCDF-duet-1-4CL+pET-22b-DCS: : ZoCURS;④ pRSF-duet-1-ACC-4CL+ pET-22b-DCS: : ZoCURS。
表 1 用于PCR扩增的引物
Table 1 Primers used for PCR amplification
| Gene |
Primer
sequence (5′–3′) |
| AtACC-1F |
CCGGAATTCATGGACGCTTCTATGATTAC |
(EcoR Ⅰ) |
| AtACC-1B |
CCGCTCGAGCTAAACCGTTGCGTTTGTC |
(Xho Ⅰ) |
| AtACC-2F |
CGGAATTCGATGGACGCTTCTATGATTACCAATTC |
(EcoR Ⅰ) |
| AtACC-2B |
TTGCGGCCGCCTAAACCGTTGCGTTTGTCAGATC |
(Not Ⅰ) |
| At4CL-1F |
GGGGTACCATGGCGCCACAAGAACAAGCAGTTTC |
(Kpn Ⅰ) |
| At4CL-1B |
CCCTCGAGTCAGTGAAGAACACTTTGTTGATTC |
(Xho Ⅰ) |
| The underlined sequences are the restriction sites. |
1.2.2 培养方法及产物分析定量 将含有不同质粒的重组菌株按1︰50 (V/V) 的比例接种到具有相应抗性的LB培养基中培养过夜,活化后的菌液按照1︰100 (V/V) 比例接种到含有50 μg/mL卡那霉素的LB培养基(第一培养基/生长培养基) 中,在37 ℃下培养至菌液OD600值为0.4-0.6时加入终浓度为0.8 mmol/L的IPTG,调节温度至20 ℃继续振荡培养5 h;4 ℃、4 000 r/min离心,收集菌体。将收集的菌体接种到LB培养基(第二培养基/发酵培养基) 中(0.5 g菌体/20 mL培养基),并加入CaCO3 (40 g/L)、山梨醇(5 g/L)、阿魏酸(3 mmol/L)。继续振荡培养48 h,上清液和沉淀分别经乙酸乙酯和甲醇提取,合并提取液,减压蒸干溶剂,甲醇复溶,利用岛津LCMS-IT-TOF液质联用仪(Shimadzu,Japan) 分析姜黄素的生成及含量。液相检测条件:色谱柱Agilent Eclipse XDB C18色谱柱(250 mm×4.6 mm,I.D. 5 μm),流速:1.0 mL/min,流动相:乙腈-水(含0.1%甲酸)。梯度洗脱:-5 min,5%乙腈;5-40 min,5%-40%乙腈;40-45 min,40%-80%乙腈。为计算姜黄素的含量,利用液相色谱法建立姜黄素标准品的标准曲线,以峰面积(y) 和化合物浓度(x) 进行线性回归(y=0.020 1x+3 679.3,R2=0.999 5),根据标准曲线计算姜黄素的含量。姜黄素的制备与纯化:50 mL发酵培养后的培养液,超声破碎,乙酸乙酯提取3次,合并提取液浓缩蒸干,经柱色谱纯化后,利用HRESIMS、1H NMR数据进行鉴定。
姜黄素:1H NMR (DMSO-d6,500 MHz):3.82 (6H,s),6.03 (2H,s),6.72 (2H,d,J =16.0 Hz),6.80 (2H,d,J = 8.0 Hz),7.12 (2H,brd,J=8.0 Hz),7.30 (2H,brs),7.51 (2H,d,J=16.0 Hz);HRESIMS m/z 369.135 7 [M+H]+ (calcd for C21H21O6, 369.133 8)。
1.2.3 培养条件考查 分别考查诱导培养阶段诱导剂加入时菌体OD600值(培养至OD600值分别为0.4、0.5和0.6)、诱导剂IPTG浓度(0.4 mmol/L、0.6 mmol/L和1 mmol/L)、诱导表达时间(4 h、5 h、6 h和8 h),发酵培养阶段发酵培养基组成(LB、TB、M9、改良M9),上述考查实验平行开展,每个因素实验重复3次,且仅保持被考查因素为变量,其他因素固定,其余操作同1.2.2进行。
取上述综合最佳参数组合,对发酵培养阶段的相关因素包括接种菌体量(每20 mL培养0.5 g、1.0 g、2.0 g和2.5 g菌体)、发酵培养时间(8 h、12 h、24 h、36 h、48 h、60 h) 进行平行考查,即保持被考查因素为变量,其他因素固定,其余实验操作同1.2.2。
2 结果与分析
2.1 姜黄素关键合成步骤在大肠杆菌中的构建 日本学者研究发现姜黄素是由β-酮酰辅酶A合酶(Diketide-CoA synthase,DCS) 和姜黄素合酶(Curcumin synthase,CURS) 两个植物聚酮合酶共同作用的结果。首先,DCS催化阿魏酰辅酶A与一分子丙二酰辅酶A缩合形成β-酮酰辅酶A之后,CURS再催化β-酮酰辅酶A与另一分子阿魏酰辅酶A缩合生成姜黄素[18]。然而,由DCS和CURS体外催化合成姜黄素,需要以价格昂贵、合成困难的酰基辅酶A硫酯为底物,无法用于姜黄素的大量合成与生产。在植物体内,4-香豆酰辅酶A连接酶(4CL) 可以将阿魏酸转化为阿魏酰辅酶A,乙酰辅酶A羧化酶(ACC) 可以将代谢过程中的乙酰辅酶A转化为丙二酰辅酶A。因此,将编码4CL、ACC、DCS、CURS四个关键酶的基因引入大肠杆菌(图 1),再加上大肠杆菌自身代谢过程中产生的部分丙二酰辅酶A,可以实现在大肠杆菌中以阿魏酸为底物合成姜黄素。课题组在前期研究中发现非天然融合蛋白DCS: : ZoCURS能够催化阿魏酸辅酶A和丙二酰辅酶A缩合生成姜黄素,与DCS、CURS分步催化合成姜黄素相比,姜黄素的产率提高1倍。因此,本研究中尝试将前期构建的非天然融合蛋白DCS: : ZoCURS引入大肠杆菌,而4-香豆酰辅酶A连接酶基因(4CL) 则克隆自拟南芥。为了提高大肠杆菌体内丙二酰辅酶A的合成能力,我们还在大肠杆菌中引入从拟南芥中克隆的能够催化乙酰辅酶A转化为丙二酰辅酶A的乙酰辅酶A羧化酶基因ACC。
将上述3个基因连接到表达载体pET-28a中,并构建不同质粒组合的工程菌株①和菌株②,考察姜黄素的生成量。结果表明,携带ACC基因的工程菌株②合成姜黄素的量为(15.74±2.27) mg/L,约为未携带ACC基因工程菌株① ((3.07±1.14) mg/L) 的3倍。因此,我们以菌株②对相关参数逐个进行考察。
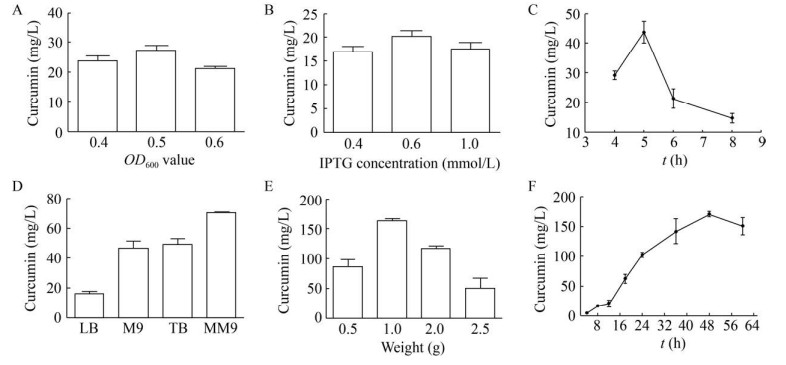
2.2 培养条件考察 首先利用菌株②考察在生长培养阶段,诱导剂加入时间、浓度及诱导表达时间长短对姜黄素合成的影响。结果显示(图 2A–C),在生长培养阶段,当OD600值在0.5时加入诱导剂0.6 mmol/L继续诱导表达5 h的条件下,有利于姜黄素的合成。
接下来对第二培养基(发酵培养基) 的组成进行考察,结果表明菌株②在生长培养基相同(均为LB培养基) 的情况下,第二培养基组成不同,对姜黄素的合成影响非常明显(图 2D),其中在M9改良培养基中合成姜黄素最高,达到(70.09±1.02) mg/L。相比之下,虽然改良后的M9培养基仅仅是在M9培养基的基础上加入了Fe2+、Zn2+、Cu2+等金属离子以及VB5、VB6、核黄素等,但在发酵培养阶段,菌体的生长状态及成活时间明显改善,因此有充裕时间促使姜黄素合成,这可能是姜黄素产率明显提高的原因。
选择上述优化后的最佳诱导培养条件,利用改良的M9培养基作为发酵培养基,以菌株②进一步对其发酵培养阶段接种菌体量和发酵培养时间对姜黄素合成的影响进行考察,结果(图 2E–F)显示加入菌体量为1 g/20 mL,发酵培养48 h有利于获得较高产率的姜黄素,姜黄素的产率均在150 mg/L以上。
非常意外的是,当我们将构建的含有pRSF-duet-1-ACC、pCDF-duet-1-4CL、pET-22b- DCS: : ZoCURS质粒的菌株③在上述优化后的条件下进行姜黄素的合成时,姜黄素的含量明显升高,同样条件下,培养液的黄色和菌株②相比显示肉眼可见的加深,考虑到姜黄素的累积浓度过高,可能会影响菌体细胞的存活,我们在发酵培养8 h后,向培养液中加入了经灭菌的AB-8大孔吸附树脂(每100 mL加入树脂2 g),培养液中的姜黄素被大孔吸附树脂吸附而迅速变浅,进一步振荡培养72 h,发现大量菌体死亡,此时按1.2.2中的方法测定姜黄素的产率达到386.8 mg/L (图 3),高出文献报道的最高值353 mg/L (同一菌株,作者在未进行系列条件优化前,姜黄素的产率仅为70 mg/L)。因此,我们进一步构建了含有ACC和4CL基因融合表达的载体pRSF-duet-1-ACC-4CL,并将其与pET-22b-DCS: : ZoCURS一起引入大肠杆菌构建了菌株④,在上述同样的条件下进行发酵培养,希望进一步提高姜黄素的产率,但遗憾的是,菌株④合成姜黄素的产率没有升高,反而降低。这可能是由于构建融合载体降低了ACC和4CL基因的表达。考虑到所用菌株均为大肠杆菌BL21(DE3),并且本研究的主要目的是想考察将非天然融合基因DCS: : ZoCURS引入大肠杆菌后合成姜黄素的可行性,我们没有再继续对菌株③的发酵培养条件作深入系统的考察与优化。
在前期研究中[25],我们发现非天然融合蛋白DCS: : ZoCURS不仅能够接受以阿魏酰辅酶A为底物生成姜黄素,同时还可以接受以4-羟基苯丙酰辅酶A、肉桂酰辅酶A、4-香豆酰辅酶A为底物,生成姜黄素的类似物,而4CL也可以催化合成上述阿魏酰辅酶A类似物的合成。因此,我们尝试以4-羟基苯丙酸、4-香豆酸为底物,同样可以检测到相应的姜黄素类似物的合成,但由于缺乏相关的对照品,其产率没有进行进一步的准确测定。
3 结论 通过构建含有乙酰辅酶A羧化酶基因ACC、4-香豆酰辅酶A连接酶基因4CL、非天然融合基因DCS: : ZoCURS的大肠杆菌,成功实现从阿魏酸合成姜黄素,最高产率达到386.8 mg/L。证明非天然融基因DCS: : ZoCURS可以用于构建合成姜黄素及其类似物的相关工程菌,为后续通过进一步复杂的代谢网络优化、构建姜黄素合成能力更强的工程菌提供参考。
 2021, Vol. 37
2021, Vol. 37