
中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘娟娟, 张妍, 赫卫清
- Liu Juanjuan, Zhang Yan, He Weiqing
- 利用CRISPR-Cas9系统与核糖体工程获得新型可利霉素产生菌
- Construction of a novel carrimycin-producing strain by using CRISPR-Cas9 and ribosome engineering techniques
- 生物工程学报, 2021, 37(6): 2116-2126
- Chinese Journal of Biotechnology, 2021, 37(6): 2116-2126
- 10.13345/j.cjb.200763
-
文章历史
- Received: November 28, 2020
- Accepted: February 9, 2021
- Published: February 23, 2021
2. 沈阳同联集团有限公司,辽宁 沈阳 110042
2. Shenyang Tonglian Group Co., Ltd., Shenyang 110042, Liaoning, China
可利霉素(Carrimycin,CAM),原名为必特螺旋霉素(Bitespiramycin),是将耐热链霉菌Streptomyces thermotolerans中的4″-O-异戊酰基转移酶基因(4″-O-isovaleryltransferase gene,ist)整合至螺旋链霉菌Streptomyces spiramyceticus的基因组中所获得的基因工程菌株的发酵产物[1]。CAM的主组分是异戊酰螺旋霉素(Isovalerylspiramycin,ISP)Ⅰ、Ⅱ和Ⅲ,它对革兰氏阳性菌有较强的活性,尤其是对肺炎链球菌、肺炎支原体和衣原体的活性较强[2]。由于CAM较螺旋霉素(Spiramycin,SP) 在结构上增加了4″-O-异戊酰基侧链,使其亲脂性与体内的抗菌活性增强,且半衰期延长[3-5]。Ⅲ期临床试验证实,CAM的药效和安全性都优于阿奇霉素。2019年,CAM作为国家1类创新药获得国家药品监督管理局批准(国药证字:H20190009),并于同年上市。
原有的CAM工程菌由于经过多轮遗传改造和诱变育种,已具有安普霉素和硫链丝菌素两种抗性基因,难以通过抗性基因再对其进行遗传操作。CRISPR-Cas9系统可以高效地对基因组特定位点进行靶向编辑,包括敲除、修复和替换等[6],而且能够不利用抗性基因同时敲除多个基因。近年来,CRISPR-Cas9系统已经广泛应用于链霉菌基因组遗传改造中[7],包括基因定点突变[8]、验证基因功能[9]、激活沉默次级代谢产物生物合成基因簇[10]等。利用该系统,我们成功将S. spiramyceticus中负责SP 3位的酰化基因bsm4替换为ermEp*启动子控制下的ist基因,获得了只产ISPI且不带有抗性基因的新型工程菌[11]。
微生物次级代谢产物的生物合成与核糖体密切相关,通过核糖体工程技术对核糖体、RNA聚合酶及转录因子进行修饰改造可以提高次级代谢产物的合成能力[12-13]。该方法常用靶向核糖体或RNA聚合酶的抗生素来筛选抗性突变株[14],一般采用的抗生素包括链霉素(Streptomycin,STR)、利福平(Rifampicin,RFP)、庆大霉素(Gentamicin,GEN)、巴龙霉素(Paromomycin,PRM) 和夫西地酸(Fusidic acid) 等。因其获得突变株的过程简单、获得高产菌株效率高、应用时无需了解菌株的遗传背景等优点,该技术已被广泛应用于菌种改良等方面[15]。
为了满足对CAM工程菌进一步遗传改造的需要,本研究采用CRISPR-Cas9系统将ist-acyB2连锁基因克隆至SP生物合成基因簇的下游附近,构建不带任何抗性标记的新型CAM工程菌。并且通过核糖体工程技术对构建成功的CAM工程菌株进行菌种诱变,筛选获得新型CAM高产菌株。
1 材料与方法 1.1 材料 1.1.1 菌种和质粒螺旋霉素产生菌S. spiramyceticus 1941由本实验室保藏。耐热链霉菌S. thermotolerans ATCC 11416T,用于提取基因组DNA扩增ist基因和正调控基因acyB2,购于美国模式培养物集存库(American type culture collection,ATCC)。新型无抗性CAM产生菌54IA-1和54IA-2,由本研究室构建。大肠杆菌Escherichia coli DH5α感受态细胞,用于基因克隆和重组质粒构建,购自北京全式金生物技术有限公司。E. coli ET 12567/pUZ8002甲基化缺陷型大肠杆菌,用于重组质粒去甲基化;枯草芽胞杆菌Bacillus subtilis CPCC 100029,用于工程菌发酵产物的活性检测,均为本实验室保存。
CRISPR-Cas9基因组编辑质粒pKCcas9do[8],由中国科学院上海生命科学研究院姜卫红研究员惠赠。重组质粒pKCas-ia-1和pKCas-ia-2由本研究室构建。
本研究所用引物序列见表 1。
| Oligos | Sequences (5′–3′) |
| orf54-sgRNA-F1 | GGACTAGTCTTGCGCACCGCTGCCCCCAGTTTTAGAGCTAGAAAT |
| orf54-sgRNA-F2 | GGACTAGTGCGCGGTGCCCGCACCACCAGTTTTAGAGCTAGAAAT |
| orf54-Down | GCTCTAGACTCAAAAAAAGCACCGACTCGG |
| orf54-LF | GCTCTAGAGCCACTGGCGAGGATGTTCA |
| orf54-LR | CGGAATTCTGCGGCGGTATCGGGAAGA |
| ist-acyB2-F | CGGAATTCCACCCAGCTCGAATACGCC |
| ist-acyB2-R | CCGGATATCGACCTTGACAGTCCTCGCCTC |
| orf54-RF | CCGGATATCCCCTCGGAATCCTTCTCACAG |
| orf54-RR | CCCAAGCTTGTGCTGCTGCCCATTCTCATC |
| 54IA-YF | GGCCTCGGTGCTCGATAAG |
| 54IA-YR | GCATGGACACCATCACCCTCT |
| IA-F | GCGAACACCGCTGAGAACA |
| IA-R | AGATCGCCGCTCACCCTTA |
| 16S RNA-F | GGTAGAGCTTGTTGACGCAGAG |
| 16S RNA-R | ATGAGGGCGAGGACAGCGATGC |
| ist-F | GTCTCCATCCCCCTGGTCGCAC |
| ist-R | CTGGATGATCAGGTAGTGCACG |
| acyB2-F | GCCCAGCACCTCATGGAACA |
| acyB2-R | ACCCCGAACAGCAGGAGCGT |
| bsm3-F | AGACGTTGCCTGGAGTTGGG |
| bsm3-R | GCTGGGTGAACCGCTGATAG |
| bsm5-F | GACGGCCTGCTGCGTGAACT |
| bsm5-R | GGGCCACTTGTCGCTGATGT |
| bsm9-F | ATGTTCAGCTCCATAGGCCACC |
| bsm9-R | GCACAGAATGCGAGACCCG |
| bsm42-F | TCTCGTGGGTCTGCCCTTCA |
| bsm42-R | AAGTCGGCGTCCACCTGCT |
| rpoB-F | GAGCGCATGACCACCCAG |
| rpoB-R | TCGTAGTTGTGACCCTCCC |
氯霉素(Chloramphenicol,CM) 购自北京市医药公司制药厂。安普霉素(Apramycin,AM) 购自武汉远城科技发展有限公司。硫链丝菌素(Thiostrepton,TSR)、氨苄青霉素(Ampicillin,AMP) 和利福平(RFP) 购自Sigma公司。SP对照品,购自河南天方药业股份有限公司。CAM标准品,沈阳同联集团有限公司提供。溶菌酶购自生工生物工程(上海) 股份有限公司。链霉菌基因组快速提取试剂盒购自广州东盛生物科技有限公司。限制性内切酶、T4 DNA连接酶购自TaKaRa公司。KOD FX Neo DNA聚合酶(TOYOBO) 购自北京百灵克生物科技有限公司。
1.1.3 培养基R2YE培养基用于S. spiramyceticus 1941原生质体的制备和再生[16],S. spiramyceticus 1941的斜面、种子、发酵和生物检定培养基均按参考文献[17]配制。
1.2 实验方法 1.2.1 CRISPR-Cas9基因编辑质粒的构建链霉菌基因组DNA的提取按照文献[18]进行。E. coli ET12567/pUZ8002感受态细胞的制备,目的片段的扩增,PCR产物纯化,限制性酶切,DNA连接与转化,重组转化子的筛选与鉴定等分子克隆操作按照文献[19]进行。DNA测序由中美泰和生物技术(北京) 有限公司完成。
以提取的S. spiramyceticus 1941基因组DNA为模板,用引物orf54-LF/LR、orf54-RF/RR进行扩增获得ist-acyB2基因整合位点两端的左和右同源臂。大小为988 bp的左同源臂用Xba Ⅰ和EcoR Ⅰ进行酶切回收,大小为980 bp的右同源臂用EcoR Ⅴ和Hind Ⅲ酶切回收。以S. thermotolerans ATCC 11416T基因组DNA为模板,用ist-acyB2-F/R引物扩增出3 335 bp ist-acyB2基因片段,并用EcoR Ⅰ和EcoR Ⅴ酶切回收。分别用引物orf54-sgRNA-F1、orf54-sgRNA-F2与orf54-Down配对,以pKCcas9do质粒为模板进行PCR,获得2条150 bp左右的引导RNA (Single-guide RNA, sgRNA)的DNA序列sgDNA-1和sgDNA-2,用Spe Ⅰ和Xba Ⅰ酶切回收。再分别将sgDNA-1、sgDNA-2与左右同源臂和ist-acyB2基因片段克隆至pKCcas9do载体的Spe Ⅰ和Hind Ⅲ位点获得重组质粒pKCas-ia-1和pKCas-ia-2。
1.2.2 目的菌株的筛选将构建好的重组质粒pKCas-ia-1和pKCas-ia-2通过原生质体转化法[16]分别导入到S. spiramyceticus 1941中,利用1 μg/mL TSR诱导Cas9表达,由sgRNA引导Cas9对SP生物合成基因簇下游orf54基因的靶位点进行切割形成双链断裂,重组质粒上带有同源臂的ist-acyB2基因通过同源重组插入到靶位点。将发生同源双交换的目的菌株于37 ℃培养,使质粒停止复制,通过AM抗性和无抗性平板进行筛选,获得ist-acyB2基因插入到靶位点且不带有抗性基因的菌株,并进行PCR鉴定和测序验证。
1.2.3 菌株发酵及发酵产物的提取挑取阳性转化子接种于斜面培养基上28 ℃培养5–7 d,挖块面积为1 cm2左右的菌落,接种到装有50 mL发酵培养基的250 mL三角瓶,每个菌株接种3瓶,28 ℃振荡培养96–120 h后,发酵液经离心取上清液稀释,进行抗菌活性检测。
发酵产物的提取:发酵液室温8 000 r/min离心10 min,上清液用5 mol/L NaOH调pH至8.5–9.0之间,用1/2体积的乙酸乙酯萃取,吹干后用100 μL乙酸乙酯或甲醇溶解于1.5 mL离心管中。
1.2.4 抗生素生物效价测定以Bacillus subtilis CPCC 100029为检定菌,参考《中华人民共和国国药典》2020年版(二部)乙酰螺旋霉素含量测定项下微生物检定法[20]。采用管碟法,用标准曲线法进行测定。
1.2.5 qPCR分析用TSB培养基[16]培养54IA-1和54IA-2菌株,取培养72 h的菌丝分别提取总RNA,进行反转录PCR (Reverse transcription PCR,RT-PCR) 和qPCR分析。以16S rRNA作为内参基因(引物16S RNA-F/R,表 1),对每个样品中ist和acyB2及与SP生物合成相关基因表达进行定量分析,每个样品做3个重复。使用SYBR Green Ⅰ染料法进行定量分析,2–ΔΔCt方法计算不同菌株中的基因表达量。qPCR反应条件:96 ℃预变性3 min;96 ℃变性30 s,61 ℃退火30 s,72 ℃延伸1 min,共计35个循环;72 ℃延伸10 min (罗氏公司LightCycler 96)。
1.2.6 高效液相色谱(High performance liquid chromatography,HPLC) 分析发酵液经提取、挥干后溶于色谱纯乙腈,0.22 μm的滤膜过滤后进样10 μL。色谱仪:岛津LC-10ATvp液相色谱仪,色谱柱:Kromasil C18 (4.5 mm×150 mm,5 μm),流动相:CH3OH/1% CH3COONH4 (65︰35),检测波长:231 nm,流速:1 mL/min,柱温:25 ℃。以CAM标准品为对照,检测并计算ISPⅠ、Ⅱ和Ⅲ的峰面积,确定CAM主组分的相对含量。
1.2.7 抗药性突变株筛选取扩大培养的54IA-2菌丝体,放入带玻璃珠和无菌水的试管中,剧烈振荡5 min,制成孢子悬液。制备不同浓度(20、40、80 μg/mL) 的RFP筛选平板,将孢子悬液涂布在筛选平板上,并设置空白对照组,28 ℃培养3–4 d。再将生长起来的单菌落用同样浓度的抗生素传代培养,进行摇瓶复筛:挑选传代后的菌株接种于种子培养基,28 ℃培养48–52 h,以10%的接种量转入发酵培养基(每个500 mL三角瓶装100 mL发酵培养基),28 ℃培养96 h,提取发酵产物进行HPLC检测。
1.2.8 RFP抗性突变株的突变位点分析RFP抗性突变株是由于RNA聚合酶b亚基的基因(rpoB) 发生突变从而获得抗性。为研究54IA-2的RFP抗性突变株中rpoB基因的碱基变化部位,提取RFP突变株DNA,利用rpoB-F/R引物扩增出在RFP抗性压力下rpoB基因容易发生变化的区域,分析其中发生突变的碱基,找出发生突变的对应氨基酸残基。
2 结果与分析 2.1 ist-acyB2基因片段的插入位点分析CAM生物合成基因簇[21]中负责SP生物合成的部分为89 kb左右,包含在一个约110 kb的序列中(GenBank登录号:MH460451)。这个序列中包含了54个预测的开放阅读框(Open reading frame,ORF),经鉴定与SP生物合成相关的有42个,按顺序依次命名为bsm1至bsm42[22],它们与生二素链霉菌Streptomyces ambofaciens ATCC 23877中公布的SP生物合成基因簇[23]同源性较高。bsm42与S. ambofaciens中位于整个基因簇边界附近的SP正调控基因srm40[24]是同源基因;其下游的orf43-orf48是一些未知功能的基因和与ATP酶驱动运输相关的基因,推测是负责SP的运输;orf49-orf53绝大部分是功能未知基因;orf54基因编码一种包含WD40重复序列的蛋白[25],在真核细胞中WD40蛋白家族成员常作为生物大分子相互作用的支架蛋白,在原核细胞中这类蛋白常参与信号传导和营养物质的合成,因此这个基因应该与SP的生物合成无关。在原CAM产生菌中ist基因整合在一个Ⅱ型PKS基因中间,距离SP的生物合成基因簇3 Mb以上,空间上的距离会影响异戊酰基转移酶对SP的后修饰效率。若将ist基因整合在SP生物合成基因簇附近,可能提高SP转化为ISP的效率。因此将带有ist和其正调控基因acyB2的重组质粒pKCas-ia-1和pKCas-ia-2通过CRISPR-Cas9系统插入到SP生物合成基因簇的相邻基因orf54的下游(图 1A)。

|
| 图 1 在SP生物合成基因簇下游插入ist-acyB2基因变株的构建(A) 和验证(B) Fig. 1 Construction (A) and validation (B) of a mutant with ist-acyB2 genes inserted downstream of the SP biosynthetic gene cluster. M: Trans 15k DNA marker; 1: pKCas-ia-1 (SpeⅠ/Hind Ⅲ); 2: pKCas-ia-2 (SpeⅠ/Hind Ⅲ); 3–5: IA-F and IA-R primer, 3: wildtype strain, 4: 54IA-1, 5: 54IA-2; 6–8: 54IA-YF and 54IA-YR primers, 6: wildtype strain, 7: 54IA-1, 8: 54IA-2. |
| |
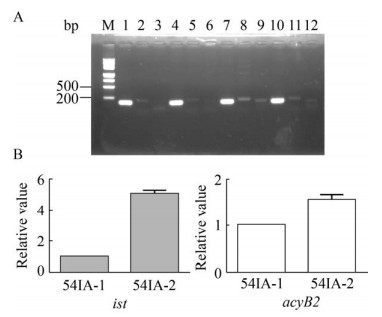
利用Spe Ⅰ和Hind Ⅲ对构建成功的质粒pKCas-ia-1和pKCas-ia-2进行酶切鉴定,结果如图 1B中泳道1和2所示,13 kb左右的条带为pKCcas9dO载体,5 kb左右的条带为带有同源臂的ist-acyB2基因片段,片段大小与预期相符,并经测序确证。将两个质粒导入S. spiramyceticus 1941中,通过TSR诱导、抗性和温度筛选出两种目的菌株54IA-1和54IA-2。利用鉴定引物IA-F/R和54IA-YF/R对原株S. spiramyceticus 1941和ist-acyB2基因片段插入后的54IA-1、54IA-2变株进行鉴定(图 1B)。泳道3–5显示IA-F/R鉴定引物在原株中没有扩增出产物,在54IA-1、54IA-2变株中扩增出约预期大小的1.8 kb目的片段;泳道6–8显示54IA-YF/R鉴定引物在原株中扩增出约2.7 kb的特异性片段,在两种变株中扩增出6 kb的特异性片段,二者之差为3.3 kb左右,符合ist-acyB2片段的大小,证明ist-acyB2基因片段已成功插入到orf54基因的下游。同时,将54IA-YF/R鉴定引物PCR扩增产物进行测序,证实两种变株中ist-acyB2片段在orf54基因下游的插入位点完全一致。
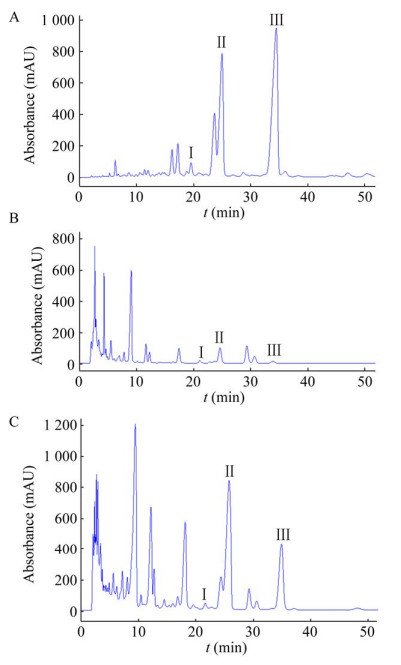
2.3 新型CAM工程菌株的发酵检测将54IA-1和54IA-2菌株进行发酵,对发酵产物进行鉴定。虽然设计了两种不同的Cas9切割位点,但是通过测序证实,两种工程菌株插入目的基因的位置与序列完全相同。但对两种变株在发酵培养基中的发酵上清进行抗菌活性测定发现,54IA-1上清液形成的抑菌圈明显小于54IA-2的抑菌圈。54IA-1的效价为(57±5.1) μg/mL,而54IA-2的效价达到(179±8.2) μg/mL,明显高于54IA-1的效价。通过HPLC检测(图 2)也发现54IA-2菌株中ISP的产量明显高于54IA-1。
|
| 图 2 菌株54IA-1和54IA-2发酵产物的HPLC检测 Fig. 2 HPLC analysis of the fermentation products of strains 54IA-1 and 54IA-2. (A) Control of CAM. (B) 54IA-1. (C) 54IA-2. Ⅰ: ISPⅠ; Ⅱ: ISPⅡ; Ⅲ: ISPⅢ. |
| |
通过qPCR检测54IA-1和54IA-2中ist和acyB2以及与SP生物合成相关基因的表达情况,结果显示,54IA-2中ist和acyB2的表达量分别比在54IA-1中高5倍和1.5倍左右(图 3),且bsm3、bsm5、bsm9和正调控基因bsm42的表达量(表 2)也明显高于54IA-1。其中bsm3编码23S rRNA的甲基转移酶,是一种修饰23S rRNA而获得抗生素抗性的基因,抗性基因的高表达通常能提高抗生素的产量[11, 26];bsm5编码O-甲基转移酶,参与聚酮合酶底物的合成;bsm9编码SAM依赖的甲基转移酶,参与NDP-脱氧糖基的生物合成;bsm42是SP生物合成的正调控基因[22-23]。菌株54IA-2中ist、acyB2以及部分与SP生物合成相关基因的表达情况与其CAM产量高于54IA-1相符,表明工程菌株54IA-1的ISP产量低于54IA-2可能是由于相关基因表达受影响引起的。
|
| 图 3 菌株54IA-1和54IA-2中ist和acyB2基因表达情况 Fig. 3 The expression level of ist and acyB2 in 54IA-1 and 54IA-2 strains. (A) RT-PCR detection of ist and acyB2 in 54IA-1 and 54IA-2 strains. 1–6: 54IA-1, 1 & 4:16S rRNA, 2 & 5: ist, 3 & 6: acyB2; 7–12: 54IA-2, 7 & 10:16S rRNA, 8 & 11: ist, 9 & 12: acyB2. (B) qPCR detection of ist and acyB2 in 54IA-1 and 54IA-2 strains. |
| |
| Gene | Proposed function | Proposed role in SP biosynthesis | Fold change (54IA-2/54IA-1) |
| bsm3 | 23S ribosomal RNA methyltransferase | Resistance | 4.99 |
| bsm5 | O-methyltransferase | Provision of extender units for PKS | 12.47 |
| bsm9 | SAM-dependent methyltransferase | Sugar biosynthesis | 8.69 |
| bsm42 | DDE_5 superfamily, transcriptional regulator | Positive regulation | 73.52 |
由于新构建菌株发酵水平远远达不到工业需求,所以通过核糖体工程方法来提高菌株的CAM产量。对新构建的菌株54IA-2进行核糖体工程常用抗生素的敏感性测试发现,该菌株对RFP比较敏感,其MIC为0.2 μg/mL,因此选择RFP作为抗性压力筛选54IA-2突变株。对一系列RFPR菌株进行发酵,并对发酵产物进行检测,发现在40 μg/mL RFP平板上生长的抗性突变株RFP40-6的ISP产量最高,为290.4 μg/mL。将突变菌株RFP40-6进行自然分离,选取15个由单个孢子长成的菌落进行摇瓶复筛。HPLC检测结果显示,ISP产量最高的为RFP40-6-8,可达842.9 μg/mL,较原始菌株提高了约6倍(表 3)。将产量变化较大的7株菌进行rpoB基因测序,结果发现所有菌株都存在576位的丝氨酸(Serine,Ser) 突变为丙氨酸(Alanine,Ala),突变情况见表 3。RFP40-6-1、RFP40-6-5均在基因序列的1 319位发生突变,碱基鸟嘌呤G突变为腺嘌呤A,使得440位精氨酸(Arginine,Arg) 变为组氨酸(Histidine,His)。RFP40-6-2、RFP40-6-3和RFP40-6-7均在基因序列的1 281位发生突变,碱基A突变为G,427位天门冬氨酸(Aspartic acid,Asp) 变为谷氨酸(Glutamic acid,Glu)。突变株RFP40-6-12中基因序列的1 310位A碱基突变为胞嘧啶C,使得437位His突变为脯氨酸(Proline,Pro)。CAM产量提高最为明显的RFP40-6-8中基因序列的1 271位A碱基突变为胸腺嘧啶T,对应的424位谷氨酰胺(Glutamine,Gln) 突变为亮氨酸(Leucine,Leu)。
| Strains | Position of mutations in rpoB | Amino acid exchange | Production of ISP (μg/mL) | The proportion of ISP to total (%) |
| 54IA-2 | – | – | 120.1 | 12.3 |
| RFP40-6 | – | – | 290.4 | 28.3 |
| RFP40-6-1 | 1 319 (G→A)/1 726 (T→G) | Arg440His/Ser576Ala | 329.5 | 12.9 |
| RFP40-6-2 | 1 281 (A→G)/1 726 (T→G) | Asp427Glu/Ser576Ala | 482.5 | 18.7 |
| RFP40-6-3 | 1 281 (A→G)/1 726 (T→G) | Asp427Glu/Ser576Ala | 329.5 | 12.9 |
| RFP40-6-5 | 1 319 (G→A)/1 726 (T→G) | Arg440His/Ser576Ala | 457.2 | 17.9 |
| RFP40-6-7 | 1 281 (A→G)/1 726 (T→G) | Asp427Glu/Ser576Ala | 365.3 | 14.3 |
| RFP40-6-8 | 1 271 (A→T)/1 726 (T→G) | Gln424Leu/Ser576Ala | 842.9 | 33.0 |
| RFP40-6-12 | 1 310 (A→C)/1 726 (T→G) | His437Pro/Ser576Ala | 472.5 | 18.5 |
CAM是利用基因工程方法获得的工程菌发酵产物,具有良好的抗菌活性,已经取得国家1类新药证书和生产批文,并在抗击新冠肺炎疫情中进入科技部发布的国家新冠治疗方案。本研究应用CRISPR-Cas9系统成功构建了没有任何抗性标记的新型CAM工程菌株54IA-1和54IA-2,并进一步通过核糖体工程技术筛选获得了新型CAM高产菌株RFP40-6-8,为CAM工程菌株的优化奠定了基础。
原有的CAM工程菌,由于经过多次的基因改造使其带有两种抗性基因,很难进行其他的基因改造。而CRISPR-Cas9系统可以通过sgRNA指导Cas9蛋白对靶基因进行双链剪切,利用细胞自身的同源定向修复机制完成基因的编辑,目前已广泛应用于链霉菌基因组的修饰或改造。本研究在应用CRISPR-Cas9系统构建新型CAM工程菌时,设计了两条切割位点不同的sgRNA,成功构建了没有任何抗性标记的新型CAM菌株54IA-1和54IA-2。测序结果显示目的片段在两株菌中的插入位点与插入序列完全一致,但是发酵结果却显示两株菌合成CAM的能力大不相同,菌株54IA-2的产量明显高于菌株54IA-1。qPCR结果显示54IA-2中ist、acyB2、bsm3、bsm5、bsm9和bsm42的表达量均高于54IA-1。推测可能是sgRNA-1引导Cas9蛋白对目的基因切割时产生脱靶,影响CAM的生物合成,导致了菌株54IA-1产量偏低。后续将对两株工程菌株进行全基因组测序,以确定是否由于脱靶现象对菌株基因组产生影响而导致的CAM产量差异。总之,利用CRISPR-Cas9系统成功构建的新型CAM工程菌54IA-1和54IA-2为进一步遗传改造提供了菌种基础。
新型CAM工程菌54IA-2的发酵水平虽然高于54IA-1,但产量还比较低。本实验选取RFP为筛选压力通过核糖体工程来筛选高产菌株。研究证实,RFP抗性突变株是由于rpoB基因(编码RNA聚合酶β亚基) 发生突变所导致的[27]。通过RFP抗性筛选54IA-2突变株发现,40 μg/mL RFP筛选获得的抗性突变株的CAM产量提高比较明显,且所有测序菌株均存在rpoB基因突变。RFP40-6-1、RFP40-6-5菌株中Arg440变为His,与Hu等[27]报道的Streptomyces lividans 66正突变株RH-1的突变位点相同。RFP40-6-2、RFP40-6-3和RFP40-6-7的Asp427变为Glu,与Hu等[27]报道的S. lividans 66正突变株DE-1和Ma等[28]报道的Toyocamycin产生菌1628正突变株1628-T09的突变位点相同。文献报道[27-30]另一个出现频率较高的rpoB基因突变位点是1 309位的C碱基和1 310位的A碱基,即His437可突变为Leu、天冬酰胺(Asparagine,Asn)、酪氨酸(Tyrosine,Tyr)、Arg、半胱氨酸(Cysteine,Cys) 或Asp,但在突变株RFP40-6-12中发生了一个新的突变His437Pro。CAM产量提高最为明显的RFP40-6-8中发生了Gln424Leu突变,与经过5轮抗性筛选(一次PRM抗性突变、3次STR抗性突变、一次RFP抗性突变) 的高产四烯类突变株G5-59[31]发生在rpoB基因上的突变位点相同。结果表明,本次实验所得的RFP抗性突变株除Ser576Ala突变外,其他突变位点均位于rpoB基因RFP抗性突变易发生区域。并且rpoB基因不同的突变位点会导致CAM产量的不同,通过RFP抗性筛选能够快速有效地获得CAM高产菌株。进一步研究rpoB基因突变与CAM产量之间的关系,将为CAM工程菌株的优化指明方向。
| [1] |
Shang GD, Dai JL, Wang YG. Construction and physiological studies on a stable bioengineered strain of Shengjimycin. J Antibiot (Tokyo), 2001, 54(1): 66-73. DOI:10.7164/antibiotics.54.66
|
| [2] |
He WQ, Yang CP, Zhao XF, et al. Antimicrobial activity of bitespiramycin, a new genetically engineered macrolide. Bioorg Med Chem Lett, 2017, 27(19): 4576-4577. DOI:10.1016/j.bmcl.2017.08.046
|
| [3] |
孙丽文, 朱锦桃, 林赴田. 生技霉素药代动力学性能研究. 中国药理学通报, 2000, 16(6): 694-698. Sun LW, Zhu JT, Lin FT. Pharmacokinetics of SJ-SPM. Chin Pharmacol Bull, 2000, 16(6): 694-698 (in Chinese). DOI:10.3321/j.issn:1001-1978.2000.06.029 |
| [4] |
Shi XG, Zhong DF, Sun NL. Pharmacokinetics of a novel antibiotic bitespiramycin in rats. Asian J of Drug Metab Pharmacokinet, 2003, 3(2): 134-137.
|
| [5] |
Shi XG, Sun YM, Zhang YF, et al. Tissue distribution of bitespiramycin and spiramycin in rats. Acta Pharmacol Sin, 2004, 25(11): 1396-1401.
|
| [6] |
Gupta D, Bhattacharjee O, Mandal D, et al. CRISPR-Cas9 system: a new-fangled dawn in gene editing. Life Sci, 2019, 232: 116636. DOI:10.1016/j.lfs.2019.116636
|
| [7] |
Ran FA, Hsu PD, Wright J, et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc, 2013, 8(11): 2281-2308. DOI:10.1038/nprot.2013.143
|
| [8] |
Huang H, Zheng GS, Jiang WH, et al. One-step high-efficiency CRISPR/Cas9-mediated genome editing in Streptomyces. Acta Biochim Biophys Sin, 2015, 47(4): 231-243. DOI:10.1093/abbs/gmv007
|
| [9] |
Qin ZW, Munnoch JT, Devine R, et al. Formicamycins, antibacterial polyketides produced by Streptomyces formicae isolated from African Tetraponera plantants. Chem Sci, 2017, 8(4): 3218-3227. DOI:10.1039/C6SC04265A
|
| [10] |
Zhang MM, Wong FT, Wang YJ, et al. CRISPR-Cas9 strategy for activation of silent Streptomyces biosynthetic gene clusters. Nat Chem Biol, 2017, 10: 10.
|
| [11] |
张晓婷, 张妍, 戴剑漉, 等. 利用CRISPR-Cas9系统构建新型异戊酰螺旋霉素Ⅰ产生菌. 生物工程学报, 2019, 35(3): 472-481. Zhang XT, Zhang Y, Dai JL, et al. Construction of a new isovalerylspiramycin Ⅰ producing strain by CRISPR-Cas9 system. Chin J Biotech, 2019, 35(3): 472-481 (in Chinese). |
| [12] |
Shima J, Hesketh A, Okamoto S, et al. Induction of actinorhodin production by rpsL (encoding ribosomal protein S12) mutations that confer streptomycin resistance in Streptomyces lividans and Streptomyces coelicolor A3(2). J Bacterial, 1996, 178(24): 7276-7281. DOI:10.1128/jb.178.24.7276-7284.1996
|
| [13] |
Ochi K. From microbial differentiation to ribosome engineering. Biosci Biotechnol Biochem, 2007, 71(6): 1373-1386. DOI:10.1271/bbb.70007
|
| [14] |
Ochi K, Okamoto S, Tozawa Y, et al. Ribosome engineering and secondary metabolite production. Adv Appl Microbiol, 2004, 56: 155-184.
|
| [15] |
Zhu SB, Duan YW, Huang Y. The application of ribosome engineering to natural product discovery and yield improvement in Streptomyces. Antibiotics (Basel), 2019, 8(3): 133. DOI:10.3390/antibiotics8030133
|
| [16] |
Kieser T, Bibb MJ, Buttner MJ, et al. Practical Streptomyces genetics. Norwich: The John Innes Foundation, 2000.
|
| [17] |
王以光, 金莲舫, 金文藻, 等. 麦迪霉素4″-酰化酶基因的克隆及在螺旋霉产生菌中的表达. 生物工程学报, 1992, 8(1): 1-14. Wang YG, Jin LF, Jin WZ, et al. Cloning of midecamycin 4"-acyltransferase and expression spiramycin producing strains. Chin J Biotech, 1992, 8(1): 1-14 (in Chinese). |
| [18] |
徐平, 李文均, 徐丽华, 等. 微波法快速提取放线菌基因组DNA. 微生物学通报, 2003, 30(4): 82-84. Xu P, Li WJ, Xu LH, et al. A microwave-based method for genomic DNA extraction from Actinomycetes. Microbiol China, 2003, 30(4): 82-84 (in Chinese). DOI:10.3969/j.issn.0253-2654.2003.04.020 |
| [19] |
Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning: A Laboratory Manual. 2nd ed. New York: Cold Spring Harbor Laboratory Press, 1989: 20-25.
|
| [20] |
国家药典委员会. 中华人民共和国药典(2020版, 第二部). 北京: 中国医药科技出版社. 2020: 12. National Pharmacopoeia Commission. Pharmacopoeia of the People's Republic of China (2020 ed, Vol Ⅱ). Beijing: China Medical Science Press, 2020: 12 (in Chinese). |
| [21] |
王以光, 姜洋, 赵小峰, 等. 可利霉素生物合成基因簇: 中国201511028754.9, 2019-01-22. Wang YG, Jiang Y, Zhao XF, et al. Bitespiramycin biosynthetic gene cluster: CN, 201511028754.9.2019-01-22 (in Chinese). |
| [22] |
Dai JL, Wang YG, Liu JJ, et al. The regulatory genes involved in spiramycin and bitespiramycin biosynthesis. Microbiol Res, 2020, 240: 126532. DOI:10.1016/j.micres.2020.126532
|
| [23] |
Karray F, Darbon E, Oestreicher N, et al. Organization of the biosynthetic gene cluster for the macrolide antibiotic spiramycin in Streptomyces ambofaciens. Microbiology, 2007, 153(12): 4111-4122. DOI:10.1099/mic.0.2007/009746-0
|
| [24] |
Karray F, Darbon E, Nguyen HC, et al. Regulation of the biosynthesis of the macrolide antibiotic spiramycin in Streptomyces ambofaciens. J Bacteriol, 2010, 192(21): 5813-5821. DOI:10.1128/JB.00712-10
|
| [25] |
Hu XJ, Li T, Wang Y, et al. Prokaryotic and highly-repetitive WD40 proteins: a systematic study. Sci Rep, 2017, 7: 10585. DOI:10.1038/s41598-017-11115-1
|
| [26] |
Jin ZH, Jin X, Jin QC. Conjugal transferring of resistance gene ptr for improvement of pristinamycin-producing Streptomyces pristinaespiralis. Appl Biochem Biotechnol, 2010, 160(6): 1853-1864. DOI:10.1007/s12010-009-8691-z
|
| [27] |
Hu H, Zhang Q, Ochi K. Activation of antibiotic biosynthesis by specified mutations in the rpoB gene (encoding the RNA polymerase β subunit) of Streptomyces lividans. J Bacteriol, 2002, 184(14): 3984-3991. DOI:10.1128/JB.184.14.3984-3991.2002
|
| [28] |
Ma Z, Luo S, Xu XH, et al. Characterization of representative rpoB gene mutations leading to a significant change in toyocamycin production of Streptomyces diastatochromogenes 1628. J Ind Microbiol Biotechnol, 2016, 43(4): 463-471. DOI:10.1007/s10295-015-1732-4
|
| [29] |
Liu L, Pan J, Wang ZL, et al. Ribosome engineering and fermentation optimization leads to overproduction of tiancimycin A, a new enediyne natural product from Streptomyces sp. CB03234. J Ind Microbiol Biotechnol, 2018, 45(3): 141-151. DOI:10.1007/s10295-018-2014-8
|
| [30] |
Hosaka T, Ohnishi-Kameyama M, Muramatsu H, et al. Antibacterial discovery in actinomycetes strains with mutations in RNA polymerase or ribosomal protein S12. Nat Biotechnol, 2009, 27(5): 462-464. DOI:10.1038/nbt.1538
|
| [31] |
汤谷. 利用核糖体工程技术提高淀粉酶产色链霉菌1628产量的研究[D]. 杭州: 中国计量大学, 2017. Tang G. Antibiotic overproduction of Streptomyces diastatochromogenes 1628 by ribosome engineering[D]. Hangzhou: China Jiliang University, 2017 (in Chinese). |
 2021, Vol. 37
2021, Vol. 37




