中国科学院微生物研究所、中国微生物学会主办
文章信息
- 杨建花, 苏晓岚, 朱蕾蕾
- Yang Jianhua, Su Xiaolan, Zhu Leilei
- 高通量筛选系统在定向改造中的新进展
- Advances of high-throughput screening system in reengineering of biological entities
- 生物工程学报, 2021, 37(7): 2197-2210
- Chinese Journal of Biotechnology, 2021, 37(7): 2197-2210
- 10.13345/j.cjb.200431
-
文章历史
- Received: July 19, 2020
- Accepted: September 11, 2020
- Published: May 18, 2020
2. 中国科学技术大学,安徽 合肥 230026
2. University of Science and Technology of China, Hefei 230026, Anhui, China
随着生物技术的快速发展,酶和细胞工厂越来越多地应用于食品、化工、环境、能源等各个行业,推动了工业与能源的可持续发展[1-3]。一般天然酶和细胞的性能难以满足工业需求,需要通过定向改造对其性能进行改进,如提高酶的活性、立体选择性、稳定性、底物特异性和细胞工厂的生产效率等[4-6]。
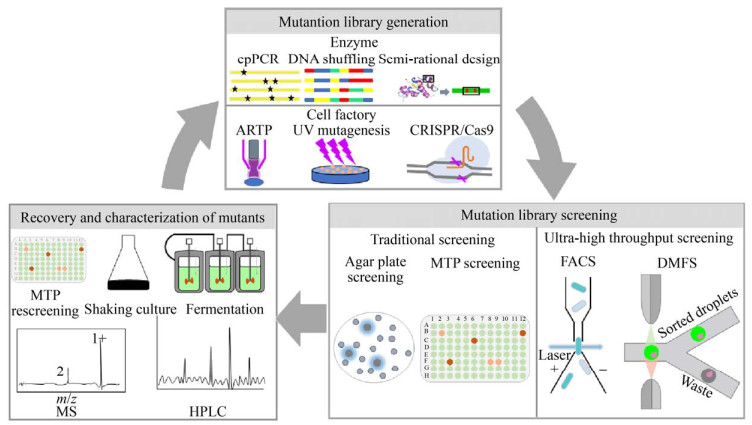
定向改造(图 1) 是在实验室模拟自然进化的过程[7-8]。其关键步骤包括:(1) 突变库的建立:通过不同方法人为地引入大量突变,构建目的蛋白基因或全细胞基因组突变库;(2) 突变库的筛选:通过合适的筛选方法快速地从突变库中筛选出符合要求的突变体;(3) 突变体性能表征与分析:回收和鉴定突变体,并选择优势突变体作为下一轮进化的起点,如此循环迭代,快速提高酶的性能或细胞工厂的生产效率[9-10]。在定向改造过程中,突变库的多样性和可靠的高通量筛选方法是决定其成败的两个关键因素[11-14]。目前,突变库的构建方法已得到广泛开发,如易错PCR[15]、基因重组[16]、Casting易错PCR[17]、序列饱和突变(Sequence saturation mutagenesis,SeSaM)[18]、基因编辑[19]、常压室温等离子体诱变[20]等,能够在较短时间构建容量大于107的突变库。然而,灵敏、可靠的超高通量筛选方法(> 106) 仍然是一个瓶颈,制约着酶和细胞工厂的高效进化[21-22]。
|
| 图 1 定向进化流程图(定向进化过程包括多轮突变库的构建、突变库筛选和优良突变体的回收与鉴定) Fig. 1 The procedure of directed evolution. Directed evolution includes generation of mutation library, screening of mutation library, recovery and characterization of excellent mutants. ARTP: atmospheric and room temperature plasma; MS: mass spectrometry; HPLC: high performance liquid chromatography. |
| |
传统筛选方法,如琼脂平板和微孔板筛选法,是目前应用最广泛的两种筛选技术。琼脂平板法操作简单,但难以准确定量,主要用于突变库的初筛。微孔板筛选方法能够精准定量,可以准确评价突变体的性能,但通量较低。自动化液体处理设备的应用使得微孔板筛选通量有所提升,但仍无法满足快速定向改造中筛选的需求。随着仪器设备的改进和生物技术的快速发展,基于流式细胞仪的荧光激活细胞分选(Fluorescence-activated cell sorting,FACS) 和基于微流控芯片和分选设备的液滴微流控分选(Droplet-based microfluidic sorting,DMFS) 技术大幅度提高了筛选通量(图 1)[23-24]。FACS是一种发展较成熟的技术,比较适用于细胞内或细胞膜上目标产物的筛选,而液滴微流控高通量筛选方法则可弥补FACS的不足,可用于胞外酶和代谢产物的筛选[25]。高通量筛选获得的突变体一般需要通过液相色谱、气相色谱等进行产物的进一步验证。
本文主要从不同通量筛选方法及其信号检测策略的优缺点出发,结合近3年超高通量筛选技术在酶和细胞工厂定向改造方面的应用实例,综述了高通量筛选方法在酶和细胞工厂定向改造中的最新研究进展,并对其发展作出展望。
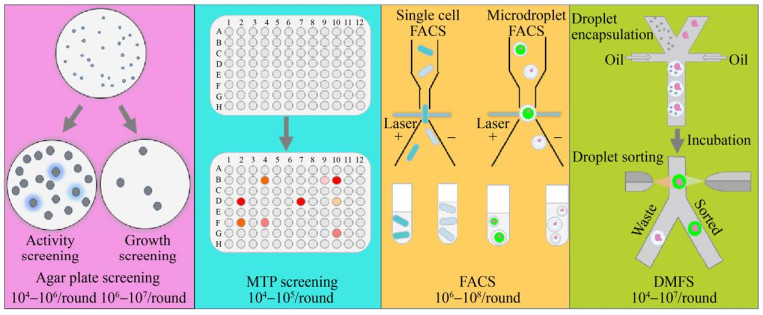
1 传统筛选方法与超高通量筛选方法的比较传统的琼脂平板筛选法和微孔板筛选方法是目前最常用的两种筛选方法(图 2)。基于透明圈、颜色圈的琼脂平板活性筛选或基于营养缺陷型或抗性的琼脂平板生长选择可作为简单易行的初筛方法,用于排除大量无活性和极低活性的突变体。但并不是所有的改造目标都能建立琼脂平板筛选法,更由于其难以准确定量的限制,根据荧光或吸光度精确检测目标产物的微孔板(Microtiter plate,MTP) 筛选方法应运而生,并已广泛应用于酶和细胞工厂的定向改造中[26-27]。但微孔板筛选法存在通量低、操作耗时等缺点。为解决以上问题,近年来开发了荧光激活细胞分选(FACS)和液滴微流控分选(DMFS) 等超高通量筛选方法(> 107),用于酶和细胞工厂定向改造中大容量突变库的筛选(图 2)[28-30]。
|
| 图 2 不同通量筛选方法的比较 Fig. 2 The comparison of different screening methods. |
| |
琼脂平板法是一种简单直接的筛选方法,已用于多种水解酶(如脂肪酶、酯酶、蛋白酶) 和氧化还原酶(如漆酶) 等突变库的初步筛选中[31]。琼脂平板法可分为表型活性筛选和表型生长选择。表型活性筛选利用菌落周围产生的水解圈、颜色圈或荧光产物等进行酶活或目标产物筛选;表型生长选择根据细胞对抗生素或其他有害物质的抗性或营养缺陷型互补,在选择培养基中依据生长情况进行筛选。但是琼脂平板法对突变体间差异可视化较弱,仅适用于突变库的初步筛选,筛选后的突变体仍需要其他检测方法如微孔板法进行准确定量。数字影像分光光度计在琼脂平板筛选方法中的应用使琼脂平板法的灵敏度提高且通量达到105克隆/d[32]。若可根据目标酶或代谢产物的特性建立琼脂平板筛选方法,则无需使用依赖复杂仪器设备的超高通量筛选法。
1.2 微孔板筛选微孔板筛选方法通过检测微孔板中底物或目标产物所引起的吸光度或荧光变化对其进行定量分析,可以保证筛选的精确性和灵敏度,是目前最常用的筛选方法[33]。反应体积一般在100-200 μL,反应液用量较少从而节约试剂成本。目前常用的微孔板一般为96孔板,其与自动单克隆采集系统(Automatic colony picker)、液体自动处理系统(Liquid handling system) 兼容,能够提高筛选通量最高达到104克隆/d,但机械自动化设备的引入增加了实验成本[12]。此外为增加筛选通量且节省筛选时间,384、1 536、3 456孔微孔板也已应用于目标代谢产物和酶的筛选,也可节约昂贵底物的使用[34]。总体来说,微孔板筛选的通量仍然受限,不利于对大容量突变库的快速筛选。
1.3 荧光激活细胞分选(FACS)FACS是一种可对单细胞进行高效分选的荧光激活细胞分选技术[11]。FACS可以根据细胞大小或荧光以高达107克隆/h的速率对细胞进行分选[35]。此外,FACS可以直接将筛选到的优势突变体分配到微孔板中进行回收与鉴定。为进行FACS筛选,首先必须建立酶活性表型与其编码基因的偶联,即将酶活性转化为可检测的荧光信号,并与酶所在的细胞构建物理联系,保持表型与基因型的一致性[36-37]。根据荧光产物与酶及其编码基因偶联形式的不同,现有的FACS酶活性筛选体系可分为细胞膜表面展示、胞内荧光产物的富集、荧光蛋白表达活性报告等类型。当检测目标为胞外分泌酶或代谢产物时,可将细胞包埋在水/油/水双液滴或水凝胶中从而保证基因型和表型的关联[38-39]。液滴包埋拓宽了FACS的应用范围。
1.4 液滴微流控筛选(DMFS)DMFS方法通过在芯片上持续高频(> 10 kHz)地将单个细胞包埋在液滴中实现基因型与表型的偶联,并通过检测液滴内的物质信号进行定量分析与分选[23, 25],其筛选通量高达105克隆/h。油包水液滴提供的纳升至皮升级反应区室,使此筛选方法不仅适用于细胞内酶或代谢产物的筛选,也适用于胞外分泌酶或代谢产物的筛选[40]。此方法相比于常规的微升至毫升级反应体系缩小了百万倍以上,对试剂的需求量大大降低,在用到昂贵底物或试剂时具有明显优势。此外,单层液滴包埋后仍可进行分析试剂的注入、液滴融合、分裂等,大大提高了操作的灵活性。
将油包水单液滴再进行水相包埋后形成的水/油/水双液滴也可用FACS进行筛选,通量进一步提高到107克隆/h[38]。此外,利用微流控芯片形成的水凝胶微滴也可以利用FACS进行筛选[39]。水凝胶微滴比较稳定,可以用于长时间的细胞培养。但双液滴和水凝胶较难进行液滴形成后的后续操作如分析试剂的注入、液滴融合、分裂等,不利于整个筛选过程的灵活操控。
DMFS结合了精密的液滴操作和快速分选系统,已经成为定向改造胞外酶和代谢物突变库筛选的有力工具。自2010年Agresti等首次利用液滴微流控成功地改造辣根过氧化物酶后,DMFS已广泛地应用到其他酶和细胞工厂的定向改造中[41]。DMFS针对胞外产物的高通量筛选具有显著优势,但整个DMFS筛选过程较复杂且技术性较强,对于初学者来说较难操作,且针对不同的目标产物或酶,DMFS筛选体系参数变化较大,需要进行大量系统优化。
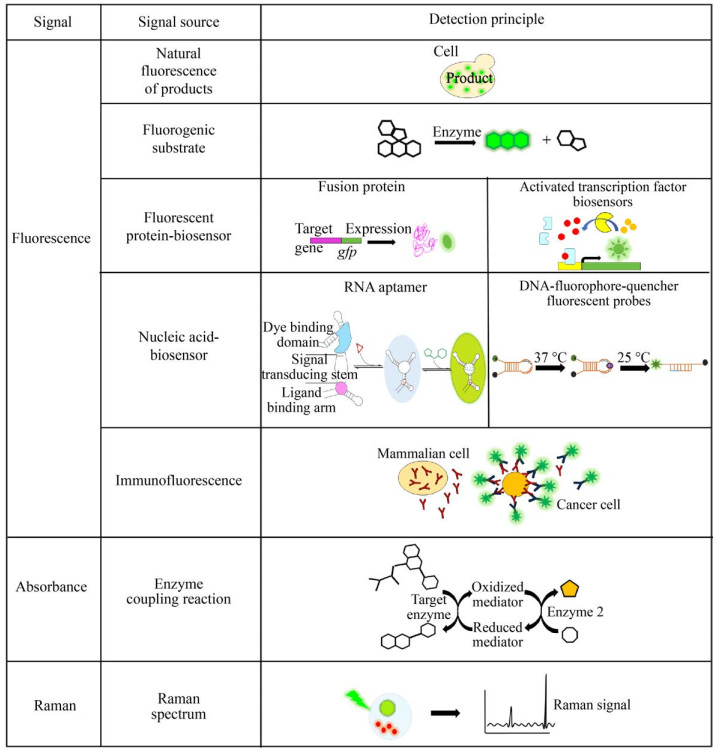
2 高通量筛选方法的信号检测策略合适的信号检测策略是筛选方法建立的核心问题,目前突变库筛选中常用的检测主要基于荧光信号。近年来吸光度和拉曼光谱等开始应用于液滴微流控筛选体系中(图 3)。通过荧光检测目标产物可以比较灵敏、可靠地进行定量分析[42-43]。由于其超敏性、高速响应能力以及拥有较成熟的检测器,荧光成为超高通量筛选方法中最常用的检测信号[44]。近年来发展的根据吸光光度值、拉曼光谱和质谱的检测方法开始应用于超高通量筛选中,但这些技术仍不成熟,需要进一步的发展,提高其灵敏性、易操作性、筛选通量等。下面我们将对每种检测方法的原理及应用进行详细的讨论。
|
| 图 3 高通量筛选方法中的信号产生原理(主要包括基于荧光、吸光度和拉曼光谱的检测方法及其原理) Fig. 3 Principle of signal generation in high-throughput screening methods, including detection methods and their principles based on fluorescence, absorbance and Raman spectrum. |
| |
由于荧光灵敏度高、响应能力强,且拥有成熟的检测器,因此,荧光检测法已成为酶和细胞工厂改造中最常用的突变库筛选方法。根据荧光产生方式不同,可分为产物自荧光、荧光底物、荧光蛋白、核酸生物传感器和免疫荧光等。
2.1.1 产物自荧光当目标产物如核黄素、甜菜碱、喜树碱等自身具有荧光时,可通过直接检测该荧光信号进行筛选。基于核黄素自身荧光检测的高通量DMFS系统已成功应用于乳酸链球菌和解脂耶氏酵母全基因组突变库的筛选,并分别获得了核黄素生产产量提高4倍和1.9倍的乳酸链球菌和解脂耶氏酵母突变体[45-46]。虽然基于自身荧光的检测相对简单,遗憾的是大多数目标产物自身并不具备荧光,需借助其他来源的荧光信号。
2.1.2 荧光底物荧光基团标记的底物可以在酶催化下释放荧光基团产生荧光信号,因此可根据荧光信号的强弱检测相关酶的活性。目前用于标记底物的荧光基团主要有试卤灵、香豆素、荧光素、BODIPY和二甲基硼等。大多数荧光基团标记的底物适用于水解酶和氧化还原酶,仅有少数荧光底物用于其他酶类,如连接酶、裂合酶、转移酶等。以二甲膦酰二氟-荧光素为底物筛选芳香基硫酸酯酶突变库,Kintses等获得了总酶活和表达量提高6倍的突变体[47]。以BODIPY-淀粉为底物,筛选全基因组突变库,Huang等获得了8个淀粉酶分泌量提高6倍的酿酒酵母突变体[48-50]。当目标酶缺少可直接催化的荧光标记底物时,可通过偶联其他反应催化荧光底物产生荧光信号。在筛选纤维素酶突变库中,真实底物羧甲基纤维素钠在纤维素酶催化下形成的单糖被己糖胺苷酶氧化形成H2O2。随后,H2O2与荧光底物荧光素-氨基苯反应产生荧光产物,根据产生的荧光信号来测定纤维素酶突变体的活性[51]。
2.1.3 荧光报告蛋白荧光蛋白是一种基因编码的荧光发色团,自维多利亚发光水母Aequorea victoria中绿色荧光蛋白GFP首次被发现后,其他来源的荧光蛋白及其衍生物等被陆续开发[52-53]。荧光蛋白的吸收和发射波长覆盖了从400到700 nm的所有波段,可以用于不同的酶或细胞工厂的筛选需求。
首先,荧光蛋白可作为融合蛋白检测目标蛋白表达量。通过将GFP与糖皮质激素受体配体结合域融合,利用FACS筛选到了稳定性和溶解度提高的突变体。其次,荧光蛋白也可作为报告蛋白(Reporter protein) 用于酶或目标代谢产物的检测,其中应用较广泛的是转录因子生物传感器[54]和核糖体开关生物传感器[55]。转录因子生物传感器是将异源转录因子移植到新的目标宿主中,并与基因的启动子或者增强子区域结合。目标酶的底物、产物或目标代谢产物可结合转录因子进而促进或阻断RNA聚合酶参与的转录过程,使得荧光报告基因的转录和表达与目标酶活性或目标代谢产物的浓度产生关联,据此可进行检测。核糖体开关生物传感器是通过目标产物与RNA核糖体开关的结合引起其构象改变,从而开启或阻断报告基因的转录和表达。根据荧光强度可以检测目标产物的产量,因此荧光蛋白-传感器适用于超高通量筛选体系中。利用基于荧光蛋白的传感器可以检测乙醇、糖类、氨基酸、芳香物质、抗生素、脂肪酸等[55]。随着研究与技术的不断发展与深入,越来越多转录因子和核糖体开关生物传感器被开发与应用。
总体来说,基于荧光蛋白的检测方法操作简单、应用广泛,但仍然需要突破以下局限:作为融合蛋白,由于荧光蛋白分子量大,可能会影响目标蛋白功能性表达;作为转录因子或核糖体开关生物传感器,其关键是需要通过条件优化提高生物传感器的灵敏度,使其适合所需的产物检测范围[54-55]。
2.1.4 核酸传感器核酸传感器是一种具有特定构象的基因序列,相比于荧光蛋白传感器,核酸传感器如核酸适配体和DNA-fluorophore-quencher荧光探针,具有特异性高、容易修饰、稳定性好等特点。核酸适配体能与多种目标代谢物特异性结合,因此被广泛应用于生物传感器领域。RNA适配体由荧光染料结合区、信号传导区和配体结合区组成。据报道,RNA适配体已被用于筛选高产酪氨酸的细胞工厂,当大量的酪氨酸与荧光染料同时结合到RNA适配体上时,适配体构象变化从而引起荧光强度增强[56]。RNA适配体是一种RNA分子,可通过改造来识别其他的代谢产物,因此随着新适配体的不断开发,其在代谢产物检测与细胞工厂筛选方面会有更广阔的应用。DNA-fluorophore-quencher荧光探针可用于检测DNA相关酶类,如DNA聚合酶、DNA连接酶、DNA限制性内切酶[57]。基于荧光探针的DMFS平台已经被成功应用于筛选高活性的KOD聚合酶突变体[57]。
2.1.5 免疫荧光免疫荧光法是将免疫学和荧光染色法结合在一起的方法,即用荧光标记的抗体或抗原与被检测样品中相应的抗原或抗体结合,通过激光器的激发检测荧光,并对样品进行分析的方法,常用于抗体的检测中,具有特异性强、灵敏度高和快速可靠等特点[58]。基于这一原理,Shembekar等开发了一种免疫荧光DMFS系统,用于选择高产抗体的哺乳动物细胞[58]。这种方法可以筛选对某一种癌细胞或细菌具有杀伤作用的抗体,通用性强,具有巨大的药物挖掘潜力。
2.2 分光光度检测法分光光度法是一种通过测定物质在特定波长或一定波长范围内的吸光度对该物质进行定性和定量分析的方法。由于其原理简单,已被广泛应用于微孔板筛选中。近年来,基于分光光度检测方法的超高通量DMFS系统已有报道。此系统通过使用1-甲氧基-5-甲基吩嗪硫酸甲酯(mPMS) 作为氧化还原介体,将苯丙氨酸脱氢酶(PheDH)对NAD+的消耗与蓝紫色染料结晶甲臜fomazan的形成联系起来,大幅度放大了吸光度信号,增强了检测的准确性和灵敏度[42]。但相比于荧光信号,基于吸光度的信号检测灵敏度低(比荧光信号低3-4个数量级),需要使用大液滴来增加光路从而收集足够的信号。随着技术的进一步发展,分光光度检测法将大大拓展DMFS的应用范围[42]。
2.3 拉曼光谱检测法当目标产物无法标记时,需用其他方法来对其进行检测。2018年,Wang等建立了一个集成的拉曼液滴微流控分选系统,该系统将拉曼信号采集和分析集成到一个细胞包埋和分选的自动系统中,用于筛选产虾青素的微藻细胞[43]。此方法基于对单细胞内化学物质拉曼图谱的获取以及与数据库中细胞拉曼数据进行比对来鉴别目标细胞,不需要标记细胞,因此对细胞无侵害性,具有广泛的应用前景。但由于拉曼信号较弱,导致信号采集时间较长,筛选通量(260液滴/min) 远低于荧光分选(600液滴/s)。进一步提高拉曼信号检测的灵敏度,减少信号采集时间,将会使其拥有更广阔的应用前景。
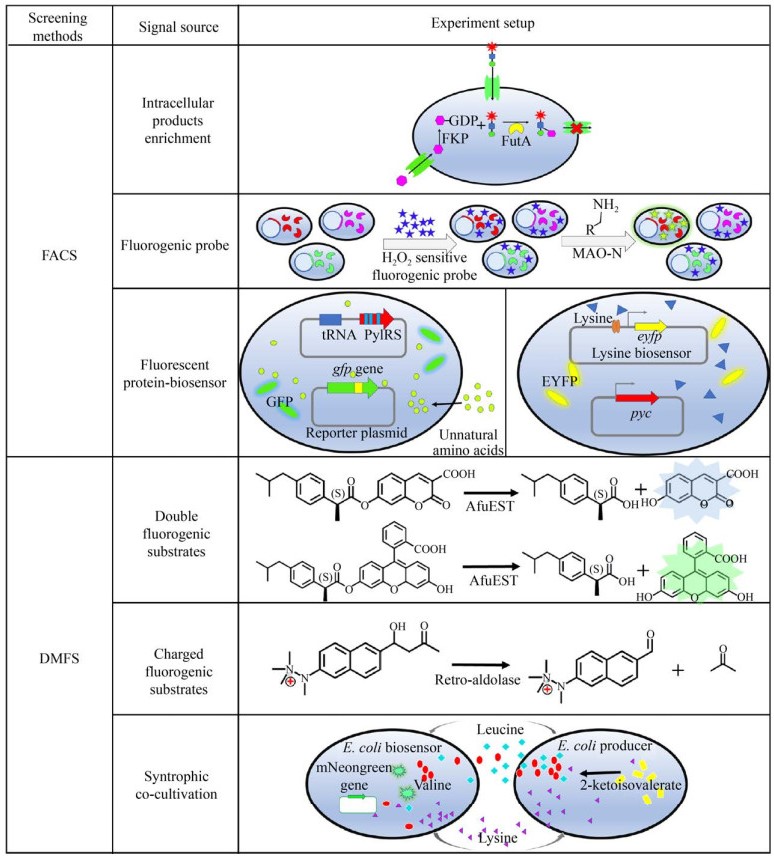
3 超高通量筛选方法在酶和细胞工厂定向改造中的最新应用近10年来超高通量FACS和DMFS迅速发展[22-23, 59],大大提高了酶和细胞工厂定向改造的效率。下面主要介绍近3年的最新案例,阐明超高通量筛选方法在酶与细胞工厂定向改造应用中的最新研究进展,以及各案例的实验设计、研究思路等(图 4)。
|
| 图 4 近3年高通量筛选在酶和细胞工厂定向改造应用中的信号产生策略 Fig. 4 The signal production strategies of high throughput screening in the applications of enzyme and cell factory engineering in the past three years. The signal generation strategies of FACS in the applications of enzyme and cell factory engineering including intracellular products enrichment, fluorogenic probe and fluorescent protein biosensor. The signal generation strategies of DMFS in the applications of enzyme and cell factory engineering including double fluorogenic substrates, charged fluorogenic substrate and syntrophic co-cultivation of biosensor strain and products producing strain. FKP: GDP-fucose synthase; FutA: α1, 3-fucosyltransferase; MAO-N: monoamine oxidase; PylRS: pyrrolysyl-tRNA synthetase; EYFP: enhanced yellow fluorescent protein; pyc: pyruvate carboxylase gene; AfuEST: esterase from Archaeoglobus fulgidus. |
| |
FACS技术的高灵敏性及高通量等特点使其能以极高的分选频率(10-15 kHz) 对大容量突变库进行高通量筛选,已广泛应用在新酶发现、高酶活筛选及细胞工厂的定向改造中。
由于FACS是全细胞水平的筛选,因此必须通过细胞膜表面展示、胞内荧光产物的富集、荧光报告基因等方法保证在筛选过程中基因型和表型的一致性。Tan等利用胞内荧光产物的富集原理构建了超高通量FACS筛选方法,并成功筛选到活性提高14倍的α-1, 3-岩藻糖基转移酶突变体[60]。通过合理设计,使细胞膜上的转运蛋白识别特殊荧光底物并将其转运到细胞内进行反应,但形成的荧光产物无法被特定的跨膜转运体识别而在细胞内富集引起荧光的积累。
单胺氧化酶是一类极具应用潜力的生物催化剂,可选择性氧化一种构型的胺生成亚胺。Sadler等开发了一种基于H2O2荧光探针的高通量筛选法用于筛选单胺氧化酶(Monoamine oxidase,MAO-N) 突变库[61]。将突变的MAO-N基因在大肠杆菌中成功表达后,加入检测H2O2的荧光探针(C-H2DCFDA) 进行孵育后加入仲胺底物,处理后的细胞催化氧化仲胺底物产生H2O2,后者与荧光探针反应释放荧光信号。通过筛选MAO-N突变库,获得了对仲胺底物具有催化活性的突变体。因此,基于活性氧检测探针的高通量筛选可以用于筛选其他可催化底物产生H2O2的氧化酶。
吡咯赖氨酸-tRNA合成酶(Pyrrolysyl-tRNA synthetases,PylRS) 可以在蛋白质合成过程中引入非天然氨基酸。Lin等开发了一种用于快速发现细菌功能性PylRS的FACS方法[62]。将两个分别含有PylRS基因和绿色荧光蛋白(GFP)-204TAG报告基因的兼容性质粒共转化至大肠杆菌宿主细胞,培养基中加入非天然氨基酸BocK和SphK,当PylRS突变体可以成功引入非天然氨基酸到绿色荧光蛋白TAG处时,绿色荧光蛋白才能顺利完成表达。通过此方法成功筛选到了可以将两个非天然氨基酸(BocK和SphK) 插入到蛋白质特定位点的功能性PylRS突变体。
丙酮酸羧化酶是l-赖氨酸合成途径中的一个关键酶,其活性影响l-赖氨酸的合成效率。为获得高产l-赖氨酸的谷氨酸棒状杆菌,Kortmann等构建了基于赖氨酸转录因子生物传感器的FACS筛选体系筛选以谷氨酸棒状杆菌为宿主菌的丙酮酸羧化酶(Pyruvate carboxylase) 突变体,从而提高谷氨酸棒状杆菌利用葡萄糖生产l-赖氨酸的能力[63]。分别含有编码丙酮酸羧化酶的基因(pyc)和编码eYFP荧光蛋白报告基因的两个质粒共转化至谷氨酸棒状杆菌中,转录调节因子LysG控制着荧光蛋白eYFP的表达。当谷氨酸棒状杆菌高产l-赖氨酸时,LysG能感知l-赖氨酸的浓度并激活荧光蛋白的表达,产生荧光信号。通过此方法筛选丙酮酸羧化酶突变库,成功获得了两个能引起宿主细胞高产l-赖氨酸的丙酮酸羧化酶突变体。
总体来讲,使用FACS进行筛选必须保持表型与基因型的统一,虽然已有很多方法将荧光偶联在细胞内或细胞膜上,如细胞膜荧光吸附、细胞表面展示、胞内荧光产物富集、荧光报告蛋白等。随着调节元件数据库的不断扩充,在数据库中挖掘合适的转录因子、核糖体开关等调节元件来建立基于荧光报告蛋白的FACS超高通量筛选方法已成为目前研究的热点。
3.2 DMFS在酶和细胞工厂改造中的最新应用随着微流控技术的发展,自2010年液滴微流控筛选技术成功应用于过氧化物酶的定向改造以来,该技术已经广泛应用于大容量突变库(> 106)的筛选中[41]。我们主要对自2017年以来的新案例进行阐述与分析。
对映异构体固有的物理化学相似性限制了高通量筛选方法在酶的对映选择性定向改造中的应用,也在一定程度上限制了酶的对映选择性方面的研究进展,长时间来一直缺乏高通量筛选的成功案例。2018年,Yang团队成功开发了基于两种独立荧光信号检测的双通道DMFS平台用于古菌酯酶的对映立体选择性筛选[64]。整个筛选过程分两个阶段:首先,从文库中筛选出对两种不同荧光标记的(S)-profens均有催化活力的突变体,以排除对某荧光基团具有偏好性的古菌酯酶。然后,将一种荧光基团标记的(S)-profens和另外一种荧光基团标记的(R)-profens同时作为底物,从第一步获得的突变体中筛选具有高催化(S)-profens活力和低催化(R)-profens活力的突变体。经过5轮进化,获得了对(S)-profens选择性比野生型高700倍的突变体。此案例首次同时利用两种荧光标记底物对酶进行进化,为提高筛选的准确性开辟了新方向。
为解决荧光产物在液滴间的扩散而互相污染的问题,Obexer等开发了一种基于带电荷的荧光底物衍生物进行逆醛缩酶酶活检测的DMFS系统,提高了筛选的准确性[65]。仅通过一轮筛选,筛选获得活性大幅提高(80倍) 的人工设计逆醛缩酶突变体。因此通过对荧光底物的设计提高其极性,从而减少产物在液滴间的扩散,会大幅度提升DMFS筛选效率及准确率。
此外,DMFS还被用于从自然界中挖掘具有独特底物特异性和高稳定性的新型酶。为了从自然界中高效筛选新型脂肪酶,Qiao等在2019年开发了一种流水线式的DMFS系统,包括4个步骤:液滴的生成、液滴的孵育、荧光素二丁酸底物的注入以及液滴的分选[66]。利用此系统,从不同地方采集到的样品中筛选出隶属于7个属的47株产脂肪酶的细菌菌株。因此DMFS也是一个从自然界中高效挖掘新酶的重要工具。
生物传感器菌株与生产菌株共包埋策略也已成功应用到DMFS系统的构建。为提高大肠杆菌生产2-酮异戊酸的能力,Saleski等建立了一个2-酮异戊酸营养缺陷型大肠杆菌生物传感器细胞和赖氨酸营养缺陷型2-酮异戊酸生产菌株共培养DMFS体系[67]。2-酮异戊酸是合成缬氨酸和亮氨酸的必要前体物质,由于作为传感器的大肠杆菌细胞无法生产2-酮异戊酸,进而无法生成胞内蛋白合成所需的缬氨酸和亮氨酸,因此,mNeongreen荧光蛋白无法表达。当与其共包埋的生产菌株可以生产2-酮异戊酸时,传感器菌株可以吸收并利用生产菌株产生的缬氨酸和亮氨酸来表达荧光蛋白。而生产菌株是赖氨酸营养缺陷型大肠杆菌,其必须吸收来自传感器菌株的赖氨酸供自身生长和2-酮异戊酸的合成。这种互利共培养模式快速放大了荧光信号,利于精准筛选。Siedler等开发了用于筛选高产S-对香豆酸的酿酒酵母细胞工厂的菌株共培养DMFS系统[68]。在此系统中,带有转录因子的大肠杆菌传感器细胞与生产对香豆酸的酿酒酵母细胞共包埋在同一个液滴中。转录因子传感器由枯草芽孢杆菌的酚酸脱羧酶的转录调节子和黄色荧光蛋白(YFP) 基因组成。在同一液滴中,酿酒酵母突变体生产的对香豆酸与抑制YFP表达的阻遏物紧密结合失去了对YFP表达的抑制作用,从而引起YFP表达释放荧光信号。生物传感器菌株与目标生产菌株共包埋的DMFS方法已经成为一种辅助细胞工厂改造的重要策略之一。
随着技术的发展,DMFS也已经应用于高等动植物细胞的筛选中。植物属于多细胞生物且原生质体脆弱而难以长期保持完整,直到2018年才报道了第一例用DMFS筛选植物原生质体的案例,但该筛选通量仍较低(28细胞/s)[69]。在该系统中,原生质体在直径大于50 μm的液滴中能够保持完整超过12 h,满足了液滴孵育的时间要求。虽然2018年之前已经建立了在液滴中分析哺乳动物细胞抗体生产的技术,但这些方法不适用于分泌特异性抗体的哺乳动物细胞的高通量分选。为了实现这一目标,Shembekar等成功开发了哺乳动物细胞和肿瘤细胞共包埋模式的DMFS高通量筛选系统用于筛选分泌针对K562肿瘤细胞抗体的哺乳动物细胞[58]。液滴微流控植物和动物细胞高通量筛选的开发与应用将大大拓宽DMFS在获得高产植物次级代谢产物以及动物蛋白细胞工厂中的应用。
4 展望目前,FACS和DMFS等超高通量筛选方法的研究和应用已取得很大的进展,已在定向改造实验中发挥了不可替代的作用[70-71]。与传统筛选方法相比,FACS和DMFS技术不仅可以对大容量样本进行超高通量的分析和筛选,而且能够对样品的多项指标同时进行定量分析,因此,具有显著的优势。FACS较高的仪器成本和难以对胞外产物进行筛选等缺点制约其应用范围。DMFS虽在筛选通量上比FACS低一个数量级,但由于其可以保持胞外分泌产物与基因型的一致性,成为研究胞外分泌产物及其工程菌株实验室改造的强有力的超高通量筛选平台。然而,目前微流控与液滴分选设备刚进入商业化阶段,随着微流控芯片及分选设备的广泛商业化,以及更灵活的检测系统的开发,相信其将进一步推动酶和细胞工厂的快速开发。
此外,FACS和DMFS主要依赖荧光信号进行检测,虽然基于吸光度和拉曼光谱的DMFS已有报道,但技术不成熟,灵敏度和通量均较低,仍需要进一步的优化与设计提高其灵敏度和筛选通量。相信随着各方面技术的不断发展,基于其他高精度的检测方法,如荧光共振能量转移、荧光偏振、中红外光谱或质谱的超高通量技术会不断涌现并日趋成熟,将进一步推动酶和细胞工程领域的快速发展。
| [1] |
Bornscheuer UT, Huisman GW, Kazlauskas RJ, et al. Engineering the third wave of biocatalysis. Nature, 2012, 485(7397): 185-194. DOI:10.1038/nature11117
|
| [2] |
Gavrilescu M, Chisti Y. Biotechnology—a sustainable alternative for chemical industry. Biotechnol Adv, 2005, 23(7/8): 471-499.
|
| [3] |
Ferrer-Miralles N, Domingo-Espín J, Corchero JL, et al. Microbial factories for recombinant pharmaceuticals. Microb Cell Fact, 2009, 8: 17. DOI:10.1186/1475-2859-8-17
|
| [4] |
Nielsen J. Cell factory engineering for improved production of natural products. Nat Prob Rep, 2019, 36(9): 1233-1236. DOI:10.1039/C9NP00005D
|
| [5] |
Turner NJ. Directed evolution drives the next generation of biocatalysts. Nat Chem Biol, 2009, 5(8): 567-573. DOI:10.1038/nchembio.203
|
| [6] |
Cheng F, Yang JH, Bocola M, et al. Loop engineering reveals the importance of active-site-decorating loops and gating residue in substrate affinity modulation of arginine deiminase (an anti-tumor enzyme). Biochem Biophys Res Commun, 2018, 499(2): 233-238. DOI:10.1016/j.bbrc.2018.03.134
|
| [7] |
Arnold FH. The nature of chemical innovation: new enzymes by evolution. Q Rev Biophys, 2015, 48(4): 404-410. DOI:10.1017/S003358351500013X
|
| [8] |
Bornscheuer UT, Hauer B, Jaeger KE, et al. Directed evolution empowered redesign of natural proteins for the sustainable production of chemicals and pharmaceuticals. Angew Chem Int Ed Engl, 2019, 58(1): 36-40. DOI:10.1002/anie.201812717
|
| [9] |
Cheng F, Zhu LL, Schwaneberg U. Directed evolution 2.0: improving and deciphering enzyme properties. Chem Commun, 2015, 51(48): 9760-9772. DOI:10.1039/C5CC01594D
|
| [10] |
Cheng F, Yang JH, Schwaneberg U, et al. Rational surface engineering of an arginine deiminase (an antitumor enzyme) for increased PEGylation efficiency. Biotechnol Bioeng, 2019, 116(9): 2156-2166. DOI:10.1002/bit.27011
|
| [11] |
Yang GY, Withers SG. Ultrahigh-throughput FACS-based screening for directed enzyme evolution. Chem Bio Chem, 2009, 10(17): 2704-2715. DOI:10.1002/cbic.200900384
|
| [12] |
Xiao H, Bao ZH, Zhao HM. High throughput screening and selection methods for directed enzyme evolution. Ind Eng Chem Res, 2015, 54(16): 4011-4020. DOI:10.1021/ie503060a
|
| [13] |
Longwell CK, Labanieh L, Cochran JR. High-throughput screening technologies for enzyme engineering. Curr Opin Biotechnol, 2017, 48: 196-202. DOI:10.1016/j.copbio.2017.05.012
|
| [14] |
Wong TS, Zhurina D, Schwaneberg U. The diversity challenge in directed protein evolution. Comb Chem High Throughput Screen, 2006, 9(4): 271-288. DOI:10.2174/138620706776843192
|
| [15] |
Lin-Goerke JL, Robbins DJ, Burczak JD. PCR-based random mutagenesis using manganese and reduced dNTP concentration. Bio Techniques, 1997, 23(3): 409-412.
|
| [16] |
刘晔, 耿建华, 刘庆军. 基因重组技术在啤酒酵母育种、改进啤酒风味方面的应用. 酿酒, 2003(4): 47-48. Liu Y, Geng JH, Liu QJ. The application of recombinant DNA technology in beer breeding and improving beer flavor. Liquor Making, 2003(4): 47-48 (in Chinese). |
| [17] |
Yang JH, Ruff AJ, Arlt M, et al. Casting epPCR (cepPCR): a simple random mutagenesis method to generate high quality mutant libraries. Biotechnol Bioeng, 2017, 114(9): 1921-1927. DOI:10.1002/bit.26327
|
| [18] |
Mundhada H, Marienhagen J, Scacioc A, et al. SeSaM-Tv-Ⅱ generates a protein sequence space that is unobtainable by epPCR. Chem Bio Chem, 2011, 12(10): 1595-1601. DOI:10.1002/cbic.201100010
|
| [19] |
Clark J, Ding S. Generation of RNAi libraries for high-throughput screens. J Biomed Biotechnol, 2006, 2006: 45716.
|
| [20] |
金丽华, 方明月, 张翀, 等. 常压室温等离子体快速诱变产油酵母的条件及其突变株的特性. 生物工程学报, 2011, 27(3): 461-467. Jin LH, Fang MY, Zhang C, et al. Operating conditions for the rapid mutation of the oleaginous yeast by atmospheric and room temperature plasmas and the characteristics of the mutants. Chin J Biotech, 2011, 27(3): 461-467 (in Chinese). |
| [21] |
Ye LD, Yang CC, Yu HW. From molecular engineering to process engineering: development of high-throughput screening methods in enzyme directed evolution. Appl Microbiol Biotechnol, 2018, 102(2): 559-567. DOI:10.1007/s00253-017-8568-y
|
| [22] |
Markel U, Essani KD, Besirlioglu V, et al. Advances in ultrahigh-throughput screening for directed enzyme evolution. Chem Soc Rev, 2020, 49(1): 233-262. DOI:10.1039/C8CS00981C
|
| [23] |
Weng LD, Spoonamore JE. Droplet microfluidics-enabled high-throughput screening for protein engineering. Micromachines, 2019, 10(11): 734. DOI:10.3390/mi10110734
|
| [24] |
Becker S, Schmoldt HU, Adams TM, et al. Ultra-high-throughput screening based on cell-surface display and fluorescence-activated cell sorting for the identification of novel biocatalysts. Curr Opin Biotechnol, 2004, 15(4): 323-329. DOI:10.1016/j.copbio.2004.06.001
|
| [25] |
吕彤, 涂然, 袁会领, 等. 毕赤酵母液滴微流控高通量筛选方法的建立与应用. 生物工程学报, 2019, 35(7): 1317-1325. Lü T, Tu R, Yuan HL, et al. Development and application of a droplet-based microfluidic high-throughput screening of Pichia pastoris. Chin J Biotech, 2019, 35(7): 1317-1325 (in Chinese). |
| [26] |
Zhu LL, Tee KL, Roccatano D, et al. Directed evolution of an antitumor drug (arginine deiminase PpADI) for increased activity at physiological pH. ChemBioChem, 2010, 11(5): 691-697. DOI:10.1002/cbic.200900717
|
| [27] |
Zhu LL, Verma R, Roccatano D, et al. Inside cover: a potential antitumor drug (arginine deiminase) reengineered for efficient operation under physiological conditions (Chem Bio Chem 16/2010). Chem Bio Chem, 2010, 11(16): 2194. DOI:10.1002/cbic.201090078
|
| [28] |
Ruff AJ, Dennig A, Wirtz G, et al. Flow cytometer-based high-throughput screening system for accelerated directed evolution of P450 monooxygenases. ACS Catal, 2012, 2(12): 2724-2728. DOI:10.1021/cs300115d
|
| [29] |
Lim SW, Abate AR. Ultrahigh-throughput sorting of microfluidic drops with flow cytometry. Lab Chip, 2013, 13(23): 4563-4572. DOI:10.1039/c3lc50736j
|
| [30] |
Kaminski TS, Scheler O, Garstecki P. Droplet microfluidics for microbiology: techniques, applications and challenges. Lab Chip, 2016, 16(12): 2168-2187. DOI:10.1039/C6LC00367B
|
| [31] |
Popovic A, Tchigvintsev A, Tran H, et al. Metagenomics as a tool for enzyme discovery: hydrolytic enzymes from marine-related metagenomes//Krogan N, Babu M, Eds. Prokaryotic Systems Biology. Cham: Springer, 2015, 883: 1-20.
|
| [32] |
Turner NJ. Directed evolution of enzymes for applied biocatalysis. Trends Biotechnol, 2003, 21(11): 474-478. DOI:10.1016/j.tibtech.2003.09.001
|
| [33] |
Leemhuis H, Kelly RM, Dijkhuizen L. Directed evolution of enzymes: library screening strategies. IUBMB Life, 2009, 61(3): 222-228. DOI:10.1002/iub.165
|
| [34] |
Mewis K, Taupp M, Hallam SJ. A high throughput screen for biomining cellulase activity from metagenomic libraries. J Vis Exp, 2011(48): 2461.
|
| [35] |
马富强, 冯雁, 杨广宇. 基于荧光激活细胞分选技术的超高通量酶活性筛选方法及其应用. 生物化学与生物物理进展, 2012, 39(4): 299-306. Ma FQ, Feng Y, Yang GY. Ultrahigh-throughput enzymatic screening method based on fluorescence-activated cell sorting and its applications. Prog Biochem Biophys, 2012, 39(4): 299-306 (in Chinese). |
| [36] |
Aharoni A, Thieme K, Chiu CPC, et al. High-throughput screening methodology for the directed evolution of glycosyltransferases. Nat Methods, 2006, 3(8): 609-614. DOI:10.1038/nmeth899
|
| [37] |
Copp JN, Williams EM, Rich MH, et al. Toward a high-throughput screening platform for directed evolution of enzymes that activate genotoxic prodrugs. Protein Eng Des Sel, 2014, 27(10): 399-403. DOI:10.1093/protein/gzu025
|
| [38] |
Zinchenko A, Devenish SRA, Kintses B, et al. One in a million: flow cytometric sorting of single cell-lysate assays in monodisperse picolitre double emulsion droplets for directed evolution. Anal Chem, 2014, 86(5): 2526-2533. DOI:10.1021/ac403585p
|
| [39] |
Fischlechner M, Schaerli Y, Mohamed MF, et al. Evolution of enzyme catalysts caged in biomimetic gel-shell beads. Nat Chem, 2014, 6(9): 791-796. DOI:10.1038/nchem.1996
|
| [40] |
Wang BL, Ghaderi A, Zhou H, et al. Microfluidic high-throughput culturing of single cells for selection based on extracellular metabolite production or consumption. Nat Biotechnol, 2014, 32(5): 473-478. DOI:10.1038/nbt.2857
|
| [41] |
Agresti JJ, Antipov E, Abate AR, et al. Ultrahigh-throughput screening in drop-based microfluidics for directed evolution. Proc Natl Acad Sci USA, 2010, 107(9): 4004-4009. DOI:10.1073/pnas.0910781107
|
| [42] |
Gielen F, Hours R, Emond S, et al. Ultrahigh-throughput-directed enzyme evolution by absorbance-activated droplet sorting (AADS). Proc Natl Acad Sci USA, 2016, 113(47): E7383-E7389. DOI:10.1073/pnas.1606927113
|
| [43] |
Wang XX, Ren LH, Su YT, et al. Raman-activated droplet sorting (RADS) for label-free high-throughput screening of microalgal single-cells. Anal Chem, 2017, 89(22): 12569-12577. DOI:10.1021/acs.analchem.7b03884
|
| [44] |
Baret JC, Miller OJ, Taly V, et al. Fluorescence-activated droplet sorting (FADS): efficient microfluidic cell sorting based on enzymatic activity. Lab Chip, 2009, 9(13): 1850-1858. DOI:10.1039/b902504a
|
| [45] |
Chen J, Vestergaard M, Jensen TG, et al. Finding the needle in the haystack-the use of microfluidic droplet technology to identify vitamin-secreting lactic acid bacteria. mBio, 2017, 8(3): e00526-17.
|
| [46] |
Wagner JM, Liu LQ, Yuan SF, et al. A comparative analysis of single cell and droplet-based FACS for improving production phenotypes: riboflavin overproduction in Yarrowia lipolytica. Metab Eng, 2018, 47: 346-356. DOI:10.1016/j.ymben.2018.04.015
|
| [47] |
Kintses B, Hein C, Mohamed MF, et al. Picoliter cell lysate assays in microfluidic droplet compartments for directed enzyme evolution. Chem Biol, 2012, 19(8): 1001-1009. DOI:10.1016/j.chembiol.2012.06.009
|
| [48] |
Huang MT, Bai YP, Sjostrom SL, et al. Microfluidic screening and whole-genome sequencing identifies mutations associated with improved protein secretion by yeast. Proc Natl Acad Sci USA, 2015, 112(34): E4689-E4696. DOI:10.1073/pnas.1506460112
|
| [49] |
Sjostrom SL, Bai YP, Huang MT, et al. High-throughput screening for industrial enzyme production hosts by droplet microfluidics. Lab Chip, 2014, 14(4): 806-813. DOI:10.1039/C3LC51202A
|
| [50] |
Huang MT, Joensson HN, Nielsen J. High-throughput microfluidics for the screening of yeast libraries//Jensen MK, Keasling JD, Eds. Synthetic Metabolic Pathways. New York, NY: Humana Press, 2018, 1671: 307-317.
|
| [51] |
Ostafe R, Prodanovic R, Ung WL, et al. A high-throughput cellulase screening system based on droplet microfluidics. Biomicrofluidics, 2014, 8(4): 041102. DOI:10.1063/1.4886771
|
| [52] |
Rodriguez EA, Campbell RE, Lin JY, et al. The growing and glowing toolbox of fluorescent and photoactive proteins. Trends Biochem Sci, 2017, 42(2): 111-129. DOI:10.1016/j.tibs.2016.09.010
|
| [53] |
Kremers GJ, Gilbert SG, Cranfill PJ, et al. Fluorescent proteins at a glance. J Cell Sci, 2011, 124(2): 157-160. DOI:10.1242/jcs.072744
|
| [54] |
Mannan AA, Liu D, Zhang FZ, et al. Fundamental design principles for transcription-factor-based metabolite biosensors. ACS Synth Biol, 2017, 6(10): 1851-1859. DOI:10.1021/acssynbio.7b00172
|
| [55] |
Fowler CC, Brown ED, Li YF. Using a riboswitch sensor to examine coenzyme B12 metabolism and transport in E. coli. Chem Biol, 2010, 17(7): 756-765. DOI:10.1016/j.chembiol.2010.05.025
|
| [56] |
Abatemarco J, Sarhan MF, Wagner JM, et al. RNA-aptamers-in-droplets (RAPID) high-throughput screening for secretory phenotypes. Nat Commun, 2017, 8(1): 332. DOI:10.1038/s41467-017-00425-7
|
| [57] |
Vallejo D, Nikoomanzar A, Paegel BM, et al. Fluorescence-activated droplet sorting for single-cell directed evolution. ACS Synth Biol, 2019, 8(6): 1430-1440. DOI:10.1021/acssynbio.9b00103
|
| [58] |
Shembekar N, Hu HX, Eustace D, et al. Single-cell droplet microfluidic screening for antibodies specifically binding to target cells. Cell Rep, 2018, 22(8): 2206-2215. DOI:10.1016/j.celrep.2018.01.071
|
| [59] |
Bunzel HA, Garraboul X, Pott M, et al. Speeding up enzyme discovery and engineering with ultrahigh-throughput methods. Curr Opin Struct Biol, 2018, 48: 149-156. DOI:10.1016/j.sbi.2017.12.010
|
| [60] |
Tan YM, Zhang Y, Han YB, et al. Directed evolution of an α1, 3-fucosyltransferase using a single-cell ultrahigh-throughput screening method. Sci Adv, 2019, 5(10): eaaw8451. DOI:10.1126/sciadv.aaw8451
|
| [61] |
Sadler JC, Currin A, Kell DB. Ultra-high throughput functional enrichment of large monoamine oxidase (MAO-N) libraries by fluorescence activated cell sorting. Analyst, 2018, 143(19): 4747-4755. DOI:10.1039/C8AN00851E
|
| [62] |
Lin AE, Lin Q. Rapid identification of functional pyrrolysyl-tRNA synthetases via fluorescence-activated cell sorting. Int J Mol Sci, 2018, 20(1): 29. DOI:10.3390/ijms20010029
|
| [63] |
Kortmann M, Mack C, Baumgart M, et al. Pyruvate carboxylase variants enabling improved lysine production from glucose identified by biosensor-based high-throughput fluorescence-activated cell sorting screening. ACS Synth Biol, 2019, 8(2): 274-281. DOI:10.1021/acssynbio.8b00510
|
| [64] |
Ma FQ, Chung MT, Yao Y, et al. Efficient molecular evolution to generate enantioselective enzymes using a dual-channel microfluidic droplet screening platform. Nat Commun, 2018, 9(1): 1030. DOI:10.1038/s41467-018-03492-6
|
| [65] |
Obexer R, Pott M, Zeymer C, et al. Efficient laboratory evolution of computationally designed enzymes with low starting activities using fluorescence-activated droplet sorting. Protein Eng Des Sel, 2017, 30(7): 531. DOI:10.1093/protein/gzx032
|
| [66] |
Qiao YX, Zhao XY, Zhu J, et al. Fluorescence-activated droplet sorting of lipolytic microorganisms using a compact optical system. Lab Chip, 2018, 18(1): 190-196. DOI:10.1039/C7LC00993C
|
| [67] |
Saleski TE, Kerner AR, Chung MT, et al. Syntrophic co-culture amplification of production phenotype for high-throughput screening of microbial strain libraries. Metab Eng, 2019, 54: 232-243. DOI:10.1016/j.ymben.2019.04.007
|
| [68] |
Siedler S, Khatri NK, Zsohár A, et al. Development of a bacterial biosensor for rapid screening of yeast p-coumaric acid production. ACS Synth Biol, 2017, 6(10): 1860-1869. DOI:10.1021/acssynbio.7b00009
|
| [69] |
Yu ZY, Boehm CR, Hibberd JM, et al. Droplet-based microfluidic analysis and screening of single plant cells. PLoS ONE, 2018, 13(5): e0196810. DOI:10.1371/journal.pone.0196810
|
| [70] |
Chiu FWY, Stavrakis S. High-throughput droplet-based microfluidics for directed evolution of enzymes. Electrophoresis, 2019, 40(21): 2860-2872. DOI:10.1002/elps.201900222
|
| [71] |
Autour A, Ryckelynck M. Ultrahigh-throughput improvement and discovery of enzymes using droplet-based microfluidic screening. Micromachines, 2017, 8(4): 128. DOI:10.3390/mi8040128
|
 2021, Vol. 37
2021, Vol. 37




