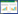中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈涛, 彭欣怡, 覃剑峰, 覃旭, 吴密, 黄锦媛, 黄显雅, 危丹妮, 王丽萍, 金刚
- Chen Tao, Peng Xinyi, Qin Jianfeng, Qin Xu, Wu Mi, Huang Jinyuan, Huang Xianya, Wei Danni, Wang Liping, Jin Gang
- 普通烟草NtODB基因的电子克隆、表达及生物信息学分析
- In silico cloning, expression and bioinformatics analysis of NtODB from common tobacco
- 生物工程学报, 2021, 37(8): 2836-2844
- Chinese Journal of Biotechnology, 2021, 37(8): 2836-2844
- 10.13345/j.cjb.200669
-
文章历史
- Received: October 19, 2020
- Accepted: January 20, 2021
- Published: February 7, 2021
生物有机体的DNA损伤修复途径包括碱基切除修复、核苷酸切除修复、错配修复、双链断裂修复等形式[1-2]。已有的研究表明,DNA错配修复缺陷对植物诱变育种具有重要意义。该系统可作为构建高效诱变体系的靶标,或直接利用于创制突变体[3]。而依赖于同源重组的双链断裂修复系统缺陷对植物诱变也具有潜在的应用价值,但目前对此类诱变体系的研究较少[4]。
辐射敏感蛋白52 (Radiation sensitive protein 52, RAD52) 在依赖同源性的DNA双链断裂修复和重组中起着至关重要的作用,这一点已通过酿酒酵母Saccharomyces cerevisiae的遗传研究得以证明[5-10]。rad52突变体对电离辐射极为敏感,在体细胞重组路径(包括单链退火、断裂诱导的修复以及霍利迪连结体中间物) 中表现出表型变异,并且出现减数分裂重组的异常[11]。RAD52蛋白主要参与修复DNA双链断裂,也参与修复其他类型的DNA损伤(如停滞的复制叉)[12-13]。
拟南芥Arabidopsis thaliana类RAD52蛋白细胞器DNA结合蛋白(Organellar DNA-binding protein,ODB) 在核和线粒体内室中的同源重组依赖的DNA修复中起作用[9, 14]。拟南芥中的ODB1和ODB2两个旁系同源基因通过可差异剪接编码4个开放阅读框(Open reading frame,ORF),每个ORF都特异性地定位于细胞核、线粒体或叶绿体。拟南芥ODB1A ORF为酵母rad52突变体提供了部分互补。ODB1的功能缺失会抑制由遗传毒性诱导的DNA断裂的同源重组修复,进而促进微同源重组[15]。与野生型相比,ODB1或ODB2功能缺陷的拟南芥突变体和RNAi转基因植株表现为育性降低,对丝裂霉素C的敏感性和染色体内重组水平降低[15-16]。
类RAD52蛋白ODB1蛋白在拟南芥线粒体中还参与剪接,并促进两个顺式剪接Ⅱ型内含子(nad1内含子2和nad2内含子1) 的剪切[17]。odb1突变体因ODB1蛋白的功能缺陷而超量积累不完全剪接的nad1和nad2转录本。类RAD52蛋白ODB1是通过第一步水解有效切除两个线粒体内含子所必需的[17]。
ODB1蛋白是植物线粒体中同源重组依赖性DNA修复的组成部分。在线粒体中,WHIRLY2 (WHY2) 和ODB1共同参与核蛋白复合物的构建,两种蛋白以DNA结合的方式免疫共沉淀,被认为是核体的重要组成部分[18-19]。染色质免疫共沉淀表明ODB1可结合整个线粒体基因组的序列。ODB1可促进互补DNA序列的退火,暗示了其具有类RAD52重组介体的功能。拟南芥odb1突变体在形态学上与野生型无明显区别,但在遗传毒性应激导致DNA损伤后,其线粒体同源重组活性降低。在相同的条件下,odb1突变体显示微同源介导重组产生的非正常修复旁路增加[12, 20]。将重组酶加载到单链DNA (Single-stranded DNA,ssDNA) 上以形成联会前纤丝需要替换ssDNA结合蛋白。ODB是酵母RAD52的同源物,可以分别在线粒体和叶绿体中执行此功能[14-15]。与RAD52一样,ODB1在体外促进互补ssDNA序列的杂交,从而支持了ODB1在重组中的介导作用[14, 19]。WHIRLY2蛋白还可以通过促进ssDNA上重组酶的加载来发挥介体的作用。然后,ODB1和WHIRLY2的功能可能是冗余的,也可能在不同替代重组路径中竞争性募集其他因子参与[14, 20-22]。
近年来随着植物基因组测序工作的不断推进,植物生物学的研究早已发展到了后基因组时代。电子克隆技术便是依托现有的EST数据库、基因组数据库等资源,采用同源性序列检索、聚类、重叠区域拼接组装等方法延伸已知EST序列,最终不可继续延伸后获得部分或全长cDNA的一种方法。电子克隆与传统的克隆方法相比,更具有操作简便、成本低效率高、针对性强等优势;但电子克隆结果仍需传统克隆的实验验证。由于ODB基因在植物诱变育种中具有潜在的利用价值,因此本研究拟通过电子克隆技术获取模式植物烟草NtODB基因的编码序列,通过生物信息学分析预测其编码蛋白的理化性质和结构特征,并研究该基因的时空表达和亚细胞定位特征。这将为该基因的生物学功能以及对植物的诱变分子机制研究提供参考。
1 材料与方法 1.1 供试材料本研究所用普通烟草Nicotiana tabacum var. Samsun和本生烟N. benthamiana Domin盆栽于广西亚热带作物研究所晾晒烟种质资源圃(东经108° 20′,北纬22° 53′)。普通烟草材料为广西晾晒烟地方品种“牛利烟”。植物组织材料经液氮速冻后,用干冰运送至-80 ℃超低温冰箱保存。
1.2 方法 1.2.1 总RNA提取与逆转录取1 g烟草组织放至研钵,倒入液氮研磨至粉末状。然后用RNAprep Pure多糖多酚植物总RNA提取试剂盒(TIANGEN) 提取RNA。琼脂糖凝胶电泳检测总RNA质量,然后用微量核酸蛋白测定仪检测RNA质量和浓度。再将烟草总RNA利用Oligo(dT)18和HiScript 1st Strand cDNA Synthesis试剂盒(Vazyme)进行逆转录。
1.2.2 烟草ODB基因的克隆根据已报道的拟南芥ODB1基因(At1g71310) 的氨基酸序列作为起始查询序列[9],通过BlastP同源比对检索烟草蛋白组数据库,选择与查询序列的相似度、覆盖度、e期望值最高的一条序列(GenBank登录号:XM_016606610.1)作为研究对象。PCR扩增烟草ODB基因的反应条件为95 ℃ 4 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min,32个循环;72 ℃ 10 min。1.0%琼脂糖凝胶电泳检测PCR产物,目标片段经切胶回收纯化后与pMD19-T载体连接,转化大肠杆菌Escherichia coli DH5α感受态。筛选阳性转化子送至苏州金唯智生物科技有限公司完成Sanger测序。
1.2.3 生物信息学分析分别利用在线软件ProtParam和ProtScale分析表达蛋白的基本理化指数、亲水性/疏水性;利用在线软件ProtComp v9.0和TMHMM工具分析该蛋白亚细胞定位和跨膜区域;利用NetPhos 3.1和TargetP 2.0在线软件分析磷酸化位点与信号肽;使用SWISS-MODEL在线软件分析结构域的高级结构[23]。
1.2.4 qRT-PCR分析提取普通烟草生殖生长期的茎、花瓣、叶、萼片的总RNA,并将提取的RNA反转录成cDNA。根据克隆测序获得的烟草NtODB基因设计qRT-PCR引物,以烟草Actin为内参基因(表 1)。使用LightCycler 480 Ⅱ荧光定量PCR仪(Roche) 检测目标基因的相对表达水平,每个样品设3次生物学重复,采用2-ΔΔCt 法计算差异表达[24]。
| Primer name | Sequence (5′-3′) | Purpose |
| ODB-F | ATGGCCTTTTCTTCATTGCG | Cloning |
| ODB-R | CTATGACATGTCCTCATGATAAAGATG | |
| pBI121-ODB-F | GACTCTAGAATGGCCTTTTCTTCATTGC | Vector construction |
| pBI121-ODB-R | TCCCCCGGGTGACATGTCCTCATGATAAAGAT | |
| Oligo(dT)18 | TTTTTTTTTTTTTTTTTT | Reverse transcription |
| ODB-RTF | TACACAGCAGAGCGAACTAAGGA | qRT-PCR |
| ODB-RTR | GCAGAGAAAGAGCACTGGCAA | |
| NtActin F1 | ACCTCTATGGCAACATTGTGCTCAG | Reference gene |
| NtActin R2 | CTGGGAGCCAAAGCGGTGATT | |
| The underlined nucleotides represent the introduced restriction endonuclease sites. | ||
设计构建NtODB亚细胞定位载体的引物pBI121-ODB-F/R (表 1),以pMD19-T-NtODB为模板,扩增NtODB完整的编码序列。扩增产物经Kpn Ⅰ和Xba Ⅰ双酶切后,连入同样被Xba Ⅰ与Kpn Ⅰ双酶切再回收纯化的空白表达载体pBI121-GFP,得到pBI121-NtODB/GFP融合表达载体。构建好的亚细胞定位载体转化农杆菌EHA105感受态细胞,挑取阳性转化子至含卡那霉素的YEB液体培养基中振荡培养12-16 h。制备农杆菌悬浮液,调整各菌液OD600为0.1-0.8,采用即时表达的方法,用成功转化目的质粒的农杆菌注入本生烟草叶片,暗培养48 h。切取侵染区域,撕表皮制片,利用激光共聚焦显微镜观察GFP荧光在烟草细胞中的分布。以不连接NtODB基因的pBI121-GFP空载体为对照。
2 结果与分析 2.1 NtODB基因的cDNA克隆利用拟南芥ODB1蛋白的氨基酸序列,在NCBI数据库中进行普通烟草蛋白序列的BlastP同源比对,相似度、覆盖度、e期望值最高的一条序列在GenBank登录号为XM_016606610.1。此cDNA的完整ORF框为第75-653 bp (图 1),起始和终止密码子分别是ATG和TGA,表达蛋白含有192个氨基酸。该序列与旱芹Apium graveolens、牵牛Ipomoea nil的DNA修复类RAD52蛋白具有较高的亲缘关系,序列同一性分别为69.59%和71.88%,且能比对到同源蛋白的C端和N端,命名基因为NtODB,预测蛋白为NtODB。
|
| 图 1 烟草NtODB基因的克隆 Fig. 1 Cloning of NtODB. M: DL2000 DNA marker; lane 1 and 2: PCR product of NtODB. |
| |
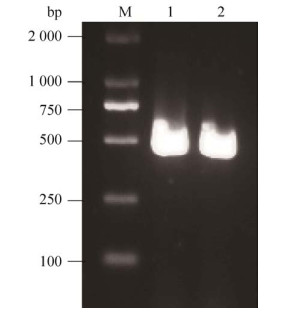
使用ProtParam tool预测NtODB蛋白的理化参数,该蛋白质分子式、分子量和理论等电点分别为C943H1479N269O295S8、21.56 kDa和8.38,是碱性蛋白质;脂肪系数、负电荷残基数(天冬氨酸+谷氨酸) 和正电荷残基数(精氨酸+赖氨酸) 分别是67.50、25.00和27.00;不稳定系数和平均总亲水性分别是47.91和-0.596,属于不稳定亲水蛋白。预测NtODB蛋白含有20种氨基酸,含量最多的是丝氨酸(Ser),占15.1%。利用ProtScale对烟草NtODB蛋白的一级结构进行亲水和疏水性分析,该多肽链的第135位酪氨酸(Tyr) 和第42位的赖氨酸(Lys) 分别具有最高(1.478) 和最低(-3.722) 分值,前者疏水性最强,后者亲水性最强(图 2)。
|
| 图 2 烟草ODB蛋白疏水性/亲水性预测 Fig. 2 Prediction of the hydrophilicity/hydrophobicity of NtODB protein in tobacco. |
| |
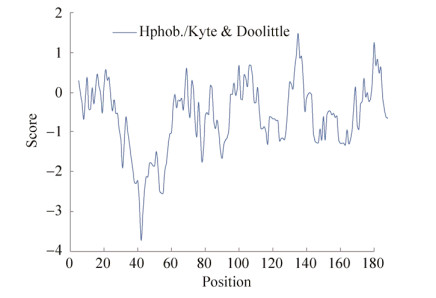
利用ProtComp 9.0软件预测NtODB蛋白的亚细胞定位,NtODB蛋白质在细胞外(2.22)、叶绿体(2.19) 和内质网(2.11) 中具有较高整体分数;而根据五聚体算法,该蛋白在线粒体(3.62)中分数较高(表 2)。据此预测NtODB蛋白可能分布于细胞器。利用TMHMM v.2.0软件在线分析NtODB蛋白跨膜区,该蛋白没有明显的跨膜区,不属于膜结合蛋白(图 3)。
| Location | LocDB | PotLocDB | Neural nets | Pentamers | Integral |
| Nuclear | 0 | 0 | 0 | 0 | 0.11 |
| Golgi | 0 | 0 | 0.11 | 0.25 | 0.41 |
| Extracellular | 0 | 0 | 0.96 | 0.02 | 2.22 |
| Cytoplasmic | 0 | 0 | 0 | 0.65 | 0 |
| Peroxisomal | 0 | 0 | 0.96 | 0.22 | 2.03 |
| Plasma membrane | 0 | 0 | 0.96 | 0 | 0 |
| Vacuolar | 0 | 0 | 0 | 0 | 0 |
| Mitochondrial | 0 | 0 | 0 | 3.62 | 0.94 |
| Endoplasmic reticulum | 0 | 0 | 0 | 0.05 | 2.11 |
| Chloroplast | 0 | 0 | 0 | 0.33 | 2.19 |
|
| 图 3 NtODB蛋白的跨膜区分析 Fig. 3 Prediction of the transmembrane regions of NtODB protein. |
| |
蛋白质磷酸化处于信号传递链的终端,可通过对转录因子的磷酸化达到调节基因活性的目的。利用NetPhos 3.1在线分析NtODB磷酸化位点,此蛋白含有的Ser、Thr和Tyr磷酸化位点数目分别是18、4、4 (图 4)。经TargetP 2.0分析,NtODB为线粒体转运蛋白的可能性是93.92%,切除位点在第24-25个氨基酸残基之间。暗示NtODB蛋白的细胞器定位性质。
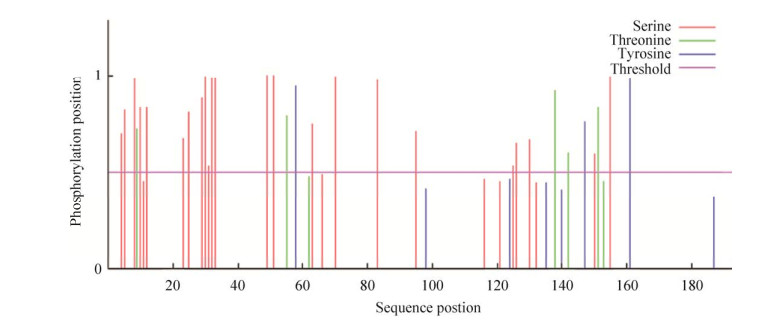
|
| 图 4 NtODB蛋白的磷酸化位点预测 Fig. 4 Prediction of the phosphorylation sites of NtODB protein. |
| |
使用在线软件CD-Search对NtODB蛋白进行保守结构域分析(图 5A),该蛋白第70-185位氨基酸残基之间存在高度保守的RAD52结构域,表明本研究所克隆NtODB基因的编码蛋白属于DNA修复蛋白RAD52家族成员。使用SOPMA分析NtODB蛋白的二级结构,包括35.94%的α螺旋、16.67%的延伸链、5.73%的β-折叠和41.67%的无规则卷曲序列(图 5B)。利用SWISS-MODEL预测该蛋白的三级结构,空间结构主要由α螺旋和无规则卷曲构成(图 5C)。
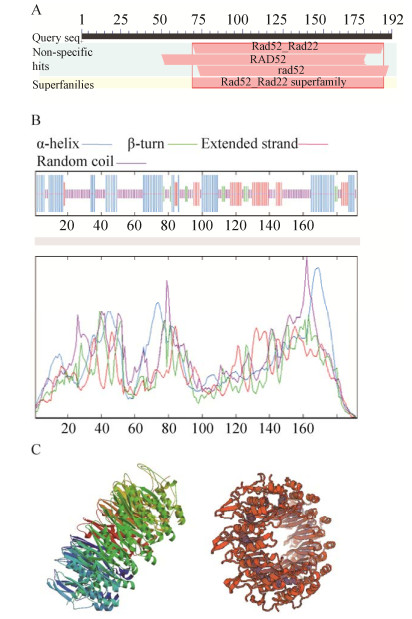
|
| 图 5 烟草NtODB蛋白保守结构域和高级结构预测 Fig. 5 Prediction of the conservative region and advanced structure of NtODB protein. (A) Conserved domain. (B) Secondary structure. (C) Tertiary structure. |
| |
从盛花期普通烟草的茎、叶、花瓣和萼片等器官中提取总RNA。以普通烟草NtActin基因为内参基因进行qRT-PCR检测NtODB基因的相对表达水平。结果表明,NtODB在盛花期普通烟草的不同器官中虽呈现组成型表达特征,但其相对表达量在不同器官之间差异显著。在叶片中表达丰度最高,茎和萼片表达丰度次之,在花瓣中表达丰度最低(图 6)。
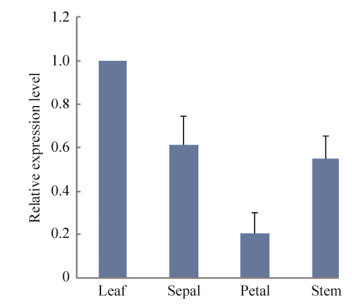
|
| 图 6 NtODB在烟草中的时空表达模式 Fig. 6 Temporal and spatial expression pattern of NtODB in tobacco. |
| |
为了进一步研究NtODB蛋白的亚细胞定位情况,以空载体为对照,在激光共聚焦显微镜下观察绿色荧光蛋白的分布,结果发现,转pBI121-GFP的荧光信号分布于整个细胞中;而NtODB-GFP融合蛋白的绿色荧光信号定位于叶绿体和细胞膜上(图 7)。
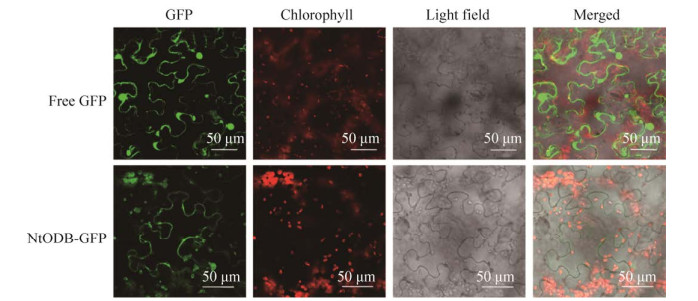
|
| 图 7 NtODB-GFP在本生烟表皮细胞中的定位 Fig. 7 Subcellular localization of NtODB fused with GFP in the epidermal cells of N. benthamiana. |
| |
发掘依赖于同源重组的双链断裂修复系统缺陷的相关基因并进行功能研究是对其开展植物诱变育种应用的前提。本研究表明,NtODB在烟草不同器官中呈组成型表达。但在不同器官中的相对表达量存在一定差异,在生殖器官花瓣中的表达丰度低于其他器官。Samach等通过亚细胞定位实验,发现拟南芥ODB1蛋白在叶绿体、细胞核和叶绿体中均有定位[15]。而其他植物ODB同源蛋白的亚细胞定位结论未见报道。亚细胞定位研究表明,烟草NtODB在叶绿体和细胞膜中表达。这与基于ProtComp v.9.0软件预测的NtODB亚细胞定位结果一致。烟草NtODB在叶绿体中表达的特征与拟南芥通过差异剪接编码的开放阅读框之一ODB2B的叶绿体定位具有一致性[15],暗示了本研究所克隆的烟草NtODB与拟南芥ODB2B在叶绿体中可能执行了相似的生物学功能。但在拟南芥中未见ODB1和ODB2细胞膜的定位[15]。
基于why2-1突变体在环丙沙星处理后的亚化学计量转换效应以及WHIRLY2和ODB1共结合这两点原因,初步判断ODB1也与细胞质异质性的亚化学计量转换密切相关,这仍有待于实验论证。
在拟南芥中,和ODB1共同参与线粒体大型核蛋白复合物组成的WHIRLY2已被证实与DNA胁迫诱变有关[26]。WHY蛋白与ssDNA的结合有利于DNA双链断裂的精确修复。对拟南芥why2-1突变体的研究表明,环丙沙星胁迫处理条件下的植株线粒体中含有以亚化学计量水平存在的异型DNA分子。在环丙沙星处理后,微观同源介导的DNA重排分子形式以亚化学计量在野生型、why2-1突变体以及why1 why3双突变体中积累,并且一些重排DNA以亚基因组长度分子形式存在[25-26]。并且在环丙沙星处理条件下,why2突变体比野生型材料更容易在线粒体基因组中积累DNA重排分子。
ODB1是植物线粒体中同源重组依赖性DNA修复的组成部分[14]。在拟南芥的染色质免疫共沉淀研究表明,ODB1可结合于整个线粒体基因组的序列。ODB1促进互补DNA序列的退火,提示具有类似RAD52重组介体的功能。拟南芥odb1突变体在形态学上与野生型无明显区别,但在遗传毒性应激导致DNA损伤后,显示出降低的线粒体同源重组活性。在相同的条件下,odb1突变体展示了由微同源介导重组产生的不合理修复旁路的增加[14]。
本研究克隆的烟草NtODB更近似于拟南芥ODB2,因为亚细胞定位结果展示了NtODB具有与ODB2相同的叶绿体定位特征。拟南芥叶绿体定位的ODB2可能参与到叶绿体DNA的修复路径[15]。推测与已在拟南芥中证实的ODB1和WHIRLY2协同参与线粒体DNA的修复机制类似,NtODB与具有叶绿体定位特征的WHY1及WHY3直系同源基因共同参与叶绿体DNA的修复。拟南芥ODB1或ODB2功能缺陷出现的表型变化,如育性降低、对丝裂霉素C的敏感性和染色体内重组水平降低等,可能均与这一类DNA修复机制有关[14-15]。这也暗示了NtODB在叶绿体DNA同源重组修复中的作用,其功能缺陷可能会导致叶绿体DNA变异,对植物DNA诱变具有潜在的研究意义[20, 25-26]。
本研究为进一步通过转化验证NtODB基因的生物学功能提供了可能,丰富了ODB基因在烟草中的功能实验证据。
| [1] |
Bray CM, West CE. DNA repair mechanisms in plants: crucial sensors and effectors for the maintenance of genome integrity. New Phytol, 2005, 168(3): 511-528. DOI:10.1111/j.1469-8137.2005.01548.x
|
| [2] |
董隽, 张天, 文碧秀. DNA双链断裂与同源重组修复的研究进展. 中国医学创新, 2015, 12(3): 143-146. Dong J, Zhang T, Wen BX. Advance in research of homologous recombination repair in DNA double strands breakage. Med Innovat China, 2015, 12(3): 143-146 (in Chinese). DOI:10.3969/j.issn.1674-4985.2015.03.049 |
| [3] |
崔海瑞, 宋悦, 袁兵, 等. 植物DNA错配修复缺陷及其对诱变育种的意义. 核农学报, 2014, 28(9): 1606-1614. Cui HR, Song Y, Yuan B, et al. Plant mismatch repair deficiency and its significance for mutation breeding. J Nucl Agric Sci, 2014, 28(9): 1606-1614 (in Chinese). |
| [4] |
Krejci L, Altmannova V, Spirek M, et al. Homologous recombination and its regulation. Nucleic Acids Res, 2012, 40(13): 5795-5818. DOI:10.1093/nar/gks270
|
| [5] |
Wright WD, Shah SS, Heyer WD. Homologous recombination and the repair of DNA double-strand breaks. J Biol Chem, 2018, 293(27): 10524-10535. DOI:10.1074/jbc.TM118.000372
|
| [6] |
Arai N, Kagawa W, Saito K, et al. Vital roles of the second DNA-binding site of Rad52 protein in yeast homologous recombination. J Biol Chem, 2011, 286(20): 17607-17617. DOI:10.1074/jbc.M110.216739
|
| [7] |
Shi I, Hallwyl SCL, Seong C, et al. Role of the Rad52 amino-terminal DNA binding activity in DNA strand capture in homologous recombination. J Biol Chem, 2009, 284(48): 33275-33284. DOI:10.1074/jbc.M109.057752
|
| [8] |
Xue CY, Greene EC. New roles for RAD52 in DNA repair. Cell Res, 2018, 28(12): 1127-1128. DOI:10.1038/s41422-018-0105-8
|
| [9] |
Piazza A, Heyer WD. Homologous recombination and the formation of complex genomic rearrangements. Trends Cell Biol, 2019, 29(2): 135-149. DOI:10.1016/j.tcb.2018.10.006
|
| [10] |
Dhingra N, Zhao XL. Intricate SUMO-based control of the homologous recombination machinery. Genes Dev, 2019, 33(19/20): 1346-1354.
|
| [11] |
Symington LS. Role of RAD52 epistasis group genes in homologous recombination and double-strand break repair. Microbiol Mol Biol Rev, 2002, 66(4): 630-670. DOI:10.1128/MMBR.66.4.630-670.2002
|
| [12] |
Gangavarapu V, Prakash S, Prakash L. Requirement of RAD52 group genes for postreplication repair of UV-damaged DNA in Saccharomyces cerevisiae. Mol Cell Biol, 2007, 27(21): 7758-7764. DOI:10.1128/MCB.01331-07
|
| [13] |
Shinohara A, Ogawa T. Stimulation by Rad52 of yeast Rad51-mediated recombination. Nature, 1998, 391(6665): 404-407. DOI:10.1038/34943
|
| [14] |
Janicka S, Kühn K, Le Ret M, et al. A RAD52-like single-stranded DNA binding protein affects mitochondrial DNA repair by recombination. Plant J, 2012, 72(3): 423-435. DOI:10.1111/j.1365-313X.2012.05097.x
|
| [15] |
Samach A, Melamed-Bessudo C, Avivi-Ragolski N, et al. Identification of plant RAD52 homologs and characterization of the Arabidopsis thaliana RAD52-like genes. Plant Cell, 2011, 23(12): 4266-4279. DOI:10.1105/tpc.111.091744
|
| [16] |
Samach A, Gurevich V, Avivi-Ragolsky N, et al. The effects of AtRad52 over-expression on homologous recombination in Arabidopsis. Plant J, 2018, 95(1): 30-40. DOI:10.1111/tpj.13927
|
| [17] |
Gualberto JM, Le Ret M, Beator B, et al. The RAD52-like protein ODB1 is required for the efficient excision of two mitochondrial introns spliced via first-step hydrolysis. Nucleic Acids Res, 2015, 43(13): 6500-6510. DOI:10.1093/nar/gkv540
|
| [18] |
Gualberto JM, Kühn K. DNA-binding proteins in plant mitochondria: implications for transcription. Mitochondrion, 2014, 19 Pt B: 323-328.
|
| [19] |
Pfalz J, Pfannschmidt T. Essential nucleoid proteins in early chloroplast development. Trends Plant Sci, 2013, 18(4): 186-194. DOI:10.1016/j.tplants.2012.11.003
|
| [20] |
Cappadocia L, Maréchal A, Parent JS, et al. Crystal structures of DNA-Whirly complexes and their role in Arabidopsis organelle genome repair. Plant Cell, 2010, 22(6): 1849-1867. DOI:10.1105/tpc.109.071399
|
| [21] |
Wild P, Susperregui A, Piazza I, et al. Network rewiring of homologous recombination enzymes during mitotic proliferation and meiosis. Mol Cell, 2019, 75(4): 859-874. DOI:10.1016/j.molcel.2019.06.022
|
| [22] |
Chevigny N, Schatz-Daas D, Lotfi F, et al. DNA repair and the stability of the plant mitochondrial genome. Int J Mol Sci, 2020, 21(1): 328. DOI:10.3390/ijms21010328
|
| [23] |
杜尚广, 余波. 木薯HSP21基因的电子克隆与生物信息学分析. 生物工程学报, 2020, 36(7): 1422-1430. Du SG, Yu B. In silico cloning and bioinformatics analysis of HSP21 in Manihot esculenta. Chin J Biotech, 2020, 36(7): 1422-1430 (in Chinese). |
| [24] |
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262
|
| [25] |
Krause K, Kilbienski I, Mulisch M, et al. DNA-binding proteins of the Whirly family in Arabidopsis thaliana are targeted to the organelles. FEBS Lett, 2005, 579(17): 3707-3712. DOI:10.1016/j.febslet.2005.05.059
|
| [26] |
Gualberto JM, Newton KJ. Plant mitochondrial genomes: dynamics and mechanisms of mutation. Annu Rev Plant Biol, 2017, 68: 225-252. DOI:10.1146/annurev-arplant-043015-112232
|
 2021, Vol. 37
2021, Vol. 37