多组学技术及其在生命科学研究中应用概述
刘景芳
,
李维林
,
王莉
,
李娟
,
李二伟
,
罗元明
生物工程学报  2022, Vol. 38 2022, Vol. 38 Issue (10): 3581-3593 Issue (10): 3581-3593 |

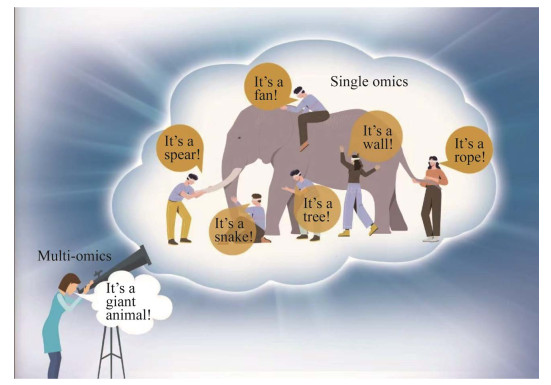
随着1986年美国遗传学家Thomas H. Roderick首次提出基因组学概念以来,基因组学得到了飞速发展,并带动了表观基因组学、转录组学、蛋白质组学、代谢组学等后基因组学时代相关组学的蓬勃发展。然而,单一组学具有明显的局限性[1],只能从单一层面去进行研究,即基因组学主要从DNA角度,转录组从RNA的角度,蛋白质组学从蛋白质的角度,代谢组学从代谢物的角度等去研究细胞或者生物体,不能从整体上揭示生物体功能,以及阐明生物和环境因素的关系。为了形象说明单组学和多组学的关系,并阐明多组学在生命科学研究中的系统性和重要性,在此借用“盲人摸象”的成语来加以说明(图 1)。单组学就像盲人摸象,摸到鼻子认为是蛇、摸到腿认为是大树,摸到象牙认为是标枪,摸到尾巴认为是绳子,摸到耳朵认为是扇子,而多组学就像人拿着放大镜去作全面整体的观察和分析,从而得出正确的结论为“大型动物-大象”。
|
| 图 1 以盲人摸象比喻单组学与多组学关系 Fig. 1 Illustration of the relationship between single-omics and multi-omics. |
| |
当利用多组学进行人体疾病研究时,为了判断人体究竟是健康状态还是疾病状态,仅从单组学数据分析是不够的,而是要从基因组、表观基因组、转录组、蛋白质组及代谢组等多维组学层面进行综合分析,才能得出正确结论(图 2)[2]。因为单组学只是利用单一技术,只能阐明生命体系中单一分子的单一功能,而生命体中,从编码蛋白的DNA序列,到具有功能的蛋白质及具有活性的代谢物,是由多个层面的调节机理共同完成的,如转录调节、翻译调节、RNA/多肽降解、翻译后修饰及差别运输等。因此,只是强调单组学层面的功能会缺失重要的信息,特别是不同组学之间的交叉和互补信息,比如,通过基因组层面的全基因组关联分析(genome-wide association study, GWAS) 可以成功鉴定人类疾病的遗传易感性基因,却不能捕获随着时间的进展,内部个体间的功能差异信息,以及这些信息和疾病风险之间的关系。然而从蛋白质组学层面,蛋白质组数据却能弥补基因组学所缺失的关键功能信息,即不同样本中蛋白及多肽信息,机体的生物学功能就是通过这些功能性的蛋白或者多肽来完成的[2],因此,通过蛋白质组学来补充和完善基因组学不能解决关于疾病的生物学机理等问题,这就是多组学研究带来的关于解决生命科学问题的新思路。
多组学是一种全新的系统研究生物学的方法和技术,以无偏差的方式去整合基因组学、表观基因组学、转录组学、蛋白质组学及代谢组学等,从而系统解析复杂生命系统的机理和表型[1, 3]。本文简要综述了各单组学的概况、多组学技术平台、多组学应用及展望,以期为从事多组学相关研究的科研工作者提供参考。
1 单组学简介 1.1 基因组学基因组学是对生物体所有基因进行集体表征、定量研究及不同基因组比较研究的一门交叉生物学学科。基因组学主要研究基因组的结构、功能、进化、定位和编辑等,以及它们对生物体的影响。主要工作包括基因组测序、插入、缺失、单核苷酸多态性、基因拷贝数变化等研究。基因组的变化和疾病发生直接相关[4]。
1.2 表观基因组学表观基因组学是研究表观基因组,即生物体中所有表观修饰的遗传物质的学科[5]。两个最具特征的表观遗传修饰是DNA甲基化和组蛋白修饰,通过修饰调控基因表达活性或者突变[6]。DNA甲基化随着年龄而变化,因此被用作反映生物学年龄与衰老的标志物[7-8],而且甲基化异常和肿瘤发生直接相关[9]。
1.3 转录组学转录组的概念由Charles Auffray在1999年首次提出[10]。转录组学为研究转录组的学科,主要是测定某一时期生物体在特定条件下基因的表达谱,即对RNA进行鉴定和定量,表达谱能反映机体的生理状态,如通过分析差别表达情况,去判定机体癌症相关的病变[11]。
1.4 蛋白质组学蛋白质组学概念由Marc Wilkins在1994年首次提出,主要研究细胞、组织或生物体蛋白质组成及其变化规律。生物质谱为探索蛋白质组结构和功能的关键工具[12],主要包括蛋白鉴定、蛋白翻译后修饰鉴定、蛋白复合物鉴定、比较蛋白质组学、修饰组学、糖组学及靶向蛋白质组学研究等。目前,蛋白质组学技术广泛应用于生命科学研究的各个领域,如用于药物靶标鉴定[13]、抗肿瘤药物设计[14]、合成生物学领域酶动力学研究[15]等。
1.5 代谢组学代谢组学概念最初是由英国伦敦大学的Nicholson教授于1999年首次提出,主要研究生命个体对外源性的刺激、环境变化或遗传修饰所作出的所有代谢应答,并绘制出整体的代谢物动态变化谱图[16]。代谢组学分为非靶向代谢组学及靶向代谢组学(包括脂质组学),常用的分析技术包括气相色谱质谱联用法(gas chromatography and mass spectrometry, GC-MS)、液相色谱质谱联用法(liquid chromatography and mass spectrometry, LC-MS) 及核磁共振法(nuclear magnetic resonance, NMR) 等,主要数据分析方法包括主成分分析(principal component analysis, PCA)、偏最小二乘法判别分析(partial least squares-discrimination analysis, PLS-DA) 及正交偏最小二乘法判别分析(orthogonal partial least squares-discrimination analysis, OPLS-DA) 等多元模式识别方法。目前,代谢组学广泛应用于阐明新冠肺炎病毒致病机理[17]、个性化医疗[18]、标志物发现[19]及合成生物学[20]等研究。
2 多组学技术平台由于最新高通量测序技术的发展[21],超高分辨质谱仪的应用[22],新的统计学工具和计算方法的发展[23],以及组学数据库的不断丰富[24],加快了多组学技术的快速发展,可真正实现从系统生物学角度去解析生物体功能和机理。
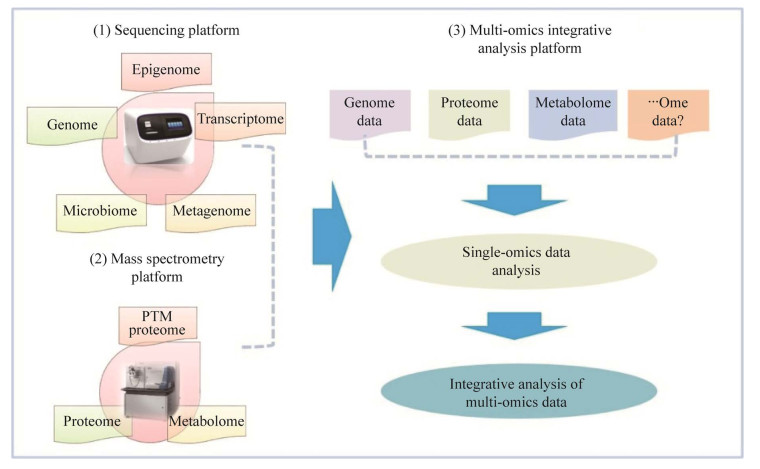
多组学技术平台主要包括3个部分,即:(1) 基于测序技术的测序平台,主要支撑的组学包括基因组学、表观基因组学、转录组及宏基因组等;(2) 基于质谱技术的质谱平台,支撑的组学包括蛋白质组学、代谢组学及修饰组学等;(3) 基于测序数据及质谱数据的多组学整合分析平台(图 3)[25-26]。多组学技术平台除了核心设备测序仪及质谱仪外,还应包含其他配套设备,如样品制备、分离纯化等设备,以及用于代谢组学研究的核磁共振波谱仪[27],用于结构蛋白质组学领域分析天然蛋白结构的冷冻电镜[28]等。
|
| 图 3 多组学技术平台的组成示意图 Fig. 3 Technology platforms for multi-omics. (1) Sequencing platform. (2) Mass spectrometry platform. (3) Integrated platform for multi-omics data analysis. |
| |
测序技术平台支撑的组学研究包括基因组、表观基因组(epigenome)、转录组及宏基因组(metagenome) 等。具体包括DNA测序[29]、RNA测序(RNA-seq)[30]、DNA-蛋白相互作用分析(ChIP-seq)[31]及甲基化测序(methyl-seq)[32]等。
最早的测序技术为第一代测序,又称Sanger双脱氧核苷酸测序[33];Sanger测序法通过引入ddNTP,第一次为研究者开启了深入了解遗传密码的大门。其原理是双脱氧核苷酸缺少3′-OH基团,不能与下一个dNTP连接。当添加放射性同位素标记的引物时,在聚合酶作用下ddNTP被合成到链上,但其后的核苷酸无法连接,合成反应也随之终止,后续再根据各个合成片段的大小不同进行聚丙烯酰胺凝胶电泳分离,放射自显影后,便可根据片段大小排序及相应泳道的末端核苷酸信息读出整个片段的序列信息。通过调节加入的dNTP和ddNTP的相对量即可获得较长或较短的末端终止片段。
第二代测序技术,又称高通量测序(next generation sequencing, NGS),为目前在全球占据主要市场的测序技术[34-35],该技术基于大规模平行测序(massive parallel analysis, MPS),可同时完成测序模板互补链的合成和序列数据的获取。相比第一代测序技术,其成本花费更低,一次检测的量更多。第二代测序技术主要有5种方法,即454焦磷酸测序技术、Solexa测序技术、SOLiD测序技术、Complete Genomics测序方法及半导体(ion torrent) 测序技术。以Illumina Solexa测序平台为例,工作过程可分为4步:(1) 文库准备:提取的DNA和cDNA通过物理或其他方法随机切割成相同大小的片段,并在5′和3′末端添加接头(adapter)。该接头主要用于提高PCR效率,在测序时提供标签索引信息(barcode/index)。(2) 生成簇:通过桥式PCR,单片段序列被大量扩增成簇。(3) 测序:每个片段基于边合成边测序(sequencing by synthesis, SBS) 原理,由计算机读取read。(4) 数据比对和分析。
第三代测序,又称单分子测序(single- molecule sequencing, SMS),在测序时,不需要经过PCR扩增,实现了对每一条DNA分子的单独测序。第三代测序具有明显优点[36],如样品制备简单,读长比较长,可达100 kb,可直接测序RNA等。
第四代测序技术,又称单分子纳米孔测序(nanopore sequencing),是最近几年兴起的新一代测序技术,在测序准确性、测序长度和通量上都有较大突破[37],该方法数据分析相对简单,成本较低,广泛应用于基因组组装、天然RNA全长测序[38]及碱基修饰分析[39]等。
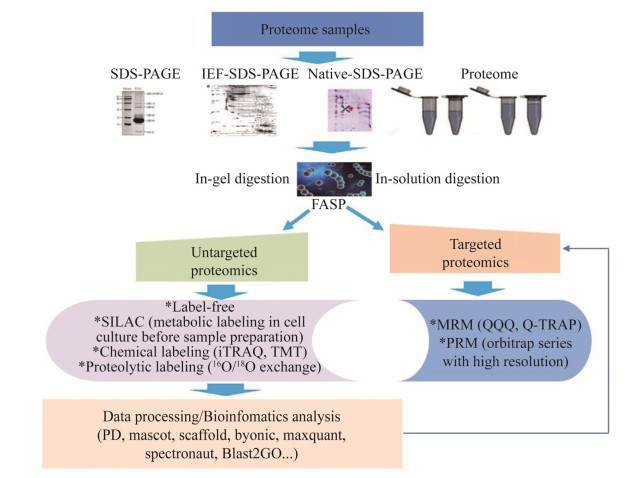
2.2 质谱平台基于质谱的技术平台主要支撑蛋白质组学及代谢组学,以及相关的组学,如糖蛋白质组学、糖组学、修饰组学、脂质组学等。目前,基于质谱技术的蛋白质组学平台的主要方法分为2类,即非靶向蛋白质组学及靶向蛋白质组学,具体技术路线如图 4所示。
|
| 图 4 基于质谱的蛋白质组学技术平台流程图 Fig. 4 Flow-chart for mass spectrometry-based proteomics platform. [L2] |
| |
如图 4所示,蛋白质组样品首先经过SDS-PAGE纯化或者通过2DE纯化,然后经过胶内酶切(in-gel digestion);或者蛋白质组样品,经过溶液内酶切(in-solution digestion) 或者膜上酶切(filter-aided sample preparation, FASP)。酶切后产生的多肽样品,用质谱进行分析。非靶向蛋白质组学分析的主要目标是对蛋白质组进行鉴定或差别表达蛋白分析,采用的技术路线包括:(1) 对酶切肽段不进行标记的Label-free技术路线[40];(2) 采用稳定同位素进行代谢标记的SILAC技术路线[41],即在细胞培养时利用13C和15N标记的氨基酸(Arg及Lys) 对合成蛋白质进行标记,然后再进行样品制备及后续的差别表达蛋白分析;(3) 酶促标记,即对蛋白进行酶切的同时进行稳定同位素标记,如18O标记[42];(4) 对酶切肽段用稳定同位素进行的化学标记,如采用iTRAQ[43]及TMT标记[44]。在非标记或者稳定同位素标记的非靶向蛋白组学方案中,蛋白组经质谱分析生成的原始数据(raw file),需要进一步数据库搜索进行蛋白鉴定(如用软件PD、Mascot、Byonics) 及定量(采用软件Scaffold、Maxquant、Spectronaut),以及对差别表达蛋白进行后续功能富集和功能注释(如Blast2GO软件)。在靶向蛋白质组学分析中,主要目标是对已知的目标蛋白进行定量分析,或对在非靶向蛋白质组分析中产生的差别表达蛋白进行确证[45]。对于目标蛋白酶切肽段的质谱分析,一般采用多反应监测(multiple reaction monitoring, MRM) 采集模式[46]。如果在Orbitrap等高分辨质谱仪上,则采用平行反应监测(parallel reaction monitoring, PRM) 采集模式[47]。采集后的原始质谱数据,需要用软件(如Maxquant、Skyline) 对目标蛋白进行进一步定量和统计学分析。
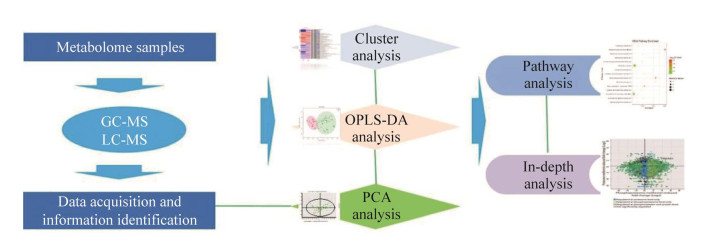
基于质谱的代谢组学技术平台主要支撑非靶向代谢组学及靶向代谢组学(含脂质组学)。代谢组学平台的技术路线如图 5所示。
|
| 图 5 基于质谱的代谢组学技术平台技术路线 Fig. 5 Flow-chart of mass spectrometry-based metabolomics platform. |
| |
如图 5所示,代谢组分析分为两个方面,即非靶向代谢组学和靶向代谢组学,其中,非靶向代谢组包括代谢组鉴定和比较代谢组分析。靶向代谢组主要对已经通过比较代谢组筛选出的差异代谢物进行确证和定量筛选。通过质谱分析得到的代谢组原始数据,需要进行数据库搜索,相关的PCA分析、OPLS-OA分析、cluster分析、pathway分析及其他功能相关的深度分析等。
2.3 多组学整合分析平台多组学整合分析平台是整个多组学技术平台的核心子平台,是将从测序平台和质谱平台等获得的单组学数据,进行同步交叉整合分析。代表性的多组学整合分析平台如Galaxy[48]和O-Miner[49]。典型的多组学整合分析平台(软件包) 应满足3个标准[50]:(1) 可同步处理多组学数据,而不是先后处理;(2) 必须整合分析2个及以上层次的组学数据;(3) 必须以软件包的方式处理任意格式的数据。
多组学整合分析的策略就是功能注释[51],即针对生物体的特定生物学功能(如脂肪酸代谢、特定疾病指标等),对来自基因组、转录组、蛋白质组和代谢组等不同组学层次的批量数据在同一整合分析软件中进行归一化处理、比较分析和相关性分析等,建立不同层次分子间数据相关性;同时结合Gene Ontology (GO) 功能分析、代谢通路富集、分子互作等生物功能分析,系统全面地解析生物分子功能和调控机制。比如,在对甲状腺乳头状癌(papillary thyroid carcinoma, PTC) 患者的脂肪酸代谢进行多组学分析时[52],将蛋白质组数据、代谢组数据及脂质组数据输入Ingenuity Pathway Analysis (IPA) 整合分析软件,采用P值和Z-score值对多组学数据进行功能富集筛选。多组学数据整合分析结果表明,在PTC患者实验组中,和脂肪酸利用相关的氧化磷酸化途径、TCA循环途径Ⅱ及脂肪酸-氧化途径显著激活,表明PTC患者体内的脂肪酸运输和氧化比较活跃。
理想的多组学整合分析是整合来自同一组样本的多组学数据,然而很难实现,如果采用不同样本组的数据,就需要对这些数据进行标准化和统计学处理[2]。目前,满足多组学整合分析数据平台标准的公共平台资源越来越多,如癌症基因组数据库(the cancer genome atlas, TCGA) 已经可以对30多种人类肿瘤进行整合分析[53]。组学发现索引数据库(omics discovery index) (https://www.omicsdi.org/),由11个统一格式的数据库合并而成,包含基因组、转录组、蛋白质组及代谢组数据等。常见代表性的多组学数据库如表 1所示[50]。代表性的多组学整合分析机器学习方法和算法如iOmicsPASS[23]、AMARETTO[54]、DrugComboExplorer[55]、Deep Network[56]、Clusternomics[57]、MixOmics[58]。
| Data repository | Web link | Disease | Types of multi-omics data available |
| The cancer genome atlas (TCGA) | https://cancergenome.nih.gov/ | Cancer | RNA-seq, DNA-seq, miRNA-seq, SNV, CNV, DNA methylation, and RPPA |
| Clinical proteomic tumor analysis | https://cptac-data-portal.georgetown.edu/cptacPublic/ | Cancer | Proteomics data corresponding to TCGA cohorts |
| International cancer genomics consortium (ICGC) | https://icgc.org/ | Cancer | Whole genome sequencing, genomic variations data (somatic and germline mutation) |
| Cancer cell line encyclopedia (CCLE) | https://portals.broadinstitute.org/ccle | Cancer cell line | Gene expression, copy number, and sequencing data; pharmacological profiles of 24 anticancer drugs |
| Molecular taxonomy of breast cancer international consortium (METABRIC) | http://molonc.bccrc.ca/aparicio-lab/research/metabric/ | Breast cancer | Clinical traits, gene expression, SNP, and CNV |
| TARGET | https://ocg.cancer.gov/programs/target | Pediatric cancers | Gene expression, miRNA expression, copy number, and sequencing data |
| Omics discovery index | https://www.omicsdi.org | Consolidated data sets from 11 repositories in a uniform framework | Genomics, transcriptomics, proteomics, and metabolomics |
| CNV: copy number variation; miRNA: microRNA; RPPA: reverse phase protein array; SNP: single-nucleotide polymorphism; SNV: single-nucleotide variant. | |||
目前,多组学技术已经成为生命科学研究热点,以multi-omics为关键词在NCBI上进行检索,从2014年到2021年,发表文章的数量逐年增加,特别是2019年以来,文章数量成倍增加(图 6)。

|
| 图 6 以multi-omics为关键词在NCBI网站上检索到的2014−2021年的文章数量 Fig. 6 Number of multi-omics articles that can be found at NCBI website from 2014‒2021. |
| |
多组学技术的特点是将基因组、表观基因组、转录组、蛋白组及代谢组等多维层次的信息进行有机整合,构建基因的调控网络,全面探索和深层次理解各生物分子之间的调控及因果关系,从而正确解析生命体的生物功能和生理机制。多组学技术在生命科学多个领域的应用日趋广泛,比如在前列腺癌的研究中,通过整合转录组和代谢组数据,揭示引起前列腺癌增生的主要代谢途径和潜在的生物标记分子[59];在新冠机理研究中,通过整合新冠病毒SARS-CoV-2数据库,新冠患者的基因组数据、转录组数据、微生物组数据及药物治疗等数据信息,探索新冠致病机理[60];通过整合转录组、宏基因组、蛋白质组及代谢组数据进行衰老机理研究,评估生物年龄及筛选衰老相关的生物标志物等[61];在合成生物学领域,通过结合基因组测序和代谢物图谱技术,发现新的天然活性代谢物[62-63],通过对转录组、蛋白质组及代谢组数据进行整合分析,以提高异戊烯醇、苧烯、甜没药烯等生物质燃料的产量[64];在微生物多组学领域,通过对比较基因组、比较转录组、比较蛋白质组及表型组的数据的整合交叉分析,去鉴别和表征具有不同表型的大肠杆菌菌株B及K12[65],通过整合基因组、宏基因组、宏转录组及宏蛋白组信息,重构代谢途径,研究微生物群落内的合作和拮抗关系等指导微生物分离培养[66]。
4 总结与展望随着高通量测序技术、高分辨质谱技术及数据整合分析技术的发展,推动了以多组学为特征的系统生物学研究新突破,颠覆了生命科学研究的传统范式,即从传统的各自分离的单组学层面的生物化学反应和代谢途径模式,向对整体细胞系统进行大规模研究的多组学整合分析模式转变。因此,多组学数据整合分析是从系统生物学角度去全面解析生物体功能的必要手段。
随着多组学技术的发展和日益成熟,科学家们便可以从系统生物学角度去解决生物医学,合成生物学等领域的关键技术问题和关键科学问题。下面以生命科学领域的两个热点前沿领域,即合成生物学及单细胞多组学为例对多组学技术的应用进行展望。
在合成生物学领域,合成代谢产物产量直接与酶活性和含量相关,而酶活性和含量直接与基因结构及活性相关,因此,多组学整合分析,去研究合成酶组是必要和有效的[67]。为了从多组学角度有效提升细胞合成效率,有必要合理利用多组学技术在合成生物学领域的应用(图 7)。
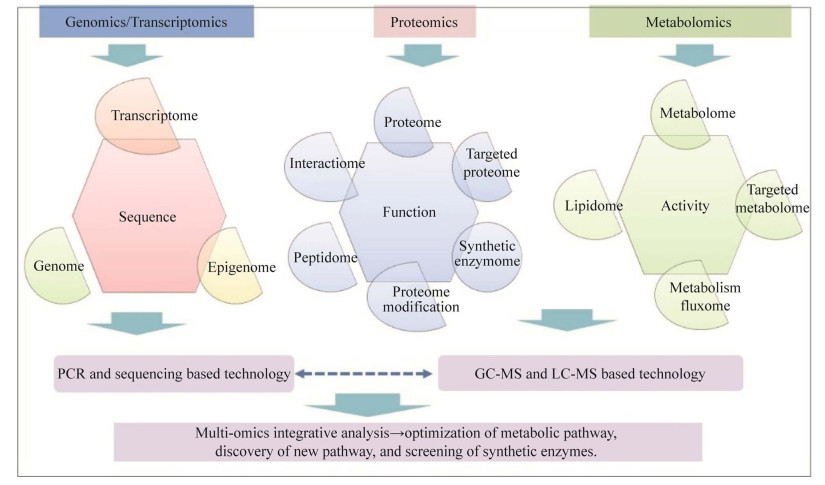
|
| 图 7 多组学在合成生物学领域应用技术路线 Fig. 7 Roadmap of multi-omics applications in synthetic biology. |
| |
如图 7所示,在基因组及转录组水平,以DNA或者RNA序列为基础,进行元件库构建。在蛋白质组水平可以进行合成酶组的组成、含量和功能分析。在代谢组水平可进行代谢物含量和活性分析。通过整合多组学数据的关联分析,可解决合成生物学领域重大关键的技术及工艺问题:(1) 如从生长阶段(初级代谢) 到生产阶段(次级代谢),其蛋白质组(酶组) 和代谢组的关系;(2) 底物转化效率和酶的相关性;(3) 初次代谢和次级代谢转化时机的评估等。可具体应用于合成生物学领域工程菌株筛选、代谢途径优化、隐性代谢途径发现及合成酶的筛选等。
目前,组学领域的单细胞组学已经在生命科学领域广泛应用,如单细胞基因组学[68]、单细胞转录组学[69]、单细胞蛋白质组[70]及单细胞代谢组学[71]等。但是,最为值得期待的是如何将基因组、表观基因组、转录组、蛋白质组及代谢组整合进行单细胞多组学研究,用于系统阐明细胞多样性、细胞谱系示踪、鉴定新的细胞类型、组织分型、肿瘤分期等成为当今多组学领域的亟待解决的挑战[72]。这些挑战主要包括:(1) 单细胞基因组测序覆盖率低及假阳性等问题;(2) 单细胞蛋白质组学中质谱仪的分辨率、灵敏度及采集速率问题;(3) 基于多组学数据的整合分析软件及数据库等。具体挑战包括基于不同层次组学数据格式的多样性,数据归一化、标准化及质量控制,多个组学大数据的高强度计算和处理对平台服务器要求,数据结果的专业性解读,涉及到诸多学科背景信息,如临床数据和病理资料[73]等。
可以预期,对新一代以多组学整合分析为特征的系统生物学研究新范式,主要任务将集中在模型构建,从而以动态变化的方式协同处理成千上万条转录组数据、蛋白质组数据及代谢组数据等,以期在生物医药领域实现个性化的癌症治疗方案选择[74],通过个性化的多组学大数据,实现精准医学领域对肿瘤病人的个性化靶向治疗[75],在有效增加抗癌药物疗效的同时,克服癌细胞化疗及免疫治疗的耐药表型,从而有效提高病人的生活质量。
| [1] |
Manzoni C, Kia DA, Vandrovcova J, et al. Genome, transcriptome and proteome: the rise of omics data and their integration in biomedical sciences. Brief Bioinform, 2016, 19(2): 286-302.
|
| [2] |
Sun Y, Hu YJ. Integrative analysis of multi-omics data for discovery and functional studies of complex human diseases. Adv Genet, 2016, 93: 147-190.
|
| [3] |
Hasin Y, Seldin M, Lusis A. Multi-omics approaches to disease. Genome Biol, 2017, 18(1): 1-15. DOI:10.1186/s13059-016-1139-1
|
| [4] |
Stratton MR, Campbell PJ, Futreal PA. The cancer genome. Nature, 2009, 458(7239): 719-724. DOI:10.1038/nature07943
|
| [5] |
Francis RC, Epigenetics: the Ultimate Mystery of Inheritance. New York: WW Norton, 2011: ISBN 978-0-393-07005-7.
|
| [6] |
Bernstein BE, Meissner A, Lander ES. The mammalian epigenome. Cell, 2007, 128(4): 669-681. DOI:10.1016/j.cell.2007.01.033
|
| [7] |
Horvath S, Raj K. DNA methylation-based biomarkers and the epigenetic clock theory of ageing. Nat Rev Genet, 2018, 19(6): 371-384.
|
| [8] |
Jones MJ, Goodman SJ, Kobor MS. DNA methylation and healthy human aging. Aging Cell, 2015, 14(6): 924-932. DOI:10.1111/acel.12349
|
| [9] |
Köhler F, Rodríguez-Paredes M. DNA methylation in epidermal differentiation, aging, and cancer. J Invest Dermatol, 2020, 140(1): 38-47. DOI:10.1016/j.jid.2019.05.011
|
| [10] |
Piétu G, Mariage-Samson R, Fayein NA, et al. The Genexpress IMAGE knowledge base of the human brain transcriptome: a prototype integrated resource for functional and computational genomics. Genome Res, 1999, 9(2): 195-209. DOI:10.1101/gr.9.2.195
|
| [11] |
Jeong E, Moon SU, Song ME, et al. Transcriptome modeling and phenotypic assays for cancer precision medicine. Arch Pharm Res, 2017, 40(8): 906-914.
|
| [12] |
Aebersold R, Mann M. Mass-spectrometric exploration of proteome structure and function. Nature, 2016, 537(7620): 347-355. DOI:10.1038/nature19949
|
| [13] |
Jiang Y, Sun A, Zhao Y, et al. Proteomics identifies new therapeutic targets of early-stage hepatocellular carcinoma. Nature, 2019, 567(7747): 257-261. DOI:10.1038/s41586-019-0987-8
|
| [14] |
Haymond A, Davis JB, Espina V. Proteomics for cancer drug design. Expert Rev Proteomics, 2019, 16(8): 647-664. DOI:10.1080/14789450.2019.1650025
|
| [15] |
Davidi D, Milo R. Lessons on enzyme kinetics from quantitative proteomics. Curr Opin Biotechnol, 2017, 46: 81-89. DOI:10.1016/j.copbio.2017.02.007
|
| [16] |
Nicholson J, Lindon JC, Holmes E. 'Metabonomics': understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data. Xenobiotica, 1999, 29(11): 1181-1189. DOI:10.1080/004982599238047
|
| [17] |
Lee JW, Su YP, Baloni P, et al. Integrated analysis of plasma and single immune cells uncovers metabolic changes in individuals with COVID-19. Nat Biotechnol, 2022, 40(1): 110-120. DOI:10.1038/s41587-021-01020-4
|
| [18] |
Jacob M, Lopata AL, Dasouki M, et al. Metabolomics toward personalized medicine. Mass Spectrom Rev, 2019, 38(3): 221-238. DOI:10.1002/mas.21548
|
| [19] |
Wu X, Ao HX, Gao H, et al. Metabolite biomarker discovery for human gastric cancer using dried blood spot mass spectrometry metabolomic approach. Sci Rep, 2022, 12(1): 14632.
|
| [20] |
Wang WS, Li SS, Li ZL, et al. Harnessing the intracellular triacylglycerols for titer improvement of polyketides in Streptomyces. Nat Biotechnol, 2020, 38(1): 76-83. DOI:10.1038/s41587-019-0335-4
|
| [21] |
Hu TS, Chitnis N, Monos D, et al. Next-generation sequencing technologies: an overview. Hum Immunol, 2021, 82(11): 801-811. DOI:10.1016/j.humimm.2021.02.012
|
| [22] |
Couvillion SP, Zhu Y, Nagy G, et al. New mass spectrometry technologies contributing towards comprehensive and high throughput omics analyses of single cells. Analyst, 2019, 144(3): 794-807. DOI:10.1039/C8AN01574K
|
| [23] |
Koh HWL, Fermin D, Vogel C, et al. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl, 2019, 5: 22. DOI:10.1038/s41540-019-0099-y
|
| [24] |
Vasaikar SV, Straub P, Wang J, et al. LinkedOmics: analyzing multi-omics data within and across 32 cancer types. Nucleic Acids Res, 2018, 46(D1): D956-D963. DOI:10.1093/nar/gkx1090
|
| [25] |
Lancaster SM, Sanghi A, Wu S, et al. A customizable analysis flow in integrative multi-omics. Biomolecules, 2020, 10(12): 1606. DOI:10.3390/biom10121606
|
| [26] |
Pinu FR, Beale DJ, Paten AM, et al. Systems biology and multi-omics integration: viewpoints from the metabolomics research community. Metabolites, 2019, 9(4): 76. DOI:10.3390/metabo9040076
|
| [27] |
Tan AH, Chong CW, Lim SY, et al. Gut microbial ecosystem in parkinson disease: new clinicobiological insights from multi-omics. Ann Neurol, 2021, 89(3): 546-559. DOI:10.1002/ana.25982
|
| [28] |
McCafferty CL, Verbeke EJ, Marcotte EM, et al. Structural biology in the multi-omics era. J Chem Inf Model, 2020, 60(5): 2424-2429. DOI:10.1021/acs.jcim.9b01164
|
| [29] |
Ribeiro FJ, Przybylski D, Yin SY, et al. Finished bacterial genomes from shotgun sequence data. Genome Res, 2012, 22(11): 2270-2277.
|
| [30] |
Wang Z, Gerstein M, Snyder M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet, 2009, 10(1): 57-63.
|
| [31] |
Nakato R, Sakata T. Methods for ChIP-seq analysis: a practical workflow and advanced applications. Methods, 2021, 187: 44-53.
|
| [32] |
Wold B, Myers RM. Sequence census methods for functional genomics. Nat Methods, 2008, 5(1): 19-21.
|
| [33] |
Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. PNAS, 1977, 74(12): 5463-5467.
|
| [34] |
Metzker ML. Sequencing technologies-the next generation. Nat Rev Genet, 2010, 11(1): 31-46.
|
| [35] |
Pareek CS, Smoczynski R, Tretyn A. Sequencing technologies and genome sequencing. J Appl Genet, 2011, 52(4): 413-435.
|
| [36] |
Van Dijk EL, Jaszczyszyn Y, Naquin D, et al. The third revolution in sequencing technology. Trends Genet, 2018, 34(9): 666-681.
|
| [37] |
Wang Y, Zhao Y, Bollas A, et al. Nanopore sequencing technology, bioinformatics and applications. Nat Biotechnol, 2021, 39(11): 1348-1365.
|
| [38] |
Garalde DR, Snell EA, Jachimowicz D, et al. Highly parallel direct RNA sequencing on an array of nanopores. Nat Methods, 2018, 15(3): 201-206.
|
| [39] |
Rand AC, Jain M, Eizenga JM, et al. Mapping DNA methylation with high-throughput nanopore sequencing. Nat Methods, 2017, 14(4): 411-413.
|
| [40] |
Goeminne LJE, Gevaert K, Clement L. Experimental design and data-analysis in label-free quantitative LC/MS proteomics: a tutorial with MSqRob. J Proteomics, 2018, 171: 23-36.
|
| [41] |
Graumann J, Hubner NC, Kim JB, et al. Stable isotope labeling by amino acids in cell culture (SILAC) and proteome quantitation of mouse embryonic stem cells to a depth of 5, 111 proteins. Mol Cell Proteomics, 2008, 7(4): 672-683.
|
| [42] |
Heller M, Mattou H, Menzel C, et al. Trypsin catalyzed 16O-to-18O exchange for comparative proteomics: tandem mass spectrometry comparison using MALDI-TOF, ESI-QTOF, and ESI-ion trap mass spectrometers. J Am Soc Mass Spectrom, 2003, 14(7): 704-718.
|
| [43] |
Kumar D, Varshney S, Sengupta S, et al. A comparative study of the proteome regulated by the Rpb4 and Rpb7 subunits of RNA polymerase II in fission yeast. J Proteomics, 2019, 199: 77-88.
|
| [44] |
Pagel O, Kollipara L, Sickmann A. Tandem mass tags for comparative and discovery proteomics. Methods Mol Biol, 2021, 2228, 117-131.
|
| [45] |
Harlan R, Zhang H. Targeted proteomics: a bridge between discovery and validation. Expert Rev Proteomics, 2014, 11(6): 657-661.
|
| [46] |
Bertsch A, Jung S, Zerck A, et al. Optimal de novo design of MRM experiments for rapid assay development in targeted proteomics. J Proteome Res, 2010, 9(5): 2696-2704.
|
| [47] |
Rauniyar N. Parallel Reaction Monitoring: a targeted experiment performed using high resolution and high mass accuracy mass spectrometry. Int J Mol Sci, 2015, 16(12): 28566-28581.
|
| [48] |
Afgan E, Baker D, Batut B, et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Res, 2018, 46(W1): W537-W544.
|
| [49] |
Sangaralingam A, Dayem Ullah AZ, Marzec J, et al. 'Multi-omic' data analysis using O-miner. Brief Bioinform, 2017, 20(1): 130-143.
|
| [50] |
Subramanian I, Verma S, Kumar S, et al. Multi-omics data integration, interpretation, and its application. Bioinform Biol Insights, 2020, 14: 1177932219899051.
|
| [51] |
Tasan M, Musso G, Hao T, et al. Selecting causal genes from genome-wide association studies via functionally coherent subnet works. Nat Methods, 2015, 12(2): 154-159.
|
| [52] |
Lu J, Zhang Y, Sun M, et al. Multi-omics analysis of fatty acid metabolism in thyroid carcinoma. Front Oncol, 2021, 11: 737127.
|
| [53] |
Network CGAR, Weinstein JN, Collisson EA, et al. The cancer genome atlas pan-cancer analysis project. Nat Genet, 2013, 45(10): 1113-1120.
|
| [54] |
Champion M, Brennan K, Croonenborghs T, et al. Module analysis captures pancancer genetically and epigenetically deregulated cancer driver genes for smoking and antiviral response. EBioMedicine, 2018, 27: 156-166.
|
| [55] |
Huang L, Brunell D, Stephan C, et al. Driver network as a biomarker: systematic integration and network modeling of multi-omics data to derive driver signaling pathways for drug combination prediction. Bioinformatics, 2019, 35(19): 3709-3717.
|
| [56] |
Tang BH, Pan ZX, Yin K, et al. Recent advances of deep learning in bioinformatics and computational biology. Front Genet, 2019, 10: 214.
|
| [57] |
Gabasova E, Reid J, Wernisch L. Clusternomics: integrative context-dependent clustering for heterogeneous datasets. PLoS Comput Biol, 2017, 13(10): e1005781.
|
| [58] |
Rohart F, Gautier B, Singh A, et al. mixOmics: an R package for omics feature selection and multiple data integration. PLoS Comput Biol, 2017, 13(11): e1005752.
|
| [59] |
Ren S, Shao Y, Zhao X, et al. Integration of metabolomics and transcriptomics reveals major metabolic pathways and potential biomarker involved in prostate cancer. Mol Cell Proteomics, 2016, 15(1): 154-163.
|
| [60] |
Zhu ZJ, Zhang SN, Wang P, et al. A comprehensive review of the analysis and integration of omics data for SARS-CoV-2 and COVID-19. Brief Bioinform, 2021, 23(1): bbab446.
|
| [61] |
Solovev I, Shaposhnikov M, Moskalev A. Multi-omics approaches to human biological age estimation. Mech Ageing Dev, 2020, 185: 111192.
|
| [62] |
Goering AW, McClure RA, Doroghazi JR, et al. Metabologenomics: correlation of microbial gene clusters with metabolites drives discovery of a nonribosomal peptide with an unusual amino acid monomer. ACS Cent Sci, 2016, 2(2): 99-108.
|
| [63] |
Paulus C, Rebets Y, Tokovenko B, et al. New natural products identified by combined genomics metabolomics profiling of marine Streptomyces sp. MP131-18. Sci Rep, 2017, 7: 42382.
|
| [64] |
Brunk E, George KW, Alonso-Gutierrez J, et al. Characterizing strain variation in engineered E. coli using a multi-omics-based workflow. Cell Syst, 2016, 2(5): 335-346.
|
| [65] |
Yoon SH, Han MJ, Jeong H, et al. Comparative multi-omics systems analysis of Escherichia coli strains B and K-12. Genome Biol, 2012, 13(5): R37.
|
| [66] |
Gutleben J, Chaib de Mares M, Van Elsas JD, et al. The multi-omics promise in context: from sequence to microbial isolate. Crit Rev Microbiol, 2018, 44(2): 212-229.
|
| [67] |
Nilsson A, Nielsen J, Palsson BO. Metabolic models of protein allocation call for the kinetome. Cell Syst, 2017, 5(6): 538-541.
|
| [68] |
Gawad C, Koh W, Quake SR. Single-cell genome sequencing: current state of the science. Nat Rev Genet, 2016, 17(3): 175-188.
|
| [69] |
Kolodziejczyk AA, Kim JK, Svensson V, et al. The technology and biology of single-cell RNA sequencing. Mol Cell, 2015, 58(4): 610-620.
|
| [70] |
Yu J, Zhou J, Sutherland A, et al. Microfluidics-based single-cell functional proteomics for fundamental and applied biomedical applications. Annu Rev Anal Chem (Palo Alto Calif), 2014, 7: 275-295.
|
| [71] |
Zenobi R. Single-cell metabolomics: analytical and biological perspectives. Science, 2013, 342(6163): 1243259.
|
| [72] |
Bock C, Farlik M, Sheffield NC. Multi-omics of single cells: strategies and applications. Trends Biotechnol, 2016, 34(8): 605-608.
|
| [73] |
López de Maturana E, Alonso L, Alarcón P, et al. Challenges in the integration of omics and non-omics data. Genes, 2019, 10(3): 238.
|
| [74] |
Werner HMJ, Mills GB, Ram PT. Cancer systems biology: a peek into the future of patient care. Nat Rev Clin Oncol, 2014, 11(3): 167-176.
|
| [75] |
GuhaThakurta D, Sheikh NA, Meagher TC, et al. Applications of systems biology in cancer immunotherapy: from target discovery to biomarkers of clinical outcome. Expert Rev Clin Pharmacol, 2013, 6(4): 387-401.
|