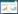中国科学院微生物研究所、中国微生物学会主办
文章信息
- 郭圆圆, 孙雅如, 张和平
- GUO Yuanyuan, SUN Yaru, ZHANG Heping
- 微生物转录组学技术研究进展
- Advances in microbial transcriptomics techniques
- 生物工程学报, 2022, 38(10): 3606-3615
- Chinese Journal of Biotechnology, 2022, 38(10): 3606-3615
- 10.13345/j.cjb.220527
-
文章历史
- Received: July 7, 2022
- Accepted: August 18, 2022
2. 内蒙古农业大学 农业农村部奶制品加工重点实验室, 内蒙古 呼和浩特 010018;
3. 内蒙古农业大学 内蒙古乳品生物技术与工程重点实验室, 内蒙古 呼和浩特 010018
2. Key Laboratory of Dairy Products Processing, Ministry of Agriculture and Rural Affairs, Inner Mongolia Agricultural University, Hohhot 010018, Inner Mongolia, China;
3. Inner Mongolia Key Laboratory of Dairy Biotechnology and Engineering, Inner Mongolia Agricultural University, Hohhot 010018, Inner Mongolia, China
分子生物技术自20世纪90年代开始逐渐发展起来。研究人员利用DNA高通量测序技术对微生物基因序列进行可视化分析研究,增加了人们对生态环境中微生物多样性的认识。微生物转录组研究主要是对特定环境或特定时期下微生物的RNA进行分析,以此来获得基因转录情况,为研究微生物基因差异表达的调控规律提供有力的证据[1]。Auffray于1996年提出了转录组(transcriptome) 这一术语[2]。广义上转录组是指细胞中的转运RNA (transfer RNA, tRNA)、核糖体RNA (ribosome RNA, rRNA) 以及非编码RNA (noncoding RNA, ncRNA) 等所有转录产物。而狭义上转录组指的是细胞在特定环境下负责蛋白质转录和翻译的所有信使RNA (messenger RNA, mRNA)[3]。转录组学(transcriptomics) 主要研究细胞整体的基因转录情况以及调控规律,进而揭示某特定生物学过程分子机理的一门学科。转录组学发展时间较早、可应用领域的范围比较广泛。利用转录组学技术不仅发现了两株可影响癌症发展和癌症治疗效果的短双歧杆菌(Bifidobacterium breve)[4],还为分析特定环境微生物群落组成[5]、监测微生物感染[6]、了解细菌与昆虫之间共生的相互作用[7]提供了有利的工具。
随着分子生物技术的快速发展,相继出现了多种可用于转录组学研究的相关技术。微阵列技术是最早用于检测微生物已知序列表达情况的研究方法,但在发现新的基因表达序列方面存在一定限制。后续发展起来的转录组学测序技术特别是RNA测序技术(RNA sequencing, RNA-seq) 具有测序速度快、分辨率高等优点,现已被广泛使用。近年来,三代测序技术、单细胞转录组学技术以及空间转录组学等技术逐渐兴起,但由于成本高、操作难度大等缺点仍需完善。因此,本文主要分析探讨了传统和新型的微生物转录组学相关技术,为后续微生物转录组的研究提供了参考。
2 传统的微生物转录组学方法传统的微生物转录组学研究方法大致包括微阵列和测序技术。前者是利用分离自样品中的RNA与已知序列杂交进行定性定量分析,主要是指微阵列技术(microarray)。后者则是对提取得到的RNA进行前处理后直接进行测序,测序方法主要包括对序列标签的测序、对序列片段进行测序以及对全长RNA直接进行测序[8]。
2.1 基于杂交的微阵列技术微阵列技术是将成千上万个已知序列的DNA或寡核苷酸富集排列在固体支撑物上,如硅片、玻片、尼龙膜等。用来固定DNA的固体支撑物性质不同,微阵列常被称为印迹(blotting)、膜(membrane)、芯片(chip) 或玻片(slide)。微生物提取得到的RNA做荧光或放射性标记处理形成“探针”。将微阵列与探针在一定条件下进行杂交,用相应的设备获取图像信息,最后对结果进行分析比对。早期利用微阵列技术主要是对基因表达序列是否表达或表达量进行定性、定量分析研究[9]。根据排列在固体支撑物上的DNA序列不同,微阵列技术可大致分为利用已知的cDNA序列或寡核苷酸排列形成的两种微阵列。
2.1.1 互补脱氧核糖核苷酸(complementary DNA, cDNA) 微阵列互补脱氧核糖核苷酸(cDNA) 微阵列是将探针进行荧光标记后,与固定于固体支撑物上已知序列的cDNA进行杂交。Selinger等开发了一个具有高分辨率的“基因组阵列”并对对数生长期和稳定期的大肠杆菌总RNA进行定性鉴定[10]。结果发现,在稳定期存在许多已知参与饥饿反应的基因和以前未被识别的生长阶段调控基因被表达,并且参与翻译的基因在对数生长期有较高的表达水平。微阵列技术检测转录组表达技术较为烦琐,目前构建微阵列芯片的操作大多是由委托公司或研究所制得。由于RNA存在不稳定性,为了验证转录因子测序的正确性,大部分转录组分析实验会采用逆转录-聚合酶链式反应(reverse transcription-polymerase chain reaction, RT-PCR) 技术对基因表达情况进行反向检测。通过利用RT-PCR技术,Kim等通过定量分析证实了在强毒株中存在两个差异表达基因的转录水平明显高于弱毒株[11]。Lockyer等利用cDNA微阵列定量鉴定出耐药和易感蜗牛感染曼氏链球菌后2−24 h的98个差异表达基因等,并应用RT-PCR技术分析验证了结果[12]。
2.1.2 寡核苷酸微阵列寡核苷酸微阵列可以直接在芯片表面固定预先合成或原位合成的寡核苷酸[13]。Chizhikov等设计了人轮状病毒VP7蛋白基因特异性寡核苷酸探针与PCR扩增制备的荧光标记的单链杂交探针,分别与猴肾细胞中培养的轮状病毒株RNA杂交,通过检测20个编码轮状病毒的分离株,发现寡核苷酸芯片杂交技术优于传统PCR技术[14]。但利用此种方法生产高密度微阵列,不但价格昂贵,还需要高度精密的工业设备,在应用方面受到了限制。Wang等[15]通过进行原位定制寡核苷酸微阵列,设计了2 906个基因的寡核苷酸探针,总计6 208个探针,其中396个寡核苷酸为对照,结果发现,豆奶生态系统对干酪乳酪杆菌Zhang (Lacticaseibacillus casei Zhang) 的生长具有复杂的促进作用。
最早的转录组研究中通常使用微阵列技术检测微生物基因的表达水平,但由于该技术只能检测已知序列的基因的表达量,且操作烦琐、特异性低等缺点,使其在转录组学中的应用受到了限制。
2.2 基于测序的转录组学技术随着测序技术的出现,针对转录组学相关测序技术也随之发展起来。该技术主要是将RNA进行前处理后直接进行测序分析,利用这种方法可以快速地获得微生物在特定环境下的基因表达信息[16]。根据转录组学测序技术的发展可分为第一代测序技术、RNA-seq技术和第三代测序技术[17]。
2.2.1 第一代转录组学测序第一代转录组学测序技术获取基因表达水平的方式是将样品中的mRNA反转录合成cDNA,对cDNA两端的序列标签进行测序。该测序技术主要包括早期的基因表达序列分析技术(serial analysis of gene expression, SAGE)、表达序列标签技术(expression sequence tags technology, EST) 和大规模平行测序技术(massively parallel signature sequencing, MPSS)等[18]。
(1) 基因表达序列分析技术(serial analysis of gene expression, SAGE):Velculescu等于1995年建立了SAGE技术。每9个碱基的序列标签代表一种转录特征序列,并且标签出现的概率能够反映出对应mRNA的丰度。SAGE技术所产生的标签首先要从待测样品总RNA中分离纯化得到mRNA,将mRNA反转录合成cDNA,经过酶切处理后对得到的cDNA片段进行扩增、测序即可得到相应转录组信息(图 1)。SAGE技术的优点在于可以对未知序列进行测序,从而在鉴定代谢机制等的同时也可以鉴定到新表达基因序列[19]。鉴于SAGE技术的不断完善,目前,已被成功应用于酵母全基因表达谱的鉴定[20]。
|
| 图 1 第一代测序技术原理 Fig. 1 Principle of the first generation sequencing technique. |
| |
(2) 大规模平行测序技术(massively parallel signature sequencing, MPSS):MPSS技术是由Brenner等在2000年建立,先将预测到的所有可能表达的已知序列与直径为5 μm的微球相结合并构建文库,在微珠流动池中组装平面模板阵列,分析每个微珠上克隆模板的自由端序列,最后与待测样本进行结合就可得到16−20个碱基的特征序列信息[21] (图 1)。MPSS技术原理与SAGE技术相似但不相同,前者则可以得到更长的标签序列,在酵母cDNA文库中,单个操作就可得到的数十万个标签序列,并且能够检测到低水平的基因表达序列[22]。
(3) 表达序列标签技术(expression sequence tags technology, EST):与前两种测序技术相比,EST技术首先要构建某特定微生物的cDNA文库,从中选择部分cDNA片段进行扩增、测序,最后再与基因表达序列标签(expressed sequence tags, ESTs) 数据库进行比对并分析相关基因的表达丰度。EST技术可以得到约200−800 bp的cDNA片段并进行测序[18, 22] (图 1)。Zhang等通过对真菌角毛壳菌(Chaetomium cupreum) 寄生条件下基因表达的研究,阐明了真菌寄生的分子机制,推动了开发植物真菌病害生物防治的新策略[23]。然而对ESTs直接测序存在的缺点在于不能定量检测,并且通量低、成本高,不是转录组学分析的最佳方法[24]。
2.2.2 第二代测序技术随着测序技术的不断发展和进步,开始出现第二代测序技术——RNA-seq技术。RNA-seq技术可以全面、快速地检测特定环境和时间条件下的微生物基因表达信息。Illumina公司的新一代测序仪采用边合成边测序技术,该测序仪最大的特点在于测序时间短且得到的数据量大,现已被转录组学相关研究工作普遍应用。Guo等利用Illumina HiSeq 2500对酿酒酵母mRNA逆转录得到的cDNA进行测序,探讨了化学突变体酿酒酵母BY23-195菌株高核酸合成的遗传机制[25]。此外,Roe等对4对具有不同易感性的系统发育相关鲍曼不动杆菌分离株的转录组进行二代测序。确定了鲍曼不动杆菌5种不同的潜在耐药机制,为诊断治疗感染鲍曼不动杆菌患者的治疗和预后提供了基础[26]。
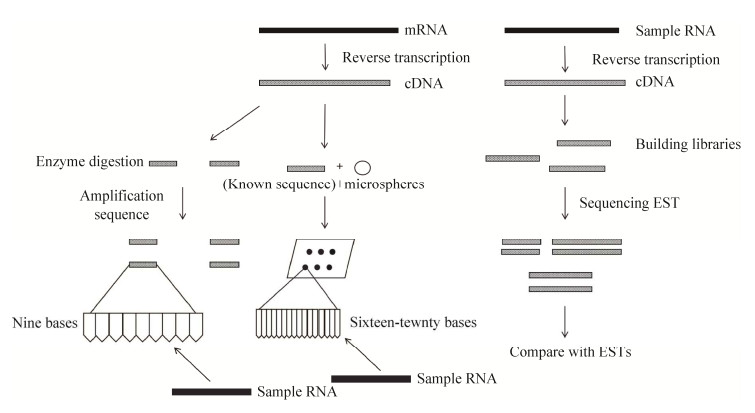
RNA-seq技术测序首先需要在微生物中提取所有的转录产物,将纯化后的mRNA片段化后反转录形成cDNA,对构建的cDNA文库进行PCR扩增、测序(图 2)。与微阵列技术和第一代测序技术相比较,RNA-seq技术在转录组学研究中的应用显得尤为广泛。利用该技术可以对酵母发酵过程中酿酒酵母(Saccharomyces cerevisiae) 和旧金山乳杆菌(Lactobacillus sanfranciscense) 在纯培养和混合培养条件下的代谢特征和转录变化进行探讨[27]。但根据Deng等对牙周关键口腔微生物的转录组特征分析发现,牙周生态位中的核胞体与实验室培养中的核胞体的基因表达情况有所不同[28]。微生物群落的多样性是其适应自然的一个重要特征,所以从实验室数据推断微生物相关功能性质需要谨慎对待。

|
| 图 2 第二代(左) 和第三代(右) 测序技术原理 Fig. 2 Principle of the second (left) and the third (right) generation sequencing technique. |
| |
RNA-seq测序仪高通量的特点是微阵列和第一代测序技术无法达到的。该技术的优点在于不仅准确率高,而且测序成本也远小于传统Sanger测序技术。另外,RNA-seq测序技术还具有对背景干扰具有高分辨率,对低表达量基因具有高灵敏度的特点,研究人员对乳酸乳球菌(Lactococcus lactis) 进行差异RNA测序(differential RNA-seq, dRNA-seq),发现了375种新型RNA,包括小RNA (small regulatory RNAs, sRNAs)、反义RNA (antisense RNA, asRNA)、新型(小) 开放阅读框(open reading frames, ORFs)、转录起始位点(transcription start site, TSS) 和操纵子结构等[29]。RNA-seq技术不仅能够检测出差异基因,还可以检测出相关基因的表达丰度[30]。Sethiya等比较了光滑念珠菌(Candida glabrata) 暴露于过氧化氢后不同时间点的转录组,发现在即时反应期间,基因表达会发生局部瞬时变化。但随着微生物逐渐适应氧化环境,转录组又重新表达翻译以此来恢复关键的细胞功能、蛋白质稳态、碳水化合物等的生物合成[31]。
2.2.3 第三代测序技术二代测序技术测序读数较短(通常为100− 300 bp),只能得到序列片段,然而第三代测序技术是以单分子测序为基础,可以对全长RNA进行直接测序,无需将RNA进行片段化处理,目前三代测序技术主要包括Pacific Biosciences (PacBio) 测序仪和Oxford Nanopore Technologies (ONT) 纳米孔测序两种方法[32]。两种方法虽然都可以对长DNA或RNA直接进行测序,但测序原理不同。PacBio测序仪是根据RNA逆转录得到的核苷酸产生独特荧光信号来识别碱基序列[33](图 2);而ONT纳米孔测序是基于生物工程纳米孔的单分子测序,这些纳米孔嵌入在施加电压的电阻膜中。当单链DNA或RNA片段通过时,膜上的电流发生变化,并使用基于递归神经网络(recursive neural network, RNN) 的算法将其转化为特定的核苷酸序列。由于测序是通过将电信号转化为核苷酸序列来介导的,所以纳米孔测序无需合成cDNA和PCR扩增就可以直接对RNA进行测序,并且产生的读长相对较长(> 2 Mb)[34]。
Uemura等通过运用第三代测序技术发现了氨基酸被单个核糖体串联的过程[35]。PacBio和ONT测序被广泛用于真核生物和病毒RNA的测序,而应用于原核生物转录组测序的实验技术流程相对较少。为此,Grünberger等为细菌模式生物大肠杆菌中的ONT RNA-seq测序设计了相关实验和生物信息分析的工作流程,且该流程适用于任何微生物[36]。虽然第三代测序技术存在许多优势,但测序错误率高、价格高昂的问题也一直影响着第三代测序技术在转录组学中的应用。为此,经过PacBio公司对技术的不断优化,在2016年发布的Sequel系统实现了新的突破,该系统提高测序通量的同时减少了测序所需的时间,并且其公司的Sequel System 6.0准确率较高且读长最长达300 kb[37]。
3 新型转录组学技术 3.1 单细胞转录组学与传统的群体转录组分析相比,单细胞转录组技术主要是分析特定环境下单细胞的基因表达水平,单细胞转录组测序(single-cell RNA sequencing, scRNA-seq) 就是其中技术之一,现已被广泛应用于分析各种生物学过程。早在1992年单细胞转录组学工作就已经被报道,但针对单细胞转录组学测序的相关技术直到2009年才被首次公开[38](图 3)。然而,单细胞转录组技术目前没有广泛应用于微生物领域中主要是因为在对微生物细胞进行裂解时操作难度大,缺少多聚腺苷酸尾保护mRNA分子,而且微生物在不同环境和生态位进行的转录表达也有差异。要想利用单细胞转录组技术在细胞水平上获得微生物的生物学机制,继续改进或发展相关技术是很重要的。截至目前已经开发了一种微生物分裂池连接转录组学方法(microbial split-Pool ligation transcriptomics, MicroSPLiT)[39],与前期高通量scRNA-seq方法相比,克服了细菌特有的低mRNA含量、细胞大小的多样性以及细胞壁结构等的挑战。该方法适用于革兰氏阴性和革兰氏阳性细菌,低成本、高通量,在一次实验中仅使用基本的设备就可对数万个细胞进行分析。
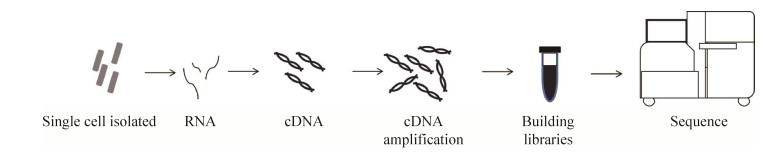
|
| 图 3 单细胞转录组技术原理 Fig. 3 Principle of single cell transcriptome technique. |
| |
单细胞转录组学改变了传统微生物群体系统转录水平的研究,逐渐向单个微生物细胞水平进行动力学和生长发育方面的研究。并且该技术对自然群落的研究也具有潜在的价值,不仅对物种分类多样性,而且对微生物的生长发育情况、细胞生理状态和生态相互作用特征的描述提供了新的见解[40]。通过对技术的继续改进和发展,运用单细胞转录组技术进行微生物领域研究的趋势也会逐渐增加。
3.2 空间转录组学通过上述的单细胞转录组学可以对特定环境下单个微生物细胞的差异表达基因进行研究,但在同一环境中微生物细胞的基因表达水平也会出现显著差异[41-43],这就表示微生物的基因表达也可能与所在生态位的不同出现差异。近年来,对于转录组研究逐渐进入到了空间转录组(spatial transcriptome) 的研究阶段。与之前的转录组学研究相比,进行空间转录组的研究不仅可以获得特定环境下单个微生物细胞基因表达水平信息,还可以了解在空间位置上微生物细胞之间基因表达的差异水平,为进一步细化在时间和空间两个特定条件下微生物真实基因表达的研究提供了重要的研究手段。Dar等开发了一种适用于环境中复杂的微生物群落或人体微生物群,名为平行序列荧光原位杂交(parallel sequential fluorescence in situ hybridization, par-seqFISH) 的转录组成像方法[44]。他们将这种技术应用于条件致病菌铜绿假单胞菌,分析了浮游和生物膜培养中数十种生理条件下的60万个个体。绘制了生物膜相关过程的空间背景,包括运动和亲属排斥机制,并识别广泛和高度空间分辨的代谢异质性。另外,mRNA标记技术和序列荧光原位杂交(sequential fluorescence in situ hybridization,seqFISH) 技术组合可用于在同一样本中以亚微米分辨率分析数百甚至数千个基因。到目前为止,seqFISH技术已经不仅应用于研究哺乳动物细胞和组织的生理生化系统,也适用于对环境和人体微生物群基因表达方面的分析鉴定。
随着细胞通量、转录本数量和质量检测技术的不断发展,空间转录组学技术也产生了巨大的进步,对微生物细胞空间定位信息也逐渐准确。对于微生物基因表达水平的研究已经日渐成熟,研究人员的目光开始逐渐转入到表观转录组学方面的研究。
3.3 表观转录组学根据表观转录组学研究发现,大多数RNA动态可逆的化学修饰是在tRNA、rRNA和小核RNA (small nuclear RNA, snRNA) 等当中[45],Kouvela等研究了在人类受到细菌感染过程中起关键作用的tRNA表观转录组的形成[46]。由于mRNA占细胞中总RNA含量的百分比较低、检测技术受限等原因,mRNA被检测到的化学修饰较少[47]。然而,随着检测技术的不断进步,mRNA存在的化学修饰在最近几年也逐渐被挖掘,并形成了关于表观转录组的研究学科[48]。mRNA上目前已经被鉴定到的化学修饰主要包括7-甲基鸟嘌呤(7-methylguanosine, m7G)、N6, 2′-O-二甲基腺嘌呤(N6, 2′-O-dimethyladenosine, m6Am)、2′-氧甲基化(2′-O-methylation, Nm) 以及内部的N6-甲基腺嘌呤(N6-methyladenosine, m6A) 等[49]。随着表观转录组学的发展进步,针对RNA修饰的研究方法逐渐被研究人员开发出来,目前以质谱(mass spectrometry, MS) 为基础的方法是检测不同RNA修饰的唯一通用方法。Wang等使用MS方法分析了敏捷乳杆菌(Lactobacillus agilis) 的表观转录组,并确定了L. agilis的表观转录组是否参与了菌株适应益生元菊粉的生理代谢过程[50]。
4 展望在生物信息分析中,通过转录组学可研究特定时空下的生物基因表达水平,从而揭示其内在机理,为各领域的深入研究提供了新思路。随着转录组学发展至今,主要广泛应用于真核生物等领域,而针对微生物的应用还存在着许多待解决的问题。例如,RNA对微环境变化的不稳定性;三代测序技术的准确率相对较低、成本高;单细胞转录组学研究中的微生物细胞的裂解;以及对RNA表观修饰的深入研究技术的缺乏等。随着相关生物技术的改进,微生物转录组学的不足也被日益完善。随着转录组学技术的不断成熟,势必加速微生物在不同生境的转录表达及调控信息了解的深入,将极大地推动微生物领域的进步。
| [1] |
马述, 刘虎虎, 田云, 等. 宏转录组技术及其研究进展. 生物技术通报, 2012(12): 46-50. Ma S, Liu HH, Tian Y, et al. Advances of metatranscriptomics technology. Biotechnol Bull, 2012(12): 46-50 (in Chinese). DOI:10.13560/j.cnki.biotech.bull.1985.2012.12.024 |
| [2] |
李宗瑾. 基于转录组学和代谢组学研究大肠杆菌K12碳降解物阻遏作用[D]. 深圳: 深圳大学, 2017. Li ZJ. Research of carbon catabolite repression in Escherichia coli K12 based on transcriptome and metabolomics[D]. Shenzhen: Shenzhen University, 2017 (in Chinese). |
| [3] |
文雅. 铬还原菌Lysinibacillus fusiformis 15-4的转录组学研究[D]. 兰州: 兰州交通大学, 2021. Wen Y. Transcriptomic characteristics of a chromium reducing Lysinibacillus fusiformis strain 15-4[D]. Lanzhou: Lanzhou Jiatong University, 2021 (in Chinese). |
| [4] |
Yoon Y, Kim G, Jeon BN, et al. Bifidobacterium strain-specific enhances the efficacy of cancer therapeutics in tumor-bearing mice. Cancers, 2021, 13(5): 957. DOI:10.3390/cancers13050957
|
| [5] |
Zheng T, Zhang KK, Zhu XD, et al. Integrated metatranscriptome and transcriptome reveals the microbial community composition and physiological function of xylem sap on grapevine during bleeding period. Genes Genomics, 2019, 41(9): 1095-1111. DOI:10.1007/s13258-019-00841-7
|
| [6] |
Pan WY, Ngo TTM, Camunas-Soler J, et al. Simultaneously monitoring immune response and microbial infections during pregnancy through plasma cfRNA sequencing. Clin Chem, 2017, 63(11): 1695-1704. DOI:10.1373/clinchem.2017.273888
|
| [7] |
Mazumdar T, Teh BS, Murali A, et al. Transcriptomics reveal the survival strategies of Enterococcus mundtii in the gut of Spodoptera littoralis. J Chem Ecol, 2021, 47(2): 227-241. DOI:10.1007/s10886-021-01246-1
|
| [8] |
赵乐, 孟祥晨. 转录组测序技术在植物乳杆菌细菌素合成研究中的应用研究进展. 乳业科学与技术, 2021, 44(5): 32-37. Zhao L, Meng XC. A review of the application of transcriptome sequencing in research on bacteriocin synthesis by Lactobacillus plantarum. J Dairy Sci Technol, 2021, 44(5): 32-37 (in Chinese). DOI:10.15922/j.cnki.jdst.2021.05.007 |
| [9] |
乔纳森. 佩夫斯纳, 著. 田卫东, 赵兴明, 主译. 生物信息学与功能基因组学. 第3版. 北京: 化学工业出版社, 2006: 379-382. Jonathan Pevsner, write. Tian WD, Zhao XM, main translator. Bioinformatics and Functional Genomics. Third edition. Beijing: Chenmical Industry Press, 2006: 379-382 (in Chinese). |
| [10] |
Selinger DW, Cheung KJ, Mei R, et al. RNA expression analysis using a 30 base pair resolution Escherichia coli genome array. Nat Biotechnol, 2000, 18(12): 1262-1268. DOI:10.1038/82367
|
| [11] |
Kim IH, Kim BS, Lee KS, et al. Identification of virulence factors in Vibrio vulnificus by comparative transcriptomic analyses between clinical and environmental isolates using cDNA microarray. J Microbiol Biotechnol, 2011, 21(12): 1228-1235. DOI:10.4014/jmb.1111.11016
|
| [12] |
Lockyer AE, Spinks J, Kane RA, et al. Biomphalaria glabrata transcriptome: cDNA microarray profiling identifies resistant-and susceptible-specific gene expression in haemocytes from snail strains exposed to Schistosoma mansoni. BMC Genomics, 2008, 9(1): 1-17. DOI:10.1186/1471-2164-9-1
|
| [13] |
杨劲松, 陈诗书. 微阵列(microarrays)技术及其应用. 生命科学, 2001, 13(2): 85-88. Yang JS, Chen SS. Microarray technology and its application. Chin Bull Life Sci, 2001, 13(2): 85-88 (in Chinese). DOI:10.3969/j.issn.1004-0374.2001.02.010 |
| [14] |
Chizhikov V, Wagner M, Ivshina A, et al. Detection and genotyping of human group A rotaviruses by oligonucleotide microarray hybridization. J Clin Microbiol, 2002, 40(7): 2398-2407. DOI:10.1128/JCM.40.7.2398-2407.2002
|
| [15] |
Wang JC, Zhang WY, Zhong Z, et al. Transcriptome analysis of probiotic Lactobacillus casei Zhang during fermentation in soymilk. J Ind Microbiol Biotechnol, 2012, 39(1): 191-206. DOI:10.1007/s10295-011-1015-7
|
| [16] |
田瑞杰, 张勇, 冯大鸿, 等. 基于宏转录组学的浓香型白酒酒醅活性微生物群落空间异质性研究. 轻工学报, 2022, 37(1): 1-11. Tian RJ, Zhang Y, Feng DH, et al. Study on spatial heterogeneity of active microbial community in strong-flavor baijiu fermented grains based on metatranscriptome. J Light Ind, 2022, 37(1): 1-11 (in Chinese). |
| [17] |
步雨珊, 杨慧, 易华西, 等. 转录组学在乳酸菌研究中的应用. 食品安全质量检测学报, 2019, 10(13): 4370-4375. Bu YS, Yang H, Yi HX, et al. Application of transcriptomics in the research of lactic acid bacteria. J Food Saf Qual, 2019, 10(13): 4370-4375 (in Chinese). DOI:10.3969/j.issn.2095-0381.2019.13.057 |
| [18] |
赵圣明, 赵岩岩, 马汉军, 等. 转录组学在抑菌机制中的应用研究进展. 食品与发酵工业, 2017, 43(7): 259-264. Zhao SM, Zhao YY, Ma HJ, et al. Applications of transcriptomics in research of antimicrobial mechanism. Food Ferment Ind, 2017, 43(7): 259-264 (in Chinese). DOI:10.13995/j.cnki.11-1802/ts.013619 |
| [19] |
付畅, 黄宇. 转录组学平台技术及其在植物抗逆分子生物学中的应用. 生物技术通报, 2011(6): 40-46. Fu C, Huang Y. Transcriptome platforms and their applications in molecular biology of plant stress resistance. Biotechnol Bull, 2011(6): 40-46 (in Chinese). DOI:10.13560/j.cnki.biotech.bull.1985.2011.06.031 |
| [20] |
Anisimov SV. Serial Analysis of gene expression (SAGE): 13 years of application in research. Curr Pharm Biotechnol, 2008, 9(5): 338-350. DOI:10.2174/138920108785915148
|
| [21] |
Brenner S, Johnson M, Bridgham J, et al. Gene expression analysis by massively parallel signature sequencing (MPSS) on microbead arrays. Nat Biotechnol, 2000, 18(6): 630-634. DOI:10.1038/76469
|
| [22] |
刘伟, 郭光艳, 秘彩莉. 转录组学主要研究技术及其应用概述. 生物学教学, 2019, 44(10): 2-5. Liu W, Guo GY, Bei CL. A summary of the main research techniques of transcriptology and their applications. Biol Teach, 2019, 44(10): 2-5 (in Chinese). DOI:10.3969/j.issn.1004-7549.2019.10.001 |
| [23] |
Zhang HY, Yang Q, Wang G, et al. Analysis of expressed sequence tags from Chaetomium cupreum grown under conditions associated with mycoparasitism. Lett Appl Microbiol, 2009, 48(3): 275-280. DOI:10.1111/j.1472-765X.2008.02487.x
|
| [24] |
陈杰. 大规模平行测序技术(MPSS)研究进展. 生物化学与生物物理进展, 2004, 31(8): 761-765. Chen J. A novel gene identification approach: massively parallel signature sequencing. Prog Biochem Biophys, 2004, 31(8): 761-765 (in Chinese). DOI:10.3321/j.issn:1000-3282.2004.08.018 |
| [25] |
Guo XW, Zhao B, Zhou XR, et al. Analysis of the molecular basis of Saccharomyces cerevisiae mutant with high nucleic acid content by comparative transcriptomics. Food Res Int, 2021, 142(1): 110188.
|
| [26] |
Roe C, Williamson CHD, Vazquez AJ, et al. Bacterial genome wide association studies (bGWAS) and transcriptomics identifies cryptic antimicrobial resistance mechanisms in Acinetobacter baumannii. Front Public Heal, 2020, 8: 451. DOI:10.3389/fpubh.2020.00451
|
| [27] |
Yang HY, Sadiq FA, Liu TJ, et al. Use of physiological and transcriptome analysis to infer the interactions between Saccharomyces cerevisiae and Lactobacillus sanfranciscensis isolated from Chinese traditional sourdoughs. LWT, 2020, 126(1): 109268.
|
| [28] |
Deng ZL, Sztajer H, Jarek M, et al. Worlds apart-transcriptome profiles of key oral microbes in the periodontal pocket compared to single laboratory culture reflect synergistic interactions. Front Microbiol, 2018, 9: 124. DOI:10.3389/fmicb.2018.00124
|
| [29] |
Van Der Meulen SB, Jong AD, Kok J. Transcriptome landscape of Lactococcus lactis reveals many novel RNAs including a small regulatory RNA involved in carbon uptake and metabolism. RNA Biol, 2016, 13(3): 353-366. DOI:10.1080/15476286.2016.1146855
|
| [30] |
范凯. 华根霉脂肪酶液态发酵不同形态菌体的比较转录组学分析[D]. 无锡: 江南大学, 2017. Fan K. Comparative transcriptome analysis of Rhizopus chinensis with different mycelia morphology in submerged fermentation for lipase production[D]. Wuxi: Jiangnan University, 2017 (in Chinese). |
| [31] |
Sethiya P, Rai MN, Rai R, et al. Transcriptomic analysis reveals global and temporal transcription changes during Candida glabrata adaptation to an oxidative environment. Fungal Biol, 2020, 124(5): 427-439. DOI:10.1016/j.funbio.2019.12.005
|
| [32] |
Ciuffreda L, Rodríguez-Pérez H, Flores C. Nanopore sequencing and its application to the study of microbial communities. Comput Struct Biotechnol J, 2021, 19(1): 1497-1511.
|
| [33] |
Vilfan ID, Tsai YC, Clark TA, et al. Analysis of RNA base modification and structural rearrangement by single-molecule real-time detection of reverse transcription. J Nanobiotechnology, 2013, 11(1): 1-11. DOI:10.1186/1477-3155-11-1
|
| [34] |
Soneson C, Yao Y, Bratus-Neuenschwander A, et al. A comprehensive examination of Nanopore native RNA sequencing for characterization of complex transcriptomes. Nat Commun, 2019, 10(1): 3359. DOI:10.1038/s41467-019-11272-z
|
| [35] |
Uemura S, Aitken CE, Korlach J, et al. Real-time tRNA transit on single translating ribosomes at codon resolution. Nature, 2010, 464(7291): 1012-1017. DOI:10.1038/nature08925
|
| [36] |
Grünberger F, Ferreira-Cerca S, Grohmann D. Nanopore sequencing of RNA and cDNA molecules in Escherichia coli. RNA, 2022, 28(3): 400-417. DOI:10.1261/rna.078937.121
|
| [37] |
李玉梅, 李书娴, 李向上, 等. 第三代测序技术在转录组学研究中的应用. 生命科学仪器, 2018, 16(Z1): 114-121, 113. Li YM, Li SX, Li XS, et al. Transcriptome studies with the third-generation sequencing technology. Life Sci Instrum, 2018, 16(Z1): 114-121, 113 (in Chinese). |
| [38] |
Zhang Y, Gao JX, Huang YY, et al. Recent developments in single-cell RNA-seq of microorganisms. Biophys J, 2018, 115(2): 173-180. DOI:10.1016/j.bpj.2018.06.008
|
| [39] |
Kuchina A, Brettner LM, Paleologu L, et al. Microbial single-cell RNA sequencing by split-pool barcoding. Science, 2021, 371(6531): eaba5257. DOI:10.1126/science.aba5257
|
| [40] |
Ku C, Sebé-Pedrós A. Using single-cell transcriptomics to understand functional states and interactions in microbial eukaryotes. Philos Trans R Soc Lond B Biol Sci, 2019, 374(1786): 20190098. DOI:10.1098/rstb.2019.0098
|
| [41] |
Ackermann M. A functional perspective on phenotypic heterogeneity in microorganisms. Nat Rev Microbiol, 2015, 13(8): 497-508.
|
| [42] |
Schreiber F, Ackermann M. Environmental drivers of metabolic heterogeneity in clonal microbial populations. Curr Opin Biotechnol, 2020, 62(1): 202-211.
|
| [43] |
Schreiber F, Littmann S, Lavik G, et al. Phenotypic heterogeneity driven by nutrient limitation promotes growth in fluctuating environments. Nat Microbiol, 2016, 1(6): 1-7.
|
| [44] |
Dar D, Dar N, Cai L, et al. Spatial transcriptomics of planktonic and sessile bacterial populations at single-cell resolution. Science, 2021, 373(6556): eabi4882.
|
| [45] |
Milanowska K, Mikolajczak K, Lukasik A, et al. RNApathwaysDB—a database of RNA maturation and decay pathways. Nucleic Acids Res, 2012, 41(D1): D268-D272.
|
| [46] |
Kouvela A, Zaravinos A, Stamatopoulou V. Adaptor molecules epitranscriptome reprograms bacterial pathogenicity. Int J Mol Sci, 2021, 22(16): 8409.
|
| [47] |
Desrosiers R, Friderici K, Rottman F. Identification of methylated nucleosides in messenger RNA from Novikoff hepatoma cells. PNAS, 1974, 71(10): 3971-3975.
|
| [48] |
He C. Grand challenge commentary: RNA epigenetics?. Nat Chem Biology, 2010, 6(12): 863-865.
|
| [49] |
彭金英, 伊成器. 蓬勃发展的表观转录组学. 中国生物化学与分子生物学报, 2021, 37(4): 407-418. Peng JY, Yi CQ. Flourishing epitranscriptomics research. Chin J Biochem Mol Biol, 2021, 37(4): 407-418 (in Chinese). |
| [50] |
Wang HZ, Simpson JH, Kotra ME, et al. Epitranscriptomic profile of Lactobacillus agilis and its adaptation to growth on inulin. BMC Res Notes, 2021, 14(1): 1-6.
|
 2022, Vol. 38
2022, Vol. 38