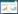中国科学院微生物研究所、中国微生物学会主办
文章信息
- 邱燕华, 翟斌涛, 白玉彬, 陈树林, 张继瑜
- QIU Yanhua, ZHAI Bintao, BAI Yubin, CHEN Shulin, ZHANG Jiyu
- 热蛋白质组学分析:一种全面评估蛋白质状态的技术
- Thermal proteome profiling: a technique for a comprehensive assessment of protein status
- 生物工程学报, 2022, 38(10): 3628-3637
- Chinese Journal of Biotechnology, 2022, 38(10): 3628-3637
- 10.13345/j.cjb.220206
-
文章历史
- Received: March 17, 2022
- Accepted: July 1, 2022
- Published: July 6, 2022
2. 西北农林科技大学 动物医学院, 陕西 杨凌 712100
2. College of Veterinary Medicine, Northwest A & F University, Yangling 712100, Shaanxi, China
药物在治疗疾病的过程中,会与机体内的靶蛋白结合,从而改变靶蛋白和下游蛋白的活性,最终终止导致疾病的生物过程[1]。了解与药物结合的蛋白质靶标对于新药研发、药物的临床应用、药物的作用机制和疾病的致病机理都有重要意义[2-3]。目前研究药物靶点的方法多基于化学修饰和表型分析,但基于化学修饰的方法可能会导致药物活性的改变,所以基于表型分析的方法越来越受到重视,热蛋白质组学分析(thermal proteome profiling, TPP) 就是其中一种[4]。TPP发明于2014年,最初用于表征活细胞中抗癌药物作用的靶点和下游信号通路,随着方法的不断改进,TPP已不仅仅只用于确定药物的靶点和脱靶点[5]。
1 热蛋白质组学分析的原理TPP是基于细胞热漂移测定(cellular thermal shift assay, CETSA) 的原理和使用多重定量质谱的蛋白质组学来监测数千种表达蛋白质的熔解曲线[6]。TPP的原理同CETSA,都是基于配体诱导的靶蛋白热稳定的生物物理原理,配体与靶蛋白结合,通过改变靶蛋白的构象、增强靶蛋白分子内的疏水性、靶蛋白分子内或亚单位间产生化学交联等方式改变靶蛋白的热稳定性(通常增强)[7]。蛋白质的热稳定性可通过热变性中点温度(melting temperature, Tm),即蛋白质解折叠50%时对应的温度来表示。蛋白质在加热过程中会发生热变性解折叠,有配体结合的蛋白与无配体结合的蛋白相比一般更稳定,Tm也更高,通过对比有无配体结合蛋白的Tm和熔解曲线是否存在差异即可确定靶蛋白(图 1)[8]。
|
| 图 1 CETSA的原理 Fig. 1 Principle of CETSA. |
| |
蛋白质在加热条件下会发生变性且溶解性降低,CETSA通过蛋白质免疫印迹测定每个温度下剩余可溶性蛋白的含量,以此获得每种检测到的蛋白质的熔解曲线[9]。但蛋白质免疫印迹需基于抗体,因此CETSA仅限于同时研究少量蛋白质,Savitski等将CETSA与定量质谱相结合,建立了TPP的方法,让TPP的测定范围可以涉及到全蛋白质组[5]。
2 热蛋白质组学分析的方法目前为止,TPP主要包括温度范围的热蛋白质组学分析(TPP temperature range, TPP- TR)、配体浓度范围的热蛋白质组学分析(TPP compound concentration range, TPP-CCR) 和二维热蛋白质组学分析(two-dimensional TPP, 2D-TPP)。广义上说,TPP的方法步骤主要包括:(1) 样品制备;(2) 热处理;(3) 收集可溶性蛋白组分;(4) 基于质谱的蛋白组学分析;(5) 数据分析[8]。其中样品制备包括要研究的细胞材料和条件改变,细胞材料可以是细胞提取物、完整细胞、组织、生物体液(如血液) 和微生物(如古菌、细菌、原生生物、真菌)[10-16];条件变化通常是化学(如药物或代谢物)、遗传(如基因敲除或过表达)、环境的变化和不同的细胞状态(如细胞周期的不同阶段),也能在无条件变化下研究蛋白质在原位的熔解行为[17-21]。热处理是对样品进行短暂的热处理以诱导蛋白质变性聚集,时间通常为3 min[5]。可溶性蛋白组分通常通过超速离心收集上清液或使用多孔滤板以低速离心收集[22]。可溶性蛋白提取后用蛋白酶将蛋白质消化成肽,然后一般通过稳定同位素标记试剂串联质量标签(tandem mass tags, TMT) 对不同样本进行标记,通过质谱法对每个样品中肽的丰度进行量化[5]。最后用蛋白质组学数据库处理获得的原始质谱数据以识别和量化测定的蛋白质,TPP数据分析的最终目标是找到在两个或多个条件间热稳定性发生改变的蛋白质,具体的分析方法取决于所进行的TPP类型(TPP-TR、TPP-CCR或2D-TPP)[8]。
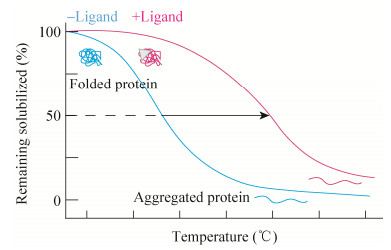
2.1 温度范围的热蛋白质组学分析TPP-TR是TPP最原始的方法,TPP的其他方法基本基于此发展而来。首先将有无配体处理的细胞或细胞提取物分成10等份,分别在递增的10个温度下加热,提取可溶性蛋白质,并用稳定同位素标记试剂串联质量标签(TMT10) 分别标记不同温度下的蛋白质,然后混合所有样品并通过LC-MS/MS进行分析,最后使用获得的报告离子强度生成有无配体情况下每种蛋白质的熔解曲线,同时计算有无配体条件下每种蛋白的Tm (图 2)[5]。加热温度一般为37–67 ℃,这是基于绝大多数蛋白质(> 90%) 的熔点范围,具体还需根据目标蛋白的实际熔解温度进行调整[5]。
|
| 图 2 TPP-TR方法步骤 Fig. 2 Protocol of TPP-TR method. 1: cell materials treated with or without ligands were divided into ten equal parts, orange hexagon: ligand; 2: samples are heated at different temperatures; 3: TMT10 labeled soluble proteins; 4: LC-MS/MS analysis; 5: calculation of the Tm of proteins. |
| |
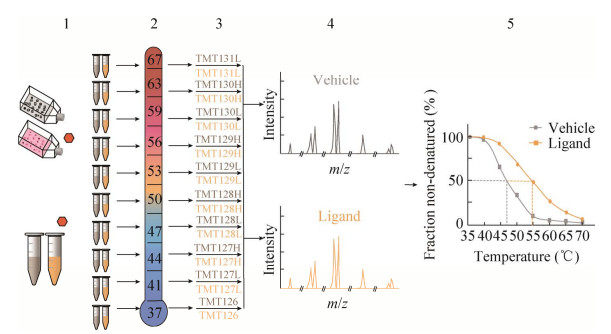
为了研究配体浓度对靶蛋白的影响,研究人员建立了一个等温剂量反应程序,即TPP- CCR。与TPP-TR不同,TPP-CCR用不同浓度配体处理细胞材料,并在相同的温度下加热,然后进行蛋白标记、鉴定和定量,最后通过数据处理可以获得每种蛋白质的剂量反应曲线,靶蛋白因与配体结合,其热稳定性被改变,所以靶蛋白的剂量反应曲线会随着配体浓度的增大而升高或降低,而未与配体结合蛋白的剂量反应曲线通常为一条直线,并不会随着配体浓度的变化而改变(图 3)[21]。TPP-CCR侧重于配体浓度的重要性,虽然不能同TPP-TR一样可以获得每种蛋白质的熔解曲线和Tm值,但是可通过测定EC50对不同蛋白质靶标的结合亲和力进行排序,这是TPP-TR不能达到的。TPP-CCR于单一温度下进行,所以温度是最关键的参数,温度选择不合适靶蛋白就不能被识别,TPP- CCR实验通常在略高于目标蛋白质的解链温度(未经配体处理) 下进行[23]。
|
| 图 3 TPP-CCR方法步骤 Fig. 3 Protocol of TPP-CCR method. 1: cell materials treated with different ligand concentrations, orange hexagon: ligand; 2: heating of samples with different ligand concentrations at a single temperature; 3: TMT10 labeled protein; 4: LC-MS/MS analysis; 5: protein identification, quantification, and data analysis. |
| |
2D-TPP可以说是TPP-TR和TPP-CCR的结合,同时也结合了两者的优势,是当前TPP方法中最准确灵敏的。细胞材料在n个不同浓度配体下孵育一定时间,每个样品被分成m个等份试样,每个试样都通过m个温度中的一个加热,并提取剩余的可溶性蛋白。蛋白质用蛋白酶消化并用TMT标记,一组TMT标记可用于所有浓度和两个相邻温度。执行w=m/2多重MS分析,通过数据库搜索识别肽,并在蛋白质水平聚合量化信号。因此,对于每个蛋白质i,可获得汇总报告离子强度的m×n数据矩阵Yi (图 4),最终生成由每个浓度和温度下可溶性蛋白质丰度强度着色的热图[24]。在2D-TPP方法中,温度和配体浓度都系统地发生变化,这样可以立刻估计配体对靶蛋白的亲和力,并且在识别目标时更加灵敏。因为与TPP-TR不同,2D-TPP所有变化的条件都在同一质谱中进行比较,从而产生更精确的量化。此外,在2D-TPP方法中,靶蛋白的热稳定性与配体浓度的关系预计呈剂量依赖性,这对数据增加了额外的质量要求,并可排除一定的假阳性[22, 24]。
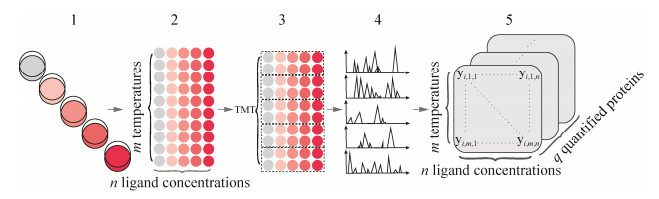
|
| 图 4 2D-TPP方法步骤 Fig. 4 Protocol of 2D-TR method. 1: cell materials treated with n different ligand concentrations; 2: each sample is heated at m temperatures; 3: TMT labels the proteins of all samples at two adjacent temperatures; 4: LC-MS/MS analysis; 5: protein identification, quantification, and data analysis. |
| |
经过近几年的发展,TPP的应用范围在不断地扩大。在药物发现中,TPP可以识别药物的靶点和脱靶点,后者可以解释药物的不良反应和再利用[22, 25];TPP还可用于通过研究翻译后修饰、蛋白质-蛋白质相互作用、核酸-蛋白质相互作用或基本蛋白质的功能来探索代谢途径[26-27];此外,TPP可以与限制性蛋白水解或成像相结合,以进一步了解蛋白质折叠动力学(图 5)[28]。除了以上应用,还有很多未知的应用正在探索中。
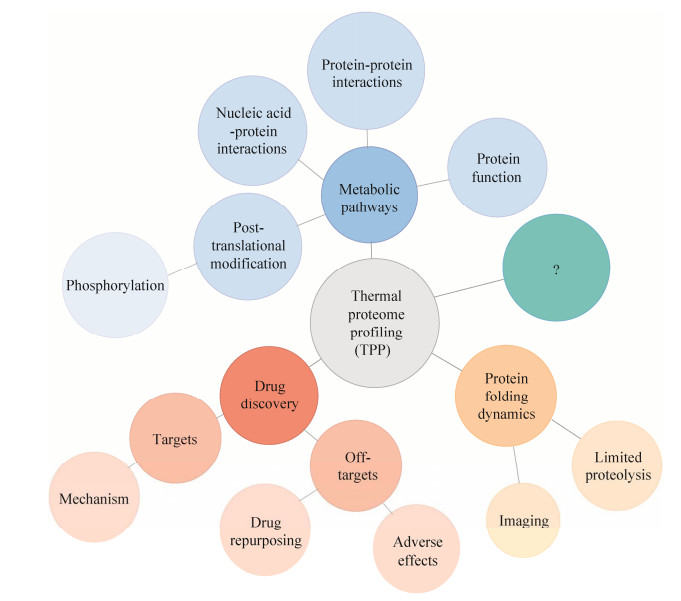
|
| 图 5 当前TPP的应用范围 Fig. 5 Current application scope of TPP. |
| |
TPP可以实现全面的药物靶点分析,因此在药物发现中具有很高的应用价值[28]。通过分析靶向和脱靶蛋白质结合来分析药物作用机制代表了目前TPP的主要应用[5, 29-30]。TPP最初就是被开发用于寻找药物类分子的靶点和脱靶点,截至目前,TPP已成功应用于大量药物靶向和脱靶筛查案例。比如TPP结果显示苯丙氨酸羟化酶是帕比司他的脱靶点,提示应慎重用该药物治疗酪氨酸血症[22]。Goroshchuk等通过TPP将会影响免疫反应和脂肪酸代谢的PIP4K2A和ZADH2鉴定为伏拉塞替的脱靶点,成功地解释了在接受伏拉塞替治疗的患者中出现的一些副作用[31]。此外,TPP在细菌中的应用有望通过筛选新化合物的靶点来加速新抗生素的发现,并能更好地了解耐药机制[11, 32-33]。
3.2 代谢途径除了药物发现,TPP也可以成为绘制代谢途径的重要工具,因为它可以研究翻译后修饰、蛋白质-蛋白质相互作用、核酸-蛋白质相互作用和蛋白质的基本功能[34-36]。对于翻译后修饰的研究,已有研究表明磷酸化会影响蛋白质的热稳定性[19]。还可以使用TPP检测蛋白质-蛋白质的相互作用,因为可以识别复合物中存在的蛋白质的稳定性变化,比如激酶抑制剂星形孢菌素稳定了含有细胞周期蛋白的激酶复合物[5]。为了评估蛋白质的功能,基因被敲除的细胞蛋白质组的热稳定性可能有助于深入了解蛋白质的机制。因为TPP的结果不仅可以看到哪些蛋白质稳定,还可以看到哪些蛋白质不稳定。当蛋白质复合物受到干扰或代谢物浓度降低时,就会发生不稳定。例如,帕比司他降低胆固醇水平就会导致载脂蛋白B的不稳定[23]。
3.3 其他方面的应用此外,TPP可以与补充蛋白质热稳定性方法的其他方法相结合。例如,最近的一项研究在裂解物环境中使用有限的蛋白质水解对熔解蛋白质组进行了分析,有助于深入了解序列水平的热解折叠[37]。结合单分子成像和热应力,可以进一步了解蛋白质的折叠和展开动力学。TPP也可用于酶的研究,尤其是耐热酶,Oztug等通过对嗜热杆菌ARTRW1的热蛋白质组学分析说明TPP在加速耐热酶研究和创新方面具有重要的工业和生物医学潜力[32, 38]。由于TPP位于蛋白质组学和代谢组学的边界,将来TPP应该还可以应用于研究许多其他生物学问题。
4 热蛋白质组学分析的优势及局限性TPP与当前很多原理相似的方法相比具有相对的优势,虽然在某些方面还存在一些不可避免的局限性,但随着技术的发展,该方法的局限范围在不断地缩小,针对于不同的局限性,研究人员都提出了不同的解决方式。
4.1 优势TPP在当前药物发现中的应用有相对的优势,当前的药物发现通常从使用基于靶标或基于表型的方法进行化合物筛选开始,其中许多策略使用基于质谱的蛋白组学,并且基于化合物结合后靶标稳定性的变化[39-40]。这些包括药物亲和力反应靶标稳定性技术(drug affinity responsive target stability, DARTS)、蛋白质氧化速率稳定性技术(stability of proteins from rates of oxidation, SPROX)、限制性蛋白水解(limited proteolysis, LiP)、羟自由基足迹法(hydroxyl radical footprinting, HRF) 和TPP等技术[4, 41-42]。其中TPP仍然是迄今为止唯一一种不需要对化合物进行修饰就可识别活细胞中细胞内靶标的方法[34, 43]。
除了识别直接靶点外,细胞中的TPP还可以揭示在直接药物靶点的任何调节活性下游已经改变稳定性的蛋白质。在大多数蛋白质复合物被破坏或减弱的裂解物中进行的TPP可以显示直接与化合物相互作用的蛋白质[44-45]。
TPP可以在体外、原位和体内进行[8]。TPP最初局限于体外细胞水平上,随着技术的发展,目前TPP已可在原位和体内中进行。原位使用TPP测量蛋白质组范围内的丰度和热稳定性可以了解蛋白质活性的全面变化。Selkrig等在原位使用TPP将其应用范围扩展到重要人类病原体的领域,并为SARS-CoV-2感染引起的分子变化提供更深入的解决方案[46]。在体内,Perrin等建立了组织热蛋白质组学分析(tissue-TPP) 和血液热蛋白质组学分析(blood-TPP) 的方法,揭示了帕比司他在大鼠组织和血液中的靶标谱[14]。近期Schirle用来自模型生物和患者的全血复杂组织样本进行了TPP方法的研究,首次证明TPP能够在药物发现的后期阶段,甚至在临床环境中实现器官特异性药物靶标的参与和识别研究[47]。
4.2 局限性虽然TPP越来越受欢迎,但基于原理它还是存在一些不可避免的局限性。首先该方法对膜蛋白检测困难,其次是不适用于热不敏感蛋白,而且不能显示蛋白结合位点[1]。TPP的方法依赖于用不含洗涤剂的缓冲液提取可溶性蛋白质,但许多配体结合受体和药物靶标是跨膜蛋白,而绝大多数跨膜蛋白是不可溶的,这是该方法的最大局限[48]。但目前已有很多研究表明在TPP中使用浓度为0.4%的温和去污剂NP-40足以溶解绝大多数的膜蛋白,同时不影响准确的蛋白质-药物亲和力的测定[11, 48]。Carnero等就利用TPP技术将吲哚素的靶点鉴定为与膜结合的嘌呤受体P2X4[49]。Kalxdorf等也介绍了用于全面表征配体诱导的蛋白质丰度和质膜热稳定性变化的细胞表面热蛋白质组学分析(cell surface thermal proteome profiling, CS-TPP)[50]。其实,TPP中不溶性蛋白质也可被分析,方法包括配体稳定化目标识别法(target identification by ligand stabilization, TILS)、微粒辅助沉淀筛选法(microparticle-assisted precipitation screening, MAPS) 和热蛋白组图谱沉淀支持测定法(precipitate-supported thermal proteome profiling, PSTPP)[51-53]。一些蛋白质需要在极端的温度条件下才会表现出热稳定性变化,还有一些蛋白质与配体结合后不会明显改变其热稳定性,这种情况下用TPP的方法不可能将这些蛋白质识别,但是可以从其抑制作用的下游效应来推断靶标[5, 11]。TPP是基于热稳定性的方法检测一般蛋白质性质的变化,因此无法揭示配体结合域的详细信息[1]。
TPP的方法中还存在一些硬件上的局限性,由于该方法是基于质谱的蛋白组学测定,因此仍然是一种低通量方法,并且缺乏对低丰度蛋白质检测的敏感度[54]。当前已有很多研究人员正在开发更快、更灵敏的质谱仪器以及新的采集模式,以满足对蛋白质组低丰度区域的研究[55]。近期Zinn等就使用稳定同位素标记试剂串联质量标签TMTpro实现了对多达16种条件下的蛋白质组样品同时分析的可能性[56-57]。
传统的TPP数据分析是通过比较有无配体结合蛋白的熔解曲线中的参数Tm的差异来确定靶蛋白的,这种数据处理方法往往会导致不必要的假阴性[58-59]。Childs等提出了一种替代方法,即响应曲线的非参数分析(nonparametric analysis of response curves, NPARC),它比较整个熔解曲线而不是汇总参数,并且不依赖于Tm,其敏感性和特异性都优于传统的方法[58]。Fang等建立了用于TPP数据分析的贝叶斯半参数模型,可对偏离预测的S形的蛋白质进行全面分析,已通过一系列已发表的数据发现了潜在的双相现象[60]。Kurzawa等也开发了一种针对2D-TPP数据进行错误发生率(false discovery rate, FDR) 控制分析的统计方法,显示出比定制阈值的传统方法更好的敏感性和特异性,该方法应用于先前发表的帕比司他的2D-TPP数据集中发现了新靶点[22, 24]。
| [1] |
Lyu JW, Wang KY, Ye ML. Modification-free approaches to screen drug targets at proteome level. Trac Trends Anal Chem, 2020, 124: 115574. DOI:10.1016/j.trac.2019.06.024
|
| [2] |
Santos R, Ursu O, Gaulton A, et al. A comprehensive map of molecular drug targets. Nat Rev Drug Discov, 2017, 16(1): 19-34. DOI:10.1038/nrd.2016.230
|
| [3] |
Ramsay RR, Popovic-Nikolic MR, Nikolic K, et al. A perspective on multi-target drug discovery and design for complex diseases. Clin Transl Med, 2018, 7(1): 3.
|
| [4] |
马婕, 刘强. 无化学修饰的小分子药物靶蛋白鉴定技术及其应用进展. 生物工程学报, 2021, 37(4): 1131-1138. Ma J, Liu Q. Identification techniques of small molecule drug target proteins without chemical modification and its applications: a review. Chin J Biotech, 2021, 37(4): 1131-1138 (in Chinese). DOI:10.13345/j.cjb.200426 |
| [5] |
Savitski MM, Reinhard FBM, Franken H, et al. Tracking cancer drugs in living cells by thermal profiling of the proteome. Science, 2014, 346(6205): 1255784. DOI:10.1126/science.1255784
|
| [6] |
Tan CSH, Go KD, Bisteau X, et al. Thermal proximity coaggregation for system-wide profiling of protein complex dynamics in cells. Science, 2018, 359(6380): 1170-1177. DOI:10.1126/science.aan0346
|
| [7] |
Martinez Molina D, Jafari R, Ignatushchenko M, et al. Monitoring drug target engagement in cells and tissues using the cellular thermal shift assay. Science, 2013, 341(6141): 84-87. DOI:10.1126/science.1233606
|
| [8] |
Mateus A, Kurzawa N, Becher I, et al. Thermal proteome profiling for interrogating protein interactions. Mol Syst Biol, 2020, 16(3): e9232.
|
| [9] |
Jafari R, Almqvist H, Axelsson H, et al. The cellular thermal shift assay for evaluating drug target interactions in cells. Nat Protoc, 2014, 9(9): 2100-2122. DOI:10.1038/nprot.2014.138
|
| [10] |
Jarzab A, Kurzawa N, Hopf T, et al. Meltome atlas-thermal proteome stability across the tree of life. Nat Methods, 2020, 17(5): 495-503. DOI:10.1038/s41592-020-0801-4
|
| [11] |
Mateus A, Bobonis J, Kurzawa N, et al. Thermal proteome profiling in bacteria: probing protein state in vivo. Mol Syst Biol, 2018, 14(7): e8242.
|
| [12] |
Ochoa D, Jarnuczak AF, Viéitez C, et al. The functional landscape of the human phosphoproteome. Nat Biotechnol, 2020, 38(3): 365-373. DOI:10.1038/s41587-019-0344-3
|
| [13] |
Dziekan JM, Yu H, Chen D, et al. Identifying purine nucleoside phosphorylase as the target of quinine using cellular thermal shift assay. Sci Transl Med, 2019, 11(473): eaau3174. DOI:10.1126/scitranslmed.aau3174
|
| [14] |
Perrin J, Werner T, Kurzawa N, et al. Identifying drug targets in tissues and whole blood with thermal-shift profiling. Nat Biotechnol, 2020, 38(3): 303-308. DOI:10.1038/s41587-019-0388-4
|
| [15] |
Corpas-Lopez V, Wyllie S. Utilizing thermal proteome profiling to identify the molecular targets of anti-leishmanial compounds. STAR Protoc, 2021, 2(3): 100704. DOI:10.1016/j.xpro.2021.100704
|
| [16] |
Herneisen AL, Lourido S. Thermal proteome profiling to identify protein-ligand interactions in the api complexan parasite Toxoplasma gondii. Bio-protocol, 2021, 11(21): e4207.
|
| [17] |
Mateus A, Hevler J, Bobonis J, et al. The functional proteome landscape of Escherichia coli. Nature, 2020, 588(7838): 473-478. DOI:10.1038/s41586-020-3002-5
|
| [18] |
Becher I, Andrés-Pons A, Romanov N, et al. Pervasive protein thermal stability variation during the cell cycle. Cell, 2018, 173(6): 1495-1507.e18. DOI:10.1016/j.cell.2018.03.053
|
| [19] |
Banzhaf M, Yau HC, Verheul J, et al. Outer membrane lipoprotein NlpI scaffolds peptidoglycan hydrolases within multi-enzyme complexes in Escherichia coli. EMBO J, 2020, 39(5): e102246.
|
| [20] |
Dai L, Zhao T, Bisteau X, et al. Modulation of protein-interaction states through the cell cycle. Cell, 2018, 173(6): 1481-1494.e13. DOI:10.1016/j.cell.2018.03.065
|
| [21] |
Herneisen AL, Sidik SM, Markus BM, et al. Identifying the target of an antiparasitic compound in Toxoplasma using thermal proteome profiling. ACS Chem Biol, 2020, 15(7): 1801-1807. DOI:10.1021/acschembio.0c00369
|
| [22] |
Becher I, Werner T, Doce C, et al. Thermal profiling reveals phenylalanine hydroxylase as an off-target of panobinostat. Nat Chem Biol, 2016, 12(11): 908-910. DOI:10.1038/nchembio.2185
|
| [23] |
Franken H, Mathieson T, Childs D, et al. Thermal proteome profiling for unbiased identification of direct and indirect drug targets using multiplexed quantitative mass spectrometry. Nat Protoc, 2015, 10(10): 1567-1593. DOI:10.1038/nprot.2015.101
|
| [24] |
Kurzawa N, Becher I, Sridharan S, et al. A computational method for detection of ligand-binding proteins from dose range thermal proteome profiles. Nat Commun, 2020, 11(1): 5783. DOI:10.1038/s41467-020-19529-8
|
| [25] |
Peuget S, Zhu JW, Sanz G, et al. Thermal proteome profiling identifies oxidative-dependent inhibition of the transcription of major oncogenes as a new therapeutic mechanism for select anticancer compounds. Cancer Res, 2020, 80(7): 1538-1550. DOI:10.1158/0008-5472.CAN-19-2069
|
| [26] |
Sridharan S, Kurzawa N, Werner T, et al. Proteome-wide solubility and thermal stability profiling reveals distinct regulatory roles for ATP. Nat Commun, 2019, 10(1): 1155. DOI:10.1038/s41467-019-09107-y
|
| [27] |
Leijten NM, Bakker P, Spaink HP, et al. Thermal proteome profiling in zebrafish reveals effects of napabucasin on retinoic acid metabolism. Mol Cell Proteomics, 2021, 20: 100033. DOI:10.1074/mcp.RA120.002273
|
| [28] |
Frantzi M, Latosinska A, Mischak H. Proteomics in drug development: the dawn of a new era. Proteomics Clin Appl, 2019, 13(2): e1800087. DOI:10.1002/prca.201800087
|
| [29] |
Carrasco Del Amor A, Freitas S, Urbatzka R, et al. Application of bioactive thermal proteome profiling to decipher the mechanism of action of the lipid lowering 13 2-hydroxy-pheophytin isolated from a marine cyanobacteria. Mar Drugs, 2019, 17(6): 371. DOI:10.3390/md17060371
|
| [30] |
Johnson FD, Ferrarone J, Liu A, et al. Characterization of a small molecule inhibitor of disulfide reductases that induces oxidative stress and lethality in lung cancer cells. Cell Rep, 2022, 38(6): 110343. DOI:10.1016/j.celrep.2022.110343
|
| [31] |
Goroshchuk O, Kolosenko I, Kunold E, et al. Thermal proteome profiling identifies PIP4K2A and ZADH2 as off-targets of Polo-like kinase 1 inhibitor volasertib. FASEB J, 2021, 35(7): e21741.
|
| [32] |
Oztug M, Kilinc E, Akgoz M, et al. Thermal proteome profiling and meltome analysis of a thermophilic bacterial strain, Geobacillus thermoleovorans ARTRW1: toward industrial applications. OMICS, 2020, 24(12): 756-765. DOI:10.1089/omi.2020.0115
|
| [33] |
Martin JK 2nd, Sheehan JP, Bratton BP, et al. A dual-mechanism antibiotic kills Gram-negative bacteria and avoids drug resistance. Cell, 2020, 181(7): 1518-1532.e14. DOI:10.1016/j.cell.2020.05.005
|
| [34] |
Mateus A, Määttä TA, Savitski MM. Thermal proteome profiling: unbiased assessment of protein state through heat-induced stability changes. Proteome Sci, 2017, 15: 13.
|
| [35] |
Peck Justice SA, Barron MP, Qi GD, et al. Mutant thermal proteome profiling for characterization of missense protein variants and their associated phenotypes within the proteome. J Biol Chem, 2020, 295(48): 16219-16238. DOI:10.1074/jbc.RA120.014576
|
| [36] |
Huang JX, Lee G, Cavanaugh KE, et al. High throughput discovery of functional protein modifications by hotspot thermal profiling. Nat Methods, 2019, 16(9): 894-901. DOI:10.1038/s41592-019-0499-3
|
| [37] |
Leuenberger P, Ganscha S, Kahraman A, et al. Cell-wide analysis of protein thermal unfolding reveals determinants of thermostability. Science, 2017, 355(6327): eaai7825. DOI:10.1126/science.aai7825
|
| [38] |
Türkowsky D, Lohmann P, Mühlenbrink M, et al. Thermal proteome profiling allows quantitative assessment of interactions between tetrachloroethene reductive dehalogenase and trichloroethene. J Proteomics, 2019, 192: 10-17. DOI:10.1016/j.jprot.2018.05.018
|
| [39] |
Swinney DC, Anthony J. How were new medicines discovered. Nat Rev Drug Discov, 2011, 10(7): 507-519. DOI:10.1038/nrd3480
|
| [40] |
Saxena C. Identification of protein binding partners of small molecules using label-free methods. Expert Opin Drug Discov, 2016, 11(10): 1017-1025. DOI:10.1080/17460441.2016.1227316
|
| [41] |
Ha J, Park H, Park J, et al. Recent advances in identifying protein targets in drug discovery. Cell Chem Biol, 2021, 28(3): 394-423. DOI:10.1016/j.chembiol.2020.12.001
|
| [42] |
Kaur U, Meng H, Lui F, et al. Proteome-wide structural biology: an emerging field for the structural analysis of proteins on the proteomic scale. J Proteome Res, 2018, 17(11): 3614-3627. DOI:10.1021/acs.jproteome.8b00341
|
| [43] |
Mateus A, Kurzawa N, Perrin J, et al. Drug target identification in tissues by thermal proteome profiling. Annu Rev Pharmacol Toxicol, 2022, 62: 465-482. DOI:10.1146/annurev-pharmtox-052120-013205
|
| [44] |
Saei AA, Gullberg H, Sabatier P, et al. Comprehensive chemical proteomics for target deconvolution of the redox active drug auranofin. Redox Biol, 2020, 32: 101491. DOI:10.1016/j.redox.2020.101491
|
| [45] |
Wilke J, Kawamura T, Xu H, et al. Discovery of a σ 1 receptor antagonist by combination of unbiased cell painting and thermal proteome profiling. Cell Chem Biol, 2021, 28(6): 848-854.e5. DOI:10.1016/j.chembiol.2021.01.009
|
| [46] |
Selkrig J, Stanifer M, Mateus A, et al. SARS-CoV-2 infection remodels the host protein thermal stability landscape. Mol Syst Biol, 2021, 17(2): e10188.
|
| [47] |
Schirle M. Characterizing drug-target interactions: shifting towards the clinic. Trends Pharmacol Sci, 2020, 41(5): 295-297. DOI:10.1016/j.tips.2020.03.001
|
| [48] |
Reinhard FBM, Eberhard D, Werner T, et al. Thermal proteome profiling monitors ligand interactions with cellular membrane proteins. Nat Methods, 2015, 12(12): 1129-1131. DOI:10.1038/nmeth.3652
|
| [49] |
Carnero Corrales MA, Zinken S, Konstantinidis G, et al. Thermal proteome profiling identifies the membrane-bound purinergic receptor P2X4 as a target of the autophagy inhibitor indophagolin. Cell Chem Biol, 2021, 28(12): 1750-1757.e5. DOI:10.1016/j.chembiol.2021.02.017
|
| [50] |
Kalxdorf M, Günthner I, Becher I, et al. Cell surface thermal proteome profiling tracks perturbations and drug targets on the plasma membrane. Nat Methods, 2021, 18(1): 84-91. DOI:10.1038/s41592-020-01022-1
|
| [51] |
Peng H, Guo HB, Pogoutse O, et al. An unbiased chemical proteomics method identifies FabI as the primary target of 6-OH-BDE-47. Environ Sci Technol, 2016, 50(20): 11329-11336. DOI:10.1021/acs.est.6b03541
|
| [52] |
Lyu JW, Ruan CF, Zhang XL, et al. Microparticle- assisted precipitation screening method for robust drug target identification. Anal Chem, 2020, 92(20): 13912-13921. DOI:10.1021/acs.analchem.0c02756
|
| [53] |
Ruan CF, Ning WS, Liu Z, et al. Precipitate-supported thermal proteome profiling coupled with deep learning for comprehensive screening of drug target proteins. ACS Chem Biol, 2022, 17(1): 252-262. DOI:10.1021/acschembio.1c00936
|
| [54] |
Peck Justice SA, McCracken NA, Victorino JF, et al. Boosting detection of low-abundance proteins in thermal proteome profiling experiments by addition of an isobaric trigger channel to TMT multiplexes. Anal Chem, 2021, 93(18): 7000-7010. DOI:10.1021/acs.analchem.1c00012
|
| [55] |
Meier F, Geyer PE, Virreira Winter S, et al. BoxCar acquisition method enables single-shot proteomics at a depth of 10 000 proteins in 100 minutes. Nat Methods, 2018, 15(6): 440-448. DOI:10.1038/s41592-018-0003-5
|
| [56] |
Thompson A, Wölmer N, Koncarevic S, et al. TMTpro: design, synthesis, and initial evaluation of a proline-based isobaric 16-plex tandem mass tag reagent set. Anal Chem, 2019, 91(24): 15941-15950. DOI:10.1021/acs.analchem.9b04474
|
| [57] |
Zinn N, Werner T, Doce C, et al. Improved proteomics-based drug mechanism-of-action studies using 16-plex isobaric mass tags. J Proteome Res, 2021, 20(3): 1792-1801. DOI:10.1021/acs.jproteome.0c00900
|
| [58] |
Childs D, Bach K, Franken H, et al. Nonparametric analysis of thermal proteome profiles reveals novel drug-binding proteins. Mol Cell Proteomics, 2019, 18(12): 2506-2515. DOI:10.1074/mcp.TIR119.001481
|
| [59] |
McCracken NA, Peck Justice SA, Wijeratne AB, et al. Inflect: optimizing computational workflows for thermal proteome profiling data analysis. J Proteome Res, 2021, 20(4): 1874-1888. DOI:10.1021/acs.jproteome.0c00872
|
| [60] |
Fang SQ, Kirk PDW, Bantscheff M, et al. A Bayesian semi-parametric model for thermal proteome profiling. Commun Biol, 2021, 4(1): 810. DOI:10.1038/s42003-021-02306-8
|
 2022, Vol. 38
2022, Vol. 38