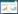中国科学院微生物研究所、中国微生物学会主办
文章信息
- 尹业师, 陈浒, 张美红, 曹林艳, 陈华海
- YIN Yeshi, CHEN Hu, ZHANG Meihong, CAO Linyan, CHEN Huahai
- 人体肠道4 644株代表菌次级代谢产物生物合成基因簇挖掘与耐药及毒力基因分析
- Mining of gene clusters for biosynthesis of secondary metabolites and analysis of genes encoding antibiotic resistance and virulence in 4 644 representative human gut strains
- 生物工程学报, 2022, 38(10): 3682-3694
- Chinese Journal of Biotechnology, 2022, 38(10): 3682-3694
- 10.13345/j.cjb.220220
-
文章历史
- Received: March 21, 2022
- Accepted: June 22, 2022
- Published: June 27, 2022
2. 中国农业大学 动物科学技术学院 动物营养学国家重点实验室, 北京 100193
2. State Key Laboratory of Animal Nutrition, College of Animal Science and Technology, China Agricultural University, Beijing 100193, China
天然产物,尤其是微生物次级代谢产物一直是药物先导化合物的重要来源。在过去几十年,超过一半的新型小分子药物来源于微生物天然产物。微生物天然产物具有巨大的结构和化学多样性及各种各样的生物特性。然而,值得注意的是无法培养菌株和沉默基因簇等所带来的限制越来越大,传统自上而下的筛选方法发现新的微生物天然产物变得越来越困难。采用先进的分析方法并以生物信息学分析为指导的自下而上的靶向筛选方法为前所未有的规模化解锁微生物代谢组提供了一种有希望的替代方法[1]。哺乳动物肠道中居住着大量微生物,人体肠道微生物组编码的基因数量是人类基因组所编码基因数量的400多倍[2],这将是一个挖掘候选新药的重要资源宝库[3]。
人体肠道菌群大多数为共生微生物。它们有助于人体宿主的基本功能和生理发育。相反,少部分病原微生物可以通过表达毒力因子,微调宿主反应,从而有效感染宿主并导致疾病。毒力因子是在细胞水平上帮助细菌定殖宿主的分子,包括分泌性的、膜相关的和细胞质性的。分泌因子是细菌武器库的重要组成部分,帮助细菌逃脱宿主体内的先天性和适应性免疫反应。膜相关毒力因子有助于细菌粘附和逃避宿主排斥。胞质因子促进细菌快速适应代谢、生理和形态变化[4]。因此,对毒力因子的研究不仅可以更好地了解病原微生物的感染和致病机制,也可以作为疾病防治的重要靶标。但到目前为止,除了对大肠杆菌[5]和结核分枝杆菌[4]等常见病原菌的毒力因子有较系统的研究外,肠道中丰富多样的微生物所编码毒力因子的种类和功能还有待进一步研究。
随着抗生素的过度使用和滥用,全球抗生素耐药性日趋严重,许多致病菌对主要类别的抗生素均产生了耐药,导致无法治疗的感染急剧增加,从而出现了所谓的“超级细菌”。因此,抗生素耐药对公共卫生构成了紧迫且日益严重的威胁。多重耐药细菌的出现和传播严重威胁着人类健康。估计到2050年,每年由抗生素耐药引起的死亡人数将超过1 000万[6]。人体肠道微生物是一个抗生素耐药基因混合池,分析和研究肠道微生物组编码的耐药基因情况,将为更好认识耐药基因的流行情况及指导抗生素有效使用提供参考。
随着测序技术的不断发展和生物信息学分析方法的不断更新,科学家们可以不受微生物体外培养的限制,更好地认识和分析肠道菌群组成。为了建立一个统一的人类肠道微生物基因组非冗余数据集,Almeida等收集了大量从人类胃肠道分离获得的细菌基因组序列和人体肠道微生物宏基因组数据,通过拼接和筛选获得了286 997个肠道微生物参考基因组[7]。为了进一步确定肠道微生物参考基因组数据库中包含多少物种,Almeida等使用基于距离的多步法对所有基因组进行了聚类,在至少30%的对齐分数,平均核苷酸同一性(average nucleotide identity, ANI) 大于95%的分析标准下,共鉴定到4 644个原核物种(4 616种细菌和28种古菌)[7]。人体肠道微生物代表性菌株的获得将为更好地认识肠道菌群及简化生物信息学分析奠定基础。本研究将基于这4 644个代表性菌株的基因组信息对其编码次级代谢产物情况、编码抗生素耐药基因情况和编码毒力因子情况进行预测分析。
1 材料与方法 1.1 材料从美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI) 下载人体肠道微生物4 644株代表性菌株[7]的基因组序列。
1.2 基因组注释在标准模式下使用快速原核基因组注释工具(Prokka v1.14.6) 对基因组序列进行注释[8]。
1.3 次级代谢产物生物合成基因簇预测结合基因组注释文件,使用基因组次级代谢产物分析工具antiSMASH v6.0.0 (其数据库已更新到最新版antiSMASH database v3) 进行基因簇编码基因预测[9]。经antiSMASH分析,从注释结果中提取次级代谢产物合成基因簇核心基因蛋白质序列,用于后续序列相似性比对分析。使用比对搜索工具BLASTP将基因簇中的核心基因蛋白质序列与NCBI中的非冗余蛋白序列数据库(non-redundant) 进行序列比对,以一致性30%为阈值对比对结果进行筛选和分类。
1.4 潜在新次级代谢产物生物合成基因簇鉴定在核心基因相似性分析基础上,对核心基因蛋白质序列相似性低于30%的基因簇,使用在线版antiSMASH (V6.0.0) 对其整个生物合成基因簇与antiSMASH数据库(v3) 和MIBiG标准数据库进行基因簇相似性比对,进一步筛选相似性均少于30%的基因簇被认定为潜在的新生物合成基因簇。
1.5 毒力因子预测使用最新发布的PathoFact (v1.0) 对抗生素耐药基因、毒素和毒力因子进行预测分析[10]。
1.6 Circos图绘制使用R软件circlize安装包对代表性菌株与耐药基因型之间的数量关系进行Circos图绘制。
2 结果与分析 2.1 4 644株代表菌的分类与组成分析4 644株代表菌有20株属于尚未被分类的新细菌门,其他4 624株分布在15个已知的细菌门和1个古菌门,其中54%的代表菌株分布在厚壁菌门(Firmicutes),其他菌株分布较多的细菌门分别为放线菌门(Actinobacteria) (18%)、拟杆菌门(Bacteroidea) (13%) 和变形菌门(Proteobacteria) (10%)。在细菌属的水平,3 033株代表菌被注释到319个细菌属,其他1 611个代表菌株未能在细菌属水平进行分类。含量较高的10个细菌属分别为柯林斯氏菌属(Collinsella,580株代表菌)、梭菌属(Clostridium,273株代表菌)、普雷沃氏菌属(Prevotella,126株代表菌)、拟杆菌属(Bacteroides,107株代表菌)、瘤胃球菌属(Ruminococcus,67株代表菌)、另枝杆菌属(Alistipes,66株代表菌)、优杆菌属(Eubacterium,65株代表菌)、链球菌属(Streptococcus,55株代表菌)、乳杆菌属(Lactobacillus,54株代表菌) 和萨特氏菌属(Sutterella,43株代表菌)。在细菌种的水平,只有约20%的代表菌株(933株) 被注释到734个细菌种,其中分布到产气柯林斯菌(Collinsella aerofaciens)、简明弯曲菌(Campylobacter concisus) 和普拉梭菌(Faecalibacterium prausnitzii) 的菌株数较多,分别为61株、12株和10株。根据Almeida等的分析发现,894株代表菌的基因组序列与人肠道来源的可培养菌具有大于95%的同源性,3 312株仍属于尚未可培养细菌[7]。
为进一步鉴定4 644代表菌株中哪些可能是具有传染性的病原微生物,参照中华人民共和国国家卫生健康委员会官网发布的“人间传染的病原微生物目录(征求意见稿)” (http://www.nhc.gov.cn/qjjys/s3590/202112/94fcc4480ea2403e9c51c641645d6c20.shtml),从被注释到的734种细菌中检测到50种80个代表菌株为潜在的病原微生物。包括鲍氏不动杆菌(Acinetobacter baumannii)、鲁氏不动杆菌(Acinetobacter lwoffi)、嗜水气单胞菌(Aeromonas hydrophila)、蜡状芽孢杆菌(Bacillus cereus)、脆弱拟杆菌(Bacteroides fragilis)、短螺旋体属(Brachyspira)、大肠弯曲菌(Campylobacter coli)、胎儿弯曲菌(Campylobacter fetus)、空肠弯曲菌(Campylobacter jejuni)、艰难梭菌(Clostridioides difficile)、肉毒杆菌(Clostridium botulinum)、产气荚膜梭菌(Clostridium perfringens)、破伤风梭菌(Clostridium tetani)、纹带棒杆菌(Corynebacterium striatum)、迟缓爱德华氏菌(Edwardsiella tarda)、侵蚀艾肯菌(Eikenella corrodens)、产气肠杆菌/阴沟肠杆菌(Enterobacter aerogenes/Enterobacter cloacae)、肠杆菌(Enterobacter spp.)、坏死梭杆菌(Fusobacterium necrophorum)、阴道嗜血杆菌(Gardnerella vaginalis)、流感嗜血杆菌(Haemophilus influenzae)、幽门螺旋杆菌(Helicobacter pylori)、产酸克雷伯氏菌(Klebsiella oxytoca)、李斯特氏菌(Listeria monocytogenes)、摩氏摩根菌(Morganella morganii)、鸟分枝杆菌(Mycobacterium avium)、淋病奈瑟菌(Neisseria gonorrhoeae)、其他分枝杆菌(other Mycobacterium species)、索氏梭菌(Paeniclostridium sordellii)、大肠杆菌(Escherichia coli)、厌氧消化链球菌(Peptostreptococcus anaerobius)、类志贺邻单胞菌(Plesiomonas shigelloides)、普雷沃氏菌(Prevotella spp.)、奇异变形杆菌(Proteus mirabilis)、彭氏变形菌(Proteus penneri)、普通变形杆菌(Proteus vulgaris)、产碱普罗威登斯菌(Providencia alcalifaciens)、雷氏普罗威登斯菌(Providencia rettgeri)、铜绿假单胞菌(Pseudomonas aeruginosa)、沙门氏菌(Salmonella)、液化沙雷氏菌(Serratia liquefaciens)、粘质沙雷氏菌(Serratia marcescens)、金黄色葡萄球菌(Staphylococcus aureus)、表皮葡萄球菌(Staphylococcus epidermidis)、链球菌(Streptococcus spp.)、非霍乱弧菌(non-cholerae Vibrio spp.)、创伤弧菌(Vibrio vulnificus)、小肠结肠炎耶尔森菌(Yersinia enterocolitica)、分枝结核杆菌(Mycobacterium tuberculosis)、霍乱弧菌(Vibrio cholerae)、鼠疫耶尔森菌(Yersinia pestis)。“人间传染的病原微生物目录”将病原微生物按危害程度由高到低分为第四类、第三类、第二类、第一类,除Mycobacterium tuberculosis、Vibrio cholerae、Yersinia pestis属于第三类外,其他肠道代表菌中潜在的传染性病原微生物均属于第二类。
2.2 4 644株代表菌编码的次级代谢产物分析使用antiSMASH预测分析发现,4 644株代表菌中有2 800株含有至少1个编码次级代谢产物的基因簇,共预测到5 766个基因簇。其中24株代表菌的基因组中预测发现含有10个及以上编码次级代谢产物的基因簇(表 1)。在细菌门的水平,厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria) 和疣微菌门(Verrucomicrobia) 编码较多次级代谢产物基因簇,分别预测到3 793、1 109、363、308和139条编码次级代谢产物的基因簇。在细菌纲的水平,梭菌纲(Clostridia)、芽孢杆菌纲(Bacilli)、γ-变形菌纲(Gammaproteobacteria)、拟杆菌纲(Bacteroidia)、放线菌纲(Actinobacteria)、厚壁菌纲(Negativicutes)、β-变形菌纲(Betaproteobacteria)、α-变形菌纲(Alphaproteobacteria) 分别编码2 559、818、744、350、255、170、157、111条基因簇。从代谢产物类型来看,分别有1 210、898、587、481、390、337、291、257、224、206条基因簇编码塞克肽类、非核糖体多肽合酶、细菌素、芳基多烯类化合物、萜烯、β-丙内酯、NRPS-like、内酯类自诱导剂、羊毛硫肽化合物、Ⅲ型聚酮合酶等次级代谢产物。具体肠道菌群在纲水平与微生物编码的次级代谢产物类型之间的对应关系如图 1所示。肠道古菌编码的次级代谢产物生物合成基因簇数量较少。肠道细菌编码的次级代谢产物生物合成基因簇主要由梭菌纲(Clostridia)、芽孢杆菌纲(Bacilli)、γ-变形菌纲(Gammaproteobacteria)、拟杆菌纲(Bacteroidia)、放线菌纲(Actinobacteria) 和厚壁菌纲(Negativicutes) 6类细菌编码的非核糖体多肽合成酶、细菌素、芳基多烯类化合物、萜烯、β-丙内酯、NRPS-样酶组成。
| Species representative | Taxonomy | Status | Cluster No. |
| GUT_GENOME239690 | s__Paenibacillus polymyxa | Cultured (human gut) | 24 |
| GUT_GENOME140361 | s__Mycobacterium avium | Cultured (human gut) | 23 |
| GUT_GENOME001364 | s__Bacillus sonorensis | Cultured (human gut) | 20 |
| GUT_GENOME096381 | g__Streptomyces | Cultured (human gut) | 20 |
| GUT_GENOME143709 | s__Paenibacillus thiaminolyticus | Cultured (human gut) | 20 |
| GUT_GENOME143383 | s__Pseudomonas aeruginosa | Cultured (human gut) | 17 |
| GUT_GENOME141727 | s__Bacillus wiedmannii | Cultured (human gut) | 16 |
| GUT_GENOME000711 | s__Bacillus paralicheniformis | Cultured (human gut) | 15 |
| GUT_GENOME207869 | g__Bradyrhizobium | Cultured (unknown) | 15 |
| GUT_GENOME000031 | s__Bacillus subtilis | Cultured (human gut) | 14 |
| GUT_GENOME000295 | s__Roseburia intestinalis | Cultured (human gut) | 14 |
| GUT_GENOME095592 | g__Pseudomonas | Cultured (unknown) | 14 |
| GUT_GENOME096372 | g__Paenibacillus | Cultured (human gut) | 14 |
| GUT_GENOME141763 | s__Mycobacterium tuberculosis | Cultured (human gut) | 14 |
| GUT_GENOME171359 | g__Paenibacillus | Uncultured | 14 |
| GUT_GENOME200288 | s__Paenibacillus amylolyticus | Cultured (unknown) | 14 |
| GUT_GENOME001578 | g__Clostridium | Cultured (human gut) | 12 |
| GUT_GENOME243082 | g__Bradyrhizobium | Cultured (unknown) | 12 |
| GUT_GENOME001770 | s__Roseburia inulinivorans | Cultured (human gut) | 11 |
| GUT_GENOME096513 | s__Paenibacillus ihumii | Cultured (human gut) | 11 |
| GUT_GENOME141425 | s__Serratia marcescens | Cultured (human gut) | 11 |
| GUT_GENOME213384 | s__Pseudomonas extremaustralis | Cultured (unknown) | 11 |
| GUT_GENOME096393 | s__Aneurinibacillus aneurinilyticus | Cultured (human gut) | 10 |
| GUT_GENOME232192 | g__Serratia | Cultured (human) | 10 |
|
| 图 1 4 644株代表菌可能产生的次级代谢产物预测与分析 Fig. 1 Prediction and analysis of secondary metabolites that might be produced by 4 644 representative strains. |
| |
对预测发现的基因簇进一步进行核心基因的BLASTp同源性比对分析发现,有9条基因簇的核心基因其同源性与数据库中已有基因的氨基酸序列同源性低于30%,推测可能编码新颖次级代谢产物。在整个基因簇的水平再次进行同源性分析发现,除代表菌株GUT_GENOME157951编码的羊毛硫肽化合物基因簇与优杆菌(Eubacteriaceae bacterium) NZ_CABIWW010000011具有大于30%的同源性外,其他8条基因簇均与已知基因簇的同源性低于30%。从表 2可以看出,这9条基因簇除了GUT_ GENOME187394曾从未知环境中分离获得过培养菌外,其他8株菌仍属于未被分离培养的微生物。
| No. | Cluster length (bp) | Code genes | Product | Species representative | Taxonomy | Status |
| Cluster 1 | 9 873 | 6 | NRPS | GUT_GENOME064659 | f__Lachnospiraceae | Uncultured |
| Cluster 2 | 57 398 | 38 | NRPS/T1PKS /NRPS-like |
GUT_GENOME104570 | f__Ruminococcaceae | Uncultured |
| Cluster 3 | 24 204 | 35 | NRPS | GUT_GENOME112856 | g__Prevotella | Uncultured |
| Cluster 4 | 19 673 | 10 | NRPS/NRPS-like | GUT_GENOME121457 | g__Eubacterium | Uncultured |
| Cluster 5 | 22 228 | 25 | Ranthipeptide | GUT_GENOME157951 | o__Clostridiales | Uncultured |
| Cluster 6 | 22 908 | 23 | Lanthipeptide | GUT_GENOME187394 | s__Peptoniphilus lacydonensis | Cultured (unknown) |
| Cluster 7 | 26 044 | 20 | NRPS | GUT_GENOME233270 | f__Lachnospiraceae | Uncultured |
| Cluster 8 | 2 071 | 2 | NRPS | GUT_GENOME233276 | f__Porphyromonadaceae | Uncultured |
| Cluster 9 | 5 584 | 8 | Other: LmbU | GUT_GENOME234141 | f__Akkermansiaceae | Uncultured |
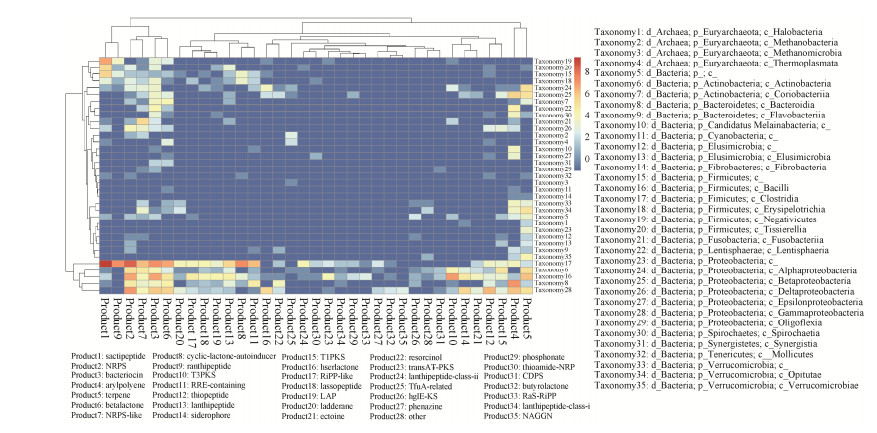
使用PathoFact整合的抗生素耐药基因预测功能对4 644株代表性菌株编码的抗生素耐药基因进行分析。结果发现所有4 644株肠道代表菌共编码30 675个ARGs,平均每个代表菌株编码6–7个ARGs,被分类到31个抗生素耐药基因型。图 2展示了各代表菌株所编码的抗生素耐药基因型与细菌分类之间的对应关系。28个代表性古菌共编码16个ARGs,平均每个菌编码ARGs的概率较低;81个潜在的传染性病原微生物共编码4 213个ARGs,平均每个细菌编码50–51个ARGs,编码ARGs的概率较高;其他4 535个代表菌株共编码26 446个ARGs,平均每个细菌编码5–6个ARGs,编码ARG的概率较潜在的传染性病原微生物低10倍。如表 3所示,每个潜在病原菌编码的多重耐药、多肽、β-内酰胺、氨基糖苷类ARGs数量较多,潜在病原菌中每个细菌平均编码四烯霉素、氨基香豆素、氨基糖苷(氨基香豆素)、艾法霉素、磺胺、抗菌游离脂肪酸的ARGs数量比非病原菌高20倍以上。
|
| 图 2 代表菌株编码的抗生素耐药基因分布情况 Fig. 2 The prevalence of different resistance gene categories within the representative species. |
| |
| Type of antibiotic resistant genes | Non-pathogen | Pathogen | Pathogen/non-pathogen |
| Multidrug | 1.95 | 15.60 | 8.01 |
| Peptide | 0.43 | 7.06 | 16.42 |
| β-lactam | 0.42 | 4.96 | 11.95 |
| Aminoglycoside | 0.28 | 4.91 | 17.52 |
| Unclassified | 0.34 | 3.06 | 8.95 |
| Fuoroquinolone | 0.27 | 2.80 | 10.24 |
| MLS | 0.70 | 2.20 | 3.16 |
| Aminoglycoside: aminocoumarin | 0.08 | 2.10 | 27.83 |
| Bacitracin | 0.12 | 1.90 | 15.82 |
| Tetracycline | 0.23 | 1.88 | 8.04 |
| Aminocoumarin | 0.04 | 1.47 | 35.25 |
| Elfamycin | 0.03 | 0.78 | 26.13 |
| Fosfomycin | 0.12 | 0.78 | 6.63 |
| Phenicol | 0.06 | 0.67 | 11.58 |
| Glycopeptide | 0.65 | 0.58 | 0.89 |
| Nitroimidazole | 0.03 | 0.46 | 16.84 |
| Sulfonamide | 0.01 | 0.26 | 22.61 |
| Triclosan | 0.01 | 0.12 | 13.02 |
| Diaminopyrimidine | 0.02 | 0.12 | 6.51 |
| Rifamycin | 0.02 | 0.11 | 5.48 |
| Antibacterial free fatty acids | 0.00 | 0.06 | 21.53 |
| Nucleoside | 0.01 | 0.05 | 5.21 |
| Mupirocin | 0.01 | 0.02 | 2.80 |
| Tetracenomycin_C | 0 | 0.01 | 55.99 |
| Bicyclomycin | 0 | 0.01 | 8.00 |
| Polymyxin | 0 | 0.01 | 8.00 |
| Fusidic-acid | 0 | 0.01 | 7.00 |
| Acridine_dye | 0 | 0 | 0 |
| Pleuromutilin | 0 | 0 | 0 |
| Polyamine: peptide | 0 | 0 | 0 |
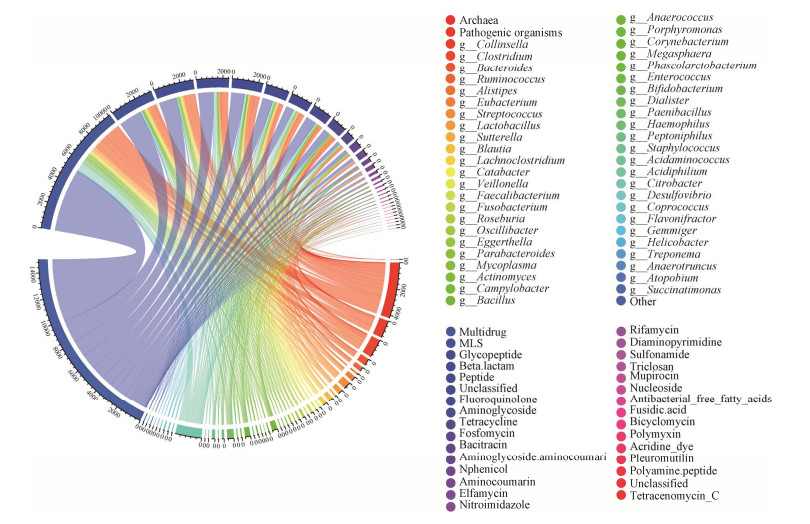
使用PathoFact对4 644株代表性细菌编码的毒力因子和毒素进行分析。如图 3所示,所有4 644个菌株编码的参考基因数量差异较大,最少的只编码425个参考基因,最多的编码9 054个参考基因,平均每个菌株编码2 154个基因。其中平均5.26%、12.24%和49.34%的基因分别编码分泌型毒力因子、非分泌型毒力因子和潜在毒力因子。
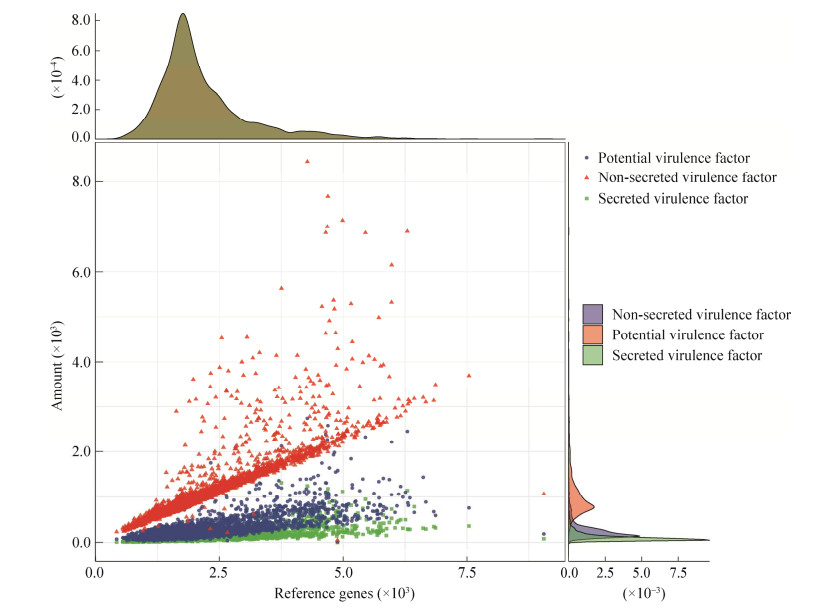
|
| 图 3 代表菌株参考基因数目和编码毒力因子数量 Fig. 3 Number of reference genes and virulence factors encoded in the representative strains. |
| |
所有4 644个肠道代表菌株共编码56 096个分泌型毒素(211个毒素类型) 和156 446个非分泌型毒素(302个毒素类型),平均每个代表菌株编码12个分泌型毒素和34个非分泌型毒素。81个潜在传染性病原微生物共编码8 359个分泌型毒素(平均每个细菌编码103个) 和10 899非分泌型毒素(平均每个细菌编码135个)。4 535个非病原菌共编码47 674个分泌型毒素(平均每个细菌编码11个) 和144 914非分泌型毒素(平均每个细菌编码32个),编码分泌型毒素和非分泌型毒素的概率均明显低于潜在传染性病原微生物。如表 4所示,平均每个潜在传染性病原微生物中编码5个以上外膜蛋白、PapC N端结构域、PapC C端结构域、肽酶M16非活性结构域等分泌型毒素。尽管潜在传染性病原微生物编码的非分泌型毒素的数量平均只是非病原菌的4倍左右,但如表 5所示,平均每个潜在传染性病原微生物编码5个以上硝基还原酶家族、AcrB/AcrD/AcrF家族、PLD样结构域、Cupin结构域、假定溶血素、肽酶S24样蛋白、磷酸转移酶家族、核酸内切酶/核酸外切酶/磷酸酶家族、乙二醛酶/博莱霉素抗性等非分泌型毒素。值得注意的是,在分泌型毒素中,尽管每个非病原菌编码的外膜蛋白基因簇数量明显比潜在病原菌低,但每个非病原菌可以编码约2个外膜蛋白基因簇。在非分泌型毒素中,虽然非病原菌与潜在病原菌之间的差异比分泌型毒素小,但每个非病原菌基因组序列中预测到可以编码约2个肽酶S24样蛋白和3个硝基还原酶家族基因。
| Secretory toxins | Non-pathogen | Pathogen | Pathogen / non-pathogen |
| AcrB/AcrD/AcrF family | 0.45 | 3.25 | 7.14 |
| Endonuclease/Exonuclease/Phosphatase family | 0.43 | 3.05 | 7.07 |
| Gram-negative porin | 0.21 | 3.31 | 15.50 |
| Hyaluronoglucosaminidase | 0.31 | 3.07 | 9.83 |
| Insulinase | 0.30 | 4.90 | 16.07 |
| Outer membrane protein | 1.77 | 14.91 | 8.44 |
| PapC C-terminal domain | 0.15 | 6.20 | 41.27 |
| PapC N-terminal domain | 0.17 | 7.20 | 41.63 |
| Peptidase C10 family | 0.16 | 3.51 | 22.24 |
| Peptidase M16 inactive domain | 0.35 | 5.25 | 15.01 |
| Presequence protease | 0.30 | 4.35 | 14.56 |
| Sialidase-1 | 0.21 | 2.07 | 9.90 |
| Streptopain | 0.26 | 6.65 | 26.04 |
| Non-secretory toxins | Non-pathogen | Pathogen | Pathogen/non-pathogen |
| Nitroreductase family | 2.85 | 9.95 | 3.50 |
| AcrB/AcrD/AcrF family | 1.35 | 9.62 | 7.15 |
| PLD-like domain | 1.54 | 7.99 | 5.17 |
| Cupin domain | 1.67 | 7.70 | 4.62 |
| putative hemolysin | 1.25 | 6.56 | 5.25 |
| Peptidase S24-like | 2.31 | 6.52 | 2.82 |
| Phosphotransferase enzyme family | 1.53 | 6.43 | 4.20 |
| Endonuclease/exonuclease/phosphatase family | 1.28 | 6.28 | 4.92 |
| Glyoxalase/bleomycin resistance protein/dioxygenase superfamily | 1.02 | 5.73 | 5.63 |
| Peptidase M16 inactive domain | 1.43 | 3.22 | 2.26 |
| UDP-glucose 4-epimerase C-term subunit | 0.92 | 2.80 | 3.04 |
| Presequence protease | 1.01 | 2.77 | 2.74 |
| Insulinase (peptidase family M16) | 0.75 | 2.68 | 3.55 |
| Insecticidal toxin complex protein TccC | 0.17 | 2.57 | 14.74 |
| Hemolysin Ⅲ | 0.63 | 2.07 | 3.31 |
虽然已有多个研究团队对人体肠道微生物进行了培养组学研究[11-14],但基因组同源性分析表明,从人体肠道中拼接获得的4 644株代表菌仍有71%属于未可培养菌[7]。尽管高通量测序和大数据生物信息学分析为我们认识肠道菌群的功能及其与疾病的相关性创造了条件。但最近Tierney等使用数百万种不同的关联建模策略,评估了肠道菌群-疾病关联的一致性,涉及6种疾病、15个队列和2 343名个体[15]。很多情况下,对于同一个分类群-疾病的配对,不同的建模产生了相互矛盾的关联结果。大量潜在的混杂因素(如测序深度、血糖水平、胆固醇、身体质量指数等) 和DNA提取方法、生物信息学分析流程等均可能影响菌群关联研究的结果[15]。通过与“人间传染的病原微生物目录”比对,在4 644株代表菌中找到81株是潜在传染性微生物,但也很难排除其他条件致病菌和非传染性病原微生物。因此,分离培养与疾病相关菌株,并遵从“科赫法则”,对其在疾病发生发展中的作用进行验证十分必要。目前已经基本公认,人体肠道菌群分为拟杆菌肠型、普雷沃氏菌肠型和瘤胃球菌肠型,拟杆菌和普雷沃氏菌是人体肠道中的优势菌群[16-17]。但分析发现,柯林斯氏菌(Collinsella) 和梭菌(Clostridium) 的菌株多样性高于拟杆菌和普雷沃氏菌,其生物学意义尚有待进一步研究。
肠道菌群是一个有待挖掘的资源宝库,其编码的蛋白质、酶、多肽和合成的次级代谢产物等具有非常重要的生理功能[18-20]。最近,中国科学院微生物研究所王军团队与陈义华团队合作,使用人工智能方法从人体肠道微生物组数据中挖掘出了181个新型抗菌肽,其中10多种新型抗菌肽在体内外对多重耐药菌表现出较好的抑制作用[21]。本研究从未培养菌中预测到了8个与次级代谢产物基因簇同源性低于30%的基因簇,主要编码NRPS。通过细菌分离和合成生物学异源表达等进一步对这些次级代谢产物进行分离和鉴定,将为发现新型NRPS提供指导。另外,肠道菌群也参与饮食代谢,其转化代谢的产物对维持宿主健康具有非常重要的作用。越来越多的证据表明,肠道微生物衍生代谢产物氧化三甲胺(trimethylamine oxide, TMAO) 在许多疾病的发病机制中起着至关重要的作用,可以作为多种心血管疾病的预后生物标志物[22]。由于肠道环境中代谢网络的复杂性,肠道微生物自身分泌的次级代谢产物及参与代谢产生的代谢产物还有待进一步研究。
抗生素耐药已经成为全球关注的公共卫生问题,肠道中由于微生物数量多,接触抗生素的频率较高,已被认为是抗生素耐药基因产生和传播的重要混合池[23-25]。而且抗生素耐药基因不仅会影响细菌的耐药性,也可能与疾病的发生发展相关。最近研究报道,肠道抗生素抗性组的结构在健康人、糖尿病前期和2型糖尿病患者中存在显著差异,抗生素抗性组的多样性与2型糖尿病的发生发展呈正相关[26]。通过对人体肠道4 644株代表菌株的耐药基因进行预测发现,古菌编码的耐药基因较少,潜在传染性病原微生物编码的耐药基因明显高于非传染性病原微生物,这也是导致目前临床上需要开发新型抗生素来对抗耐药病原菌的重要原因之一。同时,这些数据也暗示我们,通过监测抗生素耐药基因的丰度也可能在一定程度上反映病原微生物的数量。
毒力因子顾名思义与致病性有较大关联,它有助于致病微生物能够在宿主生态位定居,最终导致组织损伤以及局部和全身炎症。这些因素对病原体建立感染很重要,从而直接或间接地影响疾病进程[27]。尽管科学家们对病原微生物的毒力因子已关注较多,但对非病原微生物及人体肠道菌群毒力因子的认识和研究还有待进一步加强。本研究对人体肠道4 644株代表菌株编码的毒力因子和毒素进行了较全面的分析和统计,结果发现平均每个代表菌株编码12个分泌型毒素和34个非分泌型毒素。虽然对分泌型毒素研究相对较多,在致病过程中可能发挥更直接的作用,但对非分泌型毒素的功能研究还有待进一步加强。尽管在潜在传染性病原微生物基因组中能预测到更多的编码毒素的基因,但有些非传染性病原微生物中也能预测到较多的毒素编码基因。这些非病原微生物编码的毒素基因是否与正常肠道菌群促进免疫系统发育成熟和调控免疫平衡等有关仍有待进一步研究。
总之,通过对人体肠道中4 644株代表菌进行分析发现,人体肠道中70%以上的菌株仍属于未可培养菌,尽管拟杆菌属和普雷沃氏菌属细菌在人体肠道中含量较高,但柯林斯氏菌(Collinsella) 和梭菌(Clostridium) 可能具有更丰富的多样性。超过60%的代表菌株编码至少1个次级代谢产物基因簇,并从8个未可培养菌中鉴定到了8个与已有基因簇同源性低于30%的潜在新颖次级代谢产物基因簇。抗生素耐药基因和毒力因子在代表性菌株中分布广泛,但潜在传染性病原微生物编码的抗生素耐药基因和毒力因子频率显著高于非传染性病原微生物。
| [1] |
Sukmarini L. Recent advances in discovery of lead structures from microbial natural products: genomics- and metabolomics-guided acceleration. Molecules, 2021, 26(9): 2542. DOI:10.3390/molecules26092542
|
| [2] |
Li JH, Jia HJ, Cai XH, et al. An integrated catalog of reference genes in the human gut microbiome. Nat Biotechnol, 2014, 32(8): 834-841. DOI:10.1038/nbt.2942
|
| [3] |
Kalkreuter E, Pan GH, Cepeda AJ, et al. Targeting bacterial genomes for natural product discovery. Trends Pharmacol Sci, 2020, 41(1): 13-26. DOI:10.1016/j.tips.2019.11.002
|
| [4] |
Sharma AK, Dhasmana N, Dubey N, et al. Bacterial virulence factors: secreted for survival. Indian J Microbiol, 2017, 57(1): 1-10. DOI:10.1007/s12088-016-0625-1
|
| [5] |
Pakbin B, Brück WM, Rossen JWA. Virulence factors of enteric pathogenic Escherichia coli: a review. Int J Mol Sci, 2021, 22(18): 9922. DOI:10.3390/ijms22189922
|
| [6] |
尹业师, 陈华海, 曹林艳, 等. 细菌耐药性应对策略研究进展. 生物工程学报, 2018, 34(8): 1346-1360. Yin YS, Chen HH, Cao LY, et al. Progress in strategies to combat antimicrobial resistance. Chin J Biotech, 2018, 34(8): 1346-1360 (in Chinese). DOI:10.13345/j.cjb.180223 |
| [7] |
Almeida A, Nayfach S, Boland M, et al. A unified catalog of 204, 938 reference genomes from the human gut microbiome. Nat Biotechnol, 2021, 39(1): 105-114. DOI:10.1038/s41587-020-0603-3
|
| [8] |
Seemann T. Prokka: rapid prokaryotic genome annotation. Bioinformatics, 2014, 30(14): 2068-2069. DOI:10.1093/bioinformatics/btu153
|
| [9] |
Blin K, Shaw S, Kloosterman AM, et al. antiSMASH 6.0: improving cluster detection and comparison capabilities. Nucleic Acids Res, 2021, 49(W1): W29-W35.
|
| [10] |
De Nies L, Lopes S, Busi SB, et al. PathoFact: a pipeline for the prediction of virulence factors and antimicrobial resistance genes in metagenomic data. Microbiome, 2021, 9(1): 49. DOI:10.1186/s40168-020-00993-9
|
| [11] |
Traoré SI, Bilen M, Cadoret F, et al. Study of human gastrointestinal microbiota by culturomics in Africa. Med Sante Trop, 2019, 29(4): 366-370.
|
| [12] |
Forster SC, Kumar N, Anonye BO, et al. A human gut bacterial genome and culture collection for improved metagenomic analyses. Nat Biotechnol, 2019, 37(2): 186-192. DOI:10.1038/s41587-018-0009-7
|
| [13] |
Zou YQ, Xue WB, Luo GW, et al. 1 520 reference genomes from cultivated human gut bacteria enable functional microbiome analyses. Nat Biotechnol, 2019, 37(2): 179-185. DOI:10.1038/s41587-018-0008-8
|
| [14] |
Raymond F, Boissinot M, Ouameur AA, et al. Culture- enriched human gut microbiomes reveal core and accessory resistance genes. Microbiome, 2019, 7(1): 56. DOI:10.1186/s40168-019-0669-7
|
| [15] |
Tierney BT, Tan YX, Yang Z, et al. Systematically assessing microbiome-disease associations identifies drivers of inconsistency in metagenomic research. PLoS Biol, 2022, 20(3): e3001556. DOI:10.1371/journal.pbio.3001556
|
| [16] |
De Filippo C, Cavalieri D, Di Paola M, et al. Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa. PNAS, 2010, 107(33): 14691-14696. DOI:10.1073/pnas.1005963107
|
| [17] |
Arumugam M, Raes J, Pelletier E, et al. Enterotypes of the human gut microbiome. Nature, 2011, 473(7346): 174-180. DOI:10.1038/nature09944
|
| [18] |
Jia BL, Han X, Kim KH, et al. Discovery and mining of enzymes from the human gut microbiome. Trends Biotechnol, 2022, 40(2): 240-254. DOI:10.1016/j.tibtech.2021.06.008
|
| [19] |
Wang LL, Ravichandran V, Yin YL, et al. Natural products from mammalian gut microbiota. Trends Biotechnol, 2019, 37(5): 492-504. DOI:10.1016/j.tibtech.2018.10.003
|
| [20] |
Bhattarai K, Bhattarai K, Baral B. Next-generation microbial drugs developed from microbiome's natural products. Adv Genet, 2021, 108: 341-382.
|
| [21] |
Ma Y, Guo ZY, Xia BB, et al. Identification of antimicrobial peptides from the human gut microbiome using deep learning. Nat Biotechnol, 2022, 40(6): 921-931. DOI:10.1038/s41587-022-01226-0
|
| [22] |
Huang R, Yan L, Lei YH. The gut microbial-derived metabolite trimethylamine N-oxide and atrial fibrillation: relationships, mechanisms, and therapeutic strategies. Clin Interv Aging, 2021, 16: 1975-1986. DOI:10.2147/CIA.S339590
|
| [23] |
Hu YF, Yang X, Qin JJ, et al. Metagenome-wide analysis of antibiotic resistance genes in a large cohort of human gut microbiota. Nat Commun, 2013, 4: 2151. DOI:10.1038/ncomms3151
|
| [24] |
Hu YF, Yang X, Li J, et al. The bacterial mobile resistome transfer network connecting the animal and human microbiomes. Appl Environ Microbiol, 2016, 82(22): 6672-6681. DOI:10.1128/AEM.01802-16
|
| [25] |
Hu YF, Gao GF, Zhu BL. The antibiotic resistome: gene flow in environments, animals and human beings. Front Med, 2017, 11(2): 161-168. DOI:10.1007/s11684-017-0531-x
|
| [26] |
Shuai ML, Zhang GQ, Zeng FF, et al. Human gut antibiotic resistome and progression of diabetes. Adv Sci (Weinh), 2022, 9(11): e2104965. DOI:10.1002/advs.202104965
|
| [27] |
Wu HJ, Wang AHJ, Jennings MP. Discovery of virulence factors of pathogenic bacteria. Curr Opin Chem Biol, 2008, 12(1): 93-101. DOI:10.1016/j.cbpa.2008.01.023
|
 2022, Vol. 38
2022, Vol. 38