桃儿七叶绿体比较基因组学分析
马录花
,
宁佳奇
,
王永杰
,
赵敏
,
李以康
,
周华坤
生物工程学报  2022, Vol. 38 2022, Vol. 38 Issue (10): 3695-3712 Issue (10): 3695-3712 |

桃儿七(Sinopodophyllum hexandrum (Royle) Ying) 为多年生草本植物,隶属小檗科(Berberidaceae) 桃儿七属(Sinopodophyllum)[1],分布在喜马拉雅及附近区域,可适应高海拔(2 000–4 600m) 寒冷干旱气候[2]。桃儿七是常用的传统药材,常用于解毒、止痛、活血等[3-4],其根茎的成分鬼臼毒素有抗癌功效[5]。近年,由于人为无节制挖掘,桃儿七野生资源逐渐枯竭濒临灭绝[6-7],20世纪末已列入《中国生物多样性红色名录》《中国植物红皮书》和《中国珍稀濒危植物名录》等[8-10],为国家二级保护植物。目前,关于桃儿七的研究主要集中在化学成分[11]、生物学特性[12]、人工栽培技术[13]等方面,桃儿七不同叶绿体基因组序列特征很少关注。
叶绿体基因组(chloroplast genome) 具有进化慢,基因组成和结构保守性强[14-15]特征,因此叶绿体基因组常用于植物物种鉴定和谱系进化研究[16]。植物叶绿体基因组包括4个区域:一个大单拷贝区(large single-copy, LSC) 和一个小单拷贝区(small single-copy, SSC) 以及两个反向重复序列区(inverted repeat, IRa和IRb)[17],叶绿体基因组的基因主要参与光合作用、转录、翻译等过程[18]。叶绿体基因最早从烟草[19]和地钱[20]获得,近年来随着测序技术和分析方法的发展,系统发育基因组新兴学科出现[21-22],而且在药理作用及生物分子学方面都取得了重大进展[23],如可食用植物细胞生产药用蛋白[24]、莴苣叶绿体表达蛋白质药物[25]在疾病治疗方面发挥作用,生物大分子可以推断生物进化史[26-27]。段义忠等[28]、张倩等[29]和李述成等[30]分别基于沙冬青(Ammopiptanthus)、白花刺续断(Acanthocalyx alba) 和小檗属探究叶绿体全基因组特征及系统发育关系,以及‘苟当3号’水稻叶绿体基因组特征分析[31],均揭示了叶绿体基因组在医学、物种进化和系统发育方面的重要性。
本研究以桃儿七为研究对象,从美国国家生物技术信息中心(National Center for Biotechnology Information) 数据库选择桃儿七5个叶绿体基因组,探究同物种不同叶绿体基因组的差异性,通过该研究了解桃儿七基因组间的关系,为桃儿七叶绿体基因工程、物种鉴定和分子遗传学研究奠定理论基础并为系统发育进化研究提供参考。
1 材料与方法 1.1 材料本研究以叶绿体基因组为研究对象,从NCBI (https://www.ncbi.nlm.nih.gov/)下载已公布的5个桃儿七叶绿体基因组(GenBank登录号分别为KT445939.1、NC_027732.1、MT982396.1、MW331536.1、MG593048.1,其中MG593048.1样本采集于田间植物新鲜绿叶,其标本目前在浙江大学植物标本馆[32],NC_027732.1、MW331536.1样品采集于西藏灵芝新鲜叶子,其标本存放于西藏大学农牧学院、青藏高原生态研究所[33]。KT445939.1样品采集于滇西北香格里拉桃儿七的鲜叶,标本保存于中国云南师范大学[34],MT982396.1样品由甘肃省农业科学院采集于甘肃省,目前MW331536.1、MT982396.1样本仅获得了基因数据尚未发表文章,由于桃儿七5个样本的采集地区不同,其基因组成和基因结构存在差异),利用在线工具CHLOROPLOT (https://irscope.shinyapps.io/Chloroplot/) 绘制基因组谱图,用Excel和Origin2021做数据统计与绘图。
1.2 重复序列分析利用在线软件MISA-web (https://webblast. ipk-gatersleben.de/misa/),设置参数为1−10 (单核苷酸至少重复10次),2−6 (二核苷酸至少重复6次),3−4 (三核苷酸至少重复4次),四五六核苷酸均至少重复3次[30-31],获得SSR位点碱基序列。在REPuter (https://bibiserv. cebitec.uni-bielefeld.de/reputer) 在线软件中,上传5种叶绿体基因组数据,参数设置为:汉明距离为3、最小重复值为30、最大重复无上限,选择4种重复类型进行散在重复序列在线分析。
1.3 密码子偏好分析叶绿体密码子偏好分析常用CodonW1.4.2软件。在软件CodonW1.4.2中对桃儿七5个叶绿体基因组的蛋白编码基因分别进行密码子偏好分析,参数设置为软件默认值,获得密码子偏好性指数包括相对同义密码子使用度(relative synonymous codon usage, RSCU)、密码子适应指数(codon adaptation index, CAI)、ENc、GC、GC3s[35]。
1.4 IR/SC边界收缩和扩张分析IR/SC边界变化是植物叶绿体基因组变化的主要原因[28],从NCBI数据库下载水稻(Oryza sativa)、鼠耳芥(Arabidopsis thaliana) 叶绿体基因组,利用IRscope在线软件(https://irscope.shinyapps.io/irapp/) 绘制桃儿七5个叶绿体基因组与水稻、鼠耳芥IR/SC边界对比图,分析桃儿七基因组与水稻、鼠耳芥IR/SC边界收缩与扩张特征。
1.5 桃儿七基因组序列分析首先利用mVISTA将GenBank格式的文件转换成mVISTA网站输入的注释文件,然后在在线软件mVISTA Input (lbl.gov)中,将序列数设置为7,输入5个桃儿七叶绿体基因组、水稻基因组和鼠耳芥基因组,获得基因组序列比较图。
1.6 桃儿七的系统发育分析从NCBI数据库下载桃儿七5个叶绿体基因组和15个小檗科植物叶绿体基因组,其中包括小檗亚科(Berberidoideae) 和裸花草属(Achlys)、鬼臼属(Dysosma)、山荷叶属(Diphylleia)、北美桃儿七属(Podophyllum) 和桃儿七属,利用MAFFT在线软件对20个叶绿体基因序列进行对齐,然后将数据导入MEGA 7软件利用最大似然(maximum likelihood, ML) 法[37-38],boostrap参数设置为200次构建系统发育树。
2 结果与分析 2.1 桃儿七叶绿体基因组结构的基本特征桃儿七叶绿体基因组结构为一个大单拷贝区LSC、一个小单拷贝区SSC,以及两个反向重复区IRa和IRb组成的四分体双链环形结构,5个叶绿体基因组的全长在157 203–157 940 bp之间,其中最长的是基因组NC_027732.1,最短的是基因组KT445939.1,基因组长度差为737 bp,基因组长度差值较小(图 1)。桃儿七5个叶绿体基因组的GC含量均为38.48%– 38.50%,AT含量为61.50%–61.52%,无明显差异。
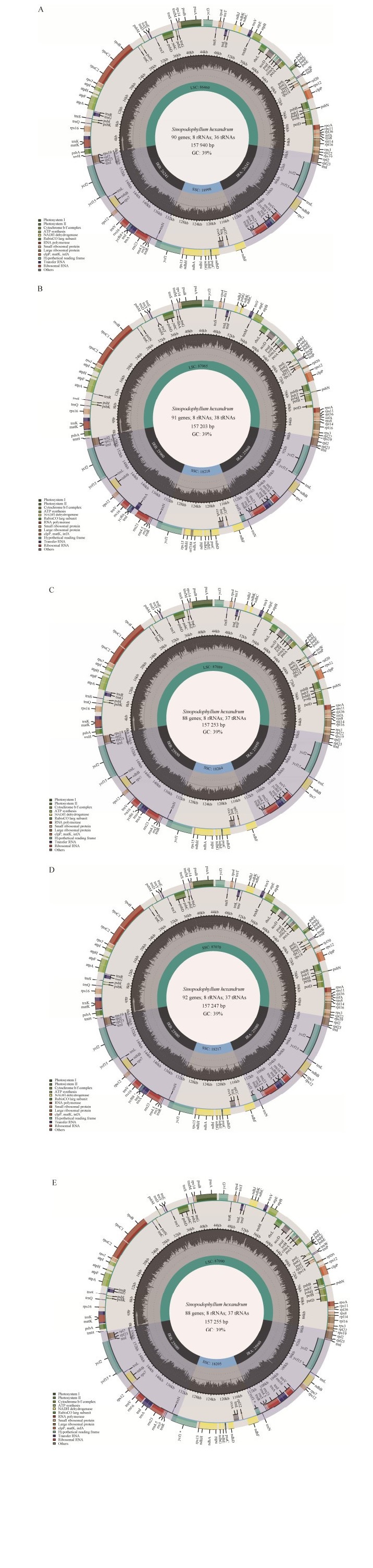
|
| 图 1 桃儿七5个叶绿体基因组图谱 Fig. 1 Genome atlas of five chloroplast genome in S. hexandrum. 圈内侧的是反向编码的基因,圈外侧的是正向编码的基因,颜色表示功能基因。内部的深灰色对应于GC含量,浅灰色对应于AT含量。A代表基因组NC_027732.1,B代表基因组KT445939.1,C代表基因组MG593048.1,D代表基因组MT98236.1,E代表基因组MW331536.1 The inside of the circle is the reverse-coding gene, while the outside of the circle is the forward-coding gene, the colors here represent functional genes. The inner dark gray corresponds to the GC content and the light gray corresponds to the AT content. (A) Genome NC_027732.1. (B) Genome KT445939.1. (C) Genome MG593048.1. (D) Genome MT98236.1. (E) Genome MW331536.1. |
| |
以基因组NC_027732.1为参照,桃儿七5个叶绿体基因组比较结果表明(表 1),桃儿七5个叶绿体基因组基因组成有差异,总基因数为133–137个,基因组MW331536.1和MG593048.1基因总数为133,缺失ycf68、infA基因,新增rps12基因,基因组KT445939.1、MT982396.1基因总数为137个,但是缺失基因和新增基因种类有很大差异。核糖体RNA (ribosomal RNA, rRNA) 的数量均为8个,最为恒定保守,转运RNA (transfer RNA, tRNA) 的数量为36–38个,蛋白编码基因数为88–92个,其中基因组KT445939.1的蛋白编码基因为91个,基因组MW331536.1和MG593048.1的蛋白编码基因为88个,基因组NC_027732.1的蛋白编码基因为90个,基因组MT98236.1的蛋白编码基因为92个。
| GenBank accession No. | Absence of genes | Additional genes | Total gene number |
| KT445939.1 | trnG, psbZ | trnM, trnN, psbG, lhbA, orf188 | 137 |
| MG593048.1 | ycf68, infA | rps12 | 133 |
| MT98236.1 | ycf1* | rps12 | 137 |
| MW331536.1 | ycf68, infA | rps12 | 133 |
| “*” indicates that the gene contains introns. | |||
桃儿七5个叶绿体基因组中散在重复序列存在差异。基因组NC_027732.1中共检测到50个散在重复序列,在5个叶绿体基因组类型中重复序列值最高,但仅包括35个正向重复序列(F型) 和15个回文重复序列(P型) 两种类型。其他4个叶绿体基因组中均检测到26个散在重复序列,包括正向重复序列(F型)、回文重复序列(P型) 和反向重复序列(R型) 三种类型(图 2)。桃儿七叶绿体基因组中缺失互补重复(C型),正向重复序列(F型) 和回文重复序列(P型) 为桃儿七叶绿体基因组的主要类型。
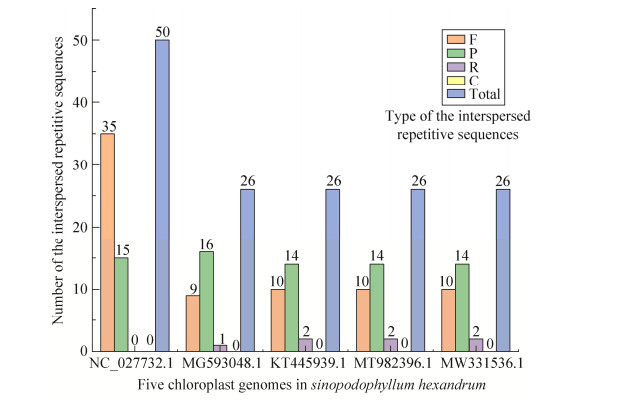
|
| 图 2 桃儿七叶绿体基因组散在重复序列 Fig. 2 Interspersed repetitive sequences of chloroplast genomes in S. hexandrum. 图中颜色表示不同的散在重复序列类型 The colors represent type of interspersed repetitive sequences. |
| |
在桃儿七叶绿体基因组简单重复序列(simple sequence repeat, SSR) 中,共检测到简单重复序列位点个数分别为59、56、58、55和56个。单核苷酸重复序列类型A和T最多,A的数量分别为13、15、15、12和12个,基因组KT445939.1、MW331536.1中A的数量相同均为15个,MG593048.1、MT982396.1的数量相同均为12个,T的数量分别为21、19、21、20和20个,基因组NC_027732.1和KT445939.1中T数量相同,基因组MG593048.1和MW331536.1中T数量相同,而G在5个叶绿体基因组中仅含有1个;双核苷酸重复序列均为2个;三核苷酸重复序和四核苷酸重复序列除了基因组NC_027732.1为7个和13个之外,其他4种基因组中均分别为6个和5个;五核苷酸序列和六核苷酸重复序列为AATGG和TAAGAA各有1个(图 3)。简单重复序列最丰富的类型为A/T,SSR位点碱基特征与桃儿七叶绿体基因组中富含的碱基特征有一致性。
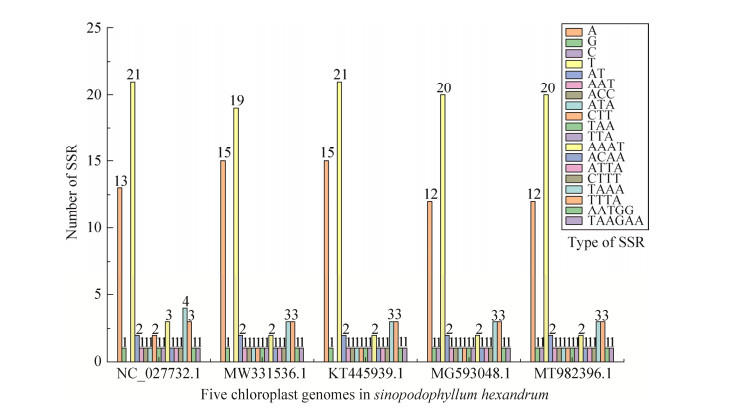
|
| 图 3 桃儿七叶绿体基因组SSR类型和数量 Fig. 3 Simple sequence repeat (SSR) of chloroplast genomes in S. hexandrum. 图中颜色表示不同的SSR类型 The colors here represent type of SSR. |
| |
桃儿七叶绿体基因组密码子使用性检测结果表明:桃儿七叶绿体基因组密码子使用偏好性不同,相对同义密码子使用度(relative synonymous codon usage, RSCU) 值大于1和小于1的密码子均有31个,均以碱基A/U结尾,说明桃儿七叶绿体基因组密码子都倾向以碱基A/U结尾。蛋氨酸(methionine, Met)、色氨酸(tryptophan, Trp) 的RSCU值为1,无密码子偏向性(图 4)。由于有效密码子(effective number of codon, ENc) 值介于51.14–51.17,说明叶绿体基因组中的密码子偏好性一般,并且密码子中GC的含量以及GC位于密码子末端的值分别为38.9%–39.4%、28.7%–29.0%均小于50% (表 3)。
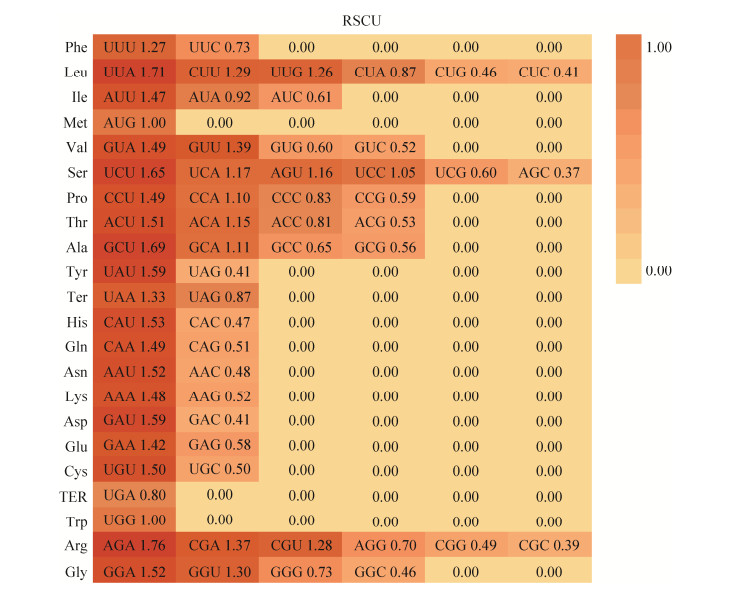
|
| 图 4 桃儿七叶绿体基因组密码子使用率 Fig. 4 RSCU values of chloroplast genomes in S. hexandrum. 图中颜色表示RSCU值,随着颜色变深密码子使用率增加 The colors here represent RSCU values, and codon usage increases as the color gets darker. |
| |
| Gene function classification | Group of genes | Gene name |
| Genes involve in self-replication | Ribosomal RNA genes | rrn4.5, rrn5, rrn16, rrn23 |
| Transfer RNA genes | trnA, trnC, trnD, trnE, trnF, trnfM, trnG, trnH, trnK, trnL, trnL, trnL, trnM, trnN, trnP, trnR, trnR, trnS, trnS, trnS, trnT, trnV, trnW, trnY, trnQ, trnT, trnG, trnI, trnI, trnR, trnV |
|
| RNA polymerase | rpoA, rpoB, rpoC1, rpoC2 | |
| Small subunits of ribosome | rps2, rps3, rps4, rps7, rps8, rps11, rps12, rps14, rps15, rps16, rps18, rps19, | |
| Lange subunits of ribosome | rpl2, rpl14, rpl16, rpl20, rpl22, rpl23, rpl32, rpl33, rpl36 | |
Gene involved in photosynthesis |
Photosystem I |
psaA, psaB, psaC, psaI, psaJ |
| Photosystem Ⅱ | psbA, psbB, psbC, psbD, psbE, psbF, psbH, psbJ, psbK, psbL, psbI, psbM, psbT, psbZ, psbN | |
| RuBisCO large subunits | rbcL | |
| Cytochrome b/f complex | petA, petB, petD, petG, petL, petN | |
| Subunits of ATP synthase | atpA, atpB, atpE, atpF, atpI, atpH | |
| NADH dehydrogenase | ndhA, ndhB, ndhC, ndhD, ndhE, ndhF, ndhG, ndhH, ndhJ, ndhI, ndhK | |
| Other genes | Protease | clpP |
| Maturase | matK | |
| Envelop membrane protein | cemA | |
| Subunits of acetyl-CoA-carboxylase | accD | |
| Translation initiation factor |
infA | |
| C-type cytochrome synthesis | ccsA | |
| Genes with unknown function | Hypothetical chloroplast reading frames |
ycf1, ycf2, ycf3, ycf4, ycf15, ycf68* |
| GenBank accession No. | CAI | ENc | GC3s | GC |
| NC_027732.1 | 0.167 | 51.16 | 0.287 | 0.389 |
| MG593048.1 | 0.167 | 51.17 | 0.290 | 0.394 |
| KT445939.1 | 0.167 | 51.17 | 0.289 | 0.391 |
| MT982396.1 | 0.167 | 51.14 | 0.287 | 0.389 |
| MW331536.1 | 0.167 | 51.17 | 0.290 | 0.394 |
植物叶绿体基因组IR/SC边界的收缩和扩展决定了植物叶绿体基因组的大小[36],是全基因组大小变化的主要机制[28]。在叶绿体基因组中存在JLB、JSB、JSA、JLA 4个边界,基因进化伴随着4个边界的扩张与收缩。本研究对桃儿七5个基因组和水稻、鼠耳芥IR/SC边界区域的碱基分布情况进行比较分析(图 5),发现鼠耳芥和桃儿七5个基因组序列边界基因的位置和类型差异很小,与水稻有明显差异。在桃儿七和鼠耳芥JLB边界分布的基因rps19横跨JLB边界,rpl2基因位于IRb区域,水稻JLB边界rpl22基因位于LSC区域,rps19基因位于IRb区域内。JSB边界上,NC_027732.1和水稻在IRb区域内基因与其他5种不同,基因组NC_027732.1在IRb区域内的基因为trnR,水稻在IRb区域内的基因为ndhH,其余5种在IRb区域内的基因为ycf1,在SSC区域内的基因为ndhF。在JSA边界上,桃儿七和鼠耳芥中ycf1基因横跨JSA,trnN基因位于IRa区域内,水稻JSA边界中ndhH基因横跨JSA,rps15基因位于IRa区域内。JLA边界基因差异较大,除水稻外trnH基因位于LSC区域内,psbA基因位于水稻LSC区域内,rps19、rpl2基因分布也有差异。
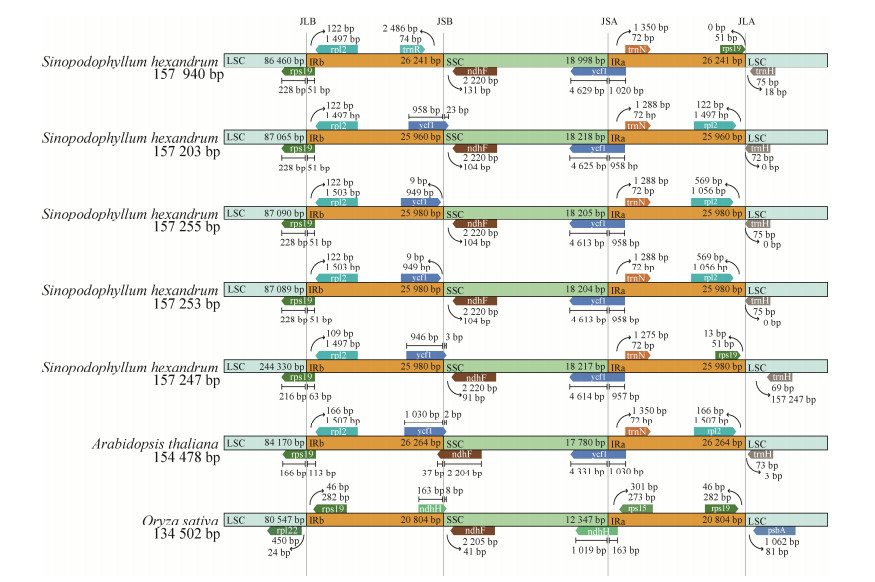
|
| 图 5 叶绿体基因组的IR/SC边界分析 Fig. 5 IR/SC boundary analysis of chloroplast genomes. 图中不同颜色表示叶绿体基因组的4个区域以及基因类型 The colors here represent four regions and type of genes of chloroplast genomes. |
| |
本研究运用mVISTA在线工具,以桃儿七叶绿体基因组NC_027732.1为参照,对桃儿七其他4个叶绿体基因组、水稻叶绿体基因组、鼠耳芥叶绿体基因组进行了全基因组的比较。结果表明,桃儿七5个叶绿体基因组基因结构和排列顺序整体上相似,在IRb和IRa区无明显变异,具有较高的保守性,在非基因编码区存在有较明显的差异,其中LSC区的变异程度最高。trnC-GCA、rps16、trnS-GGA、accD、psal、trnL-UAG、ndhA、rps15、ycf1基因有较大的变异,其他基因有较高的保守度。与水稻和鼠耳芥相比,在基因组4个区域差异性很大,不同层序和变异基因类型与桃儿七基因组序列有很大差异(图 6)。
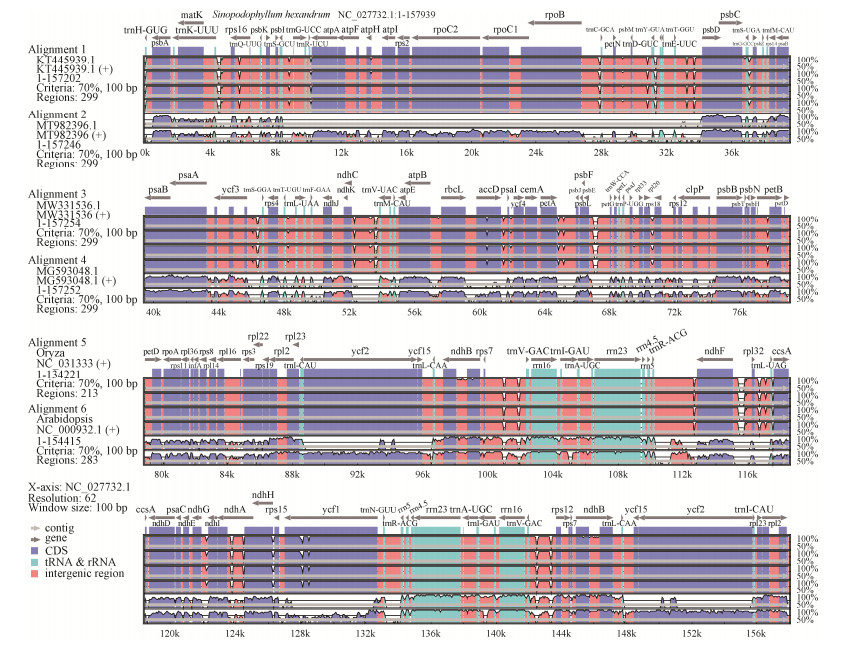
|
| 图 6 桃儿七叶绿体基因序列比对分析 Fig. 6 Alignment of chloroplast genome sequences in S. hexandrum. 图中浅灰色箭头表示重叠区,深灰色表示基因,紫色表示基因编码序列(coding sequence, CDS),浅蓝色表示tRNA和rRNA,红色表示基因间隔区 Light gray arrows represent overlapping regions, dark gray represents genes, purple represents CDs, light blue represents tRNA and rRNA, and red represents gene spacer regions. |
| |
为揭示桃儿七在小檗科中的进化位置,本研究基于桃儿七5个基因组与其他15个小檗科植物叶绿体基因组构建系统发育树(图 7)。结果表明,所构建的ML系统发育树分支的检验值较高,3种小檗亚科外类群植物独立聚为1支,3种植物分别为黄芦木(Berberis amurensis)、山槐叶(Bongardia chrysogonum) 和红毛七(Caulophyllum robustum)。鬼臼亚科植物则聚成2个大支,第1大支由裸花草属裸花草(Achlys triphylla) 构成;第2大支由鬼臼属、山荷叶属、北美桃儿七属、桃儿七属共16种植物构成,其中桃儿七5个叶绿体基因组独立聚成一支,传统的分类方法也得到了相同的结果。桃儿七位置紧邻北美桃儿七,同北美桃儿七的亲缘关系最近,所有物种最先分开的是山槐叶和红毛七,二者与其他物种的亲缘关系最远,在鬼臼亚科中与桃儿七亲缘关系由远递进的顺序为鬼臼属、山荷叶属、北美桃儿七属,其进化顺序同亲缘关系密切。
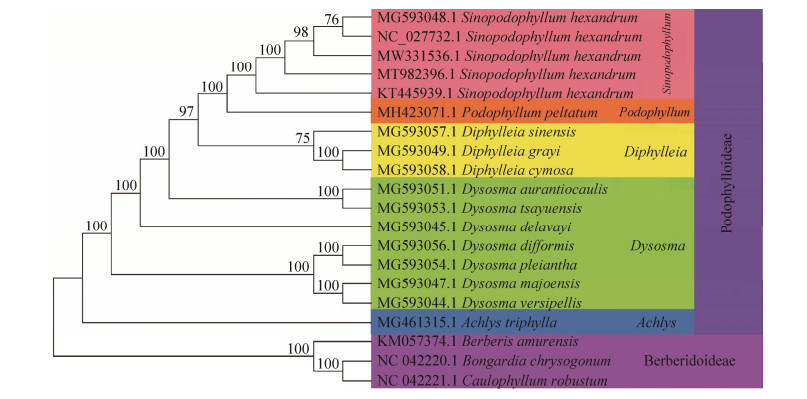
|
| 图 7 叶绿体全基因组系统发育树 Fig. 7 Maximum likelihood tree constructed by complete chloroplast genomes. 图中颜色代表不同科植物类型 The colors represent plant types of different families. |
| |
桃儿七属仅桃儿七一个物种,隶属于小檗科鬼臼亚科[39],作为传统藏中药材,具有利气活血、驱寒祛湿、抗病毒、抗炎等药理作用[12]。桃儿七主要分布在我国青藏高原及周边地区,大量开采使其生境遭到破坏,现被列入国家级二级保护植物[40],本研究以桃儿七5个叶绿体基因组为研究对象,对其结构特征和物种进化关系进行比较分析。
研究结果表明,桃儿七5个叶绿体基因组结构和长度与被子植物叶绿体基因组一致[41],不同基因组间差异小,说明桃儿七叶绿体基因组恒定,有较高的保守性;叶绿体全基因组中GC含量有细微差异,这种差异与rRNA基因全部位于IR区有关[42];并且叶绿体基因组组成数量和类型有明显的差异,植物叶绿体基因组基因在数量和类型上存在较小差异可能由假基因或基因组重组引起基因组变异[43],导致叶绿体基因结构的变化,其影响程度类似于IR/SC边界扩张和收缩[44]。
基因组重复序列是研究物种进化进程以及遗传特征的重要依据[45],本研究结果显示桃儿七5个叶绿体基因组SSR位点个数有较小差异,可能桃儿七在进化过程中部分基因缺失、增加、突变或者IR/SC边界收缩所致,A/T单核苷酸占主要优势,被子植物SSR位点也有相同的特征[46],同时证明A和T为构成叶绿体基因组SSR的主要碱基类型、C和G重复较少的观点[47],与基因组中AT占比高的结论相一致。高含量的A/T碱基体现出碱基偏好性,主要与A/T碱基在重组时容易发生断裂有关[31]。5个叶绿体基因组散在重复序列主要以正向和回文重复为主,与王文斌等关于黄芩(Scutellaria baicalensis) 基因组序列的研究结论吻合[48],除基因组NC_027732.1外其他4种基因组包括反向重复序列,李冉郡等[49]在唐古特大黄(Rheum tanguticum) 和掌叶大黄(Rheum palmatum) 也检测到这3种重复类型,并且在数量上存在很大的差异,这种在数量和类型上的差异这可能是基因增加或变异的结果[50],地理和生态因素也是影响其差异的一个原因。因此散在重复序列可用于鉴别不同种类基因组以及物种环境特征,可为物种遗传提供科学的依据。
密码子偏好分析结果表明,ENc值大于44,GC和GC3s小于50%,说明桃儿七叶绿体密码子偏好性弱,A和U为桃儿七叶绿体基因组密码子偏向使用碱基,证明了桃儿七5个叶绿体基因组的密码子偏好性相同,紫花苜蓿(Medicago sativa)[51]和樟树(Cinnamomum camphora)[52]叶绿体基因组密码子使用偏好性也有相同的结果。影响密码子偏好性的因子为自然选择和基因突变[53],GC含量还能进行基因组识别,判断不同基因组的差异性[54],通过基因组密码子使用偏好性以及因素的分析,可以用于确定最优密码子和基因功能预测[55]。
即使桃儿七叶绿体基因组在结构及大小极其保守,但基因组边界不稳定,基因组边界扩张和缩减是进化过程的表现之一[28]。研究结果表明,桃儿七IR/SC边界分布的基因类型相同,基因组基因结构和排列顺序整体上相似,说明桃儿七叶绿体基因组IR/SC边界较为保守。同物种不同基因组间IR/SC边界存在一定的差异,在非基因编码区发生了变异,这些容易变异的位点适合作为多态性位点开发为分子标记,用于种内遗传多样性分析。不同物种间边界的差异性很大,以水稻为标准,通过对比发现桃儿七和鼠耳芥基因有明显减少,表明桃儿七叶绿体基因组IR/SC边界有收缩现象,边界收缩可能是受到生长环境影响,水稻生长环境为半湿润区[56],鼠耳芥和桃儿七生长环境比较干旱[57]。基于桃儿七叶绿体基因的系统进化分析显示,桃儿七和北美桃儿七的亲缘关系最近,节点支持率达100%,进化上具有共线性,属于平行进化物种,在小檗属[13]和鬼臼亚科系统发育的研究发现相同的进化关系[29],说明在进化中桃儿七与北美桃儿七的基因结构特征相似,该结论有助于分析近缘物种间的遗传特征。
本研究结果表明桃儿七叶绿体基因组NC_027732.1除了散在重复序列与其他4个桃儿七叶绿体基因组存在较大的差异外,在其他4类分析中桃儿七5个叶绿体基因组无明显的差异,这种差异由内含子、编码序列长度,在边界基因新增与缺失以及反向重复区基因的变化引起,长期地理隔离和生长区域环境的差异也会使其叶绿体基因组发生变异,在基因组特征分析中表现出差异性。与模式植物鼠耳芥和水稻在边界和基因序列层面有很大的差异,物种类型和生长环境特征是引起这种差异的主要原因。基于叶绿体基因组散在重复序列表现出的差异性,可以进行桃儿七物种鉴别、产地溯源、群体遗传分析研究。
致谢: 感谢青海民族大学的王久利博士在数据分析方面给予指导。
| [1] |
沈奇, 张盾, 臧春鑫, 等. 基于ISSR分子标记的野生桃儿七遗传多样性研究. 植物遗传资源学报, 2019, 20(1): 129-136. Shen Q, Zhang D, Zang CX, et al. Genetic diversity analysis of Sinopodophyllum hexandrum (royle) T. S. Ying germplasm accessions using ISSR markers. J Plant Genet Resour, 2019, 20(1): 129-136 (in Chinese). |
| [2] |
郭彦龙, 卫海燕, 顾蔚, 等. 基于模糊物元模型的桃儿七潜在地理分布研究. 生态学报, 2015, 35(3): 770-778. Guo YL, Wei HY, Gu W, et al. Potential distributions of Sinopodophyllum hexandrum based on fuzzy matter element model. Acta Ecol Sin, 2015, 35(3): 770-778 (in Chinese). |
| [3] |
李忠超, 王小兰, 葛学军. 濒危药用植物桃儿七的生物学特性及其保育措施. 广西植物, 2005, 25(2): 179-185. Li ZC, Wang XL, Ge XJ. The biology and conservation of Sinopodophyllum hexandrum, an endangered medicinal herb. Guihaia, 2005, 25(2): 179-185 (in Chinese). DOI:10.3969/j.issn.1000-3142.2005.02.022 |
| [4] |
赵纪峰, 刘翔, 王昌华, 等. 珍稀濒危药用植物桃儿七的资源调查. 中国中药杂志, 2011, 36(10): 1255-1260. Zhao JF, Liu X, Wang CH, et al. Resources of survey in rare and endangered medicinal plant Sinopodophyllum emodi. China J Chin Mater Med, 2011, 36(10): 1255-1260 (in Chinese). |
| [5] |
李经纬. 中医大辞典. 2版. 北京: 人民卫生出版社, 2004. Li JW. Dictionary of Traditional Chinese Medicine. Beijing: People's Medical Publishing House, 2004 (in Chinese). |
| [6] |
马绍宾, 胡志浩. 桃儿七分布格局与生态适应的初步研究. 武汉植物学研究, 1996, 14(1): 47-54. Ma SB, Hu ZH. Preliminary studies on the distribution pattern and ecological adaptation of Sinopodophyllum hexandrum (Royle) Ying (Berberidaceae). J Wuhan Bot Res, 1996, 14(1): 47-54 (in Chinese). |
| [7] |
尚海琳. 桃儿七形态与生理特征的地理变异及种子萌发生理[D]. 西安: 西北大学, 2009. Shang HL. Geographic variation of morphology and physiology characteristics and seed germination physiology of Sinopodophyllum hexandrum[D]. Xi'an: Northwest University, 2009 (in Chinese). |
| [8] |
傅立国. 中国植物红皮书-第一册: 稀有濒危植物. 北京: 科学出版社, 1991: 184. Fu LG. China Plant Red Book-volume 1: Rare and Endangered Plants. Beijing: Science Press, 1991: 184 (in Chinese). |
| [9] |
虞泓. 珍稀植物桃儿七. 植物杂志, 1999(3): 6-7. Yu H. Rare plant Taoerqi. Plants, 1999(3): 6-7 (in Chinese). |
| [10] |
陈大霞, 赵纪峰, 刘翔, 等. 濒危药用植物桃儿七野生居群遗传多样性与遗传结构的SCoT分析. 中国中药杂志, 2013, 38(2): 278-283. Chen DX, Zhao JF, Liu X, et al. Genetic diversity and genetic structure of endangered wild Sinopodophyllum emodi by start codon targeted polymorphism. China J Chin Mater Med, 2013, 38(2): 278-283 (in Chinese). |
| [11] |
刘伟. 桃儿七化学多样性研究及鬼臼毒素生物合成相关基因发掘[D]. 杨凌: 西北农林科技大学, 2015. Liu W. Phytochemical diversity of Sinopodophyllum hexdrum and miniing of genes involved in podophyllotoxin biosynthesis[D]. Yangling: Northwest A & F University, 2015 (in Chinese). |
| [12] |
刘艳杰, 王健, 刘丽歌. 桃儿七化学成分和药理作用研究进展. 北方药学, 2016, 13(4): 105-106. Liu YJ, Wang J, Liu LG. Research progress on chemical constituents and pharmacological effects of Ardisia japonica thunb. J North Pharm, 2016, 13(4): 105-106 (in Chinese). |
| [13] |
叶文清. 抗癌药用植物鬼臼亚科的系统发育基因组学和生物地理学研究[D]. 杭州: 浙江大学, 2020. Ye WQ. Phylogenomic and biogeographic study of an anticancer herbaceous subfamily podophylloideae[D]. Hangzhou: Zhejiang University, 2020 (in Chinese). |
| [14] |
Shaw J, Lickey EB, Schilling EE, et al. Comparison of whole chloroplast genome sequences to choose noncoding regions for phylogenetic studies in angiosperms: the tortoise and the hare III. Am J Bot, 2007, 94(3): 275-288. DOI:10.3732/ajb.94.3.275
|
| [15] |
Olejniczak SA, Lojewska E, Kowalczyk T, et al. Chloroplasts: state of research and practical applications of plastome sequencing. Planta, 2016, 244(3): 517-527. DOI:10.1007/s00425-016-2551-1
|
| [16] |
Du XY, Zeng T, Feng Q, et al. The complete chloroplast genome sequence of yellow mustard (Sinapis alba L.) and its phylogenetic relationship to other Brassicaceae species. Gene, 2020, 731: 144340. DOI:10.1016/j.gene.2020.144340
|
| [17] |
Mower JP, Vickrey TL. Structural diversity among plastid genomes of land plants. Adv Bot Res, 2018, 85: 263-292.
|
| [18] |
Yarra R. Plastome engineering in vegetable crops: current status and future prospects. Mol Biol Rep, 2020, 47(10): 8061-8074. DOI:10.1007/s11033-020-05770-3
|
| [19] |
Ohyama K, Fukuzawa H, Kohchi T, et al. Chloroplast gene organization deduced from complete sequence of liverwort Marchantia polymorpha chloroplast DNA. Nature, 1986, 322(6079): 572-574. DOI:10.1038/322572a0
|
| [20] |
Shinozaki K, Ohme M, Tanaka M, et al. The complete nucleotide sequence of the tobacco chloroplast genome: its gene organization and expression. EMBO J, 1986, 5(9): 2043-2049. DOI:10.1002/j.1460-2075.1986.tb04464.x
|
| [21] |
Eisen JA. Phylogenomics: improving functional predictions for uncharacterized genes by evolutionary analysis. Genome Res, 1998, 8(3): 163-167. DOI:10.1101/gr.8.3.163
|
| [22] |
Eisen JA, Hanawalt PC. A phylogenomic study of DNA repair genes, proteins, and processes. Mutat Res Repair, 1999, 435(3): 171-213. DOI:10.1016/S0921-8777(99)00050-6
|
| [23] |
Dobrogojski J, Adamiec M, Luciński R. The chloroplast genome: a review. Acta Physiol Plant, 2020, 42(6): 1-13.
|
| [24] |
Daniell H, Jin SX, Zhu XG, et al. Green giant-a tiny chloroplast genome with mighty power to produce high-value proteins: history and phylogeny. Plant Biotechnol J, 2021, 19(3): 430-447. DOI:10.1111/pbi.13556
|
| [25] |
Kumar SRP, Wang XM, Avuthu N, et al. Role of small intestine and gut microbiome in plant-based oral tolerance for hemophilia. Front Immunol, 2020, 11: 844. DOI:10.3389/fimmu.2020.00844
|
| [26] |
Li WH. Molecular Evolution. Sunderland, Mass. : Sinauer Associates, 1997.
|
| [27] |
Nei M, Kumar S. Molecular Evolution and Phylogenetics. New York: Oxford University Press, 2000.
|
| [28] |
段义忠, 张凯. 沙冬青属植物叶绿体基因组对比和系统发育分析. 西北植物学报, 2020, 40(8): 1323-1332. Duan YZ, Zhang K. Comparative analysis and phylogenetic evolution of the complete chloroplast genome of Ammopiptanthus. Acta Bot Boreali Occidentalia Sin, 2020, 40(8): 1323-1332 (in Chinese). |
| [29] |
张倩, 张德全. 白花刺续断野生居群的叶绿体全基因组特征解析究. 广西植物, 2022, 1-16. Zhang Q, ZhanG DQ. Analysis of complete chloroplast genomes from wild populations of Acanthocalyx alba. Guihaia, 2022, 1-16 (in Chinese). |
| [30] |
李述成, 郭生虎, 贝盏临. 小檗属植物叶绿体基因组序列结构及系统发育分析. 中草药, 2022, 53(3): 818-826. Li SC, Guo SH, Bei ZL. Chloroplast genome sequences and phylogenetic analysis of Berberis genus. Chin Tradit Herb Drugs, 2022, 53(3): 818-826 (in Chinese). |
| [31] |
吴朝昕, 徐海峰, 刘雪薇, 等. '苟当3号'水稻叶绿体基因组特征分析. 分子植物育种, 2022, 1-29. Wu CX, Xu HF, Liu XW, et al. Analysis of chloroplast genome of 'Goudang 3'. Molecular Plant Breeding, 2022, 1-29 (in Chinese). |
| [32] |
Ye WQ, Yap ZY, Li P, et al. Plastome organization, genome-based phylogeny and evolution of plastid genes in Podophylloideae (Berberidaceae). Mol Phylogenet Evol, 2018, 127: 978-987.
|
| [33] |
Li HE, Guo QQ. The complete chloroplast genome of Sinopodophyllum hexandrum (Berberidaceae). Mitochondrial DNA A DNA Mapp Seq Anal, 2016, 27(4): 2955-2956.
|
| [34] |
Meng LH, Liu RJ, Chen JB, et al. The complete chloroplast genome of Sinopodophyllum hexandrum Ying (Berberidaceae). Mitochondrial DNA A, 2017, 28(3): 342-343.
|
| [35] |
张东旭, 孔丽丽. 连香树叶绿体基因组测序与特征分析. 分子植物育种, 2022, 1-15. Zhang DX, Kong LL. Genome sequencing and characteristic Analysis of Cercidiphyllum japonicum. Molecular Plant Breeding, 2022, 1-15 (in Chinese). |
| [36] |
Huang H, Shi C, Liu Y, et al. Thirteen Camellia chloroplast genome sequences determined by high-throughput sequencing: genome structure and phylogenetic relationships. BMC Evol Biol, 2014, 14: 151.
|
| [37] |
Tamura K, Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol Biol Evol, 1993, 10(3): 512-526.
|
| [38] |
Kumar S, Stecher G, Tamura K. MEGA 7: molecular evolutionary genetics analysis version 7. 0 for bigger datasets. Mol Biol Evol, 2016, 33(7): 1870-1874.
|
| [39] |
郁文彬. 桃儿七属的命名学考订(英文). 植物分类与资源学报, 2014(1): 37-40. Yu WB. Nomenclatural clarifications of Sinopodophyllum (Berberidaceae). Plant Divers Resour, 2014(1): 37-40 (in Chinese). |
| [40] |
郭其强, 高超, 李慧娥, 等. 药用濒危植物桃儿七种群生存状况及种实特性分析. 中国中药杂志, 2018, 43(6): 1104-1110. Guo QQ, Gao C, Li HE, et al. Survival state and fruiting characteristics of endangered medicinal plant Sinopodophyllum hexandrum plant in Tibet plateau. China J Chin Mater Med, 2018, 43(6): 1104-1110 (in Chinese). |
| [41] |
Wang XW, Rhein HS, Jenkins J, et al. Chloroplast genome sequences of Carya illinoinensis from two distinct geographic populations. Tree Genet Genomes, 2020, 16(4): 1-14.
|
| [42] |
张靖雯. 濒危植物羽叶丁香叶绿体全基因组及系统发育研究[D]. 杨凌: 西北农林科技大学, 2019. Zhang JW. The complete chloroplast genome and phylogenetic analysis of the endangered species Syringa pinnatifolia (Oleaceae)[D]. Yangling: Northwest A & F University, 2019 (in Chinese). |
| [43] |
李杰, 蔡嘉慧, 王慧中, 等. 药用植物基因组测序及功能基因组学研究进展. 杭州师范大学学报(自然科学版), 2021, 20(4): 364-373, 397. Li J, Cai JH, Wang HZ, et al. Advances in genome sequencing and functional genomics of medicinal plants. J Hangzhou Norm Univ (Nat Sci Ed), 2021, 20(4): 364-373, 397 (in Chinese). |
| [44] |
李岩, 吕光辉, 张雪妮, 等. 十字花科植物叶绿体基因组结构及变异分析. 西北植物学报, 2017, 37(6): 1090-1101. Li Y, Lü GH, Zhang XN, et al. Chloroplast genome structure and variation analysis of Brassicaceae species. Acta Bot Boreali Occidentalia Sin, 2017, 37(6): 1090-1101 (in Chinese). |
| [45] |
Hu JH, Gui ST, Zhu ZX, et al. Genome-wide identification of SSR and SNP markers based on whole-genome re-sequencing of a Thailand wild sacred Lotus (Nelumbo nucifera). PLoS One, 2015, 10(11): e0143765.
|
| [46] |
Guo HJ, Liu JS, Luo L, et al. Complete chloroplast genome sequences of Schisandra chinensis: genome structure, comparative analysis, and phylogenetic relationship of basal angiosperms. Sci China Life Sci, 2017, 60(11): 1286-1290.
|
| [47] |
Kuang DY, Wu H, Wang YL, et al. Complete chloroplast genome sequence of Magnolia kwangsiensis (Magnoliaceae): implication for DNA barcoding and population genetics. Genome, 2011, 54(8): 663-673.
|
| [48] |
王文斌, 于欢, 邱相坡. 黄芩叶绿体基因组重复序列及密码子偏好性分析. 分子植物育种, 2018, 16(8): 2445-2452. Wang WB, Yu H, Qiu XP. Analysis of repeat sequence and codon bias of chloroplast genome in Scutellaria baicalensis. Mol Plant Breed, 2018, 16(8): 2445-2452 (in Chinese). |
| [49] |
李冉郡, 武立伟, 辛天怡, 等. 大黄药材基原物种叶绿体基因组分析与特异DNA条形码开发. 药学学报, 2022, 57(5): 1495-1505. Li RJ, Wu LW, Xin TY, et al. Analysis of chloroplast genomes and development of specific DNA barcodes for identifying the original species of Rhei Radix et Rhizoma. Acta Pharm Sin, 2022, 57(5): 1495-1505 (in Chinese). |
| [50] |
Ammiraju JSS, Zuccolo A, Yu Y, et al. Evolutionary dynamics of an ancient retrotransposon family provides insights into evolution of genome size in the genus Oryza. Plant J, 2007, 52(2): 342-351.
|
| [51] |
喻凤, 韩明. 紫花苜蓿叶绿体基因组密码子偏好性分析. 广西植物, 2021, 41(12): 2069-2076. Yu F, Han M. Analysis of codon usage bias in the chloroplast genome of alfalfa (Medicago sativa). Guihaia, 2021, 41(12): 2069-2076 (in Chinese). |
| [52] |
秦政, 郑永杰, 桂丽静, 等. 樟树叶绿体基因组密码子偏好性分析. 广西植物, 2018, 38(10): 1346-1355. Qin Z, Zheng YJ, Gui LJ, et al. Codon usage bias analysis of chloroplast genome of Camphora tree (Cinnamomum camphora). Guihaia, 2018, 38(10): 1346-1355 (in Chinese). |
| [53] |
Suzuki Y. Statistical methods for detecting natural selection from genomic data. Genes Genet Syst, 2010, 85(6): 359-376.
|
| [54] |
原晓龙, 李云琴, 张劲峰, 等. 降香黄檀叶绿体基因组密码子偏好性分析. 广西植物, 2021, 41(4): 622-630. Yuan XL, Li YQ, Zhang JF, et al. Analysis of codon usage bias in the chloroplast genome of Dalbergia odorifera. Guihaia, 2021, 41(4): 622-630 (in Chinese). |
| [55] |
李薇, 王倩, 胡晓艳, 等. 银杏叶绿体基因组密码子使用偏性分析. 林业调查规划, 2021, 46(6): 28-34. Li W, Wang Q, Hu XY, et al. Codon usage bias in chloroplast genome of Ginkgo biloba. For Invent Plan, 2021, 46(6): 28-34 (in Chinese). |
| [56] |
Feng Q, Song WC, Zhang YJ, et al. The complete chloroplast genome sequence of Oryza sativa Temperate Japonica. Mitochondrial DNA B Resour, 2021, 6(3): 927-928.
|
| [57] |
Stadermann KB, Holtgräwe D, Weisshaar B. Chloroplast genome sequence of Arabidopsis thaliana accession Landsberg erecta, assembled from single-molecule, real-time sequencing data. Genome Announc, 2016, 4(5): e00975-e00916.
|