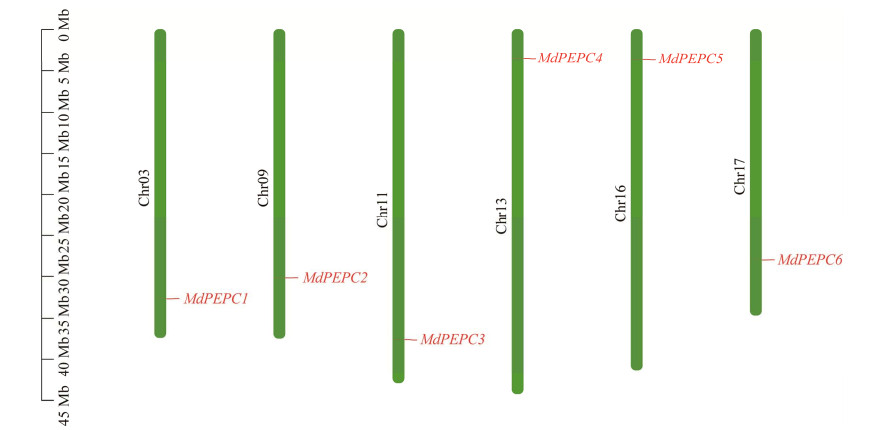
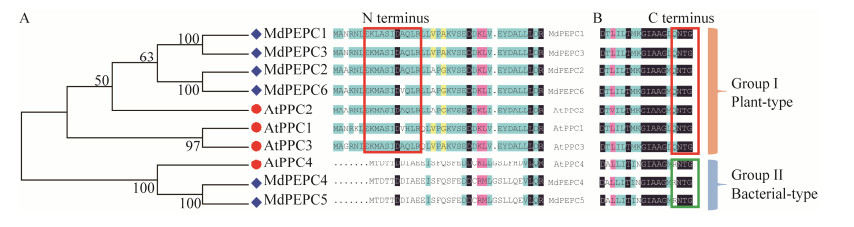
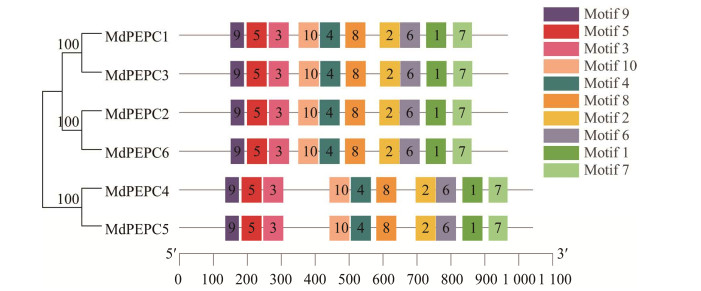
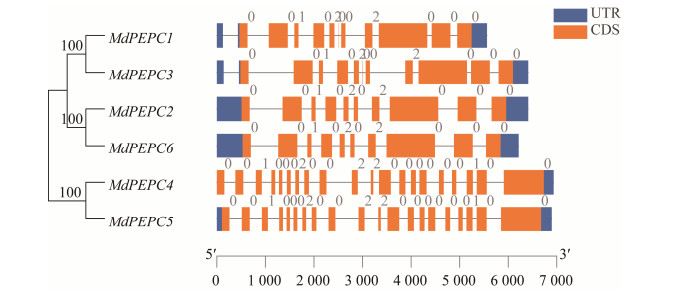
| [1] |
O'Leary B, Park J, Plaxton WC. The remarkable diversity of plant PEPC (phosphoenolpyruvate carboxylase): recent insights into the physiological functions and post-translational controls of non-photosynthetic PEPCs. Biochem J, 2011, 436(1): 15-34. DOI:10.1042/BJ20110078
|
|
| [2] | |
|
| [3] |
Guillet C, Just D, Bénard N, et al. A fruit-specific phosphoenolpyruvate carboxylase is related to rapid growth of tomato fruit. Planta, 2002, 214(5): 717-726. DOI:10.1007/s00425-001-0687-z
|
|
| [4] |
Sweetman C, Deluc LG, Cramer GR, et al. Regulation of malate metabolism in grape berry and other developing fruits. Phytochemistry, 2009, 70(11/12): 1329-1344.
|
|
| [5] |
Cousins AB, Baroli I, Badger MR, et al. The role of phosphoenolpyruvate carboxylase during C4 photosynthetic isotope exchange and stomatal conductance. Plant Physiol, 2007, 145(3): 1006-1017. DOI:10.1104/pp.107.103390
|
|
| [6] |
Cheng G, Wang L, Lan HY. Cloning of PEPC-1 from a C4 halophyte Suaeda aralocaspica without Kranz anatomy and its recombinant enzymatic activity in responses to abiotic stresses. Enzyme Microb Technol, 2016, 83(11): 57-67.
|
|
| [7] |
Perotti VE, Figueroa CM, Andreo CS, et al. Cloning, expression, purification and physical and kinetic characterization of the phosphoenolpyruvate carboxylase from orange ( Citrus sinensis osbeck var. Valencia) fruit juice sacs. Plant Sci, 2010, 179(5): 527-535. DOI:10.1016/j.plantsci.2010.08.003
|
|
| [8] |
Østergaard L, Kempin SA, Bies D, et al. Pod shatter-resistant Brassica fruit produced by ectopic expression of the FRUITFULL gene. Plant Biotechnol J, 2006, 4(1): 45-51. DOI:10.1111/j.1467-7652.2005.00156.x
|
|
| [9] |
彭文丽, 刘照宇, 李晓波, 等. 杧果PEPC基因家族全基因组鉴定及表达分析. 中国南方果树, 2021, 50(6): 37-46. Peng WL, Liu ZY, Li XB, et al. Genome-wide identification and expression analysis of PEPC gene family in mango. South China Fruits, 2021, 50(6): 37-46 (in Chinese).
|
|
| [10] |
Sánchez R, Cejudo FJ. Identification and expression analysis of a gene encoding a bacterial-type phosphoenolpyruvate carboxylase from Arabidopsis and rice. Plant Physiol, 2003, 132(2): 949-957. DOI:10.1104/pp.102.019653
|
|
| [11] | |
|
| [12] |
门小鹏, 许雪峰, 王玉斌, 等. 我国苹果生产的现状、问题与发展对策. 农村实用技术, 2022(1): 25-27+59. Men XP, Xu XF, Wang YB, et al. The present situation, problems and development countermeasures of apple production in our country. Rural Practical Technology, 2022(1): 25-27, 59 (in Chinese).
|
|
| [13] |
Kim HK, Van Oosterom E, Dingkuhn M, et al. Regulation of tillering in Sorghum: environmental effects. Ann Bot, 2010, 106(1): 57-67. DOI:10.1093/aob/mcq079
|
|
| [14] |
Kebrom TH, Mullet JE. Photosynthetic leaf area modulates tiller bud outgrowth in Sorghum. Plant Cell Environ, 2015, 38(8): 1471-1478. DOI:10.1111/pce.12500
|
|
| [15] |
Chen CJ, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data. Mol Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009
|
|
| [16] |
Gasteiger E, Gattiker A, Hoogland C, et al. ExPASy: the proteomics server for in-depth protein knowledge and analysis. Nucleic Acids Res, 2003, 31(13): 3784-3788. DOI:10.1093/nar/gkg563
|
|
| [17] |
Lescot M, Déhais P, Thijs G, et al. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences. Nucleic Acids Res, 2002, 30(1): 325-327. DOI:10.1093/nar/30.1.325
|
|
| [18] |
Da LL, Liu Y, Yang JT, et al. AppleMDO: a multi-dimensional omics database for apple co-expression networks and chromatin states. Front Plant Sci, 2019, 10: 1333. DOI:10.3389/fpls.2019.01333
|
|
| [19] |
Wang R, Zhao P, Kong N, et al. Genome-wide identification and characterization of the potato bHLH transcription factor family. Genes (Basel), 2018, 9(1): E54. DOI:10.3390/genes9010054
|
|
| [20] | |
|
| [21] |
Chollet R, Vidal J, O'Leary MH. Phosphoenolpyruvate carboxylase: a ubiquitous, highly regulated enzyme in plants. Annu Rev Plant Physiol Plant Mol Biol, 1996, 47: 273-298. DOI:10.1146/annurev.arplant.47.1.273
|
|
| [22] | |
|
| [23] |
Hartwell J, Gill A, Nimmo GA, et al. Phosphoenolpyruvate carboxylase kinase is a novel protein kinase regulated at the level of expression. Plant J, 1999, 20(3): 333-342. DOI:10.1046/j.1365-313X.1999.t01-1-00609.x
|
|
| [24] | |
|
| [25] |
Yamaguchi-Shinozaki K, Shinozaki K. Organization of cis-acting regulatory elements in osmotic- and cold-stress-responsive promoters. Trends Plant Sci, 2005, 10(2): 88-94. DOI:10.1016/j.tplants.2004.12.012
|
|
| [26] |
Rameau C, Bertheloot J, Leduc N, et al. Multiple pathways regulate shoot branching. Front Plant Sci, 2015, 5: 741.
|
|
| [27] |
Carmo-Silva AE, Bernardes Da Silva A, Keys AJ, et al. The activities of PEP carboxylase and the C 4 acid decarboxylases are little changed by drought stress in three C 4 grasses of different subtypes. Photosynth Res, 2008, 97(3): 223-233. DOI:10.1007/s11120-008-9329-7
|
|
| [28] |
Bandyopadhyay A, Datta K, Zhang J, et al. Enhanced photosynthesis rate in genetically engineered indica rice expressing PEPC gene cloned from maize. Plant Sci, 2007, 172(6): 1204-1209. DOI:10.1016/j.plantsci.2007.02.016
|
|
| [29] |
Sánchez R, Flores A, Cejudo FJ. Arabidopsis phosphoenolpyruvate carboxylase genes encode immunologically unrelated polypeptides and are differentially expressed in response to drought and salt stress. Planta, 2006, 223(5): 901-909.
|
|
| [30] |
马海洋, 赵秋芳, 陈曙, 等. 菠萝 PEPC基因家族生物信息学分析. 热带作物学报, 2020, 41(1): 97-103. Ma HY, Zhao QF, Chen S, et al. Bioinformatics analysis of PEPC gene family in pineapple. Chin J Trop Crops, 2020, 41(1): 97-103 (in Chinese).
|
|
| [31] |
彭文丽, 杨成坤, 崔志富, 等. 榴莲 PEPC基因家族全基因组鉴定及表达分析. 分子植物育种, 2022, 20(1): 76-85. Peng WL, Yang CK, Cui ZF, et al. Genome-wide identification and expression analysis of PEPC gene family in durian ( Durio zibethinus Murr. ). Mol Plant Breed, 2022, 20(1): 76-85 (in Chinese).
|
|
| [32] |
王岳坤, 阳江华, 丁丽, 等. 橡胶树 PEPC基因的克隆、生物信息学和表达分析. 西南农业学报, 2017, 30(11): 2436-2443. Wang YK, Yang JH, Ding L, et al. Cloning, bio-informatics and expression analysis of PEPC gene from Hevea brasiliensis. Southwest China J Agric Sci, 2017, 30(11): 2436-2443 (in Chinese).
|
|
| [33] |
Law RD, Plaxton WC. Purification and characterization of a novel phosphoenolpyruvate carboxylase from banana fruit. Biochem J, 1995, 307(3): 807-816.
|
|
| [34] |
高凯, 朱铁霞, 邓波, 等. 顶端优势去除对菊芋物质分配规律的影响. 中国草地学报, 2016, 38(1): 14-19. Gao K, Zhu TX, Deng B, et al. The influence of removal of apical dominance on matter allocation in Jerusalem artichoke. Chin J Grassland, 2016, 38(1): 14-19 (in Chinese).
|
|
| [35] |
武慧, 吕杰, 付智坤, 等. TDZ对微型月季不定芽生长的影响. 湖北农业科学, 2013, 52(13): 3178-3179, 3202. Wu H, Lv J, Fu ZK, et al. Effect of TDZ on adventitious bud growth of Rosa chinensis minima. Hubei Agric Sci, 2013, 52(13): 3178-3179, 3202 (in Chinese).
|
|
| [36] | |
|
| [37] |
孙洪雁, 陶吉寒, 辛力, 等. TDZ和BA对金花梨和鸭梨叶片不定芽再生的影响. 山东农业科学, 2016, 48(1): 26-28, 39. Sun HY, Tao JH, Xin L, et al. Effects of TDZ and BA on adventitious bud regeneration from leaf explants of pear cultivars Jinhua and yali. Shandong Agric Sci, 2016, 48(1): 26-28, 39 (in Chinese).
|
|
 2022, Vol. 38
2022, Vol. 38