中国科学院微生物研究所、中国微生物学会主办
文章信息
- 刘丹, 王柯蔼, 倪蓬, 王秋艳, 朱康, 危文亮
- LIU Dan, WANG Keai, NI Peng, WANG Qiuyan, ZHU Kang, WEI Wenliang
- 大豆GolS基因家族鉴定及盐旱胁迫下的表达分析
- Identification of soybean GolS gene family and analysis of expression patterns under salt and drought stresses
- 生物工程学报, 2022, 38(10): 3757-3772
- Chinese Journal of Biotechnology, 2022, 38(10): 3757-3772
- 10.13345/j.cjb.220449
-
文章历史
- Received: June 7, 2022
- Accepted: July 22, 2022
干旱、盐分和极端温度等非生物胁迫会导致植物发生一系列形态、生理和生化变化,严重影响植物的生长发育和产量品质[1-3]。研究发现,许多植物在干旱胁迫期间会积累大量可溶性糖,包括蔗糖、海藻糖和棉子糖家族寡糖等[4-7]。棉子糖家族寡糖(raffinose family oligosaccharides, RFOs) 作为渗透保护剂在植物中发挥着非常重要的生理作用,例如提高种子活力[8],提高植物对生物和非生物胁迫的耐受性[9-13]等。肌醇半乳糖苷合成酶(galactinol synthase, GolS) 是RFOs生物合成途径中的关键酶,其所催化的UDP-半乳糖与肌醇合成肌醇半乳糖苷的反应是RFO代谢途径的第一步[14]。
迄今为止,在很多植物中开展了GolS基因的全基因组鉴定、克隆和功能研究。在拟南芥[15]、水稻[16]、大豆[17]、杨树[18]、烟草[19]、柳枝稷[20]和芝麻[21]等物种中,均有GolS基因被发现和克隆的研究报道,并发现GolS基因的表达与非生物胁迫密切相关。在拟南芥中已鉴定出7个GolS成员,其中,AtGolS1、AtGolS2和AtGolS3均在成熟的种子中表达,AtGolS1和AtGolS2受干旱、盐和热胁迫诱导,而AtGolS3仅受冷胁迫诱导[15]。AtGolS2在水稻中过表达显著提高了转基因植株的抗旱性,并增加了干旱条件下的产量[22]。在木薯中已鉴定出8个GolS家族成员,并分析了这些家族成员在旱盐胁迫下的表达模式,发现MeGolS5和MeGolS6对干旱胁迫的响应相似,均在3 h表达量达到峰值;MeGolS2在盐胁迫下24 h呈现高表达、MeGolS4在6 h和24 h相比对照表达量显著增加[23]。烟草中MfGolS1的过度表达导致转基因植物对冷冻和低温的耐受性增强[24]。在山茶科茶树中克隆出了3个GolS基因,并进一步对其进行功能鉴定,结果发现CsGolS1对水分亏缺、低温和脱落酸敏感,而CsGolS2和CsGolS3对虫害和植物激素敏感[25]。矮沙冬青AnGolS1的表达量在盐、旱胁迫处理下比对照明显增加,呈上调表达模式[26]。综上所述,GolS基因在应对非生物胁迫中起重要作用。
大豆(Glycine max) 是重要粮食、油料和饲料作物,是我国重要的经济作物之一。近年来,干旱、盐渍化、高温等非生物胁迫发生的频率逐年增加,对大豆的产量和质量造成了很大的影响[27]。加强大豆抗旱耐盐机理研究,挖掘大豆抗逆相关基因,对于培育大豆抗逆品种、振兴我国大豆产业,具有极为重要的指导作用。本研究在全基因组水平上鉴定了大豆GolS基因家族成员,并利用生物信息学对其进化关系、基因结构、保守基序、染色体位置、蛋白质二级结构和三级结构、组织表达模式等进行分析,同时还对该家族成员进行盐旱胁迫处理下的表达量分析和基因功能的初步鉴定,为后续解析大豆GolS家族基因功能提供科学依据。
1 材料与方法 1.1 大豆GolS基因家族的全基因组鉴定下载已知的拟南芥(Arabidopsis thaliana) GolS1–7蛋白序列,在Phytozome (http://www.phytozome.org) 数据库中对大豆GolS基因家族成员进行BlastP搜索与筛选,筛选E-value≤ 1e-5、score≥200且同源性较高的序列,手动删去冗余数据。并将候选基因的蛋白序列提交NCBI网站在线工具Conserved Domain Search (http://www.ncbi.nlm.nih.gov/cdd/) 和Smart (http://smart.emblheidelberg.de/) 工具检查是否含有GolS蛋白序列的结构域,并剔除无GolS结构域的基因,最终获得大豆GolS基因家族成员。大豆的基因组序列、CDS序列和蛋白质序列信息均在Phytozome数据库中下载获得。
1.2 多序列比对与系统发育树的构建根据文献报道,下载了拟南芥(7个)、水稻(2个)、木薯(8个)、油菜(20个) 等4个物种GolS家族成员的蛋白质序列(共37个)。将拟南芥和大豆两个物种的GolS蛋白序列导入DNAMAN软件中进行序列比对;同时,将前述下载的4个物种(37个) 以及本文中鉴定的大豆(6个) GolS家族成员蛋白质序列在MEGA7.0软件中使用邻接法(neighbor-joining, NJ) 构建系统发育树,氨基酸替换模型为JTT+ G,bootstrap设置为1 000,其他参数默认[28-29]。并使用在线工具Evolview编辑进化树。
1.3 染色体位置、基因结构及保守基序鉴定分析利用大豆基因组的GFF3文件提取GolS基因家族成员在染色体上的位置,并将提取结果和大豆GolS基因成员的ID提交至TBtools[30]软件,绘制大豆GolS基因在染色体上的分布图。外显子-内含子结构图也由TBtools软件绘制。通过MEME在线工具(http://meme-suite.org/tools/meme) 预测大豆GolS蛋白的保守基序。
1.4 GmGolS基因启动子中顺式作用元件的分析为了研究大豆GmGolS基因家族成员的启动子中可能存在的顺式元件,从大豆基因组中提取GmGolS基因启动子上游2 kb序列。利用PlantCARE网站(http://bioinformatics.psb.ugent.be/webtools/PlantCARE/html/) 检测各基因的启动子序列,并借助TBtools软件绘制顺式元件分布图。
1.5 大豆GolS蛋白质二级结构和三级结构分析使用在线工具SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html) 对大豆GolS蛋白进行蛋白质二级结构预测。利用SWISS-MODEL (https://swissmodel.expasy.org/interactive) 预测蛋白的三级结构。
1.6 GmGolS基因在不同组织中的表达模式分析从Phytozome数据库中检索转录组数据,分析GmGolS基因在不同组织中的表达模式,植物组织包括花、叶、根瘤、豆荚、根、根毛、种子和茎。使用TBtools软件构建基因表达热图。
1.7 植物生长条件及盐旱胁迫处理为了明确大豆GolS基因对非生物胁迫的响应,分别在高盐和干旱胁迫下进行处理,检测高盐和干旱胁迫下大豆GolS基因的表达水平。实验材料是威廉姆斯82 (Williams 82)。将种植于花盆混合土(土︰蛭石=1︰1) 中的Williams 82大豆置于可控温室(光周期为16 h光照/8 h黑暗,温度为25 ℃,生长室相对湿度为60%) 正常生长至15 d后,将大豆幼苗从土壤中轻轻拔出根部,用自来水冲洗根部表面泥土、再用滤纸吸干根部表面水分,用于干旱和盐胁迫处理。干旱处理:将大豆根部的营养土清洗干净置于10% PEG6000的Hoagland营养液进行旱胁迫。盐处理:将清洗干净的大豆根部置于250 mmol/L NaCl的Hoagland营养液中[31]。两种胁迫处理的取样:分别在处理的0、1、2、4、8、12和24 h的时间点取植株叶片样品,做好样品编号。每个取样时间点取3个生物学重复。所有样品均用液氮快速冷冻,并在−80 ℃保存备用。
1.8 GmGolS基因在盐旱胁迫下的表达分析使用植物总RNA提取试剂盒Plant RNApure Kit (Zomanbio, Beijing) 提取上述大豆叶片样品的总RNA,并电泳检测RNA质量。使用反转录试剂盒EasyScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (Transgen, Beijing) 合成cDNA。qRT-PCR用于检测盐和干旱胁迫后不同时期大豆GolS家族成员的基因表达水平。qRT-PCR反应的体系为20 µL:10 µL 2× Real Universal PreMix;1 µL模板cDNA;正反向引物各0.6 µL (10 μmol/L);7.8 μL ddH2O。qRT-PCR反应程序为:预变性95 ℃ 5 min;变性95 ℃ 10 s,退火53 ℃ 15 s,延伸72 ℃ 30 s,40个循环。每个样品设3个技术重复,基因相对表达量采用2−ΔΔCt算法进行计算。采用Excel 2003进行数据统计和绘图。采用SPSS软件进行相对表达量显著性分析(P < 0.05)。所有GmGolS基因及内参基因Actin (U60506) 的引物信息均列于表 1。
| Gene ID | Primer sequence (5′→3′) | Size (bp) |
| Actin (U60506) | F: ACATTGTTCTTAGTGGTGGCT | 21 |
| R: CTGTTGGAAGGTGCTGAG | 18 | |
| Glyma.03G222000 | F: TACCCACCCGAAAACCAAA | 19 |
| R: TCGCAGAAACAATCCATCA | 19 | |
| Glyma.03G229800 | F: TGCTGGAAACGGTGATTAT | 19 |
| R: GGTCTGGTTCTTAGGAGGG | 19 | |
| Glyma.10G145300 | F: TCTAAGCCTTGGAGGTACACTGG[32] | 23 |
| R: GGCACGGACGAACTTGACTTC | 21 | |
| Glyma.19G219100 | F: GCGAGATCGAACCCGTTTA | 19 |
| R: CCTGAATGTCTCCGTCCAA | 19 | |
| Glyma.19G227800 | F: GCGGTGATGGATTGTTTCTG[17] | 20 |
| R: GTGGGCTTGGTGAGTTGGA | 19 | |
| Glyma.20G094500 | F: GTGACTATGTGAAAGGTGTCGTTGG | 25 |
| R: GATTCTCAGGAGGGTACACGGGTTC | 25 |
利用已知拟南芥GolS蛋白序列在大豆数据库共筛选出6个大豆GolS基因,并获得了这些基因(蛋白) 的理化特征和分子信息(表 2)。6个大豆GolS基因编码的蛋白质长度为324− 339 aa氨基酸,其中,Glyma.03G222000蛋白序列最长(339 aa),Glyma.20G094500蛋白序列最短(324 aa);分子量大小在37 567.07 Da (Glyma.20G094500) 到38 817.59 Da (Glyma. 03G222000) 之间。6个蛋白质的等电点变幅为5.45 (Glyma.19G219100)−6.08 (Glyma.03G229800)。利用WoLF PSORT (http://.genscript.com/wolf-psort.html) 在线工具对6个蛋白进行亚细胞定位预测,预测结果显示,Glyma.03G222000、Glyma.03G229800、Glyma.10G145300、Glyma. 19G227800这4个蛋白位于叶绿体上,Glyma. 19G219100、Glyma.20G094500均位于质膜上。
| Gene | Length /aa | Gene position | MW (Da) | pI | Subcellular localization |
| Glyma.03G222000 | 339 | Chr03:42 494 622-42 497 111 | 38 817.59 | 5.47 | Chloroplast |
| Glyma.03G229800 | 331 | Chr03:43 172 456-43 175 687 | 38 097.66 | 6.08 | Chloroplast |
| Glyma.10G145300 | 328 | Chr10:38 014 452-38 016 396 | 38 027.61 | 5.79 | Chloroplast |
| Glyma.19G219100 | 335 | Chr19:47 148 224-47 150 373 | 38 362.12 | 5.45 | Cytoplasm |
| Glyma.19G227800 | 330 | Chr19:47 911 129-47 914 214 | 38 052.51 | 5.48 | Chloroplast |
| Glyma.20G094500 | 324 | Chr20:33 759 416-33 761 555 | 37 567.07 | 5.79 | Cytoplasm |
6个大豆GolS基因的详细位置信息来自Phytozome (表 2),借助TBtools软件制作了染色体位置图(图 1)。染色体定位结果表明,6个GmGolS基因分布在4条大豆染色体上。3号和19号染色体上各有2个基因,10号和20号染色体上各有1个基因。

|
| 图 1 大豆GolS基因家族的染色体定位分析 Fig. 1 Chromosomal localization of GmGolS genes. |
| |
使用DNAMAN软件对拟南芥和大豆GolS基因家族成员的蛋白序列进行比对(图 2)。比对结果表明,6种大豆GolS蛋白的氨基酸序列均包含GolS蛋白的共同特性,即DXD基序。DXD基序是许多糖基转移酶(glycosyl transferase, GT) 家族中的保守基序,它与Mn2+相互作用并与NDP-糖(二磷酸核苷酸-糖) 供体结合[33-35]。此外,6种大豆GolS蛋白的羧基末端均包含五肽基序APSAA。意味着6种大豆GolS蛋白的氨基酸序列是保守的。

|
| 图 2 利用DNAMAN软件生成的拟南芥和大豆GolS蛋白质氨基酸序列比对图 Fig. 2 The result of BLASTp of GolS proteins of Arabidopsis thaliana and soybean generated by DNAMAN software. DXD and C-terminal pentapeptide (APSAA) were highlighted with yellow squares. |
| |
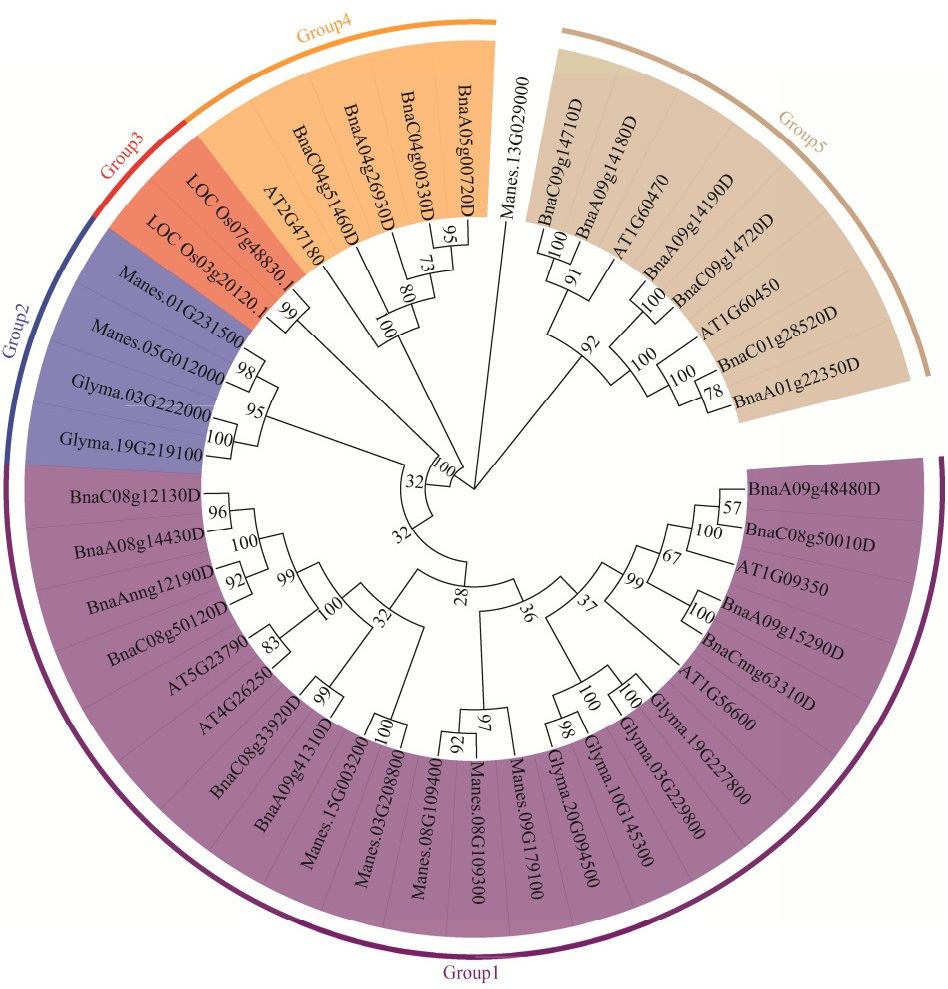
根据文献报道,下载了拟南芥(7个)、水稻(2个)、木薯(8个)、油菜(20个) 以及本文中鉴定的大豆(6个) GolS蛋白质序列,总共43个蛋白序列,构建系统发育树(图 3)。这43个GolS蛋白可被分为5组,即Group 1、Group 2、Group 3、Group 4和Group 5。Group 1中包含的成员最多,包括了来自油菜(10个)、拟南芥(4个)、木薯(5个)、大豆(4个) 等共23个成员。Group 2包括了来自于木薯(2个) 和大豆(2个) 这两个物种的4个GolS成员,Group 3则只包含2个水稻GolS成员,Group 4和Group 5均由拟南芥和油菜这两个物种的GolS成员组成,分别包括1个、2个拟南芥GolS成员和4个、6个油菜GolS成员。进化树聚类结果发现,大豆GolS家族基因成员在进化树上呈现出两两紧邻的现象,这从一定程度上说明大豆GolS基因家族成员之间的保守性较高。
|
| 图 3 来自5个物种43个GolS家族成员的进化树分析 Fig. 3 Phylogenetic tree of 43 members of GolS family in 5 species. At: Arabidopsis thaliana; Bna: Brassica napus; LOC: Oryza sativa; Manes: Manihot esculenta Crantz; Gm: Glycine max. |
| |
对大豆GolS家族基因进行了外显子-内含子基因结构分析(图 4)。4个基因(Glyma.03G229800、Glyma.10G145300、Glyma. 19G227800和Glyma.20G094500) 均含有3个内含子、4个外显子;另外2个基因(Glyma. 03G222000和Glyma.19g219100) 均有2个内含子、3个外显子。表明GmGolS基因在结构上也是高度保守的。一般认为,外显子和内含子的长度、数量一致的基因,可能具有相似的功能。

|
| 图 4 六个大豆GolS家族成员的基因结构分析 Fig. 4 Analysis of gene structure of six GolS members in soybean. |
| |
此外,利用MEME在线工具分析了GmGolS蛋白的氨基酸序列保守结构域(图 5)。结果发现,6个大豆GolS蛋白都含有1−10个不等的Motifs,其中,Motif 1、Motif 2、Motif 3、Motif 4、Motif 5、Motif 6、Motif 7和Motif 8均存在于6个大豆GolS蛋白中,意味着这8个保守基序可能是大豆GolS家族蛋白的特征基序。推测具有相同保守基序的GmGolS蛋白可能执行相同或相似的功能。

|
| 图 5 六个大豆GolS家族成员蛋白的保守基序分析 Fig. 5 Analysis of conserved motifs of six GolS proteins in soybean. |
| |
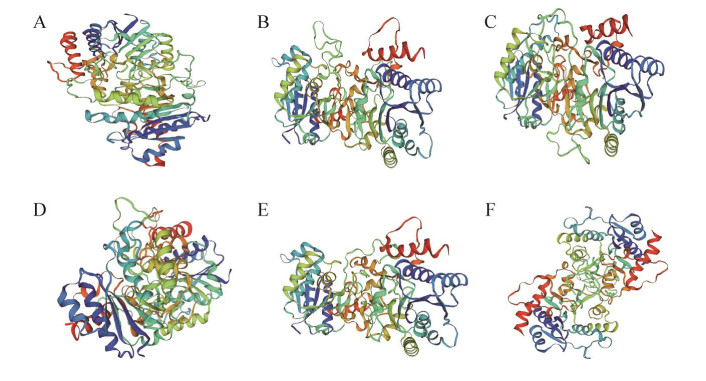
对大豆GolS蛋白质进行了二级结构和三级结构预测分析(表 3和图 6)。结果表明,6个大豆GolS蛋白质的二级结构中均含有α螺旋、延伸链、β转角和无规则卷曲结构。其中,α螺旋结构数量比例为39.02%−43.81%,延伸链结构数量比例为10.91%−12.96%,β转角结构数量比例为2.78%−4.55%,无规则卷曲结构数量比例为41.39%−44.21%。可见,6个大豆GolS蛋白的二级结构均是以α螺旋和无规则卷曲结构为主要组成部分。三级结构预测结果进一步揭示,该家族所有成员空间结构主要由α-螺旋和无规则卷曲结构组成,有较少的β转角结构和延伸链结构(图 6)。
| Protein | Protein length/Percentage (%) | |||
| α-helical structure | Extended chain structure | β-turn structure | Random coil structure | |
| Glyma.03G222000 | 136/40.12 | 42/12.39 | 15/4.42 | 146/43.07 |
| Glyma.03G229800 | 145/43.81 | 39/11.78 | 10/3.02 | 137/41.39 |
| Glyma.10G145300 | 128/39.02 | 42/12.80 | 13/3.96 | 145/44.21 |
| Glyma.19G219100 | 134/40.00 | 42/12.54 | 14/4.18 | 145/43.28 |
| Glyma.19G227800 | 140/42.42 | 36/10.91 | 15/4.55 | 139/42.12 |
| Glyma.20G094500 | 137/42.28 | 42/12.96 | 9/2.78 | 136/41.98 |
|
| 图 6 GmGolS家族蛋白三级结构预测 Fig. 6 Prediction of tertiary structure of GmGolS proteins. (A) Glyma.03G222000. (B) Glyma.03G229800. (C) Glyma.10G145300. (D) Glyma.19G219100. (E) Glyma.19G227800. (F) Glyma.19G227800. |
| |
为明确GolS基因家族成员可能的生物学功能和响应特性,利用PlantCARE对家族各成员启动子序列中包含的顺式作用元件进行了分析。结果表明,在6个大豆GolS基因启动子中鉴定出17个胁迫响应元件,包括CCAAT-box、G-Box、ABRE、TGACG-motif、CGTCA-motif、TCCC-motif、TCT-motif、ARE、Box 4、TCA-element、circadian、MBS、TC-rich repeats、ATCT-motif、MRE、CCAAT-box和AAAC-motif (表 4和图 7)。大豆GolS家族成员具有多种与激素和非生物胁迫响应相关的顺式作用元件,推测GmGolS基因可能通过不同的激素调控途径参与大豆的生长发育,并与多种非生物胁迫响应调控有关。
| Site name | Sequence | Function of the cis-elements |
| CCAAT-box | CAACGG | MYBHv1 binding site |
| G-Box | CACGTG | Cis-acting regulatory element involved in light responsiveness |
| ABRE | CACGTG | Cis-acting element involved in the abscisic acid responsiveness |
| TCCC-motif | TCTCCCT | Part of a light responsive element |
| CGTCA-motif | CGTCA | Cis-acting regulatory element involved in the MeJA-responsiveness |
| TCCC-motif | TCTCCCT | Part of a light responsive element |
| Box 4 | ATTAAT | Part of a conserved DNA module involved in light responsiveness |
| ARE | AAACCA | Cis-acting regulatory element essential for the anaerobic induction |
| TCA-element | CCATCTTTTT | Cis-acting element involved in salicylic acid responsiveness |
| circadian | CAAAGATATC | Cis-acting regulatory element involved in circadian control |
| MBS | CAACTG | MYB binding site involved in drought-inducibility |
| TC-rich repeats | GTTTTCTTAC | Cis-acting element involved in defense and stress responsiveness |
| ATCT-motif | AATCTAATCC | Part of a conserved DNA module involved in light responsiveness |
| MRE | AACCTAA | MYB binding site involved in light responsiveness |
| AAAC-motif | CAATCAAAACCT | Light responsive element |
| TGACG-motif | TGACG | Cis-acting regulatory element involved in the MeJA-responsiveness |
| TCT-motif | TCTTAC | part of a light responsive element |

|
| 图 7 大豆GolS基因家族成员的顺式作用元件分析 Fig. 7 Cis-acting element analysis of GolS genes in soybean. |
| |
为了研究大豆GolS基因家族成员在各组织器官中的表达情况,利用Phytozome数据库下载大豆GolS基因家族成员在花、叶、根瘤、豆荚、根、根毛、种子和茎等8种组织中的转录数据(图 8)。结果表明,大豆GolS基因在不同组织器官中的表达模式存在差异,Glyma.19G227800在花中表达量最高,在茎中表达量最低;Glyma.10G145300在根中的表达量最高;Glyma.03G222000在豆荚和茎中表达量最高;Glyma.20G094500在豆荚和根中表达量相对较高;Glyma.03G229800在花和茎中的表达量相对较高,在叶中表达量最低;Glyma.19G219100在8个不同组织器官中表达量均比较低。可见GmGolS基因家族成员在大豆组织中具有不同的表达模式,这些基因表达的组织特异性意味着它们在不同组织中行使不同的功能。

|
| 图 8 大豆GolS基因家族的组织表达分析 Fig. 8 Tissue expression analysis of soybean GolS gene family. |
| |
为鉴定大豆GolS基因可能在响应盐/旱胁迫方面的功能,进一步分析了6个GmGolS基因在干旱和盐胁迫下的表达情况(图 9–10)。结果表明,在干旱胁迫下,Glyma.03G222000和Glyma.19G219100基因均在8 h表达量达到峰值,之后表达量有所下降、但仍维持显著上调水平;而Glyma.03G229800、Glyma.10G145300和Glyma.19G227800在胁迫1−24 h的表达量均显著上调,但期间的波动变化较大;Glyma.20G094500基因则仅在2 h和4 h相对于对照0 h表达量显著增加,在其余时间点的表达量与对照0 h相比无显著变化。在盐胁迫下,Glyma.20G094500、Glyma.10G145300、Glyma.03G229800和Glyma.19G227800这4个基因的表达分别在盐胁迫处理2 h (约90倍)、2 h (约17倍)、12 h (约42倍) 和24 h (约28倍) 达到峰值。Glyma.03G222000和Glyma.19G219100这2个基因均在盐处理12 h显著上调(分别约为18倍和40倍)。总体来说,研究结果揭示这些GmGolS基因对干旱和盐胁迫的响应具有复杂而重要的作用。

|
| 图 9 GolS基因家族成员在干旱胁迫下的表达模式分析不同小写字母表示差异显著性(P < 0.05) Fig. 9 Expression pattern analysis of GmGolS genes under drought stress. Different lowercase letters indicate significant differences (P < 0.05). |
| |

|
| 图 10 GolS基因家族成员在盐胁迫下的表达模式分析 Fig. 10 Expression pattern analysis of GmGolS genes under salt stress. Different lowercase letters indicate significant differences (P < 0.05). |
| |
GolS参与RFO生物合成的初始阶段,在植物中发挥着重要作用。在拟南芥[15]、烟草[19]、小麦[36]、番茄[37]、鹰嘴豆[12]等植物中开展了很多关于GolS基因克隆、基因功能鉴定等相关研究。在大豆中,也有研究报道了GmGolS2-1基因(对应本文基因Glyma.19G227800)[17]和GmGolS1基因(对应本文基因Glyma. 03G222000)[38]能提高转基因烟草的耐热性。研究表明,对野生型和GmGolS2-1转基因烟草植株进行高温胁迫处理,转基因烟草的电解质渗透率和丙二醛含量均低于野生型烟草,从而提高了转基因烟草的耐热性[17]。在高温胁迫下的大豆幼苗中GmGolS1表达量明显升高。同时,分析了野生型与GmGolS1转基因烟草植株在高温胁迫下的长势及电解质渗透率、可溶性糖含量及丙二醛含量等生理指标,结果表明GmGolS1提高了转基因烟草的耐高温能力[38]。然而,关于大豆GolS家族成员的全基因组鉴定及相关生物信息学特征、家族成员耐逆功能分析等相关研究尚未见报道。本研究首次对大豆GolS基因家族成员进行生物学信息分析,并对其耐盐抗旱功能进行了初步鉴定。
利用生物信息学共鉴定出6个大豆GolS家族成员,分布于4条染色体上。综合前人研究结果可以发现,不同作物GolS基因家族成员的基因结构及其蛋白质的理化性质相差不大。比如,在本研究中,大豆GolS蛋白的氨基酸数目在324−339 aa之间,等电点在5.45−6.08之间,基因的内含子在1−3个之间(表 2和图 4);而番茄的4个GolS蛋白的氨基酸数目在318−347 aa之间,等电点在5.35− 6.40之间,基因的内含子在3−4个之间[37];苹果的8个GolS蛋白的氨基酸长度在298−359 aa之间,基因的内含子在2−4个之间[39]。据此推测,GolS基因及其所编码蛋白质在植物中具有较高的保守性。进一步基于拟南芥、水稻、油菜、木薯、大豆等5个物种的43个GolS蛋白序列进行了系统进化分析。结果表明,6个GmGolS基因家族成员与其余4个物种的GolS成员聚为5组(图 3),大豆GolS家族基因成员在进化树上呈现出两两紧邻的现象,这从一定程度上说明大豆GolS家族成员之间的保守性较高。基因结构分析发现,6个大豆GolS家族成员中,有4个成员含有3个内含子、4个外显子,2个成员含有2个内含子、3个外显子。保守基序分析表明,motif基序数量从1到10个不等。该结果与前人在烟草[19]、油菜[19]、番茄[37]中的研究结果相近。表明了该基因家族在物种间具有较高的保守性。
亚细胞定位预测结果显示,GmGolS蛋白在细胞质和叶绿体上均有分布。前人对木薯[23]、油菜[19]、烟草[19]GolS蛋白成员进行亚细胞定位预测,发现其GolS蛋白在细胞质、叶绿体、线粒体以及质膜均有分布。GolS蛋白的不同亚细胞定位结果,预示着它们可能具有不同的生物学功能。蛋白质的二级结构和三级结构预测结果揭示α螺旋、无规则卷曲是大豆GolS空间结构的主要组成部分。顺式作用元件分析在GmGolS基因启动子区域上检测到多个与激素和非生物胁迫相关的顺式作用元件,推测GmGolS基因可能通过不同的激素调控途径参与大豆的生长发育,并与多种非生物胁迫响应调控有关。
大豆GolS家族成员组织表达模式分析结果表明,GmGolS基因具有不同的组织表达模式,暗示着它们在大豆生长发育的过程中可能行使不同的功能。如Glyma.10G145300在根中的表达量最高,表明该基因可能在大豆根发育过程中起着重要作用。本研究分析了6个大豆GolS基因响应盐旱胁迫的表达模式,结果与毛果杨PtrGolS基因家族成员在盐、旱胁迫下的表达模式[40]相似,这些基因均在盐旱胁迫下表现不同程度的显著上调表达,表明这些基因在植物响应盐旱胁迫过程中发挥了比较复杂而重要的作用。研究结果为深入解析大豆GolS基因功能提供了科学信息。
综上,本研究在大豆基因组中鉴定出6个GolS基因,分布在4条染色体上,具有相对保守的基因结构和保守基序,并检测出多个与激素和非生物胁迫相关的顺式作用元件。所有大豆GolS基因都在盐和旱处理后出现显著上调表达、但上调表达变化模式各有不同,推测大豆GolS基因在响应盐旱胁迫的过程中发挥着复杂而重要的作用。研究结果为进一步解析大豆GolS基因功能奠定了基础。
| [1] |
Wang WX, Vinocur B, Altman A. Plant responses to drought, salinity and extreme temperatures: towards genetic engineering for stress tolerance. Planta, 2003, 218(1): 1-14. DOI:10.1007/s00425-003-1105-5
|
| [2] |
Theocharis A, Clément C, Barka EA. Physiological and molecular changes in plants grown at low temperatures. Planta, 2012, 235(6): 1091-1105. DOI:10.1007/s00425-012-1641-y
|
| [3] |
Wang WX, Vinocur B, Shoseyov O, et al. Biotechnology of plant osmotic stress tolerance physiological and molecular considerations. Acta Hortic, 2001(560): 285-292.
|
| [4] |
Albini FM, Murelli C, Finzi PV, et al. Galactinol in the leaves of the resurrection plant Boea hygroscopica. Phytochemistry, 1999, 51(4): 499-505. DOI:10.1016/S0031-9422(99)00031-X
|
| [5] |
Collett H, Shen A, Gardner M, et al. Towards transcript profiling of desiccation tolerance in Xerophyta humilis: construction of a normalized 11 k X. humilis cDNA set and microarray expression analysis of 424 cDNAs in response to dehydration. Physiol Plant, 2004, 122(1): 39-53. DOI:10.1111/j.1399-3054.2004.00381.x
|
| [6] |
Gechev TS, Dinakar C, Benina M, et al. Molecular mechanisms of desiccation tolerance in resurrection plants. Cell Mol Life Sci, 2012, 69(19): 3175-3186. DOI:10.1007/s00018-012-1088-0
|
| [7] |
Peters S, Mundree SG, Thomson JA, et al. Protection mechanisms in the resurrection plant Xerophyta viscosa (Baker): both sucrose and raffinose family oligosaccharides (RFOs) accumulate in leaves in response to water deficit. J Exp Bot, 2007, 58(8): 1947-1956. DOI:10.1093/jxb/erm056
|
| [8] |
余箬芊, 王福祥, 郑燕梅, 等. 植物棉子糖家族寡糖(RFOs)在种子活力及非生物胁迫中的生物学功能研究进展. 福建农业学报, 2022, 37(1): 114-122. Yu RQ, Wang FX, Zheng YM, et al. Research advances on the biological function of raffinose families oligosaccharides in seed vigor and abiotic stress. Fujian J Agric Sci, 2022, 37(1): 114-122 (in Chinese). DOI:10.19303/j.issn.1008-0384.2022.01.015 |
| [9] |
Cho SM, Kang EY, Kim MS, et al. Jasmonate- dependent expression of a galactinol synthase gene is involved in priming of systemic fungal resistance in Arabidopsis thaliana. Botany, 2010, 88(5): 452-461. DOI:10.1139/B10-009
|
| [10] |
Kim MS, Cho SM, Kang EY, et al. Galactinol is a signaling component of the induced systemic resistance caused by Pseudomonas chlororaphis O6 root colonization. Mol Plant Microbe Interact, 2008, 21(12): 1643-1653. DOI:10.1094/MPMI-21-12-1643
|
| [11] |
Gu L, Zhang YM, Zhang MS, et al. ZmGOLS2, a target of transcription factor ZmDREB2A, offers similar protection against abiotic stress as ZmDREB2A. Plant Mol Biol, 2016, 90(1-2): 157-170. DOI:10.1007/s11103-015-0403-1
|
| [12] |
Salvi P, Kamble NU, Majee M. Stress-inducible galactinol synthase of chickpea (CaGolS) is implicated in heat and oxidative stress tolerance through reducing stress-induced excessive reactive oxygen species accumulation. Plant Cell Physiol, 2017, 59(1): 155-166.
|
| [13] |
Nishizawa A, Yabuta Y, Shigeoka S. Galactinol and raffinose constitute a novel function to protect plants from oxidative damage. Plant Physiol, 2008, 147(3): 1251-1263. DOI:10.1104/pp.108.122465
|
| [14] |
ElSayed AI, Rafudeen MS, Golldack D. Physiological aspects of raffinose family oligosaccharides in plants: protection against abiotic stress. Plant Biol, 2014, 16(1): 1-8. DOI:10.1111/plb.12053
|
| [15] |
Taji T, Ohsumi C, Iuchi S, et al. Important roles of drought- and cold-inducible genes for galactinol synthase in stress tolerance in Arabidopsis thaliana. Plant J, 2002, 29(4): 417-426. DOI:10.1046/j.0960-7412.2001.01227.x
|
| [16] |
Mukherjee S, Sengupta S, Mukherjee A, et al. Abiotic stress regulates expression of galactinol synthase genes post-transcriptionally through intron retention in rice. Planta, 2019, 249(3): 891-912. DOI:10.1007/s00425-018-3046-z
|
| [17] |
邱爽, 张军, 何佳琦, 等. 大豆GmGolS2-1基因高温胁迫诱导表达及转基因烟草鉴定. 江苏农业学报, 2021, 37(1): 38-43. Qiu S, Zhang J, He JQ, et al. Expression of soybean GmGolS2-1 induced by heat stress and identification of transgenic tobacco. Jiangsu J Agric Sci, 2021, 37(1): 38-43 (in Chinese). DOI:10.3969/j.issn.1000-4440.2021.01.005 |
| [18] |
Liu L, Wu XL, Sun WB, et al. Galactinol synthase confers salt-stress tolerance by regulating the synthesis of galactinol and raffinose family oligosaccharides in poplar. Ind Crops Prod, 2021, 165: 113432. DOI:10.1016/j.indcrop.2021.113432
|
| [19] |
Fan YH, Yu MN, Liu M, et al. Genome-wide identification, evolutionary and expression analyses of the GALACTINOL SYNTHASE gene family in rapeseed and tobacco. Int J Mol Sci, 2017, 18(12): 2768. DOI:10.3390/ijms18122768
|
| [20] |
De Gois EHB, Menegazzo RF, dos Santos TB, et al. Identification, evolutionary and expression analysis of the galactinol synthase (GolS) genes in Panicum virgatum L. and Panicum hallii: an in silico approach. Plant Gene, 2020, 24: 100262. DOI:10.1016/j.plgene.2020.100262
|
| [21] |
You J, Wang Y, Zhang Y, et al. Genome-wide identification and expression analyses of genes involved in raffinose accumulation in sesame. Sci Reports, 2018, 8(1): 4331.
|
| [22] |
Selvaraj MG, Ishizaki T, Valencia M, et al. Overexpression of an Arabidopsis thaliana galactinol synthase gene improves drought tolerance in transgenic rice and increased grain yield in the field. Plant Biotechnol J, 2017, 15(11): 1465-1477. DOI:10.1111/pbi.12731
|
| [23] |
Huang TW, Luo XL, Fan ZP, et al. Genome-wide identification and analysis of the sucrose synthase gene family in cassava (Manihot esculenta Crantz). Gene, 2021, 769: 145191. DOI:10.1016/j.gene.2020.145191
|
| [24] |
Zhuo CL, Wang T, Lu SY, et al. A cold responsive galactinol synthase gene from Medicago falcata (MfGolS1) is induced by myo-inositol and confers multiple tolerances to abiotic stresses. Physiol Plant, 2013, 149(1): 67-78. DOI:10.1111/ppl.12019
|
| [25] |
Zhou Y, Liu Y, Wang SS, et al. Molecular cloning and characterization of galactinol synthases in Camellia sinensis with different responses to biotic and abiotic stressors. J Agric Food Chem, 2017, 65(13): 2751-2759. DOI:10.1021/acs.jafc.7b00377
|
| [26] |
Liu YD, Zhang L, Chen LJ, et al. Molecular cloning and expression of an encoding galactinol synthase gene (AnGolS1) in seedling of Ammopiptanthus nanus. Sci Rep, 2016, 6: 36113. DOI:10.1038/srep36113
|
| [27] |
盖钧镒. 发展我国大豆遗传改良事业解决国内大豆供给问题. 中国工程科学, 2003, 5(5): 1-6. Gai JY. Expanding and enhancing the research allocation on soybean breeding and genetics for the establishment of market supply based on domestic production. Eng Sci, 2003, 5(5): 1-6 (in Chinese). DOI:10.3969/j.issn.1009-1742.2003.05.001 |
| [28] |
Saitou N, Nei M. The neighbor-joining method: a new method for reconstructing phylogenetic trees. Mol Biol Evol, 1987, 4(4): 406-425.
|
| [29] |
Kumar S, Stecher G, Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Mol Biol Evol, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054
|
| [30] |
Chen C, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data. Mol Plant, 2020(8): 1194-1202.
|
| [31] |
Du YT, Zhao MJ, Wang CT, et al. Identification and characterization of GmMYB118 responses to drought and salt stress. BMC Plant Biol, 2018, 18(1): 320. DOI:10.1186/s12870-018-1551-7
|
| [32] |
张军, 邱爽, 何佳琦, 等. 大豆GmGolS基因植物表达载体构建及烟草遗传转化. 齐齐哈尔大学学报(自然科学版), 2020, 36(6): 22-25. Zhang J, Qiu S, He JQ, et al. Plant expression vector construction and tobacco genetic transformation of soybean GmGolS gene. J Qiqihar Univ (Nat Sci Ed), 2020, 36(6): 22-25 (in Chinese). DOI:10.3969/j.issn.1007-984X.2020.06.006 |
| [33] |
Wiggins CA, Munro S. Activity of the yeast MNN1 alpha-1, 3-mannosyltransferase requires a motif conserved in many other families of glycosyltransferases. PNAS, 1998, 95(14): 7945-7950. DOI:10.1073/pnas.95.14.7945
|
| [34] |
Gibbons BJ, Roach PJ, Hurley TD. Crystal structure of the autocatalytic initiator of glycogen biosynthesis, glycogenin. J Mol Biol, 2002, 319(2): 463-477. DOI:10.1016/S0022-2836(02)00305-4
|
| [35] |
Persson K, Ly HD, Dieckelmann M, et al. Crystal structure of the retaining galactosyltransferase LgtC from Neisseria meningitidis in complex with donor and acceptor sugar analogs. Nat Struct Biol, 2001, 8(2): 166-175. DOI:10.1038/84168
|
| [36] |
Wang YG, Liu HH, Wang SP, et al. Overexpression of a common wheat gene GALACTINOL SYNTHASE3 enhances tolerance to zinc in Arabidopsis and rice through the modulation of reactive oxygen species production. Plant Mol Biol Report, 2016, 34(4): 794-806. DOI:10.1007/s11105-015-0964-9
|
| [37] |
Filiz E, Ozyigit II, Vatansever R. Genome-wide identification of galactinol synthase (GolS) genes in Solanum lycopersicum and Brachypodium distachyon. Comput Biol Chem, 2015, 58: 149-157. DOI:10.1016/j.compbiolchem.2015.07.006
|
| [38] |
李铭杨, 邱爽, 何佳琦, 等. 大豆GmGolS1的克隆及转基因烟草耐高温性鉴定. 植物遗传资源学报, 2022, 23(2): 575-582. Li MY, Qiu S, He JQ, et al. Cloning of soybean GmGolS1 and identification of heat resistance in transgenic tobacco. J Plant Genet Resour, 2022, 23(2): 575-582 (in Chinese). |
| [39] |
Falavigna VDS, Porto DD, Miotto YE, et al. Evolutionary diversification of galactinol synthases in Rosaceae: adaptive roles of galactinol and raffinose during apple bud dormancy. J Exp Bot, 2018, 69(5): 1247-1259. DOI:10.1093/jxb/erx451
|
| [40] |
Zhou J, Yang Y, Yu J, et al. Responses of Populus trichocarpa galactinol synthase genes to abiotic stresses. J Plant Res, 2014, 127(2): 347-358. DOI:10.1007/s10265-013-0597-8
|
 2022, Vol. 38
2022, Vol. 38