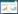中国科学院微生物研究所、中国微生物学会主办
文章信息
- 许凯龙, 聂巍巍, 童倩雯, 马立新, 刘洁, 王洋
- XU Kailong, NIE Weiwei, TONG Qianwen, MA Lixin, LIU Jie, WANG Yang
- 基于单细胞转录组的视网膜母细胞瘤进展特征分析
- Analysis of progress characteristics of retinoblastoma based on single cell transcriptome sequencing
- 生物工程学报, 2022, 38(10): 3809-3824
- Chinese Journal of Biotechnology, 2022, 38(10): 3809-3824
- 10.13345/j.cjb.220491
-
文章历史
- Received: June 23, 2022
- Accepted: September 26, 2022
2. 哈尔滨第四医院 眼科, 黑龙江 哈尔滨 150001
2. Department of Ophthalmology, Harbin Fourth Hospital, Harbin 150001, Heilongjiang, China
视网膜母细胞瘤(retinoblastoma, RB) 是一种源于光感受器前体细胞的恶性肿瘤,多见于3岁以下儿童,是婴幼儿最常见的原发性眼内恶性肿瘤,在世界范围内发病率约为1∶2 0 000[1]。RB早期无临床症状,只能通过眼底检查发现较小的肿瘤。其临床症状复杂多样,多表现为结膜充血水肿、虹膜新生血管、玻璃体混浊、眼压升高及斜视等,且该病具有颅内转移和远处转移的特点,会威胁患儿生命[2],治疗RB需按个体化治疗原则并结合RB病理分期。在肿瘤学上,肿瘤分期对治疗方法的选择及疗效评价极其重要[3]。因此,探究不同进展时期RB的特点、识别与其进展密切相关基因,对RB进展研究及早期诊断具有一定的参考意义。
Cobrinik及其同事们在早期的研究中指出,RB肿瘤起源于视锥细胞前体,RB肿瘤是响应RB1单基因突变而形成[4]。而RB1基因编码了一种肿瘤抑制蛋白Rb,Rb可以通过在细胞做好分裂准备之前抑制细胞周期进程来阻止过度的细胞生长[5],防止细胞恶性增殖进而转化为癌细胞[6]。然而,肿瘤的发生是一个复杂的、多因素过程,同时,在肿瘤整个增殖、扩散、转移的过程当中,必然还存在其他关键因素,进而引起组织在细胞水平发生变化,最终发生病变。因此,挖掘RB肿瘤进展过程中视锥细胞前体显著表达的一些促进增殖及抑制凋亡的关键癌相关蛋白就变得十分重要。
近年来,单细胞转录组测序(single-cell RNA sequencing, scRNA-seq) 技术的出现极大地推动了肿瘤发病机制研究及异质性[7]的解析。RB中的肿瘤细胞具有高度异质性,从单细胞层面解析肿瘤内异质性将更好地揭示肿瘤细胞的来源,识别RB进展相关的细胞组分及关键基因[8]。因此,本研究基于公共基因表达数据库(gene expression omnibus, GEO) 资源,下载RB肿瘤单细胞转录组测序数据,对不同患病时长的RB组织及癌旁组织的转录特征进行解析,详细阐述RB在不同病理时间、不同组织部位的细胞组成、比例差异,并探索其在RB肿瘤进展过程中的关键作用。研究结果显示,视锥前体G1期细胞群、G2期细胞群以及小胶质细胞群在RB肿瘤与癌旁组织中的分布比例存在明显差异。同时,结果显示,随着肿瘤发病时间的增加,小胶质细胞群显著地表达一些促进细胞增殖、转移及免疫应答相关的蛋白,如B2M、RPL23及HLA家族基因。本研究结果可为RB肿瘤发病机制及进展研究提供新的数据资源。
1 材料与方法 1.1 数据的下载与整理本课题的数据下载于GEO数据库,数据编号GSE166173[9]。数据包括1例患病4个月的RB肿瘤癌旁组织(GSM5065164)、1例患病4个月的RB肿瘤实质部分(GSM5065165)、1例患病2年的RB肿瘤癌旁组织(GSM5065166)、1例患病2年的RB肿瘤实质部分(GSM5065167),共4个样本。
1.2 数据分析 1.2.1 数据预处理及质控采用SEURAT[10]R包进行数据处理及相关分析,分析软件及程序版本见表 1。设置质控参数筛选高质量细胞用于后续分析,其中基因数目设定阈值为500−4 000、线粒体基因 < 15%、核糖体基因 < 20%。质控结果已提交国家微生物科学数据中心(编号:NMDCX0000144)。
| Software/package | Version |
| R | 4.1.2 |
| Seurat | 4.0.5 |
| ggplot2 | 3.3.5 |
| dplyr | 1.0.7 |
| tidyverse | 1.3.1 |
| GSEABase | 1.56.0 |
| patchwork | 1.1.1 |
| clusterProfiler | 4.2.0 |
| KEGG.db | 1.0 |
| harmony | 0.1.0 |
| SingleR | 1.8.0 |
采用FINDVARIABLEFEATURES函数寻找前2 000个高变基因,可视化结果见附图 1C (详见《生物工程学报》网络版附件)。采用RUNPCA进行线性降维(N=20),聚类过程中RESOLUTION设定为0.5,对数据集进行SCT标准化[11],使用RUNHARMONY函数[12]消除批次效应。使用UMAP非线性降维可视化聚类结果。采用FindAllMarkers函数识别每个细胞亚群的Marker基因,应用SingleR[13]、Cellmarker数据库[14],并结合参考文献进行人工校正,对细胞亚群进行鉴定。
|
| 图 1 细胞分群及注释 Fig. 1 Cell clustering and annotation. (A) The results after batch effect corrected (by orig.ident). (B) The results after batch effect corrected (by group). (C) Dimensional reduction and clustering on the data after QC. (D) Add annotation for clustering results. (E) The heatmap of top5 marker gene in each cell type. (F) The violinplot of top1 marker gene in each cell types. |
| |
本课题最终选取了3组对象进行差异基因的识别及富集分析:(1) 针对患病4个月肿瘤实质和患病2年肿瘤实质部分的视锥细胞G1期细胞簇(cones: G1 phase);(2) 针对患病4个月肿瘤实质和患病2年肿瘤实质部分的视锥细胞G2期细胞簇(cones: G2 phase);(3) 针对患病4个月肿瘤实质和患病2年肿瘤实质部分的小胶质细胞。针对3组数据中两两对象之间取marker基因得到差异基因集,显著性阈值设置为p_val_adj < 0.05,|avg_log2FC| > 0.5。针对3组数据中识别到的差异基因进行基因本体论(gene ontology, GO)[15]、京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes, KEGG)[16]和基因集富集分析(gene set enrichment analysis, GSEA)[17],其中GSEA分析中的基因集来自MSigDB[18]数据库中癌症特征基因集数据(h: hallmark gene sets, http://www.gsea-msigdb.org/gsea/msigdb/download_file.jsp?filePath=/msigdb/release/7.5.1/msigdb.v7.5.1.symbols.gmt)。
2 结果与分析 2.1 RB肿瘤微环境单细胞图谱的刻画通过对数据进行低质量细胞过滤及相关质控,共保留5 260个细胞用于后续相关分析。其中,3 626个细胞来自RB肿瘤组织(其中来自4个月RB肿瘤组织的共3 044个细胞,来自2年RB肿瘤组织的共581个细胞);1 635个来自RB癌旁组织(来自4个月RB癌旁组织的335个细胞,来自2年RB癌旁组织的1 300个细胞),采用统一流形逼近与投影(uniform manifold approximation and projection, UMAP) 对细胞群体分布进行投影可视化,结果显示整合后的细胞群不存在批次效应,整体分布相一致(图 1A–B)。无监督聚类分析将5 260个细胞划分为19个细胞亚群(图 1C)。
进一步,对每个细胞亚群进行标记基因的识别,结合SINGELR、CELLMARKER数据库,以及相关文献中细胞的标志基因进行人工校正,最终将19个无监督聚类的细胞亚群注释为11类细胞群(图 1D)。包括G1、G2、G1+S+G2/M、胶质细胞团(MULLER GLIA)、星形胶质细胞团(ASTROCYTES)、PMEL+、G1+S、小胶质细胞团(MICROGLIA)、G1+G2/M、内皮细胞(ENDOTHELIUM)、RGS5+。其中,G1、S、G2/M分别指视锥细胞前体处于G1期、S期、G2/M期的细胞团;PMEL+与RGS5+分别代表PMEL和RGS5基因上调表达的细胞团。结果显示,RB肿瘤不同组织部位的细胞分布整体相似,但个别细胞群的丰度会因肿瘤发生阶段不同而存在差异,这些细胞群主要是一些细胞周期相关的细胞亚群,如G1亚群、G2亚群、G1+G2/M亚群等。各细胞亚群的前5个标志基因热图可视化结果及代表基因的小提琴图分布见图 1E–F。
2.2 RB肿瘤异质性分析及特征细胞亚群的识别本研究统计了RB肿瘤不同发病时间、不同组织采样部位中各类型细胞的含量,用于比较各组别间细胞组成及比例的差异,识别差异显著的细胞类型(表 2)。
| 4M_normal | 4M_tumor | 2Y_normal | 2Y_tumor | |
| G1 | 0 (0) | 1 065 (0.349) | 126 (0.096 9) | 120 (0.206 5) |
| G2 | 29 (0.080) | 1 060 (0.348) | 247 (0.190 0) | 289 (0.497 4) |
| Muller Glia | 138 (0.410) | 222 (0.072 9) | 514 (0.395 4) | 50 (0.086 1) |
| G1+S+G2/M | 5 (0.010) | 430 (0.141) | 27 (0.020 8) | 29 (0.049 9) |
| PMEL+ | 0 (0) | 90 (0.029 5) | 163 (0.125 4) | 2 (0.003 4) |
| Astrocytes | 129 (0.385) | 35 (0.011 5) | 10 (0.007 7) | 3 (0.005 2) |
| Microglia | 10 (0.030) | 61 (0.020) | 99 (0.076 2) | 30 (0.051 6) |
| G1+S | 3 (0.009) | 51 (0.017) | 17 (0.013 1) | 15 (0.025 8) |
| G1+G2/M | 0 (0) | 1 (0.000 3) | 46 (0.035 4) | 32 (0.051 6) |
| Endothelium | 16 (0.040) | 16 (0.005 3) | 22 (0.073 3) | 8 (0.013 8) |
| RGS5+ | 5 (0.010) | 13 (0.004 3) | 29 (0.022 3) | 3 (0.005 2) |
| Total | 335 | 3 044 | 1 300 | 581 |
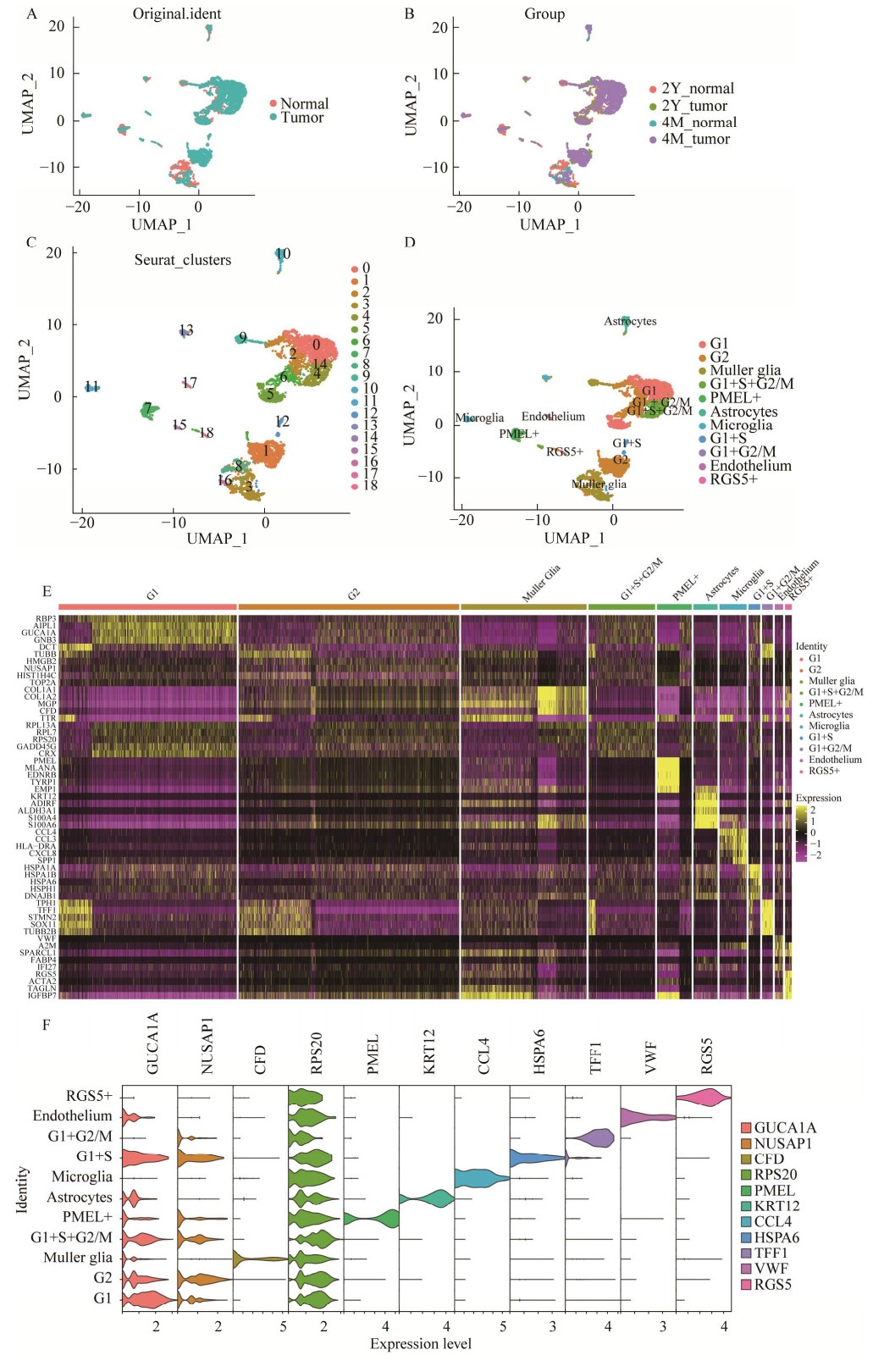
结果显示,随着RB肿瘤的进展,肿瘤组织及癌旁组织中各类细胞亚群比例发生了改变。其中,4个月的癌旁组织中含量最多的是星形胶质细胞(astrocytes),占比38.5%;2年的肿瘤组织中含量最多的是穆勒胶质细胞(Muller Glia),占比39.5%;4个月的肿瘤组织中含量最多的是G1和G2亚群(占比分别为34.9%和34.8%),2年的肿瘤组织中含量最高的细胞类型是G2细胞亚群(占比49.7%)。
与此同时,本研究又根据所处细胞周期阶段对细胞状态进行了统计。结果表明,超过50%的视锥前体细胞处于G2/M期或S期,与Collin等[9]结论一致,揭示RB肿瘤的发生引起了不受抑制的细胞分裂增殖,最终发生癌变。
进一步,本研究按照肿瘤组织、癌旁组织以及肿瘤的持续时间进行分组,统计不同组别的细胞所处周期状态(表 3)。结果表明,癌旁组织中60%−90%的细胞处于G1期,而在肿瘤组织中超过60%的细胞处于G2/M期或S期,且这种细胞比例在不同时期会有明显差异,揭示RB肿瘤组织中大量细胞在异常增殖。
| Original.ident | Group | G1 phase | G2M phase | S phase |
| Normal (1 635) |
4M_normal | 322 (96.1%) | 11 (3.3%) | 2 (0.6%) |
| 2Y_normal | 828 (63.7%) | 287 (22.1%) | 185 (14.2%) | |
| Tumor (3 625) |
4M_tumor | 1 114 (36.6%) | 742 (24.4%) | 1 188 (39.0%) |
| 2Y_tumor | 183 (31.5%) | 217 (37.3%) | 181 (31.2%) |
最后,本研究展示了不同组织、不同进展时间下各细胞类型的分布情况及含量差异(图 2A–B)。结果显示,相较于癌旁组织,肿瘤组织中与细胞周期相关细胞亚群(G1、G2、G1+S+G2/M细胞亚群) 大幅增多,而穆勒胶质细胞、小胶质细胞大幅减少。
|
| 图 2 RB细胞丰度柱状图 Fig. 2 Cell abundance stackbarplot. Cell type composition and content between tumor and normal tissue (A) and among four small samples (B). |
| |
对比4个月肿瘤与2年肿瘤可发现,G1细胞亚群含量减少而G2细胞亚群含量增多。有研究指出[9],RB的发生极有可能是某些基因失活导致视锥前体细胞绕过了G1/S阻滞而进入G2乃至分裂期,引起恶性增殖,进而形成肿瘤。而这两类细胞的比例在肿瘤发生的前后分别呈现降低与升高的趋势,揭示大量处于G1期的视锥细胞无视抑癌基因阻滞而向G2期转化增殖。
2.3 G1期细胞亚群中RB肿瘤进展相关特征基因的识别及筛选提取G1期视锥前体细胞亚群,采用FindMarkers函数对4个月肿瘤及2年肿瘤进行差异分析,共识别到差异基因1 587个,其中129个基因显著上调,其中297个基因显著下调(|avg_log2FC| > 1,p_val_adj < 0.05)。
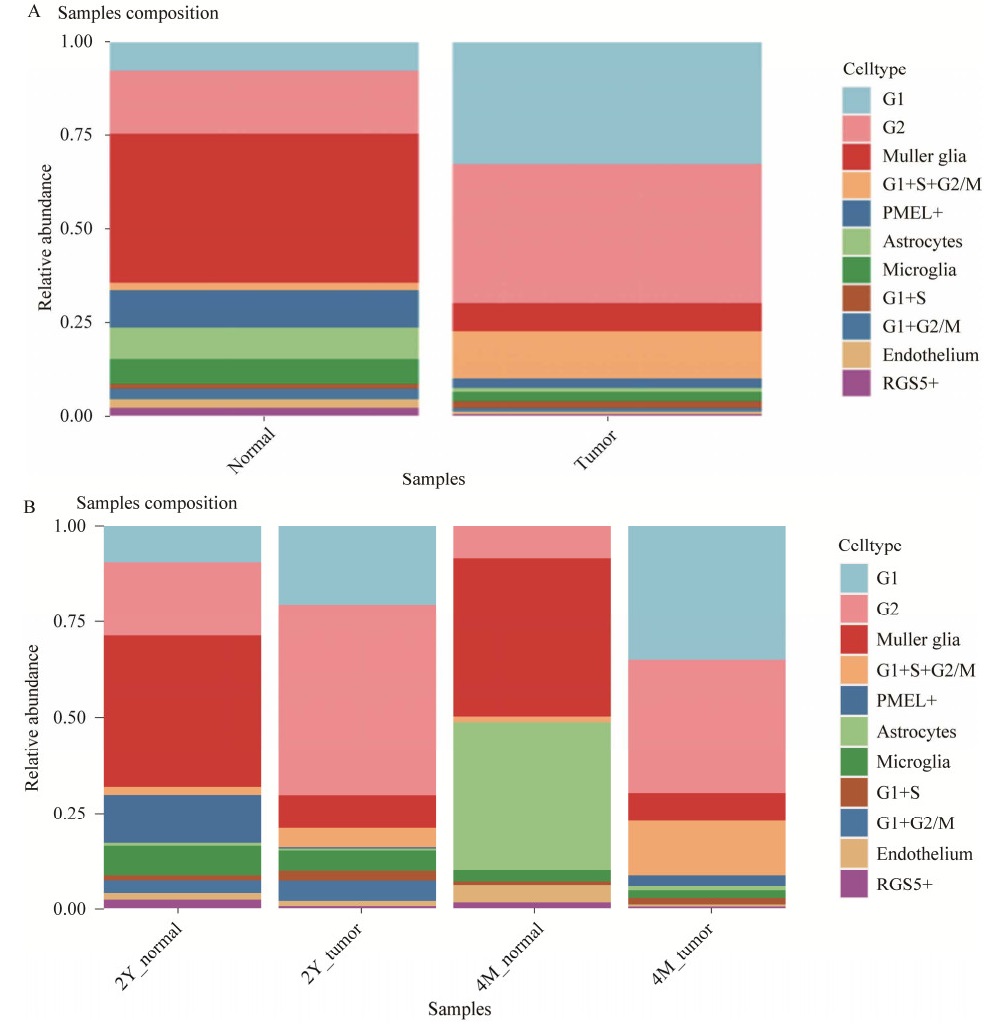
GO功能分析结果显示,差异基因主要富集的生物学过程为ATP代谢过程、细胞质内的翻译等;主要富集的细胞组分为线粒体内膜、核糖体等;主要富集的分子功能为核糖体的结构成分等(图 3A)。这些都是与细胞增殖密切相关,进一步揭示了肿瘤进展过程中细胞功能上的差异主要体现在细胞增殖方面。
|
| 图 3 视锥前体G1期细胞亚群在不同发育年龄的RB肿瘤样本中富集分析 Fig. 3 Enrichment analysis for cones cell in G1 phase of different RB developmental stage. (A) Visualization for GO analysis of differential expressed genes. (B) Visualization for KEGG enrichment analysis of up-regulated and down regulated. (C) Visualization for GSEA analysis of differential exgenes. (D) Gseaplot for G2/M_CHECKPOINT. (E) Gseaplot for E2F_TARGETS. |
| |
KEGG富集分析结果显示,上调基因主要参与的通路包括抗原处理和呈递、吞噬体、甲状腺激素合成、内质网中的蛋白质加工等通路(图 3B),说明这些通路是响应G1期视锥前体在肿瘤发展中的关键通路;下调基因主要富集到核糖体相关通路、氧化磷酸化等生物学过程。其中,氧化磷酸化过程能够以ATP形式产生95%以上的细胞能量,如果该通路被抑制,则会造成ATP合成受阻,进而在一定程度上破坏能量的平衡,最终导致了肿瘤的发生和进展。
进一步,GSEA富集基因结果发现,G1期视锥前体细胞在肿瘤进展过程中,多数富集到的通路表现为激活作用。关键被激活的通路包括E2F靶标、G2/M检查点、未折叠蛋白反应等;关键被抑制的通路为氧化磷酸化通路等(图 3C),其中氧化磷酸化通路在KEGG富集分析的结果中亦有体现。值得注意的是,E2F转录因子家族是参与细胞周期及细胞凋亡重要的调节因子[19],它会以特异方式与RB1基因结合,调节G1到S期细胞周期进程,在S期细胞中表达最高,在G2/M期表达较低[9]。本研究结果发现,E2F靶标、G2/M检查点在肿瘤进展过程受到激活(图 3D–E),导致患者肿瘤细胞在G2/M期失调,未能成功阻止细胞进入M期,无法使细胞的DNA得到修复,癌症患者的病情就得不到控制。
2.4 G2期细胞亚群中RB肿瘤进展相关特征基因的识别及筛选针对G2期细胞亚群进行GSEA富集分析,结果发现未折叠蛋白质反应、MYC TARGETS v1、G2/M_CHECKPOINT等通路在2年的RB肿瘤组织中的是被激活的(图 4A)。而有研究指出,MYC基因在70%的肿瘤中存在过表达或突变,是最常见的高度扩增的癌基因之一[20],揭示G2期的RB肿瘤细胞处于异常不受控制的增殖状态。KEGG富集分析结果表明,上调基因主要参与的通路包括吞噬体、抗原处理和呈递等通路(图 4B),说明此处存在较为活跃的免疫反应,在肿瘤发展过程中其肿瘤微环境发生了变化;下调基因主要参与的通路包括松弛素信号通路、核糖体和PI3K-Akt信号通路等。
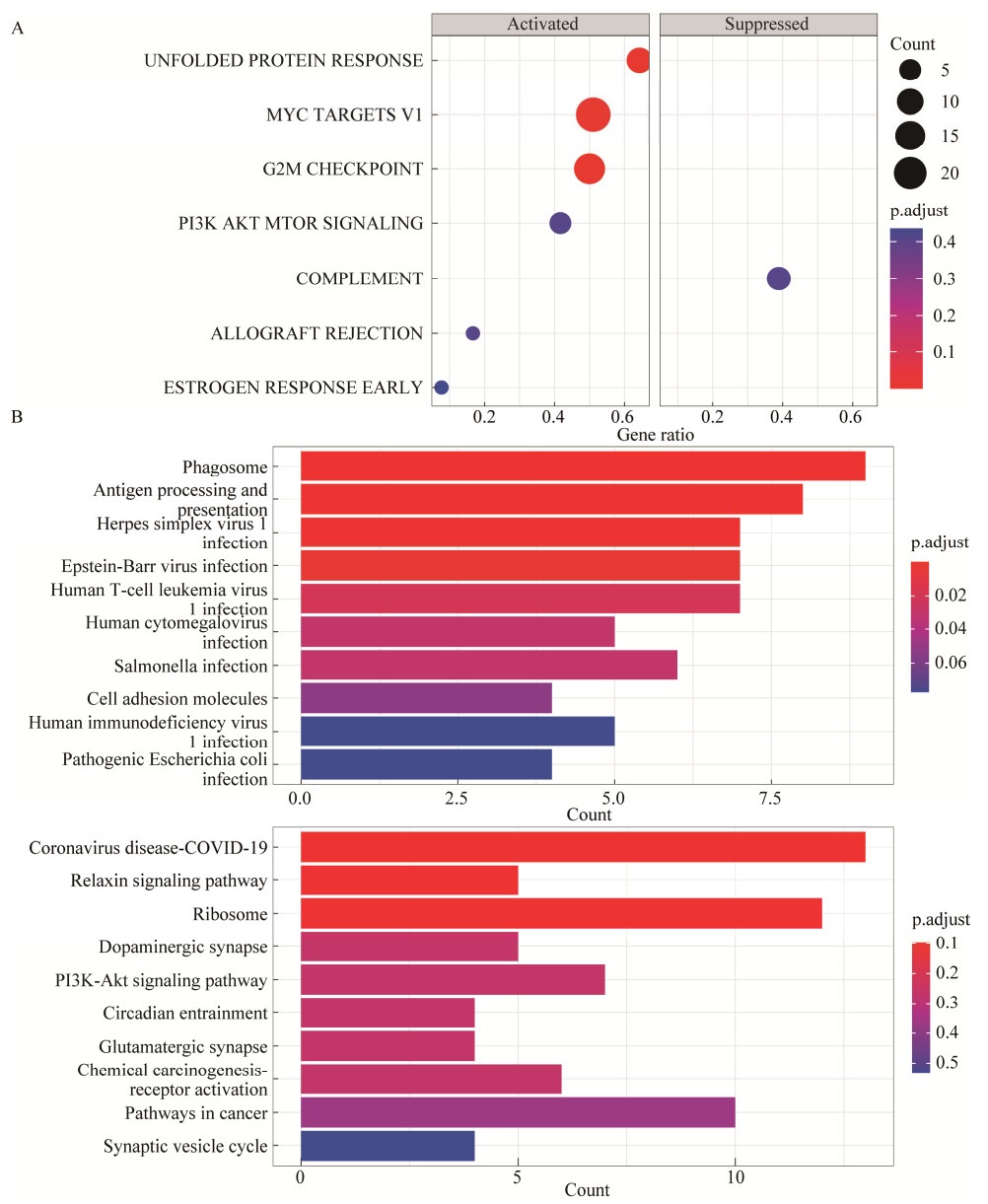
|
| 图 4 视锥前体G2期细胞亚群在不同发育年龄的RB肿瘤样本中富集分析 Fig. 4 Enrichment analysis for cones cell in G2 phase of different RB developmental stage. (A) Visualization for GSEA analysis of differential genes. (B) Visualization for KEGG enrichment analysis of up-regulated and down regulated genes. |
| |
最后,本研究重点分析了4个月RB肿瘤与两年RB肿瘤组织中microglia细胞亚群异质性,共提取差异基因1 410个,其中显著上调基因16个,显著下调基因17个(|avg_log2FC| > 1, p_val_adj < 0.05)。显著上调和下调的Top6基因集及相关信息见表 4。
| TOP6 up-regulated and down regulated gene |
Avg_log2FC |
| Up-regulated gene | |
| HLA-C | 2.450 1 |
| IFI6 | 2.430 7 |
| HLA-A | 2.330 2 |
| B2M | 2.265 1 |
| HLA-B | 2.144 3 |
| ANXA1 | 1.924 3 |
| Down-regulated gene | |
| RPL13A | –1.422 4 |
| RPS20 | –1.634 9 |
| HSP90AA1 | –1.748 3 |
| TGFBI | –1.788 3 |
| ARL4C | –1.921 0 |
| HSPA1A | –2.285 1 |
| FC refers to fold change, log2 FC can represent the relative change trend of the difference gene, negative numbers represent downward regulation, positive numbers represent upward regulation. | |
通过上调基因/下调基因的基因-通路网络图(图 5A–B)可知,上调基因富集到的通路关联最多的基因是HLA家族基因(如HLA-A和HLA-B) 和细胞增殖、转移相关基因——B2M基因,关联的通路为MHC I类抗原处理和肽抗原的呈递,抗原处理和内源性抗原的呈递以及肽抗原的抗原处理和呈递等(图 5A)。其中,HLA家族基因是控制细胞间相互识别、调节免疫应答的一组紧密连锁基因群,其随着RB肿瘤的进展而上调表达,揭示了HLA家族基因在RB肿瘤进展过程中呈现了强烈的免疫应答过程。同时,有研究指出,B2M基因在T细胞杀伤肿瘤过程中发挥着关键作用,并且与肿瘤的增殖和转移密切相关[21]。然而目前基因B2M在视网膜母细胞瘤中的相关研究非常少,并无B2M抑制肿瘤细胞迀移、侵袭的报道,因此本研究推测这可能是RB肿瘤发展过程中的一个重要标志物。
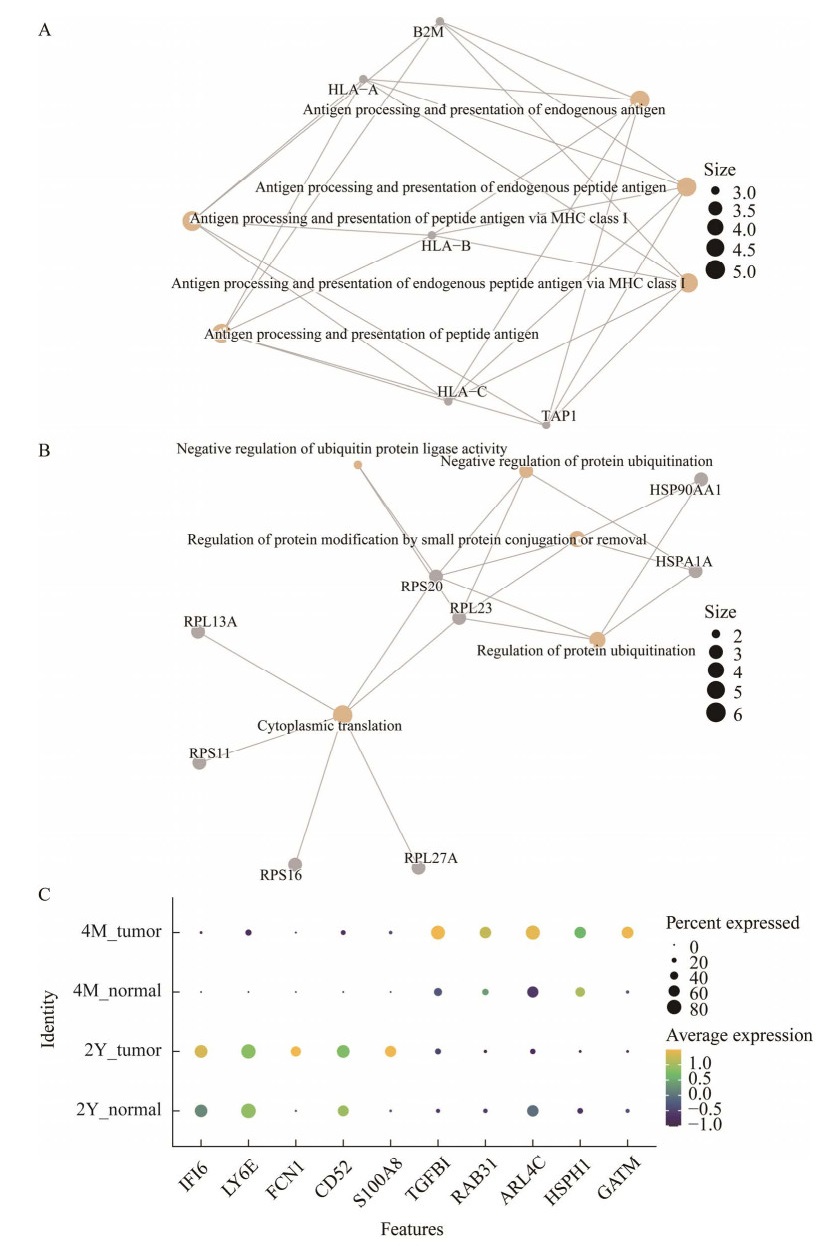
|
| 图 5 小胶质细胞亚群在不同发育年龄的RB肿瘤样本中富集分析 Fig. 5 Enrichment analysis for microglia of different RB developmental stage. (A) Centplot for up-regulated gene. (B) Centplot for down-regulated gene. (C) Dotplot for the top 5 differential genes. |
| |
下调基因富集到的通路关联最多的基因是RPS20和RPL23。其中RPL23编码的核糖体蛋白L23是已报道过的肿瘤基因治疗的靶点,是肿瘤患者潜在的临床预后因子[22]。值得注意的是,有研究指出,RPL23能够间接影响c-MYC的致癌活性,在正常细胞中,c-MYC以非激活的形式存在,当c-MYC异常失控时,会诱导包括RPL23在内的大量核糖体蛋白转录,通过加快蛋白质合成速度以满足肿瘤细胞所需要的高增殖活性[23]。与此同时,从Vo等[24]在小脑髓母细胞瘤的研究以及Wanzel等[25]的工作中可以发现,RPL23通过核磷蛋白介导作用直接抑制Myc相关锌脂蛋白活性,间接影响了c-MYC的表达。而RPL23是否具有反向调控的能力也存在多方争论,其临床数据并不充分,Zhou等[26]发现核糖体蛋白S14能负反馈调控c-Myc,与此同时RPL23也拥有这样的活性,然而Liao等[27]使用免疫沉淀却未能观察到RPL23能够结合Myc-TRBP2区域从而进一步诱导c-Myc凋亡的现象,认为c-Myc确实诱导了RPL23等核糖体蛋白的高表达,但仅仅只有核糖体蛋白L5和L11才具有特定的负反馈活性。在未来,探索RPL23对RB肿瘤防治也是一个值得深入的研究方向。
综合考虑分别选出5个最具代表意义的上调基因和下调基因做气泡图(图 5C)。已知基因IFI6和TGFBI[28]都是已报道过的与肿瘤进展或是靶向治疗有关的重要标志物,但它在不同发育年龄的RB肿瘤中有着不同的表达情况,因此这些基因在RB肿瘤进展过程中同样发挥着不可忽视的作用。
3 讨论本研究发现,随着RB肿瘤的进展,3种细胞亚群在RB肿瘤和癌旁组织中的比例发生了显著改变,包括视锥细胞G1期细胞簇、视锥细胞G2期细胞簇以及小胶质细胞簇。其中小胶质细胞中一些关键基因(如RPL23、HLA家族基因、IFI6和TGFBI等) 的异常表达,与肿瘤细胞的增殖、能量代谢以及免疫应答密切相关,可能是预测RB进展的重要标志基因。
为了证实我们的研究结果,我们下载另一套RB肿瘤单细胞转录组测序数据(GSE68434) 进行了相关验证[29]。该数据集源于7例不同年龄的RB肿瘤患者,该数据集多数细胞团有表达视锥前体marker基因的特征(图 6A)。提取小胶质细胞亚群,按照患者年龄进行分组。其中,3Y+microglia表示患者年龄在3年或3年以上,定为高年龄组;而3Y-microglia代表患者年龄低于3年,定为低年龄组。通过对本研究中表 4的基因进行可视化的验证,发现本课题样本里在两年肿瘤患者小胶质细胞团中上调表达的基因,同样也在参考集的高年龄组中明显表达(图 6B),而下调基因在参考数据中也处于较低的平均表达量。这进一步证实我们研究的准确性,说明HLA家族基因、B2M基因的表达的确与RB肿瘤进展密切相关。
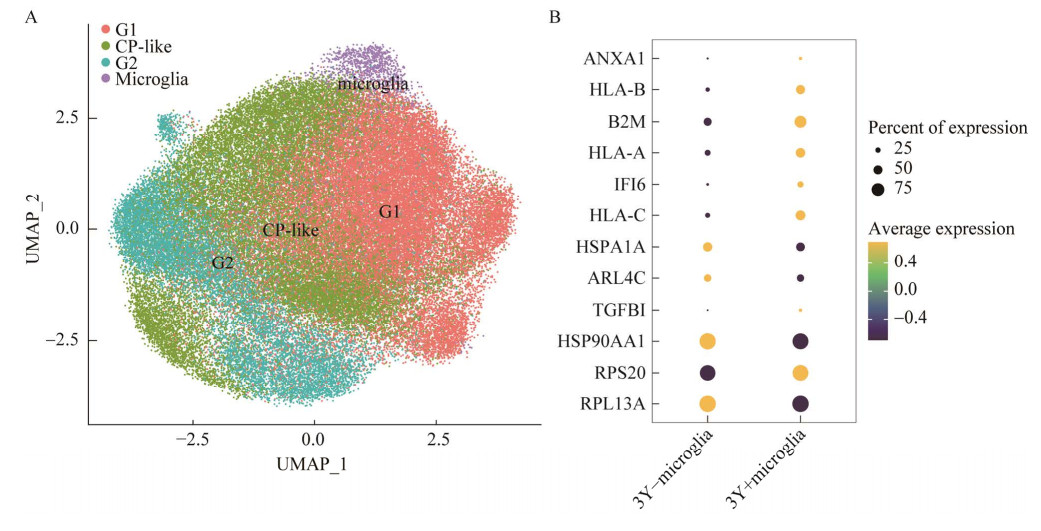
|
| 图 6 在验证数据集中对特定基因进行可视化 Fig. 6 Visualization of specific genes in reference datasets. (A) ADD CELL ANNOTATION FOR reference datasets. (B) DOTPLOT FOR specific genes of microglia from different age. |
| |
本研究的创新点在于,基于原发RB肿瘤个体样本,选择来自4个不同阶段、不同部位的样本数据,解析RB肿瘤在同时段的不同部位、或同部位的不同时段上细胞组成及含量上的差异,从而更清晰直观地说明肿瘤异质性现象及肿瘤动态发展的特征。其次,通过锁定3组RB肿瘤进展密切相关的细胞组分做深入性的差异分析,识别与之相关的差异基因及相关生物学过程,验证了B2M及HLA家族基因(HLA-A、HLA-B和HLA-C等) 与RB肿瘤进展密切相关,可以为后续研究RB的关键基因和关键通路提供新的方向,也为将来RB肿瘤的治疗以及发病进展机制的理论研究提供了一些参考思路。
| [1] |
Fabian ID, Onadim Z, Karaa E, et al. The management of retinoblastoma Oncogene, 2018, 37(12): 1551-1560.
|
| [2] |
Ortiz MV, Dunkel IJ. Retinoblastoma. J Child Neurol, 2016, 31(2): 227-236. DOI:10.1177/0883073815587943
|
| [3] |
Linn MA. Intraocular retinoblastoma: the case for a new group classification. Ophthalmol Clin North Am, 2005, 18(1): 41-53, viii. DOI:10.1016/j.ohc.2004.11.003
|
| [4] |
Xu XL, Singh HP, Wang L, et al. Rb suppresses human cone-precursor-derived retinoblastoma tumours. Nature, 2014, 514(7522): 385-388. DOI:10.1038/nature13813
|
| [5] |
McEvoy JD, Dyer MA. Genetic and epigenetic discoveries in human retinoblastoma. Crit Rev Oncog, 2015, 20(3/4): 217-225.
|
| [6] |
Giacinti C, Giordano A. RB and cell cycle progression. Oncogene, 2006, 25(38): 5220-5227. DOI:10.1038/sj.onc.1209615
|
| [7] |
Prasetyanti PR, Medema JP. Intra-tumor heterogeneity from a cancer stem cell perspective. Mol Cancer, 2017, 16(1): 41. DOI:10.1186/s12943-017-0600-4
|
| [8] |
Papalexi E, Satija R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nat Rev Immunol, 2018, 18(1): 35-45. DOI:10.1038/nri.2017.76
|
| [9] |
Collin J, Queen R, Zerti D, et al. Dissecting the transcriptional and chromatin accessibility heterogeneity of proliferating cone precursors in human retinoblastoma tumors by single cell sequencing-opening pathways to new therapeutic strategies. Invest Ophthalmol Vis Sci, 2021, 62(6): 18. DOI:10.1167/iovs.62.6.18
|
| [10] |
Hao YH, Hao S, Andersen-Nissen E, et al. Integrated analysis of multimodal single-cell data. Cell, 2021, 184(13): 3573-3587.e29. DOI:10.1016/j.cell.2021.04.048
|
| [11] |
Hafemeister C, Satija R. Normalization and variance stabilization of single-cell RNA-seq data using regularized negative binomial regression. Genome Biol, 2019, 20(1): 296-310. DOI:10.1186/s13059-019-1874-1
|
| [12] |
Korsunsky I, Millard N, Fan J, et al. Fast, sensitive and accurate integration of single-cell data with harmony. Nat Methods, 2019, 16(12): 1289-1296. DOI:10.1038/s41592-019-0619-0
|
| [13] |
Aran D, Looney AP, Liu L, et al. Reference-based analysis of lung single-cell sequencing reveals a transitional profibrotic macrophage. Nat Immunol, 2019, 20(2): 163-172. DOI:10.1038/s41590-018-0276-y
|
| [14] |
Zhang XX, Lan YJ, Xu JY, et al. CellMarker: a manually curated resource of cell markers in human and mouse. Nucleic Acids Res, 2018, 47(D1): D721-D728.
|
| [15] |
Consortium GO. The gene ontology resource: enriching a gold mine. Nucleic Acids Res, 2021, 49(D1): D325-D334. DOI:10.1093/nar/gkaa1113
|
| [16] |
Kanehisa M, Goto S. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Res, 2000, 28(1): 27-30. DOI:10.1093/nar/28.1.27
|
| [17] |
Subramanian A, Tamayo P, Mootha VK, et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. PNAS, 2005, 102(43): 15545-15550. DOI:10.1073/pnas.0506580102
|
| [18] |
Liberzon A, Subramanian A, Pinchback R, et al. Molecular signatures database (MSigDB) 3.0. Bioinformatics, 2011, 27(12): 1739-1740. DOI:10.1093/bioinformatics/btr260
|
| [19] |
Dimova DK, Dyson NJ. The E2F transcriptional network: old acquaintances with new faces. Oncogene, 2005, 24(17): 2810-2826. DOI:10.1038/sj.onc.1208612
|
| [20] |
Schulze A, Oshi M, Endo I, et al. MYC targets scores are associated with cancer aggressiveness and poor survival in ER-positive primary and metastatic breast cancer. Int J Mol Sci, 2020, 21(21): 8127-8139. DOI:10.3390/ijms21218127
|
| [21] |
Jongvilaikasem S, Sampao S, Kanjanapradit K, et al. Serum β-2 microglobulin levels are associated with distant metastasis in patients with breast cancer. Mol Clin Oncol, 2021, 14(6): 118-125. DOI:10.3892/mco.2021.2280
|
| [22] |
Wu LY, Li X, Xu F, et al. Over-expression of RPL23 in myelodysplastic syndromes is associated with apoptosis resistance of CD34+cells and predicts poor prognosis and distinct response to CHG chemotherapy or decitabine. Ann Hematol, 2012, 91(10): 1547-1554. DOI:10.1007/s00277-012-1486-2
|
| [23] |
Van Riggelen J, Yetil A, Felsher DW. MYC as a regulator of ribosome biogenesis and protein synthesis. Nat Rev Cancer, 2010, 10(4): 301-309. DOI:10.1038/nrc2819
|
| [24] |
Vo BT, Wolf E, Kawauchi D, et al. The interaction of myc with Miz1 defines medulloblastoma subgroup identity. Cancer Cell, 2016, 29(1): 5-16. DOI:10.1016/j.ccell.2015.12.003
|
| [25] |
Wanzel M, Russ AC, Kleine-Kohlbrecher D, et al. A ribosomal protein L23-nucleophosmin circuit coordinates Mizl function with cell growth. Nat Cell Biol, 2008, 10(9): 1051-1061. DOI:10.1038/ncb1764
|
| [26] |
Zhou X, Hao Q, Liao JM, et al. Ribosomal protein S14 negatively regulates c-Myc activity. J Biol Chem, 2013, 288(30): 21793-21801. DOI:10.1074/jbc.M112.445122
|
| [27] |
Liao JM, Zhou X, Gatignol A, et al. Ribosomal proteins L5 and L11 co-operatively inactivate c-Myc via RNA-induced silencing complex. Oncogene, 2014, 33(41): 4916-4923. DOI:10.1038/onc.2013.430
|
| [28] |
Zhao HY, Li ZF, Gao Y, et al. Single-cell RNA-sequencing portraying functional diversity and clinical implications of IFI6 in ovarian cancer. Front Cell Dev Biol, 2021, 9: 677697. DOI:10.3389/fcell.2021.677697
|
| [29] |
Wu C, Yang JQ, Xiao W, et al. Single-cell characterization of malignant phenotypes and microenvironment alteration in retinoblastoma. Cell Death Dis, 2022, 13(5): 438-449. DOI:10.1038/s41419-022-04904-8
|
 2022, Vol. 38
2022, Vol. 38