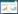中国科学院微生物研究所、中国微生物学会主办
文章信息
- 翁涵, 刘霞, 陶思齐, 梁英梅
- WENG Han, LIU Xia, TAO Siqi, LIANG Yingmei
- 山田胶锈菌和亚洲胶锈菌吸器的比较转录组分析
- Comparative transcriptomic analysis of the haustoria of Gymnosporangium yamadae and G. asiaticum
- 生物工程学报, 2022, 38(10): 3825-3843
- Chinese Journal of Biotechnology, 2022, 38(10): 3825-3843
- 10.13345/j.cjb.220379
-
文章历史
- Received: May 10, 2022
- Accepted: August 4, 2022
2. 北京林业大学 博物馆, 北京 100083
2. Museum of Beijing Forestry University, Beijing 100083, China
苹果和梨产业是中国水果的支柱[1-2],但其产量和品质会受到由山田胶锈菌(Gymnosporangium yamadae Miyabe ex G. Yamada) 和亚洲胶锈菌(Gymnosporangium asiaticum Miyabe ex G. Yamada) 所致锈病的影响。这两种胶锈菌不仅在亚洲地区广泛分布[3],还被欧洲和地中海植物保护组织(European and Mediterranean Plant Protection Organization, EPPO) 列入防疫名单[4]。虽然山田胶锈菌和亚洲胶锈菌的冬孢子阶段能寄生于同一株圆柏上,但冬孢子萌发后产生的担孢子却只能分别侵染各自的转主寄主,不能发生交叉侵染[5],即山田胶锈菌寄生于苹果属植物,亚洲胶锈菌寄生于皱皮木瓜、榅桲属和梨属植物[6],并分别在各自寄主的叶片上产生性孢子,再发育形成锈孢子去侵染圆柏,从而完成整个生活史循环[7],这可能暗示两种胶锈菌与寄主形成了某种特殊的专化性选择机制。然而,目前仍缺乏对这两种胶锈菌致病机理及其寄主专化性选择机制的深入研究。
作为专性寄生病原体,胶锈菌的担孢子经气流传播到达寄主植物表面,在适宜条件下萌发产生芽管,芽管生长延伸通过气孔进入寄主植物,在植物表皮细胞下形成胞间菌丝,进一步形成一种称为“吸器”的复杂细胞结构[8],使植物细胞壁和细胞膜凹陷从而从活细胞中吸收营养[9],并通过吸器分泌大量效应蛋白以调控寄主细胞各项生命活动,从而促进锈菌定殖[10]。吸器作为病原菌直接与寄主接触的结构,对其进行研究能有效破译病原菌的致病机制。早期对吸器和效应子进行分离研究较为困难[11],而伴刀豆球蛋白A能吸附锈菌吸器的发现使分离提取锈菌吸器成为了可能[12]。随后,蚕豆单胞锈菌[Uromyces viciae-fabae (Pers.) Schroet.][13]、亚麻栅锈菌[Melampsora lini (Ehrenb.) Lév.][14]、条锈菌(Puccinia striiformis Westend.)[15]、大豆层锈菌(Phakopsora pachyrhizi Syd. & P. Syd.) 和菜豆单胞锈菌(Uromyces appendiculatus F. Strauss)[16]等多种锈菌的吸器被成功分离,并通过转录组测序研究了吸器在锈菌早期侵染阶段的生物学功能。通过分析小麦条锈菌吸器的转录组数据发现,吸器吸收宿主营养物质,为生物合成途径产生大量能量并最终产生孢子[17]。与萌发的担孢子相比,编码分泌蛋白的基因大部分在吸器中上调表达,说明锈菌在吸器形成时期与寄主植物的互作更为强烈,同时暗示了吸器产生的分泌蛋白是解析锈菌与寄主协同进化机制的关键因子[18]。从蚕豆单胞锈菌的吸器和未产生吸器的侵染结构中分别鉴定得到62个和42个分泌蛋白,在吸器形成时期,许多与发病机制相关的分泌蛋白被上调表达并靶向了寄主植物细胞[19-20]。分泌蛋白中属于蛋白酶的类别往往与致病机制相关,而除了协助锈菌侵染寄主植物外,吸器的部分分泌蛋白能够协助锈菌利用寄主植物体内的营养物质,例如专性寄生菌依赖吸器和胞间菌丝吸收氨基酸,依赖吸器吸收糖分[21],除此之外还发现丝状真菌能够利用脂质[22]。然而分泌蛋白在锈菌的不同孢子阶段、不同侵染时期及侵染结构吸器的表达呈现阶段特异性[23],而吸器中表达的分泌蛋白可能是决定锈菌寄主专化性选择的重要因素[24]。迄今为止,尚未有木本植物锈菌的吸器转录组学相关研究的报道。
本课题组前期对山田胶锈菌和亚洲胶锈菌的冬孢子进行了比较转录组学分析[25],发现冬孢子阶段的基因表达模式及功能分布趋势相对一致,且分泌蛋白的数量及种类大致相同,这从一定程度上解释了这两种胶锈菌在冬孢子阶段能够寄生于同一个寄主植物的原因。而吸器作为山田胶锈菌和亚洲胶锈菌选择分别侵染苹果叶片和梨叶片时形成的关键侵染结构,可能是解析这两种胶锈菌实现对寄主专化性选择的关键因素。因此本研究基于前期建立的山田胶锈菌和亚洲胶锈菌吸器提取体系,成功获得了二者的吸器并进行了转录组测序,旨在解析山田胶锈菌和亚洲胶锈菌的致病机理及其寄主专化性选择分子机制,为锈病有效防控提供理论依据。
1 材料与方法 1.1 材料供试用3年生嘎啦苹果苗和水晶梨苗各20株(株高80 cm,直径1−1.5 cm)。
供试的山田胶锈菌(G. yamadae, GY) 冬孢子采自北京海淀公园,亚洲胶锈菌(G. asiaticum, GA) 冬孢子采自山东省枣庄市。
1.2 方法 1.2.1 接种及吸器样本收集将山田胶锈菌和亚洲胶锈菌的冬孢子角分别在无菌水中浸泡约12 h,用接种针将部分膨胀的冬孢子置于玻片上,在显微镜下观察,当萌发率高于65%时,参照刘霞等[5]的方法接种。接种后叶片产生大量花斑时分别采集苹果和梨的发病叶片,分别提取山田胶锈菌和亚洲胶锈菌的吸器[5],每个吸器样品各收集3个重复(分别为GY1, GY2, GY3和GA1, GA2, GA3)。
1.2.2 RNA的提取、质检、文库构建和测序使用Trizol法(Trizol试剂来自Invitrogen, Carlsbad) 分别提取各样本的RNA[26]。采用1%琼脂糖凝胶电泳、NanoPhotometer®分光光度计(IMPLEN)、Qubit® 2.0 Flurometer (Life Technologies) 和Agilent Bioanalyzer 2100系统(Agilent Technologies) 检测RNA的质量并评估完整性。确认RNA质量满足建库要求后,用NEBNext® Ultra™ RNA Library Prep Kit for Illumina® (NEB) 生成测序文库。文库片段用AMPure XP系统(Beckman Coulter) 纯化,由北京诺禾致源科技股份有限公司在Illumina HiSeq 2000平台进行测序。用Hisat2[27]获得并计算clean reads的Q20、Q30、GC含量和序列重复水平。
1.2.3 转录本从头组装、基因筛选及编码蛋白预测使用Trinity[28]默认参数分别进行山田胶锈菌和亚洲胶锈菌吸器转录组的组装。用寄主苹果[Malus domestica (Suckow) Borkh.]和梨(Pyrus bretschneideri Rehder) 的基因组及蛋白质组数据与该实验中的转录组数据进行比对(BLAST, E-value < 10−5) 以去除寄主序列的影响,用桃金娘锈菌(Austropuccinia psidii (G. Winter) Beenken)、栎柱锈(Cronartium quercuum (Berk.) Miyabe ex Shirai)、小麦杆锈菌(Puccinia graminis f. sp. tritici Pers.)、小麦条锈菌(Puccinia striiformis f. sp. tritici Westend.)、小麦叶锈菌(Puccinia triticina Erikss.)、落叶松-杨栅锈菌(Melampsora larici-populina Kleb.)、大豆层锈菌(Ph. Pachyrhizi)、咖啡驼孢锈菌(Hemileia vastatrix Berk. & Broome) 和双色蜡蘑(Laccaria bicolor (Maire) P. D. Orton) 9种真菌的基因组及蛋白质组数据以及Pucciniales的表达序列标签(expressed sequence tag,EST) 与转录组数据进行同源序列比对(BLAST, E-value < 10−5) 以确定目标物种的基因序列。上述数据均来自JGI (https://jgi.doe.gov/) 和NCBI (https://www.ncbi.nlm.nih.gov/)。
通过RSEM[29]默认参数估计每个重复的映射率和基因表达水平,以及计算每个样品的FPKM (fragments per kilobase million)。使用TransDecoder v5.5.0默认参数分别预测山田胶锈菌和亚洲胶锈菌unigenes的开放阅读框(open reading frames, ORFs) 和蛋白质编码区(coding sequence, CDS)。
1.2.4 实时荧光定量PCR验证基因表达为了验证转录组测序数据的准确性,在山田胶锈菌和亚洲胶锈菌吸器转录组数据中分别随机选取了5个基因进行实时荧光定量PCR (quantitative real-time polymerase chain reaction, qRT-PCR) 验证,并分别以EF1 (translation elongation factor EF-1 alpha/Tu, KOG id为KOG0052) 和GAPDH (glyceraldehyde 3-phosphate dehydrogenase, KOG id为KOG0657) 为内参基因。使用Primer3在线网站(https://primer3.ut.ee/) 的默认参数设计特异性引物,引物由睿博兴科(北京) 生物技术有限公司合成(表 1)。采用FastKing RT Kit (with gDNase) cDNA第一链合成试剂盒(天根生化科技(北京) 有限公司) 分别将山田胶锈菌和亚洲胶锈菌吸器的总RNA反转录合成cDNA,将其作为模板,利用2×HQ SYBR qPCR Mix (without ROX) 试剂盒(北京庄盟国际生物基因科技有限公司) 在CFX ConnectTM荧光定量PCR检测系统(Bio-Rad) 中进行qRT-PCR检测。每次检测包含3次生物与技术重复,在检测结果中利用内参基因校正目的基因的相对表达量。使用SPSS 16.0通过Kolmogorov-Smirnov检验(P > 0.05) 判断基因的qRT-PCR数据和转录组测序数据是否符合正态分布,如不符合则对数据进行转化,随后使用Pearson相关性分析计算P值和相关系数r。
| Primer name | Primer sequence (5′→3′) | Size (bp) |
| 1086_c0_g1_i7 | F: ACCAGAAACCCTAACCCGAG R: AGTGGAAGTGTTGGTGGTGA |
94 |
| 28430_c0_g4_i7 | F: TCCATCAACTACCGCCTCTC R: TGATTACGTCCCTGCCCTTT |
131 |
| 50_c1_g1_i2 | F: CCTCGGAAGCACAACCTTTC R: GACGTTGAGGAGAAGTTCGC |
158 |
| 22244_c0_g1_i5 | F: ATGTTCCCCGAGAGCTTTGA R: TCCCATTCACTGTCCTGCTT |
80 |
| 666_c0_g1_i9 | F: TGGCTTCTTGGTCTCGGTTT R: GTGGTCAGCGCATGTCTAAC |
112 |
| 517_c0_g1_i18 | F: CACCTTTCCCTCACGGTACT R: ACCTTTGATCTCCCGAGTGG |
133 |
| 25850_c0_g1_i1 | F: TCGTATTCCCCGTGTCCAAA R: CCGTAAGCAACAGCCTCATC |
93 |
| 2089_c1_g1_i1 | F: TACTTGACCGCTTCCACTGT R: TGTCCTCCCCAGTCATTGAC |
119 |
| 512_c0_g2_i4 | F: CATCCCGCTGAAGAATCGC R: TGGCTCCTCGATATTGGCTT |
85 |
| 293480_c0_g1_i1 | F: TGTTCGTGTTGGTCAAGCTG R: AGGGGAAGTATGCGTTTGGA |
85 |
| EF1 | F: AGGAGGCTCAATAGCGTCAA R: CAACATGCAATGGTTCAAGG |
97 |
| GAPDH | F: TCCTGCCTTTGAAATTTTGG R: TTGCTTTACGCTTGATGTGC |
101 |
使用BLAST程序(E-value < 10−5) 将蛋白编码基因与国家生物技术信息中心非冗余蛋白数据库(NCBI-Nr; http://www.ncbi.nlm.nih.gov)、基因本体论数据库(GO; http://geneontology.org/)、蛋白质序列数据库(Swiss-prot; http://www.expasy.ch/sprot)、蛋白质原核/真核同源数据库(COG/KOG; http://www.ncbi.nlm.nih.gov/COG)、蛋白质序列数据库(Uniprot-Swiss-prot; https://www.uniprot.org/) 进行同源性比对;使用KofamKOALA[30]默认设置将蛋白编码基因注释到京都基因与基因组百科全书数据库(KEGG; http://www.genome.jp/kegg) 以获取KO编号;使用dbCAN2程序[31]默认设置将蛋白编码基因注释到碳水化合物活性酶数据库(CAZymes; http://www.cazy.org/) 以判断是否为水解酶;使用HMMER程序[32]将蛋白编码基因注释到蛋白质家族数据库(Pfam; https://pfam.xfam.org/) 以判断是否存在结构域。
下载菜豆单胞锈菌[16]、小麦条锈菌[17]和大麦布氏白粉菌(Blumeria graminis f. sp. hordei M. Liu & Hambl.)[33]的吸器转录组数据,获取其基因对应的KO编号,随后与山田胶锈菌和亚洲胶锈菌吸器的转录组数据比较功能富集通路及富集比例的差异。
对山田胶锈菌和亚洲胶锈菌KEGG占比最高的4条通路中的基因进行表达量化,将各个基因的FPKM值依次从高到低排序,并绘制曲线图。FPKM > 1 000 (即lgFPKM > 3) 的基因被视为高表达基因,而FPKM < 10 (即lgFPKM < 1) 的基因被视为低表达基因。
1.2.6 候选效应子预测剔除FPKM低于所选阈值(50%) 的基因,根据效应子的一般特征[34]使用SignalP v3.0[35]预测编码蛋白是否存在信号肽,用TMHMM v2.0[36]排除具有跨膜结构域的蛋白,删除氨基酸数量(amino acids, aa)≥300和冗余的蛋白序列。
由于碳水化合物活性酶和蛋白酶可能与致病相关,脂酶可能与营养吸收利用相关,因此分别使用dbCAN2程序[31]、BLAST程序、HMMER程序[32](均默认E-value < 10−5) 将效应子比对到碳水化合物活性酶数据库、蛋白酶数据库(Merops; https://www.ebi.ac.uk/merops/) 和脂酶数据库(lipase engineering databases; http://www.led.uni-stuttgart.de/)。
1.2.7 序列聚类、同源群分析与物种进化使用OrthoFinder[37]对山田胶锈菌和亚洲胶锈菌的蛋白质组进行同源性分析,将包含两个物种蛋白的聚类视为同源群,并根据系统发育关系鉴定出山田胶锈菌和亚洲胶锈菌之间的一对一直系同源基因。使用BLAST程序以40%为阈值计算山田胶锈菌和亚洲胶锈菌的同源基因序列相似性,同时存在直系同源基因和旁系同源基因的基因被单独列出计算。使用KaKs Calculator[38]计算一对一直系同源基因的非同义替换率(nonsynonymous substitution rate, Ka) 与同义替换率(synonymoussubstitution rate, Ks) 的比值:Ka/Ks > 1的基因被认为是正选择,Ka/Ks=1的基因为中性选择,Ka/Ks < 1的基因为纯化选择[39]。
2 结果与分析 2.1 山田胶锈菌和亚洲胶锈菌吸器转录组的组装与编码蛋白预测分别对获得的山田胶锈菌和亚洲胶锈菌吸器各3个重复构建相应的cDNA文库(NCBI登录号为SRR17660644–SRR17660646和SRR17652436–SRR17652438)。山田胶锈菌和亚洲胶锈菌的clean reads分别约为8 200万和4 300万条。主成分分析发现GY1存在显著偏差,因此在后续的分析中将GY1剔除,转录组组装结果见表 2。为了确保约50%的基因被定义为表达,选择FPKM > 0.3作为阈值。每个样本的平均映射率(mapped rates) 为73.3%,表明转录组组装对于没有基因组的非模式生物是相对可靠的。
| Samples | Raw reads | Clean reads | Q20 (%) | Q30 (%) | Mapped reads | Expressed unigenes (FPKM > 0.3) |
| GY2 | 88 291 610 | 86 875 518 | 93.00 | 87.72 | 52 261 672 (60.16%) | 13 541 (63.83%) |
| GY3 | 78 319 004 | 77 042 716 | 96.00 | 91.20 | 60 354 304 (78.34%) | 16 046 (75.64%) |
| GA1 | 46 114 474 | 45 963 146 | 98.50 | 95.57 | 34 084 841 (74.16%) | 6 119 (47.01%) |
| GA2 | 43 108 202 | 42 965 560 | 98.50 | 95.54 | 32 924 754 (76.63%) | 7 038 (54.08%) |
| GA3 | 41 630 708 | 41 497 642 | 98.47 | 95.48 | 32 032 000 (77.19%) | 6 635 (50.98%) |
山田胶锈菌和亚洲胶锈菌分别获得553 420条和469 905条unigenes,N50分别为272和294,平均长度为309 bp和325 bp。此外,获得了1 055 746个山田胶锈菌的转录本(平均长度为410 bp),N50为469,以及607 956个亚洲胶锈菌的转录本(平均长度为357 bp),N50为355 (表 3)。吸器阶段的转录本和unigenes平均长度较短,N50较低,但两个物种间的差异不大。经序列比对筛选后分别获得21 213条和10 315条unigenes,最后分别预测到山田胶锈菌和亚洲胶锈菌的全长CDS序列13 677个(64.47%) 和6 273个(48.20%),分别对应12 210条和6 018条unigenes。
| Parameter | G. yamadae | G. asiaticum | |
| Transcripts | Number | 1 055 746 | 607 956 |
| Max length (bp) | 27 461 | 26 613 | |
| Average length (bp) | 410 | 357 | |
| N50 | 469 | 355 | |
| Total residues | 433 239 086 | 217 273 590 | |
| Unigenes | Number | 553 420 | 469 905 |
| Max length (bp) | 27 461 | 26 612 | |
| Average length (bp) | 309 | 325 | |
| N50 | 272 | 294 | |
| Total residues | 171 079 823 | 152 803 226 | |
| Full-length CDS | Number | 13 677 | 6 273 |
| Max length (bp) | 11 355 | 4 464 | |
| Average length (bp) | 263 | 188 | |
山田胶锈菌和亚洲胶锈菌各5个基因的转录组测序结果与qRT-PCR验证结果的相关性分析(图 1) 表明,其相关性均约等于0.9,即RNA-seq测序结果与qRT-PCR扩增结果存在显著的线性关系,表明转录组测序结果可靠(P < 0.05),可用于后续的生物信息学分析。

|
| 图 1 山田胶锈菌(A) 和亚洲胶锈菌(B) 基因的qRT-PCR验证结果 Fig. 1 qRT-PCR verification results of G. yamadae (A) and G. asiaticum (B) genes. |
| |
从各数据库的注释结果(表 4) 发现:在山田胶锈菌和亚洲胶锈菌的蛋白编码序列中,255条(2.09%) 和90条(1.50%) 序列均能在7个数据库中得到注释,11 738个(96.13%) 和5 826个(96.81%) 基因在至少1个数据库中被注释。
| No. of unigenes annotated to the database respectively | G. yamadae | G. asiaticum |
| Annotated in Nr | 11 758 (96.30%) | 5 817 (96.66%) |
| Annotated in GO | 9 533 (78.08%) | 4 914 (81.66%) |
| Annotated in KO | 10 197 (83.51%) | 5 470 (90.89%) |
| Annotated in UniProt | 8 662 (70.94%) | 4 625 (76.85%) |
| Annotated in CAZy | 329 (2.69%) | 123 (2.04%) |
| Annotated in KOG | 10 917 (89.41%) | 5 427 (90.18%) |
| Annotated in Pfam | 10 786 (88.34%) | 5 415 (89.98%) |
| Annotated in all databases | 208 (1.70%) | 88 (1.46%) |
| Annotated in at least one database | 11 805 (96.68%) | 5 864 (97.44%) |
| Total unigenes | 12 210 (100.00%) | 6 018 (100.00%) |
在7个数据库中选择了具有代表性的4个数据库注释结果以进一步分析。在Nr数据库中,二者注释最多的类群是柄锈菌亚门(Pucciniomycotina)、银耳目(Tremellales) 和座囊菌亚纲(Dothideomycetidae),将注释到柄锈菌亚门的序列进一步解析(图 2),发现山田胶锈菌注释比例最多的物种从高到低依次是禾柄锈菌(Puccinia graminis Pers.)、小麦叶锈菌和禾冠柄锈菌(Puccinia coronata Corda),而亚洲胶锈菌则依次是禾柄锈菌、落叶松-杨栅锈菌和紫萁混合菌(Mixia osmundae (Nishida) C.L. Kramer)。山田胶锈菌有约80%的序列都能注释到柄锈菌纲(Pucciniomycetes),而亚洲胶锈菌仅为60%,这种差异体现了二者进化关系上的不同。
|
| 图 2 山田胶锈菌和亚洲胶锈菌与Nr数据库匹配的物种分布 Fig. 2 Species distribution of G. yamadae and G. asiaticum matched with Nr database. |
| |
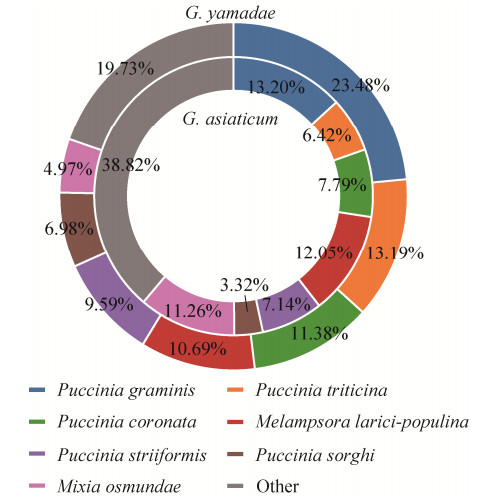
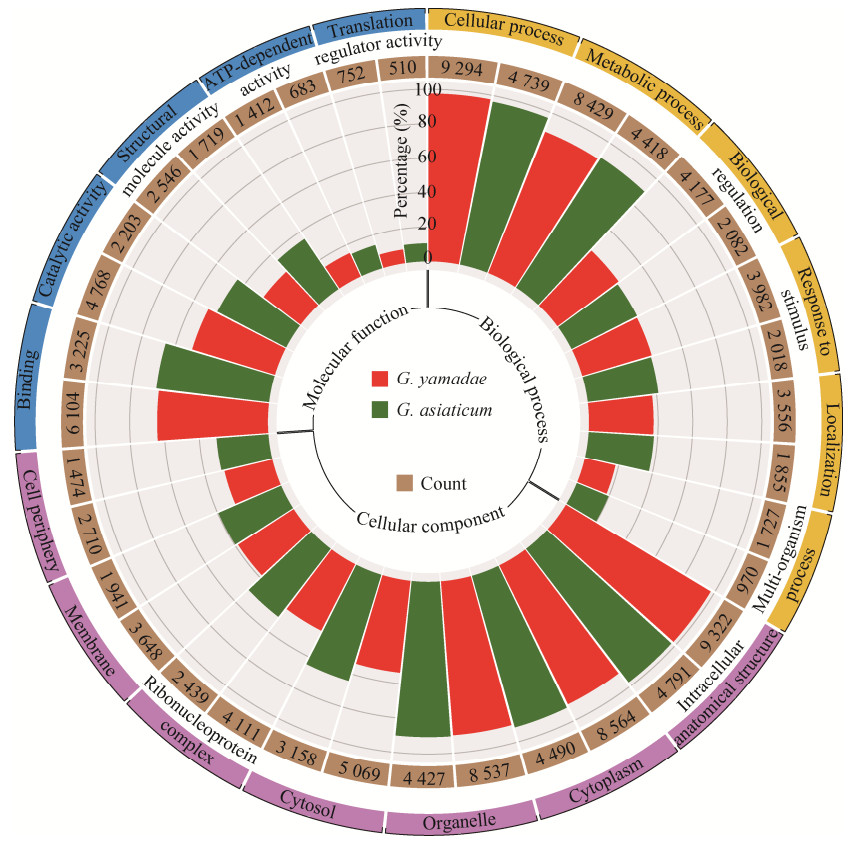
KOG富集分析(图 3) 表明,山田胶锈菌有19.2%的unigenes富集在翻译、核糖体结构和生物发生的类别,而亚洲胶锈菌为26.23%,说明亚洲胶锈菌比山田胶锈菌蛋白质生物合成更为活跃;其次,二者富集到的类别为翻译后修饰、蛋白质周转、伴侣,所占比例相差不大。在GO功能注释上(图 4),两者注释的类别及比例都没有表现出明显的差异。生物进程中,山田胶锈菌97.5%和88.4%的unigenes都能注释到细胞过程和代谢过程,亚洲胶锈菌则为96.4%和89.9%;细胞组分中则是细胞内解剖结构、细胞质、细胞器的富集程度最高;分子功能中,富集到连接、催化活性类别占比最多。KEGG通路中,注释到“核糖体” (ko03010) 的基因分别为2 354个(23.09%) 和1 672个(30.57%),注释到“内质网中的蛋白质加工” (ko04141) 的基因则分别为762个(7.47%) 和455个(8.32%),以及在山田胶锈菌中有661个(6.48%) 基因被注释到“剪接体” (ko03040)中,在亚洲胶锈菌中有386个(7.06%) 基因被注释到“氧化磷酸化” (ko00190)。整体来看,二者在二级通路中富集程度类似(图 5),但亚洲胶锈菌在遗传信息相关途径的富集要略大于山田胶锈菌。

|
| 图 3 山田胶锈菌(A) 和亚洲胶锈菌(B) 的吸器阶段分别与冬孢子阶段的KOG富集比较 Fig. 3 Comparison of KOG enrichment at the haustorial stage and teliospores stage of G. yamadae (A) and G. asiaticum (B), respectively. |
| |
|
| 图 4 山田胶锈菌和亚洲胶锈菌的GO富集比较 Fig. 4 Comparison results of GO database annotations of G. yamadae and G. asiaticum. |
| |
|
| 图 5 山田胶锈菌(GY)、亚洲胶锈菌(GA) 与小麦条锈菌(PST)、大豆层锈菌(UA) 和大麦布氏白粉菌(BGH) 的KEGG富集比较 Fig. 5 Comparison of KEGG enrichment in G. yamadae (GY), G. asiaticum (GA), P. striiformis f. sp. tritici (PST), U. appendiculatus (UA) and Blumeria graminis f. sp. hordei (BGH). |
| |
与冬孢子阶段相比,KOG注释中,吸器阶段与翻译相关的类别明显比例大幅增加;GO注释中,吸器阶段的生物进程和分子功能相差不大,但细胞组分相差较大,吸器阶段更多集中于细胞内结构和细胞质中;KEGG注释中,吸器阶段与冬孢子阶段富集“氨基酸的生物合成”的表现存在差异,更注重蛋白质的合成与加工,这也与一般锈菌吸器阶段的规律相符。
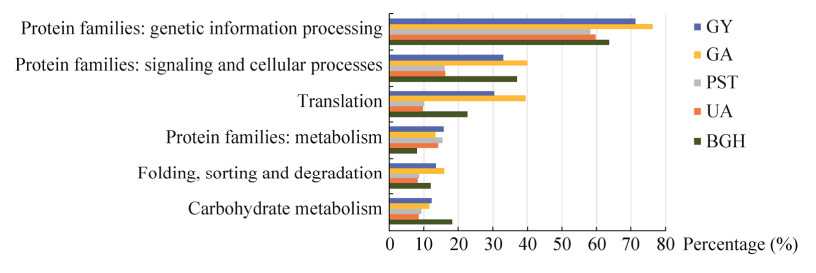
与其他活养寄生真菌的吸器阶段对比发现(图 5),山田胶锈菌、亚洲胶锈菌与大麦布氏白粉菌的富集情况较为相似,其在信号、细胞进程及翻译途径的富集比例远超小麦条锈菌和大豆层锈菌,为2−3倍,但其余通路例如蛋白质家族:基因信息进程、蛋白质家族:代谢等相差不大。而山田胶锈菌和亚洲胶锈菌富集占比前四的通路中的基因表达情况(图 6) 显示,虽然山田胶锈菌和亚洲胶锈菌各通路的高表达基因比例相似,但山田胶锈菌的低表达基因的比例远超亚洲胶锈菌,证明山田胶锈菌虽然在通路中的基因数量很多,但其中有大量基因表达量较低。

|
| 图 6 山田胶锈菌(蓝色) 和亚洲胶锈菌(黄色) KEGG通路所对应的基因表达分布 Fig. 6 Gene expression distribution of G. yamadae (blue) and G. asiaticum (yellow) in the corresponding KEGG pathways. |
| |
使用FPKM > 0.3、aa≥300、存在信号肽、不存在跨膜结构域4个指标最终得到山田胶锈菌343个(2.51%) 和亚洲胶锈菌175个(2.79%) 候选分泌蛋白。在山田胶锈菌的候选效应子中,发现了14个蛋白酶和10个脂酶,亚洲胶锈菌则为5个和3个。比较两种胶锈菌从初始unigenes到最终预测出效应蛋白过程中的序列数量变化(图 7),可以发现从筛选unigenes后开始,二者的数量一直呈现2︰1,但是相对比例基本没有改变。
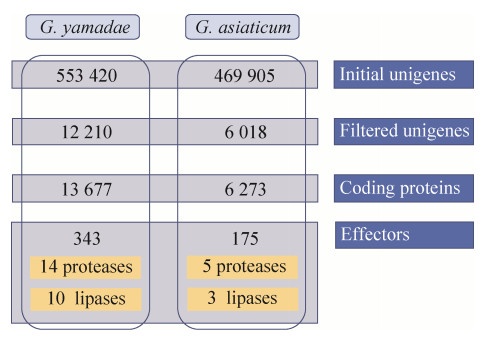
|
| 图 7 山田胶锈菌和亚洲胶锈菌unigenes中预测的蛋白质组和效应子 Fig. 7 Predicted proteomes and effectors from G. yamadae and G. asiaticum unigenes. |
| |
与冬孢子阶段的分泌蛋白相比,亚洲胶锈菌中的蛋白酶比例显著减少(冬孢子阶段为5.26%,吸器阶段为2.84%),但山田胶锈菌和亚洲胶锈菌的脂酶比例显著增加(冬孢子阶段分别为0%和0.55%,吸器阶段为2.92%和1.71%)。其中,山田胶锈菌和亚洲胶锈菌均含有脂酶家族abH01.06 (羧酸酯酶下的alpha酯酶) 和abH33 (抗原85) 的成员,而脂酶家族abH01 (羧酸酯酶) 在山田胶锈菌中分布最多,其次为abH34家族(类溶酶体保护蛋白) (表 5)。
| Species | Protein ID | Target family name | Description | E-value |
| GY | 1086_c0_g1_i7.p1 | abH01.05 | Bacillus esterases | 0.002 3 |
| GY | 177983_c0_g1_i4.p1 | abH01.06 | Alpha esterases | 1.20E-59 |
| GY | 177983_c0_g1_i10.p2 | abH01.06 | Alpha esterases | 9.30E-06 |
| GY | 136799_c0_g2_i1.p1 | abH34.01 | Lysosomal protective protein like | 6.60E-24 |
| GY | 22244_c0_g1_i5.p1 | abH34.02 | Serine carboxypeptidase Ⅱ like | 1.30E-104 |
| GY | 3056_c0_g1_i3.p1 | abH22.09 | Saccharomyces cerevisiae proteins | 0.000 14 |
| GY | 114837_c0_g1_i1.p1 | abH36 | Cutinases | 0.001 |
| GY | 28430_c0_g4_i7.p1 | abH03 | Candida rugosa lipase like | 4.70E-86 |
| GY | 145247_c0_g1_i1.p2 | abH33 | Antigen 85 | 0.007 8 |
| GY | 21970_c0_g1_i1.p1 | abH08.06 | Miscellaneous | 0.008 |
| GA | 374_c0_g1_i3.p2 | abH01.06 | Alpha esterases | 1.50E-76 |
| GA | 710465_c0_g1_i1.p2 | abH07.02 | Moraxella lipase 3 like | 0.003 6 |
| GA | 205323_c0_g2_i2.p1 | abH33 | Antigen 85 | 0.002 7 |
将去冗余后的山田胶锈菌的5 743条和亚洲胶锈菌的3 271条蛋白质序列通过OrthoMCL方法进行聚类,聚合了1 917个直系同源群,其中497个为单系群,包含1 491个蛋白。未聚类的2 019条蛋白序列中包括761对一对一直系同源基因和513对单拷贝直系同源基因,但属于同一家族的直系同源基因较少。最大的直系同源群为F型ATP酶家族,其次为多聚泛素家族和ATP依赖性RNA解旋酶,表明这是满足二者基本生命需求的共同成分;山田胶锈菌最大的单系群为线粒体载体蛋白和泛素C,亚洲胶锈菌为内质网ATP酶和泛素小亚基核糖体蛋白Rps27a,说明二者在蛋白转运、降解、修饰等方面存在各自的独特性。
在一对一的直系同源基因对中,山田胶锈菌和亚洲胶锈菌的蛋白质相似率具有双峰分布(图 8;谷值对应的比对率为82%,峰值对应的比对率为62%和95%),其他蛋白(即存在旁系同源物的蛋白) 则呈现单峰,峰值对应的比对率为97%。由于保守蛋白和其他蛋白出现峰值时的蛋白比对率十分接近,说明二者的进化速度相似;进化蛋白和保守蛋白的分界线所对应的比对率为82%,即序列相似性超过82%才能将该蛋白对定义为保守,说明山田胶锈菌和亚洲胶锈菌的基因序列差异很大。
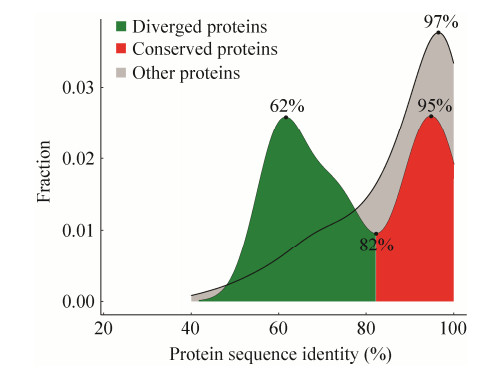
|
| 图 8 山田胶锈菌和亚洲胶锈菌的一对一直系同源物的序列相似性分布 Fig. 8 Distribution of protein sequence identity between one-to-one orthologs of G. yamadae and G. asiaticum. |
| |
同时,Ka/Ks结果(图 9) 显示,有80对基因每个同义位点的同义取代数≥0.75,15对基因的Ka/Ks > 1,这些基因对(共95对,占比12.48%) 被认为是正选择,其余666对基因的Ka/Ks < 1,被认为是纯化选择,表明有相当一部分基因近期正在快速进化。吸器阶段进化的基因比例也远高于冬孢子阶段(2.34%),表明该阶段环境选择压力较大。

|
| 图 9 山田胶锈菌和亚洲胶锈菌一对一直系同源基因的Ka和Ks值分布 Fig. 9 Scatter plots of Ka and Ks values of one-to-one orthologs of G. yamadae and G. asiaticum. |
| |
在亚洲胶锈菌效应子中发现5个蛋白处于正选择,其中一个为脂酶abH07.02 (类Moraxella脂酶3) 的家族成员,其余4个蛋白没有注释。在山田胶锈菌中暂未发现处于正选择的效应子。
3 讨论本研究利用高通量测序技术得到了山田胶锈菌和亚洲胶锈菌吸器的转录组数据,发现筛选后山田胶锈菌的unigenes约为亚洲胶锈菌的两倍。这种转录组unigenes数目因物种不同而存在显著差异可能是二者本身的生理特性或者进化机制所导致的。组装的序列平均较短表明该阶段可能更多地生成较小的分泌蛋白。
众所周知,对基因进行分类是很困难的,因为它们编码的蛋白质可能具有多种功能和/或与其生物学的各个方面有关。然而,对转录组数据进行注释通常无法识别大多数unigenes。在小麦条锈菌的吸器转录组中,约60%的基因与E-value < 10–25水平的已知基因不相似[17],但我们发现通过使用近缘种的数据进行注释的方法可以排除无法识别的序列,以保持较高的注释率。
大多数山田胶锈菌和亚洲胶锈菌序列都能被注释到柄锈菌亚门、银耳目和座囊菌亚纲。由于锈菌冬孢子产生担孢子的过程类似于子囊的形成过程,因此一些真菌学家认为锈菌是由子囊菌门进化而来的,也有学者指出锈菌来源于担子菌门的木耳目,因为木耳目和锈菌目的结构和担孢子的形成方式之间具有显著的相似性[40]。事实上,本研究的Nr注释也证明了锈菌目和银耳目、木耳目之间潜在的遗传关系。在注释到柄锈菌亚门的序列中,山田胶锈菌有约80%被注释到了柄锈菌纲,而亚洲胶锈菌为60%,这意味着山田胶锈菌和亚洲胶锈菌的亲缘关系相对存在较大差异。
从GO、KOG和KEGG的结果来看,山田胶锈菌和亚洲胶锈菌之间没有发现显著差异,表明不同锈菌的吸器期所涉及的生物学过程相对保守,而此时的致病表型也是相似的。在主要的富集通路中,虽然二者的高表达基因占比差异不大,但山田胶锈菌的低表达基因占比显著多于亚洲胶锈菌,这表明虽然山田胶锈菌的基因数量较多,但是有相当一部分基因处于低表达的状态,这可能表示亚洲胶锈菌发挥生物功能的基因模式更为精简。当寄主叶片上形成斑点时,吸器可能已经定殖,因此与寄主植物的“斗争”可能发生在担孢子侵染早期。一旦成功入侵宿主,两种锈菌进入相同的营养吸收阶段,它们与植物的免疫相互作用将大大降低。与冬孢子阶段对比发现,两者都富集于核糖体途径。先前研究发现,编码核糖体RNA和蛋白质的基因在大麦布氏白粉菌穿透寄主植物后总体上相对增加,与叶表面菌丝的增殖和分生孢子开始产生的时机一致[41]。本研究也发现大量相应的基因在山田胶锈菌和亚洲胶锈菌的吸器阶段表现出高表达。而且冬孢子阶段作为“非互作”的休眠阶段,更偏向氨基酸合成,但吸器阶段在“内质网中的蛋白质加工”相关类别上富集,说明此时锈菌处于与寄主互作阶段,可能需要大量的调控蛋白。与其他真菌的吸器阶段对比发现,山田胶锈菌、亚洲胶锈菌与大麦布氏白粉菌的富集情况较为相似,锈菌的部分途径具有与白粉菌类似的收缩现象[22],整体呈现出极不均衡的状态,且代谢相关途径相对富集较少,由此推测胶锈菌在能量利用上效率较低,这也与其吸器更为原始、侵染策略更简单、特化程度不高的推论相符[42-43]。
胶锈菌中的大多数效应子都无法注释到已知蛋白,也不包含任何与已知致病相关的结构域或基序,很难推测它们的功能。一些被认为是相对保守的效应因子,如RTP等[44]虽然存在于冬孢子阶段,但并没有在胶锈菌的吸器阶段中发现。蛋白酶中的很多家族都被证明与致病相关[45],例如蛋白酶是十字花科黑腐病菌(Xanthomonas campestris pv. Campestis) 的主要毒力因子[46],而向日葵锈菌(P. helianthi) 胞外分泌的蛋白酶通过破坏寄主细胞组织结构成分促进其侵染[47]。在冬孢子阶段,锈菌需要通过萌发而突破寄主表皮,可能蛋白酶在其中起到了降解细胞壁的作用,但不清楚为何吸器阶段蛋白酶比例减少。由于山田胶锈菌和亚洲胶锈菌寄生在活体植物上,它们不会产生太多直接攻击性的效应子[48-49],所以胶锈菌效应子的主要目的应该是调控寄主的生理进程,而不是对寄主的组织进行直接破坏,这可能解释了山田胶锈菌和亚洲胶锈菌吸器阶段效应子很多,但几乎很少发现功能已知的致病相关蛋白。
不同活体寄生菌的吸器都具有特殊的代谢功能[24]。在真菌中,糖是最常见的碳源,山田胶锈菌和亚洲胶锈菌的碳代谢途径非常简洁,缺失了许多相关基因,但仍然可以与植物保持长期共生状态,这与大麦布氏白粉菌类似,可能存在另一种策略以便在能量供应小于其他物种的情况下维持生存[22]。越来越多的研究发现,脂质在真菌侵染的过程中发挥了能量来源的作用,并观察到脂质从植物中转移到寄生病原菌中[50]。因此推测随着糖代谢途径的收缩,脂质可能是另一个潜在的能量来源,并且真菌可以直接吸收代谢的脂肪酸,这也许可以解释为什么吸器阶段需要大量脂酶的原因。虽然山田胶锈菌和亚洲胶锈菌可以合成脂肪酸,但是其脂肪酸延伸、脂肪酸降解途径都不完整,因此推测山田胶锈菌和亚洲胶锈菌可以同时使用碳水化合物和脂质,但是由于通路的收缩,利用效率非常低。
蛋白聚类表明山田胶锈菌和亚洲胶锈菌均含有F型ATP酶,也有大量的氧化磷酸化相关基因高表达,而F型ATP酶直接参与ATP水解,是线粒体供能的最后一步[51],可能意味着吸器的生理活动需要大量的能量。泛素家族在同源群和单系群中均高度富集,其蛋白质丰度随着DNA复制压力的增加而增加[52],表明这些真菌可能面临更高的进化压力。从单系群来看,山田胶锈菌富集线粒体载体蛋白,可能意味着其比亚洲胶锈菌更需要能量;有趣的是,亚洲胶锈菌富含的RPS27a,在小麦条锈菌研究中发现其在侵染后被诱导高表达,预示其可能是候选致病基因,推测RPS27a可能通过影响吸器的发育或功能参与小麦条锈菌的侵染和致病性,但遗憾的是其具体功能尚不清楚[53]。
可以发现,两个物种的同源蛋白相似性较低。在玉米黑粉菌(Ustilago maydis (D.C.) Corda.) 和玉米丝黑穗病菌(Sporisorium reilianum (J.G. Kuhn) Langdon & Full.) 的蛋白质比较中,进化基因和保守基因的分界线所对应的比对率仅为53%[54],且两种真菌不在同一属中,说明山田胶锈菌和亚洲胶锈菌的蛋白质序列存在大量变异。同时,Ka/Ks结果显示大部分位点存在同义突变,序列差异较大,进化距离较长。由于单拷贝直系同源基因是物种中最保守的[55],它们大多是结构基因,表达要求很少,因此在实际情况中,Ka/Ks比值远小于1,因为一般非同义替换会带来有害性状,只有少数情况会导致进化优势[56],但胶锈菌吸器阶段进化的基因远高于冬孢子阶段,这也说明这两个物种在吸器阶段的进化方向完全不同,且在该阶段受到的选择压力较大,这可能和频繁的寄主互作相关。Ka/Ks指标已广泛用于测定氨基酸水平选择压力[57],然而,氨基酸序列并不是基因在进化过程中经历选择压力的唯一特征,其他选择过程也可以作用于基因并影响其进化速度[58]。最近对RNA剪接的研究表明,在真核生物基因组中存在广泛的RNA选择压力[59],因此除了氨基酸水平上的选择压力差异,二者在其他尺度上的进化选择方向也有待研究。
在受到正选择的基因中,编码效应蛋白的基因仅有5个,并且全部集中于亚洲胶锈菌中。病原菌与寄主植物均在无休止的斗争中不断进化[18],因此认为直接与寄主互作的效应子可能更容易处于进化选择,由此看来吸器阶段虽然受到自然条件下相对较高的选择压力,但效应子可能并不是其用于应对的主要手段。其中一个效应子为脂酶abH07.02的家族成员,是潜在的毒力因子[60],但目前该家族在植物病原真菌致病性中的作用仍旧未知。
4 结论本研究主要对山田胶锈菌和亚洲胶锈菌吸器阶段进行了转录组测序并分析,对基因进行了功能注释,从中预测获得了候选效应子,并分析了两种锈菌的序列聚类和物种进化关系。
从基因功能注释结果来看,山田胶锈菌和亚洲胶锈菌侵染寄主植物后的基本策略相似,其作为定殖后保守的营养吸收阶段在形态和分子层面的差异不明显,尽管种间存在典型的寄主选择性,但吸器所涉及的生物学过程相对保守。
山田胶锈菌和亚洲胶锈菌的效应子与植物病原真菌中已知的大部分效应子没有相似性,且缺少已知功能的致病相关蛋白,因此其功能应该是调控寄主植物的某些生理进程,或者避开寄主的识别机制等,而不是直接对其进行攻击。山田胶锈菌和亚洲胶锈菌的脂酶比例显著增加,推测可能是山田胶锈菌和亚洲胶锈菌同时还利用植物的脂质作为碳源来提供能量。蛋白酶中的很多家族都被证明与致病相关,亚洲胶锈菌吸器阶段蛋白酶比例减少,证实了大部分效应子并非直接破坏寄主组织,这可能与锈菌是活体寄生菌相关,锈菌需要维持寄主的生命以持续吸收营养。
直系同源基因的Ka/Ks分布和蛋白质序列的相似性揭示了山田胶锈菌和亚洲胶锈菌在吸器阶段受到较大的进化选择压力而存在较大分化。尽管吸器阶段受到自然条件相对较高的选择压力,但效应子并不是其用于应对的主要手段。
致谢: 感谢西北农林科技大学康振生院士团队在吸器提取方面给予的支持,感谢西北农林科技大学黄丽丽教授、北京林业大学田呈明教授给予的建设性意见。
| [1] |
韩爱华, 贺雪娇, 张放. 2007—2016年全球苹果、梨与桃生产变动分析. 中国果业信息, 2018, 35(5): 21-35. Han AH, He XJ, Zhang F. Analysis of changes in global apple, pear and peach production from 2007 to 2016. China Fruit News, 2018, 35(5): 21-35 (in Chinese). DOI:10.3969/j.issn.1673-1514.2018.05.007 |
| [2] |
张放. 2019年我国进出口苹果与梨鲜果统计分析. 中国果业信息, 2020, 37(6): 21-31. Zhang F. Statistical analysis of China's import and export of fresh apples and pears in 2019. China Fruit News, 2020, 37(6): 21-31 (in Chinese). DOI:10.3969/j.issn.1673-1514.2020.06.005 |
| [3] |
Cummins GB, Hiratsuka Y. Illustrated genera of rust fungi// Cummins G B, Hiratsuka Y, eds. Illustrated genera of rust fungi. St. Paul, USA: American Phytopathological Society (APS Press), 2003: 240.
|
| [4] |
European and Mediterranean Plant Protection Organization. PM 8/2 (3) Coniferae. EPPO Bulletin, 2018, 48(3): 463-494. DOI:10.1111/epp.12503
|
| [5] |
刘霞, 陶思齐, 翁涵, 等. 山田胶锈菌和亚洲胶锈菌吸器提取体系建立. 菌物学报, 2019, 38(9): 1430-1439. Liu X, Tao SQ, Weng H, et al. Construction of haustorial isolation systems of Gymnosporangium yamadae and G. asiaticum. Mycosystema, 2019, 38(9): 1430-1439 (in Chinese). DOI:10.13346/j.mycosystema.190049 |
| [6] |
曹槟. 中国胶锈菌属的分类及系统发育研究[D]. 北京: 北京林业大学, 2018. Cao B. Taxonomy and phylogeny of Gymnosporangium in China[D]. Beijing: Beijing Forestry University, 2018 (in Chinese). |
| [7] |
Kern FD. A revised taxonomic account of Gymnosporangium. Pennsylvania, USA: Pennsylvania State University Press, 1991.
|
| [8] |
Szabo LJ, Bushnell WR. Hidden robbers: the role of fungal haustoria in parasitism of plants. PNAS, 2001, 98(14): 7654-7655. DOI:10.1073/pnas.151262398
|
| [9] |
Chong J, Harder DE, Rohringer R. Ontogeny of mono- and dikaryotic rust haustoria: cytochemical and ultrastructural studies. Phytopathology, 1981, 71(9): 975-983. DOI:10.1094/Phyto-71-975
|
| [10] |
Garnica DP, Nemri A, Upadhyaya NM, et al. The ins and outs of rust haustoria. PLoS Pathog, 2014, 10(9): e1004329. DOI:10.1371/journal.ppat.1004329
|
| [11] |
Varden FA, De La Concepcion JC, Maidment JH, et al. Taking the stage: effectors in the spotlight. Curr Opin Plant Biol, 2017, 38: 25-33. DOI:10.1016/j.pbi.2017.04.013
|
| [12] |
Hahn M, Mendgen K. Isolation by ConA binding of haustoria from different rust fungi and comparison of their surface qualities. Protoplasma, 1992, 170(3/4): 95-103.
|
| [13] |
Hahn M, Mendgen K. Characterization of in planta-induced rust genes isolated from a haustorium-specific cDNA library. Mol Plant Microbe Interact, 1997, 10(4): 427-437. DOI:10.1094/MPMI.1997.10.4.427
|
| [14] |
Catanzariti AM, Dodds PN, Lawrence GJ, et al. Haustorially expressed secreted proteins from flax rust are highly enriched for avirulence elicitors. Plant Cell, 2006, 18(1): 243-256.
|
| [15] |
汤春蕾. 条锈菌与小麦互作中效应蛋白及诱导寄主细胞坏死基因的鉴定与功能分析[D]. 杨凌: 西北农林科技大学, 2013. Tang CL. Characterization and functional analyses of effectors and host cell death inducing genes in wheat and Puccinia striiformis interactions[D]. Yangling: Northwest A & F University, 2013 (in Chinese). |
| [16] |
Link TI, Lang P, Scheffler BE, et al. The haustorial transcriptomes of Uromyces appendiculatus and Phakopsora pachyrhizi and their candidate effector families. Mol Plant Pathol, 2014, 15(4): 379-393. DOI:10.1111/mpp.12099
|
| [17] |
Garnica DP, Upadhyaya NM, Dodds PN, et al. Strategies for wheat stripe rust pathogenicity identified by transcriptome sequencing. PLoS One, 2013, 8(6): e67150. DOI:10.1371/journal.pone.0067150
|
| [18] |
Jones JDG, Dangl JL. The plant immune system. Nature, 2006, 444(7117): 323-329. DOI:10.1038/nature05286
|
| [19] |
Link TI, Voegele RT. Secreted proteins of Uromyces fabae: similarities and stage specificity. Mol Plant Pathol, 2008, 9(1): 59-66.
|
| [20] |
Jakupović M, Heintz M, Reichmann P, et al. Microarray analysis of expressed sequence tags from haustoria of the rust fungus Uromyces fabae. Fungal Genet Biol, 2006, 43(1): 8-19. DOI:10.1016/j.fgb.2005.09.001
|
| [21] |
Voegele RT, Mendgen KW. Nutrient uptake in rust fungi: how sweet is parasitic life. Euphytica, 2011, 179(1): 41-55. DOI:10.1007/s10681-011-0358-5
|
| [22] |
梁鹏. 白粉菌以糖类代谢途径收缩和效应分子多样化适应专性活体营养生活方式[D]. 海口: 海南大学, 2018. Liang P. Powdery mildews are characterized by contracted carbohydrate metabolism and diverse effectors to adapt to obligate biotrophic lifestyle[D]. Haikou: Hainan University, 2018 (in Chinese). |
| [23] |
Hacquard S, Delaruelle C, Legué V, et al. Laser capture microdissection of uredinia formed by Melampsora larici-populina revealed a transcriptional switch between biotrophy and sporulation. Mol Plant Microbe Interact, 2010, 23(10): 1275-1286. DOI:10.1094/MPMI-05-10-0111
|
| [24] |
范学锋, 张河山, 杨文香. 植物专性寄生菌吸器功能研究现状. 微生物学报, 2016, 56(8): 1222-1233. Fan XF, Zhang HS, Yang WX. Advances in haustoria function of plants obligate parasite-a review. Acta Microbiol Sin, 2016, 56(8): 1222-1233 (in Chinese). DOI:10.13343/j.cnki.wsxb.20150517 |
| [25] |
Tao SQ, Cao B, Tian CM, et al. Comparative transcriptome analysis and identification of candidate effectors in two related rust species (Gymnosporangium yamadae and Gymnosporangium asiaticum). BMC Genomics, 2017, 18(1): 651. DOI:10.1186/s12864-017-4059-x
|
| [26] |
杨楠, 徐正进, 周永力. 改良TRIZOL法提取真菌RNA的方法. 黑龙江科技信息, 2008(24): 20. Yang N, Xu ZJ, Zhou YL. Modified TRIZOL method for the extraction of fungal RNA. Heilongjiang Sci Technol Inf, 2008(24): 20 (in Chinese). DOI:10.3969/j.issn.1673-1328.2008.24.020 |
| [27] |
Kim D, Langmead B, Salzberg SL. HISAT: a fast spliced aligner with low memory requirements. Nat Methods, 2015, 12(4): 357-360. DOI:10.1038/nmeth.3317
|
| [28] |
Grabherr MG, Haas BJ, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol, 2011, 29(7): 644-652. DOI:10.1038/nbt.1883
|
| [29] |
Li B, Dewey CN. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics, 2011, 12: 323. DOI:10.1186/1471-2105-12-323
|
| [30] |
Aramaki T, Blanc-Mathieu R, Endo H, et al. KofamKOALA: KEGG ortholog assignment based on profile HMM and adaptive score threshold. Bioinformatics, 2020, 36(7): 2251-2252. DOI:10.1093/bioinformatics/btz859
|
| [31] |
Zhang H, Yohe T, Huang L, et al. dbCAN2: a meta server for automated carbohydrate-active enzyme annotation. Nucleic Acids Res, 2018, 46(W1): W95-W101. DOI:10.1093/nar/gky418
|
| [32] |
Mistry J, Finn RD, Eddy SR, et al. Challenges in homology search: HMMER3 and convergent evolution of coiled-coil regions. Nucleic Acids Res, 2013, 41(12): e121. DOI:10.1093/nar/gkt263
|
| [33] |
Lambertucci S, Orman KM, Das Gupta S, et al. Analysis of barley leaf epidermis and extrahaustorial proteomes during powdery mildew infection reveals that the PR5 thaumatin-like protein TLP5 is required for susceptibility towards Blumeria graminis f. sp. hordei. Front Plant Sci, 2019, 10: 1138. DOI:10.3389/fpls.2019.01138
|
| [34] |
Reid AJ, Jones JT. Bioinformatic analysis of expression data to identify effector candidates. Methods Mol Biol, 2014, 1127: 17-27.
|
| [35] |
Dyrløv Bendtsen J, Nielsen H, Von Heijne G, et al. Improved prediction of signal peptides: SignalP 3.0. J Mol Biol, 2004, 340(4): 783-795. DOI:10.1016/j.jmb.2004.05.028
|
| [36] |
Krogh A, Larsson B, Von Heijne G, et al. Predicting transmembrane protein topology with a hidden Markov model: application to complete genomes. J Mol Biol, 2001, 305(3): 567-580. DOI:10.1006/jmbi.2000.4315
|
| [37] |
Emms DM, Kelly S. OrthoFinder: phylogenetic orthology inference for comparative genomics. Genome Biol, 2019, 20(1): 238. DOI:10.1186/s13059-019-1832-y
|
| [38] |
Zhang Z, Li J, Zhao XQ, et al. KaKs_Calculator: calculating Ka and Ks through model selection and model averaging. Genom Proteom Bioinform, 2006, 4(4): 259-263. DOI:10.1016/S1672-0229(07)60007-2
|
| [39] |
Koonin EV, Rogozin IB. Getting positive about selection. Genome Biol, 2003, 4(8): 331. DOI:10.1186/gb-2003-4-8-331
|
| [40] |
庄剑云. 中国真菌志: 锈菌目. 北京: 科学出版社, 2012: 157-195. Zhuang JY. Chinese Fungi: Pucciniales. Beijing: Science Press, 2012: 157-195 (in Chinese). |
| [41] |
Both M, Eckert SE, Csukai M, et al. Transcript profiles of Blumeria graminis development during infection reveal a cluster of genes that are potential virulence determinants. Mol Plant Microbe Interact, 2005, 18(2): 125-133. DOI:10.1094/MPMI-18-0125
|
| [42] |
Gold RE, Mendgen K. Cytology of basidiospore germination, penetration, and early colonization of Phaseolus vulgaris by Uromyces appendiculatus var. appendiculatus. Can J Bot, 1984, 62(10): 1989-2002. DOI:10.1139/b84-271
|
| [43] |
周世国. 梨胶锈菌性孢子和锈孢子阶段吸器的超微结构研究. 真菌学报, 1992, 11(4): 289-293, 337-339. Zhou SG. Ultrastructural studies on the haustorium of Gymnosporangium haraeanum in the pycnio-and aeciostage. Mycosystema, 1992, 11(4): 289-293, 337-339 (in Chinese). |
| [44] |
Kemen E, Kemen AC, Rafiqi M, et al. Identification of a protein from rust fungi transferred from haustoria into infected plant cells. Mol Plant Microbe Interact, 2005, 18(11): 1130-1139. DOI:10.1094/MPMI-18-1130
|
| [45] |
董章勇, 王振中. 植物病原真菌细胞壁降解酶的研究进展. 湖北农业科学, 2012, 51(21): 4697-4700. Dong ZY, Wang ZZ. Research progress of fungal cell wall-degrading enzyme. Hubei Agric Sci, 2012, 51(21): 4697-4700 (in Chinese). DOI:10.3969/j.issn.0439-8114.2012.21.001 |
| [46] |
Dow JM, Clarke BR, Milligan DE, et al. Extracellular proteases from Xanthomonas campestris pv. campestris, the black rot pathogen. Appl Environ Microbiol, 1990, 56(10): 2994-2998. DOI:10.1128/aem.56.10.2994-2998.1990
|
| [47] |
李鑫淳. 向日葵锈菌金属蛋白酶基因的生物信息学分析及其原核表达[D]. 呼和浩特: 内蒙古农业大学, 2019. Li XC. Bioinformatics analysis and prokaryotic expression of metalloproteinase gene from Puccinia helianthi Schw[D]. Hohhot: Inner Mongolia Agricultural University, 2019 (in Chinese). |
| [48] |
Duplessis S, Cuomo CA, Lin YC, et al. Obligate biotrophy features unraveled by the genomic analysis of rust fungi. PNAS, 2011, 108(22): 9166-9171. DOI:10.1073/pnas.1019315108
|
| [49] |
Spanu PD, Abbott JC, Amselem J, et al. Genome expansion and gene loss in powdery mildew fungi reveal tradeoffs in extreme parasitism. Science, 2010, 330(6010): 1543-1546. DOI:10.1126/science.1194573
|
| [50] |
Luginbuehl LH, Menard GN, Kurup S, et al. Fatty acids in arbuscular mycorrhizal fungi are synthesized by the host plant. Science, 2017, 356(6343): 1175-1178. DOI:10.1126/science.aan0081
|
| [51] |
Cross RL, Müller V. The evolution of A-, F-, and V-type ATP synthases and ATPases: reversals in function and changes in the H+/ATP coupling ratio. FEBS Lett, 2004, 576(1/2): 1-4.
|
| [52] |
Gemayel R, Yang Y, Dzialo MC, et al. Variable repeats in the eukaryotic polyubiquitin gene ubi4 modulate proteostasis and stress survival. Nat Commun, 2017, 8: 397. DOI:10.1038/s41467-017-00533-4
|
| [53] |
庄华, 成玉林, 康振生. 小麦条锈菌泛肽-核糖体蛋白S27a基因的鉴定与表达分析. 西北农业学报, 2016, 25(12): 1775-1779. Zhuang H, Cheng YL, Kang ZS. Identification and expression analysis of an ubiquitin-ribosomal protein S27a gene from Puccinia striiformis f. sp. tritici. Acta Agric Boreali Occidentalis Sin, 2016, 25(12): 1775-1779 (in Chinese). DOI:10.7606/j.issn.1004-1389.2016.12.005 |
| [54] |
Depotter JRL, Zuo WL, Hansen MK, et al. Effectors with different gears: divergence of Ustilago maydis effector genes is associated with their temporal expression pattern during plant infection. J Fungi (Basel), 2020, 7(1): 16. DOI:10.3390/jof7010016
|
| [55] |
Kristensen DM, Wolf YI, Mushegian AR, et al. Computational methods for gene orthology inference. Brief Bioinform, 2011, 12(5): 379-391. DOI:10.1093/bib/bbr030
|
| [56] |
Nei M, Kumar S. Molecular Evolution and Phylogenetics. Oxford, UK: Oxford University Press, 2000.
|
| [57] |
Hurst LD. The Ka/Ks ratio: diagnosing the form of sequence evolution. Trends Genet, 2002, 18(9): 486.
|
| [58] |
Xing Y, Lee C. Can RNA selection pressure distort the measurement of Ka/Ks. Gene, 2006, 370: 1-5.
|
| [59] |
Pagani F, Baralle FE. Genomic variants in exons and introns: identifying the splicing spoilers. Nat Rev Genet, 2004, 5: 389-396.
|
| [60] |
Looi HK, Toh YF, Yew SM, et al. Genomic insight into pathogenicity of dematiaceous fungus Corynespora cassiicola. PeerJ, 2017, 5: e2841.
|
 2022, Vol. 38
2022, Vol. 38