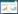1. 重庆医科大学 基础医学院 细胞生物学与遗传学教研室 分子医学与肿瘤研究中心, 重庆 400016;
2. 军事科学院军事医学研究院 生命组学研究所 中国医学科学院蛋白质组学与药物研发新技术创新单元 国家蛋白质科学中心 (北京) 北京蛋白质组研究中心 蛋白质组学国家重点实验室, 北京 102206
收稿日期:2022-03-11;接收日期:2022-05-10;网络出版时间:2022-05-13
基金项目:国家自然科学基金(32071431, 31700723);国家重点研发计划(2017YFC0906600, 2017YFA0505000)
作者简介:段昌柱 重庆医科大学教授、博士生导师。国家自然科学基金、重庆市科技攻关、重庆市自然科学基金获得者。国家自然科学基金通讯评议专家,教育部学位中心论文评审专家,重庆市科委项目通讯评议专家。长期致力于细胞增殖与调控研究,培养博士、硕士研究生30余人.
Quantitative proteomics reveal the potential biological functions of the deubiquitinating enzyme Ubp14 in Saccharomyces cerevisiae
1. Molecular Medicine and Cancer Research Center, Department of Cell Biology and Genetics, College of Basic Medical Sciences, Chongqing Medical University, Chongqing, 400016, China;
2. State Key Laboratory of Proteomics, Beijing Proteome Research Center, National Center for Protein Sciences (Beijing), Research Unit of Proteomics & Research and Development of New Drug, Chinese Academy of Medical Sciences, Beijing Institute of Lifeomics, Academy of Military Medical Sciences of Academy of Military Science, Beijing 102206, China
Received: March 11, 2022; Accepted: May 10, 2022; Published: May 13, 2022
Supported by: National Natural Science Foundation of China (32071431, 31700723); National Key Research and Development Program of China (2017YFC0906600, 2017YFA0505000)
泛素化(ubiquitination) 是真核生物中一种普遍存在的蛋白质翻译后修饰,可以在特定时空条件下精确传递不同的生物学信号,参与包括蛋白质降解、DNA修复、内吞、自噬等在内的多种生物学过程,启动特定的细胞信号转导[1]。泛素分子通过其C端的甘氨酸共价修饰到底物蛋白的赖氨酸残基上,N端的甲硫氨酸和7个赖氨酸均可作为继续链接的位点,在E1、E2、E3的级联酶促反应下共价修饰到底物蛋白的赖氨酸残基上形成多聚泛素化修饰,去泛素化酶(deubiquitin enzyme, DUB) 的存在使这一过程可逆化[2]。泛素链的生成和修剪组成了泛素化修饰的调控网络,在维持内环境稳态方面有重要的意义[3-4]。DUB功能异常可能会导致自身免疫系统疾病、传染病、癌症等疾病的发生[5-7],其具有成为药物靶点的巨大潜力。有研究表明,JAMM金属蛋白酶家族成员Psmd14为多发性骨髓瘤的潜在的治疗靶点,而USP家族成员Usp14有望成为非小细胞肺癌的潜在治疗靶点[8-11]。
DUB主要分为半胱氨酸蛋白酶和金属蛋白酶两大类。根据结构和作用机制,半胱氨酸蛋白酶又分为4类:泛素特异性蛋白酶(ubiquitin-specific protease, USP)、泛素羧基末端水解酶(ubiquitin C-terminal hydrolase, UCH)、卵巢肿瘤蛋白酶(otubain protease, OUT)、Machado-Joseph disease蛋白酶(Machado-Joseph disease protease, MJD);而属于金属蛋白酶类的DUB为MPN(+)/JAMM蛋白酶(JAMM)。结构上,前4种都含有半胱氨酸和组氨酸盒子等有催化活性的结构域。而JAMM中起着催化活性的为JAMM/MPN+结构域[12-13]。人类大约有100种DUB,而酵母中有21种DUB[4, 13]。其中,USP是迄今为止研究最多且成员最广的一类DUB,它在人体中大约有60种,而在酵母中有16种且用UBP表示[13]。
ubp14基因(ORF YBR058c) 位于酵母基因组2号染色体上,其在人体中的同源基因为usp5 (isot),负责切割细胞中大多数游离的泛素链,切割特性为每次切掉链近端的一个泛素。在哺乳动物细胞中,Usp5同样能够识别、切割未锚定泛素链并维持泛素单体稳态[14]。Usp5切割的速度会因为近端泛素C末端被酯化或移除G76而显著降低,即泛素链C端处于游离状态是Usp5识别底物的关键[15]。Usp5通过介导非锚定泛素链的水解来调节热诱导应激颗粒的组装和拆卸,下调Usp5会引起热应激颗粒的累积[16]。有研究表明,DNA双链断裂后,Usp5被招募到DNA损伤位点切割多聚泛素链,促进细胞通过同源重组途径高效修复DNA[17]。Usp5是T-型钙通道亚型Cav3.2的相互作用蛋白并调节其细胞表面表达,在慢性疼痛反应中Usp5上调,而阻止Usp5与Cav3.2结合能够缓解神经性疼痛[18]。Usp5可以稳定免疫抑制分子细胞程序性死亡-配体1 (PD-L1),使肿瘤细胞免受免疫系统破坏[19]。Usp5通过移除G1/S-特异性周期蛋白-D1 (cyclin D1)上的K48链修饰使其稳定,下调Usp5能够引起cyclin D1蛋白下降,诱导细胞周期的G1/S期阻滞,显著抑制胶质母细胞瘤的增殖和集落形成[20]。此外,Usp5可以通过其DUB活性稳定缺氧诱导因子-1 (HIF2α),促进乳腺癌细胞的增殖和转移[21]。Usp5在人体中调控多种重要的生物学过程,具有成为药靶的巨大潜力。
酵母中,去泛素化酶Ubp14负责识别并切割游离泛素链,对于细胞中的泛素分子稳态维持极为重要,其缺失会导致游离泛素链的累积[22]。细胞中的泛素分子稳态直接影响细胞中泛素化蛋白的稳态,进而对全蛋白质组产生影响,导致生物体稳态变化[23]。研究表明,Ubp14选择性切割游离的泛素链,Ubp14 (人类中的Usp5) 缺失导致酿酒酵母中K29和K48链的丰度增加了约30倍,而K63链的累积程度不足2倍[22]。Ubp14缺失会引起蛋白酶体功能受损,导致游离的泛素单体耗竭以及未切割的寡聚和多聚泛素链的累积。Ubp14和Ubp6在促进体内泛素底物降解方面的作用相似,二者的敲除皆可挽救ubp10敲除导致的细胞增殖缺陷[24-25]。过表达Ubp14能够增加细胞中泛素链的降解从而增加游离的泛素单体,使得细胞对应激易感,并且阻碍某些蛋白的蛋白酶体降解途径,影响细胞生长[26]。
细胞中泛素化蛋白的丰度较低,使得其鉴定较为困难[27-28]。由于DUB的移除效应,其对应的泛素化底物鉴定更加困难。因此,本研究利用酵母同源重组技术[29-30],构建了ubp14∆菌株,结合基于SILAC的定量蛋白质组学技术,在全蛋白质组水平分析了Ubp14缺失前后发生变化的蛋白[31]。本研究聚焦到DUB缺失之后引起变化的蛋白质和通路,从而对其功能和参与的生物学过程进行了分析。本研究共鉴定到109个差异蛋白,其中包含71个上调蛋白、38个下调蛋白。通过GO分析,Ubp14可能参与了包括氨基酸合成、氨基酸代谢、氧化还原、细胞对热反应等过程。对热应激通路进行温度敏感验证,高温使敲除菌株生长受损,与GO分析得到的结果一致。表明本研究筛选得到的ubp14∆菌株中109个潜在受Ubp14调控的蛋白以及其潜在的生物学功能可信。本研究可为深入探究去泛素化酶Ubp14的生物学功能,理解游离泛素链的稳态平衡与生物学过程调控的功能探究提供数据支持和理论依据,本实验方法也可为其他DUB的功能探究提供借鉴和思路。
1 材料与方法
1.1 材料
1.1.1 质粒和菌株 本研究用到的质粒和菌株见表 1。
表 1 本研究所用的质粒和菌株
Table 1 Strains and plasmids used in this study
| Strains & plasmids |
Description |
Types |
Background |
Sources |
| WT (JMP024) |
Endogenous ubiquitin genes are knocked out, ubiquitin expression in plasmid |
Strain |
SUB592 |
[23] |
| ubp14∆ |
ubp14 is knocked out |
Strain |
JMP024 |
This study |
| pFA6a-kanMX4 |
KanMX4 |
Plasmid |
| [32] |
1.1.2 培养基和培养条件 YPD培养基(100 mL):2 g蛋白胨,1 g酵母粉,2 g葡萄糖,1.5 g琼脂(固体培养基加入),100 mL双蒸水。SC base (100 mL):0.67 g酵母氮源基础(含硫酸铵),2 g葡萄糖,0.8 g琼脂糖(固体培养基加入),90 mL双蒸水。SC培养基(100 mL):90 mL SC base,10 mL SC-10 × aa (成分见表 2)。
表 2 SC-10×氨基酸混合物
Table 2 SC-10×aa mix
| Amino acids |
The final
concentration (mg/L) |
10×aa mix
(g/L) |
| Adenine sulfate |
20 |
0.2 |
| Uracil |
20 |
0.2 |
| L-arginine-HCl |
20 |
0.2 |
| L-tyrosine |
30 |
0.3 |
| L-leucine |
30 |
0.3 |
| L-isoleucine |
30 |
0.3 |
| L-phenylalanine |
50 |
0.5 |
| L-glutamic acid |
100 |
1.0 |
| L-aspartic acid |
100 |
1.0 |
| L-valine |
150 |
1.5 |
| L-threonine |
200 |
2.0 |
| L-serine |
400 |
4.0 |
| L-histidine-HCl |
20 |
0.2 |
| L-lysine-HCl |
30 |
0.3 |
重标培养基:90 mL SC base,10 mL SC-10×aa-K/R,300 μL K6 (1 g/100 mL),200 μL R10 (1 g/100 mL);轻标培养基:90 mL SC base,10 mL SC-10×aa-K/R,300 μL K0 (1 g/100 mL),200 μL R0 (1 g/100 mL)。
SD-ura培养基(100 mL):0.17 g酵母氮源基础(不含硫酸铵),0.5 g硫酸铵,2 g葡萄糖,0.8 g琼脂糖(固体培养基加入),10 mL SD- 10×aa mix-U (成分见表 3),90 mL双蒸水。遗传霉素(G418) 在YPD培养基中的使用浓度是50 μg/mL。WT和ubp14∆菌株在220 r/min、30 ℃振荡培养。
表 3 SD-10×氨基酸混合物(无尿嘧啶)
Table 3 SD-10×aa mix-U
| Amino acids |
The final concentration
(mg/L) |
10×aa mix
(g/L) |
| L-arginine-HCl |
20 |
0.2 |
| L-histidine-HCl |
20 |
0.2 |
| L-lysine-HCl |
30 |
0.3 |
1.1.3 主要试剂和仪器 dNTPs购自TaKaRa;遗传霉素购自AMERSCO公司;2×Taq PCR Master Mix购自BIOMIGA公司;DNA胶回收试剂盒购自天根生化科技有限公司;Ac-Trypsin蛋白酶由实验室自制[33-34]。
PCR仪、凝胶成像系统和电泳仪电源,BIO-RAD公司;涡旋混合仪,海门其林公司;生化培养箱,上海一恒;脱色摇床、电泳槽,北京市六一仪器厂;分光光度计,SHIMADZU BIOTECH公司;质谱、超高压色谱、冷冻离心机、摇床、超低温冰箱,Thermo公司。
1.2 实验方法
1.2.1 敲除菌株的构建 同源重组片段构建:在SGD数据库找到酿酒酵母中ubp14基因上下游序列,设计同源臂并合成引物ubp14-F和ubp14-R。用pFA6a-kanMX4质粒为模板进行PCR,扩增用于同源重组的替换片段卡那霉素抗性基因kan[35]。PCR引物见表 4。产物经1.2%琼脂糖凝胶电泳检验后,进行纯化。
表 4 菌株构建所用引物
Table 4 Primers used in this study
| Name |
Primer sequences (5′→3′) |
Size (bp) |
| ubp14-F |
AGGTAAATTCATCCAAAAAGAGAGCCCGAAGTCTTTCTCACATTCAGAAGACATGGAGGCCCAGAATAC |
69 |
| ubp14-R |
TATATATATATATGTGTGTGTGTGTGTGTGTGTGTGTGTAAACGTTCTTCAGTATAGCGACCAGCATTC |
69 |
| A1 |
GCTTGTTGCTGTGACTGGAGTCGGAATCGCAGAC |
34 |
| B1 |
GAGTCGGAATCGCAGAC |
17 |
| C1 |
ACATACCCACCACTAAGG |
18 |
| D1 |
GAAGAACCTCAGTGGCAA |
18 |
酵母感受态制备:配制过滤无菌冰冻的感受态细胞(freezing competent cell, FCC) 溶液(20 mL):10 mL 10%的无菌DMSO,10 mL 10%的无菌甘油。从平板上挑取WT菌株(JMP024) 单克隆接种到5 mL YPD培养基中,220 r/min、30℃摇床培养过夜。12–16 h后测定OD600,以起始OD600=0.3转接到50 mL 2×YPD培养基中,当OD600为1.5时,3 000×g离心5 min收菌。用1 mL无菌水洗细胞,重复一次,转移至无菌离心管中,4 ℃、3 000×g离心5 min弃去上清。用200 μL的FCC溶液重悬细胞沉淀。每管20 μL分装到1.5 mL离心管中,得到酵母感受态细胞。
敲除菌株的构建:配制FCC转化体系(360 mL):36 μL的1 mol/L的醋酸锂,260 mL的50%的PEG3350,50 μL的鲑精DNA以及14 μL的DNA产物。将感受态细胞在37 ℃水浴解冻30–60 s,3 000×g离心2 min,弃上清。将配好的转化体系与菌体混匀,42 ℃水浴1 h,3 000×g离心30 s,弃上清。1 mL YPD重悬细胞,并30 ℃温育2 h。3 000×g离心1 min,弃上清,取一半菌体涂板。
敲除菌株的验证:挑取抗性培养基上生长的单克隆进行PCR验证。PCR验证体系:10 μL 2 × Taq mix、0.2 μL primer-F、0.2 μL primer-R、少量菌体,最后加去离子水补足至20 μL。PCR用到的引物见表 4。PCR产物经1.2% 琼脂糖凝胶电泳检验。
1.2.2 生长曲线绘制 从平板上挑取少许菌落,接种到5 mL的SC培养基中,30 ℃、220 r/min培养过夜,第2天以起始OD600=0.26转接到50 mL SC培养基中,每隔2 h测定OD600,测定总时间为24 h。每个菌株设置3个生物学重复。
1.2.3 SILAC样品制备 用YPD平板活化WT、ubp14∆菌株,30 ℃生化培养箱培养过夜,每个菌株挑取2个单克隆分别转接到含4 mL YPD培养基的试管中,30 ℃摇床220 r/min培养过夜;第2天测定OD600,从中取出500 μL,加1 mL水洗2次,3 000×g离心5 min,弃上清,用1 mL水重悬菌体;根据OD600计算转接所需的菌体量,以起始OD600=0.001将WT菌株转接到20 mL轻标SC培养基中,将ubp14∆菌株转接到20 mL重标SC培养基中,每组2个生物学重复。在OD600为1.5时收集菌体,3 000 r/min离心5 min;每管用1 mL水洗两次,17 000×g离心,弃上清;用1 mL 0.1% NaN3重悬菌体,取少量测定OD600,根据测定的OD600值取对应的WT轻标和ubp14∆的重标各15 OD混合,两个生物学重复分别记为R1、R2。17 000×g、4 ℃离心5 min,加裂解缓冲液及与菌体体积相等的玻璃珠,涡旋1 min,冰上静置1 min,重复15次。17 000×g、4 ℃离心1 min,取上清转移至新管与5×SDS上样缓冲液混合。
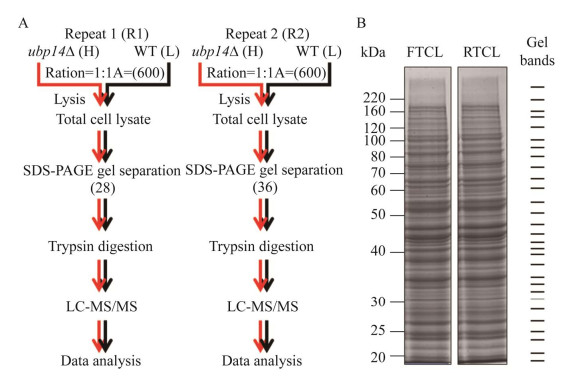
1.2.4 质谱样品制备 配置10% SDS-PAGE聚丙烯酰胺凝胶,将R1、R2样品经电泳分离,用考马斯亮蓝染液染色处理直至看到清晰的蛋白条带。根据蛋白条带的位置对其进行分区划线并切块,其中R1分为28个条带,R2分为36个条带。再将各条带切成体积为1 mm3胶粒转移到EP管中。向含有胶粒的EP管中加入300 μL脱色液,涡旋至溶液颜色不再变深,离心1 min,去掉上清。重复上述步骤至胶粒颜色变为无色。离心弃上清,加入300 μL乙腈(acetonitrile, ACN),先用手轻弹混匀将硬粒打散,再涡旋直至胶粒变为白色硬颗粒,将样品置于真空冷冻干燥仪中彻底干燥。加入酶解液,以酶液刚好浸没胶粒为准,冰上放置,胶粒溶胀后补加酶解液;冰上放置40 min,直至胶粒不再膨大;37 ℃封口放置,酶解12 h。
酶解结束后,17 000×g离心1 min,将酶液小心吸出,转移至新的EP管中。向胶中加入抽提液40 μL (5%甲酸(formic acid, FA),50% ACN),17 000×g离心1 min,静置4 min,吸出上清,转移到对应的EP管中,重复该步骤4–5次。加入ACN重复脱水,同样转移到对应的离心管中,混匀后检测肽段浓度,确定上样体积。最后,将离心管置于旋转蒸发仪中干燥以获得肽段样品。
1.2.5 质谱检测参数 用上样缓冲液溶解样品,17 000×g离心15 min。质谱检测使用超高压液相色谱(Nano Acquity, Waters) 分离梯度,上样体积为3 μL,上样流速为10 μL/min。洗脱下来的组分直接经过纳喷离子源(nanoelectrospray ionization, NSI) 进入高分辨率质谱仪(LTQ Orbitrap Velos, Thermo Fisher Scientific) 检测。流动相A:98%双蒸水+2%乙腈+0.1%甲酸;流动相B:100%乙腈+0.1%甲酸。洗脱条件:0–5 min,流动相B由3%增长至6%;5–50 min,流动相B由6%线性增长至40%;50–60 min,流动相B由40%增长至50%。流动相流速为0.3 μL/min。扫描范围300– 1 600 m/z,分辨率为30 000,自动增益控制(automatic gain control, AGC) 为1×106;数据采集模式为数据依赖模式(data-dependent acquisition, DDA);选择一级谱中丰度排名前20的离子进一步在离子阱中碎裂并检测;离子碎裂模式为高能碰撞诱导碎裂(high energy collision-induced dissociation, HCD),AGC为1×105,最大离子注射时间为100 ms。自动排除扫描正1价离子和未被识别的离子。动态排除的时间为30 s,期间不检测相同的已测序离子,提升测序覆盖度。
1.2.6 数据分析 使用MaxQuant (version 1.5.3.0) 软件搜库得到的原始定量蛋白质组学数据进行差异蛋白筛选。分别对两组的比值(ubp14∆/WT) 划分频数区间,在Excel中用NORM.DIST函数建立高斯拟合曲线。R1、R2的SD值均为0.13,以3倍SD值为卡值标准,在R1、R2中同时鉴定到且趋势一致的为差异蛋白。利用David在线网站(https://david.ncifcrf.gov/tools.jsp) 分别对上下调的差异蛋白进行GO分析,选取P < 0.05的部分差异通路进行展示。
1.2.7 点板实验 挑取WT与ubp14∆菌株单克隆,分别接种到5 mL YPD培养基中,30 ℃、220 r/min摇床培养过夜,第2天取1 mL WT与ubp14∆菌液,3 000×g离心1 min,用无菌水洗3次,再用1 mL无菌水重悬,测量OD后先稀释至1 OD/mL,再梯度稀释相应倍数,取2 μL菌体悬浊液点板。将板子分别置于30℃、37℃培养。一定时间后,拍照观察。
2 结果与分析
2.1 酵母ubp14敲除菌株构建与验证 ubp14基因位于酵母基因组2号染色体上,通过同源重组的方法用卡那霉素抗性基因kan代替ubp14。以pFA6a-kanMX4质粒为模板DNA,利用引物ubp14-F和ubp14-R扩增得到用于同源重组的kan片段。以WT菌株为模板无法扩增出任何片段,而以pFA6a-kanaMX4质粒为模板可以扩增出kan片段(图 1A)。将扩增得到的片段转化到酵母细胞中,选取部分单克隆菌落,利用表 4中两对引物A1、B1对上游片段进行验证,C1、D1对下游片段进行PCR验证,扩增出原ubp14上下游部分序列及kan部分片段(图 1B),从基因层面证明ubp14已成功敲除。对WT及ubp14∆菌株进行蛋白质组分析,Ubp14仅在WT菌株中被检测到,信号强度超过7×106,而敲除菌株中没有检测到Ubp14的蛋白信号(图 1C),该结果从蛋白质层面证明ubp14敲除成功。
2.2 酵母敲除ubp14后生长减缓 对WT和ubp14∆菌株进行生长曲线的测绘,从表型上探究Ubp14缺失是否会引起细胞生长的变化。分别将等起始量(OD600=0.26) 的WT和ubp14∆菌株接种到SC培养基中,每隔2 h测定吸光度OD600,测定总时间为24 h,每个菌株设置3个生物学重复。在指数生长期ubp14∆菌株的生长较WT菌株略为缓慢,WT菌株的平台期为4.3 OD,而ubp14∆菌株的平台期为3.6 OD,相较于野生型下降了0.7 OD (图 2A)。在指数生长期(6–8 h) 测定这两个菌株的倍增时间,ubp14∆菌株倍增时间为2.6 h,而WT菌株倍增时间为1.8 h,ubp14∆的倍增时间延长了0.8 h,出现了明显的生长阻滞(图 2B)。表明Ubp14参与菌株生长,其影响的关键蛋白和通路有待进一步研究。
2.3 定量蛋白质组学筛选ubp14∆菌株差异表达蛋白 为了得到高覆盖的蛋白质组学数据,按流程制备质谱样品(图 3A),用胰蛋白酶进行消化(图 3B)。用质谱检测标记效率,结果证明标记效率超过99% (数据未展示),符合SILAC定量比较的要求。利用基于SILAC标记技术的高覆盖定量蛋白质组学策略比较了Ubp14缺失前后酵母蛋白组中所有蛋白的含量变化。鉴定结果如表 5所示,R1共鉴定到3 205个蛋白,其中定量的蛋白有2 757个,定量变异系数(CV) 平均为13.2%;R2共鉴定到3 860个蛋白,其中定量的蛋白有3 570个,CV平均为14.7%。R1、R2中有定量信息的蛋白有2 642个重复(表 5),两次实验重复性较好,并且它们的相关性较好,两组定量的变异系数均小于15%,数值可信(图 4A、4C)。对R1、R2样品的比值(ubp14∆/WT) 取Log2进行了频数分布统计,并用样本标准差(SD) 等于0.13绘制了高斯拟合曲线(图 4B)。以3倍SD值为卡值标准,两次实验中均符合卡值区间且趋势一致的差异蛋白有109个,其中包括71个上调蛋白,38个下调蛋白(图 4C)。
表 5 质谱鉴定结果
Table 5 Identification by mass spectrometry
| Items |
R1 |
R2 |
| Protein groups |
3 205 |
3 806 |
| Peptides |
22 346 |
54 462 |
Average sequence
coverage (%) |
18.4 |
33.6 |
| MS/MS |
737 087 |
816 279 |
| PSM |
146 248 |
332 449 |
| MS/MS identified (%) |
19.8 |
40.7 |
| Quantified proteins |
2 757 |
3 570 |
| Average quantified CV (%) |
13.2 |
14.7 |
2.4 差异蛋白GO分析 为了探究Ubp14潜在的调控通路与分子功能,分别对这71个上调蛋白以及38个下调蛋白进行GO分析,包括分子功能分析、参与过程分析及亚细胞定位分析(图 5A、5B)。结果显示上调的差异蛋白主要参与氨基酸生物合成、代谢、氧化还原及细胞对热反应等过程,这些蛋白具有催化酶活性、转移酶活性、裂解酶活性和氧化还原活性,主要定位在细胞质与线粒体中。其中,敲除菌株中上调差异蛋白涉及细胞对热反应的分析与人体中ubp14的同源基因usp5耗竭引起热休克蛋白上调的报道一致[16]。下调的差异蛋白主要参与硫酸盐同化、代谢过程、氨基酸生物合成以及氧化还原过程,具有催化酶活性、还原型辅酶Ⅱ (NADPH) 活性、水解酶活性和氧化还原活性,这些蛋白主要定位在NADPH复合物中。研究表明,酿酒酵母中Met3、Met14、Met16在将硫酸盐同化还原为硫化物的过程中是必要的[36],虽然没有证据表明Ubp14是否影响这一通路,但在敲除菌株中观察到了Met3、Met14、Met16的下调。而甲硫氨酸调控通路是受到泛素化调控的,推测细胞内游离泛素链和泛素分子的含量变化,可能间接影响了甲硫氨酸合成通路相关蛋白的稳态,进而引发相关通路失调[37-38]。
2.5 ubp14∆菌株对高温敏感 GO分析发现,在ubp14∆菌株中有5个上调蛋白(Hsp26、Hsp78、Ssa4、Hsp31、Hsp42) 隶属于热休克蛋白家族成员,此类蛋白在细胞热休克时表达上调。其中,Hsp26具有恢复错误折叠蛋白的正常折叠结构的功能[39];Hsp78在酵母的线粒体基质中表达,对于线粒体热耐受性和热休克后线粒体错误折叠蛋白质的恢复至关重要[40];Hsp70可以促进错误折叠的蛋白质降解[41];除热刺激外,Hsp31在过氧化氢处理后也增加,缓解蛋白质聚集和错误折叠,并对许多的底物蛋白有效[42];Hsp42可以促进细胞中应激诱导的错误折叠蛋白隔离形成胞质聚集体,这些上调的热休克蛋白对错误折叠蛋白的修复均有作用[43]。此外,两次重复中定量到的19个热休克蛋白家族成员大部分均有上调现象(图 6A)。对ubp14∆菌株进行温度敏感实验验证GO分析的结果,在30 ℃和37 ℃条件下,用SD培养基和SC-ura培养基分别培养了WT和ubp14∆菌株并观察其生长表型。结果表明,两种菌株在30 ℃生长最佳,生长没有显著差异。37 ℃处理后ubp14∆菌株存在明显的生长阻滞现象(图 6B),ubp14敲除会导致菌株对高温敏感。
3 讨论与结论 酵母中,去泛素化酶Ubp14负责识别并切割游离泛素链,在泛素稳态维持中具有重要调控作用。其缺失导致游离泛素链的累积,进而影响整个泛素化调控网络。本研究通过同源重组用卡那霉素抗性基因kan替代酵母基因组中的ubp14,为探究Ubp14的功能奠定了基础。利用构建的ubp14∆菌株,首先观察了Ubp14缺失引发的效应,发现ubp14∆菌株存在生长阻滞现象,Ubp14可能参与调控细胞增殖。
本研究系统比较了WT和ubp14∆菌株中的差异蛋白,共筛选到109个差异蛋白。通过GO分析发现,上下调的差异蛋白均参与了氨基酸合成、氧化还原以及代谢等生物学过程。ubp14∆菌株受影响的代谢通路与氨基酸合成通路可以解释其表现出的生长阻滞现象。有研究表明,Usp5的耗竭加速热应激颗粒组装,还可以通过引起未锚定泛素链累积从而抑制热应激颗粒拆卸,这与在ubp14∆菌株中观察到的热休克蛋白上调的结果一致。不同温度下的点板实验证实ubp14∆菌株对高温(37 ℃) 敏感,Ubp14可能通过调控泛素链降解参与调控细胞内热应激过程。这条通路的成功验证说明了研究方法的可行性以及结果的可信性。本研究聚焦在Ubp14潜在生物学功能的发现,其他一些功能和精确的调控机制有待进一步探索。
本文仅探究了ubp14敲除在全蛋白质组水平引起的变化,包含Ubp14引起的直接效应以及Ubp14通过影响游离泛素稳态引起的间接效应。实验分析得到的部分生物学功能可能是由于生物副效应引起的,下一步的工作将对这些上下调的差异蛋白进行RNA-seq分析以及Ubp14相互作用组的筛选进而探索Ubp14直接调控的生物学过程。
综上所述,本研究筛选了Ubp14缺失前后产生差异的蛋白并对其进行了GO分析,揭示了Ubp14潜在的生物学功能,并选取了部分通路进行验证。这为Ubp14在人体中的同源去泛素化酶Usp5的底物和功能研究提供了新的方向,本文的研究方法也为其他去泛素化酶底物及功能研究提供了新思路。
 2022, Vol. 38
2022, Vol. 38