The albino mechanism of a new theanine-rich tea cultivar 'Fuhuang 2'
1. Key Laboratory of Tea Science at Universities in Fujian, College of Horticulture, Fujian Agriculture and Forestry University, Fuzhou 350002, Fujian, China;
2. Agriculture Genomics Institute at Shenzhen, Chinese Academy of Agricultural Sciences, Shenzhen 518120, Guangdong, China;
3. Tea Research Institute, Fujian Academy of Agricultural Sciences, Fuzhou 350013, Fujian, China
Received: January 27, 2022; Accepted: July 27, 2022; Published: August 11, 2022
Supported by: Chinese Oolong Tea Industry Innovation Center Special Project (J2015-75); Projects of the Science and Technology Plan Projects in Fujian Province (2021R1029001); Fujian Agriculture and Forestry University Tea Industry Chain Technology Innovation and Service System Construction Project (K1520005A01); Special Fund for Science and Technology Innovation of Fujian Zhang Tianfu Tea Development Foundation (FJZTF01)
植物叶片黄化是一种常见的现象,通常发生在绿色植物中,一般来说,叶片黄化是一种异常表型表达,在大多数植物中被认为是不良性状[1]。然而在茶树中,黄化茶树具有重要的产业应用价值,可产生高茶氨酸等功能成分代谢产物[2]。研究表明,黄化茶树品种中含有较低的儿茶素和较高的氨基酸含量是其良好风味产生的原因[3]。
茶树(Camellia sinensis (L.) O. Kuntze) 是一种具有经济价值的木本作物[4]。近年来,我国已育成了‘白叶1号’‘黄金芽’‘中黄1号’‘中黄2号’等黄化茶树新品种[5]。在黄化茶树突变体中发现,叶绿素生物合成或降解途径中相关基因的突变或阻断都可能导致茶树叶片色泽的变化,如叶绿素a镁脱氢酶(stay-green,SGR) 可能引发‘黄金水仙’叶绿素降解的加速,叶绿素含量降低,导致叶色变化[6]。此外,叶片色泽还受到其他色素和代谢途径的调控,如类黄酮和类胡萝卜素生物合成的综合作用决定‘黄金芽’的叶片颜色[7]。在‘中黄2号’的研究中,鉴定到差异表达基因被定位到与氨基酸代谢、叶绿素生物合成和类黄酮生物合成相关的途径,这些途径被认为是‘中黄2号’表型变化的原因[8]。
茶具有良好的风味品质,是儿茶素、生物碱和氨基酸等物质综合作用决定的,特别是游离氨基酸[9]。茶树芽叶中较为丰富的游离氨基酸有茶氨酸、谷氨酰胺、谷氨酸和精氨酸等[10]。通常茶氨酸在干茶中的含量为1%−2%,占总氨基酸的40%−70%[11-12]。而茶树芽叶中的茶氨酸含量受到茶树品种、营养和生态环境等的影响,选育高茶氨酸茶树资源难度较大[11]。在黄化茶树中发现较高茶氨酸对高茶氨酸茶树种质的发掘利用具有重要作用,为选育高茶氨酸茶树品种提供新方向。
‘福云6号’为国家级茶树优良品种(编号GS13003-1987),是我国的主要茶树栽培品种,其芽叶黄化突变体‘福黄2号’是本研究团队挖掘的高茶氨酸茶树新品系。本研究以‘福云6号’和‘福黄2号’为供试材料,利用多组学技术初步探究其黄化变异形成及高茶氨酸累积的机制,为高茶氨酸茶树黄化变异茶树资源选育的提供新的理论依据。
1 材料与方法
1.1 材料处理 供试材料为‘福云6号’和‘福黄2号’,取自福建省宁德市蕉城区八都镇茶树品种示范茶园。2021年3月,选取一部分表型完整的春梢第二片叶片,并利用2.5%戊二醛将其固定,用于后续超微结构的鉴定。此外,采集的其余春梢一芽二叶立即用液氮固定,并存放于–80 ℃冰箱中,用于后续实验,设置3次重复。
1.2 叶绿体超微结构观察 将‘福云6号’和‘福黄2号’成熟、无虫口二叶进行固定、漂洗、渗透、包埋和聚合后,利用超薄切片机(EM UC7, Leica) 切片并染色,并通过电镜(TEM Hitachi HT7700) 观察叶绿体结构。具体透射电镜样品的制备和实验步骤参考Hao等[13]的方法。
1.3 叶绿素和类胡萝卜素含量测定 使用UV-3200分光光度法(Mapada) 测定‘福云6号’和‘福黄2号’的叶绿素a、叶绿素b和总类胡萝卜素的含量,设置3次生物学重复。叶绿素和类胡萝卜素的具体提取以及操作步骤参考Wang等[14]的方法。利用超高效液相色谱质谱联用仪(ExionLCTMAD,https://sciex.com.cn/; QTRAP® 6500+,https://sciex.com.cn/) 对‘福云6号’和‘福黄2号’的类胡萝卜素组分进行了定量,其具体方法参考林馨颖等[15]的方法。液相条件:流动相A甲醇/乙腈(0.01% BHT和0.1%甲酸),流动相B甲基叔丁基醚(0.01% BHT),色谱柱YMC C30 (3 mm, 100 mm× 2.0 mm i.d.)。梯度洗脱条件:0−3 min A/B为100∶0 (V/V),3 min为100∶0 (V/V),5 min为30∶70 (V/V),9 min为5∶95 (V/V),10 min为100∶0 (V/V),11 min为100∶0 (V/V);流速0.8 mL/min;柱温28 ℃;进样量2 μL。质谱条件主要包括:大气压化学离子源(atmospheric pressure chemical ionization source, APCI) 温度350 ℃,气帘气(curtain gas, CUR) 25 psi。在Q-Trap 6500+中,每个离子对是根据优化的去簇电压(declustering potential, DP) 和碰撞能(collision energy, CE) 进行扫描检测。
1.4 主要生化成分含量的测定 利用液质联用仪(Sciex 4500 QTrap质谱仪;Nexera X2LC-30A) 对‘福云6号’和‘福黄2号’游离氨基酸含量进行测定,具体方法参考陈思肜等[16]。儿茶素和生物碱的含量测定参考陈潇敏等[17]方法,对8种儿茶素(GC、EGC、C、EGCG、EC、GCG、ECG、CG) 与3种生物碱(咖啡碱、可可碱、茶叶碱) 进行测定。
1.5 代谢物的LC-MS/MS分析 利用超高效液相色谱质谱联用仪(SHIMADZU Nexera X2, https://www.shimadzu.com.cn/; Applied Biosystems 4500 QTRAP,http://www.appliedbiosystems.com.cn/) 对‘福云6号’和‘福黄2号’进行物质鉴定,液相条件和质谱条件具体参考Lin等[18]的报道。液相条件:流动相A为超纯水(0.1%的甲酸),流动相B为乙腈(0.1%的甲酸);洗脱梯度条件:0 min B相为5%,9 min B相为95% (维持1min),10– 14 min B相为5%;色谱柱:Agilent SB-C18 1.8 µm,2.1 mm´100 mm;流速0.35 mL/min;柱温40 ℃;进样量4 μL。质谱条件:质谱条件主要包括:电喷雾离子源(electrospray ionization,ESI) 温度550 ℃,离子喷雾电压(ion spray voltage, IS) 5 500 V (正离子模式)/–4 500 V (负离子模式);离子源气体Ⅰ (ion source gas Ⅰ, GSⅠ),气体Ⅱ (ion source gas Ⅱ,GSⅡ) 和帘气(curtain gas, CUR) 分别设置为50、60和25 psi,碰撞诱导电离参数设置为高。差异代谢物筛选分析是通过多元统计分析VIP值(variable importance in projection,VIP)、P值(P-value,P)及差异倍数,并对差异代谢成分进行聚类分析、绘制热图、KEGG (Kyoto encyclopedia of genes and genomes,KEGG) 富集分析。
1.6 转录组测序及分析 对‘福云6号’和‘福黄2号’的总RNA的提取及质量控制参考Zheng等方法[19]。利用Illumina HiSeq测序平台(迈特维尔生物科技有限公司,武汉) 对文库片段进行测序。所获得的结果与茶树‘黄棪’基因组[4]进行序列比对。对所获得的基因序列进行计数(featureCounts[20])、计算(FPKM) 及校正(DESeq2、Benjamini-Hochberg法)。筛选差异表达基因,其条件为|log2fold change|≥1,且FDR < 0.05。差异基因的KEGG富集分析使用clusterProfiler软件[21]。
1.7 转录组数据验证 随机挑选7个差异表达基因验证转录组数据的可靠性,通过Primer3Plus设计荧光定量引物(表 1)。根据已提取的总RNA,以CsGAPDH (GenBank登录号:GE651107) 作为内参基因[22],利用全式金Easyscript One-step gDNA Removal and cDNA synthesis superMix试剂盒(北京全式金生物技术股份有限公司) 合成‘福黄2号’和‘福云6号’的cDNA作为实时荧光定量PCR模板。具体方法参照林馨颖等[23]方法。
表 1 引物序列
Table 1 Primer sequences
| Gene ID |
Gene name |
Forward primer sequence (5′→3′) |
Reverse primer sequence (5′→3′) |
| HD.13G0020620 |
HEMA |
ATCACTGGCTTTGGCAGGAA |
CAGCAGCAATGTTCGTCTCG |
| HD.08G0023950 |
NCED |
AGCCCAAACCCTCTACTCCA |
CGGAGACAACGATGACGGAT |
| HD.07G0015710 |
LHCB1 |
CTTGGGCAACCCTAGCTTGA |
GCTAGGCCCAATGGATCGAA |
| HD.10G0001820 |
CAR |
CACGTCTCTCACTACGGCAT |
AGACGCGTCCATCTGAATCC |
| HD.06G0022520 |
CYNT |
TCCATCTCGTGTCCTCGACT |
CGACTGTGTCCGATGACCAA |
| HD.03G0016260 |
LRR |
ACTTTGTCGCCTCTCCCATC |
GGTGAAGCTAGAAGCGTGGA |
| HD.02G0006170 |
PBP1 |
TCAATGGTTGAGAGGCTCGG |
CAGACCCTTTTCCACGTCCA |
2 结果与分析
2.1 ‘福黄2号’芽叶表型特征和超微结构分析 采集‘福黄2号’和‘福云6号’的新梢一芽二叶(图 1A1、B1)。对其进行超微结构观察,‘福云6号’细胞结构清晰、液泡充盈、叶绿体(chloroplast, Ch) 细胞膜结构完整,沿着细胞壁(cell wall, CW) 分布,多呈梭形或长椭圆形,类囊体(thylakoid, Th) 结构较为完整,基粒(grana, Gr) 层数较多,连接各基粒片层的基质类囊体膜结构清晰可见,有少许嗜锇颗粒(osmophilic granule, OG) (图 1A2、A3、A4)。‘福黄2号’较绿叶而言,其细胞中产生较多液泡(vacuole, Va),细胞结构模糊,叶绿体细胞膜结构不完整,与细胞壁分散较远,游离在细胞内,呈现不规则形状。叶绿体内部结构发育不良,基粒片层大部分排列散乱、间隙大,类囊体呈丝状(图 1B2、B3、B4)。以上结果说明茶树色泽变化的叶片叶绿体超微结构发生较大变异。
2.2 ‘福黄2号’春梢芽叶代谢组分析
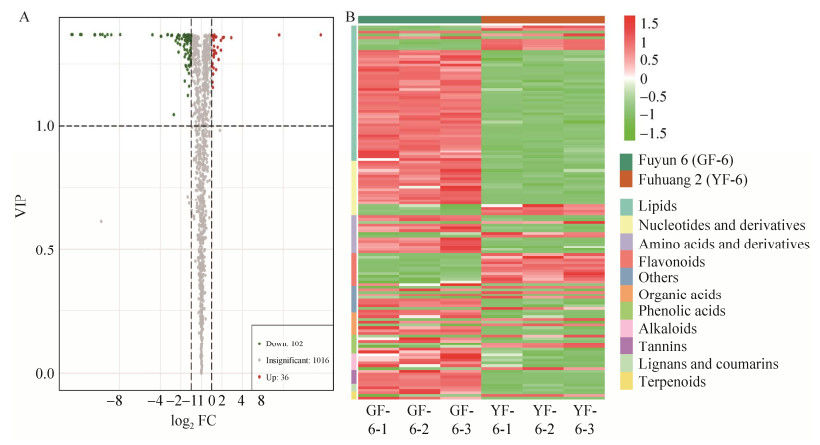
2.2.1 ‘福黄2号’代谢物分析及差异代谢物筛选 在‘福黄2号’和‘福云6号’中共鉴定出1 154种代谢物,主成分分析和正交偏最小二乘判别分析结果显示(图已提交国家微生物科学数据中心(编号:NMDCX0000139)),两者代谢物明显分离。以VIP≥1,fold change≥2和fold change≤0.5为筛选条件,筛选到138种差异代谢物,其中有36种含量显著上调,102种显著下调,这些差异代谢物可分为11类(图 2A、2B)。
为了更清楚地观察‘福云6号’和‘福黄2号’差异代谢物的变化,构建138个差异代谢物的热图生成(图已提交国家微生物科学数据中心(编号:NMDCX0000139))。发现20个核苷酸和衍生物在‘福云6号’和‘福黄2号’中存在差异积累,其中16个在‘福黄2号’中显著降低,包括腺苷、鸟苷、尿嘧啶核苷等。12种黄酮代谢物在‘福黄2号’中有差异积累,尤其是槲皮素类和芹菜素类,黄酮合成前体物之一的肉桂酸显著上调,上调了3倍。在‘福黄2号’中鉴定到大量脂质,共50种,并呈显著下调趋势,共有43种下调,有趣的是,上调的7种代谢物均为游离脂肪酸和甘油酯,而下调的基本为溶血磷脂酰胆碱。与‘福云6号’相比,检测到8种有机酸在‘福黄2号’中发生显著变化,其中增加了6个,减少了2个,其中酮戊二酸显著上调2.24倍。
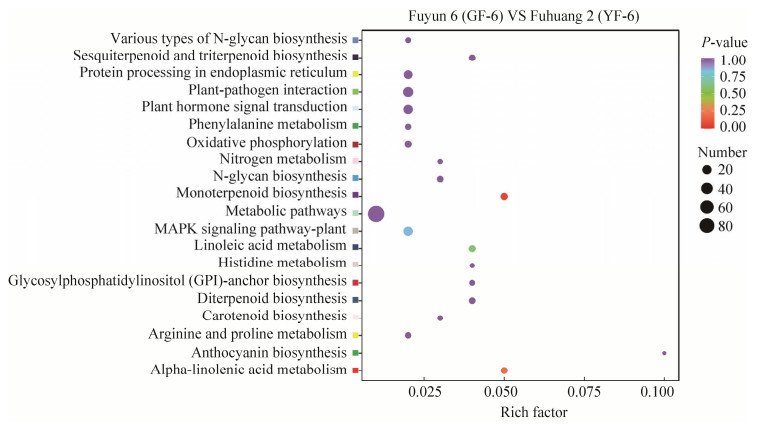
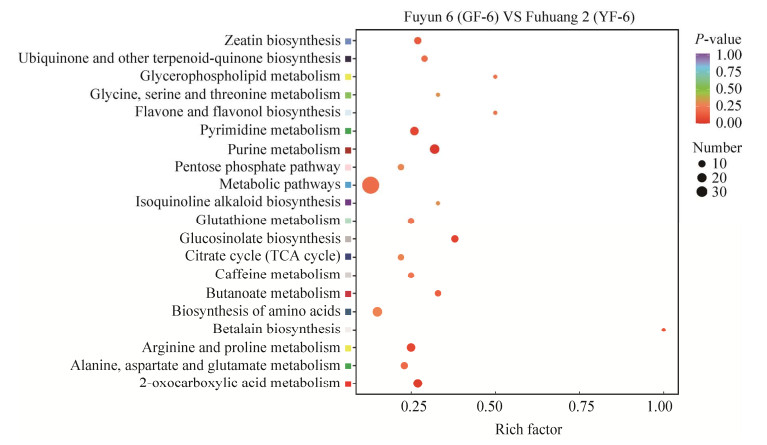
2.2.2 ‘福黄2号’和‘福云6号’差异代谢物KEGG功能富集分析 ‘福黄2号’和‘福云6号’春梢芽叶差异代谢物KEGG富集结果表明,138种差异代谢物共富集到58条通路,其中主要富集到氨基酸生物合成途径、TCA循环、黄酮生物合成、嘌呤代谢等途径(图 3)。与碳和氮相关的代谢通路发生代谢重编事件,如各氨基酸代谢途径和磷酸戊糖途径等。这些结果表明在‘福黄2号’中为应对外部环境胁迫,在黄化叶片中可能发生大规模代谢重编事件。
2.3 ‘福黄2号’芽叶转录组分析
2.3.1 ‘福黄2号’转录组数据质量分析及差异表达基因筛选 如表 2所示,在‘福云6号’和‘福黄2号’获得的序列数据质量好,其中过滤序列Q20在96.09%以上,Q30在89.29%以上,整体测序错误率在0.03%,可用于后续分析,并筛选(|log2 fold change|≥1,且FDR < 0.05) 出658个差异表达基因,包括283个上调基因和376个下调基因。
表 2 ‘福云6号’ (GF-6) 和‘福黄2号’ (YF-6) 转录组数据
Table 2 The transcriptome data of 'Fuyun 6' and 'Fuhuang 2'
| Sample |
Raw reads |
Clean reads |
Clean base (G) |
Error rate (%) |
Q20 (%) |
Q30 (%) |
GC content (%) |
| GF-6-1 |
49 101 520 |
48 091 016 |
7.21 |
0.03 |
96.52 |
90.16 |
44.93 |
| GF-6-2 |
45 410 156 |
44 409 254 |
6.66 |
0.03 |
96.42 |
89.99 |
44.77 |
| GF-6-3 |
48 141 540 |
47 228 224 |
7.08 |
0.03 |
96.45 |
90.03 |
44.76 |
| YF-6-1 |
46 992 978 |
46 053 770 |
6.91 |
0.03 |
96.26 |
89.65 |
44.20 |
| YF-6-2 |
47 683 114 |
46 663 040 |
7.00 |
0.03 |
96.49 |
90.07 |
44.70 |
| YF-6-3 |
51 634 474 |
50 410 604 |
7.56 |
0.03 |
96.09 |
89.29 |
44.53 |
| Total |
288 963 782 |
282 855 908 |
42.42 |
| | | |
2.3.2 ‘福黄2号’差异表达基因KEGG功能富集分析 ‘福云6号’和‘福黄2号’差异表达基因共富集到86条途径(图 4)。差异表达基因主要富集到碳氮相关的途径,包括:内质网中的蛋白质加工、氮代谢、苯丙氨酸代谢、聚糖生物合成等途径,这些途径与差异代谢物的分类密切相关,暗示了茶树黄化的产生与代谢物和基因调控密切相关。
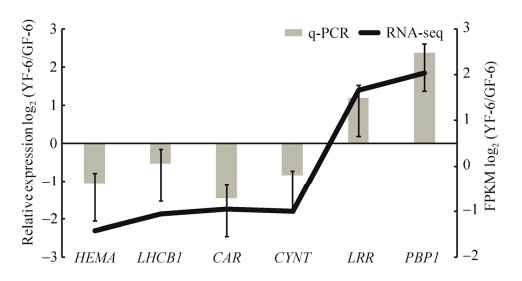
2.3.3 ‘福黄2号’差异表达基因RT-qPCR验证分析 为验证转录组数据的可靠性,选取6个差异表达基因,进行qRT-PCR验证(表 1,图 5)。将荧光定量表达水平与转录组基因表达丰度进行Log2转换后,结果发现,荧光定量表达与转录组基因表达的变化趋势相同,转录组数据具有可靠性。
2.4 ‘福黄2号’生化成分含量分析 对‘福黄2号’芽叶游离氨基酸进行靶向测定,共鉴定到20种游离氨基酸、5种儿茶素和2种生物碱,色谱图已提交国家微生物科学数据中心(编号:NMDCX0000139)。20种游离氨基酸在‘福黄2号’中含量上调,包括茶氨酸、天冬氨酸、精氨酸等在‘福黄2号’中均显著高于‘福云6号’,尤其是茶氨酸,达到57.37 mg/g (表 3)。对8种儿茶素进行测定,共检测出5种,包括儿茶素(catechin, C)、表没食子儿茶素(epigallocatechin, EGC)、表儿茶素没食子酸酯(epicatechin gallate, ECG)、表儿茶素(epicatechin, EC)、表没食子儿茶素没食子酸酯(epigallocatechin gallicacid, EGCG),其中黄化种EGC和EC含量显著高于绿叶品种。在‘福黄2号’中可可碱含量显著低于‘福云6号’,咖啡碱含量的差异未达到显著水平。
表 3 ‘福云6号’和‘福黄2号’的主要生化成分含量(mg/g)
Table 3 Biochemical content of 'Fuyun 6' and 'Fuhuang 2'
| Content |
Fuyun 6 |
Fuhuang 2 |
| Content |
Fuyun 6 |
Fuhuang 2 |
| GABA |
0.11±0.01b |
0.58±0.04a |
| Valine |
0.12±0.01a |
0.27±0.03a |
| β-ABA |
0.13±0.01b |
0.26±0.02a |
Isoleucine |
0.15±0.02b |
0.40±0.06a |
| Alanine |
0.30±0.03b |
0.86±0.12a |
Histidine |
0.07±0.01b |
0.27±0.01a |
| Theanine |
27.88±2.30b |
57.37±1.95a |
Glycine |
0.05±0.01b |
0.12±0.01a |
| Proline |
0.24±0.02b |
1.15±0.19a |
Glutamate |
2.30±0.20b |
4.75±0.16a |
| Lysine |
0.18±0.03b |
0.76±0.10a |
Glutamine |
0.89±0.12b |
2.92±0.30a |
| Tyrosine |
0.22±0.01a |
0.30±0.03a |
Arginine |
0.15±0.02b |
3.28±0.41a |
| Leucine |
0.15±0.02b |
0.37±0.04a |
Serine |
0.84±0.19b |
2.85±0.24a |
| Tryptophan |
0.33±0.01a |
0.24±0.02a |
Asparagine |
0.14±0.01b |
0.57±0.04a |
| Threonine |
0.22±0.03b |
0.80±0.10a |
Total |
34.77±3.08b |
82.07±3.86a |
| Aspartate |
0.30±0.08b |
4.01±0.28a |
| | |
| C |
2.67±0.21a |
2.03±0.49a |
EGC |
13.00±0.97b |
27.32±3.36a |
| ECG |
23.96±0.83a |
18.37±0.80b |
EC |
11.78±0.37b |
15.59±0.39a |
| EGCG |
36.74±1.44a |
36.36±0.67a |
Total |
88.15±3.74b |
99.67±4.12a |
| Theobromine |
1.55±0.11a |
0.22±0.01b |
Total |
24.38±4.01a |
20.60±0.60a |
| Caffeine |
22.82±3.90a |
22.62±1.11a |
| | |
| a, b: indicated significant difference at the P < 0.05 level. |
2.5 ‘福黄2号’DEGs和SCMs的联合分析
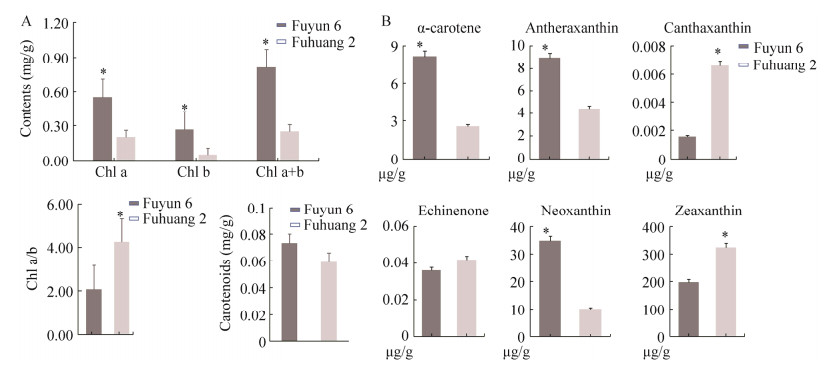
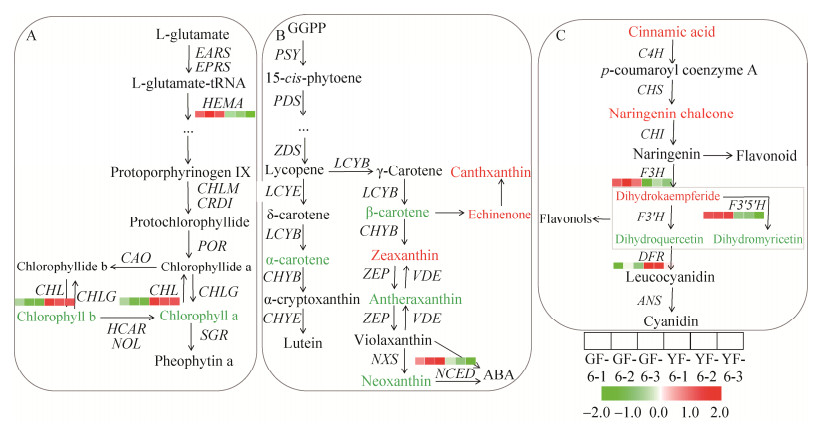
2.5.1 色素代谢相关的DEGs和SCMs联合分析 叶绿素、类胡萝卜素和类黄酮对茶树叶片色泽有影响。本研究测定了‘福云6号’和‘福黄2号’的叶绿素a、叶绿素b和总叶绿素含量,结果如图 6A所示,‘福黄2号’的叶绿素a、叶绿素b和总叶绿素含量较‘福云6号’显著下降,正常绿叶表型的形成主要由叶绿素含量决定。在叶绿素合成途径中,谷氨酰tRNA还原酶(glutamyl-tRNA reductase, HEMA) 表达量显著下调,下调约1/3,叶绿素酶(chlorophyllase, CLH) 表达水平显著上调,上调4.32倍(图 7A)。CLH是编码叶绿素降解相关的酶基因,其表达显著上调影响叶绿素的合成降解效率,是茶树叶色发生变化的重要原因。
类胡萝卜素是植物中另一种重要的捕光色素。类胡萝卜素总量和类胡萝卜素各组分进行测定,其结果表明,‘福黄2号’中的α-胡萝卜素、花药黄质和新黄质的含量显著下降,角黄质和玉米黄质的含量显著增加,海胆烯酮的含量上升但未达到显著水平(图 6B),但总含量未达到显著差异水平(图 6A)。在类胡萝卜素生物合成途径中(图 7B),发现9-顺式-环氧类胡萝卜素双加氧酶(9-cis-epoxycarotenoid dioxygenase, NCED) 的表达显著下调,可能会调节类胡萝卜素的降解。
类黄酮代谢途径中,与‘福云6号’相比,‘福黄2号’中关键合成前体肉桂酸的表达提高了3倍(图 7C)。随后的合成步骤,包括柚皮苷查尔酮和二氢山奈素,以及大多数类黄酮化合物(类黄酮、黄烷酮和黄酮醇) (数据已提交国家微生物科学数据中心(编号:NMDCX0000139)) 在‘福黄2号’中明显积累。类黄酮生物合成途径相关的差异表达基因发生变化,类黄酮3β-羟化酶(flavonoid 3β-hydroxylase, F3H) 和类黄酮3′, 5′-羟化酶(flavonoid 3′, 5′-hydroxylase, F3′5′H) 是类黄酮合成途径的关键酶,表达显著下降,分别下调了17/25、1/2,二氢黄烷醇4-还原酶(dihydroflavanol 4-reductase, DFR) 的表达显著上调,上调了1.80倍。
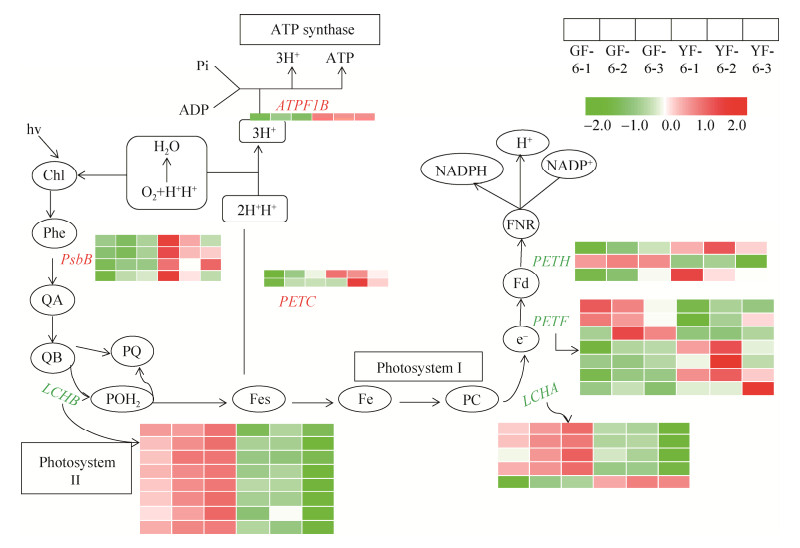
2.5.2 光合作用相关的DEGs和SCMs联合分析 黄化茶树中与光合作用相关的DEGs表达水平发生差异变化(图 8)。LHCA和LHCB是叶绿素a和叶绿素b的结合蛋白基因(light- harvesting complex Ⅱ chlorophyll a/b binding protein, LHCA和LHCB),并能激发能量转移到光系统Ⅰ (photosystem Ⅰ, PSⅠ) 和光系统Ⅱ(photosystem Ⅱ, PSⅡ) 的反应中心,在‘福黄2号’中表达水平显著降低。此外,与PSⅠ反应中心相关的10个DEGs:铁氧还蛋白-NADP+还原酶(ferredoxin-NADP+ reductase, PETH) 3个,铁氧还蛋白酶(ferredoxin, PETF) 7个,与PSⅡ反应中心相关的6个DEGs:光系统Ⅱ CP47叶绿素脂蛋白(photosystem ⅡCP47 chlorophyll apoprotein, PsbB) 4个,细胞色素b6-f复合铁硫亚基(cytochrome b6-f complex iron-sulfur subunit, PETC) 2个,与正常绿叶相比,黄化种中的表达水平发生变化。
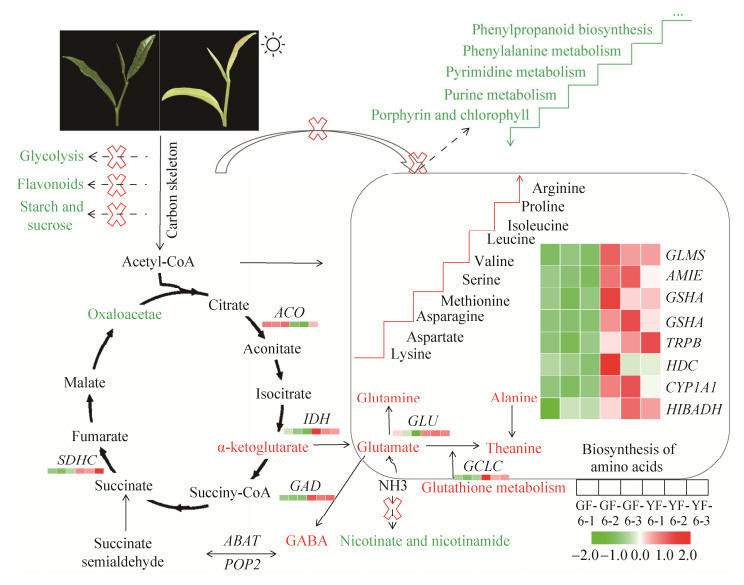
2.5.3 氨基酸途径及其他代谢途径相关的DEGs和SCMs联合分析 图 9为一个简化的氨基酸代谢网络,说明了黄化茶树突变体氨基酸代谢的紊乱。参与氨基酸合成途径的相关差异表达基因呈上调趋势,谷氨酸脱羧酶(glutamate decarboxylase, GAD) 是γ-氨基丁酸(gamma-aminobutyric acid, GABA) 合成途径中重要酶,其表达上调。谷氨酰胺合成酶(glutamine synthase, GLU)、酰胺酶(amidase, AMIE) 等表达均在黄化种中上调。
通过结合差异代谢物和差异表达基因的KEGG富集分析,发现与‘福云6号’相比,‘福黄2号’中与碳骨架相关的物质积累均下降(表 4),包括淀粉和蔗糖代谢、糖酵解、类黄酮途径。蔗糖合酶、海藻糖6-磷酸、果糖二磷酸醛缩酶表达显著下调,D-果糖-1, 6-二磷酸、木糖醇、半乳糖醇、阿东糖醇、D-甘露醇含量显著下降。叶绿素和其他含氮分子生物合成减少,如表 4所示,嘌呤代谢、嘧啶代谢等氮素代谢途径中相关的代谢物和差异表达基因下调(表 4)。与苯丙烷生物合成和苯丙氨酸代谢相关的3个差异表达基因和2个差异代谢物的水平显著降低。在谷胱甘肽代谢途径中,γ-谷氨酰半胱氨酸合成酶(γ-glutamylcysteine synthase, GCLC)的转录水平上调了1.50倍,脱氢抗坏血酸也显著增加了1.68倍。有趣的是,在三羧酸循环途径中,观察到乌头酸水合酶和草酰乙酸呈现下调,而异柠檬酸脱氢酶和α-酮戊二酸上调,其中α-酮戊二酸含量的上调与氨基酸合成途径密切相关。
表 4 其他代谢途径中涉及的DEG和SCM
Table 4 DEGs and SCMs involved in other metabolic pathways
| Pathway name |
ko ID |
Genes (log2 fold change) |
Metabolites (log2 fold change) |
| Glutathione metabolism |
ko00480 |
g-glutamylcysteine synthetase (1.50) |
Dehydroascorbic acid (1.68) |
| Purine metabolism |
ko00230 |
Adenylate cyclase (–4.43) |
Adenosine (–2.12)
Cyclic 3′, 5′-adenylic acid (–1.04)
Adenosine 5′-mono phosphate (–2.95)
3′-adenylic Acid (–1.27)
2′-deoxya denosine-5′-monophosphat (–1.61)
Guanosine 5′-mon ophosphate (–2.38)
Inosine 5′-monophosphate (–2.87) |
| Pyrimidine metabolism |
ko00240 |
Uridine kinase (1.89)
Carbamoyl-phosphate synthase (–1.05) |
Cytidine 5′-monophosphate (–2.23)
Uridine 5′-monophosphate (–1.54)
Uridine (–1.13)
2-deoxyribose-1-phosphate (–2.37)
Orotic acid (–2.92) |
Starch and sucrose
metabolism |
ko00500 |
Sucrose synthase (1.98)
Trehalose 6-phosphate phosphatase (–1.32) |
|
Glycolysis/
gluconeogenesis |
ko00010 |
Fructose-bisphosphate aldolase (–0.19) |
d-fructose-1, 6-biphosphate (–1.19)
Xylitol (–0.90)
Dulcitol* (–0.69)
Ribitol* (–1.64)
d-mannitol* (–1.39) |
| Phenylalanine metabolism |
ko00360 |
Primary-amine oxidase (–1.20) |
l-tyrosine (–1.38) |
| l-phenylalanine (–0.66) |
| Phenylpropanoid biosynthesis |
ko00940 |
Shikimate O-hydroxycinnam
oyltransferase (–1.43)
Scopoletin glucosyltransferase (–1.31) |
l-tyrosine (–1.38)
Sinapinaldehyde (–1.04) |
| Nitrogen metabolism |
ko00910 |
Carbonic anhydrase (CYNT) (–1.10)
Carbonic anhydrase (CAH) (–2.23) |
|
| Citrate cycle (TCA cycle) |
ko00020 |
Aconitate hydratase (–0.41)
Isocitrate dehydrogenase (0.62) |
Oxaloacetic acid (–1.62)
α-ketoglutaric acid (1.16) |
3 讨论
3.1 ‘福黄2号’的叶色变异机理分析 植物叶片的色泽与叶绿体的发育和分裂,以及叶绿素的生物合成和运输相关[24]。‘福黄2号’是茶树‘福云6号’自然黄化突变种,新梢呈现黄色表型。对其进行超微结构观察,发现黄化叶片较绿叶而言,细胞结构模糊,细胞膜发育不完整,脱离细胞壁,游离在细胞内,并产生较多液泡(空泡),叶绿体结构发育不良,基粒片层大部分排列散乱、间隙大,类囊体呈丝状。研究表明,类囊体膜规则排列并堆积成基粒的叶绿体是植物光能吸收与转化的重要场所,大多数叶色突变体类囊体膜存在变异,银杏叶片中含有破裂的类囊体膜,形成分散的液泡[25]。因此,叶片黄化表型的产生可能与叶绿体发育异常有关。植物叶片颜色还受到色素的影响,叶绿素和类胡萝卜素是以光捕获能量的主要色素[6]。叶绿素代谢是导致黄化表型的重要因素之一,许多叶绿素缺乏的叶色突变体的表型是由参与叶绿素生物合成的基因表达改变引起的[25]。色素测定分析表明,‘白叶1号’ ‘黄叶肉桂’ ‘黄金水仙’ ‘福黄1号’等黄化茶树中叶绿素a和叶绿素b含量降低[4, 14, 26-27],这与本研究结果一致,‘福黄2号’叶绿素a和叶绿素b显著低于‘福云6号’,且参与叶绿素代谢途径的基因在黄叶中也表现出不同表达水平,其中编码叶绿素降解相关酶CHL在黄叶中显著上调,上调4.32倍,这与在‘白叶1号’的研究相似[26]。这暗示了CHL在‘福黄2号’叶绿素降解过程中加快了叶绿素的降解效率,从而使得叶绿素a和b含量在黄叶中显著下降。
光合作用发生在叶绿体中,叶绿体的异常发育和叶绿素生物合成的改变可能会影响‘福黄2号’的光合能力。黄化种中与光合作用相关的DEGs表达水平差异变化(图 8),LHCA和LHCB是叶绿素a和b的结合蛋白基因,在‘福黄2号’中表达水平显著降低。此外,与PSⅠ反应中心相关的10个DEGs和与PSⅡ反应中心相关的6个DEGs在黄化种中的表达水平发生变化。PSⅠ、PSⅡ和细胞色素b6/f参与光反应,这是光合作用的初始阶段,LHC在PSⅠ和PSⅡ的捕光中起着重要作用。在拟南芥中LHC表达减少导致叶绿体中基粒堆积受损[28]。‘中黄1号’也观察到LHC基因的下调可能导致叶绿体发育异常和叶绿素含量降低[9]。由此推测,‘福黄2号’黄化表型可能是光合作用蛋白、叶绿素代谢基因和叶绿素含量的缺乏所致。
3.2 ‘福黄2号’高茶氨酸代谢机理分析 氨基酸合成主要在叶绿体中[29],黄化茶树中叶绿体结构缺失破坏了碳氮平衡,进而导致游离氨基酸的积累。通过对‘福黄2号’和‘福云6号’研究发现,叶绿体发育异常(图 1)、类黄酮含量降低、光合作用受损(图 8)、碳水化合物含量降低以及游离氨基酸积累(图 9和表 4),这表明了在‘福黄2号’叶片中与碳、氮代谢相关的途径受到抑制或者加强,叶片中碳氮失衡。已有研究表明,光合速率和碳代谢降低使得氮代谢增强[30]。而在‘黄金芽’的研究中也发现,黄化茶树处于缺碳状态,TCA循环被激活,氮积累和碳供应短缺共同作用引起蛋白质降解,膜结构的破坏和大分子的分解导致了细胞的自噬,从而增加了黄化叶片中氨基酸的含量[3]。茶树叶片的主要含氮化合物是蛋白质、叶绿素和咖啡碱[31-32]。‘福黄2号’和‘福云6号’的咖啡碱含量较高,但两者间的差异未达到显著水平,这表明咖啡碱不是黄化茶树中氮消耗的一个因素。在‘福黄2号’中苯丙氨酸代谢、苯丙素生物合成等氮素代谢途径表达呈下调,氮源消耗降低。α-酮戊二酸在黄化茶树中能够促进氮元素的再循环,其含量在‘福黄2号’中显著上升,参与氨基酸合成途径的相关差异基因呈上调趋势(图 9),如谷氨酸脱羧酶(GAD)、谷氨酰胺合成酶(GLU)、酰胺酶(AMIE),茶氨酸、精氨酸、谷氨酸、谷氨酰胺等游离氨基酸含量显著上升,尤其是茶氨酸含量达到53.37 mg/g。在茶树中,谷氨酸是茶氨酸的合成前体之一,当谷氨酸含量积累,会加速茶氨酸的合成,这暗示了在‘福黄2号’中游离氨基酸的积累可能是低氮消耗,如叶绿素和其他含氮分子生物合成减少,氮源积累被用于氨基酸合成,茶氨酸显著积累[33-34]。
4 结论 本研究以‘福云6号’ (GF-6) 及其黄化品系‘福黄2号’ (YF-6) 为供试材料,利用多组学联合分析茶树叶色黄化和高茶氨酸累积的潜在分子机制。其中,‘福黄2号’黄化表型可能是光合作用蛋白、叶绿素代谢基因和叶绿素含量的缺乏所致。具有较高含量的茶氨酸以及其他多种游离氨基酸,其积累可能是氮资源、氨基在叶片中被更有效储存,使得氨基酸合成通路中的代谢物及相关基因表达上调,茶氨酸成为黄化叶中显著积累的含氮化合物。高茶氨酸黄化茶树新品系‘福黄2号’具有科学研究与利用价值,采用多组学技术探究黄化变异形成的机制,为高茶氨酸茶树资源选育提供新的材料。
 2022, Vol. 38
2022, Vol. 38