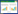中国科学院微生物研究所、中国微生物学会主办
文章信息
- 褚鑫, 王力为, 许虹, 张燕飞
- CHU Xin, WANG Liwei, XU Hong, ZHANG Yanfei
- 工业生物技术的前沿科技
- Frontier science for industrial biotechnology
- 生物工程学报, 2022, 38(11): 4019-4026
- Chinese Journal of Biotechnology, 2022, 38(11): 4019-4026
- 10.13345/j.cjb.220717
-
文章历史
- Received: September 7, 2022
- Accepted: September 22, 2022
2. 中国科学院天津工业生物技术研究所, 天津 300308
2. Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China
随着经济社会高速发展以及全球人口数量日益增长,资源供需矛盾日益尖锐;同时温室气体大量排放,造成全球气温升高和极端灾害性天气频发,气候变化已成为人类发展面临的最大威胁。解决能源和资源持续供给、应对全球气候变化,实现绿色可持续发展已成为世界各国共同面对的重大挑战。工业生物技术是以生物质、二氧化碳等可再生碳资源为原料,以生物细胞或工业酶为工具,生产医药、化工、能源、材料等产品的物质合成与加工模式,具有典型的绿色、可持续等特征[1]。工业生物技术是现代社会由化石经济向生物经济过渡的必要手段,也是生物经济的重点发展方向[2]。
1 生命科学突破促进了工业生物技术的快速发展从20世纪后半叶开始,以分子生物学为代表的生命科学突破性成就孕育和促进了全球生物技术的飞速发展。工业生物技术的发展是继医药生物技术、农业生物技术之后生物技术发展的“第三次浪潮”[3],对未来制造业将产生巨大的影响。相比医药和农业生物领域的长研发周期和高研发投入,主要以微生物为研究对象的工业生物技术对生命科学新发现、新理论、新知识、新技术的吸收、拓展和运用更加迅速和高效,工业生物技术已经突破了简单改造生物系统的阶段,正在大步迈向工程生物学这个将生物学工程化、数字化和自动化的发展新阶段(图 1)。
|
| 图 1 工业生物技术的学科基础变迁 Fig. 1 The transition of the discipline basis of industrial biotechnology. |
| |
1928年,Fleming首先发现了青霉素[4],证明其是一种由青霉菌产生的可以选择性抑制其他微生物的次级代谢产物,开启了抗生素研究时代,后续研究又进一步开发出了卡那霉素、链霉素、氯霉素等。抗生素化学本质的确定和抗性基因的挖掘形成了丰富的工具库,成为工业微生物菌株重构和筛选的必要手段。1953年,Watson和Crick提出DNA双螺旋结构模型吹响了现代分子生物学发展的号角[5]。中心法则和基因学说等基础理论的发展支撑和促进了工业微生物育种技术的发展[6]。转座元件(生物体内非游离的、能自复制或自剪切拷贝的、并能在生物体基因组内不断移动位置的功能性DNA片段) 的发现和研究至今依旧支撑着工业微生物的基因组扰动和编辑,并拓展出与成簇的规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats, CRISPR) 联用的精确基因编辑技术[7]。法国科学家Jacob和Monod于1961年提出的操纵子学说[8],揭示了蛋白质合成中的调节机制,开创了“中心法则”以外的基因调控研究新领域。操纵子概念的普适性在后续研究中得到充分证实,基于此发展出了众多适用于不同工业微生物的基因表达调控系统,在工业生物细胞工厂生理代谢的时空调控、蛋白质生产等方面都发挥了重要作用。DNA合[A3] 成技术是生命科学和分子生物学研究的重要基础技术,从单链寡核苷酸链合成到双链DNA合成,主要经历了柱合成、芯片合成、超高通量芯片合成和酶促合成等几个阶段,有力驱动了工业生物领域中基因密码子优化、代谢途径优化、基因组合成等研究,在抗体生产及蛋白工程等领域也发挥了重要作用。1985年,Mullis发明了聚合酶链式反应(polymerase chain reaction, PCR) 技术[9],通过体外扩增特定DNA片段,实现了微量DNA的低成本高通量扩增,为工业生物技术进入基因工程时代奠定了基础。基因编辑是一种新兴的、能对生物体基因组特定目标基因进行修饰和编辑的基因工程技术。先后发展出多种基于同源重组(如λ-Red)、核酸酶(如TALEN、CRISPR-Cas等) 的基因编辑技术,通过基因组编辑、重写和优化为工业微生物在基因层面的理性设计和改造奠定了理论和技术基础。
2 生物工程等学科的交叉创新显著推动了工业生物技术的发展紧随分子生物学发展,具有融合特征的生物工程(Biological Engineering or Bioengineering) 作为一门综合性应用学科于20世纪70年代开始兴起并得到迅速发展,并先后发展出了代谢工程与合成生物学等更加体现“融合”与“汇聚”特征的新学科、新方法,促进了工业生物技术在过去50年的高速发展与产业化进程。生物工程是生物学与工程学及其他学科的融合,它将生物学的理论和技术与工程学(化工、机械、电子计算机等现代工程技术) 进行融合,充分运用分子生物学的最新技术手段,定向地改造生物体或/及其功能元件,创造出具有超远缘性状的新物种,再将构建的“工程菌”进行大规模的发酵培养,以生产特定代谢产物或发挥特定生理功能。
1986年,Liao等率先开展了酶分子的功能进化研究[10]。1993年,Arnold团队的工作让“定向进化” (directed evolution) 概念为大众接受[11]。该技术通过随机突变和DNA重组制造大量的蛋白质突变体,按照特定的需求和目的给予选择压力,筛选出具有期望特征的突变体,为高效生物催化剂的开发提供了强有力的工具。20世纪90年代,Bailey和Stephanopoulos等以微生物代谢网络和途径为研究对象,首先提出代谢工程(metabolic engineering) 概念[12-13],旨在利用DNA重组技术有目的地改造微生物细胞的代谢途径、催化功能、转运系统和调控功能,从而改善细胞活性,有效推进了工业微生物细胞工厂的构建和微生物发酵产业的发展。同时期,自动化DNA测序和计算机辅助基因组测序数据处理技术的进步,以及高通量转录组、蛋白质组、代谢组研究成果积累催生了系统生物学(Systems Biology),它旨在系统研究细胞内组分组成及其相互作用,最终指导细胞代谢网络的反向改造(反向代谢工程,inverse metabolic engineering)。20世纪90年代末期,逐步形成与“自上而下” (top-down) 的系统生物学互补的“自下而上” (bottom-up) 的合成生物学领域,“目标导向性地设计和构建新型生物学元件(parts)、装置(devices) 和系统(systems),或者对现有的生物系统进行重新设计”,旨在阐明并模拟生物合成的基本规律,达到人工设计并构建新的具有特定生理功能的生物系统,从而建立药物、化学品、功能材料和替代燃料等的生物制造途径。2000年初,Collins等首先报道了一个可以交互抑制的转录抑制因子的遗传可拨动开关,可以使宿主细胞响应外界信号,在两种稳定的表达状态之间自由切换,实现了对生物系统的重设计[14]。2003年,Keasling等应用合成生物学理念实现了抗疟药物前体青蒿酸在酿酒酵母中合成[15]。2013年,Amyris公司进一步将其提升到产业化规模[16]。2010年,张学礼等采用代谢工程与合成生物学方法改造大肠杆菌,率先实现l-丙氨酸的厌氧发酵,显著降低生产成本和三废排放[17]。经过多年积累,基于合成生物学逐步形成了“设计-构建-测试-学习” (design-build-test-learn, DBTL) 的微生物细胞工厂开发流程,结合生物铸造自动化平台(biofoundry, 功能包括DNA设计、DNA组装、DNA质量控制、基因编辑、菌株转化、克隆挑选、菌株质量控制、表型测试、高通量筛选、菌株保藏、数据分析、工艺放大等) 的助力,大大加快了微生物细胞工厂的构建进程。
3 新阶段工业生物技术的学科基础是工程生物学随着现代生物制造的智能化、工程化和自动化发展,原有的以“拼接式、应用式”为主的生物工程学科体系难以支撑大规模生物经济的发展需求,发展和建立新的学科基础和研究范式成为支撑现代工业生物技术发展、推动生命科学工业化、生物技术产业化的迫切需求。
工程生物学是生物学工程化理念的延伸,符合新阶段工业生物技术学科基础的特征和需求。工程生物学(Engineering Biology) 是利用生命科学知识,对生物体系的结构与功能进行工程化设计再造的生物学新学科,它使传统的生物学研究进入了工程化、数字化和自动化的新范式,将从根本上提升生物体系的物质合成能力与效率。工程生物学可深入认识理解生物进化、遗传规律、生理和代谢本质,揭示不同环境和条件下生物功能机制与生物行为规律,催生新一代生物技术,是支撑高性能工业菌种和工业酶创制、促进生物产业走向规模化的核心学科基础。在前沿基础方面,工程生物学汇聚计算生物学、系统生物学、工业蛋白质科学、生物有机化学、生物能量学、合成生物学等前沿方向,为生物合成提供基础理论支撑;在方法工具方面,工程生物学发展工程生物信息学(生物组学)、生物数据科学(人工智能)、生物设计与实验自动化(高通量自动化筛选、高通量基因合成与编辑等) 等方向,为生物工具创制形成高效的方法支撑;在应用基础方面,工程生物学发展蛋白质工程、生物催化、代谢工程、发酵生理工程、底盘工程(包括原核底盘、真核底盘,植物底盘工程等)、智能发酵工程、微生物组工程等方向,为生物制造产业发展形成核心技术支撑。
4 工业生物技术前沿突破和发展趋势近年来,工程生物学前沿基础研究取得了一系列重要进展,为解决工业生物技术所面临的基础科学问题奠定了坚实基础。使用机器学习可以精准构建可解释的转录调控模型[18],开发计算模型可以设计非重复性遗传元件[19],人工智能辅助蛋白质的精准人工设计[20-21],深度学习助力酿酒酵母核心真核蛋白质复合物的精准建模[22];酶法DNA合成技术和芯片式原位DNA合成技术取得了突破,其中基于末端脱氧核苷酸转移酶(terminal transferase, TdT) 的酶法DNA合成技术在合成精度、长度、成本、效率等方面具有可以颠覆现有化学方法的优势和巨大潜力[23-24];CRISPR-Cas基因组编辑技术为实现高效、低成本的生物体改造提供了重要技术支撑,人们已经可以实现基因组水平和规模的精准、高通量的改造[25-26];利用机器学习可以实现蛋白结构的快速预测[27]和从头设计[21, 28],快速定向进化蛋白质,优化其功能[29];适应性进化可以应用于研究影响菌株表型、性能和稳定性的进化潜力以及快速获取含有有益突变的工业生产菌株[30],进一步应用全基因组扰动[31-32]、途径特异性原位突变[33-34]以及基因特异性原位突变[35-36]工具,可以有效提高突变几率,配合高通量筛选技术,可以高效推进工业生产菌株的开发;遗传密码扩展技术可以通过增加碱基种类[37-38]或者改造氨酰-tRNA的合成酶[39]向蛋白质中引入非天然氨基酸,对生命科学研究、蛋白质功能进化、蛋白/多肽药物研发等领域产生深刻的影响。
工程生物学前沿的基础研究也促进了二氧化碳的固定与转化利用等低碳生物合成技术陆续获得突破。首次将大肠杆菌[40]和巴斯德毕赤酵母[41]转变为自养型生物的研究,为二氧化碳工业生物转化利用提供了可能。光驱动大肠杆菌二氧化碳固定技术[42],理性设计和高通量定向进化开发新的固碳羧化酶[43]进一步推动了低碳生物制造工业生物技术的发展。我国科学家在国际上首次实现了从二氧化碳到淀粉的人工全合成[44],开辟了以二氧化碳为原料合成淀粉的制造新模式,为二氧化碳到复杂化工产品的工艺路线奠定了基础。未来基础研究需要寻求多种形式的能量(如:电能、光能、氢能、非粮生物质能和其他化能等),更加高效地向细胞导入和定向转换,构建光能、电能或氢能驱动的细胞合成与物质定向合成新范式,以应对生物制造关于如何实现高效物质定向转化和能量转移转换的挑战,并促进生物固碳与低碳合成的工业生物技术发展。
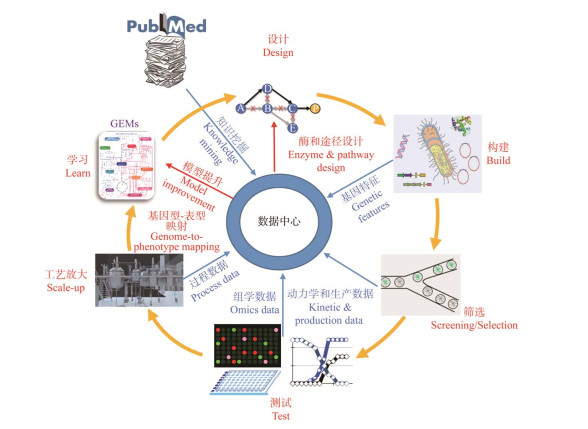
工程生物学需要继续加强前沿基础研究以支撑和促进工业生物技术创新,促进人工智能与生物技术融合,促进数字细胞与代谢网络模型指导的新反应、新途径的设计,促进蛋白质合成与分泌过程预测的研究。同时,工程生物学也需要加强重大科技设施的发展,集成人工智能、自动化、高通量等前沿技术,发展实验自动化、生物铸造自动化平台和智能发酵平台等,以支撑工业生物技术的研发,加速“设计-构建-筛选-测试-工艺放大-学习(design-build- screen-test-process-learn, D-B-S-T-P-L)”工业生物技术研发循环新范式的发展(图 2)。
|
| 图 2 工业生物技术研发“设计-构建-筛选-测试-工艺放大-学习(D-B-S-T-P-L)”循环新范式 Fig. 2 Novel paradigm of "design-build-screen- test-process-learn (D-B-S-T-P-L)" cycle for industrial biotechnology. |
| |
工程生物学需要继续加强前沿应用基础研究,进一步发展无细胞系统应用、生物传感、微生物群落设计与应用,以促进和支撑工业生物技术在低碳绿色制造、工业固碳与固氮、环境监测与修复、高通量高灵敏度的代谢物检测和灵敏传感系统开发等方面的拓展。
| [1] |
李寅. 中科院李寅: 工业生物技术可有效推动绿色经济发展[EB/OL]. [2017-09-26]. https://www. chinaventure.com.cn/cmsmodel/news/detail/319954.html. Li Y. Li Yin from Chinese Academy of Sciences: industrial biotechnology can effectively promote the development of green economy[EB/OL]. [2017-09-26]. https://www.chinaventure.com.cn/cmsmodel/news/detail/319954.html. |
| [2] |
马延和. 生物制造产业是生物经济重点发展方向. 中国生物工程杂志, 2022, 42(5): 4-5. Ma YH. Biomanufacturing is a key area to accelerate the development of bioeconomy. China Biotechnol, 2022, 42(5): 4-5 (in Chinese). DOI:10.13523/j.cb.2205102 |
| [3] |
王钦宏, 马延和. 2019工业生物学专刊序言. 生物工程学报, 2019, 35(10): 1801-1805. Wang QH, Ma YH. Preface for special issue on industrial biology (2019). Chin J Biotech, 2019, 35(10): 1801-1805 (in Chinese). DOI:10.13345/j.cjb.190449 |
| [4] |
Ligon BL. Penicillin: its discovery and early development. Seminars in Pediatric Infectious Diseases, 2004, 15(1): 52-57. DOI:10.1053/j.spid.2004.02.001
|
| [5] |
Watson JD, Crick FHC. Molecular structure of nucleic acids: a structure for deoxyribose nucleic acid. Nature, 1953, 171(4356): 737-738. DOI:10.1038/171737a0
|
| [6] |
戴四发, 黎观红, 吴石金. 现代工业微生物育种技术研究进展. 微生物学杂志, 2000, 20(2): 48-50, 53. Dai SF, Li GH, Wu SJ. Advances in modern industrial microorganism breeding techniques. J Microbiol, 2000, 20(2): 48-50, 53 (in Chinese). |
| [7] |
Klompe SE, Vo PLH, Halpin-Healy TS, et al. Transposon-encoded CRISPR-Cas systems direct RNA-guided DNA integration. Nature, 2019, 571(7764): 219-225. DOI:10.1038/s41586-019-1323-z
|
| [8] |
Jacob F, Monod J. Genetic regulatory mechanisms in the synthesis of proteins. Journal of Molecular Biology, 1961, 3(3): 318-356. DOI:10.1016/S0022-2836(61)80072-7
|
| [9] |
Mullis KB. The unusual origin of the polymerase chain reaction. (0036-8733 (Print)).
|
| [10] |
Liao H, McKenzie T, Hageman R. Isolation of a thermostable enzyme variant by cloning and selection in a thermophile. PNAS, 1986, 83(3): 576-580. DOI:10.1073/pnas.83.3.576
|
| [11] |
Wang YJ, Xue P, Cao MF, et al. Directed evolution: methodologies and applications. Chem Rev, 2021, 121(20): 12384-12444. DOI:10.1021/acs.chemrev.1c00260
|
| [12] |
Bailey JE. Toward a science of metabolic engineering. Science, 1991, 252(5013): 1668-1675. DOI:10.1126/science.2047876
|
| [13] |
Stephanopoulos G, Vallino JJ. Network rigidity and metabolic engineering in metabolite overproduction. Science, 1991, 252(5013): 1675-1681. DOI:10.1126/science.1904627
|
| [14] |
Gardner TS, Cantor CR, Collins JJ. Construction of a genetic toggle switch in Escherichia coli. Nature, 2000, 403(6767): 339-342. DOI:10.1038/35002131
|
| [15] |
Martin VJJ, Pitera DJ, Withers ST, et al. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat Biotechnol, 2003, 21(7): 796-802. DOI:10.1038/nbt833
|
| [16] |
Paddon CJ, Westfall PJ, Pitera DJ, et al. High-level semi-synthetic production of the potent antimalarial artemisinin. Nature, 2013, 496(7446): 528-532. DOI:10.1038/nature12051
|
| [17] |
Zhang XL, Jantama K, Moore JC, et al. Production of l-alanine by metabolically engineered Escherichia coli. Appl Microbiol Biotechnol, 2007, 77(2): 355-366. DOI:10.1007/s00253-007-1170-y
|
| [18] |
La MG. From single-cell RNA-seq to transcriptional regulation. Nat Biotechnol, 2019, 37(12): 1421-1422. DOI:10.1038/s41587-019-0327-4
|
| [19] |
Hossain A, Lopez E, Halper SM, et al. Automated design of thousands of nonrepetitive parts for engineering stable genetic systems. Nat Biotechnol, 2020, 38(12): 1466-1475. DOI:10.1038/s41587-020-0584-2
|
| [20] |
Anishchenko I, Pellock SJ, Chidyausiku TM, et al. De novo protein design by deep network hallucination. Nature, 2021, 600(7889): 547-552. DOI:10.1038/s41586-021-04184-w
|
| [21] |
Huang B, Xu Y, Hu XH, et al. A backbone-centred energy function of neural networks for protein design. Nature, 2022, 602(7897): 523-528. DOI:10.1038/s41586-021-04383-5
|
| [22] |
Baek M, DiMaio F, Anishchenko I, et al. Accurate prediction of protein structures and interactions using a three-track neural network. Science, 2021, 373(6557): 871-876. DOI:10.1126/science.abj8754
|
| [23] |
Palluk S, Arlow DH, De Rond T, et al. De novo DNA synthesis using polymerase-nucleotide conjugates. Nat Biotechnol, 2018, 36(7): 645-650. DOI:10.1038/nbt.4173
|
| [24] |
Lu X, Li J, Li C, et al. Enzymatic DNA synthesis by engineering terminal deoxynucleotidyl transferase. ACS Catalysis, 2022, 12(5): 2988-2997. DOI:10.1021/acscatal.1c04879
|
| [25] |
Nishimasu H, Shi X, Ishiguro S, et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science, 2018, 361(6408): 1259-1262. DOI:10.1126/science.aas9129
|
| [26] |
Anzalone AV, Koblan LW, Liu DR. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol, 2020, 38(7): 824-844. DOI:10.1038/s41587-020-0561-9
|
| [27] |
Jumper J, Evans R, Pritzel A, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596(7873): 583-589. DOI:10.1038/s41586-021-03819-2
|
| [28] |
Kumar P, Paterson NG, Clayden J, et al. De novo design of discrete, stable 310-helix peptide assemblies. Nature, 2022, 607(7918): 387-392. DOI:10.1038/s41586-022-04868-x
|
| [29] |
Yang KK, Wu Z, Arnold FH. Machine-learning-guided directed evolution for protein engineering. Nat Methods, 2019, 16(8): 687-694. DOI:10.1038/s41592-019-0496-6
|
| [30] |
Matson MM, Cepeda MM, Zhang A, et al. Adaptive laboratory evolution for improved tolerance of isobutyl acetate in Escherichia coli. Metab Eng, 2022, 69: 50-58. DOI:10.1016/j.ymben.2021.11.002
|
| [31] |
Badran AH, Liu DR. Development of potent in vivo mutagenesis plasmids with broad mutational spectra. Nat Commun, 2015, 6: 8425. DOI:10.1038/ncomms9425
|
| [32] |
Wang J, Zhao DD, Li J, et al. Helicase-AID: a novel molecular device for base editing at random genomic loci. Metab Eng, 2021, 67: 396-402. DOI:10.1016/j.ymben.2021.08.005
|
| [33] |
Álvarez B, Mencía M, de Lorenzo V, et al. In vivo diversification of target genomic sites using processive base deaminase fusions blocked by dCas9. Nat Commun, 2020, 11(1): 6436. DOI:10.1038/s41467-020-20230-z
|
| [34] |
Cravens A, Jamil OK, Kong DZ, et al. Polymerase-guided base editing enables in vivo mutagenesis and rapid protein engineering. Nat Commun, 2021, 12(1): 1579. DOI:10.1038/s41467-021-21876-z
|
| [35] |
Halperin SO, Tou CJ, Wong EB, et al. CRISPR-guided DNA polymerases enable diversification of all nucleotides in a tunable window. Nature, 2018, 560(7717): 248-252. DOI:10.1038/s41586-018-0384-8
|
| [36] |
Ravikumar A, Arzumanyan GA, Obadi MKA, et al. Scalable, continuous evolution of genes at mutation rates above genomic error thresholds. Cell, 2018, 175(7): 1946-1957.e13. DOI:10.1016/j.cell.2018.10.021
|
| [37] |
Zhang Y, Ptacin JL, Fischer EC, et al. A semi-synthetic organism that stores and retrieves increased genetic information. Nature, 2017, 551(7682): 644-647. DOI:10.1038/nature24659
|
| [38] |
Zhou Y, Xu XX, Wei YF, et al. A widespread pathway for substitution of adenine by diaminopurine in phage genomes. Science, 2021, 372(6541): 512-516. DOI:10.1126/science.abe4882
|
| [39] |
Manandhar M, Chun E, Romesberg FE. Genetic code expansion: inception, development, commercialization. J Am Chem Soc, 2021, 143(13): 4859-4878. DOI:10.1021/jacs.0c11938
|
| [40] |
Gleizer S, Ben-Nissan R, Bar-On YM, et al. Conversion of Escherichia coli to generate all biomass carbon from CO2. Cell, 2019, 179(6): 1255-1263.e12. DOI:10.1016/j.cell.2019.11.009
|
| [41] |
Gassler T, Sauer M, Gasser B, et al. The industrial yeast Pichia pastoris is converted from a heterotroph into an autotroph capable of growth on CO2. Nat Biotechnol, 2020, 38(2): 210-216.
|
| [42] |
Hu G, Li Z, Ma D, et al. Light-driven CO2 sequestration in Escherichia coli to achieve theoretical yield of chemicals. Nat Catal, 2021, 4(5): 395-406.
|
| [43] |
Scheffen M, Marchal DG, Beneyton T, et al. A new-to-nature carboxylation module to improve natural and synthetic CO2 fixation. Nat Catal, 2021, 4(2): 105-115.
|
| [44] |
Cai T, Sun HB, Qiao J, et al. Cell-free chemoenzymatic starch synthesis from carbon dioxide. Science, 2021, 373(6562): 1523-1527. DOI:10.1126/science.abh4049
|
 2022, Vol. 38
2022, Vol. 38