Application-oriented structure and function study of proteins: a review
蛋白质是生命存在的基本功能单元,几乎所有细胞活动都依赖酶蛋白介导的催化反应才能发生,蛋白质结构和功能的研究是了解生命运作的关键[1];蛋白质也是许多药物的作用靶点,如异戊二烯合成酶抑制剂可以作为胆固醇血症的治疗药物等。异常的蛋白质空间结构很可能导致其生物活性的降低、丧失,甚至会导致疾病,如疯牛病、Alzheimer’s症等都是由于蛋白质折叠异常引起的疾病[2]。活体细胞内的蛋白质正常折叠、异常折叠的研究,尤其是分子伴侣和大分子等的参与是这一领域的研究热点。蛋白质的功能不仅决定于蛋白质分子的一级结构,而且与其特定的空间结构密切相关,蛋白质的折叠方式与空间构象对于蛋白质的功能有着决定性的作用。蛋白质的结构可分为活性中心、远活性区和外层,活性中心决定了催化特性,活性区口袋的形状决定了底物选择性和结合模式;远活性区的氨基酸不直接与底物发生作用,但可能间接影响底物结合区的性质;外层的区域与溶剂或者其他蛋白质接触,决定了耐温性等酶学性质[3]。所以蛋白质的结构功能研究是生命科学领域中的核心问题,许多生命奥秘和重大科学问题的揭示依赖于在整体水平上认识蛋白质的结构和功能的构效关系。
在工业相关领域,蛋白质作为生物催化剂,具有反应条件温和、效率高、立体和区域选择性强、污染少等特点,传统酶制剂在食品、医药、纺织、洗涤剂、造纸和农产品加工等方面具有广泛的应用[4];在利用微生物细胞工厂等进行天然产物以及多种化合物产品的合成中,也涉及到许多关键催化酶及关键调控、转运蛋白,对于这些蛋白质的性能研究以及性能提升,将能提高催化反应的专一性以及合成途径的特异性,提高整体生产效率[5]。以合成生物学为代表的新一代生物技术正在引领新一轮科技革命和产业变革[6],生物产业发展前景广阔、势头迅猛,并日益成为创新驱动发展战略、加快建设创新型国家的重要支撑。随着合成生物学技术的飞速发展,合成生物学领域中关键的新酶研究和高效合成路径设计,都依赖于对关键蛋白质结构和功能的理解,所以蛋白质结构和功能研究是合成生物技术领域的核心技术[7]。加强蛋白质结构功能相关支撑能力,可以提升工业蛋白质科学的研究水平,并进一步支撑生物催化和合成生物学的发展。
1 蛋白质结构解析技术进展 近几十年来,X-射线晶体学(X-ray crystallography)、核磁共振(nuclear magnetic resonance, NMR) 以及冷冻电镜(cryo-electron microscopy, Cryo-EM) 等技术都被用来确定蛋白质的构型[8-9]。X-射线晶体学是至今使用最广泛的结构测定方法,早在20世纪60年代,X-射线晶体衍射技术就已经被用来解析蛋白晶体结构如胰岛素的结构等[10]。目前在蛋白质结构数据库(RCSB protein data bank)[11]中使用X-射线晶体衍射技术解析的蛋白结构占比约85%,NMR解析的占8%,Cryo-EM的占5%。从占比来看,X-射线晶体衍射技术仍然处于绝对领先地位,随着样品处理以及数据收集等相关技术的发展,冷冻电镜技术比重呈急剧上升的趋势,并于2017年获得诺贝尔化学奖[12]。利用X-射线进行晶体衍射实验时,首先需要获得高纯度的蛋白质,并使用结晶试剂盒培养蛋白质晶体,再利用室内X-射线衍射仪或同步辐射光源对蛋白质晶体进行X-射线衍射。收集衍射图谱,随后根据衍射的方向测定晶格参数或晶胞的大小和形状,根据衍射线强度分布测定原子在晶胞中的坐标,再结合其他方法推断原子排列得到初步结构模型,然后与实验观察值比较修正并获得最终模型。蛋白质晶体的形成和其所处的环境有关,如特定的pH值、温度、蛋白质浓度、沉淀剂的种类以及金属离子和某些蛋白质的配基等,高质量晶体往往需要经过对这些参数进行耗时费力的多轮筛选优化而得,所以如何获得高质量晶体也是蛋白质X-射线晶体学研究的瓶颈[13]。而核磁共振技术需要特殊元素对蛋白质进行标记,其研究的分子量大小受到限制,主要是用来研究小分子,也可以被用来研究相关动力学信息[14]。冷冻电镜技术通过将蛋白质或其他生物分子急速冷冻,并用电子对其轰击,从而生成单个分子的显微图像,它们被用来重建分子的三维形状或结构[8],由于样品制备时使用了瞬时冷冻的技术,冷冻电镜技术可以瞬时捕捉到同个样品在不同状态下的近生理构象,最大可能观察到生物大分子的自然状态,有助于揭示蛋白质如何发挥作用,该方法也无需蛋白质结晶,比较适合研究超大分子量蛋白质复合物甚至亚细胞器的结构,但这项技术也存在局限,如它更适合于大分子量蛋白质以及蛋白质复合体等,目前相关设备的购置及使用维护费用昂贵等。近年来深度学习和人工智能等技术在蛋白质结构预测方面也取得了较大突破,DeepMind公司开发的Alpha fold[15]系列以及华盛顿大学David Baker团队的Rosetta fold[16]等蛋白质结构模拟技术,已经能够比较准确地预测蛋白质的整体结构。但在一些蛋白的关键区域,尤其是结合底物后有较大构象变化的区域,一些预测的模型仍有误差,还需要蛋白质晶体衍射或冷冻电镜等实验技术来进行确证。通过计算获得的这些模型,可以提高没有同源模型蛋白的结构解析速度,加快结构解析效率,并与分子动力学、量子力学计算等技术相交叉结合,将极大地提升蛋白质结构及功能认知的效率[17],实现对蛋白质结构与功能关系的全面分析。
2 利用蛋白质结构生物学技术解析关键酶机制应用实例 近10年来,我们研究团队面向合成生物技术产业化应用的重大需求,以解决合成生物设计构建的基本科学问题为目标,利用蛋白质结构解析技术,聚焦天津工业生物所六大科技计划,积极合作,集中攻关,获得了许多重要的原创性的成果,发表了多篇高水平论文,在蛋白质结构功能的研究领域作出了系统性的贡献。其中主要进行了以下几方面的工作:植物天然产物相关的萜类合成酶、生物质转化利用相关酶、CO2生物固定转化相关酶等领域。
2.1 萜类合成酶相关研究 萜类化合物(terpene) 是自然界中数量最大的化合物,在生物体的生命代谢和功能行使中扮演非常重要的角色。萜类化合物合成与天然产物代谢具有密切关系,许多萜类具有重要的药用价值,如紫杉醇和青蒿素[18]。目前鉴定出的萜类化合物已超过10万种,功能与形态丰富多样,负责合成萜类架构的萜类合成酶是这个领域重要的研究热点。萜类基础架构皆由最小单位的5个碳的异戊烯基二磷酸(isopentenyl diphosphate, IPP) 与二甲基烯丙基二磷酸(dimethylallyl diphosphate, DMAPP),遵循“异戊二烯规则”以“头-碰-尾” “头-碰-头” “头-碰-中”或者其他方式,组合成10个到5 000个以上的碳单元(橡胶) 的各类化合物。根据萜类的碳支架构型以及近10年来解析的各种结构,萜类合成酶可大略分为3大类(图 1):(1) 全α-折叠的反式合成酶;(2) 蝴蝶样折叠的顺式合成酶;(3) 非典型萜类合成酶[19]。深入研究萜类合成酶如何以相同的底物进行各式各样不同的催化反应一直是酶蛋白催化的重要科学问题。近10年来本研究团队,一直致力于多种萜类合成酶结构和功能的研究,面向萜类合成酶如何催化多样反应的重大科学问题,针对以上3种类型萜类合成酶的各种不同反应,如“头-碰-尾” “头-碰-头” “头-碰-中”、环化、特殊反应以及非典型萜类合成酶进行了首次的结构解析以及深入的机理研究,获得了许多成果,在萜类合成酶机理研究领域作出了贡献,也为重要萜类化合物的生物合成及代谢工程设计提供了指导意义[20]。
2.1.1 全α-折叠反式合成酶 由40个碳单元组成的八异戊二烯是细菌中泛醌和维生素K2的侧链脂链,负责电子传递链,具有重要的生理功能。我们解析了海栖热袍菌的八异戊二烯合成酶(Thermotoga maritime, TmOPPS, PDB: 3WJK, 3WJN, 3WJO) 的晶体结构,发现蛋白质采用全α-折叠方式,具有一大一小两个底物结合域,大底物结合区呈长条状,延伸的口袋区内部由非极性氨基酸组成以容纳长链底物进入,底部则由F132封住以精确控制40个碳产物合成,进行了“头-碰-尾”的催化反应。与此同时,我们还解析了来源于大肠杆菌的八异戊二烯合成酶(Escherichia coli, EcOPPS) 与底物的复合体结构,这也是此蛋白质家族的第一个底物复合体结构,完善了长链全α-折叠反式合成酶的催化机理研究[21]。
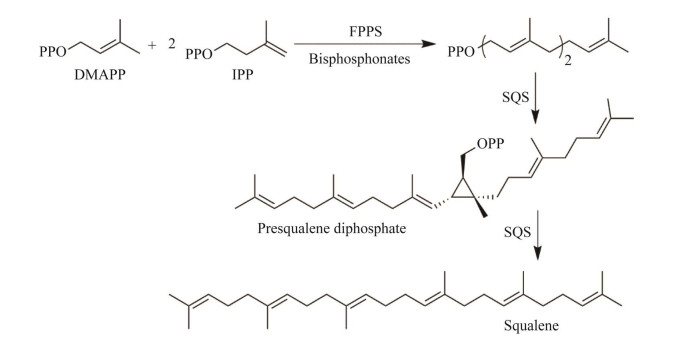
鲨烯合成酶(squalene synthase, SQS) 在真核细胞中催化两个焦磷酸法尼酯(farnesyl pyrophosphate, FPP) 以“头-碰-头”的方式对接合成角鲨烯。角鲨烯为固醇类物质的前体,因此SQS抑制剂可作为降胆固醇药物[22]。来源于克式锥虫的SQS负责合成细胞膜上麦角甾醇的前体物质,亦为新一代抗锥虫药物的重要标靶。本研究团队解析了人源和克氏锥虫的SQS与底物的复合体结构、两个人源SQS与降胆固醇药物(奎宁环) 的复合体结构,以及6个双磷酸药物与人源和克氏锥虫SQS的复合体结构(PDB: 3WC9, 3WCA, 3WCB, 3WCC, 3WCD, 3WCE, 3WCF, 3WCG, 3WCH, 3WCI, 3WCJ, 3WCL, 3WCM),建立了SQS与底物、抑制剂的竞争性结合模型,完整揭示了SQS的活性区结合两个并排的FPP以进行“头-碰-头”反应的催化机制(图 2),为进一步优化以SQS为标靶的药物奠定了重要的基础。抗结核病药物N-金刚烷-2-基-N′-3, 7-二甲基-辛-2, 6-二烯基-乙烷-1, 2-二胺(N-[(2E)-3, 7-dimethyl-2, 6-octadienyl]-N′-tricyclo[3.3.1.13, 7]dec-2-yl-1, 2-ethanediamine, SQ109)[23]可作为抗克氏锥虫的新药,而SQS正是SQ109的靶点之一。通过对克氏锥虫SQS与SQ109复合体的结构解析,发现SQ109同时占据了两条FPP的结合位,阐明了药物的结合作用模式,为设计下一代克氏锥虫SQS特异性药物提供了重要的理论基础[24]。上述相关研究成果被Science、Chemical Reviews等期刊文章引用[25-26]。
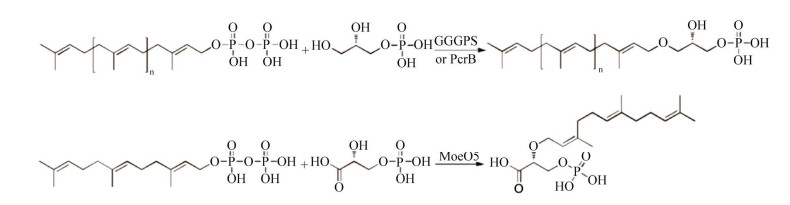
默诺霉素是目前唯一直接抑制细菌肽聚糖糖基转移酶的抗生素,抗菌活性为万古霉素的10–1 000倍。默诺霉素合成步骤中的MoeN5,负责将10个碳的香叶基焦磷酸铵盐(geranylpyrophosphate, GPP) 以“头-碰-中”的方式接合15个碳的3-糖基-2-法尼基磷酸甘油酸酯(2-(Z, E)-farnesyl-3-phosphoglycerate, FPG) 以生成25个碳的产物。由于默诺霉素的碳链长短直接与抗菌活性相关,MoeN5催化反应甚为关键。我们解析了MoeN5及与底物的复合体结构(PDB: 5GWV, 6J8W, 5GWW, 6J8V),揭示了MoeN5具有两个底物结合域。这是第一个以“头-碰-中”的全α-折叠的反式合成酶结构被完整解析出来,催化机理特殊,受到广泛关注[27]。
2.1.2 蝴蝶样折叠的顺式合成酶与环化酶 以顺式-构型对接的碳链长度长且强度大,对于细胞结构支撑非常重要,这类酶形成同源二聚体,整体结构如蝴蝶展翅。细菌特有的十一异戊二烯焦磷酸合成酶(undecaprenyl diphosphate synthase, UPPS) 最为经典,UPPS以“头-碰-尾”的方式反应生成的产物十一异戊二烯焦磷酸(undecaprenyl diphosphate synthase, UPP) 是细胞壁中合成肽聚糖的中间体,因此UPPS可作为开发新型抗生素的热门靶点。针对UPPS的新型抗生素进行开发(PDB: 5ZHE),获得可达到50–100 nmol/L抑制效果的抑制剂,有望进入药物开发的阶段[24]。
来源于薰衣草的薰衣草焦磷酸合成酶(lavandulyl diphosphate synthase, LPPS),可将两个DMAPP分子以“头-碰-中”对接方式生成薰衣草焦磷酸(lavandulyl diphosphate, LPP),经后续修饰产生的各种衍生物为组成薰衣草精油的主要成分。我们解析了LPPS的晶体结构(PDB: 5HC8, 5HC6, 5HC7),该酶为同源二聚体,蛋白质结构采取蝴蝶样折叠,最特别的是LPPS催化反应非常特殊,是目前自然界中首次发现的违反异戊二烯规则的萜类合成酶(异戊二烯规则:由IPP和DMAPP反应生成各种不同的萜类化合物,但LPPS只需DMAPP即可反应,完全违反异戊二烯规则)。透过高分辨率的LPPS与镁离子和DMAPP的复合体结构完整解析,LPPS为蝴蝶样折叠同源二聚体,其活性区具有两个小口袋,分别可容纳一个C5底物,只有两个DMAPP分子可以稳定存在于这两个口袋中,进而发生缩合反应,反应中负责攻击第一个DMAPP双磷酸基团的78位组氨酸是催化反应发生的关键位点。进一步研究发现LPPS与UPPS结构相似,但活性中心两个底物结合区皆为只能容纳5个碳的小口袋,这是首个违反异戊二烯规则的萜类合成酶[28] (VIP重点封面文章)。
针对“头-碰-中”蝴蝶样折叠的顺式合成酶进一步地深入研究,我们解析了具有不同底物结合特征的新型“头-碰-中”抗生素merochlorin的重要前体关键酶Mcl22和其底物的复合体结构(PDB: 5XK9, 5XK8, 5XK6, 5XK7, 5XK3)。以结构为基础的活性位点改造,完整阐明了作用机理[29] (图 3) (封面文章)。与Mcl22同样来源于链霉菌的衣草花青菌素前体合成酶CLDS,可将两分子的DMAPP环化成二磷酸黄烷基酯(lavandulyl diphosphate, LDP),进而生成环烷基二磷酸酯(cyclolavandulyl diphosphate, CLDP),CLDS与UPPS同为顺式构型,且氨基酸序列高度保守,但CLDS的环化功能在顺式合成酶中却是独一无二的。我们解析了CLDS的蛋白质结构及其与底物类似物的复合体结构(PDB: 5YGJ和5YGK),阐明了该酶的催化反应机理[30],为衣草花青菌素的生物合成提供了理论基础。
结核分枝杆菌(Mycobacterium tuberculosis, Mtb) 是最重要的人类传染疾病病原之一,逐年恶化的抗药性问题更加重了疾病防治的困难程度。具有致病力的Mtb中带有一个特殊的二萜合成酶Rv3378c,是Mtb感染宿主细胞不可或缺的致病因子,更是开发结核杆菌抗生素的新标靶。我们解析了Rv3378c和底物的复合体结构(PDB: 4KT8, 3WQK, 3WQN, 3WQM, 3WQL),发现Rv3378c采用与UPPS相似的蝴蝶样折叠,这也是首次发现二萜合成酶可釆用蝴蝶样折叠的结构。据此特性,我们提出了使用UPPS的抑制剂来抑制Rv3378c (具潜力的抗结核药物靶点),并研究证明了“一药多靶”的策略是可行的,同时解析了药物与Mtb两种酶的复合体结构,系统阐述了药物对不同酶的结合模型[31]。
2.1.3 非典型萜类合成酶 TIM-barrel结构的顺反式萜类合成酶:我们报道了3个此家族成员的晶体结构,负责默诺霉素生物合成第一步的关键酶MoeO5,以及来源于枯草芽孢杆菌和金黄色葡萄球菌的两个PcrB酶蛋白。MoeO5负责催化FPP的15碳单位以顺式构型转移到3-磷酸甘油酸的2位羟基上。PcrB则是催化七异戊二烯焦磷酸的35碳单位以反式构型转移到1-磷酸甘油酸的3位羟基上。同属TIM-barrel折叠的MoeO5与PcrB却可催化顺式与反式两种相反构型的反应,这也颠覆了科学界对萜类合成酶的经典认知。比对这两种酶的活性区发现,MoeO5活性区多了4个氨基酸而呈关闭状态,有利于顺式产物的形成,而PcrB的相应区域则形成一个较长口袋,利于容纳反式产物[32-33] (PDB: 3W00, 3VZZ, 3W01, 3VZX, 3W02, 3VZY) (图 4) (封面文章)。
Rossman fold的环烯醚萜合成酶:来源于药用植物长春花的环烯醚萜合成酶(iridoid synthase, IRIS) 可以利用之前从未在萜类化合物合成中报道过的麦克尔加成反应,催化十碳的线性单萜底物生成双环单萜nepetalactol[8, 34]。我们解析了IRIS与辅酶和底物的复合体结构(PDB: 5DBI, 5DBG, 5DBF),首次揭示了底物的结合模式和环烯醚萜合成酶的催化机理[35],该成果获选为当期的重点封面文章。通过以上相关研究,我们系统地揭示了一些重要萜类合成酶的催化反应机理,解释了萜类合成酶如何催化多样反应的科学问题,获得了许多重要的原创性的成果,在整个萜类合成酶机理研究领域作出了系统性的贡献。随后,O’Connor教授和我们相同的蛋白结构文章发表于Nature Chemical Biology,证明了科学界对此结构的高度关注[36],文章后续也被Chemical Reviews等文章引用[37]。
2.2 生物质转化利用相关酶 我国是农业大国,各类农作物秸秆资源十分丰富,秸秆中的天然聚合物如纤维素和木质素占很大的比例,为地球上最丰富的可再生资源。纤维素是葡萄糖的聚合物,由于结构特殊,因此有抵抗各种氧化剂的能力,只能被浓酸水解。近年来各国竞相开发生物质能源作为替代能源,寻找性质突出的纤维素酶及木质素酶为关键技术之一。除用于开发生物质能源之外,这些酶也被广泛应用在食品加工、动物饲料、废水处理、纺织、造纸等各个领域,具有重要的经济价值。多年来,天津工业生物所结构生物学平台进行了多种纤维素、半纤维素和木质素降解相关酶的研究工作,解析了多个新型酶的结构,探讨酶对于不同温度耐受性的机理与底物结合的特性。通过解析纤维素酶及半纤维素酶的晶体结构,尤其是首次解析了第120、127、131和165家族的纤维素酶、果胶酶的结构,阐明了催化反应机理,提供了针对这些酶家族进行理性设计时需要的结构基础。同时,侧重于提高工业应用中最重要的酶活力以及耐热性,开展了基于结构的酶工程改造,解决了该领域遇到的部分重大问题。
2.2.1 纤维素降解相关酶 在纤维素酶系中,内切葡聚糖酶(endo-1, 4-β-d-glucanohydrolase, EC 3.2.1.4) 是主要成分之一,它包含多种同工酶,归结在以前的Cx分类中,可以将可溶性纤维素水解成还原性的寡糖。我们对多个不同来源的葡聚糖酶进行了较系统的相关研究,来源于太瑞斯梭孢壳霉的纤维素酶12A (Theilavia terrestris, TmCel12A) 是一个高温酶,我们解析了该酶的高活性突变体以及突变体和寡糖的共晶结构(PDB: 3VHN, 3VHO, 3VHP),并针对活性区及柔性区进行了改造,获得了一些性能提升的突变子,为该酶的改造提供了理论依据[38]。葡聚糖酶被用于饲料中帮助食草动物消化植物纤维,我们解析了来源于琥珀酸丝状杆菌(Fibrobacter succinogenes) 的葡聚糖酶突变体(PDB: 3AXD) 以及突变体和纤维四糖的共晶结构(PDB: 3AXE),以此为基础的改造提升了酶活力以及热稳定性,并通过优化条件实现了在毕赤酵母中高通量表达[39]。此外,我们还解析了来源于嗜热真菌的GH 45家族的内切葡聚糖酶(Theilavia terrestris, TtCel45A) 的蛋白质结构以及和纤维二糖以及纤维四糖的复合物结构(PDB: 5GLX, 5GM9, 5GLY)[40];来源于黑曲霉的内切葡聚糖酶(Aspergillus niger, AnCel5A) 的野生型、突变体以及突变体与纤维四糖的复合物结构(PDB: 5I77, 5I78, 5I79);来源于灵芝的内切葡聚糖酶(Ganoderma lucidum, GlCel5A) 的蛋白质结构(PDB: 5D8W, 5D8Z);来源于热纤梭菌的GH16家族的β-1, 3-1, 4-内切葡聚糖酶(Clostridium thermocellum, CtLic16AA) 的蛋白质结构(PDB: 3WVJ),了解其催化机制,并在毕赤酵母里面进行了表达优化[41]。来源于嗜热拟青霉的葡聚糖酶(Paecilomyces thermophila, PtLic16A),我们通过解析其野生型以及突变体的晶体结构(PDB: 3WDT, 3WDU, 3WDV, 3WDW, 3WDX, 3WDY),揭示其催化机制及热稳定原因的机理[42]。来源于海栖热袍菌的超高温内切葡聚糖酶(Thermotoga maritima, TmCel12A),我们获得了突变体的晶体结构(PDB: 3VHN, 3VHO, 3VHP),并在其结构表面发现一个特殊的柔性区loop,通过对loop的截短进一步提升了酶活力[38]。同样来源于海栖热袍菌的超高温1, 4-内切葡聚糖酶(Thermotoga maritima, TmCel5A),通过解析其突变体的晶体结构(PDB: 3AMC, 3AMD, 3AZR, 3AZS, 3AZT, 3AMG, 3AOF)[43]及来源于棘孢曲霉的内切葡聚糖酶(Aspergillus aculeatus F-50, FI-CMCase)[44]的野生型、突变体以及突变体和纤维四糖的复合物晶体结构(PDB: 5GM3, 5GM4, 5GM5),我们阐明了催化机制,并进行了突变,提高了催化反应活性。来源于热纤梭菌的双功能纤维素酶/木质素酶(Clostridium thermocellum, CtCel5E),我们解析了其野生型以及突变体的晶体结构(PDB: 4U3A, 4U5I, 4U5K),了解了其催化机制,并进行了突变找到了活性提升的突变子[45]。上述的研究成果,为后续的工业化应用改造提供了有力支撑。
2.2.2 木质素降解相关酶 木质素是植物细胞壁中含量最丰富的半纤维素杂多糖,在用作生物燃料基质和其他原料之前,必须先被有效地分解为组成糖[46]。木聚糖1, 4-β-木糖苷酶(1, 4-β-xylosidase) 作用于低聚木糖的非还原端,将其水解为单木糖单元,来源于热解糖梭菌的木聚糖-1, 4-β-木糖苷酶(Thermoanaerobacterium saccharolyticum JW/SL- YS485, XylC) 是首个被归为CAZy数据库中(http://www.cazy.org/) GH120家族中的糖基水解酶。我们培养获得了XylC的高质量晶体[47],并解析了XylC以及该酶和木聚糖以及木二糖的晶体结构(PDB: 3VST, 3VSU, 3VSV),找到结构上的一些区域,并进行了改造获得了性能提升的突变子[48]。来源于同样热解糖梭菌中的木聚糖酶(Thermoanaerobacterium saccharolyticum JW/SL-YS485, TsXylA),属于GH10家族,通过解析野生型TsXylA及其突变体E146A、E251A和木聚二糖,木聚四糖的复合物结构(PDB: 3W24, 3W25, 3W26, 3W27, 3W28, 3W29),获知了相应底物和酶的实际结合情况,为后续进一步改造提供了理论依据。嗜冷微生物来源的GH10家族的耐低温木聚糖酶(xylanase, XynGr40),通过解析了该酶和木二糖的晶体结构(PDB: 5AY7, 5D4Y),我们发现了该酶在低温环境下维持较高活性的可能机制,即额外的两个loop区域,并进行了突变验证[49]。来源于链霉菌的GH10家族的木聚糖酶(Streptomyces sp. 9, XynAS9),有较广泛的pH以及温度耐受范围,对其进行突变后,获得了一些比较好的结果,在此基础上,我们解析了野生型和突变体的空结构,以及其分别和木二糖的复合体结构(PDB: 3WUB, 3WUE, 3WUF, 3WUG),并进行了改造,获得了活性提升的突变子[50]。来源于瘤胃厌氧真菌的GH11家族的嗜热木聚糖酶(Neocallimastix patriciarum, XynCDBFV),能在较高温度下进行催化反应,我们解析了该酶野生型、突变体以及突变体复合物的晶体结构(PDB: 3WP4, 3WP5, 3WP6)[51]。在这些木质素降解相关酶的基础上,研究团队也对国内外相关酶的相关研究进行了整理分析[52],为日后的工业化应用改造指引了新方向。
2.2.3 人造高分子聚合物降解相关酶 人造高分子聚合物的生产,给人类的生活带来了许多便利,但相关高分子聚合物的大量使用却成为了主要的环境污染源。发展对环境友善的生物处理方案,让这些人造聚合物通过生物降解回到碳循环体系,不但可解决塑料污染的问题,更有可能实现高分子原料回收再利用。聚乙烯醇(polyvinyl alcohol, PVA) 有较好的性能,并在纤维和造纸等领域有广泛应用,但大量使用对环境造成了危害,酶法生物降解是环境友好的潜在降解方法。通过对两个不同来源的鞘氨醇盒菌(Sphingopyxis sp. 113P3) pOPH以及假单胞菌(Pseudomonas sp. VM15C) sOPH的PVA降解酶进行研究,我们解析了pOPH以及突变子的复合物结构(PDB: 3WL6, 3WL7, 3WL8),同时还解析了sOPH的晶体结构(PDB: 3WL9),通过比较两种酶的结构,找到了这类酶的催化机制,以及可能影响催化反应位点和区域,并进行了突变验证[53],(PDB: 3WWC, 3WWD, 3WWE),为聚乙烯醇的酶法降解提供了思路。
2.3 甲醛裂解酶及羧酸酶 二氧化碳(CO2) 是参与自然界“CO2-有机碳-碳酸盐”循环的组分,其正常循环是全球生态环境平衡的关键因素。然而,近年来化石能源大量消耗导致了大气中CO2浓度激增,CO2的过量排放已经造成了一系列严重的环境、社会和经济问题,因此,如何高效、绿色、经济地封存和利用CO2成为了各国研究和开发的热点[54]。为了实现减少我们对化石资源的依赖和转向可持续的生物炼制的目标,必须找到能够维持化学品和燃料大规模生产的替代原料储备。二氧化碳、一氧化碳、四氧化碳、三氧化碳和六氧化碳,是具有吸引力的长期原料,可维持每年数亿吨的燃料和精细化学品的生产[55]。
碳一(C1) 分子中,甲醛是一种特别通用的反应性化合物,为生产增值化学品提供了巨大的前景[56]。甲醛在甲醛裂合酶(benzoylformate decarboxylase, BFD) 的作用下生产1, 3-二羟基丙酮(dihydroxyacetone, Dha),我们解析了来源于恶臭假单胞菌(Pseudomonas putida) 的甲醛裂合酶BFD-M3和BFD-M6的蛋白质结构(PDB: 6M2Z, 6M2Y),揭示了影响底物结合与促进Dha产生的活性中心的结合的有益位点,为甲醛转化为乳酸的两步化学酶法奠定了重要基础[57]。
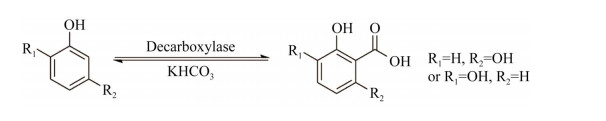
利用化学方法固定工业排放的二氧化碳,往往需要较高的反应温度,消耗大量的热能。因而利用生物方法在温和的条件下固定二氧化碳已逐渐成为人们研究的热点。芳香羧酸脱羧酶是可以降解芳香酸生成芳香烃(主要是苯酚类化合物) 和二氧化碳的一类酶,反应可以在常温和常压下进行,且不需要加入腺嘌呤核苷三磷酸(adenosine triphosphate, ATP) 及辅因子。同时其可以催化苯酚类化合物与二氧化碳反应生成有应用价值的芳香羧酸。通过与天津工业生物所朱敦明研究员、吴洽庆研究员以及天津科技大学路福平教授合作,通过研究不同来源脱羧酶对水杨酸衍生物的催化活性(PDB: 6M53、7BP1, 7BPC),拓展了对这些芳香羧酸脱酸酶的底物特异性的认识,为进一步研究脱羧反应的机理,以及利用生物法固定二氧化碳得到有价值的芳香羧酸提供了重要支撑[58-59] (图 5)。
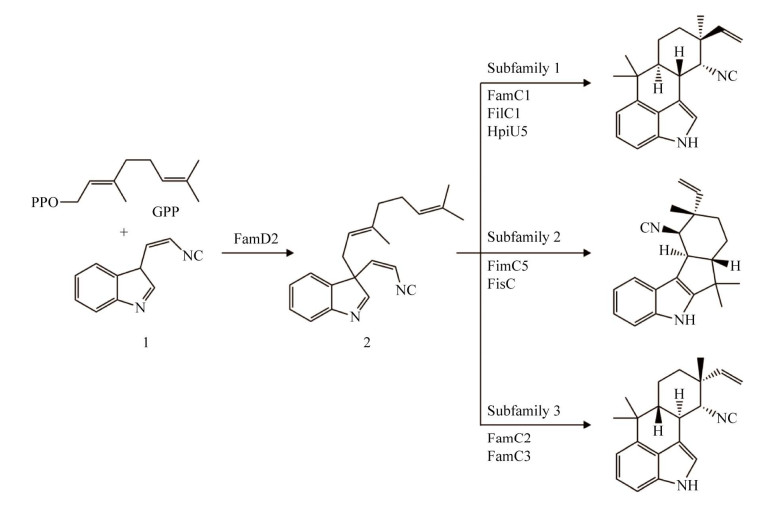
2.4 生物碱生物合成相关酶 Hapalindole类生物碱作为一种蓝藻生物碱,具有重要的抗菌、抗癌作用以及杀虫作用,而且还具有复杂的手性中心和立体异构特性。但是,它这种独特的多环结构使其化学合成尤为困难。因此,寻找hapalindole类生物碱新的生物合成路径具有非常重要的意义。虽然目前真枝藻目环化酶的结构和功能研究较少,但是stig环化酶作为科普重排反应发生的一类新型催化剂,它们在hapalindole和fischerindole吲哚生物碱合成中发挥了重要的环化作用,而且它们与已知结构化合物的序列相似性很低,因此,结构信息对深入了解这类stig环化酶的作用机制是极其重要(图 6)。我们第一次获得了第一步反应中的酶-FamD的结构(PDB: 5YNT, 5YNU, 5YNV, 5YNW),并解释了该步催化机理[60],我们随后也解析了第二步反应的酶:FamC1、FilC1、HpiU5、FilC1/CI-4, HpiU5/CI-4的5种蛋白质结构、突变体结构及复合体的结构(PDB:5YVK, 5YVL, 5YVP, 5Z53, 5Z54, 5ZFJ, 6A8X, 6A92,6A98, 6A99, 6A9F, 6ADU, 6J03)。它们像非催化碳水化合物结合基序一样采用β-三明治折叠。此外,研究中还确定了几种基于吲哚的化合物的复杂结构,这些化合物与疏水性末端腔结合,在其中保守的天冬氨酸(aspartic acid) Asp残基与吲哚N形成H键并引发酸催化的cope重排。通过分析酶与配体的相互作用和诱变实验,发现催化过程中重要的氨基酸及催化反应对Ca2+的依赖性。除了常见的底物结合方式和催化机理外,还涉及可能归因于不同产品特异性的潜在亚家族变异。这些研究结果为其他真枝藻目环化酶的研究提供了重要的理论指导,在hapalindole生物碱生物合成中也具有重要的意义[61-62]。
2.5 糖基转移异构相关酶 糖基转移酶在生物体内催化活化的糖连接到不同的受体分子,如蛋白、核酸、寡糖、脂和小分子上,糖基化的产物具有很多生物学功能[63]。葡萄糖基转移酶是在酶反应中只转移葡萄糖基的酶,葡萄糖苷转移酶是转移时连葡萄糖的糖苷键一起转移的酶。糖基转移酶起着重要作用,糖基化改变了植物中三萜化合物的一些理化性质和生物活性。植物糖基转移酶催化这种糖基化反应,是制备结构多样、价值高的三萜苷类化合物的关键步骤。我们与天津工业生物所孙媛霞研究员和孙周通研究员合作,解析了来源于三萜罗汉果的糖基转移酶中甜苷V生物合成途径中关键酶基因(Siraitia grosvenorii, UGT74ac1) 的蛋白质结构,复合体结构及突变体结构(PDB: 6L90, 6L8Z, 6L8X, 6L8W),并以此作为基础来实现定向进化和基于序列/结构的工程,由此产生的几种尿苷二磷酸糖基转移酶(UDP glycosyltransferases, UGTS) 突变体对三萜糖基化的催化效率提高了近百倍,为其他植物糖类化合物在合成有价值的三萜皂苷类化合物中的实际应用奠定了基础[64]。d-阿洛酮糖-3-差向异构酶(d-allulose-3-epimerase, DPEase) 在d-阿洛酮糖的生物合成中起重要的作用,来源于解纤梭菌(Clostridium cellulolyticum H10) 的DPEase,通过解析该酶的蛋白质结构以及和多个底物类似物复合体的晶体结构(PDB: 3VNI, 3VNK, 3VNJ, 3VNL, 3VNM),并且对相关区域进行了改造,获得了性能提升的突变子[65],该成果为糖基转移酶的定向改造提供了可借鉴的理论依据。
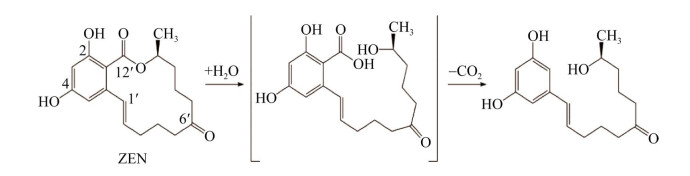
2.6 玉米赤霉烯酮毒素水解酶 霉菌毒素是一种来源于真菌的低分子量的次生代谢物,主要由镰刀菌、青霉菌和曲霉菌产生[66]。霉菌毒素严重影响了动物的生产繁殖,对畜牧业造成巨大的经济损失[67]。以高特异性的霉菌毒素降解酶除去食物中的毒素是最为理想的解毒方式。玉米赤霉烯酮(zearalenone, ZEN) 是一种由镰刀菌属真菌分泌的类雌激素霉菌毒素,最初从发霉玉米中分离得到,是目前全球污染最严重的3种霉菌毒素之一,给种植业和养殖业带来巨大的损失,进而引起人类雌激素相关的疾病,造成严重的经济损失和公共安全问题。人们已经针对玉米赤霉烯酮开发出一系列常用的物理和化学脱毒法,这些方法常会带来脱毒不彻底、引入其他毒性物质以及营养物质流失等问题,生物法脱毒专一性强,脱毒相对彻底,是目前广受关注的霉菌毒素降解方法(图 7)。
研究团队经过数年的研究,在玉米赤霉烯酮降解酶(zearalenone degradase, ZHD) 的蛋白结构和分子改造方面取得重要进展。2014年团队首次解析了来源于粉红粘帚菌的玉米赤霉烯酮水解酶(Clonostachys rosea, ZHD101) 的结构及其突变体ZHD-S102A与底物ZEN的复合体结构(PDB: 3WZL, 3WZM),揭示了该酶的催化反应机理,并根据ZHD的结构进一步进行了氨基酸定点突变的研究,验证了这些与底物相互作用关键氨基酸的重要功能[68],相关研究被《中国科学报》报道。为了提高ZHD101对ZOL (zearalenol) 的水解活力,研究团队进一步解析了ZHD101与ZOL底物的复合体结构(PDB: 5IE4, 5IE5, 5IE6, 5IE7, 2XUA)。之后针对此酶进行改造,获得能分解毒力强96倍的α-ZOL (α-zearalenol) 的高活性突变体,经结构解析,首次揭示了改变催化三联体而能提高酶活的机理[69] (封面文章)。为了开发高效的ZEN生物降解的方法,研究团队挖掘并解析出一种来源于麦氏喙枝孢霉的ZEN降解新酶(Ramichloridium mackenziei, RmZHD) 的蛋白质结构其底物ZEN和α-ZOL的复合体结构(PDB: 5XO6, 5XO7, 5XO8, 5Z5J, 5Z7J, 5Z97)。针对底物ZEN和α-ZOL,该酶降解活力分别是ZHD101的1.66倍和1.20倍,具有更高的应用价值。基于结构信息的分子改造大大提高了RmZHD对ZEN高毒性衍生物α-ZOL的活性。该研究结果为ZHD家族酶的催化机理和底物结合模式提供了深入的认识,对RmZHD在饲料工业的应用,实现玉米赤霉烯酮及其衍生物的完全脱毒具有重要的意义[70]。
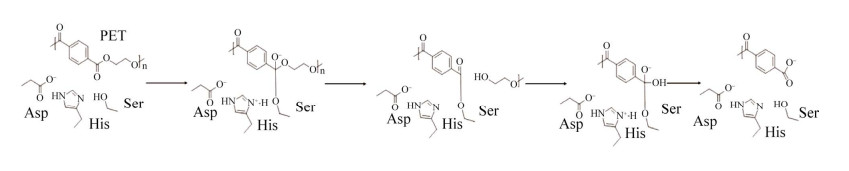
2.7 PET塑料降解酶 聚对苯二甲酸乙二酯(polyethylene terephthalate, PET) 塑料已占全球聚合物总量的18%,废弃后在自然界中极难降解,是白色污染的重要来源[71],使用生物酶法对塑料废弃物进行降解回收利用,是目前的重要研究热点之一[72]。本平台实验室近年来利用X-射线晶体衍射技术对PET塑料降解酶进行结构解析和机理研究,并对其进行理性、半理性设计改进其效能,增强它们的应用潜能。日本的研究团队于2016年报道了一种可以“吃塑料”的神奇细菌(Ideonella sakaiensis)[73-74],这株细菌能够附着在PET表面,分泌一种新型的PET水解酶将PET降解成小片段,再将降解后的产物转运入体内进一步“消化”,最终转化为乙二醇和对苯二甲酸。我们解析了首个塑料降解酶IsPETase与其底物/产物类似物的复合体结构,发现新型PET水解酶与先前报道的其他低酶活的类PET水解酶在活性区的两个结构特征明显不同[75]。这项研究阐明了PET水解酶的降解机理,为后续其他许多团队的相关研究奠定了基础,并对理解PET塑料的生物降解过程,尽快实现PET水解酶工业化应用具有重要指导意义。FEBS Journal针对本研究邀请我们在“Structural Snapshots”专栏撰稿[76],ChemistryViews于2017年12月14日以“Enzyme Breaks Down Plastics”为题对本研究进行了专题报道,《科技日报》于2018年2月7日在头版报道,《X-MOL》《中国科学报》《天津日报》《财新传媒》等也针对本研究进行了专门的报道,文章至今已被引用160余次。利用所获得的蛋白晶体结构信息,我们进一步和国内外多个研究团队合作,对PET降解酶进行进一步理性设计和定向进化改造,并对关键突变子进行结构和机制解析,阐明了提升这些酶活性和耐热性关键位点的催化机制(图 8)。至今已经获得了数十个塑料降解酶的晶体结构(PDB: 5XG0, 5XFY, 5XFZ, 5XH3, 5XH2, 6KY5, 7CWQ, 7CUV, 7E30, 7E31, 7VVC, 7VVE, 7W1N, 7W44, 7W45, 7W66, 7W69, 7W6C, 7W6O, 7W6Q等)。获得性能提升的PET降解酶,为加快PET塑料降解酶在环境保护上的应用及产业化提供了理论支撑[77-80]。
3 总结与展望 合成生物学为代表的新一代生物技术正在引领新一轮科技革命和产业变革,为促进我国经济高质量发展作出贡献。合成生物技术的发展对各种生物催化过程中蛋白质的性能提出了更高的要求,需要能快速、高效制备出高纯度蛋白质,并且通过高通量结构解析和性质研究深入了解关键蛋白质的结构、生物学功能及生理特性,并指导相关设计过程。作为合成生物技术创新中心的一个公共服务平台,中国科学院天津工业生物所结构生物学平台经过近10年来的发展,建立起蛋白质表达制备,结晶条件筛选、优化,晶体衍射数据收集解析及蛋白性质鉴定等一系列的技术体系,并对许多关键工业蛋白开展了结构与功能研究,充分发挥了对工业菌种相关的蛋白质结构及功能研究的支撑作用。在中国科学院、天津市以及天津工业生物所的大力支持下,在现有部分相关装备的基础上,平台将升级更高通量、更高灵敏度、更高精度的设备:包括高通量蛋白质制备系统,如高流速蛋白纯化系统,工艺型蛋白纯化系统,离心浓缩使用的切向流过滤系统;蛋白性质鉴定系统,如蛋白质稳定分析仪、高效率等温滴定量热仪(测定蛋白和小分子相互作用热力学参数) 等装备及结构生物学系统,如全自动结晶筛选系统、结晶优化系统,自动化蛋白晶体显微成像系统,结构解析工作站,冷冻电镜样品制备、判断以及初步数据收集等装备。进一步提高相关蛋白质研究的整体通量,形成一个比较完整的从蛋白质样品制备、性质鉴定以及结构功能研究的完整装备体系,并提升蛋白质相关技术的研究和服务能力,建立引领国内外的蛋白结构和功能研究的技术创新平台,为合成生物学与生物制造尤其是高效菌种的创制提供强有力的技术支撑。
 2022, Vol. 38
2022, Vol. 38