数字细胞模型的研究及应用
袁倩倩
,
毛志涛
,
杨雪
,
廖小平
,
马红武
生物工程学报  2022, Vol. 38 2022, Vol. 38 Issue (11): 4146-4161 Issue (11): 4146-4161 |

基因组学、转录组学和蛋白组学等一系列组学分析技术的发展大大提高了生物数据的获取能力,使人们可以更全面系统地从整体角度对生物系统进行观察分析。但至今即使针对简单的原核单细胞模式生物如大肠杆菌等,要系统地理解其细胞行为,准确预测各种环境和基因扰动对其功能的影响仍然非常困难。最主要的原因就是细胞是一个成千上万种不同分子相互作用构成的复杂系统,每一种功能表型都是细胞整体中各种类型分子综合相互作用的结果,而组学数据即使是覆盖全基因组也仅仅是在有限的条件下(如不同生长阶段) 针对某一分子层次(如转录组针对mRNA表达水平、蛋白组针对蛋白水平、代谢组则针对代谢物浓度) 进行分析的结果,数据难以对相关相互作用机理进行全覆盖。另一方面,目前生物学还主要是以观察分析为主要研究手段,没能像物理化学一样形成原理模型主导的研究模式,大部分生物学家都缺少数据分析建模的训练,即使针对单一数据,其数据量之大也超出了传统生物研究方法的处理能力。各种生物信息学方法的开发显著提升了人们对生物大数据进行分析处理的能力,但更多侧重在特定生物组件功能注释解析和机理分析,缺少对细胞从整体角度的系统组织原理分析,而这正是数字细胞模型要解决的难题。数字细胞研究目标就是基于各类生物数据,对细胞内各种代谢和调控过程进行数学方程描述,从而建立涵盖各类相互作用,能准确描述实际细胞行为的全细胞模型,进而在模型指导下揭示细胞系统层次的组织原理,并指导设计具有期望功能的人工生物系统。数字细胞研究是以合成生物学为代表的新生物学的核心研究内容。合成生物学特别强调对生物体以工程化的方式重新设计,以创建某些方面超越自然功能的人工合成生物。通过造物致知,其不仅对揭示生命基本组织规律,探索生命本质和起源具有重要意义,而且创建的具有特定性能的人工生命具有无限的应用潜力,可以极大促进生物技术和绿色生物经济的发展,为破解人类面临的资源、健康、环境和安全等重大挑战提供全新解决方案[1]。
细胞内不同生物分子间通过各种复杂相互作用构成的生物网络决定了整体细胞的功能。因此,对生物网络进行建模分析是数字细胞研究的重要内容,其中尤以对基因组尺度代谢网络模型(genome-scale metabolic network model, GEM) 的研究最为深入,方法最成熟,并已在合成生物学细胞改造中获得广泛应用[2-4]。代谢网络模型构建主要基于基因组中的酶蛋白注释信息,通过酶催化反应将各种代谢物连成一个可互相转化的复杂网络。因此其构建相对容易,且因为其中包括了基因-蛋白(酶) 和代谢物不同分子层次,使得不同类的组学数据都可以在代谢网络上进行整合分析。同时其与工业生物技术中新生物转化途径的设计构建和代谢工程菌种改造密切相关,具有重要的实际应用价值。但生物网络模型中更侧重不同分子间的相互作用关系而不是这些相互作用导致的细胞动态变化过程的描述,因此更适合对细胞在不同条件下达到稳态的模拟而难以描述细胞的动态调控过程。近年来人们从不同角度对生物网络模型进行扩展,通过整合其他生物信息和数据(如与转录组或蛋白组数据结合[5-6],整合热力学和酶约束等[7-8]),纳入更多生物过程(如转录、翻译等)[9-10],以建立更全面准确的数学模型以对细胞的动态变化行为进行更精准地模拟。数字细胞模型是传统生物网络模型的延伸发展,其涵盖了生物网络模型,同时强调涵盖更多生物过程和细胞动态行为模拟,在未来的新生物学研究中将发挥越来越重要的作用。
中国科学院天津工业生物技术研究所(以下简称天津工业生物所) 建所10年来,在基因组尺度代谢网络模型研究以及更全面的数字细胞模型研究方面做了大量的研究工作。针对一些重要工业菌种构建了高质量代谢网络模型,开发了模型分析和途径设计的新方法并应用于指导菌种改造和人工固碳新途径的设计构建,进一步提出了多约束细胞模型的构建框架和流程,建立了整合热力学和酶约束的细胞模型,显著提高了模型预测准确度。下面将对天津工业生物所在细胞模型方面研究取得的重要进展进行介绍。
1 基因组尺度代谢网络模型的构建及质控由基因组序列出发,构建细胞全局代谢网络模型已成为一种对细胞生理特性进行模拟预测的有力工具。利用GEMs可以从全局代谢角度寻找新的代谢途径、代谢工程改造策略及进行菌体生长表型预测等[11]。近年来,天津工业生物所构建了包括原核恶臭假单胞菌[12]、希瓦氏菌[13]、真核噬热毁丝酶[14]、解脂耶氏酵母[15]和氨氧化古菌[16]在内的多个GEMs (表 1)。同时,为了提升模型预测正确性,天津工业生物所在模型质量控制和检测方面做了深入研究,利用修正的高质量模型进行了代谢途径和改造策略等多种设计和应用。
| Strain | Type | Model ID | Genes | Reactions | Metabolites | References | Year |
| P. putida KT2440 | Bacteria | PpuQY1140 | 1 140 | 1 171 | 1 104 | [12] | 2017 |
| N. maritimus SCM1 | Archaea | NmrFL413 | 413 | 765 | 825 | [16] | 2018 |
| S. oneidensis MR-1 | Bacteria | iLJ1162 | 1 162 | 2 084 | 1 818 | [13] | 2022 |
| M. thermophila | Fungi | iDL1450 | 1 450 | 2 592 | 1 784 | [14] | 2022 |
| Y. lipolytica W29 | Fungi | iYli21 | 1 058 | 2 285 | 1 868 | [15] | 2022 |
GEMs构建是一个基于基因组功能注释确定基因-蛋白(酶)-反应关系并基于细胞生长代谢表型数据添加反应、修正错误的过程。2010年,Thiele等[17]在Nature Protocols发表了一篇指导GEMs的构建指南,将GEM模型构建过程分解为96个步骤并进行详细讲解,是指导GEMs构建的全面指南。2019年,Heirendt等[18]在Nature Protocols发表了针对模型构建和优化的COBRA v3.0工具包,其中包含了软件安装、模型导入导出、反应添加删除和模型缺口填补等103个实操步骤,是对96步模型构建指南的良好补充。基于该指南和COBRA工具包,可以从头构建特定菌株的GEM。尽管发表的模型构建指南为GEMs构建列出了明确的步骤,但是其中很多步骤需要人工进行仍然费时费力。为了解决此问题,研究者开发了多个模型自动化构建工具。其中,Seaver等[19]构建了目前唯一的模型在线构建平台ModelSEED。Agren等[20]开发了集模型构建、分析及可视化于一体的本地化工具箱RAVEN。相较于ModelSEED,RAVEN可以构建真核菌株代谢网络模型。Dias等[21]开发了基于Java平台的用户友好的模型构建平台Merlin,目前已用于肺炎链球菌、白色念珠菌等菌株模型构建[22-23]。Merlin于近期进行了工具的更新[24],模型构建能力有所提升。
基于上述模型构建流程,天津工业生物所构建了包括古菌、真核和原核在内的多个菌株的GEMs。例如,天津工业生物所对海洋氨氧化古菌(Nitrosopumilus maritimus SCM1) 代谢网络模型进行了从头构建[16],利用构建的模型NmrFL413计算ATP/NH4+产率,表明SCM1的能量生产效率较低,结果表明海洋氨氧化古菌对碳循环的贡献较小,而对海洋氮循环的贡献显著。天津工业生物所最近还构建了解脂耶氏酵母的代谢网络模型iYli21[15],预测营养物质利用达到了85.7%的高准确率,并与转录组学数据整合分析了6种代谢物作为唯一碳源的细胞代谢,分析了在不同底物下参与代谢的重要途径,该模型为解脂耶氏酵母研究提供了高质量工具。基于RAVEN构建了真核嗜热毁丝霉的第一个代谢网络模型iDL1450[14],在模型中引入转录组学数据,研究并解释了嗜热菌在不同生长温度下的代谢特征。近期,天津工业生物所基于ModelSEED流程,结合缺口填补等一系列模型修正流程,构建了希瓦氏菌基因组尺度代谢网络模型iLJ1162[13]。预测了可以提高希瓦氏菌产电量的改造靶点以及两种潜在的电子转移新途径,模拟微生物电合成系统中基于MR-1底盘的6种平台化学品的最优生物合成途径。
国内也有其他团队开展了代谢网络模型构建和应用的相关工作。江南大学刘立明团队围绕模型构建和模型指导实验室菌株改造进行了大量系统研究。对裂殖壶菌SR21[25]、高山被孢霉[26]和谷氨酸棒杆菌[27]等多个菌株进行了模型构建,并且利用模型预测结果指导了DHA、花生四烯酸和胞外多糖等产品的代谢工程改造,为理性代谢工程改造提供了范例。中国科学院微生物研究所温廷益课题组[28]参考96步模型构建流程和已发表的大肠杆菌模型,构建了谷氨酸棒杆菌iCW773模型,该模型是目前谷氨酸棒杆菌质量最高的模型。华东理工大学花强课题组[29]构建了解脂耶氏酵母基因组规模的代谢模型,为产油酵母生产有价值产品提供了有力工具。天津大学闻建平课题组构建了筑波链霉菌[30]、吸水链霉菌[31]等真核菌株的GEMs,并指导提高抗菌药物提高雷帕霉素产量。
尽管已发表了指导GEMs构建的流程,也开发了多种模型构建工具,但模型的质量仍良莠不齐。与国内其他研究组不同的是,天津工业生物所多年来注重模型质量的提高、模型质控流程的搭建,以构建高质量代谢网络模型为目标,开发模型构建和质控流程。天津工业生物所前期对4个已发表的恶臭假单胞菌GEMs分析发现,即使同一株菌的模型,计算得到的菌体最优生长速率差异很大,而这些不一致是由于模型构建中引入的错误导致的。因此提出了对模型边界条件、ATP生成、呼吸链构成和途径等进行质控和一致化,并构建了高质量恶臭假单胞菌代谢网络模型PpuQY1140[12]。该工作发表后,天津工业生物所与来自丹麦、德国、荷兰、瑞士、美国及加拿大等其他15个国家的研究人员一起着手解决模型质控标准,提出标准化的模型质量检测工具(metabolic model tests, MEMOTE)[32]。在MEMOTE工具开发中,天津工业生物所的具体贡献是将模型能量无限生成错误作为模型打分的评估项引入MEMOTE打分规则中(图 1),如果模型存在错误的能量合成,将降低打分分值。MEMOTE是一套由社区维护的标准化代谢模型质量自动测试流程,涵盖了从模型注释到完整性的各个方面去检测模型质量。用户提供GEMs模型后,便可调用MEMOTE的核心功能进行模型质量评估,并最终生成HTML格式的质量检测报告(图 1)。目前,MEMOTE已成为GEM质量评估的黄金标准,自2020年3月发布以来,MEMOTE被引用次数已达197次,发表的新模型都应该经过MEMOTE进行打分。

|
| 图 1 MEMOTE对大肠杆菌iML1515模型的质量检测报告输出结果 Fig. 1 Quality test report for the E. coli model iML1515 from MEMOTE. |
| |
如果把代谢网络模型比作地图,那么模拟算法就是导航系统,精准的导航离不开高准确度的地图,也需要高效的导航系统指导模型进行途径和靶点的计算挖掘。通量平衡分析(flux balance analysis, FBA)[33]是对代谢网络模型进行定量分析以及预测最优途径的常用算法,利用线性规划来求解稳态条件下的通量分布,该算法已被广泛应用于代谢工程途径和改造策略设计。为了预测l-脯氨酸生物合成的关键通量控制反应,天津工业生物所多个课题组协作[34]利用谷氨酸棒杆菌GEM模型iCW773进行FBA计算,结果显示l-脯氨酸最优合成途径需要C4回补途径,谷氨酸棒杆菌存在天然的基于磷酸烯醇丙酮酸羧化酶(Ppc) 的C4回补途径将磷酸烯醇丙酮酸(PEP) 和CO2转化为草酰乙酸,而为了节省前体PEP,葡萄糖需要通过非磷酸转移酶系统(即非PTS) 部分转运到细胞中,这种转运在谷氨酸棒杆菌中效率低下。模拟表明,使用基于丙酮酸羧化酶(Pyc) 的C4回补途径导致葡萄糖产生过量的ATP,可以支持细胞生长,提供了一种有效替代方案。另外,通过结合基于3-磷酸甘油醛脱氢酶(GapN)[A3] 的3-磷酸甘油醛氧化途径和基于Pyc的C4回补途径,可以为细胞生长和l-脯氨酸生物合成提供足够的ATP和NADPH。在模型中加入GapN催化反应后,模拟表明葡萄糖的l-脯氨酸的理论得率从0.86 mol/mol增加到0.98 mol/mol (0.63 g/g)。基于上述分析,确定了能够提高l-脯氨酸得率的改造方案,后续通过一系列的基因工程改造,最终l-脯氨酸产量达到142.4 g/L。
天津工业生物所利用FBA算法基于大肠杆菌iJO1366模型对乙酰辅酶A最优合成途径进行预测[35],发现了一条通过回收CO2从而提高乙酰辅酶A得率的新途径。该途径通过磷酸烯醇式丙酮酸固定CO2生成草酰乙酸,进而转化为苏氨酸,苏氨酸降解成甘氨酸和乙酰辅酶A,而甘氨酸再转化为丙酮酸形成固碳循环(图 2A)。该循环中苏氨酸的合成和降解是核心,因此将该途径称之为苏氨酸循环。苏氨酸循环可将乙酰辅酶A衍生产品聚羟基丁酸酯(poly(3-hydroxybutyrate), PHB) 的理论碳摩尔得率由0.67提高到0.86,对大肠杆菌菌株进行代谢工程改造的结果表明,PHB得率由出发菌株的0.20 g/g葡萄糖提高到0.36 g/g葡萄糖。除了碳再回收利用外,避开如丙酮酸脱羧等碳损失步骤也可以提高产品得率。Bogorad等[36]在大肠杆菌中引入来源于B. adolescentis的戊糖/己糖磷酸转酮酶(Fxpk),构建了非氧化酵解途径(nonoxidative glycolysis, NOG),可无碳损地将一分子葡萄糖转化为3分子乙酸。基于该结果,天津工业生物所在大肠杆菌iJO1366模型中引入Fxpk,计算了几十种不同类生化产品的理论得率,结果表明NOG途径可提高很多产品得率,其中丙酮理论得率可提高50%,PHB可提高33%[11]。基于这一模型计算结果进行了代谢工程改造,结果显示丙酮的碳摩尔得率可由出发菌株0.38 mol/mol葡萄糖提高至0.47 mol/mol葡萄糖[37],PHB碳摩尔得率从0.26 mol/mol葡萄糖提高到0.43 mol/mol葡萄糖[38]。Meadows等[39]将NOG引入酿酒酵母中生产法尼烯,通过乙酰辅酶A、还原力等途径改造,最终法尼烯的碳摩尔得率由0.52 mol/mol葡萄糖提高到0.65 mol/mol葡萄糖。
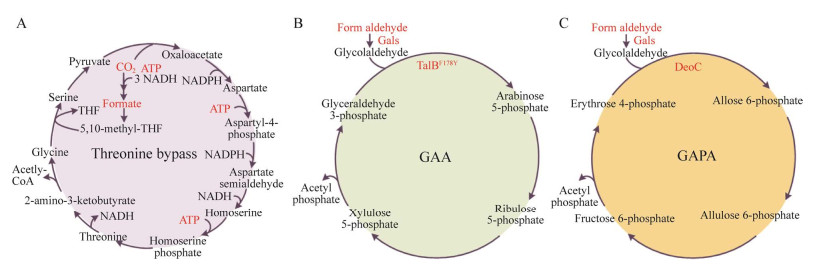
|
| 图 2 利用代谢网络模型预测的固碳循环途径 Fig. 2 CO2 assimilation pathways predicted using metabolic network models. (A) Threonine-bypass cycle predicted by E. coli iJO1366 model. (B) GAA pathway predicted by integrated model and comb-FBA algorithm. (C) GAPA pathway predicted by integrated model and comb-FBA algorithm. |
| |
利用FBA对单一菌株GEM可以进行改造策略和新途径预测,由于FBA单次计算只能得到一个最优解,无法对途径进行系统计算,同时单一菌株并未考虑数据库中海量反应数据,预测出的新途径有限。因此,天津工业生物所[40]将组合算法与FBA算法相结合,开发了comb-FBA算法,并且基于MetaCyc数据库构建了复合代谢网络模型,利用comb-FBA算法,在MetaCyc数据库中组合引入来自ATLAS数据库的非天然反应,实现了无碳损、无ATP和还原力消耗、未知反应个数可控的甲醛吸收途径的系统性挖掘,获得59条符合上述系列特征的目标途径,并通过实验验证了其中3条途径的可行性,其中乙醛酸同化途径(glycolaldehyde assimilation, GAA) 碳得率达到88% (图 2B),超过已报道的MCC途径。后续研究中,进一步预测了9条新途径,实验验证乙醇醛-阿洛糖6-磷酸同化途径(glycolaldehyde allose 6-phosphate assimilation, GAPA) 可以催化乙醇醛高效转化为乙酸(图 2C)[41]。基于上述开发的comb-FBA算法,天津工业生物所进一步对CO2产淀粉合成途径进行系统计算,设计了10条可行途径,基于此预测结果,天津工业生物所在体外成功构建了仅需要11步反应催化的ASAP途径,极大地缩减了通过天然卡尔文循环合成淀粉所需的约60步反应[42]。
2 多约束细胞模型基因组尺度代谢网络模型仅仅从生化反应速率的角度描述了细胞表型,忽视了其他的一些生物过程,比如基因调控、途径热力学限制等,而分析这些生物过程的协同效应就需要整合不同类型的数据[5]。为了提高模型预测的准确性,需要在计量学约束的基础上进一步整合其他类型的生物学数据,对模型进行限制,来体现细胞在真实代谢过程中所面临的资源约束。因此,寻找细胞表型差异背后是何种限制因素在发挥作用,也成为合成生物学领域重点关注的基础问题之一[43]。目前,主要的研究方向涉及整合转录组数据的代谢网络模型、整合资源约束的代谢网络模型、整合热力学约束的代谢网络模型,以及热力学和酶约束的整合模型,见图 3。

|
| 图 3 多约束模型研究发展路线图 Fig. 3 Development roadmap of multi-constraint models. |
| |
基因表达数据能够反应出细胞内转录本的相对或者绝对浓度,已有的研究发现转录本的浓度与代谢网络中的一些酶有较强的相关性[44]。近几年来,陆续出现了多种方法将转录组数据引入代谢网络模型约束条件中,见图 3。按照表达谱的整合方式可以分为两类:(1) 设定先验阈值或者计算相对的表达阈值判断基因表达状态[45-46],以此修改该基因对应反应的约束边界;(2) 直接将表达谱数据引入优化目标[47]或者边界条件中[48]。但是这些方法都存在一些缺陷,比如设定阈值判断基因表达状态对于判断基因是否表达的方式过于武断,会因此遗漏了一些表达值较低,但对细胞来说是必需的基因;直接引入目标函数的方法要求表达谱数据必须为绝对定量数据,由于通量和表达值间并不是同一量纲,量纲间的归一化也是需要考虑的问题;而直接利用表达谱重新定义反应上下限的方法,同样会出现表达值较低但功能重要的酶的通量被限制得很小,比如酶表达量虽然低,但其转化系数可能较高,其对应的通量同样会很高。
天津工业生物所通过分析基因本体论(gene ontology, GO) 中kcat值的分布规律,发现GO term的层次越高(描述的功能越具体详细),kcat值的标准差越小。因此,同一GO term中的kcat值可以近似用同一值替换(标记基因,在不同实验条件下表达量稳定的基因)。基于米氏方程的推导(
细胞中蛋白质的资源占比是有限的,细胞需要通过合理配置而使自身不同的生物过程,尤其是代谢过程得到高效运转[50]。2007年,通过假定固有的细胞体积会成为酶体积在空间资源上的限制,使拥挤效应成为细胞能力限制的FBAwMC模型[51]被提出。自FBAwMC以后,一系列的整合蛋白资源的模型被开发出来,发展趋势上主要分为以ME (metabolism and macromolecular expression)[52]为代表的包含酶量约束与蛋白转录翻译过程的资源约束模型(pcModel) 和以MOMENT (MetabOlic Modeling with ENzyme kineTics)[53]为代表的直接进行总酶量约束,即酶约束模型(ecModel),见图 3。
MOMENT发表于2012年,该方法直接将单位质量细胞内蛋白质的质量分数设定为细胞内的酶总量上限,并且认为所有酶的饱和度均为1,即处于完全饱和状态[53]。针对MOMENT方法的不足,GECKO做了进一步地完善,除了通过蛋白质组学数据计算出细胞内的酶占蛋白质的质量分数以外,还为每一个酶设定了一个平均的饱和度,这样计算得到的酶消耗更接近于实际值,提高了模型的预测准确度[54]。Zhou等[55]基于GECKO的建模流程构建了第一个黑曲霉的酶约束模型,通过该模型的模拟,发现酶约束能够有效提高模型的表型预测能力,并增强了模型预测潜在代谢工程改造靶点的能力。然而,GECKO在代谢网络中引入酶约束时会添加大量的假代谢物和假反应,以表示酶和酶相关的交换反应,这会增加模型的复杂性。Bekiaris等[50]基于MOMENT和GECKO,开发了一个自动化构建酶约束模型的方法(AutoPACMEN),该方法只需引入一个假反应和假代谢物,显著简化了酶约束模型的规模。
天津工业生物所于2022年提出了一个更加简易的构建酶约束模型的框架(ECMpy, https://github.com/tibbdc/ECMpy)[56]。与已有的框架相比,ECMpy考虑了蛋白亚基的真实组成,通过直接添加总酶量约束条件的方式实现蛋白资源约束,提出了基于酶用量和C13通量一致性等的校正流程,并利用该流程构建了一个高质量的大肠杆菌酶约束模型eciML1515。首先,通过eciML1515模拟,预测了大肠杆菌在高生长速率下的代谢溢流现象,从保持氧化还原平衡的角度分析了大肠杆菌与酵母在代谢溢流途径转化策略中的表现差异。接着,通过探究微生物生长过程中的3个代谢阶段,揭示了酶成本与生长之间的trade-off现象。最后,模拟了大肠杆菌在24种不同底物条件下的最大生长速率,进一步证实了酶约束模型可显著提高代谢网络模型的表型预测能力,并发现eciML1515模拟的生长速率与实验结果更接近,优于GECKO和AutoPACMEN方法构建的大肠杆菌酶约束模型。此外,还发现与C13数据相比,GECKO和AutoPACMEN构建的eciML1515预测的中心代谢途径中的代谢通量异常,尤其是EMP途径。基于这一框架,天津工业生物所还构建了首个谷氨酸棒杆菌的酶约束模型(ecCGL1),并利用该模型从酶成本角度识别出了使赖氨酸产量提升的靶点,这些靶点与已有的实验结果有较好的一致性,进一步体现了在GEM中引入酶动力学信息可以显著提升模型的表型预测能力,可以识别出途径中的关键酶,用于辅助代谢工程改造[57]。
这些模型构建流程使得构建任意物种的酶约束模型成为可能,为进一步解析某些生物学现象以及挖掘代谢工程改造靶点提供了指导。已有研究发现,整合蛋白资源约束的代谢网络模型提高了通量预测的准确度,能够正确模拟先前模型无法模拟的生物学现象[50, 54, 56],如代谢溢流、底物层级利用和途径切换等。这种基于酶约束的模型有望进一步缩小FBA的解空间,提高模型的表型预测水平,以更精准地指导代谢工程改造。基于酶约束模型的分析可以识别途径中的限速酶,进一步指导代谢工程的理性设计,已经成功应用于指导赖氨酸[58]和多聚谷氨酸[59]的生产。目前,酶约束模型的发展迅猛,已经成功构建了多个物种的酶约束模型,如酿酒酵母[54]、枯草芽孢杆菌[59]、大肠杆菌[56, 58]、黑曲霉[55]和谷氨酸棒杆菌等[57]。
2.3 热力学和酶约束整合模型的发展在代谢网络模型和酶约束模型的构建过程中,需要依据反应的热力学相关参数(如反应吉布斯自由能、代谢物浓度和酶的平衡常数等) 判断反应的可逆性。然而,这些热力学相关参数对于大部分物种来说都严重缺失,这就会导致对反应可逆性的误判,或者采用一些存在风险的默认做法对反应可逆性进行判断,比如将反应统一设置成可逆反应。这些操作都会降低代谢网络模型和酶约束的质量,从而导致预测途径的可行性降低,如产生能量和还原力的无限循环问题[12]。整合热力学因素可以将代谢物浓度及其对可逆性的影响体现出来,可以在很大程度上弥补当前酶约束模型普遍存在的过度简化造成的约束松弛、预测失真和研究角度单一等问题。例如,天津工业生物所[60]开发的Find_tfSBP算法引入了热力学可行性分析,排除了搜索网络中的热力学不可行途径,使识别地从底物到产物的最小平衡途径更具生物学意义。因此,整合更多组学规模的数据以实现约束层次增加和分析功能升级的模型构建工作开始受到关注。
2020年,Salvy等[61]将TMFA方法应用于大肠杆菌的ME模型中,开发了ETFL (expression and thermodynamics flux models) 建模方法,并将其用于探究资源限制下的生长表型和研究新的约束层次对于缩小解空间的影响。同年,天津工业生物所也公开了可将热力学约束与酶资源约束进行整合的自动化建模框架ETGEMs (图 4)[62]。天津工业生物所应用该框架构建了基于iML1515模型的具有酶学和热力学约束的大肠杆菌代谢模型EcoETM。模拟结果表明,新模型可以通过排除热力学不利或酶成本超过可用资源的途径来有效地减少解空间。ME模型和MOMENT模型在代谢网络模型的基础上都整合了酶的动力学参数信息,两者在构建原理上类似,但是他们采用的框架差异巨大,这也导致两类模型在应用层面差异很大。具体来说,ME模型更侧重于研究表型背后的生命机制和运转成本[10],而MOMENT则倾向于为代谢过程相关的通路和靶点优化提供指导和方案。这也导致了ETFL和ETGEMs方法在研究目标和应用场景上的差异,即ETGEMs更侧重于对代谢工程实践中的途径设计和靶点预测等工作提供指导。
天津工业生物所基于EcoETM和Pyomo建模包[63],通过整合多个目标和约束条件,开发了多个计算模块,包括酶成本可变性分析模块、代谢物浓度可变性分析模块、热力学驱动力可变性分析模块、最小酶量和计算模块、关键酶预测模块、瓶颈反应预测模块和限制性代谢物预测模块等。并以精氨酸合成为例,指明了多约束模型对于途径挖掘、生物学可行性评估以及优化策略提出的重要意义。具体来说,除经典代谢网络模型提供的通量分布信息外,酶热约束模型还能够提供途径的酶成本分布信息和反应的最佳热力学驱动力水平分布信息。此外,通过代谢物浓度可变性分析模块、热力学驱动力可变性分析模块以及酶成本可变性分析模块可变性分析模块可以获得精氨酸合成途径中的大量潜在靶点信息,比如途径中的关键酶CBPS和ARGSS、热力学瓶颈步骤AGCK和限制性代谢物乙酰谷氨酸等信息,这些靶点均可通过文献进行验证[64-65]。酶热约束模型提供的这些丰富信息可为人们应用基因编辑[66]、酶的定向进化[67]和解调控[68]等手段优化途径提供依据,最终为代谢产品的合成效率提升提供助力。
3 展望本文总结探讨了过去10年天津工业生物所在数字细胞模型构建方面的研究进展,这些研究工作对于理解细胞代谢组织规律,指导最优代谢途径的设计和确定菌种代谢工程改造策略具有重要意义。在常规计量学约束基础上引入热力学、酶等其他水平的约束是数字细胞模型进一步发展的方向,新约束的加入缩小了模型的解空间,进一步提高了模型预测的准确度。但目前为止发表的大多数模型仍以代谢网络为主,主要包括基因组中编码酶和传递蛋白的基因,即使在大肠杆菌等原核生物中这部分基因也仅占1/4左右,在高等生物中则低于1/10。除了基因覆盖度低外,这些基于约束优化方法的模型仍然都是基于稳态假设,只能描述细胞在一特定条件下的稳定状态,不能模拟细胞在环境条件变化时的动态调控行为,而动态调控是活的生命的最重要特征之一。针对这些问题,人们也从不同角度对细胞模型进行扩充,例如通过引入更多细胞过程,在模型中涵盖更多基因以得到所谓全细胞模型。2012年COVERT研究组[69]针对有500多个基因的生殖道支原体(Mycoplasma genitalium) 建立了第一个全细胞模型,实现了对细胞代谢、蛋白转录翻译和细胞分裂等多种生物过程的模拟。要说明的是该模型中只包含很少的调控信息,例如其仅包括5个转录调控因子相关的29个转录调控关系,而且采用了非常简化的数学形式进行描述。2020年刘立明研究组[70]针对酿酒酵母建立了一个全细胞模型WM_S288C,该模型参照了COVERT的模型结构,在模型中包含了15个细胞状态和26个细胞过程,利用该模型阐明了酿酒酵母中基因型和表现间的关系,预测了细胞周期内的资源分配和细胞行为,以及识别了细胞内核苷酸的调节机制。2021年Pelletier等[71]针对全人工合成的具有493个基因的最小细胞JCVIsyn3A构建了全细胞动力学模型,与COVERT的模型相比,其考虑了核糖体、DNA等大分子在胞内的空间分布,而且采用了随机概率(stochastic) 模型与决定论动力学模型相结合的方法以提高对低拷贝分子动态变化模拟的可靠性。全细胞模型包含的生物过程更全面,而且对很多过程采用动力学方程描述从而可以分析细胞的动态变化。但存在的问题是很多生物过程难以得到准确的定量数据确定其动力学方程,模型构建困难、投入高,因此至今只有3个全细胞模型,模型中几乎不包括任何调控过程。近年来人们提出了针对大肠杆菌等模式生物构建全细胞模型的设想,但目前除酿酒酵母外,尚无相关研究进展报道。考虑到大肠杆菌等具有比简单寄生菌更复杂的多层次调控机制,很难对所有相关调控过程都进行准确测量以确定其动力学方程,这种自下而上由基本细胞过程构建全细胞模型的方法可能并不是构建整体细胞模型的最理想方法。结合细胞系统宏观水平的组织结构特征,引入更高层次的组织调控优化原理来构建多层次模块化模型也许是全细胞模型构建最可行的路径。最近天津工业生物所通过对代谢网络中所有代谢物间的转化途径关系确定了多种生物代谢网络整体组织具有蝴蝶结结构特征,这一整体特征与细胞在波动环境中以最小调控适应新环境(如底物切换) 密切相关[72]。而这一最小调控目标与代谢网络模型模拟基因敲除后通量变化的MOMA算法的思路异曲同工。复杂系统科学的一个核心观点就是系统大于其组成成分之和,细胞作为一个典型复杂系统,其中每个组件都是非生命的,只有合成一个整体才具有生命特征,在整体层次的行为并不仅仅由组件决定,而是系统水平自组织的结果。因此要理解细胞整体行为,特别是其从非生命至生命的系统层次涌现机制,仅靠各个底层生物过程的添加和定量化建立模型是不够的。整合系统层次组织原理对基础生物过程进行概括简化可能是针对绝大多数细胞构建数字细胞模型的必要步骤。这也是天津工业生物所在数字细胞研究领域今后的主要研究方向。
除了与复杂系统原理结合外,将基于机理的细胞模型与机器学习人工智能模型相结合也是一个值得关注的发展趋势。最近天津工业生物所[73]发表的一篇人工智能在合成生物学研究中应用的综述文章对代谢网络模型与人工智能结合的研究进展也做了详细介绍。人工智能方法可以协助发现预测细胞中未知的相互作用机理(如代谢网络中的缺失反应) 和未知参数(如酶的动力学参数),从而提高模型的完整度和准确度。例如最近Nielsen研究组[74]提出了一种深度学习方法(DLKcat),仅从底物结构和蛋白序列就可以对任何生物体代谢网络中的酶进行高通量的kcat预测,提高了代谢网络中酶动力学参数的覆盖度,推动了酶约束代谢网络模型的构建。另一方面,人工智能预测方法与基于机理模型的靶点预测方法可以结合以更准确地预测代谢工程改造靶点。2019年Hon等[75]就结合人工蜂群算法(artificial bee colony algorithm, ABC) 和FBA算法,识别了大肠杆菌中提高琥珀酸和乳酸产量的敲除策略,使琥珀酸和乳酸的得率分别提高到了18.17 g/g葡萄糖和12.19 g/g葡萄糖。天津工业生物所最近也开展了应用人工智能预测酶号和新反应以完善细胞模型的研究工作并已取得初步进展。侧重机理的数字细胞模型与数据驱动的人工智能模型的结合将更好地推动人们对复杂生命现象的理解,实现更高效的人工细胞合成和改造。
| [1] |
张媛媛, 曾艳, 王钦宏. 合成生物制造进展. 合成生物学, 2021, 2(2): 145-160. Zhang YY, Zeng Y, Wang QH. Advances in synthetic biomanufacturing. Syn Biol J, 2021, 2(2): 145-160 (in Chinese). |
| [2] |
Fang X, Lloyd CJ, Palsson BO. Reconstructing organisms in silico: genome-scale models and their emerging applications. Nat Rev Microbiol, 2020, 18(12): 731-743. DOI:10.1038/s41579-020-00440-4
|
| [3] |
Gu CD, Kim GB, Kim WJ, et al. Current status and applications of genome-scale metabolic models. Genome Biol, 2019, 20(1): 121. DOI:10.1186/s13059-019-1730-3
|
| [4] |
郝彤, 马红武, 赵学明. 基因组尺度代谢网络自动重构及分析工具研究进展. 生物工程学报, 2012, 28(6): 661-670. Hao T, Ma HW, Zhao XM. Progress in automatic reconstruction and analysis tools of genome-scale metabolic network. Chin J Biotech, 2012, 28(6): 661-670 (in Chinese). |
| [5] |
Machado D, Herrgård M. Systematic evaluation of methods for integration of transcriptomic data into constraint-based models of metabolism. PLoS Comput Biol, 2014, 10(4): e1003580. DOI:10.1371/journal.pcbi.1003580
|
| [6] |
王雪亮, 张芸, 温廷益. 整合组学数据的代谢网络模型研究进展. 科学通报, 2021, 66(19): 2393-2404. Wang XL, Zhang Y, Wen TY. Progress on genome-scale metabolic models integrated with multi-omics data. Chin Sci Bull, 2021, 66(19): 2393-2404 (in Chinese). |
| [7] |
杨雪, 张培基, 毛志涛, 等. 多约束代谢网络模型的研究进展. 生物工程学报, 2022, 38(2): 531-545. Yang X, Zhang PJ, Mao ZT, et al. Development of metabolic models with multiple constraints: a review. Chin J Biotech, 2022, 38(2): 531-545 (in Chinese). DOI:10.13345/j.cjb.210335 |
| [8] |
虞思倩, 夏建业, 庄英萍. 基于热力学原理约束的代谢网络模型研究进展及其应用. 中国生物工程杂志, 2022, 42(1/2): 128-138. Yu SQ, Xia JY, Zhuang YP. Research progress and application of metabolic network model constrained by thermodynamic principles. China Biotechnol, 2022, 42(1/2): 128-138 (in Chinese). |
| [9] |
Lerman JA, Hyduke DR, Latif H, et al. In silico method for modelling metabolism and gene product expression at genome scale. Nat Commun, 2012, 3: 929. DOI:10.1038/ncomms1928
|
| [10] |
O'Brien EJ, Lerman JA, Chang RL, et al. Genome-scale models of metabolism and gene expression extend and refine growth phenotype prediction. Mol Syst Biol, 2013, 9: 693. DOI:10.1038/msb.2013.52
|
| [11] |
袁倩倩, 李斐然, 罗浩, 等. 由代谢网络分析发现菌种代谢工程改造新策略. 化工进展, 2017, 36(12): 4592-4600. Yuan QQ, Li FR, Luo H, et al. Discovery of new strain modification strategies by metabolic network analysis. Chem Ind Eng Prog, 2017, 36(12): 4592-4600 (in Chinese). DOI:10.16085/j.issn.1000-6613.2017-0557 |
| [12] |
Yuan QQ, Huang T, Li PS, et al. Pathway-consensus approach to metabolic network reconstruction for Pseudomonas putida KT2440 by systematic comparison of published models. PLoS One, 2017, 12(1): e0169437. DOI:10.1371/journal.pone.0169437
|
| [13] |
Luo JH, Yuan QQ, Mao YF, et al. Reconstruction of a genome-scale metabolic network for Shewanella oneidensis MR-1 and analysis of its metabolic potential for bioelectrochemical systems. Front Bioeng Biotechnol, 2022, 10: 913077. DOI:10.3389/fbioe.2022.913077
|
| [14] |
Liu DF, Xu ZX, Li JG, et al. Reconstruction and analysis of genome-scale metabolic model for thermophilic fungus Myceliophthora thermophila. Biotech & Bioengineering, 2022, 119(7): 1926-1937.
|
| [15] |
Guo YF, Su LQ, Liu Q, et al. Dissecting carbon metabolism of Yarrowia lipolytica type strain W29 using genome-scale metabolic modelling. Comput Struct Biotechnol J, 2022, 20: 2503-2511. DOI:10.1016/j.csbj.2022.05.018
|
| [16] |
Li FR, Xie W, Yuan QQ, et al. Genome-scale metabolic model analysis indicates low energy production efficiency in marine ammonia-oxidizing archaea. AMB Express, 2018, 8(1): 1-12. DOI:10.1186/s13568-017-0531-x
|
| [17] |
Thiele I, Palsson BØ. A protocol for generating a high-quality genome-scale metabolic reconstruction. Nat Protoc, 2010, 5(1): 93-121. DOI:10.1038/nprot.2009.203
|
| [18] |
Heirendt L, Arreckx S, Pfau T, et al. Creation and analysis of biochemical constraint-based models using the COBRA Toolbox v.3.0. Nat Protoc, 2019, 14(3): 639-702. DOI:10.1038/s41596-018-0098-2
|
| [19] |
Seaver SMD, Liu F, Zhang QZ, et al. The ModelSEED biochemistry database for the integration of metabolic annotations and the reconstruction, comparison and analysis of metabolic models for plants, fungi and microbes. Nucleic Acids Res, 2021, 49(D1): D1555. DOI:10.1093/nar/gkaa1143
|
| [20] |
Agren R, Liu LM, Shoaie S, et al. The RAVEN toolbox and its use for generating a genome-scale metabolic model for Penicillium chrysogenum. PLoS Comput Biol, 2013, 9(3): e1002980. DOI:10.1371/journal.pcbi.1002980
|
| [21] |
Dias O, Rocha M, Ferreira EC, et al. Reconstructing genome-scale metabolic models with merlin. Nucleic Acids Res, 2015, 43(8): 3899-3910. DOI:10.1093/nar/gkv294
|
| [22] |
Viana R, Dias O, Lagoa D, et al. Genome-scale metabolic model of the human pathogen Candida albicans: a promising platform for drug target prediction. JoF, 2020, 6(3): 171. DOI:10.3390/jof6030171
|
| [23] |
Dias O, Saraiva J, Faria C, et al. iDS372, a phenotypically reconciled model for the metabolism of Streptococcus pneumoniae strain R6. Front Microbiol, 2019, 10: 1283. DOI:10.3389/fmicb.2019.01283
|
| [24] |
Capela J, Lagoa D, Rodrigues R, et al. Merlin, an improved framework for the reconstruction of high-quality genome-scale metabolic models. Nucleic Acids Res, 2022, 50: 6052-6066. DOI:10.1093/nar/gkac459
|
| [25] |
Ye C, Qiao WH, Yu XB, et al. Reconstruction and analysis of the genome-scale metabolic model of Schizochytrium limacinum SR21 for docosahexaenoic acid production. BMC Genom, 2015, 16(1): 1-11. DOI:10.1186/1471-2164-16-1
|
| [26] |
Ye C, Xu N, Chen HQ, et al. Reconstruction and analysis of a genome-scale metabolic model of the oleaginous fungus Mortierella alpina. BMC Syst Biol, 2015, 9(1): 1-11. DOI:10.1186/s12918-014-0137-8
|
| [27] |
Mei J, Xu N, Ye C, et al. Reconstruction and analysis of a genome-scale metabolic network of Corynebacterium glutamicum S9114. Gene, 2016, 575(2): 615-622. DOI:10.1016/j.gene.2015.09.038
|
| [28] |
Zhang Y, Cai JY, Shang XL, et al. A new genome-scale metabolic model of Corynebacterium glutamicum and its application. Biotechnol Biofuels, 2017, 10: 169. DOI:10.1186/s13068-017-0856-3
|
| [29] |
Pan PC, Hua Q. Reconstruction and in silico analysis of metabolic network for an oleaginous yeast, Yarrowia lipolytica. PLoS One, 2012, 7(12): e51535. DOI:10.1371/journal.pone.0051535
|
| [30] |
Huang D, Li SS, Xia ML, et al. Genome-scale metabolic network guided engineering of Streptomyces tsukubaensis for FK506 production improvement. Microb Cell Fact, 2013, 12: 52. DOI:10.1186/1475-2859-12-52
|
| [31] |
Dang LQ, Liu J, Wang C, et al. Enhancement of rapamycin production by metabolic engineering in Streptomyces hygroscopicus based on genome-scale metabolic model. J Ind Microbiol Biotechnol, 2017, 44(2): 259-270. DOI:10.1007/s10295-016-1880-1
|
| [32] |
Lieven C, Beber ME, Olivier BG, et al. MEMOTE for standardized genome-scale metabolic model testing. Nat Biotechnol, 2020, 38(3): 272-276. DOI:10.1038/s41587-020-0446-y
|
| [33] |
Orth JD, Thiele I, Palsson BØ. What is flux balance analysis?. Nat Biotechnol, 2010, 28(3): 245-248. DOI:10.1038/nbt.1614
|
| [34] |
Liu J, Liu MS, Shi T, et al. CRISPR-assisted rational flux-tuning and arrayed CRISPRi screening of an L-proline exporter for L-proline hyperproduction. Nat Commun, 2022, 13(1): 1-16. DOI:10.1038/s41467-021-27699-2
|
| [35] |
Lin ZQ, Zhang Y, Yuan QQ, et al. Metabolic engineering of Escherichia coli for poly(3-hydroxybutyrate) production via threonine bypass. Microb Cell Factories, 2015, 14(1): 1-12. DOI:10.1186/s12934-014-0183-3
|
| [36] |
Bogorad IW, Chen CT, Theisen MK, et al. Building carbon-carbon bonds using a biocatalytic methanol condensation cycle. PNAS, 2014, 111(45): 15928-15933. DOI:10.1073/pnas.1413470111
|
| [37] |
Yang XY, Yuan QQ, Zheng YY, et al. An engineered non-oxidative glycolysis pathway for acetone production in Escherichia coli. Biotechnol Lett, 2016, 38(8): 1359-1365. DOI:10.1007/s10529-016-2115-2
|
| [38] |
Zheng YY, Yuan QQ, Luo H, et al. Engineering NOG-pathway in Escherichia coli for poly-(3-hydroxybutyrate) production from low cost carbon sources. Bioengineered, 2018, 9(1): 209-213. DOI:10.1080/21655979.2018.1467652
|
| [39] |
Meadows AL, Hawkins KM, Tsegaye Y, et al. Rewriting yeast central carbon metabolism for industrial isoprenoid production. Nature, 2016, 537(7622): 694-697. DOI:10.1038/nature19769
|
| [40] |
Yang X, Yuan QQ, Luo H, et al. Systematic design and in vitro validation of novel one-carbon assimilation pathways. Metab Eng, 2019, 56: 142-153. DOI:10.1016/j.ymben.2019.09.001
|
| [41] |
Mao YF, Yuan QQ, Yang X, et al. Non-natural aldol reactions enable the design and construction of novel one-carbon assimilation pathways in vitro. Front Microbiol, 2021, 12: 677596. DOI:10.3389/fmicb.2021.677596
|
| [42] |
Cai T, Sun HB, Qiao J, et al. Cell-free chemoenzymatic starch synthesis from carbon dioxide. Science, 2021, 373(6562): 1523-1527. DOI:10.1126/science.abh4049
|
| [43] |
Price ND, Reed JL, Palsson BØ. Genome-scale models of microbial cells: evaluating the consequences of constraints. Nat Rev Microbiol, 2004, 2(11): 886-897. DOI:10.1038/nrmicro1023
|
| [44] |
Vogel C, Marcotte EM. Insights into the regulation of protein abundance from proteomic and transcriptomic analyses. Nat Rev Genet, 2012, 13(4): 227-232. DOI:10.1038/nrg3185
|
| [45] |
Becker SA, Palsson BO. Context-specific metabolic networks are consistent with experiments. PLoS Comput Biol, 2008, 4(5): e1000082. DOI:10.1371/journal.pcbi.1000082
|
| [46] |
Zur H, Ruppin E, Shlomi T. iMAT: an integrative metabolic analysis tool. Bioinformatics, 2010, 26(24): 3140-3142. DOI:10.1093/bioinformatics/btq602
|
| [47] |
Collins SB, Reznik E, Segrè D. Temporal expression-based analysis of metabolism. PLoS Comput Biol, 2012, 8(11): e1002781. DOI:10.1371/journal.pcbi.1002781
|
| [48] |
Colijn C, Brandes A, Zucker J, et al. Interpreting expression data with metabolic flux models: predicting Mycobacterium tuberculosis mycolic acid production. PLoS Comput Biol, 2009, 5(8): e1000489. DOI:10.1371/journal.pcbi.1000489
|
| [49] |
Mao ZT, Ma HW. iMTBGO: an algorithm for integrating metabolic networks with transcriptomes based on gene ontology analysis. Curr Genomics, 2019, 20(4): 252-259. DOI:10.2174/1389202920666190626155130
|
| [50] |
Bekiaris PS, Klamt S. Automatic construction of metabolic models with enzyme constraints. BMC Bioinformatics, 2020, 21(1): 19. DOI:10.1186/s12859-019-3329-9
|
| [51] |
Beg QK, Vazquez A, Ernst J, et al. Intracellular crowding defines the mode and sequence of substrate uptake by Escherichia coli and constrains its metabolic activity. PNAS, 2007, 104(31): 12663-12668. DOI:10.1073/pnas.0609845104
|
| [52] |
Lerman JA, Hyduke DR, Latif H, et al. In silico method for modelling metabolism and gene product expression at genome scale. Nat Commun, 2012, 3: 929. DOI:10.1038/ncomms1928
|
| [53] |
Adadi R, Volkmer B, Milo R, et al. Prediction of microbial growth rate versus biomass yield by a metabolic network with kinetic parameters. PLoS Comput Biol, 2012, 8(7): e1002575. DOI:10.1371/journal.pcbi.1002575
|
| [54] |
Sánchez BJ, Zhang C, Nilsson A, et al. Improving the phenotype predictions of a yeast genome-scale metabolic model by incorporating enzymatic constraints. Mol Syst Biol, 2017, 13(8): 935. DOI:10.15252/msb.20167411
|
| [55] |
Zhou JR, Zhuang YP, Xia JY. Integration of enzyme constraints in a genome-scale metabolic model of Aspergillus niger improves phenotype predictions. Microb Cell Fact, 2021, 20(1): 125. DOI:10.1186/s12934-021-01614-2
|
| [56] |
Mao ZT, Zhao X, Yang X, et al. ECMpy, a simplified workflow for constructing enzymatic constrained metabolic network model. Biomolecules, 2022, 12(1): 65. DOI:10.3390/biom12010065
|
| [57] |
Niu JH, Mao ZT, Mao YF, et al. Construction and analysis of an enzyme-constrained metabolic model of Corynebacterium glutamicum. Biomolecules, 2022, 12(10): 1499. DOI:10.3390/biom12101499
|
| [58] |
Ye C, Luo QL, Guo L, et al. Improving lysine production through construction of an Escherichia coli enzyme-constrained model. Biotechnol Bioeng, 2020, 117(11): 3533-3544. DOI:10.1002/bit.27485
|
| [59] |
Massaiu I, Pasotti L, Sonnenschein N, et al. Integration of enzymatic data in Bacillus subtilis genome-scale metabolic model improves phenotype predictions and enables in silico design of poly-γ-glutamic acid production strains. Microb Cell Fact, 2019, 18(1): 3. DOI:10.1186/s12934-018-1052-2
|
| [60] |
Xu ZX, Sun JB, Wu QQ, et al. Find_tfSBP: find thermodynamics-feasible and smallest balanced pathways with high yield from large-scale metabolic networks. Sci Rep, 2017, 7(1): 17334. DOI:10.1038/s41598-017-17552-2
|
| [61] |
Salvy P, Hatzimanikatis V. The ETFL formulation allows multi-omics integration in thermodynamics-compliant metabolism and expression models. Nat Commun, 2020, 11(1): 30. DOI:10.1038/s41467-019-13818-7
|
| [62] |
Yang X, Mao ZT, Zhao X, et al. Integrating thermodynamic and enzymatic constraints into genome-scale metabolic models. Metab Eng, 2021, 67: 133-144. DOI:10.1016/j.ymben.2021.06.005
|
| [63] |
Hart WE, Watson JP, Woodruff DL. Pyomo: modeling and solving mathematical programs in Python. Math Program Comput, 2011, 3(3): 219-260. DOI:10.1007/s12532-011-0026-8
|
| [64] |
Charlier D, Bervoets I. Regulation of arginine biosynthesis, catabolism and transport in Escherichia coli. Amino Acids, 2019, 51(8): 1103-1127. DOI:10.1007/s00726-019-02757-8
|
| [65] |
Utagawa T. Production of arginine by fermentation. J Nutr, 2004, 134(10): 2854S-2857S. DOI:10.1093/jn/134.10.2854S
|
| [66] |
Wang Y, Cheng HJ, Liu Y, et al. In-situ generation of large numbers of genetic combinations for metabolic reprogramming via CRISPR-guided base editing. Nat Commun, 2021, 12(1): 1-12. DOI:10.1038/s41467-020-20314-w
|
| [67] |
Lu XY, Liu YW, Yang YQ, et al. Constructing a synthetic pathway for acetyl-coenzyme A from one-carbon through enzyme design. Nat Commun, 2019, 10(1): 1-10. DOI:10.1038/s41467-018-07882-8
|
| [68] |
Guo J, Man ZW, Rao ZM, et al. Improvement of the ammonia assimilation for enhancing L-arginine production of Corynebacterium crenatum. J Ind Microbiol Biotechnol, 2017, 44(3): 443-451. DOI:10.1007/s10295-017-1900-9
|
| [69] |
Karr JR, Sanghvi JC, Macklin DN, et al. A whole-cell computational model predicts phenotype from genotype. Cell, 2012, 150(2): 389-401. DOI:10.1016/j.cell.2012.05.044
|
| [70] |
Ye C, Xu N, Gao C, et al. Comprehensive understanding of Saccharomyces cerevisiae phenotypes with whole-cell model WM_S288C. Biotechnol Bioeng, 2020, 117(5): 1562-1574. DOI:10.1002/bit.27298
|
| [71] |
Pelletier JF, Sun LJ, Wise KS, et al. Genetic requirements for cell division in a genomically minimal cell. Cell, 2021, 184(9): 2430-2440. DOI:10.1016/j.cell.2021.03.008
|
| [72] |
Gao YJ, Yuan QQ, Mao ZT, et al. Global connectivity in genome-scale metabolic networks revealed by comprehensive FBA-based pathway analysis. BMC Microbiol, 2021, 21(1): 292.
|
| [73] |
Liao XP, Ma HW, Tang YJ. Artificial intelligence: a solution to involution of design-build-test-learn cycle. Curr Opin Biotechnol, 2022, 75: 102712.
|
| [74] |
Li F, Yuan L, Lu H, et al. Deep learning-based kcat prediction enables improved enzyme-constrained model reconstruction. Nat Catal, 2022, 5(8): 662-672.
|
| [75] |
Hon MK, Mohamad MS, Mohamed Salleh AH, et al. Identifying a gene knockout strategy using a hybrid of simple constrained artificial bee colony algorithm and flux balance analysis to enhance the production of succinate and lactate in Escherichia coli. Interdiscip Sci Comput Life Sci, 2019, 11(1): 33-44.
|