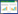中国科学院微生物研究所、中国微生物学会主办
文章信息
- 宁书晴, 巫欣欣, 罗云孜
- NING Shuqing, WU Xinxin, LUO Yunzi
- CRISPR相关转座元件的研究及应用进展
- Recent advances in CRISPR-related transposable elements
- 生物工程学报, 2022, 38(12): 4371-4384
- Chinese Journal of Biotechnology, 2022, 38(12): 4371-4384
- 10.13345/j.cjb.220197
-
文章历史
- Received: March 15, 2022
- Accepted: May 13, 2022
规律成簇的间隔短回文重复序列(clustered regularly interspaced short palindromic repeats, CRISPR) 及其相关Cas9和Cas12a蛋白所构建的CRISPR-Cas9和CRISPR-Cas12a系统是目前发展最快的基因编辑工具[1-3]。但是有限的前间隔序列邻近基序(protospacer adjacent motif, PAM) 识别范围、Cas蛋白对宿主的细胞毒性以及某些宿主细胞同源重组(homology-directed repair, HDR) 或非同源末端连接(non-homologous end joining, NHEJ) 效率较低[3-5]等问题限制了这些系统的进一步应用。长期以来,研究者们不断改造优化Cas9和Cas12a蛋白,以拓展这些系统对不同PAM序列的兼容性。如今Cas9的衍生物已经可以识别多种PAM序列[6-10],比如xCas9 (识别NG、NGG、GAA、GAT)[8]、SpCas9-NG变体(识别NG)[7-8]以及几乎不受PAM限制的SpCas9-SpRY变体(识别NRN和NYN,Y=C/T)[10]。本课题组[11-12]针对Cas12a识别TTN PAM的局限性问题构建了FnCas12a变体EP16,其PAM识别范围扩大到YN (Y=C/T)、TAC和CAA。本课题组[13]还针对CRISPR-Cas9在部分链霉菌(Streptomyces) 中的细胞毒性问题,使用工程化的CRISPR- FnCas12a3 (FnCas12a-EP16) 系统在链霉菌中精确执行位点突变和插入,为链霉
菌天然产物的发掘和激活提供了新工具;与此同时,针对CRISPR-Cas9在毕赤酵母(Pichia pastoris) 中编辑效率较低的问题,Zhang等[14]开发了用于毕赤酵母的CRISPR-Cas12a高效基因组编辑方法,加速了毕赤酵母代谢工程及基因组进化相关领域的研究。但是仍有某些宿主细胞由于HDR或NHEJ的能力较低[1-3]而无法修复断裂的DNA,使得CRISPR-Cas9和CRISPR-Cas12a系统在这些细胞中较难发挥作用。针对上述问题,研究者们开始注意“自给自足”的转座子。转座子是可以从宿主基因组中的一个位置移动到另一个位置的DNA片段,核心部分是左末端序列(left end, LE)、右末端序列(right end, RE) 以及中间的转座酶货物基因(cargo gene)[15]。转座酶表达后便会结合左右末端形成复合物,随之转座子从供体DNA完整脱落,并被转座酶带领转移到另一个位置。转座过程涉及到DNA靶点的切割和片段插入后缺口的缝合均由转座酶完成,是完全“自给自足”的。近几年,研究者在微生物体内相继鉴定出多种CRISPR系统相关的转座元件:Casposon[16-17]、TnpB[18]、IscB[19]和CAST (CRISPR-associated transposase)[20-22],这类元件依靠转座酶可将DNA有效和有针对性地整合到基因组中,无需借助HDR或NHEJ。值得注意的是,CRISPR系统相关的转座元件具有操作大片段基因的潜力,可以在基因组插入10 kb以上的片段[20-22]。这些元件的发现促使了转座酶和dCas9 (nuclease- dead mutants of Cas9, dCas9) 人工融合策略的出现并衍生出更复杂多样的应用,为基因编辑工具的开发带来了新的思路。
本文综述了近几年报道的CRISPR相关的转座元件的研究进展,并比较了其与CRISPR- Cas系统在结构和功能上的相似性(图 1和表 1)。Casposons超家族转座子编码的Cas1同源蛋白以及与原型间隔序列(protospacer) 相似的整合过程揭示了转座子和CRISPR适应模块之间的关系[23-25]。IS200/IS605家族的非自主转座子编码的转座酶TnpB和IscB具有Cas9、Cas12a同源的RuvC核酸酶结构域,在RNA的介导下可以完成基因的靶向编辑。这揭示了转座酶与CRISPR效应蛋白之间的关系[18-19, 26-28]。另外,CAST系统的基因座同时编码了转座酶基因、CRISPR阵列和cas基因,位置上的相关性和功能上的互补性[29]揭示了移动遗传元件(mobile genetic elements, MGE) 和CRISPR系统的合作关系。同时,本文还总结了近几年对于CAST系统靶向转座机制的研究进展,未来有望在此基础上对这类系统进行模块化改造和进化,以实现更高的编辑效率和更广的编辑范围。最后,本文简要介绍了人工融合dCas9-转座酶(dCas9-transposase) 的策略和应用(图 1),运用这种策略有望实现复杂的基因编辑。
|
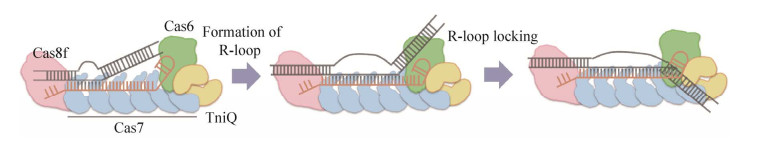
| 图 1 CRISPR相关转座元件的研究进展 Fig. 1 Recent advances in CRISPR-associated transposable elements. A:Casposon和CRISPR适应模块的同源性;B:TnpB、IscB和Cas效应核酸酶结构域的同源性;C:CRISPR相关转座因子的应用示意图 (A) Homology comparison of Casposon and CRISPR adaptation modules. (B) Homology comparison of the domains of TnpB, IscB and Cas-effector nucleases. (C) Schematic diagram of the applications of CRISPR-associated transposable elements. |
| |
| Name | Types | Composition | Relationship to the CRISPR-Cas systems |
| Casposon | Transposon | Terminal inverted repeats and casposase | Casposase is highly homologous to Cas1, the integration process of Casposon and protospacer is similar |
| TnpB | Transposase | RuvC domain | TnpB is highly homologous to Cas9 and Cas12a |
| IscB | Transposase | RuvC and HNH domain | IscB is highly homologous to Cas9 |
| CAST | Transposon | Left end, right end, CRISPR array, Cas protein and transposase | MGEs capture CRISPR-Cas systems for counter-defense |
目前,研究者们鉴定了Casposon[16-17]、TnpB[18]、IscB[19]和CAST[20-22]四种CRISPR系统相关转座元件并解析了它们的基因座结构和功能。其中,Casposon、TnpB和IscB是相对独立的转座元件,生物信息学分析表明它们与Cas蛋白具有较高同源性[17-19, 27, 30]。CAST是一种与CRISPR-Cas系统关联的类Tn7转座子,它独特的基因座结构决定了其靶向转座的功能[20-22, 31-32]。
1.1 Casposons家族转座子的结构与功能Casposon具备转座子的标准结构,即被反向重复序列(terminal inverted repeats, TIR) 包裹的货物基因以及元件两翼的靶位点重复序列(target site duplications, TSD)。Casposon的货物基因编码Cas1的同源物(Casposase) 和一种B族DNA聚合酶(DNA polymerase B, PolB),这两种蛋白在Casposon中高度保守并广泛存在,是鉴定Casposons家族转座子的重要依据[16, 24, 30]。在CRISPR-Cas系统中,Cas1核酸酶和CRISPR阵列适应模块负责捕获外源DNA并形成免疫记忆[5, 33]。Casposon能够预测整合的目标位点,使多个casposon基因形成串联结构。在某些古菌中,每两个casposon之间都会被TSD分隔开,形成Casposon阵列[34]。这类似于在适应阶段,CRISPR阵列将原型间隔序列(protospacer) 优先整合在前导重复序列(leader-repeat) 的边界处,从而形成了“间隔子-重复序列-间隔子”的阵列结构[35-37]。因此,Casposon可以和CRISPR一样,仅靠Cas1同源物就足以将casposon特异性整合到目标位点[17],且由Cas1和Casposase分别催化的protospacer和casposon整合反应非常相似(图 2)。第一次亲核攻击分别发生在CRISPR的前导重复序列和Casposon的tRNA-TSD片段边界。在中间体形成后,第二次亲核攻击分别发生在CRISPR的重复间隔区边界和Casposon的TSD片段与侧翼DNA间的连接处[17]。
之前研究者们假设CRISPR-repeats是由Casposon-TIR进化而来,但casposon和protospacer相似的整合模式将此进化场景进行了更新[25],因此推测CRISPR-repeats是直接从预先存在的casposon靶位点TSD进化而来的(图 2)。也就是说,Casposon首先整合到Cascade效应模块的附近,形成TSD重复序列,随后发生TIR的丢失和祖先Casposon在基因座上的固定,最后Cas1将异源DNA整合到原始目标中[24]。
1.2 TnpB、IscB转座酶的结构与功能除了Casposons家族转座子的结构和整合方式与CRISPR的适应模块相似,研究者们还发现IS200/IS605非自主转座子家族编码的TnpB和IscB转座酶同2类CRISPR-Cas系统的效应模块存在同源性。TnpB和IscB具有保守的RuvC样活性位点,有的RuvC样结构域中还会包含HNH结构域,这与效应核酸酶Cas9、Cas12a高度同源[27, 38]。起初只有TnpA转座酶主导的转座子的“剪切-粘贴”功能被证实[39-42],而TnpB和IscB两种蛋白被认为是可有可无的,甚至会起到下调转座的作用[27, 38]。直到近两年,TnpB和IscB的核酸酶功能才得到证实。在TnpA失活的转座子ISDra2中,TnpB蛋白与右末端回文序列(reRNA) 共表达,形成的reRNA- TnpB核糖核蛋白复合物能够识别5′-TTGAT的转座子相关基序(transposon-associated motif, TAM),并在TnpB的RuvC结构域的作用下实现基因的靶向切割[18]。该过程类似于CRISPR- Cas12a系统靶向目标基因的过程,即由crRNA引导的Cas12识别PAM基序从而完成目标基因的精准定位。同样地,编码IscB蛋白的转座子上游存在一段高度保守的序列,该序列编码一种类似于gRNA的ωRNA,它可以引导IscB蛋白识别ATAAA-3′的TAM序列,从而完成基因的靶向切割[19]。
基于TnpB、IscB和Cas9、Cas12序列和结构的比对分析,生物信息学推断TnpB、IscB可能是Cas9、Cas12核酸酶的祖先[27, 42-44],它们的蛋白结构和功能的极高相似性为该推论提供了有力的实验证据。Casposons家族转座子与CRISPR适应模块的相似性、TnpB/IscB和效应核酸酶的相似性均表明了转座元件和CRISPR- Cas系统之间的趋同关系,这说明转座元件或许也可以被重编程,成为基因编辑工具。此外,TnpB、IscB分子大小远小于Cas9,是一类新的极其紧凑的非Cas核酸酶,它们丰富了基因组编辑工具包,为更精确地诊断和更高效地治疗开辟了新的视野。
1.3 CAST系统的结构和功能虽然Casposon和IS200/IS605转座子在结构和元件功能上与CRISPR-Cas系统相似,但其在基因组中的分布与CRISPR-Cas系统并不关联。2017年,Peters等[45]从细菌和古菌中调研到许多嵌入类Tn7转座子的mini CRISPR-Cas系统,该系统由融合的cas8f-cas5f (cas8f-5f)、cas7f和cas6f基因和一个短的CRISPR阵列组成。以保守的cas7f基因和转座酶基因tnsA为探针,通过psiBLAST (position specific iterative BLAST) 可分别得到含有I-F型基因座和含有I-B型基因座的类Tn7转座子[45]。2019年,McDonald等[22]从霍乱弧菌(Vibrio cholerae) 中分离并表征了嵌入类Tn7转座子中的mini I-F CRISPR-Cas基因座。同年,Strecker等[20]表征了贺氏伪枝藻(Scytonema hofmanni) UTEX B 2349和柱孢鱼腥藻(Anabaena cylindrica) PCC 7122的嵌在类Tn7转座子中的V-K亚型CRISPR-Cas系统,此后这类元件被称为CRISPR-associated transposases (CAST)。
CAST类系统基因座包含了类Tn7转座子的基因元件以及CRISPR系统相关元件。如图 1所示,mini I-F型基因座包括tnsA、tnsB、tnsC、cas、tniQ基因和CRISPR阵列,它们共同组成货物基因,被类Tn7转座子的左端(LE) 和右端(RE) 包裹[22]。TniQ通常与Cascade效应复合体一起发挥重要作用[22]。不同于一般的Ⅰ型CRISPR-Cas系统,mini I-F型缺少许多cas基因,甚至缺少编码重要核酸酶Cas3的基因,即便如此,它依旧可以正常行使DNA切割功能,甚至完成转座基因的插入[22]。
来源于贺氏伪枝藻的ShCAST和柱孢鱼腥藻的AcCAST属于V-K型CRISPR-Cas系统,包括tnsB、tnsC、tniQ、cas12k和CRISPR阵列[20]。尽管这种系统缺乏tnsA基因,其整体功能却不受影响。Cas12k是一种无核酸酶催化活性的Cas12变体[20],与mini I-F型系统中Cas3的缺失异曲同工。mini I-F系统和CAST系统能够在Cas效应蛋白和相关转座酶的共同作用下完成转座序列在基因组的整合,因此解析Cascade-TniQ和Cas12k-TniQ主导的转座事件的分子机制能够为该系统工作效率的提高和靶向范围的扩充提供理论依据。
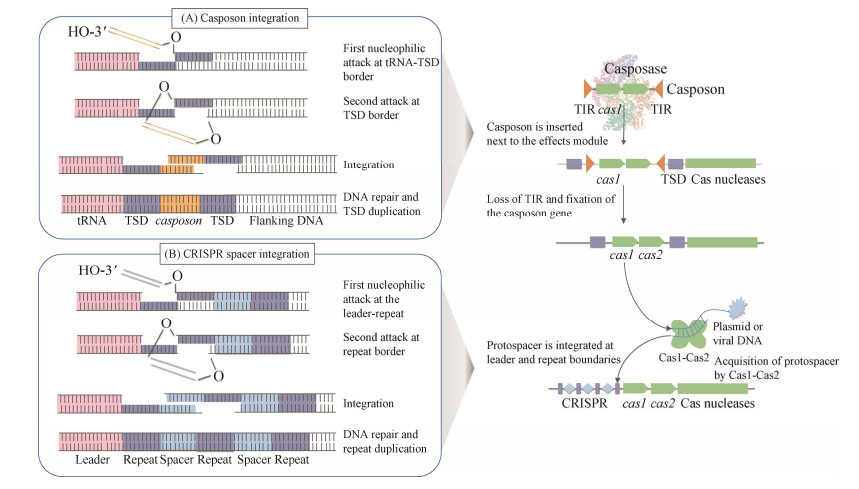
Halpin-Healy等[31]使用低温电子显微镜表征了由霍乱弧菌Tn6677转座子编码的Cascade- TniQ复合物的结构,揭示了这种功能耦合的机制基础(图 3)。天然的Cas8-Cas5融合蛋白组成的大亚基Cas8f可特异性识别crRNA的5′端[31]。6个Cas7亚基形成聚合物,结合crRNA并使核苷酸暴露在外,方便和靶DNA序列完成碱基互补配对[31]。在远离Cas8f的一端,核糖核酸酶Cas6负责加工源自CRISPR基因座的前体RNA转录物[31]。TniQ以二聚体形式存在,用于招募和结合转座元件[31]。当Cascade检测PAM序列时,底物DNA呈锐角弯曲,迫使PAM附近的DNA解链。这允许crRNA结合到dsDNA上,形成一种“R环(R-loop)”结构(图 3)。此时,Cascade-TniQ复合体的构象将发生变化,从而将dsDNA锁定。随后,在TnsB和TnsC的配合下,完成转座子在dsDNA的定点插入[31]。
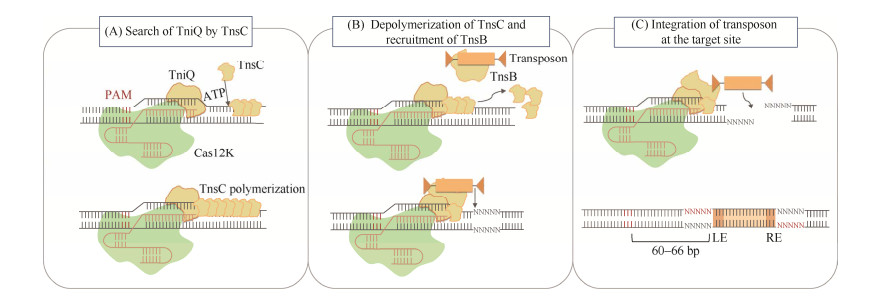
Park等[32]表征了来自ShCAST的TnsC,发现在存在三磷酸腺苷(adenosine triphosphate, ATP) 的情况下,TnsC形成一个连续的螺旋丝,与dsDNA双链的一条DNA链相互作用,从而提供一种靶位搜索机制,直到TnsC遇到Cas12k-TniQ。这时TnsB (与转座子末端结合) 被招募到目标位点,催化其他TnsC聚合体解离[32]。TnsC螺旋丝被分解成一个有限的寡聚体组件,仅留下一个TnsC单体与TniQ复合,因此转座元件插入位置总是与protospacer保持固定距离[32] (图 4)。如果TnsC螺旋丝搜索时遇到了已经整合成功的转座元件,其周围的TnsB会直接触发TnsC解聚,从而形成靶位点免疫现象[32]。上述研究为CAST系统的改造进化奠定了理论基础。
|
| 图 4 ShCAST系统完成基因的靶向整合的示意图 Fig. 4 Schematic diagram of ShCAST system for targeted integration. A:通过TnsC搜索TniQ,TnsC沿DNA方向形成螺旋丝,直至遇到Cas12k-TniQ,TnsC与TniQ形成复合物;B:TnsB的招募和TnsC的解聚,携带TnsB的转座子被招募到目标位点,同时TnsC解聚;C:在目标位点整合转座子,TnsB、TnsC和TniQ共同攻击目标位点并启动转座。转座子插入后,按照碱基互补的原则修复缺口。转座子距PAM 60–66 bp,两侧有5 bp重复 (A) Search of TniQ by TnsC. TnsC forms helical filaments along the DNA direction until encontering Cas12k-TniQ. Then, TnsC forms a complex with TniQ. (B) Recruitment of TnsB and depolymerization of TnsC. TnsB-carrying transposon is recruited to the target site with the depolymerization of TnsC. (C) Integration of transposon at the target site. TnsB, TnsC and TniQ attack the target site and initiate transposition. After insertion of transposon, the gap is repaired according to the principle of base complementarity. Eventually, the transposon is 60–66 bp from the PAM and is flanked by 5 bp repeats. |
| |
先前研究者们普遍认为移动遗传元件(MGE) 和CRISPR-Cas是在长年的宿主-入侵者“军备竞赛”中衍生的防御和反防御系统[27]。但是CAST系统以gRNA为引导完成转座这一“自私”的行为,说明MGE可以捕获宿主防御系统并将它们重新用于元件反防御机制,这符合“guns-for-hire”的概念[29]。2022年,Klompe等[46]通过生物信息学手段探索CAST的多样性。他们以Tn6677的蛋白元件作为访问序列进行了psiBLAST,报告了20个I-F3型系统,这证明“guns-for-hire”的免疫模式在细菌中是广泛存在的。实验证明,在缺少Cas核酸酶活性、不借助同源重组途径的情况下,CAST系统均可以靶向插入DNA[46]。
2 CRISPR相关转座元件高效整合目标DNA片段工程改造生物体以生成高附加值的生物分子的相关研究工作中常涉及到基因簇的遗传操作。CRISPR-Cas系统为基因簇的抓取和组装提供了多种新方法,例如CATCH技术借助Cas9的核酸酶活性和Gibson组装方法可从细菌染色体中克隆100 kb的基因[47-48];本课题组开发的iCOPE (improved CRISPR- Cas12a-assisted one-pot DNA editing) 技术借助FnCas12a变体EP15和EP16,结合消化、连接一锅法以100%的效率完成21 kb基因片段的组装编辑[11]。位点特异性重组(site-specific recombination, SSR) 介导的整合已广泛用于放线菌和其他重要工业微生物中大型代谢途径的稳定扩增[49]。同时,CRISPR-Cas介导的基因工程有效地将外源DNA直接引入细胞并显著促进靶基因的多拷贝整合[50-51]。近几年研究者们发现CRISPR相关转座元件可完成较长基因片段在基因组范围内的靶向整合,为生物合成路径的高效编辑和整合提供了新的技术支持。已经证实CAST系统可将货物基因靶向整合到细菌基因组中[52],TnpB可用于真核细胞的基因编辑[18]。无需HDR或NHEJ,CAST系统可直接将11 kb的货物基因整合到细菌基因组,甚至实现多靶点整合;工程化的dCas9融合转座酶系统(dCas9-transposase) 最高可将100 kb的货物基因直接转座到基因组,弥补了现有基因编辑工具的不足[53-55]。
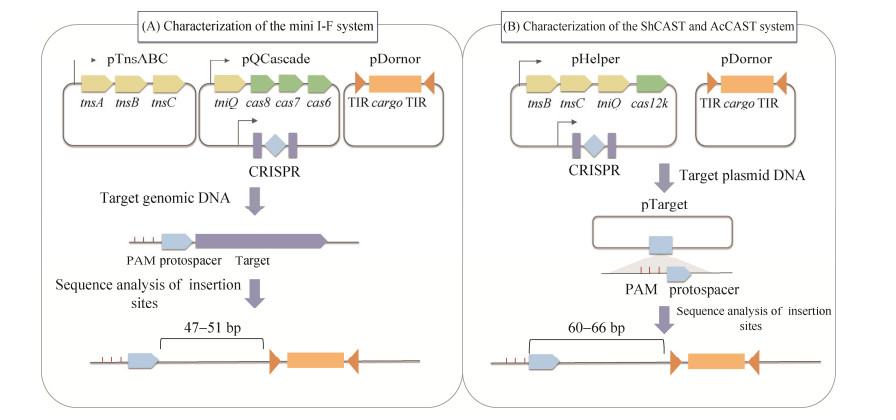
2.1 天然CRISPR系统相关转座元件的DNA整合CAST系统的基因座结构揭示了一种全新的分子机制,即由crRNA引导的靶向转座。将CAST进行模块化重组,可用于细胞内的基因编辑,但各类工具在DNA载量、插入效率、插入方向性和插入特异性均有不同(表 2)。Klompe等[21]将mini I-F型系统设计成基因编辑工具INTEGRATE,该工具包可将感兴趣的货物基因插入大肠杆菌(Escherichia coli) 基因组的目标位点,其中辅助质粒编码TnsA、TnsB和TnsC,pQCascade质粒编码CRISPR阵列、Cascade以及TniQ,供体质粒编码LE-cargo-RE的DNA底物。mini I-F系统识别-CC-的PAM基序,转座子货物基因的插入发生在protospacer末端下游47– 51 bp处,随后会在插入序列的侧翼形成5 bp的重复序列(图 5)。尽管实验表明,该系统最佳货物基因长度为775 bp,但是也能使长达10 kb的货物基因成功转座,这说明该系统具有实现大型基因整合的潜力[21]。研究者观察到INTEGRATE具有很低的脱靶效率(小于5%),且脱靶的位点没有偏好性,但由于其转座插入方向并不可控,降低了其在基因治疗方面的精准度[21]。基于上述研究,Vo等[56]将mini I-F型系统改造为pSPIN (a single-plasmid INTEGRATE),编辑效率是INTEGRATE的2–5倍。pSPIN可以100%的效率完成长达10 kb DNA片段的靶向插入。研究表明,pSPIN虽然也能检测到双向转座,但其转座子右端靠近靶位点(T-RL) 的插入事件显示出明显更强的偏差,且不存在非靶向的插入事件。由于不需要特异性的同源臂,pSPIN系统更容易一步实现多靶点的货物整合,目前可以99%的效率实现3个靶点的基因插入,但每个靶位点需间隔至少1 Mb以上[56]。据报道,ShCAST和AcCAST都需要5′-NGTN-3′ PAM。ShCAST的转座插入主要发生在PAM的3′端60–66 bp之间(图 5),AcCAST货物基因主要插入到PAM的3′端49–56 bp处。ShCAST支持插入长达10 kb的货物DNA,在大肠杆菌基因组中的48个测试靶位点中,29个被检测出成功转座,效率约60%,高于mini I-F系统[20]。ShCAST的转座具有明显的方向性,但存在一定的脱靶率(小于50%),且脱靶位点有一定的偏好性,但仍需要进一步研究Cas12k类的CAST系统在整合过程中引起脱靶现象的分子基础[20]。
| Origin | Name | PAM | Optimal DNA load and efficiency |
Maximum DNA load and efficiency | Insertion direction | Off-target efficiency (%) |
| Mini I-F | INTEGRATE | 5′-CC-3′ | 775 bp (20%−30%) | 10 kb (< 10%) | Non-directional | < 5 |
| pSPIN | 5′-CC-3′ | - | 10 kb (99%) | T-RL | 0 | |
| ShCAST | ShCAST | 5′-NGTN-3′ | 500 bp (50%−75%) | 10 kb (25%) | Directional | < 50 |
| CasTn | 5′-NGTN-3′ | - | 11 kb (91%) | Directional | - |
|
| 图 5 在大肠杆菌中测试CAST系统活性的示意图 Fig. 5 Schematic diagram of testing the activity of the CAST system in Escherichia coli. A:在大肠杆菌中测试mini I-F系统的示意图;B:在大肠杆菌中测试ShCAST和AcCAST的示意图 (A) Schematic diagram of testing the mini I-F system in Escherichia coli. (B) Schematic diagram of testing ShCAST and AcCAST in Escherichia coli. |
| |
上述工作中对CAST系统的货物基因兼容性、PAM偏好性、gRNA序列和基因座要求的探索增强了人们对这类系统的理解。最近,Rodríguez等[52]在3种变形杆菌中表征了ShCAST。他们将sgRNA和LE-cargo-RE克隆至有自杀倾向的pUC质粒上,命名为CasTn,每次编辑只需要消除和更改pUC质粒以实现迭代编辑。结果显示,CasTn在泰国伯克霍尔德菌(Burkholderia thailandensis E264) 中单基因敲除的靶向效率最高可接近100%。此外,CasTn可以在泰国伯克霍尔德菌中实现双靶位点的整合,在恶臭假单胞菌(Pseudomonas putida KT2440) 中完成11 kb的货物基因插入。该研究将CAST拓展至变形杆菌,并证明了CAST系统可以成为实现多靶点和大片段DNA编辑的高效基因编辑工具。然而,细菌基因组中已整合的DNA片段很可能会发生二次转座,因此单一的CRISPR转座酶工具很难在多个位点实现不同DNA片段的整合。为解决此问题,Vo等[56]开发了VchINT和ShoINT系统,其中VchINT来源于mini I-F系统、ShoINT均来源于ShCAST (表 2),发现两种工具联用可有效避免已整合DNA片段的重新转座和整合,大大提高了多位点不同片段整合的特异性。但ShoINT系统有很高的脱靶风险,其在目标位点的整合比例仅有5%–50%。
TnpB在reRNA的引导下,可以识别特定的5′-TTGAT TAM序列,并在其下游15–21 bp处产生粘性末端的缺口。值得注意的是,Karvelis等[18]通过修改reRNA序列将TnpB重新编程为基因组编辑工具,并以10%–20%的效率在人源细胞HEK293T中引入基因突变。
2.2 人工融合dCas9-transposase的DNA整合受到天然CAST的启发,研究者们将dCas9和不同转座酶融合,尝试开发更高效的基因编辑工具。在已知的转座元件中,来自角蝇刺激性嗜血杆菌(horn fly Haematobia irritans) 的Himar1转座子是常用的插入诱变工具,研究表明Himar1最高可以将大于7 kb的货物基因插入基因组[53]。Chen等[53]通过体外研究证明,Himar-dCas9的融合蛋白能够利用sgRNA将货物基因整合到大肠杆菌的质粒中,其转座效率比原始Himar1提高了300倍以上。研究表明,从插入位点到原型间隔区的间距可以改变转座频率,并且相对于目标TA序列的sgRNA位置和方向对转座结果具有一定的影响,这展示了dCas9-transposase独立、可编程、定点DNA插入的新模式。但是,该系统具有高频率的脱靶整合现象,且该方法目前仅在体外研究得到验证,限制了其作为靶向基因组编辑技术的实用性。
Sleeping Beauty (SB) 转座子系统是哺乳动物细胞中一种高效的基因转移工具,已被应用于功能基因组学、癌症基因发现、转基因和基因治疗[57]。基于此,人们希望开发出一种理想的基因治疗工具,以实现人类基因组中有效的定点基因整合。Kovač等[55]通过将SB100X转座酶(SB转座酶的高效突变体) 与dCas9融合,证实了该转座子可在全基因组范围内的整合。数据表明,CRISPR-Cas9的靶向机制可以有效使转座子整合到特定基因组区域,这项技术为未来开发用于精准基因工程的特定基因插入方法提供了基础。
Hew等[54]利用高效的piggyBac* (PB*) 转座子报告了第一个人类细胞中RNA引导转座的案例。piggyBac (PB) 转座酶已经被证明在跨物种和细胞类型的整合中是非常高效的,该系统的承载能力 > 100 kb,所有组件都可以用质粒传递,而人工修饰的突变体PB*的转座过程更加高效。高度活跃的PB*转座酶与dCas9融合,并利用gRNA导向人类细胞CCR5位点,这一策略使货物基因能够直接转座到细胞基因组。该研究团队通过实验发现增加gRNAs的数量可以提高靶向效率,且转座插入位点更加偏好TTAA序列[54]。这些融合表达载体扩展了PB系统在生物医学研究和基因治疗方面的应用。
3 总结与展望本文主要介绍了近几年发现的CRISPR系统相关的转座元件,并描述了这类转座元件与CRISPR-Cas系统在结构和功能上的相似特征。从转座元件中与CRISPR-Cas相关的同源基因或同源结构域出发,利用生物信息学手段,可以发现这类元件具有保守基因或保守结构域,比如Casposon中的cas1、TnpB和IscB具有保守的RuvC结构域。因此借助生物信息学手段,结合转座子末端特征可以更充分地挖掘这类基因操作元件。同时,研究者们的相关工作为CRISPR-Cas系统和转座子之间的生物多样性和神秘的遗传进化关系提供了很好的事实依据,有利于进一步了解细菌和古菌复杂的免疫防御机制。因此,阐明微生物移动元件和防御工具之间相互作用的多样性和复杂性,以及发展其共同进化的一般理论是未来几十年的研究方向。
本文中总结的几种CRISPR系统相关的转座元件具有扩展基因编辑工具包和扩充基因编辑功能的潜力,有助于提高研究效率、降低研究成本。首先是非自主转座子编码的TnpB、IscB等核酸酶的功能已经得到验证,它们相对于Cascade或Cas9、Cas12具有更小的蛋白尺寸,其改造的基因编辑工具更容易被递送至宿主细胞,大大提高了基因操作效率,尤其是非模式菌株中的基因操作。利用TnpB、IscB更容易通过修饰以实现精准地删除或大片段DNA插入、使染色体发生大面积重排等操作。因此,将TnpB、IscB类工具进行模块化和工程化改造的进程将在未来飞速发展。其次是CAST系统的相关研究展示了这类新型系统在基因编辑上的出色能力。CAST系统的最大优势是可以在不利用基因组DNA双链断裂和DNA损伤修复机制的情况下,直接通过RNA引导的转座酶进行目的DNA的插入。这表明可以利用CAST系统在基因组范围内靶向插入基因片段,辅助细胞代谢工程的研究。未来或许可以利用CAST技术辅助基因治疗,将稳定的转基因整合到基因组安全港区域(genomic safe harbors, GSHs) 以治疗人类遗传疾病。但CAST系统的转座特性也存在一定的缺陷。首先由于转座子末端序列与转座酶之间特异性识别是必不可少的[56],因此CAST不适用于点突变、无痕编辑等应用,需要基于CRISPR-Cas9和CRISPR-Cas12a的技术来实现,例如Base editor介导的点突变技术[58]、结合CRISPR和Red同源重组产生的无痕编辑技术CRMAGE (CRISPR/Cas9 and λ Red recombineering based MAGE technology)[59]以及dCas9衍生的CRISPR干扰(CRISPR interference, CRISPRi)[60]和CRISPR激活(CRISPR activation, CRISPRa)[61]技术。未来可以通过尝试缩小末端长度和将末端序列转化为功能性基因对这类系统进行改进。其次迭代DNA插入或多位点DNA插入需要考虑转座的靶标免疫和预先存在的转座插入发生再次转座的风险,虽然可以联用不同种类的CAST系统以规避风险,但仍存在一定的脱靶效应[56]。以现存的CAST系统为访问序列的psiBLAST策略可以在未来辅助挖掘更多的CAST系统,可以设想存在一组完全兼容的转座酶正交系统能够出色完成DNA的迭代插入。CAST系统分子结构的表征指出靶标免疫和TnsC螺旋丝的搜索机制有关,未来可基于此对靶标免疫现象进行进一步的研究。同时,TniQ、TnsC和Cas蛋白之间的相互作用机制决定了DNA插入窗口的大小,未来可以在分子层面上实现CAST系统的模块化重排和改造进化实现更精准的DNA插入,甚至获得更加高效的基因编辑能力、更加广泛的靶向范围和更加多样的基因操作功能。目前,天然的CAST系统并不能有效用于哺乳动物细胞基因编辑,但是dCas9-transposase系统可以在人类细胞实现大片段的基因操作。在未来,使用CAST系统实现如人类细胞等真核基因组编辑,也将标志着这些技术发展的重大突破。
| [1] |
Gasiunas G, Barrangou R, Horvath P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. PNAS, 2012, 109(39): E2579-E2586.
|
| [2] |
Jinek M, East A, Cheng A, et al. RNA-programmed genome editing in human cells. eLife, 2013, 2: e00471. DOI:10.7554/eLife.00471
|
| [3] |
Zhang F, Wen Y, Guo X. CRISPR/Cas9 for genome editing: progress, implications and challenges. Hum Mol Genet, 2014, 23(R1): R40-R46. DOI:10.1093/hmg/ddu125
|
| [4] |
Anzalone AV, Koblan LW, Liu DR. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol, 2020, 38(7): 824-844. DOI:10.1038/s41587-020-0561-9
|
| [5] |
Manghwar H, Lindsey K, Zhang XL, et al. CRISPR/Cas system: recent advances and future prospects for genome editing. Trends Plant Sci, 2019, 24(12): 1102-1125. DOI:10.1016/j.tplants.2019.09.006
|
| [6] |
Kleinstiver BP, Prew MS, Tsai SQ, et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature, 2015, 523(7561): 481-485. DOI:10.1038/nature14592
|
| [7] |
Nishimasu H, Shi X, Ishiguro S, et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science, 2018, 361(6408): 1259-1262. DOI:10.1126/science.aas9129
|
| [8] |
Hu JH, Miller SM, Geurts MH, et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature, 2018, 556(7699): 57-63. DOI:10.1038/nature26155
|
| [9] |
Miller SM, Wang TN, Randolph PB, et al. Continuous evolution of SpCas9 variants compatible with non-G PAMs. Nat Biotechnol, 2020, 38(4): 471-481. DOI:10.1038/s41587-020-0412-8
|
| [10] |
Walton RT, Christie KA, Whittaker MN, et al. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science, 2020, 368(6488): 290-296. DOI:10.1126/science.aba8853
|
| [11] |
Wang LP, Wang HJ, Liu HY, et al. Improved CRISPR-Cas12a-assisted one-pot DNA editing method enables seamless DNA editing. Biotechnol Bioeng, 2019, 116(6): 1463-1474. DOI:10.1002/bit.26938
|
| [12] |
Zhang J, Zhang D, Zhu J, et al. Efficient multiplex genome editing in Streptomyces via engineered CRISPR-Cas12a systems. Front Bioeng Biotechnol, 2020, 8: 726. DOI:10.3389/fbioe.2020.00726
|
| [13] |
Zhao QY, Wang LP, Luo YZ. Recent advances in natural products exploitation in Streptomyces via synthetic biology. Eng Life Sci, 2019, 19(6): 452-462. DOI:10.1002/elsc.201800137
|
| [14] |
Zhang XY, Gu SJ, Zheng XY, et al. A novel and efficient genome editing tool assisted by CRISPR-Cas12a/Cpf1 for Pichia pastoris. ACS Synth Biol, 2021, 10(11): 2927-2937. DOI:10.1021/acssynbio.1c00172
|
| [15] |
张孟思, 朱德康, 汪铭书. 细菌插入序列中的转座酶和转座机制. 中国生物化学与分子生物学报, 2018, 34(10): 1057-1064. Zhang MS, Zhu DK, Wang MS. Transposases in bacterial insertion sequences and their transposition mechanisms. Chin J Biochem Mol Biol, 2018, 34(10): 1057-1064 (in Chinese). DOI:10.13865/j.cnki.cjbmb.2018.10.06 |
| [16] |
Krupovic M, Makarova KS, Forterre P, et al. Casposons: a new superfamily of self-synthesizing DNA transposons at the origin of prokaryotic CRISPR-Cas immunity. BMC Biol, 2014, 12: 36. DOI:10.1186/1741-7007-12-36
|
| [17] |
Béguin P, Charpin N, Koonin EV, et al. Casposon integration shows strong target site preference and recapitulates protospacer integration by CRISPR-Cas systems. Nucleic Acids Res, 2016, 44(21): 10367-10376.
|
| [18] |
Karvelis T, Druteika G, Bigelyte G, et al. Transposon-associated TnpB is a programmable RNA-guided DNA endonuclease. Nature, 2021, 599(7886): 692-696. DOI:10.1038/s41586-021-04058-1
|
| [19] |
Altae-Tran H, Kannan S, Demircioglu FE, et al. The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonucleases. Science, 2021, 374(6563): 57-65. DOI:10.1126/science.abj6856
|
| [20] |
Strecker J, Ladha A, Gardner Z, et al. RNA-guided DNA insertion with CRISPR-associated transposases. Science, 2019, 365(6448): 48-53. DOI:10.1126/science.aax9181
|
| [21] |
Klompe SE, Vo PLH, Halpin-Healy TS, et al. Transposon-encoded CRISPR-Cas systems direct RNA-guided DNA integration. Nature, 2019, 571(7764): 219-225. DOI:10.1038/s41586-019-1323-z
|
| [22] |
McDonald ND, Regmi A, Morreale DP, et al. CRISPR-Cas systems are present predominantly on mobile genetic elements in Vibrio species. BMC Genomics, 2019, 20(1): 105. DOI:10.1186/s12864-019-5439-1
|
| [23] |
Faure G, Shmakov SA, Yan WX, et al. CRISPR-Cas in mobile genetic elements: counter-defence and beyond. Nat Rev Microbiol, 2019, 17(8): 513-525. DOI:10.1038/s41579-019-0204-7
|
| [24] |
Krupovic M, Béguin P, Koonin EV. Casposons: mobile genetic elements that gave rise to the CRISPR-Cas adaptation machinery. Curr Opin Microbiol, 2017, 38: 36-43. DOI:10.1016/j.mib.2017.04.004
|
| [25] |
Koonin EV, Makarova KS. Mobile genetic elements and evolution of CRISPR-Cas systems: all the way there and back. Genome Biol Evol, 2017, 9(10): 2812-2825. DOI:10.1093/gbe/evx192
|
| [26] |
He SS, Guynet C, Siguier P, et al. IS200/IS605 family single-strand transposition: mechanism of IS608 strand transfer. Nucleic Acids Res, 2013, 41(5): 3302-3313. DOI:10.1093/nar/gkt014
|
| [27] |
Kapitonov VV, Makarova KS, Koonin EV. ISC, a novel group of bacterial and archaeal DNA transposons that encode Cas9 homologs. J Bacteriol, 2015, 198(5): 797-807.
|
| [28] |
Pasternak C, Dulermo R, Ton-Hoang B, et al. ISDra2 transposition in Deinococcus radiodurans is downregulated by TnpB. Mol Microbiol, 2013, 88(2): 443-455. DOI:10.1111/mmi.12194
|
| [29] |
Koonin EV, Krupovic M. Evolution of adaptive immunity from transposable elements combined with innate immune systems. Nat Rev Genet, 2015, 16(3): 184-192. DOI:10.1038/nrg3859
|
| [30] |
Hickman AB, Dyda F. CRISPR-Cas immunity and mobile DNA: a new superfamily of DNA transposons encoding a Cas1 endonuclease. Mob DNA, 2014, 5: 23. DOI:10.1186/1759-8753-5-23
|
| [31] |
Halpin-Healy TS, Klompe SE, Sternberg SH, et al. Structural basis of DNA targeting by a transposon-encoded CRISPR-Cas system. Nature, 2020, 577(7789): 271-274. DOI:10.1038/s41586-019-1849-0
|
| [32] |
Park JU, Tsai AWL, Mehrotra E, et al. Structural basis for target site selection in RNA-guided DNA transposition systems. Science, 2021, 373(6556): 768-774. DOI:10.1126/science.abi8976
|
| [33] |
Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829
|
| [34] |
Krupovic M, Shmakov S, Makarova KS, et al. Recent mobility of casposons, self-synthesizing transposons at the origin of the CRISPR-Cas immunity. Genome Biol Evol, 2016, 8(2): 375-386. DOI:10.1093/gbe/evw006
|
| [35] |
Sternberg SH, Richter H, Charpentier E, et al. Adaptation in CRISPR-Cas systems. Mol Cell, 2016, 61(6): 797-808. DOI:10.1016/j.molcel.2016.01.030
|
| [36] |
Mohanraju P, Makarova KS, Zetsche B, et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems. Science, 2016, 353(6299): aad5147. DOI:10.1126/science.aad5147
|
| [37] |
Amitai G, Sorek R. CRISPR-Cas adaptation: insights into the mechanism of action. Nat Rev Microbiol, 2016, 14(2): 67-76. DOI:10.1038/nrmicro.2015.14
|
| [38] |
Bao WD, Jurka J. Homologues of bacterial TnpB_IS605 are widespread in diverse eukaryotic transposable elements. Mob DNA, 2013, 4(1): 12. DOI:10.1186/1759-8753-4-12
|
| [39] |
Berger B, Haas D. Transposase and cointegrase: specialized transposition proteins of the bacterial insertion sequence IS21 and related elements. Cell Mol Life Sci, 2001, 58(3): 403-419. DOI:10.1007/PL00000866
|
| [40] |
Chandler M, De La Cruz F, Dyda F, et al. Breaking and joining single-stranded DNA: the HUH endonuclease superfamily. Nat Rev Microbiol, 2013, 11(8): 525-538. DOI:10.1038/nrmicro3067
|
| [41] |
Han CG, Shiga Y, Tobe T, et al. Structural and functional characterization of IS679 and IS66-family elements. J Bacteriol, 2001, 183(14): 4296-4304. DOI:10.1128/JB.183.14.4296-4304.2001
|
| [42] |
Siguier P, Gourbeyre E, Chandler M. Bacterial insertion sequences: their genomic impact and diversity. FEMS Microbiol Rev, 2014, 38(5): 865-891. DOI:10.1111/1574-6976.12067
|
| [43] |
Koonin EV, Makarova KS, Zhang F. Diversity, classification and evolution of CRISPR-Cas systems. Curr Opin Microbiol, 2017, 37: 67-78. DOI:10.1016/j.mib.2017.05.008
|
| [44] |
Makarova KS, Wolf YI, Iranzo J, et al. Evolutionary classification of CRISPR-Cas systems: a burst of class 2 and derived variants. Nat Rev Microbiol, 2020, 18(2): 67-83. DOI:10.1038/s41579-019-0299-x
|
| [45] |
Peters JE, Makarova KS, Shmakov S, et al. Recruitment of CRISPR-Cas systems by Tn7-like transposons. PNAS, 2017, 114(35): E7358-E7366.
|
| [46] |
Klompe SE, Jaber N, Beh LY, et al. Evolutionary and mechanistic diversity of type I-F CRISPR-associated transposons. Mol Cell, 2022, 82(3): 616-628.e5. DOI:10.1016/j.molcel.2021.12.021
|
| [47] |
Jiang WJ, Zhao XJ, Gabrieli T, et al. Cas9-assisted targeting of chromosome segments CATCH enables one-step targeted cloning of large gene clusters. Nat Commun, 2015, 6: 8101. DOI:10.1038/ncomms9101
|
| [48] |
Jiang WJ, Zhu TF. Targeted isolation and cloning of 100-kb microbial genomic sequences by Cas9-assisted targeting of chromosome segments. Nat Protoc, 2016, 11(5): 960-975. DOI:10.1038/nprot.2016.055
|
| [49] |
Li L, Liu XC, Wei KK, et al. Synthetic biology approaches for chromosomal integration of genes and pathways in industrial microbial systems. Biotechnol Adv, 2019, 37(5): 730-745. DOI:10.1016/j.biotechadv.2019.04.002
|
| [50] |
Liu ZH, Liang YY, Ang EL, et al. A new era of genome integration-simply cut and paste!. ACS Synth Biol, 2017, 6(4): 601-609. DOI:10.1021/acssynbio.6b00331
|
| [51] |
Shi SB, Liang YY, Zhang MM, et al. A highly efficient single-step, markerless strategy for multi-copy chromosomal integration of large biochemical pathways in Saccharomyces cerevisiae. Metab Eng, 2016, 33: 19-27. DOI:10.1016/j.ymben.2015.10.011
|
| [52] |
Rodríguez LT, Ellington AJ, Reisch CR. Broad-host- range mutagenesis with CRISPR-associated transposase. bioRxiv, 2022, DOI: 10.1101/2022.01.19.475551.
|
| [53] |
Chen SP, Wang HH. An engineered cas-transposon system for programmable and site-directed DNA transpositions. CRISPR J, 2019, 2(6): 376-394. DOI:10.1089/crispr.2019.0030
|
| [54] |
Hew BE, Sato R, Mauro D, et al. RNA-guided piggyBac transposition in human cells. Synth Biol (Oxf), 2019, 4(1): ysz018. DOI:10.1093/synbio/ysz018
|
| [55] |
Kovač A, Miskey C, Menzel M, et al. RNA-guided retargeting of Sleeping Beauty transposition in human cells. eLife, 2020, 9: e53868. DOI:10.7554/eLife.53868
|
| [56] |
Vo PLH, Ronda C, Klompe SE, et al. CRISPR RNA-guided integrases for high-efficiency, multiplexed bacterial genome engineering. Nat Biotechnol, 2021, 39(4): 480-489. DOI:10.1038/s41587-020-00745-y
|
| [57] |
Querques I, Mades A, Zuliani C, et al. A highly soluble Sleeping Beauty transposase improves control of gene insertion. Nat Biotechnol, 2019, 37(12): 1502-1512. DOI:10.1038/s41587-019-0291-z
|
| [58] |
Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644
|
| [59] |
Ronda C, Pedersen LE, Sommer MOA, et al. CRMAGE: CRISPR optimized MAGE recombineering. Sci Rep, 2016, 6: 19452. DOI:10.1038/srep19452
|
| [60] |
Larson MH, Gilbert LA, Wang XW, et al. CRISPR interference (CRISPRi) for sequence-specific control of gene expression. Nat Protoc, 2013, 8(11): 2180-2196. DOI:10.1038/nprot.2013.132
|
| [61] |
Chavez A, Scheiman J, Vora S, et al. Highly efficient Cas9-mediated transcriptional programming. Nat Methods, 2015, 12(4): 326-328. DOI:10.1038/nmeth.3312
|
 2022, Vol. 38
2022, Vol. 38