中国科学院微生物研究所、中国微生物学会主办
文章信息
- 陈翠玉, 庞亚如, 陈泉冰, 李春, 吕波
- CHEN Cuiyu, PANG Yaru, CHEN Quanbing, LI Chun, LÜ Bo
- 环氧角鲨烯环化酶在三萜化合物生物合成中的进展
- Oxidosqualene cyclases in triterpenoids biosynthesis: a review
- 生物工程学报, 2022, 38(2): 443-459
- Chinese Journal of Biotechnology, 2022, 38(2): 443-459
- 10.13345/j.cjb.210169
-
文章历史
- Received: February 27, 2021
- Accepted: May 18, 2021
2. 清华大学 化学工程系, 北京 100084
2. Department of Chemical Engineering, Tsinghua University, Beijing 100084, China
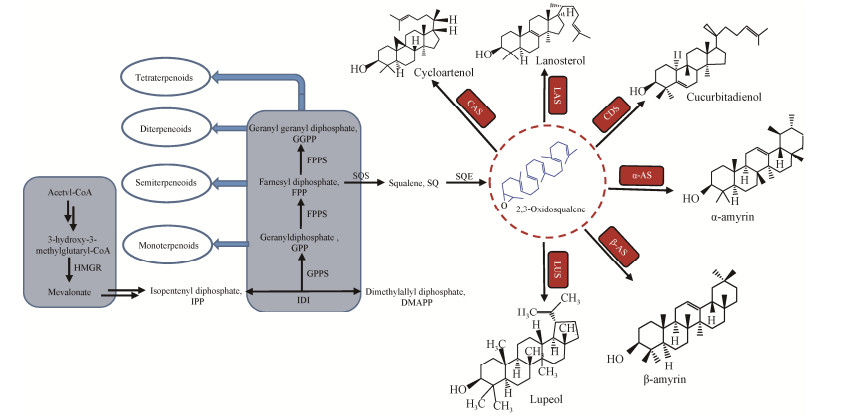
萜类化合物(terpenoids) 是自然界发现的结构最多样化的化合物之一,迄今为止,已报道的萜类化合物已超过8万种[1]。萜类化合物具有多种生物功能和药理活性,已被广泛应用于医疗、保健及食品加工等多个领域,如常用的口腔卫生产品薄荷醇(menthol)、抗疟疾药物青蒿素(artemisinin) 以及具有抗肿瘤和保肝护肝作用的熊果酸(ursolic acid, UA)、齐墩果酸(oleanolic acid, OA) 和甘草次酸(glycyrrhetinic acid, GA) 等[2]。萜类化合物的结构多样性源于异戊二烯(isoprene) 单元,包括C5结构的异戊烯焦磷酸(isopentenyl pyrophosphate, IPP) 和二甲基烯丙基焦磷酸(dimethylallyl pyrophosphate, DMAPP)。植物合成IPP和DMAPP存在两种不同的途径:甲羟戊酸途径(mevalonate pathway, MVA途径) 和甲基赤藓糖醇磷酸化途径(methylerythritol phosphate pathway, MEP途径)。IPP和DMAPP在异戊烯基转移酶(prenyltransferase) 的作用下合成萜烯的一系列前体:牦牛儿基焦磷酸(geranyl diphosphate, GPP)、法尼基焦磷酸(famesyl diphosphate, FPP) 和牦牛儿牦牛儿基焦磷酸(geranylgeranyl diphosphate, GGPP)。最后,GPP、FPP以及GGPP在不同萜烯环化酶的作用下,生成一系列单萜(monoterpenoids) C10、倍半萜(semiterpeneoids) C15、二萜(diterpeneoids) C20、三萜(triterpenoids) C30、四萜(tetraterpenoids) C40以及多萜等不同结构的萜类化合物(图 1)。其中,已知的三萜类化合物约有2万余种,是萜类化合物中数量和种类最多的一类[3]。环氧角鲨烯环化酶(oxidosqualene cyclases, OSCs) 可催化以FPP为前体生成的2, 3-氧化鲨烯(2, 3-oxidosqualene),形成多种甾醇和三萜类物质,如图 1所示。真菌和动物中的羊毛甾醇(lanoaterol),植物来源的环阿屯醇(cyloartenol)、葫芦二烯醇(cucurbitadienol)、α-香树脂醇(α-amyrin)、β-香树脂醇(β-amyrin) 以及羽扇醇(lupeol) 等,均源于OSCs酶催化2, 3-氧化鲨烯的环化反应,之后,通过各种氧化和糖基化作用形成功能多样的萜烯类衍生物。
|
| 图 1 萜类和甾醇化合物的生物合成途径 Fig. 1 Biosynthesis pathway of terpenoids and sterols. Multiple arrows represent more than one reaction step; 3-hydroxy-3-methylglutaryl coenzyme-A reductase: HMGR; isopentenyl diphosphate isomerase: IDI; geranyl diphosphate synthase: GPPS; farnesyl diphosphate synthase: FPPS; squalene synthase: SQS; squalene epoxidase: SQE; cyloartenol synthases: CAS; lanoaterol synthases: LAS; cucurbitadienol synthases: CDS; α-amyrin synthases: α-AS; β-amyrin synthases: β-AS; lupeol synthases: LUS. |
| |
OSCs酶的多样性是三萜生物合成多样性的基础。目前,已在OSCs基因的分离、纯化、克隆及表达分析上取得了一定的进展,但是对于OSCs酶催化2, 3-氧化鲨烯发生环化反应的机理尚不清楚,OSCs酶序列多样性与产物多样性的关系仍不清晰。本文对近几年OSCs酶的催化表征、基因与蛋白质的分子进化关系、OSCs酶结构基础、蛋白质模拟与分子计算等方面进行了阐述,旨在为后续的三萜环化酶的蛋白质工程和代谢工程的研究奠定基础。
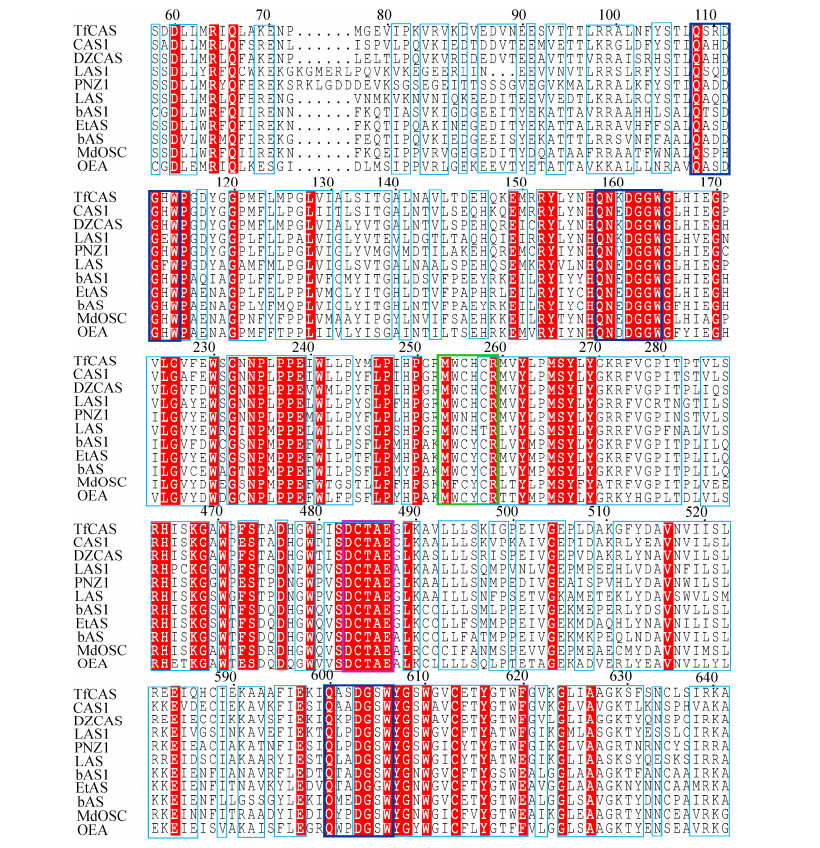
1 环氧角鲨烯环化酶的催化表征目前已经从植物中鉴定出80多种合成三萜的OSCs的功能特征,表 1列出部分功能已鉴定的OSCs。在OSCs酶催化2, 3-氧化鲨烯的环化过程中,初始底物的折叠步骤至关重要,因为这使底物易于遵循特定的环化路径。椅-船-椅(chair-boat-chair, CBC) 构象产生原甾醇阳离子(protosteryl cation) 中间体,然后在环阿屯醇合酶(cyloartenol synthases, CAS)、羊毛甾醇合酶(lanoaterol synthases, LAS) 和葫芦二烯醇合酶(cucurbitadienol synthases, CDS) 等OSCs的作用下,形成环阿屯醇、羊毛甾醇和葫芦二烯醇等四环三萜骨架(6-6-6-5)[4] (图 1)。环阿屯醇合成酶,如来自葫芦巴(Trigonella foenum-graecum L) 的TfCAS和来自水稻(Oryza sativa) 的OsOSC2[5] (表 1),能够将2, 3-氧化鲨烯转化为环阿屯醇,并以CBC构象作为类固醇生物合成的前体[3],在植物甾醇和甾体皂苷的生物合成中起着重要的作用。通过S-腺苷-L-蛋氨酸固醇C24甲基转移酶1 (S-adenosyl-L-methionine- sterol-C24-methyltransferase 1, SMT1) 和细胞色素P450 (cytochrome P450 enzymes, CYP450酶) 的催化作用,环阿屯醇可进一步转化为植物甾醇(phytosterols)[6]、胆固醇(cholesterol)[7],再进一步羟基化和环化得到具有螺旋体结构的甾体类薯蓣皂苷元(diosgenin)[8]。一些植物的OSCs酶通过CBC构象形成环阿屯醇和羊毛甾醇以外的分子。如来自西葫芦(Cucurbita pepo) 的CPQ[9] (表 1),能够催化2, 3-氧化鲨烯形成葫芦二烯醇(cucurbitadienol),是葫芦科植物中葫芦烷型四环三萜类化合物的基本前体,其在生物体内能进一步被酶修饰形成罗汉果甜苷(mogrosides) 和葫芦素(cucurbitacins) 等化合物。另外,来自水稻的OsPS[10],能够通过CBC构象形成帕克醇(parkeol)。
| OSCs | Species | Gene number | Catalytic function | Fold | Ring system | References |
| TfCAS | Trigonella foenum-graecum L. | KX148475.1 | Cycloartenol | CBC | 6/6/6/5 | – |
| CAS1 | Arabidopsis thaliana | At2g07050 | Cycloartenola | CBC | 6/6/6/5 | [12] |
| DzCAS | Dioscorea zingiberensis | AM697885 | Cycloartenol | CBC | 6/6/6/5 | [5] |
| OsOSC2 | Oryza sativa | AK121211.1 | Cycloartenol | CBC | 6/6/6/5 | [10] |
| ACX | Adiantum capillus-veneris | AB368375.1 | Cycloartenol | CBC | 6/6/6/5 | [13] |
| CASPEA | Pisum sativum | D89619.1 | Cycloartenol | CBC | 6/6/6/5 | [14] |
| CS1 | Avena strigosa | AJ311790.1 | Cycloartenol*, a | CBC | 6/6/6/5 | [15] |
| BPX1 | Betula platyphylla | AB055509.1 | Cycloartenol | CBC | 6/6/6/5 | [16] |
| CAS | Kandelia candel | AB292609.1 | Cycloartenol | CBC | 6/6/6/5 | [17] |
| PNX | Panax ginseng | AB009029.1 | Cycloartenol | CBC | 6/6/6/5 | [18] |
| CAS1 | Abies magnififica | AF216755.1 | Cycloartenol | CBC | 6/6/6/5 | – |
| BPX2 | Betula platyphylla | AB055510.1 | Cycloartenol | CBC | 6/6/6/5 | [16] |
| CAS1 | Glycyrrhiza glabra | AB025968.1 | Cycloartenol | CBC | 6/6/6/5 | [19] |
| CsOSC1 | Costus speciosus | AB058507.1 | Cycloartenol | CBC | 6/6/6/5 | [20] |
| CPX | Cucurbita pepo | AB116237.1 | Cycloartenola | CBC | 6/6/6/5 | [9] |
| CAS1 | Luffa cylindrica | AB033334.1 | Cycloartenol | CBC | 6/6/6/5 | [21] |
| CAS | Polypodiodes niponica | AB530328.1 | Cycloartenol | CBC | 6/6/6/5 | – |
| OSC5 | Lotus japonicus | AB181246.1 | Cycloartenol | CBC | 6/6/6/5 | [22] |
| CAS | Rhizophora stylosa | AB292608.1 | Cycloartenol | CBC | 6/6/6/5 | [17] |
| RcCAS | Ricinus communis | DQ268870.1 | Cycloartenola | CBC | 6/6/6/5 | [23] |
| LSS | Arabidopsis thaliana | DQ508794.2 | Lanosterol | CBC | 6/6/6/5 | [24] |
| PNZ1 | Panax ginseng | AB009031.1 | Lanosterol | CBC | 6/6/6/5 | [25] |
| LAS | Lotus japonicus | AB244671.1 | Lanosterol | CBC | 6/6/6/5 | [26] |
| CPQ | Cucurbita pepo | AB116238.1 | Cucurbitadienol | CBC | 6/6/6/5 | [9] |
| SgCbQ | Siraitia grosvenorii | HQ128567.1 | Cucurbitadienol | CBC | 6/6/6/5 | [27] |
| OsPs | Oryza sativa | AK066327.1 | Parkeol | CBC | 6/6/6/5 | [10] |
| CrMAS | Catharanthus roseus | AEX99665.1 | ≈80% α-Amyrin | CCC | 6/6/6/6/6 | [28] |
| MdOSC1 | Malus×domestica | FJ032006.1 | ≈80% α-Amyrin | CCC | 6/6/6/6/6 | [29] |
| OEA | Olea europaea | AB291240.1 | α-Amyrin | CCC | 6/6/6/6/6 | [30] |
| GgbAS1 | Glycyrrhiza glabra | AB037203.1 | β-Amyrin | CCC | 6/6/6/6/6 | [31] |
| EtAS | Euphorbia tirucalli | AB206469.1 | β-Amyrin | CCC | 6/6/6/6/6 | [32] |
| bAS | Artemisia annua | EU330197.1 | β-Amyrin | CCC | 6/6/6/6/6 | [33] |
| bAS1 | Nigella sativa | FJ013228.1 | β-Amyrina | CCC | 6/6/6/6/6 | [34] |
| OXA1 | Aster sedifolius | AY836006.1 | β-Amyrin | CCC | 6/6/6/6/6 | [35] |
| AMY1 | Lotus japonicus | AB181244.1 | β-Amyrin | CCC | 6/6/6/6/6 | [22] |
| BPY | Betula platyphylla | AB055512.1 | β-Amyrin | CCC | 6/6/6/6/6 | [16] |
| bAS | Bruguiera gymnorrhiza | AB289585.1 | β-Amyrin | CCC | 6/6/6/6/6 | [17] |
| AS | Euphorbia tirucalli | AB206469.1 | β-Amyrin | CCC | 6/6/6/6/6 | [36] |
| PSY | Pisum sativum | AB034802.1 | β-Amyrin | CCC | 6/6/6/6/6 | [14] |
| bAS1 | Avena strigosa | AJ311789.1 | β-Amyrinb | CCC | 6/6/6/6/6 | [15] |
| AMYI | Medicago truncatula | AJ430607.1 | β-Amyrin | CCC | 6/6/6/6/6 | [37] |
| PNY1 | Panax ginseng | AB009030.1 | β-Amyrin | CCC | 6/6/6/6/6 | [18] |
| PNY2 | Panax ginseng | AB014057.1 | β-Amyrin | CCC | 6/6/6/6/6 | [18] |
| bAS | Polygala tenuifolia | EF107623.1 | β-Amyrin | CCC | 6/6/6/6/6 | – |
| LUP4 | Arabidopsis thaliana | At1g78950 | β-Amyrin | CCC | 6/6/6/6/6 | [38] |
| TTS1 | Solanum lycopersicum | HQ266579.1 | β-Amyrin | CCC | 6/6/6/6/6 | [39] |
| LUS | Bruguiera gymnorrhiza | AB289586.1 | Lupeol | CCC | 6/6/6/6/5 | [17] |
| LUS1 | Glycyrrhiza glabra | AB116228.1 | Lupeold | CCC | 6/6/6/6/5 | [19] |
| RcLUS | Ricinus communis | DQ268869.1 | Lupeole | CCC | 6/6/6/6/5 | [23] |
| KdLUS | Kalanchoe daigremontiana | HM623871.1 | Lupeolc | CCC | 6/6/6/6/5 | [40] |
| OSC3 | Lotus japonicus | AB181245.1 | Lupeold | CCC | 6/6/6/6/5 | [22] |
| OEW | Olea europaea | AB025343.1 | Lupeol | CCC | 6/6/6/6/5 | [41] |
| TRW | Taraxacum offificinale | AB025345.1 | Lupeol | CCC | 6/6/6/6/5 | [41] |
| BPW | Betula platyphylla | AB055511.1 | Lupeol | CCC | 6/6/6/6/5 | [16] |
| THAS1 | Arabidopsis thaliana | AY327541.1 | Thalianolb | CCC | 6/6/5 | [42] |
| KdGLS | Kalanchoe daigremontiana | HM623869.1 | Glutinolc | CCC | 6/6/6/6/6 | [40] |
| KdFRS | Kalanchoe daigremontiana | HM623870.1 | Friedelinc | CCC | 6/6/6/6/6 | [40] |
| KdTAS | Kalanchoe daigremontiana | HM623868.1 | Taraxerolc | CCC | 6/6/6/6/6 | [40] |
| PNA | Panax ginseng | AB265170.1 | Dammarenediol Ⅱ | CCC | 6/6/6/5 | [43] |
| OsIAS | Oryza sativa | AK067451.1 | Isoarborinol | CBC | 6/6/6/6/5 | [4] |
| TTS2 | Solanum lycopersicum | HQ266580.1 | Mixed products3, 1, 5 | CCC | 6/6/6/6/6 | [39] |
| KcMS | Kandelia candel | AB257507.1 | Mixed products2, 3, 1 | CCC | [44] | |
| PEN6 | Arabidopsis thaliana | AB274959.1 | Mixed products5, 2, 3, UC | CCC | [45] | |
| AMY2 | Lotus japonicus | AF478455.1 | Mixed products2, 1, UC | CCC | [37] | |
| CsOSC2 | Costus speciosus | AB058508.1 | Mixed products2, 5, 1, UC | CCC | [20] | |
| RsM1 | Rhizophora stylosa | AB263203.1 | Mixed products5, 1, 2 | CCC | [17] | |
| Note: superscript lowercase letters denote respective OSC transcript abundance in specific tissues as determined by reverse transcription polymerase chain reaction (RT-PCR), semiquantitative PCR, or northern blot analysis. a: all tissues; b: root tip (epidermis); c: leaf and fruit epidermis; d: nodules; e: epicuticular layers of leaves and stems. Superscript numbers denote the products of OSCs that generate mixed products, with the numbers for each OSC listed in the order of the products’ abundance in gas chromatography–mass spectrometry (most abundant product listed first). 1: β-amyrin; 2: lupeol; 3: α- amyrin; 4: δ-amyrin; 5: Other cyclization products; UC: additional uncharacterized products. Asterisks denote a predicted product. Abbreviations: CB, chair-boat; CBC, chair-boat-chair; CCC, chair-chair-chair. | ||||||
此外,OSCs酶也可通过椅-椅-椅(chair- chair-chair, CCC) 构象环化形成达玛烯基阳离子(dammarenyl cation),随后产生不同的五环三萜骨架(6-6-6-6-5),例如β-香树脂醇、α-香树脂醇和羽扇醇系列及一系列其他的2, 3-氧化鲨烯环化产物[4] (表 1)。如α-香树脂醇合酶(α-amyrin synthases, α-AS) 和β-香树脂醇合酶(β-amyrin synthases, β-AS) 催化2, 3-氧化鲨烯分别形成α-香树脂醇和β-香树脂醇,这两种产物为同分异构体,它们均具有多种药理活性,分别是UA和OA的关键前体,在食品和医疗行业具有广泛的应用[46]。羽扇醇合成酶(lupeol synthases, lUS),如来自白桦(Betula platyphylla) 的BPW[16]和来自木榄(Bruguiera gymnorrhiza) 的LUS[17] (表 1),可催化2, 3-氧化鲨烯通过CCC构象生成羽扇醇阳离子,进一步经过重排和去质子化生成羽扇醇,广泛存在于可食用的水果、蔬菜和药用植物中。同样,羽扇醇也具有广泛的药理活性,如抗癌、抗氧化和抗炎等生物活性[47]。因此植物中的OSCs在单个植物物种有可能产生多个三萜骨架。例如,拟南芥(Arabidopsis thaliana)、百脉根(Lotus japonicus)和人参(Panax ginseng) 基因组均包含环阿屯醇合酶基因(cas1[12]、osc5[22]、pnx[18])、羊毛甾醇合酶基因(lss1[24]、las[26]、pnz1[25])、β-香树脂醇合酶基因(lup4[38]、amy1[22]、pny1[18]、pny2[18]) (表 1)。另外,也存在一些能够催化2, 3-氧化鲨烯同时形成β-香树脂醇、羽扇醇等多种三萜产物的多功能OSCs;或者催化2, 3-氧化鲨烯生成其他三萜产物的单功能OSCs (表 1)。
2 环氧角鲨烯环化酶的分子进化关系生物物种多样性的价值越来越被人们所认识与利用。系统发育学是组织和了解生物世界的工具[48]。通过系统发育分析所推断出来的进化关系一般用系统发育树来描述,这个系统发育树描述了同一谱系的进化关系,包括了分子进化(基因树)、物种进化以及分子进化和物种进化的综合。
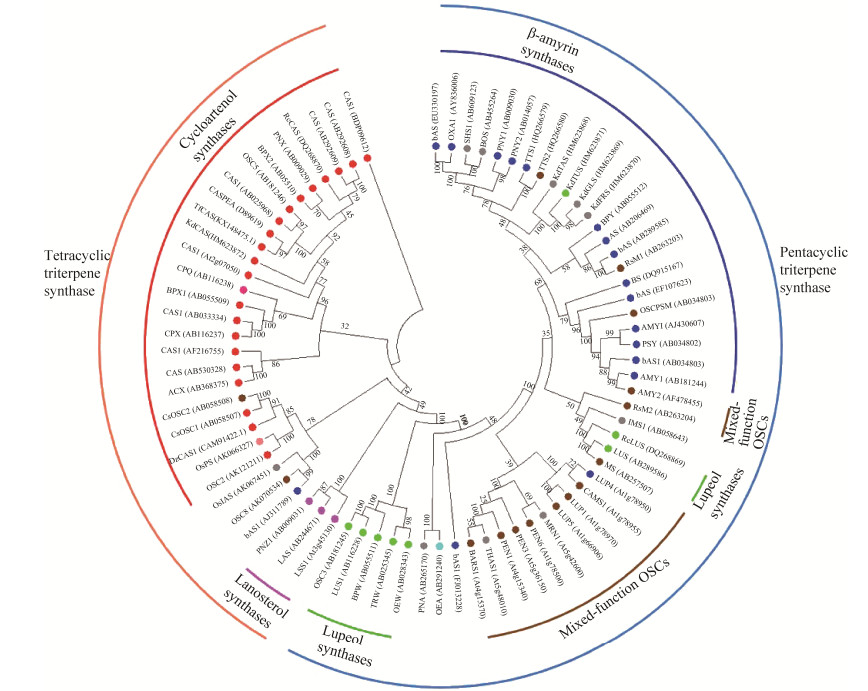
OSCs酶家族成员中不同来源的OSCs核酸序列具有高度保守性。如图 2所示,系统进化树显示了77种植物源OSCs的亲缘及进化关系。生成原甾醇阳离子的四环三萜合酶(tetracyclic triterpene synthase) 与生成达马烯基阳离子的五环三萜合成酶(pentacyclic triterpene synthase) 在分子进化上具有不同的分支途径。植物中的OSCs又是由多基因家族编码的,因此OSCs酶通常可被分为单功能酶和多功能酶[49]。由图 2可知,多功能的三萜合酶位于单功能三萜合酶的相近分支之中,这些酶很有可能通过古老的类羊毛甾醇合酶的复制而产生的,而植物的羊毛甾醇合酶很可能是从祖先的环阿屯醇合酶中分化出来的[4]。因此,三萜合酶很有可能直接或间接地从古老的环阿屯醇合酶复制和分化而来。可以想象,多功能OSCs酶可能代表进化中的中间体,这些中间体可为OSCs酶的进化与分子改造提供结构基础[49]。
|
| 图 2 不同植物的氧化角鲨烯环化酶(OSCs) 的分子进化树 Fig. 2 Neighbor-joining tree of oxidosqualene cyclases (OSCs) from diverse plant species. orange: tetracyclic triterpenoids synthase; light blue: pentacyclic triterpenoids synthase; red: cyloartenol synthase; purple: lanoaterol synthase; rose red: cucurbitadienol synthase; pink: parkeol synthases; blue: β-amyrin synthase; cyan: α-amyrin synthases; green; lupeol synthase; gray: other pure-function OSCs; brown; mixed-function OSCs. |
| |
此外,根据多种具有相同特异性的OSCs酶系统发育树分析,一些作者提出了羽扇醇合酶和β-香树脂醇合酶基因的分子进化机制,即:它们是由一个共同祖先环阿屯醇合酶进化而来[50]。Sawai等[22]对百脉根的OSCs的功能和结构进行了综合分析,并与拟南芥的OSCs基因进行比较,阐明植物三萜的多样化过程。植物OSCs酶的系统发育树表明:双子叶植物OSCs分为4类。Ⅱ、Ⅲ和Ⅳ类分别由环阿屯醇合酶,羽扇醇合酶和β-香树脂醇合酶代表。OSCs基因大多数外显子的长度是保守的,但外显子4、7和9的长度是多样化的,是区分OSCs的良好标准。3个外显子(4、7和9) 的长度对OSCs的分类与推导出的氨基酸序列的系统发育分析一致。外显子长度与OSCs功能之间的明确相关性意味着这些外显子中的3个核苷酸变化,即该区域中1个氨基酸残基的插入或缺失,这应该是OSCs分子进化中控制催化作用的主要因素之一。在百脉根和拟南芥中,OSCs基因形成了基因簇,这表明它们是通过局部基因重复复制,然后累积核苷酸替换、插入和缺失而形成的。Sawai等[22]推测百脉根OSCs基因的进化过程如下:第2染色体OSC3 (第Ⅲ类羽扇醇合酶) 是由OSC5 (第Ⅱ类环阿屯醇合酶) 的祖先进化而来。因此,三萜合酶将2, 3-氧化角鲨烯环化为CCC构象的达玛烯基阳离子的相关能力很可能源自产生CBC构象的原甾醇阳离子,底物构象的变化是酶功能多样化的主要基础。
在植物三萜化合物的细胞工厂的构建方面,通过引入不同植物来源的OSCs酶以及相应的CYP450酶和糖基转移酶(glycosyltransferases, GTs),成功实现了重要三萜化合物的生产(表 2)。在胆固醇的生物合成方面,7-脱氢胆固醇(7-dehydrocholesterol, 7-DHC) 是合成雄甾烯二酮(androstenedione, AD)、雄甾二烯二酮(androstadienedione, ADD) 等甾体药物的中间体,也是维生素D3的重要前体,7-DHC在7-脱氢胆固醇还原酶(7-dehydrocholesterol reductase, DHCR7) 作用下催化生成胆固醇。Guo等[51]通过引入外源24-脱氢胆固醇还原酶(Δ24-d ehydrocholesterol reductase, DHCR24),平衡内源的羊毛甾醇合酶ERG7 (LAS),并过表达了MVA途径的乙酰基辅酶A乙酰基转移酶ERG10 (acetyl- CoA acetyltransferase, AtoAD)、3-羟基-3-甲基戊二酰辅酶A合酶ERG13 (hydroxymethylglutaryl- CoA synthase, HMGCS)、截短的3-羟基-3-甲基戊二酰辅酶A还原酶tHMG1 (truncated 3- hydroxy-3-methylglutaryl coenzyme-A reductase1, tHMG1)、甲羟戊酸激酶ERG12 (mevalonate kinase, MVK)、磷酸甲羟戊酸激酶ERG8 (phosphomevalonate kinase, PMK)、甲羟戊酸焦磷酸脱羧酶ERG19 (mevalonate pyrophosphate decarboxylase, MPD)、异戊烯基二磷酸异构酶IDI1 (isopentenyl diphosphate isomerase1, IDI1)、法尼基焦磷酸合酶ERG20 (farnesyl diphosphate synthetase, FPPase) 等酶,最终7-DHC发酵罐产量达到1.07 g/L。
| Triterpenoids/sterol | OSCs | Products | Product ratio | Production |
| Cholesterol | TfCAS | Cycloartenol | 100% | Not reported |
| CASPEA[14] | Cycloartenol | 100% | ||
| LSS[24] | Lanosterol | 100% | ||
| ERG7[56] | 7-dehydrocholesterol | 100% | 1.07 g/L[51] | |
| PPD | PNA[43] | Dammarenediol Ⅱ | 100% | 8.09 g/L[52] |
| Cucurbitadienol | SgCbQ[27] | Cucurbitadienol | 100% | 63.00 mg/L[53] |
| CPQ[9] | Cucurbitadienol | < 100% | ||
| UA | CrMAS[28] | α-amyrin | ≈80% α-amyrin | 123.27 mg/L[28] |
| MdOSC1[29] | α-amyrin, β-amyrin | ≈80% α-amyrin | ||
| OEA[30] | α-amyrin, β-amyrin, taraxasterol | 50%–80% α-amyrin | ||
| AMY2[37] | α-amyrin, lupeol, β-amyrin | < 30% α-amyrin | ||
| GA | GgbAS1[31] | β-amyrin | 100% | (18.9±2.0) mg/L[54]/ (606.9±9.1) mg/L[55] |
| OA | ||||
| Betulin | BPW[16] | Lupeol | 100% | Not reported |
| KcMS[44] | Lupeol, α-amyrin, β-amyrin | 50% lupeol | ||
| RsM2[17] | Taraxerol, β-amyrin, lupeol | 13% lupeol | ||
| Note: the source of OSCs in table 2 can be found in table 1. | ||||
在原人参二醇(protopanaxadiol, PPD) 合成方面,PPD是人参皂苷的达玛烷型苷元的重要前体。Zhao等[52]组合过表达MVA途径的tHMG1、ERG20、ERG10等所有酶,同时过表达OSC酶达玛烷二醇-Ⅱ合酶(dammarenediol-Ⅱ synthase, DS) 催化2, 3-氧化鲨烯形成达玛烷二醇-Ⅱ (dammarenediol-Ⅱ, DMD) 骨架,利用TetR-TetO (tetracycline repressor-tetracycline operator) 调控系统微调ERG7的表达,优化三萜生物合成模块的代谢平衡;通过异源表达沙门氏菌Salmonella的乙酰辅酶A合成酶ACS突变体L641P,增加了细胞质乙酰辅酶A的供应,最终使PPD的产量达到了8.09 g/L。
在葫芦二烯醇合成方面,葫芦二烯醇是葫芦素和罗汉果甜苷等葫芦烷型植物三萜的骨架。Qiao等[53]开发了酿酒酵母Saccharomyces cerevisiae H. 作为微生物细胞工厂生产葫芦二烯醇,通过引入罗汉果Siraitia grosvenorii的OSC酶SgCbQ,过表达tHMG1、角鲨烯合酶(squalene synthase, SQS) 和角鲨烯环氧酶(squalene epoxidase, SQE) 以改善角鲨烯(squalene) 的供应,并过表达三萜合成酶的全局调控因子UPC2,敲除内源的erg7基因,最终使葫芦二烯醇的产量达63.00 mg/L。
在植物五环三萜化合物合成方面,UA是以α-香树脂醇为分子骨架的典型的乌苏烷型五环三萜化合物。Lu等[28]通过引入了长春花(Catharanthus roseus) 来源的OSC酶CrMAS、蒺藜苜蓿(Medicago truncatula) 来源的CYP450氧化酶CYP716A12和拟南芥CYP450还原酶AtCPR1,并采用葡萄糖和乙醇分批补料发酵策略,最终UA和OA产量分别为123.27和155.58 mg/L,其中UA的产量是已报道的最高产量。
GA和OA均是以β-香树脂醇为分子骨架的齐墩果烷型植物三萜化合物。Zhu等[54]在酿酒酵母中引入光果甘草(Glycyrrhiza glabra) 的OSC酶GgbAS1,以及光果甘草的CYP450基因uni25647和cpy72a63并配对乌拉尔甘草(Glycyrrhiza uralensis) 的CYP450还原酶GuCPR1。通过优化的氧化和还原模块,分批补料发酵,GA产量达到(18.9±2.0) mg/L。Zhao等[55]引入光果甘草的OSC酶GgbAS1和蒺藜苜蓿的CYP450酶MtCYP716A12、MtCPR,并过表达tHMG1、ERG9 (SQS) 和ERG1 (SQE),构建酿酒酵母细胞工厂,最终使OA产量达到(606.9±9.1) mg/L。
3 三萜环化酶的蛋白结构 3.1 三萜环化酶的蛋白结构基础根据三萜类合成过程的环化反应阶段中假定的2个碳正离子,将OSCs酶家族分成原甾醇阳离子和达玛型阳离子2个亚家族。以原甾醇阳离子作为中间体环化形成环阿屯醇、羊毛甾醇等四环三萜类化合物。以达玛型阳离子作为中间体环化合成α-香树脂醇、β-香树脂醇、羽扇豆醇等五环三萜。因此,OSCs酶可主要分为四环三萜合酶和五环三萜合酶两大类。在甾醇的生物合成中,四环三萜合酶催化2, 3-氧化鲨烯通过CBC构象合成羊毛甾醇(真菌、植物)、环阿屯醇(植物) 和葫芦二烯醇(植物) 等四环三萜。在三萜的生物合成中,五环三萜合酶与底物相互作用,底物被折叠成不同的构象-CCC构象,然后环化生成许多不同骨架类型的五环三萜。一般来说,四环三萜合酶主要包括羊毛甾醇合酶、环阿屯醇合酶和葫芦二烯醇合酶;五环三萜合酶主要包括α/β-香树脂醇合酶、羽扇醇合酶。
分别从两种OSCs酶中选择比较典型的代表进行序列比对,发现两种OSCs酶均具有DCTAE和3个QXXXGXW (QW) 等高度保守的特征功能域,其中,X代表任意氨基酸(图 3)。使用OSCs酶不可逆抑制剂和突变分析,保守序列DCTAE可能与底物结合相关[57]。DCTAE序列中的酸性残基Asp释放出的质子攻击环氧环末端,触发多环级联反应。Corey等[58]根据突变体D456N的活力分析结果,推断出酿酒酵母LAS的DCTAE序列的Asp456残基会引发成环反应。Thoma等[59]提出DCTAE序列中Asp455的酸度是通过与Cys456和Cys533形成氢键而增加的。Ito等[32]构建了4个EtAS的突变体(D485N、D485E、C486A和C564A),突变体D485N和D485E没有活性,突变体C486A的活性有所保留,而突变体C564A的活性则完全丧失;突变体D485N的活性完全丧失,这明确证明了Asp的酸性残基充当质子供体以引发成环反应;然而酸性残基Glu变体没有表现出活性,表明附加到Asp上额外的CH2部分干扰了羧基接近环氧化物的通道。因此,必须提供羧基部分和环氧化物之间的适当距离以引发多环化作用。上述实验表明,DCTAE序列参与了多环化反应的起始阶段。同样,两种OSCs酶均具有保守序列QW,该序列是带有负电性的芳香族氨基酸区域,其作用是在2, 3-氧化鲨烯环化反应中稳定碳正阳离子,这些重复序列可能也参与稳定蛋白质结构和功能[60]。
除此之外,两种OSCs酶还具有独特的功能域。在四环三萜OSCs酶中,MWCHCR为环阿屯醇合成酶的特征序列,羊毛甾醇合成酶的特征序列为MWC (N) HC (T) R;而在五环三萜OSCs酶中,β-香树脂醇合酶的特征序列为MWCYCR,α-香树脂醇合酶的特征序列为MXCYCR (图 4),这些保守序列通过阳离子-π相互作用,来稳定中间碳阳离子。Hoshino等[50]通过观察突变体Y259F、Y259A、Y259V和Y259L的活性变化,强烈表明EtAS的MWCYCR序列中Tyr259残基通过阳离子-π相互作用稳定了Baccharenyl仲阳离子,这一作用等同于酿酒酵母Erg7特征序列MWC (N) HC (T) R的His234残基。另外,EtAS的Trp257通过阳离子-π相互作用稳定了Baccharenyl仲阳离子、Lupanyl阳离子和Oleanyl阳离子,这一作用对应于Erg7中的Trp232[50]。EtAS中的Trp257残基在各种β-香树脂醇合酶中高度保守,而羽扇醇合酶中的Leu256残基在相应位置保守。Kushiro等[61]通过将这两个氨基酸定点诱变,成功地将羽扇醇合酶改造为β-香树脂醇合酶,即:将Trp259 (PNY的MWCYCR) 突变为Leu (PNY W259L突变体),该突变体以2︰1的比例将羽扇醇与β-香树脂醇一起作为主要产物。另一方面,Leu256 (OEW的MLCYCR) 被突变为Trp (OEW L256W突变体),该突变体产生大量β-香树脂醇并伴随少量羽扇醇的生成,这表明该独特功能域中单个氨基酸的突变就可以改变酶的催化特异性。Kushiro等[61]根据PNY单点定向变体:突变体W259L和Y261H的产物分布比,提出Trp259和Tyr261通过阳离子-π相互作用分别稳定Oleanyl和Lupanyl阳离子的功能。PNY中的Trp259和Tyr261分别对应EtAS中的Trp257和Tyr259[50]。另外,该独特功能域周围的脂肪族和芳香族残基也有着不可忽视的作用。如在EtAS的催化过程中,残基Val483、Phe474、Phe 413和Met729,通过疏水相互作用参与底物的紧密结合[50];残基Trp612、Tyr259、Trp257和Phe728的π电子密度对可通过阳离子-π相互作用稳定瞬态碳正离子[62]。可见,四环三萜合酶和五环三萜合酶独特的功能域对于稳定碳阳离子中间体以及产物特异性至关重要。
|
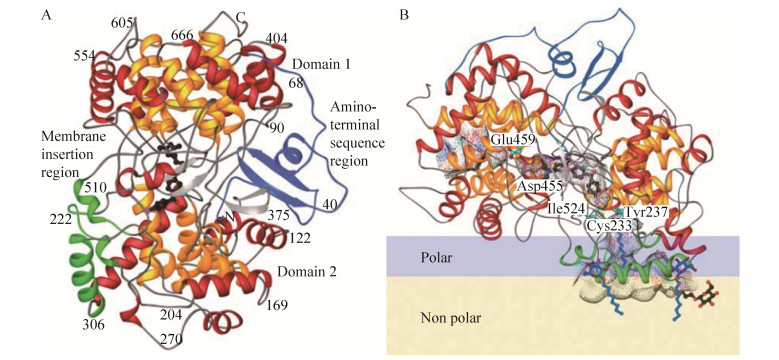
| 图 4 人源OSC酶带状图 Fig. 4 Human OSC enzyme strip chart. (A) Ro 48-8071 (黑色) 与人源OSC酶的晶体结构。(B) 羊毛甾醇与人源OSC酶的晶体结构。OSC酶相对于膜的方向,极性部分和非极性部分分别用浅蓝和浅黄表示。OSC酶内部表面和通道用以下颜色显示:蓝色,阳性;红色,阴性;青色,形成氢键供体;洋红色,其他极性部位。在晶体结构中观察到有序的洗涤剂分子或脂质片段(蓝色和黑色的线条图) 聚集在膜插入区域周围[59] (A) Crystal structure of Ro 48-8071 (black) and human OSC enzyme. (B) Crystal structure of lanosterol and human OSC enzyme. The direction of OSC enzyme relative to the membrane, the polar part and the non-polar part are represented by light blue and light yellow, respectively. The internal surface and channels of the OSC enzyme are displayed in the following colors: blue, positive; red, negative; cyan, forming hydrogen bond donors; magenta, other polar parts. Ordered detergent molecules or lipid fragments (blue and black line drawings) are observed in the crystal structure to gather around the membrane insertion area[59]. |
| |
OSCs酶的多样性与其内在的蛋白结构密切相关,从结构生物学和生物化学角度研究OSCs酶,对解析OSCs酶的催化机制、功能特点和进化关系具有重要意义。OSCs酶分子结构较大且属于膜蛋白,晶体结构解析具有一定的困难,只有人源OSC酶的晶体结构得到解析。目前,已报道两种人源OSC酶的晶体结构:(1) 人源OSC与抑制剂Ro48-8071的晶体结构(PDB登录号:1W6J);(2) 人源OSC与羊毛甾醇的晶体结构(PDB登录号:1W6K)[59]。
人源OSC酶包括两个α-螺旋结构域:Domain1和Domain2,这两个结构域被Loop环和β结构连接。活性位点位于Domain1和Domain2之间的分子中心,氨基酸末端充满了两个结构域之间的空间,并尽可能稳定其相对方向(图 4A)。人源OSC酶的晶体结构为单体,这与超离心分析数据一致[59]。OSC酶是一种单分子膜蛋白,即:它插入细胞膜但不跨越磷脂双分子层。通过结合的洗涤剂分子和其疏水性表面(1.700 Å2),检测到Domain2中的OSC酶膜结合区占酶总表面的6% (图 4B)[59]。膜插入表面由一个直径为25Å的酶二级结构平台和一个通向活性区域的通道组成。这个通道被认为是允许2, 3-氧化鲨烯进入的疏水活性部位,但是一个收缩部位将其与活性部位空腔分开(图 4B)。通过改变残基Tyr237、Cys233和Ile524的侧链构象,可以改变人源OSC酶的底物通道。人源OSC酶中的活性位点通道通向活性位点腔的“顶部”,即Asp455区域(图 4B)。在空腔的“顶部”,Asp455侧链与羊毛甾醇3-羟基形成氢键。这证实了以下假设:环化反应的启动是通过Asp455质子化预折叠的2, 3-氧化鲨烯的环氧基来实现的,这与Asp455作为一般酸的作用一致,它向底物的环氧原子提供一个质子以启动环化级联反应。在一个催化循环完成后,Asp455可以通过水分子链和Glu459的羧酸基团从本体溶剂中实现重羰化,也可以通过将质子从最终的去质子化步骤转移回Asp455。
在含有抑制剂Ro 48-8071的人源羊毛甾醇合酶的晶体结构中,Ro 48-8071的氮原子与Asp455羧酸盐在2.9 Å处形成带电氢键,该氢键将两个氧原子均引向活性位点腔,带电荷的氨基还通过与几个芳香族残基的阳离子-Π相互作用而稳定。这一结构证实了Asp455作为一般酸,而两个半胱氨酸Cys456和Cys533进一步提高了酸度。另外,该抑制剂的溴原子与通道收缩部位附近的Ile524、Tyr237和Cys233残基相互作用,而His232作为终止环化反应的残基。这与羊毛甾醇-人源OSC酶晶体结构中的催化机制一致。
由于OSCs酶的晶体结构不易获得,近年来,越来越多的研究者以已解析的晶体结构为模板,结合生物化学、有机化学和计算机辅助设计等开展OSCs催化活性的研究,以深入研究OSCs的催化反应机理、产物多样性以及功能残基。Wu等[63]以人源OSC的X射线晶体结构作为模板对酿酒酵母ERG7进行同源建模,模拟了ERG7 His234与其他氨基酸残基的取代,结果表明该位置影响了阳离子中间体与活性位点残基侧链之间空间或静电的相互作用。
4 环氧角鲨烯环化酶的催化分子机制OSCs酶的催化反应涉及环氧原子的质子化和碳正离子中间体的形成,涉及氢化物、甲基和烷基的转移以及环的膨胀。OSCs酶环化级联反应的启动依赖位于功能序列DCTAE中的Asp质子化类异戊二烯底物的末端来产生叔碳正离子[64]。随着底物的折叠、环氧原子的质子化,引发一系列亲电攻击附近双键的C-C成环反应。
目前,以人源OSC酶为基础,对OSCs酶的环化机理的研究比较普遍(图 5)。羊毛甾醇是一种具有“A-B-C-D”四个环的四环三萜化合物,是由CBC构象的原甾醇阳离子发展而来。目前,普遍认为整个A-B-C-D环的逐步形成产生原甾醇阳离子[65],环氧环的开环和A环的形成是协调一致的过程[66]。然而,在环化反应生成原甾醇阳离子的问题上仍存在许多争议[67]。目前的研究主要有3种假设,这些假设在环化的初始步骤是相同的,但是对于C和D环的形成有着截然不同的看法(图 5)。假设Ⅰ认为,C环可以通过反马氏加成直接形成[59];假设Ⅱ提出一个分步进行的马氏加成反应,首先形成五元C环的C'态,然后由环膨胀为稳定的六元C环(C状态)[68];由于自然界中普遍存在6-6-5三萜结构,而天然的6-6-6三萜结构很少存在,所以假设Ⅱ被广泛接受[69]。Corey等[46]基于从头计算提出假设Ⅲ,即:C' (6-6-5) 状态可以直接转化为D (6-6-6-6-5) 状态,而不存在C中间状态。此外,Tian等[70]的研究也指出了这种协同反应的可能性。Chen等[67]通过量子力学/分子力学-分子动力学(QM/MM-MD) 模拟表明,先前提到假设Ⅱ中的稳定中间产物C态(6-6-6) 与他们模拟中的过渡态非常相似,而C'态(6-6-5) 和D态(6-6-6-6-5) 都是催化反应中稳定的中间体。在此基础上,Chen等[71]通过二维(2D) QM/MM-MD模拟并计算整体环化自由能剖面证实了稳定的C'态(6-6-5) 可能存在,并随后转换为低能垒的D态(6-6-6-6-5),这也与Hess在气相中的计算结果一致。
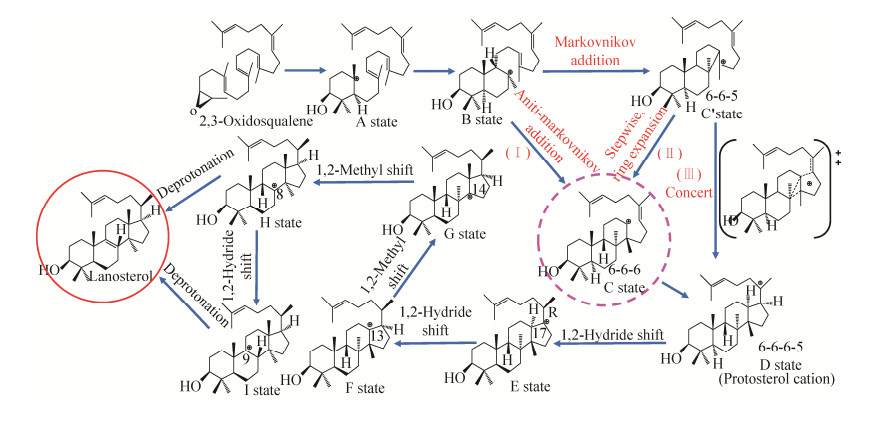
|
| 图 5 人源OSC酶催化分子机制 Fig. 5 Catalytic molecular mechanisms of human OSC enzyme. |
| |
在OSCs酶催化的整个过程中,酶和底物的构象一直处于不断变化的状态。因此,残基与碳正离子之间的相互作用也处于不断变化中。Diao等[72]利用QM/MM-MD相结合的方式研究羊毛甾醇合酶的作用机制,在氢化物从D状态转变为F状态时,为了更好地考虑富电子芳香族残基与碳正离子之间的相互作用,将残基Phe696和His232以及碳正离子中间体视为QM区域,其他原子被视为MM区域。在从F态到产物的后续步骤中,由于碳正离子化相对靠近Tyr503残基,而远离Phe696,因此吸收Tyr503残基进入QM区,并从QM区域中去除Phe696。因此,碳正离子环化和重排的变化对支架多样性有很大的影响。
除A/B/C/D环形成之外,OSCs酶催化2, 3-氧化鲨烯生成五环三萜的过程,还包括E环的形成和膨胀。Wu等[73]使用QM/MM-MD研究来自鸢尾(Iris tectorum) 的ItOSC2的作用机制,将残基Leu256、Leu258和Tyr531以及碳正离子中间体视为QM区域,其他原子被视为MM区域。Lupanyl阳离子的E环膨胀产生具有6-6-6-6-6环系统的Germanicyl阳离子,接着1, 2氢化物转移(H13到C18) 启动β-香树脂醇途径,而甲基转移(Me29到C19) 启动α-香树脂醇途径。本质上,这种环化反应是碳正离子的迁移和放热的过程。
经过初始环化后,碳正离子重排和去质子化决定了最终产物及其相关功能结构域的区域和立体化学性质。在前面工作的基础上,Diao等[72]通过QM/MM-MD,进一步研究了羊毛甾醇生物合成的机理细节,包括3个氢化物转移、2个甲基转移和1个去质子化(图 5)。通过获取羊毛甾醇生物合成的完整自由能剖面,有效证明了该假设途径是合理的。在该假设途径中,静电相互作用和CH…π相互作用等是促进碳正离子迁移的关键因素;His232是最终的质子受体,Tyr503介导去质子化。通过能量分析表明,该酶通过活性口袋的电场介导了从初始环化到后续重排的能量转移,这可能是三萜环化酶调节反应中间体相对能量并避免副产物形成的一般策略。对羊毛甾醇合酶环化机理的研究可为酶工程中生产多种融合多环三萜产物的新OSCs酶设计提供指导。
5 总结与展望萜类化合物尤其是三萜类物质的药理活性及其带来巨大经济效益以及天然资源的限制,是促使研究者不断探究如何利用代谢工程和合成生物学的方法来生产植物三萜的动力。OSCs酶的环化反应催化2, 3-氧化鲨烯进入不同的代谢分支,是甾醇和植物三萜类物质生物合成的重要调控位点。迄今为止,已经在三萜和植物甾醇的生物合成途径及相关酶的研究取得了一定的进展,并且对它们的生理功能和分子机理的研究逐渐深入。虽然目前OSCs酶的结构解析仍存在一定困难,但通过序列比对、分子进化分析和蛋白质同源建模的发展,为OSCs基因家族的系统进化研究提供了坚实基础。随着越来越多的具有新型催化特征的OSCs的出现,也为我们了解OSCs酶的蛋白结构和功能的对应关系提供了新的思路和研究方向。
| [1] |
Ohyama K, Suzuki M, Kikuchi J, et al. Dual biosynthetic pathways to phytosterol via cycloartenol and lanosterol in Arabidopsis. PNAS, 2009, 106(3): 725-730. DOI:10.1073/pnas.0807675106
|
| [2] |
常鹏程, 于洋, 王颖, 等. 酿酒酵母高效合成萜类化合物的组合调控策略. 化工进展, 2019, 38(1): 598-605. Chang PC, Yu Y, Wang Y, et al. Combinatorial regulation strategies for efficient synthesis of terpenoids in Saccharomyces cerevisiae. Chem Ind Eng Prog, 2019, 38(1): 598-605 (in Chinese). |
| [3] |
Song YD, Chen S, Wang XJ, et al. A novel strategy to enhance terpenoids production using cambial meristematic cells of Tripterygium wilfordii Hook. F. Plant Methods, 2019, 15: 129. DOI:10.1186/s13007-019-0513-x
|
| [4] |
Xue ZY, Duan LX, Liu D, et al. Divergent evolution of oxidosqualene cyclases in plants. New Phytol, 2012, 193(4): 1022-1038. DOI:10.1111/j.1469-8137.2011.03997.x
|
| [5] |
Hua WP, Kong WW, Cao XY, et al. Transcriptome analysis of Dioscorea zingiberensis identifies genes involved in diosgenin biosynthesis. Genes Genom, 2017, 39(5): 509-520. DOI:10.1007/s13258-017-0516-9
|
| [6] |
Guan HY, Su P, Zhao YJ, et al. Cloning and functional analysis of two sterol-C24-methyltransferase 1 (SMT1) genes from Paris Polyphylla. J Asian Nat Prod Res, 2018, 20(7): 595-604. DOI:10.1080/10286020.2016.1271791
|
| [7] |
Lu YD, Zhou WX, Wei L, et al. Regulation of the cholesterol biosynthetic pathway and its integration with fatty acid biosynthesis in the oleaginous microalga Nannochloropsis oceanica. Biotechnol Biofuels, 2014, 7: 81. DOI:10.1186/1754-6834-7-81
|
| [8] |
Christ B, Xu CC, Xu ML, et al. Repeated evolution of cytochrome P450-mediated spiroketal steroid biosynthesis in plants. Nat Commun, 2019, 10(1): 3206. DOI:10.1038/s41467-019-11286-7
|
| [9] |
Shibuya M, Adachi S, Ebizuka Y. Cucurbitadienol synthase, the first committed enzyme for cucurbitacin biosynthesis, is a distinct enzyme from cycloartenol synthase for phytosterol biosynthesis. Tetrahedron, 2004, 60(33): 6995-7003. DOI:10.1016/j.tet.2004.04.088
|
| [10] |
Ito R, Mori K, Hashimoto I, et al. Triterpene cyclases from Oryza sativa L.: cycloartenol, parkeol and achilleol B synthases. Org Lett, 2011, 13(10): 2678-2681. DOI:10.1021/ol200777d
|
| [11] |
Thimmappa R, Geisler K, Louveau T, et al. Triterpene biosynthesis in plants. Annu Rev Plant Biol, 2014, 65: 225-257. DOI:10.1146/annurev-arplant-050312-120229
|
| [12] |
Corey EJ, Matsuda SP, Bartel B. Isolation of an Arabidopsis thaliana gene encoding cycloartenol synthase by functional expression in a yeast mutant lacking lanosterol synthase by the use of a chromatographic screen. PNAS, 1993, 90(24): 11628-11632. DOI:10.1073/pnas.90.24.11628
|
| [13] |
Shinozaki J, Shibuya M, Masuda K, et al. Squalene cyclase and oxidosqualene cyclase from a fern. FEBS Lett, 2008, 582(2): 310-318. DOI:10.1016/j.febslet.2007.12.023
|
| [14] |
Morita M, Shibuya M, Lee MS, et al. Molecular cloning of pea cDNA encoding cycloartenol synthase and its functional expression in yeast. Biol Pharm Bull, 1997, 20(7): 770-775. DOI:10.1248/bpb.20.770
|
| [15] |
Haralampidis K, Bryan G, Qi X, et al. A new class of oxidosqualene cyclases directs synthesis of antimicrobial phytoprotectants in monocots. PNAS, 2001, 98(23): 13431-13436. DOI:10.1073/pnas.231324698
|
| [16] |
Zhang H, Shibuya M, Yokota S, et al. Oxidosqualene cyclases from cell suspension cultures of Betula platyphylla var. japonica: molecular evolution of oxidosqualene cyclases in higher plants. Biol Pharm Bull, 2003, 26(5): 642-650. DOI:10.1248/bpb.26.642
|
| [17] |
Basyuni M, Oku H, Tsujimoto E, et al. Triterpene synthases from the Okinawan mangrove tribe, Rhizophoraceae. FEBS J, 2007, 274(19): 5028-5042. DOI:10.1111/j.1742-4658.2007.06025.x
|
| [18] |
Kushiro T, Shibuya M, Ebizuka Y. Beta-amyrin synthase--cloning of oxidosqualene cyclase that catalyzes the formation of the most popular triterpene among higher plants. Eur J Biochem, 1998, 256(1): 238-244. DOI:10.1046/j.1432-1327.1998.2560238.x
|
| [19] |
Hayashi H, Huang PY, Takada S, et al. Differential expression of three oxidosqualene cyclase mRNAs in Glycyrrhiza glabra. Biol Pharm Bull, 2004, 27(7): 1086-1092. DOI:10.1248/bpb.27.1086
|
| [20] |
Kawano N, Ichinose K, Ebizuka Y. Molecular cloning and functional expression of cDNAs encoding oxidosqualene cyclases from Costus speciosus. Biol Pharm Bull, 2002, 25(4): 477-482. DOI:10.1248/bpb.25.477
|
| [21] |
Hayashi HN, Ikeshiro Y, Yazaki K, et al. Molecular cloning of a cDNA encoding cycloartenol synthase from Luffa cylindrica. Plant Physiol, 1999, 121(4): 1384-1390.
|
| [22] |
Sawai S, Shindo T, Sato S, et al. Functional and structural analysis of genes encoding oxidosqualene cyclases of Lotus japonicus. Plant Sci, 2006, 170(2): 247-257. DOI:10.1016/j.plantsci.2005.08.027
|
| [23] |
Guhling O, Hobl B, Yeats T, et al. Cloning and characterization of a lupeol synthase involved in the synthesis of epicuticular wax crystals on stem and hypocotyl surfaces of Ricinus communis. Arch Biochem Biophys, 2006, 448(1-2): 60-72. DOI:10.1016/j.abb.2005.12.013
|
| [24] |
Kolesnikova MD, Xiong QB, Lodeiro S, et al. Lanosterol biosynthesis in plants. Arch Biochem Biophys, 2006, 447(1): 87-95. DOI:10.1016/j.abb.2005.12.010
|
| [25] |
Suzuki M, Xiang T, Ohyama K, et al. Lanosterol synthase in dicotyledonous plants. Plant Cell Physiol, 2006, 47(5): 565-571. DOI:10.1093/pcp/pcj031
|
| [26] |
Sawai S, Akashi T, Sakurai N, et al. Plant lanosterol synthase: divergence of the sterol and triterpene biosynthetic pathways in eukaryotes. Plant Cell Physiol, 2006, 47(5): 673-677. DOI:10.1093/pcp/pcj032
|
| [27] |
Dai LH, Liu C, Zhu YM, et al. Functional characterization of cucurbitadienol synthase and triterpene glycosyltransferase involved in biosynthesis of mogrosides from Siraitia grosvenorii. Plant Cell Physiol, 2015, 56(6): 1172-1182. DOI:10.1093/pcp/pcv043
|
| [28] |
Lu CZ, Zhang CB, Zhao FL, et al. Biosynthesis of ursolic acid and oleanolic acid in Saccharomyces cerevisiae. AIChE J, 2018, 64(11): 3794-3802. DOI:10.1002/aic.16370
|
| [29] |
Yu Y, Chang PC, Yu H, et al. Productive amyrin synthases for efficient α-amyrin synthesis in engineered Saccharomyces cerevisiae. ACS Synth Biol, 2018, 7(10): 2391-2402. DOI:10.1021/acssynbio.8b00176
|
| [30] |
Saimaru H, Orihara Y, Tansakul P, et al. Production of triterpene acids by cell suspension cultures of Olea europaea. Chem Pharm Bull (Tokyo), 2007, 55(5): 784-788. DOI:10.1248/cpb.55.784
|
| [31] |
Hayashi H, Huang PY, Kirakosyan A, et al. Cloning and characterization of a cDNA encoding beta-amyrin synthase involved in glycyrrhizin and soyasaponin biosyntheses in licorice. Biol Pharm Bull, 2001, 24(8): 912-916. DOI:10.1248/bpb.24.912
|
| [32] |
Ito R, Masukawa Y, Hoshino T. Purification, kinetics, inhibitors and CD for recombinant β-amyrin synthase from Euphorbia tirucalli L. and functional analysis of the DCTA motif, which is highly conserved among oxidosqualene cyclases. FEBS J, 2013, 280(5): 1267-1280. DOI:10.1111/febs.12119
|
| [33] |
Kirby J, Romanini DW, Paradise EM, et al. Engineering triterpene production in Saccharomyces cerevisiae-beta-amyrin synthase from Artemisia annua. FEBS J, 2008, 275(8): 1852-1859. DOI:10.1111/j.1742-4658.2008.06343.x
|
| [34] |
Scholz M, Lipinski M, Leupold M, et al. Methyl jasmonate induced accumulation of kalopanaxsaponin I in Nigella sativa. Phytochemistry, 2009, 70(4): 517-522. DOI:10.1016/j.phytochem.2009.01.018
|
| [35] |
Cammareri M, Consiglio MF, Pecchia P, et al. Molecular characterization of β-amyrin synthase from Aster sedifolius L. and triterpenoid saponin analysis. Plant Sci, 2008, 175(3): 255-261. DOI:10.1016/j.plantsci.2008.04.004
|
| [36] |
Kajikawa M, Yamato KT, Fukuzawa H, et al. Cloning and characterization of a cDNA encoding beta-amyrin synthase from petroleum plant Euphorbia tirucalli L.. Phytochemistry, 2005, 66(15): 1759-1766. DOI:10.1016/j.phytochem.2005.05.021
|
| [37] |
Iturbe-Ormaetxe I, Haralampidis K, Papadopoulou K, et al. Molecular cloning and characterization of triterpene synthases from Medicago truncatula and Lotus japonicus. Plant Mol Biol, 2003, 51(5): 731-743. DOI:10.1023/A:1022519709298
|
| [38] |
Shibuya M, Katsube Y, Otsuka M, et al. Identification of a product specific beta-amyrin synthase from Arabidopsis thaliana. Plant Physiol Biochem, 2009, 47(1): 26-30. DOI:10.1016/j.plaphy.2008.09.007
|
| [39] |
Wang ZH, Guhling O, Yao RN, et al. Two oxidosqualene cyclases responsible for biosynthesis of tomato fruit cuticular triterpenoids. Plant Physiol, 2011, 155(1): 540-552. DOI:10.1104/pp.110.162883
|
| [40] |
Wang ZH, Yeats T, Han H, et al. Cloning and characterization of oxidosqualene cyclases from Kalanchoe daigremontiana: enzymes catalyzing up to 10 rearrangement steps yielding friedelin and other triterpenoids. J Biol Chem, 2010, 285(39): 29703-29712. DOI:10.1074/jbc.M109.098871
|
| [41] |
Shibuya M, Zhang H, Endo A, et al. Two branches of the lupeol synthase gene in the molecular evolution of plant oxidosqualene cyclases. Eur J Biochem, 1999, 266(1): 302-307. DOI:10.1046/j.1432-1327.1999.00875.x
|
| [42] |
Fazio GC, Xu R, Matsuda SPT. Genome mining to identify new plant triterpenoids. J Am Chem Soc, 2004, 126(18): 5678-5679. DOI:10.1021/ja0318784
|
| [43] |
Tansakul P, Shibuya M, Kushiro T, et al. Dammarenediol-Ⅱ synthase, the first dedicated enzyme for ginsenoside biosynthesis, in Panax ginseng. FEBS Lett, 2006, 580(22): 5143-5149. DOI:10.1016/j.febslet.2006.08.044
|
| [44] |
Basyuni M, Oku H, Inafuku M, et al. Molecular cloning and functional expression of a multifunctional triterpene synthase cDNA from a mangrove species Kandelia candel (L.) Druce. Phytochemistry, 2006, 67(23): 2517-2524. DOI:10.1016/j.phytochem.2006.09.016
|
| [45] |
Liu H, Fan JJ, Wang C, et al. Enhanced β-amyrin synthesis in Saccharomyces cerevisiae by coupling an optimal acetyl-CoA supply pathway. J Agric Food Chem, 2019, 67(13): 3723-3732. DOI:10.1021/acs.jafc.9b00653
|
| [46] |
Corey EJ, Cheng HM, Baker CH, et al. Methodology for the preparation of pure recombinant S. cerevisiae lanosterol synthase using a baculovirus expression system. Evidence that oxirane cleavage and a-ring formation are concerted in the biosynthesis of lanosterol from 2, 3-oxidosqualene. J Am Chem Soc, 1997, 119(6): 1277-1288. DOI:10.1021/ja963227w
|
| [47] |
Liu K, Zhang XM, Xie L, et al. Lupeol and its derivatives as anticancer and anti-inflammatory agents: molecular mechanisms and therapeutic efficacy. Pharmacol Res, 2021, 164: 105373. DOI:10.1016/j.phrs.2020.105373
|
| [48] |
Hug LA, Baker BJ, Anantharaman K, et al. A new view of the tree of life. Nat Microbiol, 2016, 1(5): 16048. DOI:10.1038/nmicrobiol.2016.48
|
| [49] |
Phillips DR, Rasbery JM, Bartel B, et al. Biosynthetic diversity in plant triterpene cyclization. Curr Opin Plant Biol, 2006, 9(3): 305-314. DOI:10.1016/j.pbi.2006.03.004
|
| [50] |
Hoshino T. β-Amyrin biosynthesis: catalytic mechanism and substrate recognition. Org Biomol Chem, 2017, 15(14): 2869-2891. DOI:10.1039/C7OB00238F
|
| [51] |
Guo XJ, Xiao WH, Wang Y, et al. Metabolic engineering of Saccharomyces cerevisiae for 7-dehydrocholesterol overproduction. Biotechnol Biofuels, 2018, 11(1): 192. DOI:10.1186/s13068-018-1194-9
|
| [52] |
Zhao FL, Bai P, Nan WH, et al. A modular engineering strategy for high-level production of protopanaxadiol from ethanol by Saccharomyces cerevisiae. AIChE J, 2019, 65(3): 866-874. DOI:10.1002/aic.16502
|
| [53] |
Qiao J, Luo ZL, Cui SR, et al. Modification of isoprene synthesis to enable production of curcurbitadienol synthesis in Saccharomyces cerevisiae. J Ind Microbiol Biotechnol, 2019, 46(2): 147-157. DOI:10.1007/s10295-018-2116-3
|
| [54] |
Zhu M, Wang CX, Sun WT, et al. Boosting 11-oxo-β-amyrin and glycyrrhetinic acid synthesis in Saccharomyces cerevisiae via pairing novel oxidation and reduction system from legume plants. Metab Eng, 2018, 45: 43-50. DOI:10.1016/j.ymben.2017.11.009
|
| [55] |
Zhao YJ, Fan JJ, Wang C, et al. Enhancing oleanolic acid production in engineered Saccharomyces cerevisiae. Bioresour Technol, 2018, 257: 339-343. DOI:10.1016/j.biortech.2018.02.096
|
| [56] |
Corey EJ, Matsuda SP, Bartel B. Molecular cloning, characterization, and overexpression of ERG7, the Saccharomyces cerevisiae gene encoding lanosterol synthase. PNAS, 1994, 91(6): 2211-2215. DOI:10.1073/pnas.91.6.2211
|
| [57] |
Abe I, Prestwich GD. Molecular cloning, characterization, and functional expression of rat oxidosqualene cyclase cDNA. PNAS, 1995, 92(20): 9274-9278. DOI:10.1073/pnas.92.20.9274
|
| [58] |
Corey EJ, Cheng HM, Baker CH, et al. Studies on the substrate binding segments and catalytic action of lanosterol synthase. Affinity labeling with carbocations derived from mechanism-based analogs of 2, 3-oxidosqualene and site-directed mutagenesis probes. J Am Chem Soc, 1997, 119(6): 1289-1296. DOI:10.1021/ja963228o
|
| [59] |
Thoma R, Schulz-Gasch T, D'Arcy B, et al. Insight into steroid scaffold formation from the structure of human oxidosqualene cyclase. Nature, 2004, 432(7013): 118-122. DOI:10.1038/nature02993
|
| [60] |
Sikkema J, de Bont JA, Poolman B. Interactions of cyclic hydrocarbons with biological membranes. J Biol Chem, 1994, 269(11): 8022-8028. DOI:10.1016/S0021-9258(17)37154-5
|
| [61] |
Kushiro T, Shibuya M, Masuda K, et al. Mutational studies on triterpene synthases: engineering lupeol synthase into β-amyrin synthase. J Am Chem Soc, 2000, 122(29): 6816-6824. DOI:10.1021/ja0010709
|
| [62] |
Hess BA. Concomitant C-ring expansion and D-ring formation in lanosterol biosynthesis from squalene without violation of Markovnikov's rule. J Am Chem Soc, 2002, 124(35): 10286-10287. DOI:10.1021/ja026850r
|
| [63] |
Wu TK, Liu YT, Chang CH, et al. Site-saturated mutagenesis of histidine 234 of Saccharomyces cerevisiae oxidosqualene-lanosterol cyclase demonstrates dual functions in cyclization and rearrangement reactions. J Am Chem Soc, 2006, 128(19): 6414-6419. DOI:10.1021/ja058782p
|
| [64] |
Segura MJ, Lodeiro S, Meyer MM, et al. Directed evolution experiments reveal mutations at cycloartenol synthase residue His477 that dramatically alter catalysis. Org Lett, 2002, 4(25): 4459-4462. DOI:10.1021/ol0269897
|
| [65] |
Tantillo DJ. Biosynthesis via carbocations: theoretical studies on terpene formation. Nat Prod Rep, 2011, 28(6): 1035-1053. DOI:10.1039/c1np00006c
|
| [66] |
Tantillo DJ. Recent excursions to the borderlands between the realms of concerted and stepwise: carbocation cascades in natural products biosynthesis. J Phys Org Chem, 2008, 21(7-8): 561-570. DOI:10.1002/poc.1320
|
| [67] |
Chen NH, Zhou JW, Li JB, et al. Concerted cyclization of lanosterol C-ring and D-ring under human oxidosqualene cyclase catalysis: an ab initio QM/MM MD study. J Chem Theory Comput, 2014, 10(3): 1109-1120. DOI:10.1021/ct400949b
|
| [68] |
Wu TK, Chang CH, Liu YT, et al. Saccharomyces cerevisiae oxidosqualene-lanosterol cyclase: a chemistry-biology interdisciplinary study of the protein's structure-function-reaction mechanism relationships. Chem Rec, 2008, 8(5): 302-325. DOI:10.1002/tcr.20157
|
| [69] |
Matsuda SP, Wilson WK, Xiong QB. Mechanistic insights into triterpene synthesis from quantum mechanical calculations. Detection of systematic errors in B3LYP cyclization energies. Org Biomol Chem, 2006, 4(3): 530-543. DOI:10.1039/b513599k
|
| [70] |
Tian BX, Eriksson LA. Catalytic mechanism and product specificity of oxidosqualene-lanosterol cyclase: a QM/MM study. J Phys Chem B, 2012, 116(47): 13857-13862. DOI:10.1021/jp3091396
|
| [71] |
Chen NH, Wang SL, Smentek L, et al. Biosynthetic mechanism of lanosterol: cyclization. Angew Chem Int Ed, 2015, 54(30): 8693-8696. DOI:10.1002/anie.201501986
|
| [72] |
Diao HJ, Chen NH, Wang K, et al. Biosynthetic mechanism of lanosterol: a completed story. ACS Catal, 2020, 10(3): 2157-2168. DOI:10.1021/acscatal.9b05221
|
| [73] |
Wu SD, Zhang F, Xiong WB, et al. An unexpected oxidosqualene cyclase active site architecture in the Iris tectorum multifunctional α-amyrin synthase. ACS Catal, 2020, 10(16): 9515-9520. DOI:10.1021/acscatal.0c03231
|
 2022, Vol. 38
2022, Vol. 38