中国科学院微生物研究所、中国微生物学会主办
文章信息
- 张碧飞, 吕成, 张萌, 许菲
- ZHANG Bifei, LÜ Cheng, ZHANG Meng, XU Fei
- 基于多重计算设计策略提高奇异变形杆菌脂肪酶的热稳定性
- Improving the thermal stability of Proteus mirabilis lipase based on multiple computational design strategies
- 生物工程学报, 2022, 38(4): 1537-1553
- Chinese Journal of Biotechnology, 2022, 38(4): 1537-1553
- 10.13345/j.cjb.210881
-
文章历史
- Received: September 7, 2021
- Accepted: February 22, 2022
- Published: February 24, 2022
脂肪酶(三酰基甘油酯水解酶,EC 3.1.1.3)可以介导甘油三酯在油水界面上水解成单甘油酯、脂肪酸、双甘油酯和甘油[1],也能催化非水环境中的酯交换、酯化、醇解、酸解和氨解[2]。由于其独特的催化性能,脂肪酶被广泛应用于食品工业、烘焙、洗涤剂和生物燃料等多种行业[3-4]。2020年,微生物脂肪酶市场规模已达到4.4亿美元,预计到2027年将达到6.5亿美元[5]。近年,来自多种变形杆菌的脂肪酶因具有较高的酶活、耐受有机溶剂以及可在大肠杆菌中可溶性表达的特点引起了人们的关注[6-11]。其中,来自奇异变形杆菌的脂肪酶(Proteus mirabilis lipase, PML)[12]属于Ⅰ.1家族脂肪酶,由287个氨基酸组成,其晶体结构显示:PML是带有盖子结构域的α/β水解酶,S79、H254和D232组成其催化三联体,E80的主链酰胺以及两个水分子组成其氧阴离子洞,此外,PML的α8末端附近存在Ca2+结合位点,该Ca2+的结合很可能是正确定位H254以激活亲核残基S79所必需的[12],对脂肪酶的稳定性和活性至关重要[13-14]。近年,PML在生物柴油的生产等方面展示出了极大的潜力[15-17],然而,其热稳定性并不突出,这限制了其在工业上的进一步发展。
提高酶的热稳定性一直是蛋白质工程中研究的热点之一。传统的蛋白质工程如定向进化往往以低筛选效率为代价得到符合工业需求的突变体。近年,得益于结构生物学及生物信息学的发展,基于蛋白质结构和序列信息来提高蛋白质热稳定性的计算设计方法得到了广泛应用。在蛋白质结构信息的指导下,基于不同打分策略的能量函数对蛋白能量进行计算评分可以得到自由能更小、更有利于蛋白质稳定的突变设计,如利用经验力场建立打分函数的FoldX[18-19],应用统计能量函数进行计算设计的ABACUS[20-22],基于物理能量函数的Rosetta ddg[23-24]等;以及在此基础上开发的对蛋白质空腔进行填充设计的Rosetta-VIP[25],它通过将蛋白空腔周围的氨基酸突变成侧链基团更大或非极性的氨基酸来提高酶包装的紧密性,进而提高酶的稳定性。此外,基于多序列比对(multiple sequence alignment, MSA) 的序列分析可以为蛋白质的计算设计提供有益信息,如位置特异性评分矩阵(position specific scoring matrix, PSSM)[26-27]可以筛选出同一位点上自然界中出现概率更大、更可能稳定的氨基酸种类;共进化分析(Generative REgularized ModeLs of proteINs, GREMLIN)[28-29]可以提供蛋白质序列中与功能相关的保守氨基酸对;通过祖先序列重建(ancestral sequence reconstruction, ASR)[30]的策略也有望得到热稳定性较高的突变体设计。
然而,单独的某一种计算策略可能有一定的局限性。结合多种计算方法,可以减少单一算法中的采样偏差,从而降低假阳性结果的概率[31]。其中,Goldenzweig等将序列保守性分析PSSM与蛋白质构象自由能计算Rosetta ddg相结合,使人源乙酰胆碱酯酶(hAChE) 的热熔融温度(melting temperature, Tm) 提高了20 ℃[32];Wijma等把分子动力学模拟及FoldX、Rosetta ddg能量分析及二硫键设计相结合,将环氧化物水解酶(LEH) 的Tm提高了35 ℃[33]。此外,吴边课题组[34]在多种计算策略的基础上,进一步提出GRAPE策略,对有益突变进行聚类分析和贪婪累积,实现了高效的组合路径。
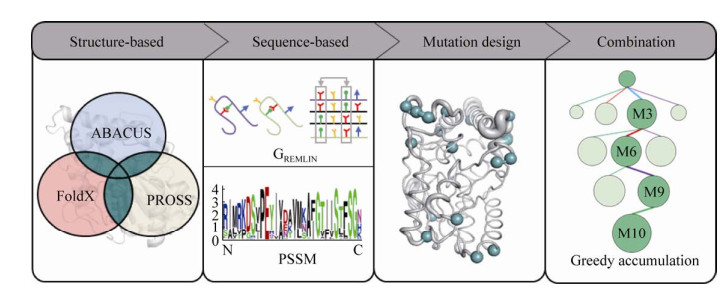
本研究以PML作为模式蛋白,利用PML的三维结构和序列信息,通过ABACUS、PROSS以及FoldX三种正向筛选以及GREMLIN和PSSM两种负向筛选,得到潜在的热稳定性提高的单点突变。通过贪婪累积策略进行组合,以期提高PML的热稳定性,进一步拓展其在工业上的应用。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒大肠杆菌(Escherichia coli) JM109、E. coli BL21(DE3) 由本实验室保存;表达载体pET-28a(+) 购自Novagen公司;奇异变形杆菌(Proteus mirabilis) 脂肪酶基因由苏州金唯智生物科技有限公司合成。
1.1.2 主要试剂Primer StarTM HS DNA聚合酶、DL-10 000 DNA marker及Premixed Protein marker (Low)均购于TaKaRa公司;琼脂糖凝胶DNA回收试剂盒和质粒小提试剂盒购于Axygen公司;BCA蛋白浓度测定试剂盒(增强型) 和聚丙烯酰胺凝胶电泳(SDS-PAGE) 试剂盒购于碧云天公司;卡那霉素(kanamycin, Kan)、异丙基-β-D-硫代半乳糖苷(IPTG) 购于生工生物工程(上海) 股份有限公司;HisTrapTM HP 1 mL Ni2+柱以及HiTripTM 5 mL Desalting柱购于美国GE医疗集团;PCR引物均由上海亦欣公司合成;ρ-nitrophenyl laurate (ρNPL) 和ρ-nitrophenol (ρNP) 购于Sigma-Aldrich公司;其余试剂无特殊说明,均为市售分析纯。
1.2 方法 1.2.1 奇异变形杆菌脂肪酶的计算设计如图 1所示,本研究基于奇异变形杆菌脂肪酶PML的三维结构和序列信息,将ABACUS、PROSS以及FoldX三种策略作为正筛,将GREMLIN和PSSM作为负筛,通过多重计算设计筛选能提高热稳定性的突变体,针对阳性突变利用贪婪累积算法进行组合。
|
| 图 1 计算设计策略示意图 Fig. 1 Schematic diagram of computational design. |
| |
首先,分别利用ABACUS 2在线服务器(http://biocomp.ustc.edu.cn/servers/abacus-design.php) 和PROSS在线服务器(https://pross.weizmann.ac.il./step/pross-terms/) 对野生型PML (WT) 进行计算设计;利用FoldX中提供的PositionScan,对PML的287个残基进行虚拟饱和突变得到所有突变的DDG (DDG=DGMut–DGWt),将DDG < –0.45 kcal/mol的突变保留。选择在上述两种及两种以上的计算中同时出现的设计作为本轮的正向筛选结果。
其次,利用PML序列信息对上一轮的单点突变进行负向筛选。通过在GREMLIN服务器(http://openseq.org/submit.php) 上提交PML-WT的序列,可以得到其中共进化的残基对及其归一性打分,剔除GREMLIN中得分 > 0.7的各保守残基对中相关位点的突变设计;PSSM得分以PROSS的比对结果为准,保留在PSSM中得分 > 0的突变设计。
对计算设计得到的单点突变,测定其稳定性和酶活,将稳定性提高的单点通过贪婪累积策略进行组合。以单点突变中热稳定性最好的突变作为下一轮突变的出发点,分别叠加其他阳性突变得到两点组合突变体,再以该轮突变中热稳定性最好的两点组合突变体为出发点,分别叠加其他单点阳性突变,得到三点组合突变,以此类推,直到组合突变的热稳定性降低或所有阳性突变的叠加均已完成为止。
1.2.2 定点突变以含有奇异变形杆菌脂肪酶基因的pET28a(+)-PML质粒作为模板,根据突变位点设计引物进行全质粒PCR扩增,琼脂糖凝胶电泳验证后回收PCR产物,利用热激转化法将其转化至E. coli BL21(DE3) 感受态细胞中。筛选得到的阳性转化子送至无锡天霖生物科技有限公司进行DNA测序验证,获得目标菌株。本研究所使用的引物如表 1所示。
| Primer name | Primer sequence (5′→3′) | Size (bp) |
| N17V-F | CTGGCGGGCTTTGTGGAAATTGTG | 24 |
| N17V-R | CACAAAGCCCGCCAGGCCATGCAC | 24 |
| E18R-F | CGGGCTTTAACCGTATTGTGGGCT | 24 |
| E18R-R | CGGTTAAAGCCCGCCAGGCCATGC | 24 |
| Q38R-F | AAGATGGCCATCGTGTGTTTACCG | 24 |
| Q38R-R | ATGGCCATCTTGGCGCAGCGCATC | 24 |
| Q66A-F | AGACCCTGCTGGCAGAAACCCAAG | 24 |
| Q66A-R | AGCAGGGTCTGCACAAACTGCCAC | 24 |
| V132I-F | TGGAAAAAATTCTGAACGCGTTTG | 24 |
| V132I-R | TCCACAATATATTCCGGAATGC | 22 |
| G148D-F | CGGCCATAGAGACGATCCGCAAG | 23 |
| G148D-R | CTCTATGGCCGCTAAAGGTGC | 21 |
| D152N-F | CCGCAACATGCGATTGCGGCG | 21 |
| D152N-R | CGCATGTTGCGGATCGCCTC | 20 |
| A175G-F | CAAATATCCGCAAGGCCTGCCG | 22 |
| A175G-R | TTGCGGATATTTGTTGTTAAATTC | 24 |
| G206N-F | GGCCTGATTGCGAACGAAAAAGGC | 24 |
| G206N-R | TCGCAATCAGGCCTTGAAT | 19 |
| G206D-F | GGCCTGATTGCGGACGAAAAAGGC | 24 |
| G206D-R | CCGCAATCAGGCCTTGAATAT | 21 |
| K208G-F | GCGGGCGAAGGCGGCAACCTGCTG | 24 |
| K208G-R | GCCCGCAATCAGGCCTTGAATA | 22 |
| G209L-F | ATTGCGGGCGAAAAACTGAACC | 22 |
| G209L-R | TCGCCCGCAATCAGGCCTTGA | 21 |
| A217R-F | GATCCGACCCATCGTGCGATGCG | 23 |
| A217R-R | ATGGGTCGGATCCAGCAGGTTG | 22 |
| N223A-F | GCGCGTGCTGGCCACCTTTTTTAC | 24 |
| N223A-R | CCAGCACGCGCATCGCCGCATG | 22 |
| A251P-F | AGATGATTATCCGCAAGATCATA | 23 |
| A251P-R | ATAATCATCTTTAATCAGTTTGC | 23 |
| I255L-F | TCATCTTTAATCAGTTTGCCCAG | 24 |
| I255L-R | ATAATCATCTTTAATCAGTTTGC | 24 |
| Q286G-F | TGGCGAGCAAAGGTCTGTAAGCG | 23 |
| Q286G-R | TGCTCGCCAGATATTTCGCATG | 22 |
将保存于甘油管的E. coli BL21(DE3)/ pET28a(+)-PML野生型及突变型菌株涂布于含25 μg/mL Kan的LB固体平板,于37 ℃恒温培养箱中培养12−16 h。挑取单菌落于20 mL含25 μg/mL Kan的LB液体培养基中,37 ℃、200 r/min振荡培养12 h。以1%接种量接入100 mL含25 μg/mL Kan的TB液体培养基,37 ℃、200 r/min振荡培养至OD600=0.6时,加入终浓度为0.5 mmol/L的IPTG诱导,16 ℃、200 r/min继续培养16 h后,8 000 r/min离心5 min收集发酵菌体。
用25 mL上样缓冲液(50 mmol/L Tris-HCl,300 mmol/L NaCl,10 mmol/L咪唑,pH 8.0) 重悬菌体,在冰浴条件下超声破碎菌体(超声2 s,间歇3 s,20 min) 后,10 000 r/min离心20 min收集上清,并用0.45 μm滤膜过滤,利用镍柱亲和层析纯化目的蛋白。纯化条件:首先用上样缓冲液平衡HisTrapTM HP 1 mL Ni2+亲和层析柱15个柱体积,将过滤后的上清液以1 mL/min的流速进样,等待进样结束后,使用5个柱体积的上样缓冲液平衡;用10%的洗脱缓冲液(50 mmol/L Tris-HCl,300 mmol/L NaCl,250 mmol/L咪唑,pH 8.0) 进行梯度洗脱除去杂蛋白,再用30%的洗脱缓冲液梯度洗脱10个柱体积,对目的蛋白进行收集。对经过亲和层析的目的蛋白用HiTripTM 5 mL Desalting凝胶柱进行脱盐处理,并更换至20 mmol/L Tris-HCl缓冲液(pH 8.0) 中,测量其浓度并用SDS-PAGE对其纯度进行验证。于0 ℃条件下储存酶液备用。
1.2.4 野生型及突变体蛋白的酶活测定奇异变形杆菌脂肪酶活力分析是通过水解ρNPL所释放的ρNP的量来测定。总体积2 mL的底物体系包括:1 880 μL反应缓冲液(20 mmol/L Tris-HCl,5 mmol/L CaCl2,pH 8.0);20 μL Triton X-100及100 μL浓度为50 mmol/L的溶于乙腈的ρNPL溶液。充分混匀后以每孔190 μL分装至96孔板中,35 ℃下孵育后加入适当稀释的酶液10 μL,用酶标仪测定2 min内410 nm处的吸光值变化。以相同条件下缓冲液的测定作为空白对照,每次设3组平行试验。
上述条件下,将每分钟水解ρNPL生成1 μmol ρNP所需的酶量定义为1个酶活单位(U)。总酶活力除以酶浓度得到比活,单位为U/mg。
1.2.5 野生型及突变体的热稳定性测定利用圆二色光谱(CD) 测定蛋白质二级结构及Tm。将纯化得到的酶液取适量稀释至0.2 mg/mL,取200 μL加入到光程为0.1 cm的石英比色皿中,用圆二色光谱仪测定其在4 ℃条件下,190–260 nm的摩尔椭圆率。根据全波长扫描来确定蛋白折叠情况。选择最低吸收值处的波长220 nm,将温度从4 ℃升温至80 ℃ (升温速率1 ℃/min),测定其摩尔椭圆率变化得到其溶解曲线。将得到的曲线通过波兹曼拟合,取蛋白解折叠至一半的温度作为Tm。
将纯化后的酶液分别在30−70 ℃ (间隔5 ℃) 下孵育10 min,放置于冰上冷却10 min后,采用上述1.2.4方法测定不同孵育温度下的酶活力。将未孵育条件下的酶活力设为100%,计算各温度条件下的相对比活。相对剩余酶活力降为50%时所对应的温度,即半失活温度(T50)。
将纯化后的酶液在50 ℃下孵育0−90 min (间隔10 min),放置于冰上冷却10 min后,采用上述1.2.4方法测定不同孵育时间下的酶活力。将未孵育条件下的酶活力设为100%,计算各孵育时间下的相对比活。相对剩余酶活力降为50%时所对应的时间,即为半失活时间(t1/2)。
1.2.6 突变体的分子动力学模拟基于PML的晶体结构(PDB ID: 4GW3),在Pymol (http://www.pymol.org) 中进行氨基酸替换得到突变体的三维结构。以该结构作为初始结构,基于AMBER99SB-ILDN力场利用Gromacs 5.0.4进行分子动力学模拟[35]。将蛋白分子放置在正方体的水盒子中,用TIP3P模型[36]的水分子填充溶剂及离子使系统呈中性后,用steepest descent方法[37]进行能量最小化。利用velocity rescaling算法保持体系温度恒定为25 ℃,并且使用弱耦合方法控制压力为1 bar[38],50 ns NPT的模型优化后进行150 ns NVT的平衡模拟,其中模拟时间步长设置为2 fs。
氢键定义:当氢供体氮原子与氢受体氧原子之间的距离小于3.5 Å,且∠HNO小于30°时氢键存在[39-40]。盐桥定义:由带正电残基羧酸酯基团的氧原子与带负电残基酰胺基团的氮原子之间的距离确定,当两原子之间的距离小于4.0 Å时盐桥存在[40-41]。分子动力学模拟的最后50 ns中,成键的时间占比为对应成键的占有率。
2 结果与分析 2.1 奇异变形杆菌脂肪酶的计算设计利用两轮筛选进行奇异变形杆菌脂肪酶的计算设计。第一轮筛选中结合了ABACUS、PROSS和FoldX三种设计策略,选取三者两两相交的部分作为正向筛选的结果,共计得到了67个突变设计。随后,利用GREMLIN和PSSM对以上设计进行负向筛选,仅保留GREMLIN归一性打分 < 0.7的位点及PSSM > 0的设计,最终获得了18个单点突变设计(表 2)。18个设计中,Q69G、A217R等5个设计来自3种正向筛选器的交集,N17V等6种设计来自ABACUS和FoldX的交集,E18R等5种设计来自PROSS和FoldX的交集,Q66A和K208G来自ABACUS和PROSS的交集。
| Mutant | ABACUS | PROSS | FoldX (kcal/mol) | Intersection | PSSM | GREMLIN |
| N17V | √ | –0.71 | ABACUS+FoldX | 3 | / | |
| E18R | √ | –0.50 | PROSS+FoldX | 6 | / | |
| Q38R | √ | –0.52 | ABACUS+FoldX | 4 | / | |
| Q66A | √ | √ | –0.13 | ABACUS+PROSS | 5 | / |
| Q69G | √ | √ | –1.86 | ABACUS+PROSS+FoldX | 7 | / |
| V132I | √ | –0.61 | ABACUS+FoldX | 3 | / | |
| G148D | √ | –1.38 | ABACUS+FoldX | 1 | / | |
| D152N | √ | –0.64 | PROSS+FoldX | 5 | / | |
| A175G | √ | –1.81 | PROSS+FoldX | 7 | / | |
| G206N | √ | –0.48 | ABACUS+FoldX | 3 | / | |
| G206D | √ | –0.48 | PROSS+FoldX | 6 | / | |
| K208G | √ | √ | –0.26 | ABACUS+PROSS | 7 | / |
| G209L | √ | –1.03 | PROSS+FoldX | 3 | / | |
| A217R | √ | √ | –0.46 | ABACUS+PROSS+FoldX | 2 | / |
| N223A | √ | √ | –1.05 | ABACUS+PROSS+FoldX | 2 | / |
| A251P | √ | √ | –1.35 | ABACUS+PROSS+FoldX | 8 | / |
| I255L | √ | √ | –0.68 | ABACUS+PROSS+FoldX | 5 | / |
| Q286G | √ | –2.01 | ABACUS+FoldX | 7 | / | |
| Note: “/” indicates that there was no co-evolution or GREMLIN score was less than 0.7 at the site where the design was located. | ||||||
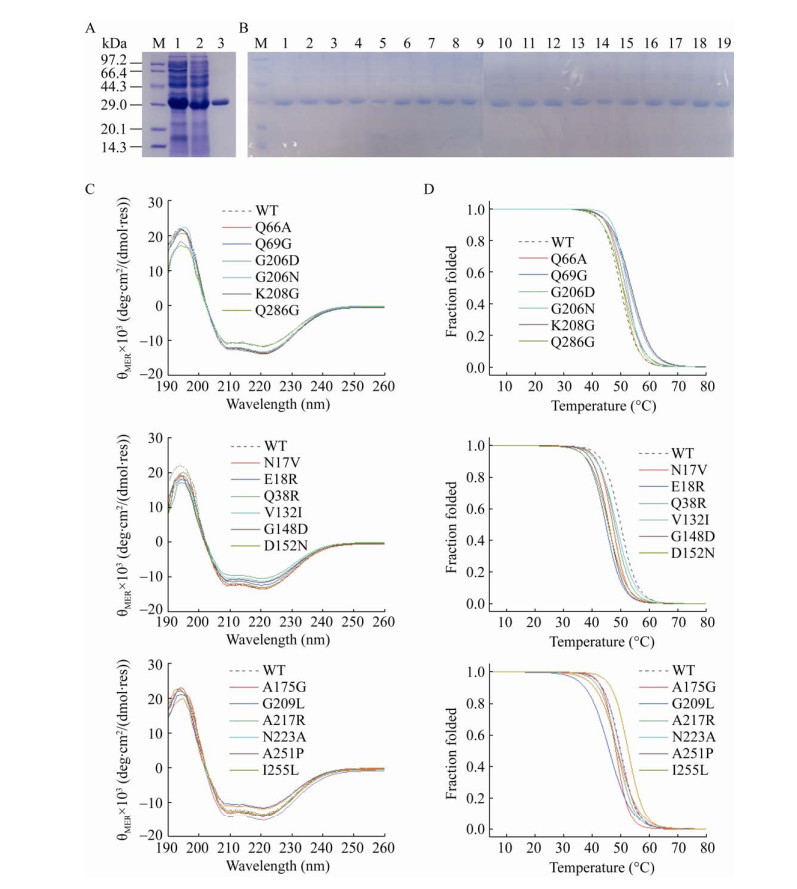
分别收集IPTG诱导后的PML-WT的全细胞、上清以及纯化后获得的酶液进行SDS-PAGE鉴定。SDS-PAGE中目的蛋白条带大小约为33 kDa (图 2A),与理论值相符。测定野生型奇异变形杆菌脂肪酶的比活为(33.48±0.68) U/mg。用表 1中的引物进行全质粒PCR突变,将突变的质粒测序验证后,进行蛋白的表达纯化。所有的单点突变均能在大肠杆菌中可溶性表达,且条带对应分子量大小正确,纯度均达到了电泳纯(图 2B)。
|
| 图 2 野生型PML及单点突变体的表达纯化及其圆二色谱分析 Fig. 2 Expression, purification, and circular dichromatography of wild-type PML and single-point mutants. (A) M: premixed protein marker; 1–2: total and soluble protein of E. coli BL21(DE3) cells harboring pET28a(+)-PML, respectively; 3: purified wild type PML. (B) M: premixed protein marker; 1: purified wild type PML; 2–19: purified protein of mutant N17V, E18R, Q38R, Q66A, Q69G, V132I, G148D, D152N, A175G, G206N, G206D, K208G, G209L, A217R, N223A, A251P, I255L, Q286G. (C) Full-wavelength scans. res: residue number. (D) Melting curves. |
| |
为表征WT与突变体在二级结构上的差异,用圆二色谱仪对WT及单点突变体在190–260 nm进行全波长扫描(图 2C)。根据全波长扫描结果可知,所有突变蛋白的扫描结果与野生型变化趋势相同,在210 nm和220 nm附近均有典型负峰,说明突变体全都折叠形成了与WT一致的二级结构。
为了表征WT和突变体热稳定性的差异,用圆二色谱仪测定其熔融曲线(图 2D),各突变体的Tm如表 3所示。根据Tm测定结果可知,Tm值上升的突变体有7个,ΔTm为0.78–3.75 ℃不等。其中提高3 ℃以上的单点突变体有K208G (3.75 ℃) 和G206D (3.21 ℃)。选择Tm高于野生型的单点突变测定其T50和t1/2,其变化趋势同Tm基本保持一致。7个单点突变体的T50较WT增加了0.66–3.83 ℃不等,t1/2均略高于WT。利用酶标仪进一步测定了单点突变体的相对比活。18个单点突变中,有12个单点突变的比活高于WT。A175G、G206N、G206D和K208G四个突变体的比活超过了WT的130%,其中G206D比活最高,达到了WT的137%。
| Mutant | Tm (℃) | ΔTm (℃) | T50 (℃) | t1/2 (min) | Relative activity (%) |
| WT | 49.96 | 0.00 | 56.70 | 59.30 | 100.00 |
| N17V | 47.20 | –2.76 | – | – | 66.92 |
| E18R | 44.76 | –5.20 | – | – | 80.93 |
| Q38R | 47.97 | –1.99 | – | – | 128.87 |
| Q66A | 51.74 | 1.78 | 58.63 | 61.36 | 118.08 |
| Q69G | 50.74 | 0.78 | 57.36 | 59.75 | 110.48 |
| V132I | 48.50 | –1.46 | – | – | 112.94 |
| G148D | 45.79 | –4.17 | – | – | 127.56 |
| D152N | 45.64 | –4.32 | – | – | 71.93 |
| A175G | 48.96 | –1.00 | – | – | 133.33 |
| G206N | 50.87 | 0.91 | 57.92 | 59.25 | 135.56 |
| G206D | 53.17 | 3.21 | 59.23 | 63.89 | 137.28 |
| K208G | 53.71 | 3.75 | 60.53 | 65.68 | 130.18 |
| G209L | 48.29 | –1.67 | – | – | 112.31 |
| A217R | 46.05 | –3.91 | – | – | 83.55 |
| N223A | 48.82 | –1.14 | – | – | 117.82 |
| A251P | 49.83 | –0.13 | – | – | 76.17 |
| I255L | 52.63 | 2.67 | 58.06 | 62.85 | 75.03 |
| Q286G | 52.36 | 2.40 | 57.87 | 60.52 | 124.54 |
| Note: “–” indicates that the data has not been determined by experiment. | |||||
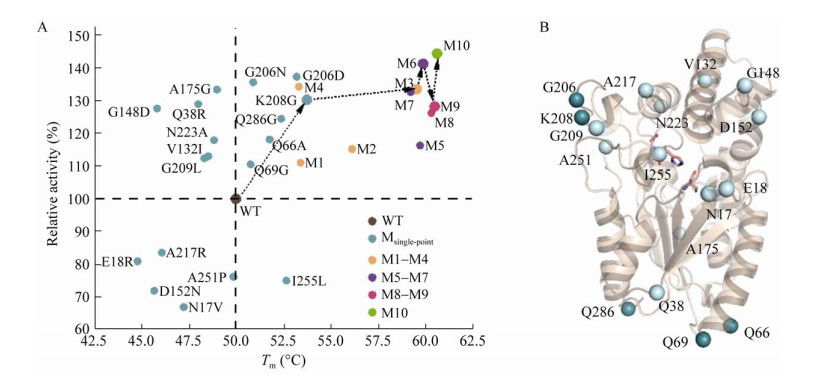
根据Tm及比活的大小,18个单点突变体被分成了4大类(图 3A)。第一大类中,Tm及比活均高于WT,包括K208G、G206D、G206N、Q286G、Q66A及Q69G六个突变体。其中,K208G的热稳定性最强(ΔTm=3.75 ℃)、相对比活也达到了WT的130%。206位相关的两个设计(G206N及G206D) 比活力相似,均为WT的135%左右,但G206D的Tm较G206N高出2 ℃以上。其他3个突变体的Tm及比活的大小关系均为Q286G > Q66A > Q69G。K208G及G206D是所有突变体中热稳定性及比活力最高的两个突变体,这可能与两个位点在PML结构中的特殊位置有关(图 3B)。两个位点均位于盖子区域附近的loop上,该区域柔性较大,且靠近Ca2+结合位点。两个位点的突变引入可能会对Ca2+结合位点的稳定起到积极作用。第二大类中,Tm低于WT,比活高于WT,包含A175G等6个突变体。第三大类中,Tm高于WT,比活低于WT,仅包含I255L一个突变体,其比活仅有WT的75%左右,猜测这可能是由于该位点过于靠近活性中心,突变可能引起活性中心构象的变化(图 3B)。第四类中的突变体有5个,其Tm和比活均低于WT。
|
| 图 3 突变体的酶活、Tm及各位点在PML晶体结构中的位置 Fig. 3 Activity and Tm of mutants and mutation sites in PML crystal structure. (A) The dots of the same color contain mutations from the same parent. The dotted arrow indicates the combined path from WT (brown) to the optimal mutant M10 (green), which contains the best mutant in each round of stacking: K208G (dark cyan), M3 (orange), M6 (purple), M9 (pink). The horizontal and vertical dotted lines represent the enzyme activity and Tm of WT, respectively. (B) Mutational sites shown in wild type PML structure. The mutants with higher Tm and enzyme activity than WT have a darker color. The catalytic triad of the enzyme are shown as stick. |
| |
通过以上酶学性质的表征发现,在18个筛选出的单点突变设计中,有7个设计热稳定性提升。这7个设计分别涵盖了来自三者共同交集的2个(Q69G及I255L);来自ABACUS和FoldX交集中的2个(G206N及Q286G);来自PROSS和FoldX交集中的1个(G206D);来自ABACUS和PROSS交集中的2个(Q66A及K208G)。这与其他利用多重计算设计取交集以富集阳性突变的研究[31, 42]一致,也说明不同设计策略之间具有一定的互补性。吴边等[34]的研究中,通过将多种算法取并集的方式,从85个候选设计中得到了21个Tm提高超过1.5 ℃的塑料降解酶(PETase) 突变体,本研究中取交集的方式在保证筛选效率相当的情况下提供了一种工作量更小的手段,当然也承担了错过其他更好突变体的可能性。此外,7个较优突变体的PSSM得分除G206N外均在5−7之间,说明PML的序列保守性分析可以一定程度辅助其热稳定性的计算设计。
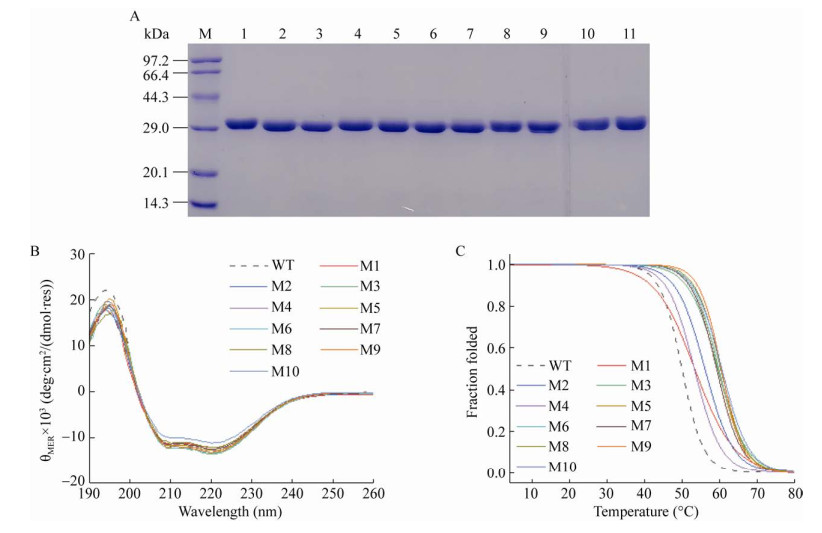
2.4 组合突变体的酶学性质为了得到热稳定性更好的突变体,同时考察突变位点之间的上位性,用贪婪累积策略对5个热稳定性及酶活均优于WT的单点突变(K208G、G206D、Q286G、Q66A及Q69G) 进行组合(由于G206D在热稳定性及酶活上均优于G206N,因此选择G206D进行组合)。经过4轮的贪婪累积后得到了10个组合突变体。所有的组合突变体均能在大肠杆菌中可溶性表达,且纯化后纯度均达到了电泳纯(图 4A)。全波长扫描结果显示,所有组合突变体均折叠形成了正确的二级结构(图 4B)。
|
| 图 4 野生型PML及组合突变体的表达纯化及其圆二色谱分析 Fig. 4 Expression, purification, and circular dichromatography of wild-type PML and its combination mutants. (A) Wild type PML and combined mutants expression in E. coli BL21(DE3). M: premixed protein marker; 1: purified wild type PML; 2–11: purified protein of mutant M1, M2, M3, M4, M5, M6, M7, M8, M9, M10. (B) Full-wavelength scans. res: residue number. (C) Melting curves. |
| |
组合突变体的熔融曲线如图 4C所示,其Tm、T50、t1/2和相对比活结果如表 4所示。第一轮两点组合突变体中,M3 (K208G+G206D)和M2 (K208G+Q69G) 的ΔTm分别为9.55 ℃和6.14 ℃,均高于其单点突变的ΔTm之和(6.96 ℃及4.53 ℃),说明K208G与G206D、K208G与Q69G之间存在一定的协同作用。M1 (K208G+Q66A) 及M4 (K208G+Q286G) 的ΔTm较K208G分别下降0.32 ℃和0.42 ℃,说明Q66A及Q286G与K208G的叠加均出现了负的上位性。四个突变体的T50较WT高出2.64–7.86 ℃,其中M3最高,比K208G高出4.03 ℃。t1/2提高的组合突变体有3个,其中M3较K208G提高了21.29 min。四个突变体的比活均高于WT,其中,M3及M4的比活与K208G接近,均达到了WT的130%以上。
| Mutant | Tm (℃) | ΔTm (℃) | T50 (℃) | t1/2 (min) | Relative activity (%) |
| WT | 49.96 | 0.00 | 56.70 | 59.30 | 100.00 |
| M1 (K208G+Q66A) | 53.39 | 3.43 | 60.35 | 66.59 | 110.96 |
| M2 (K208G+Q69G) | 56.10 | 6.14 | 59.34 | 79.46 | 115.15 |
| M3 (K208G+G206D) | 59.51 | 9.55 | 64.56 | 86.97 | 133.45 |
| M4 (K208G+Q286G) | 53.29 | 3.33 | 59.52 | 64.58 | 134.20 |
| M5 (M3+Q66A) | 59.70 | 9.74 | 63.96 | 90.52 | 116.20 |
| M6 (M3+Q69G) | 59.86 | 9.90 | 65.36 | 91.53 | 141.21 |
| M7 (M3+Q286G) | 59.20 | 9.24 | 64.58 | 88.65 | 132.50 |
| M8 (M6+Q66A) | 60.29 | 10.33 | 66.32 | 90.36 | 126.14 |
| M9 (M6+Q286G) | 60.46 | 10.50 | 66.85 | 95.66 | 128.17 |
| M10 (M9+Q66A) | 60.59 | 10.63 | 67.73 | 98.11 | 144.36 |
| Note: bold numbers indicate the maximum value of ΔTm in each round of greed accumulation, and the mutant is the parent of next round of accumulation. | |||||
选择M3作为起点进行了第二轮贪婪累积。三个组合突变体的Tm均在59–60 ℃之间。M6 (M3+Q69G) 及M5 (M3+Q66A) 的ΔTm分别为9.90 ℃和9.74 ℃,较M3分别提高了0.35 ℃和0.16 ℃,M7 (M3+Q286G) 则比M3低0.31 ℃,说明Q69G、Q66A两个突变与M3之间的协同作用并不明显,Q286G与M3之间可能存在一定负上位性。M6及M7的T50均高于M3,其中M6比M3高0.80 ℃。M5的T50较M3有所下降。三个组合突变体的t1/2均高于M3,其中M6最高,比M3提高了4.56 min,此外,M6的比活在M3基础上进一步上升,达到了WT比活的140%,M7的比活与M3相似,M5则低于M3。
选择M6作为第3轮贪婪累积的起点得到M8 (M6+Q66A) 及M9 (M6+Q286G)。两个组合突变体的ΔTm分别为10.33 ℃和10.50 ℃,较M6提高了0.63 ℃及0.43 ℃。M8及M9的T50较M6分别提高了0.96 ℃和1.49 ℃。M9的t1/2较M6提高了4.13 min,M8较M6下降了1.17 min。两个突变体的比活相近,但均低于M6,仅为WT的127%左右。
最后一轮贪婪累积得到了五点组合突变体M10 (M9+Q66A),其Tm达到了60.59 ℃,较WT提高了10.63 ℃,T50为67.73 ℃,t1/2为98.11 min,分别比WT提高了11.03 ℃和38.81 min。此外,其比活也是所有组合突变体中最高的,为WT的144.36%。
以WT为出发点,通过单点突变和四轮贪婪累积构建出了WT-K208G-M3-M6-M9-M10组合路径,突变体的热稳定性逐步提高(ΔTm:3.75-9.55-9.90-10.50-10.63 ℃),且酶活均稳定到了WT的130%左右(图 3A,表 4)。其中需要注意的是,ΔTm提高幅度最大的一步是K208G-M3,ΔΔTm为5.80 ℃,而M3–M10的ΔΔTm仅为1.08 ℃;K208G、G206D单点突变和各相关组合突变体酶活均在130%左右。从热稳定性与酶活两方面推测,K208G与G206D这两个位置相近的突变体之间的协同作用为M10性质的改善提供了主要贡献。另外,组合路径中,前两轮组合突变的效果较好,随后的叠加没有发生较大变化,这种现象与以往的发现[34, 43]一致,表现较好的突变体一般在组合的早期出现,由于上位性效应,对两个或三个以上的突变体进行组合不一定能得到持续改进酶性质的突变体。
2.5 突变体的分子动力学模拟为了进一步研究突变对蛋白质结构的影响,对WT以及五点组合变体M10进行了150 ns的分子动力学模拟。利用主链原子的均方根偏差(root mean square deviation, RMSD) 表征系统整体的稳定性(图 5A)。通常一个稳定的体系其RMSD应小于3.2 Å[44]。从RMSD的变化趋势可以看出,WT及M10在50 ns后均基本达到平衡状态。50–150 ns中,WT和M10的平均RMSD分别为(1.30±0.12) Å和(1.61±0.13) Å,均远小于3.2 Å,这意味着50–150 ns的模拟是稳定且具有代表性的,可以进行后续的分析。
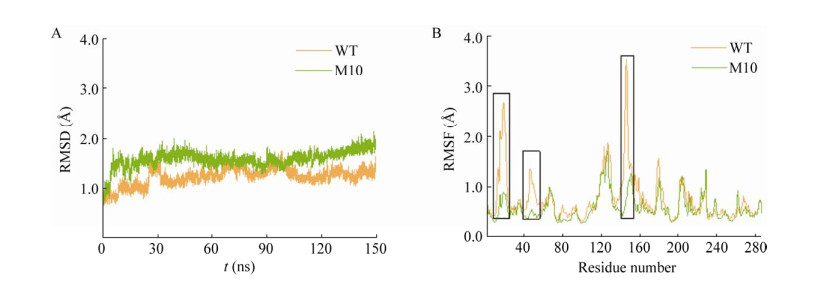
|
| 图 5 野生型PML及突变体M10的分子动力学模拟分析 Fig. 5 Molecular dynamics simulation for wild type (brown) and mutant M10 (green). (A) The root mean square deviation (RMSD). (B) The root mean square fluctuation (RMSF). |
| |
为了表征单个残基波动对蛋白质整体动力学的贡献,选择模拟的后50 ns计算得到均方根涨落(root mean square fluctuation, RMSF) (图 5B)。除了少数特殊区域(黑色方框) 外,WT和M10之间的大多数残基波动大小相似。在3个黑框区域中,M10的波动均小于WT,这3个区域均是盖子附近的柔性区域,突变的引入,减小了局部构象的波动性,更有利于PML结构稳定性的提高。
为了进一步从分子层面探析引入突变对酶学性质的影响,分别在WT和M10达到平衡后,针对突变位点附近的作用力进行分析,结果如图 6所示,所涉及的氢键及盐桥占有率如表 5所示。5个突变位点可以分为3个区域。G206/K208处于盖子α8附近的loop区,该区域靠近Ca2+结合位点,G206D/K208G两个突变位点的引入,在残基205–209附近区域引入了6个新的作用力(图 6A,6B),实现了这一区域作用力的重排。G206D突变在该区域引入了一个携带负电荷的酸性氨基酸残基,K208G在该区域消除了一个带正电的碱性氨基酸残基,这种电荷的改变可能会一方面对Ca2+的结合产生促进作用,另一方面改变所在loop的原有的运动模式,使E207发生了朝向盖子方向的偏移。这种偏移使E207与R220形成稳定的盐桥,同时与A205主链之间形成氢键。这两个相对稳定的作用力进一步促成了D206与G208主链之间、A205与E207的侧链之间新的氢键形成。G209与V265、N268形成的氢键紧靠Ca2+结合位点,可能会进一步提升Ca2+的稳定性,从而使热稳定性得到提高。这也解释了实验中发现的G206D与K208G之间的协同作用。虽然从氢键占有率看来,这种作用力重排的构象不是持续稳定存在的,但是考虑到该区域本身柔性较大,这种较WT而言出现的新构象可能为其功能的提升做出了一定贡献。Q66/Q69位于α2末端,M10中观察到Q66A与L64以及L65与T68之间形成了新的氢键(图 6C,6D),可能进一步增强了该区域α螺旋的稳定性;Q286位于C末端,在M10中,Q286G与L282、S284形成了新的氢键(图 6E,6F),增强了C末端的刚性。相较于G206/K208区域,后两区域新形成的氢键占有率较低,仅在5%左右,这与组合路径中观察到M3–M10的热稳定性的小幅度上升相吻合。以上3个区域中这些新的作用力共同提高了PML整体的稳定性。
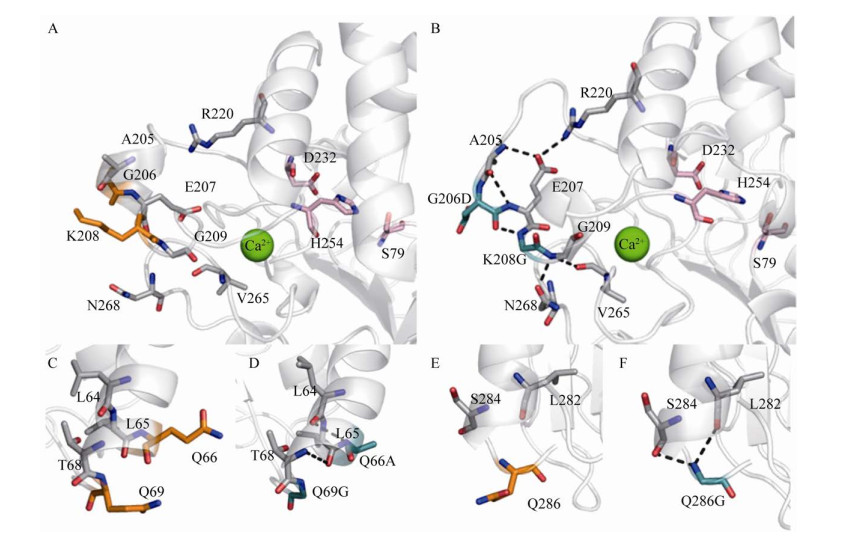
|
| 图 6 M10热稳定性提高的机理分析 Fig. 6 Mechanism for improved thermal stability of M10. (A) G206/K208 area of WT. (B) G206D/K208G area of M10. (C) Q66/Q69 area of WT. (D) Q66A/Q69G area of M10. (E) Q286 area of WT. (F) Q286G area of M10. The catalytic triad is represented by a pink stick model, and Ca2+ is represented by a green ball. The amino acids before and after the mutation are represented by orange and dark green respectively, the nearby amino acids are represented by silver, and the newly formed hydrogen bonds and salt bridges are represented by black dotted lines. |
| |
| Atom 1 | Atom 2 | Occupancy (%) | Bonding type | |
| WT | M10 | |||
| K/G208.N | G/D206.O | 0.0 | 23.5 | Hydrogen bond |
| E207.N | A205.O | 0.1 | 78.0 | Hydrogen bond |
| A205.N | E207.OE2 | 0.0 | 24.6 | Hydrogen bond |
| R220.NH2 | E207.OE2 | 7.9 | 99.0 | Salt bridge |
| G209.N | V265.O | 0.0 | 19.1 | Hydrogen bond |
| G209.N | N268.OD1 | 0.0 | 17.9 | Hydrogen bond |
| Q/A66.N | L64.O | 0.0 | 6.8 | Hydrogen bond |
| T68.N | L65.O | 0.3 | 5.0 | Hydrogen bond |
| Q/G286.N | L282.O | 0.0 | 3.9 | Hydrogen bond |
| Q/G286.N | S284.O | 0.2 | 4.7 | Hydrogen bond |
类似G206D/K208G区域作用力重排的现象在PML的其他研究中也被发现[15]:利用高通量筛选得到的高甲醇耐受性的PML变体包含G202E及K208N突变,通过X射线晶体衍射观察到聚集在Ca2+结合位点附近的相同loop被重塑,且E207与盖子α8上的R220也形成了盐桥。结合本研究结果推测,PML相较铜绿假单胞菌脂肪酶(Pseudomonas aeruginosa lipase, PAL) 在Ca2+结合位点附近多出的5个残基组成的loop[12]是与其生物学性质紧密相关的热点区域,它不仅会影响酶活、热稳定性,还会影响有机溶剂耐受性,这可以为后续PML的改造提供参考。
3 讨论与结论基于结构和序列信息构建起来的多种计算方法为天然蛋白质的热稳定性改造提供了更精确且省时省力的途径。本研究中,我们综合了多重计算设计,建立了以ABACUS、PROSS以及FoldX三种互补算法的两两交集为基准,利用GREMLIN和PSSM进一步减小突变文库的计算设计方案。以PML作为模式蛋白,利用此筛选方案得到的18个单点突变设计中,有7个热稳定性提高的突变体,突变效率达到了38.9%,且其中大部分的突变体较好地保存了酶活力,证明了这种综合已有通用计算方法和程序的计算策略在兼顾用户友好性的同时具有较高的筛选效率,可以用于其他酶的热稳定性改造。选择热稳定性和酶活均高于WT的5个突变体,采用贪婪累积策略进行组合突变,获得10个组合突变体。在组合路径中K208G和G206D的组合展现出较好的上位性,其两点组合突变体的热稳定性提高了9.55 ℃;最终五点组合突变体M10的热稳定性比WT高出10.63 ℃,同时其酶活达到了WT的140%。利用分子动力学模拟对突变引起的结构变化进行初步研究,发现M10的部分柔性区域波动小于WT;作用力分析表明突变位点附近有新的作用力生成,其中G206D与K208G突变不仅增强了所在loop的刚性,还产生了新的稳定相互作用,并潜在地稳定了对稳定性和活性至关重要的Ca2+结合位点,为M10整体热稳定性的提高提供了主要贡献,其所在loop也是经定向进化筛选出的对热稳定性有重要影响的热点区域,可以成为PML后续计算改造的重点关注区域。本研究为提高天然酶热稳定性提供一种高效精巧的计算设计方案,同时为PML的进一步工业应用提供了基础。
| [1] |
Naik S, Basu A, Saikia R, et al. Lipases for use in industrial biocatalysis: specificity of selected structural groups of lipases. J Mol Catal B Enzym, 2010, 65(1/2/3/4): 18-23.
|
| [2] |
Yu XW, Zhu SS, Xiao R, et al. Conversion of a Rhizopus chinensis lipase into an esterase by lid swapping. J Lipid Res, 2014, 55(6): 1044-1051. DOI:10.1194/jlr.M043950
|
| [3] |
Chandra P, Enespa, Singh R, et al. Microbial lipases and their industrial applications: a comprehensive review. Microb Cell Fact, 2020, 19(1): 169. DOI:10.1186/s12934-020-01428-8
|
| [4] |
Filho DG, Silva AG, Guidini CZ. Lipases: sources, immobilization methods, and industrial applications. Appl Microbiol Biotechnol, 2019, 103(18): 7399-7423. DOI:10.1007/s00253-019-10027-6
|
| [5] |
Global Microbial Lipase Industry[EB/OL]. [2021-10-27]. https://www.reportlinker.com/p05899911/Global-Microbial-LipaseIndustry.html?utm_source=GNW.
|
| [6] |
Alquati C, de Gioia L, Santarossa G, et al. The cold-active lipase of Pseudomonas fragi. Heterologous expression, biochemical characterization and molecular modeling. Eur J Biochem, 2002, 269(13): 3321-3328. DOI:10.1046/j.1432-1033.2002.03012.x
|
| [7] |
Gao B, Su EZ, Lin JP, et al. Development of recombinant Escherichia coli whole-cell biocatalyst expressing a novel alkaline lipase-coding gene from Proteus sp. for biodiesel production. J Biotechnol, 2009, 139(2): 169-175. DOI:10.1016/j.jbiotec.2008.10.004
|
| [8] |
Lu YP, Lin Q, Wang J, et al. Overexpression and characterization in Bacillus subtilis of a positionally nonspecific lipase from Proteus vulgaris. J Ind Microbiol Biotechnol, 2010, 37(9): 919-925. DOI:10.1007/s10295-010-0739-0
|
| [9] |
Whangsuk W, Sungkeeree P, Thiengmag S, et al. Gene cloning and characterization of a novel highly organic solvent tolerant lipase from Proteus sp. SW1 and its application for biodiesel production. Mol Biotechnol, 2013, 53(1): 55-62. DOI:10.1007/s12033-012-9518-7
|
| [10] |
Shao H, Hu XM, Sun LP, et al. Gene cloning, expression in E. coli, and in vitro refolding of a lipase from Proteus sp. NH 2-2 and its application for biodiesel production. Biotechnol Lett, 2019, 41(1): 159-169. DOI:10.1007/s10529-018-2625-1
|
| [11] |
Misbah A, Koraichi SI, Jouti MAT. Purification, characterization and immobilization of lipase from Proteus vulgaris OR34 for synthesis of methyl oleate. Microbiol Biotechnol Lett, 2020, 48(4): 491-505. DOI:10.48022/mbl.2003.03010
|
| [12] |
Korman TP, Bowie JU. Crystal structure of Proteus mirabilis lipase, a novel lipase from the Proteus/psychrophilic subfamily of lipase family Ⅰ.1. PLoS One, 2012, 7(12): e52890. DOI:10.1371/journal.pone.0052890
|
| [13] |
Invernizzi G, Papaleo E, Grandori R, et al. Relevance of metal ions for lipase stability: structural rearrangements induced in the Burkholderia glumae lipase by calcium depletion. J Struct Biol, 2009, 168(3): 562-570. DOI:10.1016/j.jsb.2009.07.021
|
| [14] |
El Khattabi M, van Gelder P, Bitter W, et al. Role of the calcium ion and the disulfide bond in the Burkholderia glumae lipase. J Mol Catal B Enzym, 2003, 22(5/6): 329-338.
|
| [15] |
Korman TP, Sahachartsiri B, Charbonneau DM, et al. Dieselzymes: development of a stable and methanol tolerant lipase for biodiesel production by directed evolution. Biotechnol Biofuels, 2013, 6(1): 70. DOI:10.1186/1754-6834-6-70
|
| [16] |
Heater BS, Yang ZF, Lee MM, et al. In vivo enzyme entrapment in a protein crystal. J Am Chem Soc, 2020, 142(22): 9879-9883. DOI:10.1021/jacs.9b13462
|
| [17] |
Heater BS, Chan WS, Lee MM, et al. Directed evolution of a genetically encoded immobilized lipase for the efficient production of biodiesel from waste cooking oil. Biotechnol Biofuels, 2019, 12: 165. DOI:10.1186/s13068-019-1509-5
|
| [18] |
Schymkowitz J, Borg J, Stricher F, et al. The FoldX web server: an online force field. Nucleic Acids Res, 2005, 33(Web Server issue): W382-W388.
|
| [19] |
Guerois R, Nielsen JE, Serrano L. Predicting changes in the stability of proteins and protein complexes: a study of more than 1000 mutations. J Mol Biol, 2002, 320(2): 369-387. DOI:10.1016/S0022-2836(02)00442-4
|
| [20] |
Xiong P, Hu XH, Huang B, et al. Increasing the efficiency and accuracy of the ABACUS protein sequence design method. Bioinformatics, 2020, 36(1): 136-144. DOI:10.1093/bioinformatics/btz515
|
| [21] |
Zhou XQ, Xiong P, Wang M, et al. Proteins of well-defined structures can be designed without backbone readjustment by a statistical model. J Struct Biol, 2016, 196(3): 350-357. DOI:10.1016/j.jsb.2016.08.002
|
| [22] |
Xiong P, Wang M, Zhou XQ, et al. Protein design with a comprehensive statistical energy function and boosted by experimental selection for foldability. Nat Commun, 2014, 5: 5330. DOI:10.1038/ncomms6330
|
| [23] |
Kellogg EH, Leaver-Fay A, Baker D. Role of conformational sampling in computing mutation- induced changes in protein structure and stability. Proteins, 2011, 79(3): 830-838. DOI:10.1002/prot.22921
|
| [24] |
Park H, Bradley P, Greisen PJ, et al. Simultaneous optimization of biomolecular energy functions on features from small molecules and macromolecules. J Chem Theory Comput, 2016, 12(12): 6201-6212. DOI:10.1021/acs.jctc.6b00819
|
| [25] |
向玉, 张萌, 许菲. 基于多重计算设计策略提高枯草芽孢杆菌脂肪酶的热稳定性. 生物工程学报, 2020, 36(8): 1556-1567. Xiang Y, Zhang M, Xu F. Improving the thermal stability of Bacillus subtilis lipase based on multiple computational design strategies. Chin J Biotech, 2020, 36(8): 1556-1567 (in Chinese). |
| [26] |
Altschul SF, Gertz EM, Agarwala R, et al. PSI-BLAST pseudocounts and the minimum description length principle. Nucleic Acids Res, 2009, 37(3): 815-824. DOI:10.1093/nar/gkn981
|
| [27] |
Magliery TJ. Protein stability: computation, sequence statistics, and new experimental methods. Curr Opin Struct Biol, 2015, 33: 161-168. DOI:10.1016/j.sbi.2015.09.002
|
| [28] |
Balakrishnan S, Kamisetty H, Carbonell JG, et al. Learning generative models for protein fold families. Proteins, 2011, 79(4): 1061-1078. DOI:10.1002/prot.22934
|
| [29] |
Ovchinnikov S, Kamisetty H, Baker D. Robust and accurate prediction of residue-residue interactions across protein interfaces using evolutionary information. eLife, 2014, 3: e02030. DOI:10.7554/eLife.02030
|
| [30] |
Thomas A, Cutlan R, Finnigan W, et al. Highly thermostable carboxylic acid reductases generated by ancestral sequence reconstruction. Commun Biol, 2019, 2: 429. DOI:10.1038/s42003-019-0677-y
|
| [31] |
Bi JH, Chen SH, Zhao XH, et al. Computation-aided engineering of starch-debranching pullulanase from Bacillus thermoleovorans for enhanced thermostability. Appl Microbiol Biotechnol, 2020, 104(17): 7551-7562. DOI:10.1007/s00253-020-10764-z
|
| [32] |
Goldenzweig A, Goldsmith M, Hill SE, et al. Automated structure- and sequence-based design of proteins for high bacterial expression and stability. Mol Cell, 2016, 63(2): 337-346. DOI:10.1016/j.molcel.2016.06.012
|
| [33] |
Wijma HJ, Floor RJ, Jekel PA, et al. Computationally designed libraries for rapid enzyme stabilization. Protein Eng Des Sel, 2014, 27(2): 49-58. DOI:10.1093/protein/gzt061
|
| [34] |
Cui YL, Chen YC, Liu XY, et al. Computational redesign of a PETase for plastic biodegradation under ambient condition by the GRAPE strategy. ACS Catal, 2021, 11(3): 1340-1350. DOI:10.1021/acscatal.0c05126
|
| [35] |
Lindorff-Larsen K, Piana S, Palmo K, et al. Improved side-chain torsion potentials for the Amber ff99SB protein force field. Proteins, 2010, 78(8): 1950-1958. DOI:10.1002/prot.22711
|
| [36] |
Jorgensen WL, Chandrasekhar J, Madura JD, et al. Comparison of simple potential functions for simulating liquid water. J Chem Phys, 1983, 79(2): 926-935. DOI:10.1063/1.445869
|
| [37] |
Bussi G, Donadio D, Parrinello M. Canonical sampling through velocity rescaling. J Chem Phys, 2007, 126(1): 014101. DOI:10.1063/1.2408420
|
| [38] |
Berendsen HJC, Postma JPM, Van Gunsteren WF, et al. Molecular dynamics with coupling to an external bath. J Chem Phys, 1984, 81(8): 3684-3690. DOI:10.1063/1.448118
|
| [39] |
Luzar A, Chandler D. Effect of environment on hydrogen bond dynamics in liquid water. Phys Rev Lett, 1996, 76(6): 928-931. DOI:10.1103/PhysRevLett.76.928
|
| [40] |
Sun TT, Qiang SM, Lu C, et al. Composition- dependent energetic contribution of complex salt bridges to collagen stability. Biophys J, 2021, 120(16): 3429-3436. DOI:10.1016/j.bpj.2021.05.028
|
| [41] |
Barlow DJ, Thornton JM. Ion-pairs in proteins. J Mol Biol, 1983, 168(4): 867-885. DOI:10.1016/S0022-2836(83)80079-5
|
| [42] |
Li GL, Chen Y, Fang XR, et al. Identification of a hot-spot to enhance Candida rugosa lipase thermostability by rational design methods. RSC Adv, 2018, 8(4): 1948-1957. DOI:10.1039/C7RA11679A
|
| [43] |
Cui HY, Cao H, Cai HY, et al. Computer-assisted recombination (CompassR) teaches us how to recombine beneficial substitutions from directed evolution campaigns. Chemistry, 2020, 26(3): 643-649. DOI:10.1002/chem.201903994
|
| [44] |
Carugo O, Pongor S. A normalized root-mean-square distance for comparing protein three-dimensional structures. Protein Sci, 2001, 10(7): 1470-1473.
|
 2022, Vol. 38
2022, Vol. 38




