中国科学院微生物研究所、中国微生物学会主办
文章信息
- 柯双熬, 赵胜男, 刘玉, 卓情, 童祥文, 徐瑶
- KE Shuang՚ao, ZHAO Shengnan, LIU Yu, ZHUO Qing, TONG Xiangwen, XU Yao
- 环状RNA翻译蛋白在癌症中的研究进展
- Circular RNA-encoded peptides and proteins: implications to cancer
- 生物工程学报, 2022, 38(9): 3131-3140
- Chinese Journal of Biotechnology, 2022, 38(9): 3131-3140
- 10.13345/j.cjb.210909
-
文章历史
- Received: December 8, 2021
- Accepted: April 6, 2022
- Published: April 12, 2022
最新的全球癌症统计数据显示,癌症仍然是威胁人类生命健康的重要因素[1]。研究发现,环状RNA (circular RNA, circRNA) 参与了多种细胞的生理和病理过程,如细胞增殖、分化和细胞凋亡过程,并与多种癌症的发生和发展有关[2-3]。同时,由于circRNA没有线性RNA分子特有的自由端,circRNA分子更具稳定性,可作为分子靶标用于癌症的诊断和治疗。例如临床试验的液体活组织检查是基于circRNA在人体组织和液体(血清和尿液) 中的稳定存在而进行的[4]。circRNA也常被用于开发新的生物标志物来诊断和鉴别不同的病理状态,如糖尿病[5]、细胞衰老[6]、心血管疾病[7]和人类癌症等[8]。自从circRNA被发现以来,circRNA一直被认为是非编码RNA (non-coding RNA)[9]。近年来,随着生物技术的发展,越来越多的研究证据表明,circRNA在生物体内具有翻译的潜能,并且其翻译蛋白能够参与多种重要的生理调控过程[10-12]。因此,circRNA翻译蛋白与多种恶性肿瘤的相关研究引起了研究人员的高度重视。
1 环状RNA概述1976年Sanger在植物RNA病毒中发现了第一个circRNA分子[13]。直到1991年,人们通过电子显微镜在真核细胞中观察到circRNA结构[14]。此后,circRNA一直以来被认为是异常剪接的产物。随着测序技术和生物信息学的发展,circRNA被重新认识,并且在RNA研究领域受到新的关注。研究表明,它们并非偶然的副产品或剪接错误形成的,circRNA在生命体中发挥多种重要的功能,包括肿瘤发生过程[15]、神经发育过程[16]、自身免疫反应[17]等。与线性RNA不同,circRNA是单链共价闭合的转录物,没有5′帽子结构和3′ polyA尾巴。因此,circRNA较线性RNA更加稳定。通常用RNase R酶处理总RNA对circRNA进行富集[18]。根据circRNA的来源不同,circRNA可以分为4种不同的类型:外显子circRNA (ecircRNA)、内含子circRNA (ciRNA)、外显子-内含子circRNA (eiciRNA) 和其他[19]。研究发现了一种线粒体编码的circRNA (mitochondria-encoded circRNA, mecciRNA),这种新型的circRNA在人类和小鼠细胞内广泛存在,同时研究证实了mecciRNA可促进线粒体适应各种生理环境[20]。不同于线性RNA的标准剪切模式,circRNA是通过反向剪接(back-splicing) 方式剪切形成的。现有的circRNA形成模型主要有3种(图 1):即套索驱动、内含子配对和RNA结合蛋白(RNA-binding protein, RBP) 依赖性。套索驱动主要由两个不相邻的外显子连接形成circRNA;内含子配对主要是外显子与外显子间的内含子进行配对,最终结合环化形成circRNA;RNA结合蛋白(RNA binding protein, RBP) 依赖性是通过RBP结合到pre-mRNA上,此机制可以促进相邻外显子连接环化,形成circRNA[21]。最新研究还发现,在肿瘤细胞中还存在一些融合基因,该融合基因包含反向重复互补序列,在基因重排过程中相互接近导致反向剪接,最终产生融合的环状RNA (fusion-circRNA, f-circRNA),研究发现f-circRNA和融合基因具有类似的功能。例如,促进癌症发生和发展以及具有肿瘤耐药性[22]。该研究结果进一步说明了circRNA与癌症的发生和发展存在密切联系。
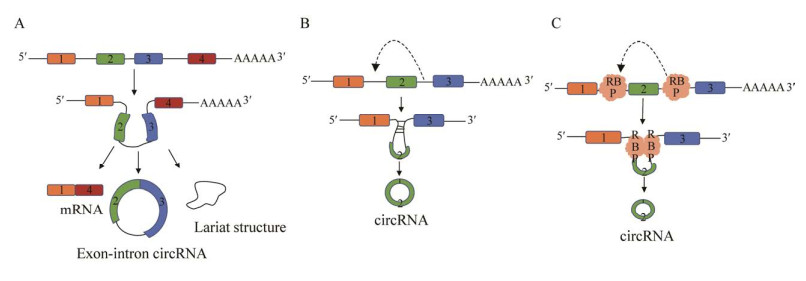
|
| 图 1 circRNA形成的3种主要方式 Fig. 1 circRNA formed by three major ways. A:套索驱动模型:由于外显子跳跃机制,可以发生反向剪接过程,导致套索形成。这个过程是一种非规范剪接途径,合成了3种不同的产物:circRNA、具有跳过外显子的mRNA和套索结构;B:内含子配对模型:内含子配对(通常是Alu重复) 可以诱导反向剪接(如图中虚线所示)。内含子共价结合在一起,合成一个circRNA;C:RNA结合蛋白依赖性模型:RNA结合蛋白(RNA-binding protein, RBP) 通常结合外显子侧翼的内含子,这些内含子将形成circRNA,RBP二聚化促进反向剪接过程(如图中虚线所示) (A) Lariat-driven circularization: due to the exon skipping mechanism, the back-splicing process takes place, which leads to lariat formation. This process is a non-canonical splicing pathway that synthesizes three distinct products: circRNAs, mRNAs with skipped exons, and lariat structures. (B) Intron-pair-driven circularization: introns pairing (usually Alu repeats) can induce back-splicing (as shown by the dotted line in the figure). The introns are covalently bound together to synthesize a circRNA. (C) RNA-binding proteins driven circularization: RNA-binding proteins (RBPs) usually bind introns flanking exons that will form circRNAs, and RBP dimerization promotes the back-splicing process (as shown by the dotted line in the figure). |
| |
长期以来,根据遗传密码的中心法则,人们认为只有mRNA可以通过起始、延伸和终止过程来进行蛋白的翻译[23]。此外,传统观点认为,5′和3′非翻译区(untranslated regions, UTR) 是真核细胞翻译的必要起始元件[24-25]。由于circRNA是环状闭合RNA,没有5′和3′非翻译区。因此,circRNA一直被认为是非编码RNA。随着研究的深入,越来越多的证据表明circRNA能够与核糖体结合,研究还发现circRNA含有AUG起始密码子序列和开放阅读框(open reading frames, ORF) 序列的基本编码翻译元件[10, 26-27]。这表明circRNA具备编码蛋白的潜力。
为了验证circRNA的翻译功能,研究通过核糖体印记证实了circRNA与核糖体相结合[12]。circRNA被发现含有特定序列可以作为核糖体介入位点(internal ribosome entry site, IRES) 元件来驱动circRNA的翻译起始[28]。此外,circRNA上还含有m6A修饰基序,研究还证实了m6A修饰作用是驱动circRNA翻译起始的一种方式[11-12]。IRES元件和m6A修饰驱动circRNA翻译起始的发现挑战了circRNA作为非编码RNA的传统观点,增加了基因调控的复杂性,并为circRNA翻译蛋白功能的研究提供了理论基础。
2.2 环状RNA的翻译方式由于circRNA是通过宿主基因反向剪接形成的,故其遗传密码与宿主基因具有相似之处。与传统RNA翻译相比,mRNA与circRNA最大的区别在于mRNA 5′端有m7G帽结构,3′端有polyA尾,而circRNA没有。另外,mRNA的5′端帽结构作为帽结合蛋白复合物(eukaryotic initiation factor 4F complex, eIF4F) 的锚定点,可以介导mRNA的40S亚基的募集[29-32]。因此,mRNA进行帽依赖性翻译,而circRNA无法进行帽依赖性翻译,不需要5′帽子结构和3′polyA尾。目前研究发现circRNA的翻译主要有IRES介导的和m6A修饰介导的2种翻译方式(图 2)。
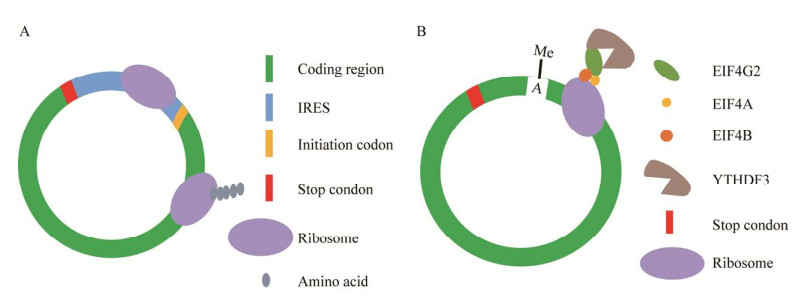
|
| 图 2 circRNA翻译的2种主要方式 Fig. 2 circRNA translated by two major ways A:通过核糖体进入位点(IRES) 起始circRNA的翻译;B:通过m6A修饰驱动核糖体结合位点起始circRNA的翻译 (A) Internal ribosome entry site (IRES) drives the initiation of circRNA translation. (B) m6A modification of ribosome binding site drives the initiation of circRNA translation. |
| |
随着研究的深入,研究发现在某些条件下,mRNA无法进行帽依赖性翻译[33-35]。由此发现一种替代机制,被称为帽非依赖性翻译,该机制通过IRES来启动mRNA翻译。通常,能够进行帽非依赖性翻译的基因参与响应细胞应激或病毒感染的生物学途径,这意味着IRES介导的帽非依赖性翻译在细胞生命活动过程中发挥着重要作用[36]。研究通过生物信息学分析发现在circRNA起始密码子上游也存在IRES序列。IRES序列能折叠成类似tRNA的结构来招募核糖体进行circRNA的翻译[28]。总而言之,IRES介导的circRNA翻译扩展了细胞生命活动的多样性,具有较大的研究价值。
2.2.2 m6A介导的环状RNA翻译N6-甲基腺嘌呤(N6-methyladenosine, m6A) 是RNA甲基化最常见的一种化学修饰[37]。m6A修饰主要发生在碱基序列RRACH中的腺嘌呤上(R=G,A;H=A,C或U),其功能由写入器(writers)、读取器(readers) 和擦除器(erasers) 来调控[38]。m6A-writers复合物被称为甲基转移酶,包括甲基转移酶样蛋白3 (METTL3)[39]、甲基转移酶样蛋白14 (METTL14)[40]、肾母细胞瘤1关联蛋白(wilms tumour 1-associated protein, WTAP)[41]和KIAA1429[42]等;m6A-erasers复合物作为去甲基化酶,包括脂肪量和肥胖相关蛋白(FTO)[43-44]和AlkB同源物5 (ALKBH5)[45]等;其他可以识别m6A修饰的结合蛋白,被称为m6A-readers蛋白,包括YTH结构域蛋白YTHDF1[44]、YTHDF2[46]、YTHDF3[47]、YTHDC1和YTHDC2[48]、RNA结合蛋白异质核蛋白A2B1 (HNRNPA2B1) 和异质核核糖核蛋白C (HNRNPC)[49]。
Yang等发现,一些circRNA序列上含有m6A基序,他们通过在m6A基序处构建突变体,成功证实了m6A修饰可以驱动circRNA的翻译[12]。研究还发现,m6A基序经甲基转移酶修饰后可以被YTHDF3蛋白识别,随后与翻译起始因子eIF4G2相互作用促进circRNA的翻译。总之,上述研究表明m6A修饰结合蛋白可以与转录起始因子相互作用来驱动circRNA的翻译。
3 环状RNA的翻译在癌症中的研究进展随着生物测序技术的发展,越来越多的circRNA被发现,某些circRNA还在癌症中特异性表达。这一现象表明circRNA与某些癌症疾病存在某种联系。近些年,circRNA翻译机制的研究进入了人们的视野,研究者发现circRNA翻译蛋白参与并调控多种癌症疾病,并且在癌症疾病的发生和发展过程中发挥着重要功能。这些理论研究的发现将有可能为癌症的疾病治疗提供强有力的支撑。本团队也在进行circRNA翻译的研究,研究首先需要通过在circRNADb和riboCIRC等生物信息学数据库中对circRNA翻译的ORF序列进行预测。然后根据ORF序列在终止密码子前插入Flag标签构建过表达载体。通过构建circRNA过表达载体,包括不含Flag、插入Flag和插入Flag但突变启动子。最后结合免疫印迹实验,验证circRNA翻译蛋白的存在。理论上,如果此circRNA能够翻译,免疫印迹实验可以检测到插入Flag的蛋白,而其他两个过表达载体翻译蛋白无法检测到。很遗憾的是,本课题组根据上述做法,对circCDYL、circMED13L翻译功能进行验证,并未检测出相关蛋白。该结果提醒我们数据库预测只是提供一种理论可能,还需要设计实验进行确认。而且,根据最近文献参考显示,circRNA翻译功能的验证还存在其他方法。因此,circRNA翻译功能的验证还是需要更多实验去验证。本文接下来将介绍circRNA翻译蛋白在近几年的研究进展,重点阐述circRNA翻译蛋白的多种验证方法及其在各种癌症中的功能(表 1)。
| circRNA | Cancer | Role in cancer | Regulations | References |
| circFBXW7 | Glioma tumorigenesis | Anti-oncogene | Down-regulation | [50] |
| circPINT | Anti-oncogene | Down-regulation | [28] | |
| circAKT3 | Anti-oncogene | Down-regulation | [51] | |
| circSHPRH | Anti-oncogene | Down-regulation | [26] | |
| circ-E-Cad | Oncogene | Up-regulation | [52] | |
| circSMO | Oncogene | Up-regulation | [53] | |
| circEGFR | Oncogene | Up-regulation | [54] | |
| circPPP1R12A | Colon cancer | Oncogene | Up-regulation | [27] |
| circFNDC3B | Anti-oncogene | Down-regulation | [55] | |
| circMAPK1 | Gastric cancer | Anti-oncogene | Down-regulation | [56] |
| circDIDO1 | Anti-oncogene | Down-regulation | [57] | |
| circE7 | Cervical cancer | Oncogene | Up-regulation | [58] |
| circHER2 | Breast cancer | Oncogene | Up-regulation | [59] |
| circBUB1B | Multiple myeloma | Oncogene | Up-regulation | [60] |
胶质瘤(glioma tumorigenesis) 是脑中常见且最恶性的原发性肿瘤,预后差和治疗难是胶质瘤致死率高的主要原因。2018年,中山大学张弩教授首次证实了circRNA翻译蛋白调控脑胶质瘤的发生、发展[50]。circFBXW7通过翻译21 kD蛋白,调控原癌基因c-Myc蛋白的稳定性,抑制恶性胶质瘤的细胞周期。同时,该团队还发现了circPINT可以翻译为87个氨基酸的多肽,该多肽可以作为转录因子与PAF1复合物相互作用,影响mRNA的转录和延伸,进一步研究显示,circPINT翻译的多肽可以作为胶质瘤的抑制因子,在胶质瘤疾病的临床研究中具有潜在的应用价值[28]。此外,还发现了circAKT3翻译蛋白可以在体内外抑制胶质瘤细胞的增殖、凋亡和肿瘤形成,证实circAKT3翻译蛋白是胶质瘤患者潜在的预后标志物,并有望应用于胶质瘤的临床诊断和预后评估[51]。Zhang等研究报道,与正常人相比,circSHPRH翻译蛋白在胶质瘤患者中表达下调,并证明了circSHPRH翻译蛋白与胶质瘤恶性程度的相关性[26]。此外,该团队发现抑癌基因E-Cadherin可以形成circRNA (circ-E-Cad),并且其翻译蛋白具有促癌作用[52]。最新研究报道,circSMO翻译蛋白可以通过增强Hedgehog信号,进而影响胶质瘤细胞的自我更新和体内致瘤性[53]。此外,circEGFR被预测并验证存在无限循环的ORF (infinite open reading frame, iORF),该特殊翻译产物具有促进脑瘤启动细胞(brain tumor initiating cell, BTIC) 成瘤的能力。研究发现,靶向EGFR的单抗药物Nimotuzumab对胶质瘤无显著疗效,但若同时干扰circEGFR,可以促进Nimotuzumab的药物敏感性,该研究结果预示circEGFR及其翻译产物有望作为胶质瘤治疗的新靶点[54]。
3.2 结肠癌结肠癌(colon cancer) 是全球范围内第四大危害人类生命的疾病。尽管结肠癌的治疗已经有了很大的改进,但超过一半的结肠癌患者最终因为无法治疗而死亡。最新研究发现,一种与结肠癌相关的circRNA (circPPP1R12A) 可以翻译出73个氨基酸多肽,该多肽和circPPP1R12A都在结肠癌中存在潜在作用,研究证实circPPP1R12A翻译蛋白在结肠癌细胞的增殖、迁移和侵袭中起关键作用,而并非circPPP1R12A本身。进一步研究发现,circPPP1R12A翻译蛋白通过激活Hippo-YAP信号通路来促进结肠癌的生长和转移。YAP特异性抑制剂多肽显著降低了circPPP1R12A翻译蛋白对结肠癌细胞的促癌作用,研究结果为结肠癌的治疗提供了新方法[27]。此外,研究还证明了circFNDC3B翻译蛋白可以抑制结肠癌的增殖、侵袭和迁移,研究发现circFNDC3B翻译蛋白通过抑制Snail基因的表达,从而增强了FBP1基因在结肠癌中的肿瘤抑制功能[55]。总而言之,以上2种机制的发现都为结肠癌的靶点治疗和临床研究提供了新方向。
3.3 胃癌胃癌(gastric cancer) 是全球最常见的恶性肿瘤之一。在过去的几十年里,尽管已经做出了巨大的努力来改善胃癌的诊断和治疗,但是胃癌的发病率和死亡率仍然很高。最近研究发现一种在胃癌中低表达的circMAPK1,该circRNA翻译蛋白通过与MAPK1竞争结合上游激酶MEK1,从而抑制了MAPK1的表达及其下游基因的磷酸化,进而影响癌症的发生,研究结果表明circMAPK1可以作为胃癌的治疗靶点[56]。此外,研究发现从人类基因DIDO1转录而来的circDIDO1翻译蛋白在胃癌的发生和发展中起重要作用,该翻译蛋白可以与PARP1相互作用并抑制其活性。同时,circDIDO1与PRDX2特异性结合可以促进RBX1介导的泛素化和PRDX2的降解,导致其下游信号通路失活。研究表明,在胃癌中具有肿瘤抑制功能的circDIDO1有望成为潜在的预后生物标志物和治疗靶点[57]。
3.4 宫颈癌宫颈癌(cervical cancer) 是全球女性第二大常见癌症,感染某些高危型人乳头瘤病毒(human papilloma virus, HPV) 是宫颈癌的最大风险因素。最近研究发现,HPV能产生多种circRNA,其中包括circE7。该circRNA通过m6A修饰作用驱动翻译产生E7癌蛋白。研究发现,在体外和肿瘤异种移植中,circE7通过降低宫颈癌细胞E7癌蛋白水平来抑制癌细胞生长。结果显示病毒衍生的circRNA编码蛋白质具有生物学功能并与某些HPV的转化特性相关。此外,高危HPV检测在宫颈癌筛查中的应用已经建立,circE7翻译蛋白能否作为高危HPV人群的敏感标记,以及circE7丰度能否在宫颈癌中具有预后意义,这些仍然需要进一步科学探究[58]。
3.5 乳腺癌乳腺癌(breast cancer) 是女性最常见的癌症。最新研究发现,circHER2及其翻译蛋白均在临床样本中高表达,敲除circHER2可以抑制乳腺癌的增殖、侵袭和体内外肿瘤发生,该结果揭示了circHER2及其翻译蛋白在乳腺癌中的致瘤性。进一步研究揭示了circHER2翻译蛋白可以促进表皮生长因子受体(epidermal growth factor receptor, EGFR)/HER3的二聚化和蛋白激酶B (AKT) 的磷酸化。此外,circHER2翻译蛋白与HER2CR1具有较高的同源性,该结构域可以被帕妥珠单抗(一种临床使用的HER2抗体) 拮抗,研究表明帕妥珠单抗显著减弱了circHER2翻译蛋白在乳腺癌细胞中的致瘤性。另外,在阴性乳腺癌细胞中,帕妥珠单抗对circHER2和circHER2翻译蛋白没有影响,该机制的发现表明circHER2及其翻译蛋白的患者可受益于帕妥珠单抗,这极大地促进了乳腺癌的临床和病例研究及其治疗方法[59]。
3.6 多发性骨髓瘤多发性骨髓瘤(multiple myeloma) 是一种起源于骨髓的分子和细胞遗传学异质性血液恶性肿瘤,其特征是染色体不稳定。尽管临床上使用靶向药物进行治疗,如免疫调节剂和蛋白酶体抑制剂,来改善多发性骨髓瘤患者的预后,但多发性骨髓瘤仍然威胁生命且无法治愈。研究发现,与正常人相比,多发性骨髓瘤患者中BUB1B基因的表达量是显著增加的,进一步研究显示BUB1B促进了骨髓瘤细胞增殖,并在体外和体内诱导了肿瘤的耐药性,而靶向基因BUB1B消除了这种影响。该机制研究揭示了BUB1B基因引起了染色体的不稳定。此外,研究还发现在外周血中,circBUB1B翻译的544个氨基酸蛋白的升高与多发性骨髓瘤密切相关,并与基因BUB1B在诱发染色体不稳定方面起协同作用,进一步研究发现多发性骨髓瘤细胞可以分泌circBUB1B翻译蛋白,该蛋白与BUB1B全长蛋白以相同的方式干扰多发性骨髓瘤的肿瘤微环境。在体外和体内研究表明,BUB1B siRNA靶向BUB1B和circBUB1B可以显著抑制多发性骨髓瘤的恶性程度。总之,该研究结果预示了BUB1B和circBUB1B翻译蛋白将有希望成为多发性骨髓瘤的预后和治疗靶点[60]。
4 总结与展望随着高通量测序技术和生物信息学的广泛应用,越来越多的circRNA被鉴定和研究。f-circRNA的发现充分证实了circRNA与癌症的发生、发展存在密切联系。与此同时,circRNA的翻译机制为揭示circRNA如何调控癌症的生理和病理过程提供了新的研究方向,已成为人类癌症研究的热点。多项circRNA翻译机制的研究表明,参与癌症相关基因调控的是circRNA翻译蛋白而非circRNA本身,充分证明circRNA翻译蛋白在癌症研究中的重要性。前期研究发现,融合蛋白的产生往往是肿瘤发生的早期事件,因此,来源于f-circRNA的融合蛋白将有望作为生物标志物或药物靶点应用于癌症诊断和治疗。另外,筛选和验证具有编码潜力的功能性circRNA具有一定的技术挑战。第一,生物信息学预测具有编码潜力的circRNA不一定翻译蛋白质,需要分子生物学实验进一步验证;第二,circRNA翻译蛋白在正常组织和癌症中的表达具有显著差异,使其易于检测和应用;第三,circRNA序列应包含一个完整的ORF;第四,circRNA翻译蛋白与其亲本基因共同调节癌症相关的信号通路。目前,大多数circRNA及其翻译蛋白仅在分子水平上被检测,尚未应用于癌症的临床诊断和治疗。相信随着新技术的应用和研究者们的不断探索,circRNA翻译蛋白将会为癌症的攻克提供新的理论支撑和指导价值。
circRNA翻译蛋白除了参与癌症的发生、发展之外,还有研究发现circRNA翻译蛋白可以控制成肌细胞的增殖和分化功能[10, 61]。同时,circRNA翻译蛋白在多种细胞信号通路中也发挥重要作用[62]。这些研究表明,circRNA翻译机制还在机体其他方面发挥作用,具有较大的研究潜力。
总之,发掘circRNA翻译蛋白在癌症生物学中的不同分子机制将有希望为癌症的临床治疗提供新的理论支撑。当然不可否认的是,circRNA翻译机制的研究道路还很长,还有许多新的理论需要被揭示和阐明,任重而道远。这就需要一代一代科学家去攻克一个又一个难题,早日为癌症的研究和临床治疗开启新的篇章。
| [1] |
Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492
|
| [2] |
Du WW, Yang WN, Liu E, et al. Foxo3 circular RNA retards cell cycle progression via forming ternary complexes with p21 and CDK2. Nucleic Acids Res, 2016, 44(6): 2846-2858. DOI:10.1093/nar/gkw027
|
| [3] |
Shen YY, Zhang MM, Da LS, et al. Circular RNA circ_SETD2 represses breast cancer progression via modulating the miR-155-5p/SCUBE2 axis. Open Med (Wars), 2020, 15(1): 940-953. DOI:10.1515/med-2020-0223
|
| [4] |
Esteller M. Non-coding RNAs in human disease. Nat Rev Genet, 2011, 12(12): 861-874. DOI:10.1038/nrg3074
|
| [5] |
Stoll L, Sobel J, Rodriguez-Trejo A, et al. Circular RNAs as novel regulators of β-cell functions in normal and disease conditions. Mol Metab, 2018, 9: 69-83. DOI:10.1016/j.molmet.2018.01.010
|
| [6] |
Wang YH, Yu XH, Luo SS, et al. Comprehensive circular RNA profiling reveals that circular RNA100783 is involved in chronic CD28-associated CD8(+)T cell ageing. Immun Ageing, 2015, 12: 17. DOI:10.1186/s12979-015-0042-z
|
| [7] |
Zhou LY, Zhai M, Huang Y, et al. The circular RNA ACR attenuates myocardial ischemia/reperfusion injury by suppressing autophagy via modulation of the Pink1/FAM65B pathway. Cell Death Differ, 2019, 26(7): 1299-1315. DOI:10.1038/s41418-018-0206-4
|
| [8] |
Braicu C, Zimta AA, Gulei DA, et al. Comprehensive analysis of circular RNAs in pathological states: biogenesis, cellular regulation, and therapeutic relevance. Cell Mol Life Sci, 2019, 76(8): 1559-1577. DOI:10.1007/s00018-019-03016-5
|
| [9] |
Patop IL, Wüst S, Kadener S. Past, present, and future of circRNAs. EMBO J, 2019, 38(16): e100836.
|
| [10] |
Legnini I, Di Timoteo G, Rossi F, et al. Circ-ZNF609 is a circular RNA that can be translated and functions in myogenesis. Mol Cell, 2017, 66(1): 22-37. e9. DOI:10.1016/j.molcel.2017.02.017
|
| [11] |
Pamudurti NR, Bartok O, Jens M, et al. Translation of CircRNAs. Mol Cell, 2017, 66(1): 9-21. e7. DOI:10.1016/j.molcel.2017.02.021
|
| [12] |
Yang Y, Fan XJ, Mao MW, et al. Extensive translation of circular RNAs driven by N6-methyladenosine. Cell Res, 2017, 27(5): 626-641. DOI:10.1038/cr.2017.31
|
| [13] |
Kolakofsky D. Isolation and characterization of Sendai virus DI-RNAs. Cell, 1976, 8(4): 547-555. DOI:10.1016/0092-8674(76)90223-3
|
| [14] |
Sanger HL, Klotz G, Riesner D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures. PNAS, 1976, 73(11): 3852-3856. DOI:10.1073/pnas.73.11.3852
|
| [15] |
Dong W, Bi JM, Liu HW, et al. Circular RNA ACVR2A suppresses bladder cancer cells proliferation and metastasis through miR-626/EYA4 axis. Mol Cancer, 2019, 18(1): 95. DOI:10.1186/s12943-019-1025-z
|
| [16] |
Dube U, Del-Aguila JL, Li ZR, et al. An atlas of cortical circular RNA expression in Alzheimer disease brains demonstrates clinical and pathological associations. Nat Neurosci, 2019, 22(11): 1903-1912. DOI:10.1038/s41593-019-0501-5
|
| [17] |
Cardamone G, Paraboschi EM, Rimoldi V, et al. The characterization of GSDMB splicing and backsplicing profiles identifies novel isoforms and a circular RNA that are dysregulated in multiple sclerosis. Int J Mol Sci, 2017, 18(3): 576. DOI:10.3390/ijms18030576
|
| [18] |
Jeck WR, Sharpless NE. Detecting and characterizing circular RNAs. Nat Biotechnol, 2014, 32(5): 453-461. DOI:10.1038/nbt.2890
|
| [19] |
Li ZY, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus. Nat Struct Mol Biol, 2015, 22(3): 256-264. DOI:10.1038/nsmb.2959
|
| [20] |
Liu X, Wang XL, Li JX, et al. Identification of mecciRNAs and their roles in the mitochondrial entry of proteins. Sci China Life Sci, 2020, 63(10): 1429-1449. DOI:10.1007/s11427-020-1631-9
|
| [21] |
Kristensen LS, Andersen MS, Stagsted LVW, et al. The biogenesis, biology and characterization of circular RNAs. Nat Rev Genet, 2019, 20(11): 675-691. DOI:10.1038/s41576-019-0158-7
|
| [22] |
Guarnerio J, Bezzi M, Jeong JC, et al. Oncogenic role of fusion-circRNAs derived from cancer-associated chromosomal translocations. Cell, 2016, 165(2): 289-302. DOI:10.1016/j.cell.2016.03.020
|
| [23] |
Gross JD, Moerke NJ, von der Haar T, et al. Ribosome loading onto the mRNA cap is driven by conformational coupling between eIF4G and eIF4E. Cell, 2003, 115(6): 739-750. DOI:10.1016/S0092-8674(03)00975-9
|
| [24] |
Lasda E, Parker R. Circular RNAs: diversity of form and function. RNA, 2014, 20(12): 1829-1842. DOI:10.1261/rna.047126.114
|
| [25] |
Yang Y, Wang ZF. IRES-mediated cap-independent translation, a path leading to hidden proteome. J Mol Cell Biol, 2019, 11(10): 911-919. DOI:10.1093/jmcb/mjz091
|
| [26] |
Zhang ML, Huang NN, Yang XS, et al. A novel protein encoded by the circular form of the SHPRH gene suppresses glioma tumorigenesis. Oncogene, 2018, 37(13): 1805-1814. DOI:10.1038/s41388-017-0019-9
|
| [27] |
Zheng X, Chen LJ, Zhou Y, et al. A novel protein encoded by a circular RNA circPPP1R12A promotes tumor pathogenesis and metastasis of colon cancer via Hippo-YAP signaling. Mol Cancer, 2019, 18(1): 47. DOI:10.1186/s12943-019-1010-6
|
| [28] |
Zhang ML, Zhao K, Xu XP, et al. A peptide encoded by circular form of LINC-PINT suppresses oncogenic transcriptional elongation in glioblastoma. Nat Commun, 2018, 9(1): 4475. DOI:10.1038/s41467-018-06862-2
|
| [29] |
Montero H, Pérez-Gil G, Sampieri CL. Eukaryotic initiation factor 4A (eIF4A) during viral infections. Virus Genes, 2019, 55(3): 267-273. DOI:10.1007/s11262-019-01641-7
|
| [30] |
Pelletier J, Sonenberg N. Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA. Nature, 1988, 334(6180): 320-325. DOI:10.1038/334320a0
|
| [31] |
Godet AC, David F, Hantelys F, et al. IRES trans-acting factors, key actors of the stress response. Int J Mol Sci, 2019, 20(4): 924. DOI:10.3390/ijms20040924
|
| [32] |
Pelletier J, Sonenberg N. The organizing principles of eukaryotic ribosome recruitment. Annu Rev Biochem, 2019, 88: 307-335. DOI:10.1146/annurev-biochem-013118-111042
|
| [33] |
Kneller EL, Rakotondrafara AM, Miller WA. Cap-independent translation of plant viral RNAs. Virus Res, 2006, 119(1): 63-75. DOI:10.1016/j.virusres.2005.10.010
|
| [34] |
Spriggs KA, Bushell M, Willis AE. Translational regulation of gene expression during conditions of cell stress. Mol Cell, 2010, 40(2): 228-237. DOI:10.1016/j.molcel.2010.09.028
|
| [35] |
Stern-Ginossar N, Thompson SR, Mathews MB, et al. Translational control in virus-infected cells. Cold Spring Harb Perspect Biol, 2019, 11(3): a033001. DOI:10.1101/cshperspect.a033001
|
| [36] |
Andreev DE, O'Connor PBF, Fahey C, et al. Translation of 5' leaders is pervasive in genes resistant to eIF2 repression. eLife, 2015, 4: e03971. DOI:10.7554/eLife.03971
|
| [37] |
Lan Q, Liu PY, Haase J, et al. The critical role of RNA m6A methylation in cancer. Cancer Res, 2019, 79(7): 1285-1292. DOI:10.1158/0008-5472.CAN-18-2965
|
| [38] |
Meyer KD, Jaffrey SR. Rethinking m6A readers, writers, and erasers. Annu Rev Cell Dev Biol, 2017, 33: 319-342. DOI:10.1146/annurev-cellbio-100616-060758
|
| [39] |
Lin SB, Choe J, Du P, et al. The m(6)A methyltransferase METTL3 promotes translation in human cancer cells. Mol Cell, 2016, 62(3): 335-345. DOI:10.1016/j.molcel.2016.03.021
|
| [40] |
Liu JZ, Yue YN, Han DL, et al. A METTL3-METTL14 complex mediates mammalian nuclear RNA N6-adenosine methylation. Nat Chem Biol, 2014, 10(2): 93-95. DOI:10.1038/nchembio.1432
|
| [41] |
Ping XL, Sun BF, Wang L, et al. Mammalian WTAP is a regulatory subunit of the RNA N6-methyladenosine methyltransferase. Cell Res, 2014, 24(2): 177-189. DOI:10.1038/cr.2014.3
|
| [42] |
Schwartz S, Mumbach MR, Jovanovic M, et al. Perturbation of m6A writers reveals two distinct classes of mRNA methylation at internal and 5' sites. Cell Rep, 2014, 8(1): 284-296. DOI:10.1016/j.celrep.2014.05.048
|
| [43] |
Mauer J, Sindelar M, Despic V, et al. FTO controls reversible m 6Am RNA methylation during snRNA biogenesis. Nat Chem Biol, 2019, 15(4): 340-347. DOI:10.1038/s41589-019-0231-8
|
| [44] |
Shi HL, Zhang XL, Weng YL, et al. M6A facilitates Hippocampus-dependent learning and memory through YTHDF1. Nature, 2018, 563(7730): 249-253. DOI:10.1038/s41586-018-0666-1
|
| [45] |
Zheng GQ, Dahl JA, Niu YM, et al. ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility. Mol Cell, 2013, 49(1): 18-29. DOI:10.1016/j.molcel.2012.10.015
|
| [46] |
Li JF, Meng S, Xu MJ, et al. Downregulation of N6-methyladenosine binding YTHDF2 protein mediated by miR-493-3p suppresses prostate cancer by elevating N6-methyladenosine levels. Oncotarget, 2017, 9(3): 3752-3764.
|
| [47] |
Shi HL, Wei JB, He C. Where, when, and how: context-dependent functions of RNA methylation writers, readers, and erasers. Mol Cell, 2019, 74(4): 640-650. DOI:10.1016/j.molcel.2019.04.025
|
| [48] |
Haussmann IU, Bodi Z, Sanchez-Moran E, et al. M6A potentiates Sxl alternative pre-mRNA splicing for robust Drosophila sex determination. Nature, 2016, 540(7632): 301-304. DOI:10.1038/nature20577
|
| [49] |
Alarcón CR, Goodarzi H, Lee H, et al. HNRNPA2B1 is a mediator of m(6)A-dependent nuclear RNA processing events. Cell, 2015, 162(6): 1299-1308. DOI:10.1016/j.cell.2015.08.011
|
| [50] |
Yang YB, Gao XY, Zhang ML, et al. Novel role of FBXW7 circular RNA in repressing glioma tumorigenesis. J Natl Cancer Inst, 2018, 110(3): 304-315. DOI:10.1093/jnci/djx166
|
| [51] |
Xia X, Li XX, Li FY, et al. A novel tumor suppressor protein encoded by circular AKT3 RNA inhibits glioblastoma tumorigenicity by competing with active phosphoinositide-dependent Kinase-1. Mol Cancer, 2019, 18(1): 131. DOI:10.1186/s12943-019-1056-5
|
| [52] |
Gao XY, Xia X, Li FY, et al. Circular RNA-encoded oncogenic E-cadherin variant promotes glioblastoma tumorigenicity through activation of EGFR-STAT3 signalling. Nat Cell Biol, 2021, 23(3): 278-291. DOI:10.1038/s41556-021-00639-4
|
| [53] |
Wu XJ, Xiao SH, Zhang ML, et al. A novel protein encoded by circular SMO RNA is essential for Hedgehog signaling activation and glioblastoma tumorigenicity. Genome Biol, 2021, 22(1): 33. DOI:10.1186/s13059-020-02250-6
|
| [54] |
Liu Y, Li ZJ, Zhang ML, et al. Rolling-translated EGFR variants sustain EGFR signaling and promote glioblastoma tumorigenicity. Neuro-oncology, 2021, 23(5): 743-756. DOI:10.1093/neuonc/noaa279
|
| [55] |
Pan ZH, Cai JY, Lin JT, et al. A novel protein encoded by circFNDC3B inhibits tumor progression and EMT through regulating Snail in colon cancer. Mol Cancer, 2020, 19(1): 71. DOI:10.1186/s12943-020-01179-5
|
| [56] |
Jiang TL, Xia YW, Lv JL, et al. A novel protein encoded by circMAPK1 inhibits progression of gastric cancer by suppressing activation of MAPK signaling. Mol Cancer, 2021, 20(1): 66. DOI:10.1186/s12943-021-01358-y
|
| [57] |
Zhang Y, Jiang JJ, Zhang JY, et al. CircDIDO1 inhibits gastric cancer progression by encoding a novel DIDO1-529 aa protein and regulating PRDX2 protein stability. Mol Cancer, 2021, 20(1): 101. DOI:10.1186/s12943-021-01390-y
|
| [58] |
Zhao JW, Lee EE, Kim J, et al. Transforming activity of an oncoprotein-encoding circular RNA from human papillomavirus. Nat Commun, 2019, 10(1): 2300. DOI:10.1038/s41467-019-10246-5
|
| [59] |
Li J, Ma MG, Yang XS, et al. Circular HER2 RNA positive triple negative breast cancer is sensitive to Pertuzumab. Mol Cancer, 2020, 19(1): 142. DOI:10.1186/s12943-020-01259-6
|
| [60] |
Tang XZ, Guo MJ, Ding PG, et al. BUB1B and circBUB1B544aa aggravate multiple myeloma malignancy through evoking chromosomal instability. Signal Transduct Target Ther, 2021, 6(1): 361. DOI:10.1038/s41392-021-00746-6
|
| [61] |
Di Timoteo G, Dattilo D, Centrón-Broco A, et al. Modulation of circRNA metabolism by m6A modification. Cell Rep, 2020, 31(6): 107641. DOI:10.1016/j.celrep.2020.107641
|
| [62] |
Liang WC, Wong CW, Liang PP, et al. Translation of the circular RNA circβ-catenin promotes liver cancer cell growth through activation of the Wnt pathway. Genome Biol, 2019, 20(1): 84. DOI:10.1186/s13059-019-1685-4
|
 2022, Vol. 38
2022, Vol. 38




