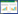中国科学院微生物研究所、中国微生物学会主办
文章信息
- 隋馨莹, 徐平, 段昌柱, 李衍常
- SUI Xinying, XU Ping, DUAN Changzhu, LI Yanchang
- p62蛋白的分子功能及其在疾病中的研究进展
- Advances in molecular function of p62 protein and its role in diseases
- 生物工程学报, 2023, 39(4): 1374-1389
- Chinese Journal of Biotechnology, 2023, 39(4): 1374-1389
- 10.13345/j.cjb.220681
-
文章历史
- Received: August 26, 2022
- Accepted: January 11, 2023
- Published: January 16, 2023
2. 军事科学院军事医学研究院 生命组学研究所国家蛋白质科学中心(北京) 北京蛋白质组研究中心 蛋白质组学国家重点实验室, 北京 102206;
3. 安徽医科大学基础医学院, 安徽 合肥 230032;
4. 中国医学科学院蛋白组学与新药研发创新单元, 北京 102206
2. State Key Laboratory of Proteomics, Beijing Proteome Research Center, National Center for Protein Sciences (Beijing), Beijing Institute of Lifeomics, Academy of Military Medical Sciences of Academy of Military Science, Beijing 102206, China;
3. School of Basic Medicine, Anhui Medical University, Hefei 230032, Anhui, China;
4. Research Unit of Proteomics & Research and Development of New Drug, Chinese Academy of Medical Sciences, Beijing 102206, China
螯合体1 (sequestosome 1, SQSTM1),又名p62,作为接头蛋白(adaptor proteins)含有多个功能结构域,可以与细胞内多种分子形成蛋白复合物进而调控不同的信号通路,包括自噬、氧化应激、细胞生长等过程[1]。其中,p62的泛素结合结构域(ubiquitin-associated domain, UBA)识别并调控泛素化修饰蛋白质参与泛素-蛋白酶体系统(ubiquitin-proteasome system, UPS)和自噬-溶酶体系统两种蛋白质降解过程。p62自身不仅是自噬体与底物之间的适配蛋白,同时也是自噬选择性降解底物,在蛋白质质量控制和细胞内稳态调控过程中发挥重要作用,其基因突变、表达含量以及翻译后修饰状态的异常与癌症、神经退行性疾病的发生发展密切相关。阿尔茨海默病(Alzheimer's disease, AD)、肌萎缩性侧索硬化症(amyotrophic lateral sclerosis, ALS)和许多慢性神经退行性疾病的发生发展与p62高度相关[2]。随着研究的深入,越来越多的p62调控的下游底物群被发现和鉴定,极大丰富了p62的生物学功能。本文系统综述了p62的分子特征与功能、介导的生物学过程以及与相关疾病的调控关系,为全面了解p62的研究进展以及将来的研究工作提供参考。
1 p62基因及蛋白的结构特征p62是在哺乳动物中发现的第一个选择性自噬衔接子[3]。最初,研究者发现p62具有形成聚集体的能力,在胞浆中以散在点状或者聚集形式存在,Shin将其命名为Sequestosome 1 (SQSTM 1)[4]。人类的p62基因位于5号染色体上,由8个外显子组成。它具有3种剪切体,其中两种5′UTR不同,但编码相同的亚型(图 1)。p62编码440个氨基酸,分子质量为47 kDa。ZIP (zeta protein kinase C-interacting protein, ZIP)和A170分别是其在大鼠和小鼠中变体的名称。氨基酸序列比对结果表明这些同源基因表达蛋白质具有高度保守性,氨基酸序列相似性高达90%[5]。
|
| 图 1 p62的3种剪切变体 Fig. 1 Three splicing variants of p62. |
| |
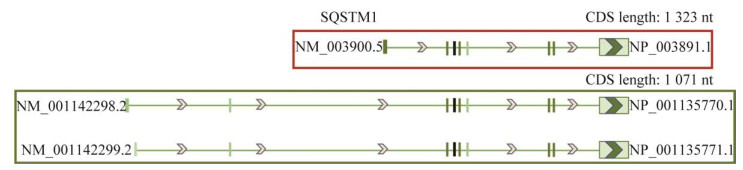
p62蛋白虽然只有47 kDa,但是包含9个功能域(图 2):phox和bem1结构域(phox and bem1, PB1)、ZZ型锌指结构域(zinc finger domain, ZZ)、TRAF6结合结构域(TRAF6 binding domain, TB)、核定位信号(nuclear localization sequence, NLS)、核输出信号(nuclear export signal, NES)、含脯氨酸-谷氨酸-丝氨酸-苏氨酸(P-E-S-T)结构域、LC3相互作用结构域(LC3-interacting region, LIR)、KEAP1相互作用结构域(Kelch like ECH associated protein 1-interacting region, KIR)和C端泛素结合结构域(UBA)结构域。丰富的功能结构域赋予了p62功能的多样性,例如p62能够通过PB1结构域实现自身寡聚化,是p62促进相分离形成所必需的[1]。另外,PB1结构域可与其他含PB1结构域的蛋白质相互作用,例如细胞外调节蛋白激酶(extracellular regulated protein kinases, ERK)、(mitogen-activated protein kinase/extracellular signal-regulated kinase kinase3, MEKK3)以及蛋白酶体亚基,进而参与互作蛋白质的降解过程[6]。ZZ结构域与受体相互作用蛋白1 (receptor-interacting protein kinase 1, RIP1)相互作用,调节核转录因子κappa B (nuclear transcription factor-κB, NF-κB)信号传导通路[7]。TB结构域可与肿瘤坏死因子受体相关因子6 (tumor necrosis factor-associated factor 6, TRAF6)相结合,促进TRAF6的寡聚并通过其K63多泛素化以激活NF-κB通路[2]。NLS域和NES域分别是核定位信号和核输出信号,使p62具有核质穿梭功能,其中NLS2的磷酸化在促进p62由细胞质运输到细胞核中起重要作用[8]。PEST结构域包含脯氨酸(P)、谷氨酸(E)、丝氨酸(S)和苏氨酸(T),所以被称为PEST序列。在真核细胞中,PEST序列被认为是直接与26S蛋白酶体相互作用的靶标,PEST的磷酸化与蛋白质快速更新有关[9]。LIR结构域包括氨基酸集群和疏水残基,促进p62与自噬体标志物LC3的结合,介导p62及其互作蛋白的自噬性降解。KIR结构域可介导p62与Kelch样环氧氯丙烷相关蛋白-1 (Kelch-like ECH-associated protein 1, KEAP1)的螯合,促进KEAP1降解并挽救核因子E2相关因子2 (NF-E2-related factor 2, NRF2),促进抗氧化应激(anti-oxidative stress)调控通路应答[1]。C端泛素结合结构域(UBA)结构域是对泛素具有高亲和力的高度保守序列[10-11],对于转运待降解的泛素化底物到自噬体中至关重要。
|
| 图 2 p62的分子功能结构域 Fig. 2 Molecular functional domains of p62. 包含9个功能域:phox和bem1结构域、ZZ型锌指结构域、TRAF6结合结构域、核定位信号、核输出信号、PEST结构域、LC3相互作用结构域、KEAP1相互作用结构域和C端泛素结合结构域 Contains nine functional domains: phox and bem1 domains (PB1), ZZ type zinc finger domain (ZZ), TRAF6 binding domain (TB), nuclear localization signal (NLS), nuclear export signal (NES), pest domain, LC3 interacting region (LIR), Kelch like ECH associated protein 1-interacting region (KIR) and a C-terminal ubiquitin binding domain (UBA). |
| |
p62丰富的功能结构域赋予其多样的分子功能,调控不同信号通路进而参与细胞生长、自噬、癌变等生理病理过程。不同的结构域如何影响p62的相互作用蛋白、细胞定位以及翻译后修饰状态值得开展更加系统深入的研究,为理解p62如何精细调控下游底物群、介导特定生物学过程提供理论参考。
2 p62的分子功能 2.1 p62与相分离为了将细胞内复杂的生物化学反应区分开,细胞形成了许多区隔或者细胞器。作为一个区隔要满足两个特点:一是它要拥有与周围环境隔开的边界;二是其中的组分可以自由流动。许多这样的区域具有膜结构,例如高尔基体、线粒体和溶酶体等[12]。然而有些没有膜结构,例如核仁以及中心体等。其中,相分离(phase separation)为细胞提供了一种相对独立的区域促使特定生化反应的有序进行。
从分子角度讲,当大分子之间相互作用的能量优势大于体系熵增大的趋势时相分离会发生。研究报道细胞中内源性或外源性表达的p62都能形成细胞质包涵体(p62小体)[13]。清华大学俞立团队发现p62介导的相分离依赖于其他多泛素化货物蛋白或游离泛素链[14]。p62通过N端PB1结构域多聚化,并通过C端UBA结构域与泛素结合。相分离发生的过程依赖泛素链长度,随着链长的增加,诱发相分离的效率随之增加[14]。除此之外,p62的互作蛋白可以促进p62小体的形成, 例如死亡结构域相关蛋白(death domain-associated protein, DAXX)、自噬受体(next to brca1 gene 1 protein, NBR1)、转录增强子辅助蛋白(ubiquitously expressed transcript, UXT)、精氨化底物等[15-18]。这些蛋白通过不同的机制促进p62聚合。例如,NBR1和K63泛素链促进p62小体的形成依赖于p62的PB1结构域,DAXX增强p62的相分离但独立于其PB1结构域[14, 17, 19]。p62的ZZ结构域与N-末端精氨酸化蛋白或其他含有N-降解子的自噬底物之间的结合可诱导p62聚集并促进其与LC3的结合[20-22]。UXT则与p62的ZZ-LB结构域结合以促进其缩合[18]。这些表明p62介导的相分离的内在结构基础不一样,是否具有功能特异性有待进一步开展研究工作。
p62小体中的蛋白质可以保持其构象和活性,并可以在聚集体内扩散到周围环境中,是一种动态可逆的过程。研究证明,自噬体标记蛋白LC3会以非脂化形式(LC3-I)均匀分布在p62小体内部。这说明除了集中和分离蛋白质货物外,p62小体也可能作为启动选择性自噬级联信号的组织中心[14]。除此以外,多种与p62相互作用蛋白也分布在小体中,例如KEAP1、组蛋白乙酰基转移酶(histone acetyltransferase, KAT5)、黏着斑激酶家族相互作用蛋白(focal adhesion kinase family interacting protein of 200 kDa, FIP200)、丝氨酸/苏氨酸蛋白激酶(serine/threonine-protein kinase, ULK1)等[1]。目前对于相分离发生的分子机制,及相分离液滴组分的研究还有待加强。鉴于p62作为参与自噬和许多信号传导途径的支架蛋白,深入研究有助于阐明p62及其互作蛋白的关系及降解机制。
2.2 p62与蛋白质稳态的维持蛋白质稳态是细胞器生物发生、细胞代谢、应激适应以及任何细胞类型和器官的长期生存和良好状态的关键要求。蛋白质稳定的一个主要挑战是防止未折叠、错误折叠或受损蛋白质的有害后果,这些蛋白质严重干扰细胞功能。该调控过程与衰老和年龄相关疾病有关,包括神经退行病变、癌症、免疫和代谢疾病。在正常条件下,细胞中近30%新合成蛋白质被错误折叠。自噬-溶酶体和泛素-蛋白酶体系统(UPS)是负责细胞稳态的两个主要质量控制途径。两个系统在多个点交叉,以协调它们在蛋白质稳态和细胞器稳态中的作用。虽然这两个系统不同,但越来越多的证据表明它们存在协同配合机制,比如两者共享一些待降解的泛素化蛋白,如亨廷顿病中的亨廷顿蛋白HttQ74[23];同时也共享降解元件,其中就包括p62。
2.2.1 p62与选择性自噬自噬主要有3种类型,即巨自噬(macroautophagy)、微自噬(microautophagy)和分子伴侣介导的自噬(chaperone-mediated autophagy, CMA),通常指巨自噬。自噬普遍存在于真核细胞中,是一自我修复的生物学过程,负责清除长寿命或错误折叠的蛋白质、脂滴和受损的细胞器,以保持细胞内稳态[24]。研究发现,自噬对被运输的货物具有高度的选择性。与非选择性自噬不同,选择性自噬以信号依赖的方式选择特定底物,并由自噬接头蛋白介导,包括p62、NBR1、TAX1结合蛋白(TAX1-binding protein1, TAX1BP1)等[25-28]。p62是参与选择性自噬重要的接头蛋白,它对于泛素化聚集体的形成及运输至关重要。底物蛋白经由多聚泛素链修饰后与p62的UBA结构域相互作用,形成p62与自噬底物蛋白质或细胞器的聚集体;p62的PB1结构域再通过自身寡聚化促进泛素化底物的包装[29],经由p62的LIR结构域靶向进入自噬溶酶体中降解。ULK1激酶介导p62的S409位点磷酸化。随后在其S403位点由TANK结合激酶1 (TANK-binding kinase 1, TBK1)磷酸化[29]。在UBA结构域的S403和S409处的磷酸化导致p62与poly-Ub结合亲和力增强,促进泛素化底物的降解。p62识别底物的特异性主要取决于UBA结构域的K63泛素链的偏好性[14]。研究证明,底物中的特定蛋白质序列,例如1型和2型N末端降解因子(N-degron),使它们被p62特异性募集并通过自噬降解。
2.2.2 p62与蛋白酶体泛素-蛋白酶体系统在短寿命、错误折叠和损坏的蛋白质降解中起关键作用。这对于维持蛋白质稳态、炎症、氧化应激及细胞凋亡是必要的。泛素化修饰的蛋白质被26S蛋白酶体识别和降解[30]。蛋白酶体是高度保守的蛋白酶复合物,20S催化核心颗粒和19S或11S调节颗粒,定位于细胞核与细胞质中。调节颗粒19S中具有ATPase环(RP triple ATPase 1–6,Rpt1–6),参与底物蛋白的去折叠并转运至20S核心颗粒。p62的N端PB1结构域可能与26S蛋白酶体的Rpt1和S5a/Rpn10相互作用,C端的UBA结构域结合泛素化蛋白促进底物降解。另外,p62使用其自身的两个核定位信号域(NLS1和NLS2)和一个核输出基序(NES)进行连续快速的核质穿梭[31],可以将泛素化货物从细胞核输出到细胞质中[8],从而参与细胞核蛋白的质量控制。此外,p62定位于核聚集体中,在将泛素化包含物募集到细胞核中的蛋白酶体中起关键作用。
蛋白酶体的抑制或过载在大多数细胞类型中诱导自噬,这提供了两个系统之间功能连接的证据。自噬主要补偿蛋白酶体降解能力的降低,并消除由受损细胞器或潜在毒性蛋白质聚集体积累造成的威胁。相反,其他研究未检测到溶酶体抑制后蛋白酶体活性的上调。抑制HeLa细胞中的自噬后,累积的p62螯合泛素化蛋白,从而延迟它们向蛋白酶体的穿梭,但蛋白酶体活性本身不受影响[23]。因此,p62作为这两种途径的调节物及底物,它如何选择底物并选择特定降解途径的机制有待阐明。
3 p62参与的生物学信号通路一定诱导条件下,p62可通过与其他蛋白质的相互作用发挥多种功能,包括核转录因子NF-E2相关转录因子2[32]、核转录因子κappa B (nuclear transcription factor-κB, NF-κB)[33]和哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin, mTOR)[34]等。通过与细胞内多种信号通路的标志分子相互作用,参与调控相关信号通路。通过与这些其他信号途径中的调节蛋白相互作用,p62参与调控细胞内多条重要信号通路,最终调节细胞生命活动。
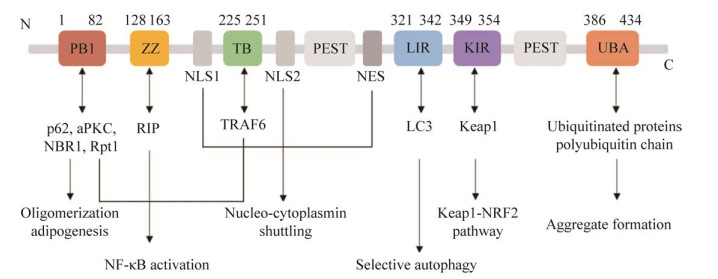
3.1 p62与NRF2-KEAP1信号通路NRF2与抗氧化反应元件(antioxidant response element, ARE)相结合,促进下游一系列抗氧化基因的表达,如抗氧化酶、谷胱甘肽合成代谢相关酶等。作为细胞内最重要的抗氧化应激调控通路发挥强大的抗氧化作用,从而稳定细胞内环境[35]。正常情况下,KEAP1作为基于Cullin 3 (CUL3)的E3连接酶的衔接子成分(用于底物识别),促进转录因子NRF2的泛素化和降解,从而使其保持低水平。当暴露于亲电试剂或活性氧(reactive oxygen species, ROS)时,KEAP1的反应性半胱氨酸残基被直接氧化修饰,降低了KEAP1-CUL3复合物的泛素E3连接酶活性并导致NRF2稳定不易被降解。新生的NRF2可以直接转移到细胞核中,与抗氧化反应元件或亲电子反应元件(electrophile response element,EpREs)结合,在其调控区域启动抗氧化酶和抗炎酶的转录。
p62主要通过3种机制调节NRF2对氧化应激的反应:(1) 磷酸化p62的KIR结构域(S351)可能识别KEAP1的(double glycine repeat, DGR)结构域[36],形成p62-KEAP1复合物,该复合物被UPS途径清除,使NRF2入核,从而激活NRF2靶基因的转录;(2) p62通过组装p62-KEAP1复合物和LC3形成LC3-p62-KEAP1复合物来控制KEAP1的周转,该复合物通过选择性自噬被消除[37];(3) 游离p62直接进入细胞核调节ARE,启动下游基因的表达。有趣的是,NRF2也能刺激自噬。当大量NRF2易位到细胞核中时,它通过小的Mafs蛋白与ARE结合,并上调自噬相关基因的转录,包括Atg5、p62和Map1Lc3b,其启动子包含在ARE核苷酸序列中,从而上调Atg5、p62和Lc3b蛋白的表达[38](图 3)。总之,NRF2途径和自噬之间通过p62-KEAP1-NRF2正反馈回路存在相互调节关系[39]。
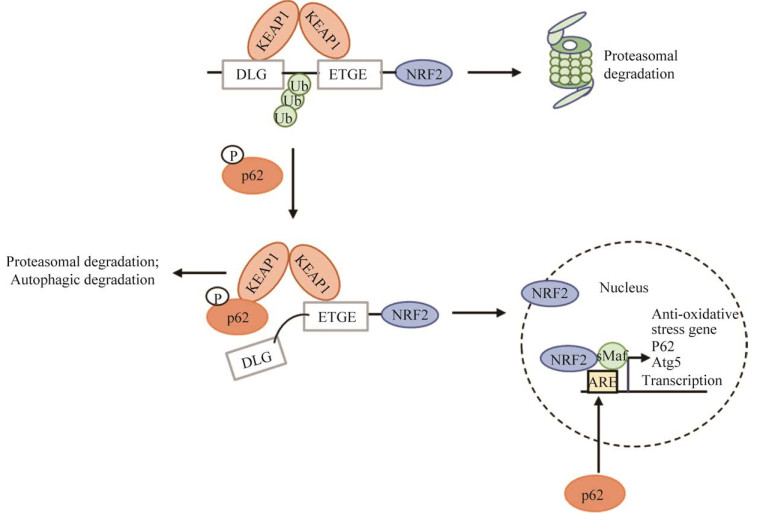
|
| 图 3 p62参与调节NRF2-KEAP1信号通路 Fig. 3 p62 is involved in regulating the NRF2-KEAP1 signaling pathway. KEAP1结合NRF2使NRF2从蛋白酶体降解;p62识别KEAP1,形成p62-KEAP1复合物被蛋白酶体/自噬-溶酶体降解,释放NRF2入核 KEAP1 binds to NRF2, allowing NRF2 to be degraded from the proteasome. p62 recognizes KEAP1, forming a p62-KEAP1 complex and degradation by the proteasome/autophagy lysosome, releasing NRF2 into the nucleus. |
| |
B细胞核因子κ-轻链增强子(NF-κB)是一类诱导型转录因子,在免疫系统中发挥多种进化保守作用,参与调节与机体免疫、炎症反应、细胞分化等相关基因的转录,同时也调控多种生理或病理相关基因的表达[40]。NF-κB家族由5种相关蛋白组成,p50 (NF-κB1)、p52 (NF-κB2)、p65 (RelA)、RelB和cRel (Rel),它们共有约300个氨基酸长的N-末端Rel同源结构域(Rel homology domain, RHD),这些蛋白质二聚化后形成有功能的NF-κB,活化的NF-κB进入细胞核内与DNA结合。细胞质中3个抑制因子(IκBa、IκBβ、IκBε)可以结合部分的NF-κB二聚体使其以无活性的状态存在。各种信号通过降解IκBs的方式来活化NF-κB。其中,IκB激酶(IκB kinase, IKK)作为一个重要角色,它是一个调节亚单位,由IKK-γ (也被称为NF-kappa-B essential modulator,NEMO)和2个催化亚单位Iκk-α、Iκk-β组成。IκBs可以被IκBs激酶(IKK)催化,使其的两个保守的丝氨酸残基磷酸化,然后在赖氨酸残基上多泛素化,最终被26S蛋白酶体降解。
p62有几种方式参与调控NF-κB信号通路。首先,肿瘤坏死因子α (tumor necrosis factor α, TNFα)与肿瘤坏死因子受体1 (tumor necrosis factor receptor 1, TNF-R1)结合,TNF-R1募集TNFR1相关的死亡结构域蛋白(TNF receptor-associated death domain, TRADD),后者又募集受体相互作用蛋白激酶1 (receptor-interacting serine/threonine-protein kinase 1, RIP1)和肿瘤坏死因子受体相关因子2 (TNF receptor-associated factor 2, TRAF2)。p62通过ZZ锌指结构参与RIP蛋白的结合[41],RIP1被E3泛素连接酶TRAF2多泛素化[42]。多泛素化RIP1招募磷酸化IKK的TAK1,这也导致IKK活化。使NF-κB被释放并转移到细胞核中,激活靶基因的转录。此外,核因子-κB受体激活剂配体(receptoractivator of nuclear factor-κB ligand, RANKL)与其受体RANK结合后会诱导TRAF6聚集,而后TRAF6通过激活IKK复合体来激活NF-κB,而p62与TRAF6结合后,p62-TRAF6复合体将与非典型性蛋白激酶C (atypical protein kinase C, aPKC)蛋白相互作用,共同调节NF-κB的活化[43]。同时,p62在NF-κB活化中的另一个作用是促进A20的降解,A20是一种去泛素酶,通过抑制NEMO的K63连接泛素化和RIP1的K63连接泛素化来防止过量的NF-κB活化,随后通过26S蛋白酶体降解[44-45]。另外,不仅作为NF-κB通路的调节因子,p62本身也会被NF-κB上调,形成正反馈环(图 4)[46]。
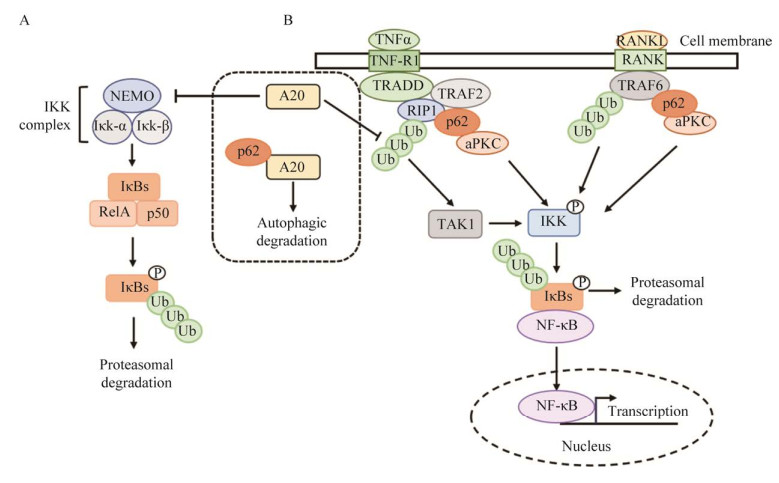
|
| 图 4 p62与NF-κB信号通路 Fig. 4 p62 and NF-κB signaling. A:IKK复合物促进IκBs的降解. p62促进A20的降解,A20抑制NF-κB活化. B:p62调控NF-κB信号通路. p62结合RIP1蛋白,RIP1被多泛素化并招募TAK1,促进IKK活化;p62-TRAF6复合体将与aPKC蛋白相互作用,共同调节NF-κB的活化 A: The IKK complex promotes degradation of IκBs. p62 promotes degradation of A20 which inhibits NF-κB activation. B: P62 regulates NF-κB signaling pathways. p62 binds to RIP1, which is polyubiquitinated and recruits TAK1, promoting IKK activation; The p62-TRAF6 complex interacts with aPKC proteins to co-regulate NF-κB. |
| |
雷帕霉素靶蛋白(mTOR)是一种丝氨酸/苏氨酸激酶,作为联系代谢上下游的关键枢纽分子,参与调控几个关键的生物学过程:蛋白质合成、脂肪合成、抑制细胞自噬。在营养充足的情况下,mTOR被激活,促进物质代谢和能量储存;而在相对匮乏的时候,其活性受到抑制以保持细胞原料和能量的相对稳定。
mTOR含有两种复合体,分别为mTORC1和mTORC2,这两个复合体在细胞内传递不同的信号。研究证明,营养感知反应与溶酶体靶向mTORC1复合体有关[47]。Rag蛋白家族是类似Ras的小GTPase,是氨基酸特异性调节因子。Rag GTPases是RagA/B与RagC或D构成的异二聚体G蛋白。p62介导mTOR与Rag GTPases体内相互作用,促进mTORC1复合物易位至溶酶体表面,从而抑制自噬[31]。p62通过连接Raptor (mTORC1复合物的亚基之一),与mTORC1和Rag GTPases形成的高分子量复合物相连,这是mTOR激活的关键步骤(图 5)。p62的高表达会激活mTORC1通路[48]。在体外细胞模型中,p62被发现是介导氨基酸诱导的mTORC1下游靶点激活所必需的,例如真核细胞翻译起始因子4E结合蛋白l (the eΙF4E-binding protein l, 4EBP1)[49]。所以p62可能是感受细胞内游离氨基酸水平的调节开关。
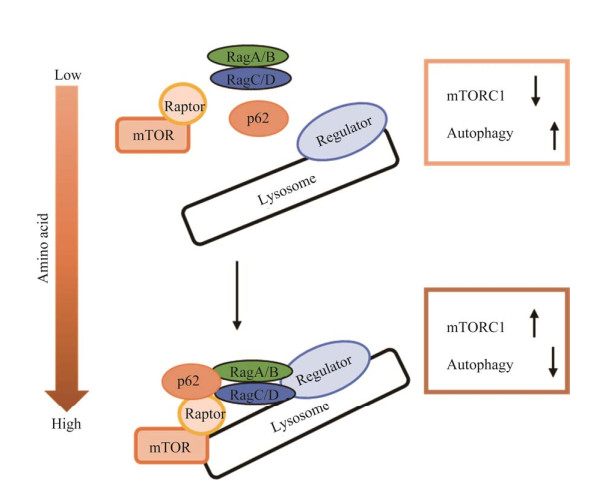
|
| 图 5 p62的高表达激活mTORC1通路 Fig. 5 High expression of p62 activates the mTORC1 pathway. p62通过连接Raptor与mTORC1和Rag GTPases复合物相连,激活mTOR P62 activates mTOR by linking Raptor to mTORC1 and the rag GTPases complex. |
| |
p62分布广,在所有组织中均有表达,在肌肉组织中表达水平较高。因此,p62可能与多种组织、器官病变的发生发展相关。研究表明,p62参与多种疾病的病理过程,如神经退行性疾病,癌症以及代谢性疾病等。已有研究证明p62是肿瘤细胞遗传、自噬、凋亡、迁移的核心因子,也是患者预后的指标,在肝癌、肾癌、前列腺癌等癌症中均与较短生存率相关。p62的功能多样性导致它在疾病中的作用是复杂且双面的。例如,以p62为中心的聚集体是神经退行性疾病及肝癌等疾病的重要标志物,在疾病的发生发展中起重要作用。然而p62在肝星状细胞(hepatic stellate cell, HSC)中起到抑制肝癌发展的作用。因此,p62在各种类型的癌症和肿瘤相关微环境中作为原癌基因或抑癌基因发挥作用,这对肿瘤的发展以及患者的治疗和预后产生较大影响。
4.1 p62与神经退行性疾病蛋白质错误折叠不能被有效降解时,被加工成小的错误折叠的低聚物,对神经元产生毒性。比如α-突触核蛋白、亨廷顿蛋白在内的细胞内聚集体分别是在帕金森氏病和亨廷顿氏病中发现的错误折叠蛋白[2, 50]。因此,蛋白质疾病(包括神经退行性疾病中发现的蛋白质疾病)的一个治疗策略是促进有毒寡聚分子的去除。鉴于p62在选择性自噬中清楚错误折叠蛋白的作用,p62可能具有神经保护作用。
阿尔茨海默病是一种以认知功能障碍和记忆障碍为主要临床特征的神经退行性疾病。病理特征是细胞外淀粉样蛋白-β (amyloid-β, Aβ)斑块的形成和神经原纤维缠结(neurofibrillary tangle, NFT)的神经内沉积[51]。早在2001年人们就发现了p62和泛素化蛋白在阿尔茨海默病患者神经元和神经胶质细胞中大量聚集[52]。虽然在阿尔茨海默病患者中未检测到p62的直接突变,但在一些阿尔茨海默病患者中检测出p62的低表达[53]。细胞内MAPT/tau (microtubule associated protein, tau)蛋白的积聚是阿尔茨海默病和其他tau蛋白相关疾病的病理学的显著特征,这种聚集大部分是源于巨自噬/自噬缺陷。p62与神经原纤维缠结以及其他聚集物的结合,抑制了p62与泛素化tau蛋白的识别与结合,进而影响tau蛋白降解的效率,这是阿尔茨海默病发病的重要原因之一。另外,p62 UBA结构域内Ser403的磷酸化增加p62与多聚泛素链的亲和力,促进其与错误折叠蛋白质和泛素标记功能失调蛋白相结合,进行促进泛素化底物的清除[54]。磷酸化的p62通过UBA结构域识别多聚泛素化tau蛋白,引导其自噬降解。研究证明Ser403磷酸化的p62在阿尔茨海默氏病患者大脑中降低[55]。因此,p62的磷酸化及其激酶成为重要的潜在治疗靶点。比如,TBK1是磷酸化p62 S403位点的激酶,PTK2 (protein tyrosine kinase 2)连接TBK1并增加其活性,促进p62的Ser403位点磷酸化。研究证实PTK2-TBK1-p62轴通过调节UPS损伤诱导的神经毒性在TAR DNA结合蛋白(TAR DNA binding protein, TARDBP)的发病机制中起关键作用。因此,靶向PTK2-TBK1-p62轴成为治疗对具有TARDBP蛋白介导的神经退行性疾病的潜在靶点[56]。
NRF2下调是神经退行性疾病的重要特征之一[57]。NRF2的表达随着年龄的增长而明显下降,导致细胞质中的活性氧(ROS)不能立即清除。p62参与调节NRF2-KEAP1信号通路。NRF2与KEAP1的结合促进其蛋白酶体降解,而p62能够竞争性识别KEAP1,形成p62-KEAP1复合物从而释放NRF2入核,并进一步促进p62的转录。p62-KEAP1-NRF2正反馈环参与消除阿尔茨海默病诱导的ROS和蛋白质聚集体。因此,维持p62-KEAP1-NRF2正反馈环的稳态可能是阿尔茨海默病治疗的潜在策略[58]。研究证明p62的过度表达挽救了转基因阿尔茨海默病小鼠的认知缺陷,Aβ水平和斑块负荷降低,自噬增加。因此p62本身可以被确定为针对神经退行性疾病的潜在治疗靶点。
4.2 p62与肝细胞癌原发性肝癌(hepatic cell carcinoma, HCC)是肝癌的主要形式,是全球癌症死亡的第4大常见原因,5年生存率约为18%[59-60]。p62已经被确定为Mallory-Denk bodies (MDBs)或细胞内透明体的关键组成部分,已知这些蛋白质聚集体在酒精性脂肪性肝炎(alcoholic steatohepatitis, ASH)、非酒精性脂肪性肝炎(non-alcoholic steatohepatitis, NASH)和肝细胞癌(HCC)患者的活检样本中经常检测到,并且与肿瘤分级和复发相关[10, 61-62]。
p62通过激活肝细胞中的NRF2、mTORC1和c-MYC原癌基因途径,促进HCC的发生和发展[62]。研究表明,p62的积累在肝癌发生发展的不同阶段发挥了关键作用。它通过支持含有ROS的肝癌祖细胞(hepatocellular carcinoma progenitor cell, HcPC)存活和积累来增加癌症发生的风险[62]。癌症起始细胞不断增殖导致氧化和代谢应激[63]。p62转录的主要激活因子,NRF2在慢性肝脏炎症和氧化应激期间被激活,促使p62的表达在各种细胞应激反应中升高。与此同时,HCC起始细胞通过累积p62聚集体获得替代性适应机制,高表达的p62竞争性结合KEAP1,释放更多NRF2,促进NRF2的稳定以及NRF2信号的上调形成正反馈环,保护氧化应激的起始细胞进而促进肝癌发生。在HCC活组织检查中,p62的S28和S349位点的磷酸化增强,分别促进p62聚集以及提高螯合KEAP1的能力,导致NRF2信号传导的过度活化,这对驱动HCC发展至关重要。此外,p62积累激活mTORC1/c-MYC信号传导以改变代谢并促进增殖。在肝脏特异性结节性硬化症1中,mTORC1信号传导的组成型活化发展自发性HCC。p62敲除的小鼠中,mTORC1活性降低并抑制HCC发展[62, 64]。p62的mTORC1依赖性磷酸化可以增强其激活NRF2信号传导的功能[36],表明p62和mTORC1之间存在正反馈环以协同促进NRF2的过度活化。除NRF2外,p62还在HCC发展过程中促进c-MYC癌基因的表达。c-MYC促进细胞增殖和代谢重编程,因此对肿瘤生长至关重要,通常在HCC中上调[65]。
慢性肝损伤启动肝细胞死亡、炎症和纤维化的恶性循环,导致肝硬化和癌症,其中肝星状细胞(HSC)起决定性作用。HSC中的维生素D受体(vitamin D receptor, VDR)活化抑制肝脏炎症和纤维化。p62直接与VDR和维甲酸X受体(retinoid X receptor, RXR)相互作用,促进其异二聚化,发挥抗纤维化和抗炎功能。HSC中p62的缺失阻滞VDR发挥作用,导致炎症、纤维化增强以及HCC的进展[66]。综上所述,p62是一种在肝实质细胞中上调但在HCC相关HSC中下调的蛋白质,说明p62可作为HSC中HCC发展的抑制因子。
4.3 p62与其他疾病研究证明,信号通路中p62和其他关键蛋白表达的综合分析可用作癌症预后的生物标志物[67-68]。2013年,p62被确定为肾癌的发病靶点[69]。在透明细胞肾细胞癌(clear cell renal cell carcinoma, RCC)系中过表达p62促进了对氧化还原应激的抗性并增加了软琼脂生长,而p62的下调降低了对氧化还原应激的抗性,损害了细胞适应性并减少了肿瘤的形成。不久之后研究表明p62在结直肠癌组织中上调[70],p62的异位过表达有助于结直肠肿瘤的发生。卵巢癌中p62的ZZ结构域调节受体相互作用蛋白1 (RIP1)介导NF-κB通路的激活,影响卵巢癌细胞的增殖和凋亡[7]。越来越多的证据表明p62也是代谢性疾病的关键调节因子,如肥胖、T2DM病、非酒精性脂肪性肝病、代谢性骨病、痛风和甲状腺疾病等[71]。脂肪是身体能量的关键储存库,肿瘤和脂肪组织建立共生作用。肿瘤促进脂肪组织的代谢活化,导致肿瘤微环境储存的营养物质减少。由于p62激活mTOR通路,因此脂肪细胞内p62的缺乏或低表达导致细胞的代谢活性降低,从而减少脂肪中正常生理的能量利用,有利于供给肿瘤的高能量需求[72]。因此,尽管p62在癌上皮中起肿瘤促进剂的作用,但它在微环境中起到肿瘤抑制因子的作用[73]。同时,研究结果显示肥胖激活的TBK1信号转导通路在脂毒性过程中促进肝细胞中p62包涵体的积累,它可能为营养过剩和肥胖促进NASH相关的HCC发展提供分子通道[74-75]。因此,如何合理平衡p62多元的生物学功能,还需要进一步努力探索蛋白质疾病的结构和病理基础。
5 结语综上所述,p62具有多个功能结构域,这决定了它能参与多个信号途径的调控,涉及许多病理生理过程,尽管其具体作用机制目前仍未阐明,但可以肯定p62在疾病中的作用是复杂、双面的。癌细胞受益于p62水平的增加,它为癌细胞提供了更好的抗氧化和解毒反应以及促血管生成和生存信号,这两者都促进了肿瘤的进展。与此同时,周围间质细胞中p62的丢失会下调这些途径,导致代谢物和其他分泌信号的分泌,从而进一步帮助癌细胞的发展[76]。因此,通过自噬依赖或独立的机制维持肿瘤和间质中p62的动态平衡将对肿瘤发生过程的最终结果起决定性作用。自从利用泛素-蛋白酶体系统降解靶蛋白的蛋白水解靶向嵌合体(proteolysis targeting chimera, PROTAC)的分子概念被报道以来,靶向蛋白质降解(targeted protein degradation, TPD)已逐渐成为治疗相关疾病的重要方法[77]。通过识别结合泛素化底物,p62将细胞内两个主要的蛋白降解机制:自噬-溶酶体系统和UPS有机联系起来,如今利用p62的功能靶向降解目的蛋白的技术逐渐发展。Ji等[78]构建了自噬靶向嵌合体(autophagy-targeting chimera, AUTOTAC),通过靶标结合配体(target-binding ligands, TBL)和自噬靶标配体(autophagy-targeting ligands, ATL)将靶标结合到p62,通过巨自噬促进靶标-p62复合物的降解,并在表达人病理性tau突变体的鼠脑模型中证实了其靶向错误折叠蛋白的治疗功效,这为基础研究和药物开发中的选择性蛋白水解提供了平台,也为p62与疾病治疗的联系提供了技术出口。随着科学的发展,p62与相分离、蛋白质稳态、肿瘤、疾病的关系将会不断得到揭示,为相关疾病治疗提供新的理论依据。结合靶向蛋白质降解技术,期望解决某些难成药性蛋白靶点引起的疾病,为疾病治疗提供更多新思路与新方法。
| [1] |
KAGEYAMA S, GUDMUNDSSON SR, SOU YS, ICHIMURA Y, TAMURA N, KAZUNO S, UENO T, MIURA Y, NOSHIRO D, ABE M, MIZUSHIMA T, MIURA N, OKUDA S, MOTOHASHI H, LEE JA, SAKIMURA K, OHE T, NODA NN, WAGURI S, ESKELINEN EL, et al. p62/SQSTM1-droplet serves as a platform for autophagosome formation and anti-oxidative stress response. Nature Communications, 2021, 12: 16. DOI:10.1038/s41467-020-20185-1
|
| [2] |
MA SF, ATTARWALA IY, XIE XQ. SQSTM1/p62: a potential target for neurodegenerative disease. ACS Chemical Neuroscience, 2019, 10(5): 2094-2114. DOI:10.1021/acschemneuro.8b00516
|
| [3] |
PANKIV S, CLAUSEN TH, LAMARK T, BRECH A, BRUUN JA, OUTZEN H, ØVERVATN A, BJØRKØY G, JOHANSEN T. p62/SQSTM1 binds directly to Atg8/LC3 to facilitate degradation of ubiquitinated protein aggregates by autophagy. Journal of Biological Chemistry, 2007, 282(33): 24131-24145. DOI:10.1074/jbc.M702824200
|
| [4] |
SHIN J. P62 and the sequestosome, a novel mechanism for protein metabolism. Archives of Pharmacal Research, 1998, 21(6): 629-633. DOI:10.1007/BF02976748
|
| [5] |
GEETHA T, WOOTEN MW. Structure and functional properties of the ubiquitin binding protein p62. FEBS Letters, 2002, 512(1/2/3): 19-24.
|
| [6] |
CHRISTIAN F, KRAUSE E, HOUSLAY MD, BAILLIE GS. PKA phosphorylation of p62/SQSTM1 regulates PB1 domain interaction partner binding. Biochimica et Biophysica Acta (BBA)-Molecular Cell Research, 2014, 1843(11): 2765-2774. DOI:10.1016/j.bbamcr.2014.07.021
|
| [7] |
YAN XY, ZHANG Y, ZHANG JJ, ZHANG LC, LIU YN, WU Y, XUE YN, LU SY, SU J, SUN LK. p62/SQSTM1 as an oncotarget mediates cisplatin resistance through activating RIP1-NF-κB pathway in human ovarian cancer cells. Cancer Science, 2017, 108(7): 1405-1413. DOI:10.1111/cas.13276
|
| [8] |
PANKIV S, LAMARK T, BRUUN JA, ØVERVATN A, BJØRKØY G, JOHANSEN T. Nucleocytoplasmic shuttling of p62/SQSTM1 and its role in recruitment of nuclear polyubiquitinated proteins to promyelocytic leukemia bodies. Journal of Biological Chemistry, 2010, 285(8): 5941-5953. DOI:10.1074/jbc.M109.039925
|
| [9] |
GARCÍA-ALAI MM, GALLO M, SALAME M, WETZLER DE, McBRIDE AA, PACI M, CICERO DO, de PRAT-GAY G. Molecular basis for phosphorylation-dependent, PEST-mediated protein turnover. Structure, 2006, 14(2): 309-319. DOI:10.1016/j.str.2005.11.012
|
| [10] |
DENK H, STUMPTNER C, ABUJA PM, ZATLOUKAL K. Sequestosome 1/p62-related pathways as therapeutic targets in hepatocellular carcinoma. Expert Opinion on Therapeutic Targets, 2019, 23(5): 393-406. DOI:10.1080/14728222.2019.1601703
|
| [11] |
KATSURAGI Y, ICHIMURA Y, KOMATSU M. p62/SQSTM1 functions as a signaling hub and an autophagy adaptor. FEBS Journal, 2015, 282(24): 4672-4678. DOI:10.1111/febs.13540
|
| [12] |
孙大晓. 多泛素链诱导的p62蛋白相分离驱动自噬降解底物的募集[D]. 北京: 清华大学博士学位论文, 2018. SUN DX. Polyubiquitin chain-induced p62 phase separation drives autophagic cargo segregation[D]. Beijing: Doctoral Dissertation of Tsinghua University, 2018 (in Chinese). |
| [13] |
BJØRKØY G, LAMARK T, BRECH A, OUTZEN H, PERANDER M, ØVERVATN A, STENMARK H, JOHANSEN T. p62/SQSTM1 forms protein aggregates degraded by autophagy and has a protective effect on huntingtin-induced cell death. Journal of Cell Biology, 2005, 171(4): 603-614. DOI:10.1083/jcb.200507002
|
| [14] |
SUN DX, WU RB, ZHENG JX, LI PL, YU L. Polyubiquitin chain-induced p62 phase separation drives autophagic cargo segregation. Cell Research, 2018, 28(4): 405-415. DOI:10.1038/s41422-018-0017-7
|
| [15] |
KEHL SR, SOOS BL A, SAHA B, CHOI SW, HERREN AW, JOHANSEN T, MANDELL MA. TAK 1 converts sequestosome 1/p62 from an autophagy receptor to a signaling platform. EMBO Reports, 2019, 20(9): e46238.
|
| [16] |
SÁNCHEZ-MARTÍN P, SOU YS, KAGEYAMA S, KOIKE M, WAGURI S, KOMATSU M. NBR 1-mediated p62-liquid droplets enhance the keap1-Nrf2 system. EMBO Reports, 2020, 21(3): e48902.
|
| [17] |
YANG Y, WILLIS TL, BUTTON RW, STRANG CJ, FU YH, WEN X, GRAYSON PRC, EVANS T, SIPTHORPE RJ, ROBERTS SL, HU B, ZHANG JK, LU BX, LUO SQ. Cytoplasmic DAXX drives SQSTM1/p62 phase condensation to activate Nrf2-mediated stress response. Nature Communications, 2019, 10: 3759. DOI:10.1038/s41467-019-11671-2
|
| [18] |
YOON MJ, CHOI B, KIM EJ, OHK J, YANG C, CHOI YG, LEE J, KANG C, SONG HK, KIM YK, WOO JS, CHO Y, CHOI EJ, JUNG H, KIM C. UXT chaperone prevents proteotoxicity by acting as an autophagy adaptor for p62-dependent aggrephagy. Nature Communications, 2021, 12: 1955. DOI:10.1038/s41467-021-22252-7
|
| [19] |
ZAFFAGNINI G, SAVOVA A, DANIELI A, ROMANOV J, TREMEL S, EBNER M, PETERBAUER T, SZTACHO M, TRAPANNONE R, TARAFDER AK, SACHSE C, MARTENS S. p62 filaments capture and present ubiquitinated cargos for autophagy. The EMBO Journal, 2018, 37(5): e98308.
|
| [20] |
CHA-MOLSTAD H, SUNG KS, HWANG J, KIM KA, YU JE, DONG YOO Y, JANG JM, HAN DH, MOLSTAD M, KIM JG, LEE YJ, ZAKRZEWSKA A, KIM SH, KIM ST, KIM SY, LEE HG, SOUNG NK, AHN JS, CIECHANOVER A, KIM BY, et al. Amino-terminal arginylation targets endoplasmic reticulum chaperone BiP for autophagy through p62 binding. Nature Cell Biology, 2015, 17(7): 917-929. DOI:10.1038/ncb3177
|
| [21] |
CHA-MOLSTAD H, YU JE, FENG ZW, LEE SH, KIM JG, YANG P, HAN B, SUNG KW, DONG YOO Y, HWANG J, McGUIRE T, SHIM SM, DONG SONG H, GANIPISETTI S, WANG NZ, JANG JM, LEE MJ, KIM SJ, LEE KH, et al. p62/SQSTM1/sequestosome-1 is an N-recognin of the N-end rule pathway which modulates autophagosome biogenesis. Nature Communications, 2017, 8: 102. DOI:10.1038/s41467-017-00085-7
|
| [22] |
ZHANG Y, MUN SR, LINARES JF, AHN J, TOWERS CG, JI CH, FITZWALTER BE, HOLDEN MR, MI WY, SHI XB, MOSCAT J, THORBURN A, DIAZ-MECO MT, KWON YT, KUTATELADZE TG. ZZ-dependent regulation of p62/SQSTM1 in autophagy. Nature Communications, 2018, 9: 4373. DOI:10.1038/s41467-018-06878-8
|
| [23] |
KOROLCHUK VI, MANSILLA A, MENZIES FM, RUBINSZTEIN DC. Autophagy inhibition compromises degradation of ubiquitin-proteasome pathway substrates. Molecular Cell, 2009, 33(4): 517-527. DOI:10.1016/j.molcel.2009.01.021
|
| [24] |
LIU J, KUANG FM, KROEMER G, KLIONSKY DJ, KANG R, TANG DL. Autophagy-dependent ferroptosis: machinery and regulation. Cell Chemical Biology, 2020, 27(4): 420-435. DOI:10.1016/j.chembiol.2020.02.005
|
| [25] |
BIRGISDOTTIR ÅB, LAMARK T, JOHANSEN T. The LIR motif-crucial for selective autophagy. Journal of Cell Science, 2013, 126(15): 3237-3247. DOI:10.1242/jcs.126128
|
| [26] |
SPARRER KMJ, GACK MU. TRIM proteins: new players in virus-induced autophagy. PLoS Pathogens, 2018, 14(2): e1006787. DOI:10.1371/journal.ppat.1006787
|
| [27] |
FARRÉ JC, SUBRAMANI S. Mechanistic insights into selective autophagy pathways: lessons from yeast. Nature Reviews Molecular Cell Biology, 2016, 17(9): 537-552. DOI:10.1038/nrm.2016.74
|
| [28] |
ZAFFAGNINI G, MARTENS S. Mechanisms of selective autophagy. Journal of Molecular Biology, 2016, 428(9): 1714-1724. DOI:10.1016/j.jmb.2016.02.004
|
| [29] |
KRAFT LJ, DOWLER J, MANRAL P, KENWORTHY AK. Size, organization, and dynamics of soluble SQSTM1 and LC3-SQSTM1 complexes in living cells. Autophagy, 2016, 12(9): 1660-1674. DOI:10.1080/15548627.2016.1199299
|
| [30] |
BHATTACHARYYA S, YU HQ, MIM C, MATOUSCHEK A. Regulated protein turnover: snapshots of the proteasome in action. Nature Reviews Molecular Cell Biology, 2014, 15(2): 122-133. DOI:10.1038/nrm3741
|
| [31] |
DURAN A, AMANCHY R, LINARES JF, JOSHI J, ABU-BAKER S, POROLLO A, HANSEN M, MOSCAT J, DIAZ-MECO MT. p62 is a key regulator of nutrient sensing in the mTORC1 pathway. Molecular Cell, 2011, 44(1): 134-146. DOI:10.1016/j.molcel.2011.06.038
|
| [32] |
YAMAMOTO M, KENSLER TW, MOTOHASHI H. The KEAP1-NRF2 system: a thiol-based sensor-effector apparatus for maintaining redox homeostasis. Physiological Reviews, 2018, 98(3): 1169-1203. DOI:10.1152/physrev.00023.2017
|
| [33] |
HAYDEN MS, GHOSH S. Regulation of NF-κB by TNF family cytokines. Seminars in Immunology, 2014, 26(3): 253-266. DOI:10.1016/j.smim.2014.05.004
|
| [34] |
TSAI LC L, XIE L, DORE K, XIE L, del RIO JC, KING CC, MARTINEZ-ARIZA G, HULME C, MALINOW R, BOURNE PE, NEWTON AC. Zeta inhibitory peptide disrupts electrostatic interactions that maintain atypical protein kinase C in its active conformation on the scaffold p62. Journal of Biological Chemistry, 2015, 290(36): 21845-21856. DOI:10.1074/jbc.M115.676221
|
| [35] |
TANG ZH, HU B, ZANG FZ, WANG JX, ZHANG XD, CHEN HJ. Nrf2 drives oxidative stress-induced autophagy in nucleus pulposus cells via a keap1/Nrf2/p62 feedback loop to protect intervertebral disc from degeneration. Cell Death & Disease, 2019, 10(7): 510.
|
| [36] |
ICHIMURA Y, WAGURI S, SOU YS, KAGEYAMA S, HASEGAWA J, ISHIMURA R, SAITO T, YANG YJ, KOUNO T, FUKUTOMI T, HOSHII T, HIRAO A, TAKAGI K, MIZUSHIMA T, MOTOHASHI H, LEE MS, YOSHIMORI T, TANAKA K, YAMAMOTO M, KOMATSU M. Phosphorylation of p62 activates the keap1-Nrf2 pathway during selective autophagy. Molecular Cell, 2013, 51(5): 618-631. DOI:10.1016/j.molcel.2013.08.003
|
| [37] |
LIAO WT, WANG ZY, FU ZJ, MA HK, JIANG MD, XU AP, ZHANG W. p62/SQSTM1 protects against cisplatin-induced oxidative stress in kidneys by mediating the cross talk between autophagy and the Keap1-Nrf2 signalling pathway. Free Radical Research, 2019, 53(7): 800-814. DOI:10.1080/10715762.2019.1635251
|
| [38] |
HO CJ, GORSKI SM. Molecular mechanisms underlying autophagy-mediated treatment resistance in cancer. Cancers, 2019, 11(11): 1775. DOI:10.3390/cancers11111775
|
| [39] |
deLa VEGA MR, CHAPMAN E, ZHANG DD. NRF2 and the hallmarks of cancer. Cancer Cell, 2018, 34(1): 21-43. DOI:10.1016/j.ccell.2018.03.022
|
| [40] |
JI Z, HE LZ, REGEV A, STRUHL K. Inflammatory regulatory network mediated by the joint action of NF-kB, STAT3, and AP-1 factors is involved in many human cancers. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(19): 9453-9462. DOI:10.1073/pnas.1821068116
|
| [41] |
SANZ L. The interaction of p62 with RIP links the atypical PKCs to NF-kappa B activation. The EMBO Journal, 1999, 18(11): 3044-3053. DOI:10.1093/emboj/18.11.3044
|
| [42] |
LEE TH, SHANK J, CUSSON N, KELLIHER MA. The kinase activity of Rip1 is not required for tumor necrosis factor-α-induced IκB kinase or p38 MAP kinase activation or for the ubiquitination of Rip1 by Traf2. Journal of Biological Chemistry, 2004, 279(32): 33185-33191. DOI:10.1074/jbc.M404206200
|
| [43] |
赵萍萍, 要乐, 蔚丹丹, 郝慧芳. p62蛋白功能及相关信号通路研究. 生命科学, 2018, 30(1): 100-106. ZHAO PP, YAO L, YU DD, HAO HF. The functions and related signaling pathways of p62. Chinese Bulletin of Life Sciences, 2018, 30(1): 100-106 (in Chinese). DOI:10.13376/j.cbls/2018014 |
| [44] |
SHEMBADE N, HARHAJ EW. Regulation of NF-κB signaling by the A20 deubiquitinase. Cellular & Molecular Immunology, 2012, 9(2): 123-130.
|
| [45] |
KANAYAMA M, INOUE M, DANZAKI K, HAMMER G, HE YW, SHINOHARA ML. Autophagy enhances NFκB activity in specific tissue macrophages by sequestering A20 to boost antifungal immunity. Nature Communications, 2015, 6: 5779. DOI:10.1038/ncomms6779
|
| [46] |
LING JH, KANG YA, ZHAO RY, XIA QH, LEE DF, CHANG Z, LI J, PENG BL, FLEMING JB, WANG HM, LIU JS, LEMISCHKA IR, HUNG MC, CHIAO PJ. KrasG12D-induced IKK2/β/NF-κB activation by IL-1α and p62 feedforward loops is required for development of pancreatic ductal adenocarcinoma. Cancer Cell, 2012, 21(1): 105-120. DOI:10.1016/j.ccr.2011.12.006
|
| [47] |
SANCAK Y, PETERSON TR, SHAUL YD, LINDQUIST RA, THOREEN CC, BAR-PELED L, SABATINI DM. The rag GTPases bind raptor and mediate amino acid signaling to mTORC1. Science, 2008, 320(5882): 1496-1501. DOI:10.1126/science.1157535
|
| [48] |
KOMATSU M, KAGEYAMA S, ICHIMURA Y. p62/SQSTM1/A170: physiology and pathology. Pharmacological Research, 2012, 66(6): 457-462. DOI:10.1016/j.phrs.2012.07.004
|
| [49] |
HONG SE, KIM EK, JIN HO, KIM HA, LEE JK, KOH JS, SEOL H, KIM JI, PARK IC, NOH WC. S6K1 inhibition enhances tamoxifen-induced cell death in MCF-7 cells through translational inhibition of Mcl-1 and survivin. Cell Biology and Toxicology, 2013, 29(4): 273-282. DOI:10.1007/s10565-013-9253-2
|
| [50] |
LIU HY, DAI CQ, FAN YL, GUO BL, REN KK, SUN TN, WANG WT. From autophagy to mitophagy: the roles of P62 in neurodegenerative diseases. Journal of Bioenergetics and Biomembranes, 2017, 49(5): 413-422. DOI:10.1007/s10863-017-9727-7
|
| [51] |
FOREST KH, NICHOLS RA. Assessing neuroprotective agents for aβ-induced neurotoxicity. Trends in Molecular Medicine, 2019, 25(8): 685-695. DOI:10.1016/j.molmed.2019.05.013
|
| [52] |
陈易斯, 宋福永. 选择性自噬接头蛋白p62/sequestosome 1的研究进展. 中国药理学与毒理学杂志, 2016, 30(3): 258-265. CHEN YS, SONG FY. Research advances in selective adaptor protein autophagy of p62/sequestosome-1. Chinese Journal of Pharmacology and Toxicology, 2016, 30(3): 258-265 (in Chinese). DOI:10.3867/j.issn.1000-3002.2016.03.012 |
| [53] |
RAMESH BABU J, LAMAR SEIBENHENER M, PENG JM, STROM AL, KEMPPAINEN R, COX N, ZHU HN, WOOTEN MC, DIAZ-MECO MT, MOSCAT J, WOOTEN MW. Genetic inactivation of p62 leads to accumulation of hyperphosphorylated tau and neurodegeneration. Journal of Neurochemistry, 2008, 106(1): 107-120. DOI:10.1111/j.1471-4159.2008.05340.x
|
| [54] |
MATSUMOTO G, SHIMOGORI T, HATTORI N, NUKINA N. TBK1 controls autophagosomal engulfment of polyubiquitinated mitochondria through p62/SQSTM1 phosphorylation. Human Molecular Genetics, 2015, 24(15): 4429-4442. DOI:10.1093/hmg/ddv179
|
| [55] |
TANJI K, MIKI Y, OZAKI T, MARUYAMA A, YOSHIDA H, MIMURA J, MATSUMIYA T, MORI F, IMAIZUMI T, ITOH K, KAKITA A, TAKAHASHI H, WAKABAYASHI K. Phosphorylation of serine 349 of p62 in Alzheimer's disease brain. Acta Neuropathologica Communications, 2014, 2(1): 1-14. DOI:10.1186/2051-5960-2-1
|
| [56] |
LEE S, JEON YM, CHA SJ, KIM S, KWON Y, JO M, JANG YN, LEE S, KIM J, KIM SR, LEE KJ, LEE SB, KIM K, KIM HJ. PTK2/FAK regulates UPS impairment via SQSTM1/p62 phosphorylation in TARDBP/TDP-43 proteinopathies. Autophagy, 2020, 16(8): 1396-1412. DOI:10.1080/15548627.2019.1686729
|
| [57] |
ALI SHAH SZ, ZHAO DM, HUSSAIN T, SABIR N, MANGI MH, YANG LF. p62-Keap1-NRF2-ARE pathway: a contentious player for selective targeting of autophagy, oxidative stress and mitochondrial dysfunction in prion diseases. Frontiers in Molecular Neuroscience, 2018, 11: 310. DOI:10.3389/fnmol.2018.00310
|
| [58] |
ZHANG WW, FENG C, JIANG H. Novel target for treating Alzheimer's diseases: crosstalk between the Nrf2 pathway and autophagy. Ageing Research Reviews, 2021, 65: 101207. DOI:10.1016/j.arr.2020.101207
|
| [59] |
JEMAL A, WARD EM, JOHNSON CJ, CRONIN KA, MA JM, RYERSON AB, MARIOTTO A, LAKE AJ, WILSON R, SHERMAN RL, ANDERSON RN, HENLEY SJ, KOHLER BA, PENBERTHY L, FEUER EJ, WEIR HK. Annual report to the nation on the status of cancer, 1975-2014, featuring survival. JNCI: Journal of the National Cancer Institute, 2017, 109(9): djx030.
|
| [60] |
VILLANUEVA A. Hepatocellular carcinoma. New England Journal of Medicine, 2019, 380(15): 1450-1462. DOI:10.1056/NEJMra1713263
|
| [61] |
INAMI Y, WAGURI S, SAKAMOTO A, KOUNO T, NAKADA K, HINO O, WATANABE S, ANDO J, IWADATE M, YAMAMOTO M, LEE MS, TANAKA K, KOMATSU M. Persistent activation of Nrf2 through p62 in hepatocellular carcinoma cells. Journal of Cell Biology, 2011, 193(2): 275-284. DOI:10.1083/jcb.201102031
|
| [62] |
UMEMURA A, HE F, TANIGUCHI K, NAKAGAWA H, YAMACHIKA S, FONT-BURGADA J, ZHONG ZY, SUBRAMANIAM S, RAGHUNANDAN S, DURAN A, LINARES JF, REINA-CAMPOS M, UMEMURA S, VALASEK MA, SEKI E, YAMAGUCHI K, KOIKE K, ITOH Y, DIAZ-MECO MT, MOSCAT J, et al. p62, upregulated during preneoplasia, induces hepatocellular carcinogenesis by maintaining survival of stressed HCC-initiating cells. Cancer Cell, 2016, 29(6): 935-948. DOI:10.1016/j.ccell.2016.04.006
|
| [63] |
TANIGUCHI K, YAMACHIKA S, HE F, KARIN M. p62/SQSTM1-Dr. Jekyll and Mr. Hyde that prevents oxidative stress but promotes liver cancer. FEBS Letters, 2016, 590(15): 2375-2397. DOI:10.1002/1873-3468.12301
|
| [64] |
MENON S, YECIES JL, ZHANG HH, HOWELL JJ, NICHOLATOS J, HARPUTLUGIL E, BRONSON RT, KWIATKOWSKI DJ, MANNING BD. Chronic activation of mTOR complex 1 is sufficient to cause hepatocellular carcinoma in mice. Science Signaling, 2012, 5(217): e2002739.
|
| [65] |
QU AJ, JIANG CT, CAI Y, KIM JH, TANAKA N, WARD JM, SHAH YM, GONZALEZ FJ. Role of myc in hepatocellular proliferation and hepatocarcinogenesis. Journal of Hepatology, 2014, 60(2): 331-338. DOI:10.1016/j.jhep.2013.09.024
|
| [66] |
DURAN A, HERNANDEZ ED, REINA-CAMPOS M, CASTILLA EA, SUBRAMANIAM S, RAGHUNANDAN S, ROBERTS LR, KISSELEVA T, KARIN M, DIAZ-MECO MT, MOSCAT J. p62/SQSTM1 by binding to vitamin D receptor inhibits hepatic stellate cell activity, fibrosis, and liver cancer. Cancer Cell, 2016, 30(4): 595-609. DOI:10.1016/j.ccell.2016.09.004
|
| [67] |
FAN LH, YIN ST, ZHANG EX, HU HB. Role of p62 in the regulation of cell death induction. Apoptosis, 2018, 23(3/4): 187-193.
|
| [68] |
SÁNCHEZ-MARTÍN P, KOMATSU M. p62/SQSTM1-steering the cell through health and disease. Journal of Cell Science, 2018, 131(21): jcs222836. DOI:10.1242/jcs.222836
|
| [69] |
LI LJ, SHEN C, NAKAMURA E, ANDO K, SIGNORETTI S, BEROUKHIM R, COWLEY GS, LIZOTTE P, LIBERZON E, BAIR S, ROOT DE, TAMAYO P, TSHERNIAK A, CHENG SC, TABAK B, JACOBSEN A, HAKIMI AA, SCHULTZ N, CIRIELLO G, SANDER C, et al. SQSTM1 is a pathogenic target of 5q copy number gains in kidney cancer. Cancer Cell, 2013, 24(6): 738-750. DOI:10.1016/j.ccr.2013.10.025
|
| [70] |
REN F, SHU GS, LIU GL, LIU DC, ZHOU JP, YUAN LW, ZHOU JP. Knockdown of p62/sequestosome 1 attenuates autophagy and inhibits colorectal cancer cell growth. Molecular and Cellular Biochemistry, 2014, 385(1/2): 95-102.
|
| [71] |
HARADA H, WARABI E, MATSUKI T, YANAGAWA T, OKADA K, UWAYAMA J, IKEDA A, NAKASO K, KIRII K, NOGUCHI N, BUKAWA H, SIOW RCM, MANN GE, SHODA J, ISHII T, SAKURAI T. Deficiency of p62/sequestosome 1 causes hyperphagia due to leptin resistance in the brain. Journal of Neuroscience, 2013, 33(37): 14767-14777. DOI:10.1523/JNEUROSCI.2954-12.2013
|
| [72] |
HUANG JF, DURAN A, REINA-CAMPOS M, VALENCIA T, CASTILLA EA, MÜLLER TD, TSCHÖP MH, MOSCAT J, DIAZ-MECO MT. Adipocyte p62/SQSTM1 suppresses tumorigenesis through opposite regulations of metabolism in adipose tissue and tumor. Cancer Cell, 2018, 33(4): 770-784.e6. DOI:10.1016/j.ccell.2018.03.001
|
| [73] |
MOSCAT J, KARIN M, DIAZ-MECO MT. p62 in cancer: signaling adaptor beyond autophagy. Cell, 2016, 167(3): 606-609. DOI:10.1016/j.cell.2016.09.030
|
| [74] |
CHO CS, PARK HW, HO A, SEMPLE IA, KIM B, JANG I, PARK H, REILLY S, SALTIEL AR, LEE JH. Lipotoxicity induces hepatic protein inclusions through TANK binding kinase 1-mediated p62/sequestosome 1 phosphorylation. Hepatology, 2018, 68(4): 1331-1346. DOI:10.1002/hep.29742
|
| [75] |
MARENGO A, ROSSO C, BUGIANESI E. Liver cancer: connections with obesity, fatty liver, and cirrhosis. Annual Review of Medicine, 2016, 67: 103-117. DOI:10.1146/annurev-med-090514-013832
|
| [76] |
SÁNCHEZ-MARTÍN P, SAITO T, KOMATSU M. p62/SQSTM 1: 'Jack of all trades' in health and cancer. The FEBS Journal, 2019, 286(1): 8-23. DOI:10.1111/febs.14712
|
| [77] |
BÉKÉS M, LANGLEY DR, CREWS CM. PROTAC targeted protein degraders: the past is prologue. Nature Reviews Drug Discovery, 2022, 21(3): 181-200. DOI:10.1038/s41573-021-00371-6
|
| [78] |
JI CH, KIM HY, LEE MIN JU, HEO AJ, PARK DY, LIM S, SHIN S, GANIPISETTI S, YANG WS, JUNG CA, KIM KY, JEONG EH, PARK SH, BIN KIM S, LEE SU JIN, NA JE, KANG JI, CHI HM, KIM HT, KIM YK, et al. Author correction: the AUTOTAC chemical biology platform for targeted protein degradation via the autophagy-lysosome system. Nature Communications, 2022, 13: 2108. DOI:10.1038/s41467-022-29845-w
|
 2023, Vol. 39
2023, Vol. 39