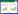中国科学院微生物研究所、中国微生物学会主办
文章信息
- 池燕飞, 李春, 冯旭东
- CHI Yanfei, LI Chun, FENG Xudong
- 机器学习在蛋白质功能预测领域的研究进展
- Advances in machine learning for predicting protein functions
- 生物工程学报, 2023, 39(6): 2141-2157
- Chinese Journal of Biotechnology, 2023, 39(6): 2141-2157
- 10.13345/j.cjb.221002
-
文章历史
- Received: December 14, 2022
- Accepted: April 4, 2023
- Published: April 20, 2023
2. 清华大学化学工程系 工业生物催化教育部重点实验室, 北京 100084
2. Key Laboratory for Industrial Biocatalysis, Ministry of Education, Department of Chemical Engineering, Tsinghua University, Beijing 100084, China
蛋白质是各种生命活动中不可或缺的物质基础之一,其生物学功能具有多样性,在各种生命活动中均发挥重要作用。绝大多数酶也属于蛋白质,其在生物制造领域扮演着重要角色[1]。研究蛋白质功能能够辅助各种生命活动的分子机制研究,有助于医学和药物研发领域的快速发展[2-3]。目前,主要通过生化实验来表征蛋白质功能,但是该方法耗时耗力、成本高昂。生物信息技术日新月异,飞速发展,数以亿计的蛋白质序列不断被发现,测序得到的蛋白序列数量远大于功能已知的蛋白序列数量,仅通过实验验证来进行蛋白质功能注释已无法满足当前的需求[4-5]。
随着计算机科学的不断发展,特别是当前处于人工智能进入高速发展的时代,借助计算机手段,高效且准确地预测并注释蛋白质功能,并为生化实验提供指导,已成为目前的研究热点。为此,本文旨在总结人工智能辅助蛋白质功能预测领域的研究进展,主要介绍现有的蛋白质功能注释方法和机器学习相关概念,重点介绍了机器学习方法在酶功能预测领域的实际应用。
1 蛋白质功能注释蛋白质是构成生命体的重要部分,是结构具有复杂性以及功能具有多样性的生物大分子,对各类生命活动均有重要意义。蛋白质参与重要的生理过程,如细胞信号传导、催化反应、调节免疫以及物质转运等,也为生命体的新陈代谢提供了能量[6]。对于蛋白质功能的探索能够辅助生命活动分子机制的研究[7],对医学领域的研究以及药物开发等都起到了重要作用[2-3]。蛋白质功能通常侧重于单个蛋白质分子的作用,如针对给定反应的催化或分子的结合,这种局部功能也被称为蛋白质的分子功能。另一方面,蛋白质也被定义为其相互作用网络中的元素,即蛋白质会在其相互作用分子的扩展网络中发挥作用,这样的情况被称为上下文功能或细胞功能[8-9]。
当今测序技术和基因组学快速发展,蛋白质被测定的序列越来越多,从蛋白质序列到其功能的准确注释能够帮助人们拓宽对自然界生命体及其生命活动的认知,对药物研究、医学以及生物绿色合成领域的发展都有极大的促进作用。目前,UniProt数据库所收录的蛋白质序列已达2亿多条[10],而通过实验进行了功能注释的蛋白质序列仅有近1%[11]。例如,UDP-糖基转移酶(uridine diphosphate glycosyltransferase, UGT)是常见的一类糖基转移酶,属于GT1家族蛋白,目前GT1成员已超过3万个,但已通过实验确定功能的UDP-糖基转移酶仅有413个[12]。此外,在目前已获得结构解析的蛋白质中,仍未获得功能注释的蛋白质还有30%以上[9]。因此,蛋白质功能的预测是当前的一个重要研究方向。
目前,主要通过生化实验对蛋白质功能进行表征及注释,但是该方法成本高昂且实验周期长,功能获得注释的蛋白与未知功能蛋白之间的数量差距越来越大。随着人工智能的高速发展,已有许多计算方法被应用于解决蛋白质功能预测的问题,相比耗时耗力的实验验证方法,通过计算机手段可以一次性对大量的蛋白质同时进行功能预测。通过人工智能对蛋白质的功能进行预测并注释是目前的研究热点之一,高效且精准地预测各类蛋白质功能可以大大推动药物靶点发现、生物活动机制以及生物合成领域的研究发展。
2 传统蛋白质功能预测方法 2.1 基于序列的方法基于序列的方法主要是从进化角度进行,蛋白质的结构决定其功能,而结构又由氨基酸序列所决定,同源性蛋白的序列具有一定的相似性,从而具有相似的功能[13]。因此,基于序列的方法主要通过序列之间的相似性比对来确定蛋白质之间的同源性,目前应用最为广泛的比对工具是BLAST[14]和HMMER[15]。BLAST主要是根据目标蛋白与功能已知蛋白的比对结果相似性高低来推定功能,是针对各类蛋白质数据库或基因数据库之间进行相似性比较的分析工具,主要由尼德曼-翁施(Needleman-Wunsch)算法和史密斯-沃特曼(Smith-Waterman)算法组成[14]。HMMER主要使用隐马尔科夫模型来对目标蛋白进行注释,相比于BLAST,其在远距离蛋白注释方面具有更高的准确性[16]。对于多个基因组或多个蛋白序列的功能预测,在经过HMMER注释后还可利用ClustalW[17]等工具进行多序列比对,通过对序列构建系统进化树来挖掘序列之间的进化关系,从而推断出序列同源性及功能相似性。目前,基于序列的方法已被广泛应用于蛋白质功能注释领域,且在许多研究中已获得了实际应用。
此外,蛋白质通常至少由一个保守的结构域组成,在结构域内较短的且保守的片段被称为基序,基序通常具有特定的生物学功能关联。基于基序的方法已成功应用于蛋白质识别和分类。例如,植物来源UDP-糖基转移酶氨基酸序列的C末端有一段由44个氨基酸组成的保守序列,该段序列为糖基转移酶与糖供体相识别的结合位点,称为植物次级代谢产物糖基转移酶序列(plant secondary product glycosyltransferase motif, PSPG box)保守区[18],在植物基因组中挖掘筛选UDP-糖基转移酶时,被视为关键性特征序列。
然而,基于序列同源性的方法来推断蛋白质功能,通常认为在序列之间有大于60%的相似度时,该方法才能达到一定的可信度[14]。此外,同源性与蛋白质功能之间并没有绝对的相关性,2条序列具有同源性只能说明其来自同一祖先,但在功能上并非绝对相似[19]。例如,目前已获得功能注释的酶序列相似性与其底物特异性之间没有呈现出明显的相关性,这很不利于酶的直接功能预测及应用。例如,UDP-糖基转移酶主要以尿苷二磷酸-糖(uridine diphosphate-糖,UDP-糖)作为糖基供体,来自葡萄的UDP-糖基转移酶VvGT5和VvGT6蛋白序列具有91%的相似性,但VvGT6以UDP-Glc和UDP-Gal为糖供体,而VvGT5仅能以UDP-GlcA为供体[20]。来自乌拉尔甘草的UGT73P12与来自大豆GmSGT2 (UGT73P2)序列相似性为75%,但GmSGT2以UDP-Gal为糖供体,将半乳糖基转移到大豆皂苷B-3-O-单葡萄糖醛酸苷以合成大豆皂苷Ⅲ,而UGT73P12将UDP-GlcA的葡萄糖醛酸基转移到甘草次酸3-O-单葡萄糖醛酸以合成甘草酸[21]。同样,低相似度序列也可能会有相似功能,如来自光果甘草的GuGT33 (UGT84F6)和GuGT37 (UGT71S4)具有相似的底物特异性,然而它们的蛋白序列相似度却只有23.7%,并且属于不同的UGT家族[22-23]。以上结果说明无法仅根据蛋白质序列相似度来准确确定UDP-糖基转移酶的生化功能特征,基于同源性获得候选UDP-糖基转移酶的系统进化分析后,仍然需要大量的实验验证才能最终确定酶的功能特征信息。因此,基于序列同源性的方法虽然已被广泛使用,但是其需要解决的问题依然很多。
2.2 基于结构的方法根据蛋白质结构能够直接决定其生物功能的理论基础,具有相似的空间结构的蛋白质往往拥有相同的功能[13]。基于结构的蛋白质功能预测方法主要通过两种方式来实现[16]:蛋白结构全局折叠相似比较和局部活性位点特征描述。蛋白质三维的空间结构相比于一维的氨基酸序列在进化上具有更强的保守性[24]。目前,大多数研究主要依据蛋白质局部状态,如三维结构上的某一特定结合区域在结构上的相似性来推断未知蛋白的功能。例如,UDP-糖基转移酶的结合位点就是酶C端和N端结构域之间的狭长口袋[25]。酶的功能几乎完全由蛋白质结构上的这些活性位点来决定,因为在进化过程中,蛋白酶活性位点周围残基能够始终保持高度保守[13]。目前,已经提出了许多将未知蛋白与蛋白质数据库(protein data bank, PDB)[26]的结构进行比对来预测蛋白功能的工具。例如,蛋白质结构比较在线网站FATCAT[27] (www.fatcat.burnham.org),具有良好性能的结构比对工具DeepAlign[28]和蛋白质结构分类数据库CATH- Gene3D[29] (www.cathdb.info),其中,FATCAT能够将目标蛋白与PDB数据库中的结构进行相似性比较及结构的叠加;DeepAlign除了对蛋白之间的刚性结构进行比较,还能够考虑到进化关系和氢键相似性等因素对蛋白之间结构差异的影响;CATH能够对PDB数据库中的蛋白质根据具体的空间结构信息进行分类。虽然基于蛋白质结构进行功能预测已经取得了较为丰富的研究成果,但是目前仍然存在较多不足,该方法需要大量已知功能的蛋白结构数据进行建模,对模型的准确性依赖程度高[13]。然而,蛋白质结构解析一直是生命科学领域一个复杂且艰巨的科学任务,目前已获得结构解析的蛋白质数据量还不能够构建一个足够准确的模型,例如,对于UDP-糖基转移酶来说,目前已获得晶体结构解析的UDP-糖基转移酶只有51个[12, 30-31]。虽然随着蛋白结构解析技术的不断发展,获得结构解析的蛋白数量越来越多,使得基于结构预测蛋白质功能的方法逐渐可靠,但这仍然存在一个漫长的时间问题。
3 基于机器学习的蛋白质功能预测方法智慧是与人类相关的一项特征,学习是人类最为显著的智能行为。机器学习作为人工智能的一个重要分支,它综合了统计学、概率论等多种学科理论,其主要目的是让机器学习并模仿人类,从而具有人类的思维方式,自动调节输入的各因素权重,综合学习并快速输出结果。深度学习(deep learning)是机器学习的子领域,以其为代表的机器学习是目前最接近人脑智能的学习和认知方法,在语音和图片的识别与转换方面已取得了显著进展,在各领域中均有着巨大的应用及商业潜力。
机器学习最初是建立在人工神经网络的研究基础上。1989年,Lecun等[32]提出了首个被成功训练的人工神经网络计算模型:卷积神经网络(convolutional neural networks, CNN)计算模型,CNN是目前计算机技术在各领域中均被广泛使用的计算模型。CNN的基本结构(图 1)主要由输入层、卷积层(激活函数)、池化层、全连接层以及输出层组成。卷积层是CNN特有的结构,其作用是对输入数据进行卷积操作,提取输入的特征信息等。激活函数是非线性函数,具有连续可微、单调性等特性,目前最常用的激活函数有Relu、Tanh和Sigmoid。池化层作用于连续的卷积层之间,对数据进行降维处理以防止数据和参数的量过大。全连接层出现在网络的最后层,主要作用为连接所有数据矩阵,把所有局部特征结合变成全局特征,最终输出数据。
|
| 图 1 CNN基本结构图 Fig. 1 The infrastructure of CNN. |
| |
20世纪末,由于暴发式增长的数据量、不断提升及优化的计算机技术和算法,人工智能热潮正式来袭[33],至此,以机器学习为代表的人工智能技术开始活跃于人类日常生活中,比如游戏、人脸识别、语音识别、自动驾驶以及医学影像技术分析等领域[34]。目前,机器学习在学术和工业领域均取得了丰富的研究和应用成果,促进了科学研究和实际应用的快速发展,人工智能已然成为了目前最热门的研究方向。如DeepMind公司在开发出了打败人类世界冠军的深度学习围棋程序AlphaGo后,开始进军生命科学领域,开发了一个基于深度神经网络算法的蛋白三维结构预测程序AlphaFold,该程序通过输入未知蛋白序列即可对该蛋白的三维结构进行预测;2020年12月,改进推出的AlphaFold2[35-36]通过与核磁共振、X射线晶体和冷冻电镜等成熟的实验技术相比较,程序输出准确率几乎达到了实验精度,输出结果与实验所测结果仅相差一个原子。随着生物信息的高速发展,生物数据逐渐积累,生命科学领域越来越多的研究者使用机器学习来辅助研究,从而促进了该学科更快更高效地发展。目前,机器学习在生命科学领域除了应用于蛋白未知结构和功能的预测,还可用于基因组学研究和药物研发等领域,以辅助药物靶点的研究[37]以及药物毒性的预测等[38];在医疗影像学中,机器学习还可基于大数据用于病灶的识别以辅助诊断[39]。
目前,机器学习辅助生命科学领域已取得了丰硕的研究成果与实际应用,随着计算机技术的不断发展,未来它也必将促进该领域更快更高效地发展。
3.1 机器学习的分类及算法目前,蛋白质功能预测主要是一个分类的问题,将序列和其特征信息作为输入映射到一个离散的功能信息输出上,可以使用的机器学习算法分为监督学习和强化学习两类。监督学习又根据数据的标签(label)存在与否主要分为3种:传统监督学习(traditional supervised learning)、无监督学习(unsupervised learning)和半监督学习(semi-supervised learning)。
传统监督学习的数据集主要包含初始训练数据和人为标注整理训练目标,这一步在整个工作流程中最为耗时但也最为重要,因为研究者们希望计算机能够通过特定算法根据标注的特征从训练数据中总结出具体对象的划分规律,然后再将这个规律应用到测试数据的预测中,最终输出有具体标签的学习结果。传统监督学习有支持向量机(support vector machine, SVM)、人工神经网络(neural networks)和深度神经网络(deep neural networks)等经典算法;主要有回归分析和任务分类等应用。无监督学习,主要应用于训练数据未被人工分类标记的情况,通过计算机算法根据相似原理自主学习总结数据之间的规律,最终获得训练数据的特征。无监督学习的典型算法有聚类(clustering)、EM算法(expectation-maximization algorithm)和主成分分析(principle component analysis)等;典型应用有:聚类和异常检测等。半监督学习即介于以上两者之间,训练数据中仅一部分数据有标签。目前,互联网生活中使用到的绝大多数软件使用的算法便是半监督学习。半监督学习主要在有标记样本数量很少的情况下,通过在模型训练中引入无标记样本来避免传统监督学习在训练样本不足时出现模型性能变弱的问题。在实际应用中,有标记样本的获取成本往往较高,而无标记样本只需重复采集即可大量收集。因此,在实际应用中当有标记样本量过少时,半监督学习算法能够获得与通过大量标记样本训练的传统监督学习相近甚至更好的效果,大大提升了生产效率。在过去几十年里,越来越多的机器学习算法被不停地更新及开发出来并应用于各个领域。
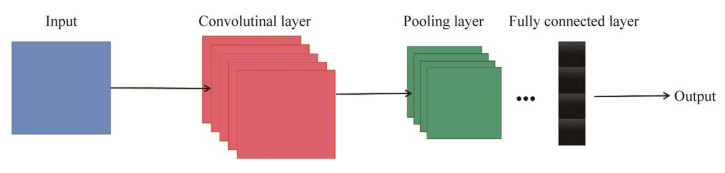
然而,机器学习实际应用过程中也存在着相应的困难(图 2),如由于特征样本量较少,模型训练中的过拟合(over fitting)与欠拟合(under fitting)[40]是机器学习方法在蛋白质功能预测领域需要面对的挑战之一。欠拟合是指由于模型使用的参数过少,得到的模型难以拟合训练数据,在训练集、验证集和测试集上均表现出性能不高的情况。过拟合则是由于使用的参数过多从而导致模型对训练数据的过度拟合,即模型在训练集上表现出很好的性能,但在验证和测试阶段就表现出性能不佳的情况。模型的构建通常需要根据实际的目的来选择并尝试多种算法,因此在蛋白质功能预测领域,目前还缺乏一种具有普适性的机器学习模型。
|
| 图 2 机器学习中容易出现的问题 Fig. 2 The problems in machine learning. |
| |
机器学习算法依赖于大量的数据,因此用于训练的数据集的数量和质量至关重要。蛋白工程中最经常使用的数据库(表 1)为InterPro[41]、UniProtKB[42]和PDB[26]。UniProtKB是最大的序列数据库,由SwissProt和TrEMBL数据集组成。SwissProt是手工注释的且非冗余的数据,这些数据的质量有一定的保证;TrEMBL包含一些仍需要手工注释的功能未知蛋白质序列。InterPro整合了多个蛋白相关的数据库,包含了38 000多条数据,该数据库具有一个蛋白序列功能注释工具interproscan,UniProtKB主要利用了该工具对未知蛋白序列进行注释。PDB收录了近20万个高分辨率蛋白三维结构信息,是最大的蛋白3D结构数据库。基因本体术语(gene ontology, GO)[43]能够系统地对物种基因及其产物属性进行注释,目前已有不少研究将GO术语数据库预测应用到蛋白质功能预测中。
| Database | Category | Website | Number |
| InterPro | Protein sequence database | www.ebi.ac.uk/interpro | More than 30 000 sequences |
| UniProtKB | Protein family and structural domain database | www.uniprot.org | More than 200 million sequences |
| PDB | Protein structure database | www.rcsb.org | Approximately 200 000 protein structures |
| Gene ontology | Gene database | www.geneontology.org | Approximately 1.5 million genes |
上述数据库中数据的数量和质量对于建立机器学习预测模型是至关重要的,其中,数量最丰富的是蛋白质序列数据库,其次是蛋白质结构数据库。然而,在蛋白功能预测实际研究中,希望氨基酸序列能够直接反映该蛋白的功能,因此,对于蛋白序列,可以从氨基酸的物理和化学性质出发去整理数据集。此外,在蛋白酶功能预测方面,由于酶反应类型、反应机制、辅因子和条件复杂多样,预测其具体功能特性是一项极具挑战的任务,目前仍然需要构建更多的数据库以应对这一挑战。
3.3 蛋白质功能特征的选择建立机器学习模型的一个关键步骤是识别合适的特征,由于蛋白质功能丰富多样,使用机器学习技术预测蛋白质功能需要根据具体需求确定合适的特征标签输入。功能相近的蛋白在序列和结构上通常表现出相似的物理化学性质,例如等电点、分子量、表面张力、极性、疏水性和电荷数等蛋白质物理和化学性质,此外还有氨基酸特征、配体性质以及结构可变性等生物学性质[19],机器学习方法主要利用计算机去捕获蛋白质的这些特征信息与功能之间的关系。氨基酸序列特征包括氨基酸的组成、分布及保守情况等[19],除了对功能预测有重要影响,其在蛋白质相互作用网络的预测中也早有应用[44]。序列特征中最显著的是序列基序,存在于绝大多数类蛋白中。此外,蛋白质-蛋白质相互作用(protein-protein interactions, PPI)网络是蛋白质之间物理接触的数学表示[9],在解析蛋白质功能之间的具体联系中有重要作用,被广泛作为特征应用于蛋白功能预测领域。
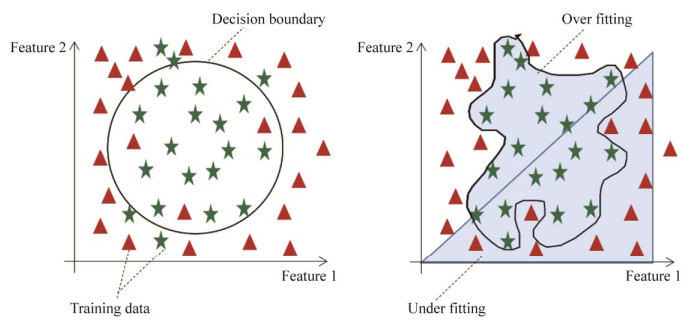
类似于同源性比对的方法,机器学习方法预测蛋白质功能也需要收集大量已知功能蛋白质数据,通过算法学习蛋白质特征与功能之间的关系并建立计算机模型,最终通过拟合的模型预测目标蛋白的具体功能(图 3)。但是,基于蛋白质序列同源性和结构的方法,都存在依赖序列相似性或结构相似性结果的局限性,而基于机器学习的方法能够利用计算机从序列、结构和蛋白网络等不同角度出发提取蛋白质特征,并映射到对应的功能上,从而改善基于序列同源性等传统方法存在的问题。比如蛋白质家族数据库Pfam在过去几年中增长了5%的蛋白质序列,但是至少有三分之一的蛋白质无法通过与已获得功能表征的序列直接进行比对来进行功能注释[7, 45],而Bileschi等[46]使用深度学习模型(ProtCNN)对来自Pfam数据库中17 929个家族未经过比对对齐的蛋白质序列进行了功能预测,相比于现有传统方法,通过对测试序列进行分类结果显示出了更高的准确性,此外,他们还将该模型和传统比对方法如BLAST和HMMER结合,对于与训练数据相似度较低的序列也进行了准确分类,表明将机器深度学习与现有方法互补可以有效提高预测效率以及改善模型性能。机器学习模型能够一次性预测大量未知功能的蛋白质序列,不仅预测效果相比于传统的比对方法获得了提升,预测速度也大大提高,节约了时间。例如,Lv等[16]从蛋白质序列和蛋白质相互作用网络中提取特征,应用深度神经网络模型对人类蛋白质功能进行预测,准确率相比传统BLAST方法高出51.3%。此外,BLAST方法还需要未知蛋白的生物背景基础,而机器学习可以在无物理或生物学等知识背景情况下,推断未知蛋白质序列的生物学特性并预测其功能。
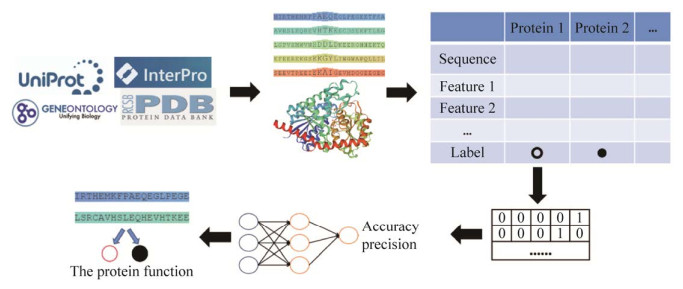
|
| 图 3 机器学习预测蛋白质功能流程图 Fig. 3 The flow chart of machine learning to predict protein function. |
| |
目前,基于机器学习对蛋白质功能进行预测主要通过关键残基的种类、理化特性以及保守程度分析等[19]手段来进行。例如,Corral-corral等[47]通过多种机器学习算法和不同的蛋白质特征表示方法来探究关键残基、蛋白质结构与蛋白功能之间的关系,最终结果表明以关键残基的物理化学性质作为特征时,机器学习模型的准确率最高。Gado等[48]利用loop中的残基数量和氨基酸类型作为训练特征,使用机器学习方法准确区分1 748个家族7糖苷水解酶的CBH和EG两种亚型。2018年发布的GT-predict[49]选择来自拟南芥的糖基转移酶超家族1的54种酶与91种底物用于功能预测,作者基于决策树算法,系统地组合了序列与底物分子物理化学性质,包括clogP、分子面积和亲核基团的数量/类型以及结构信息,在其他4种已获得功能表征的植物和微生物GT1中,模型准确率达到了69%以上。GT-predict凸显了数据特征对提高模型准确率的重要性,但是该预测工具是通过与功能特征已知的糖基转移酶进行序列比对后直接输出功能信息,没有考虑化学区域选择性的问题,仅根据基团存在与否或是数据高低与否来进行决策,存在过度拟合的问题。因此,机器学习方法对于数据的质量具有很高的依赖性,预测模型的准确性还需要进一步提升。此外,虽然机器学习在蛋白质功能预测等研究领域已有一定的研究成果及应用潜力,但根据“没有免费午餐定理”,目前还缺乏一种在所有任务中都是最优的算法[50]。如何快速获得更准确、更适用于机器学习算法的训练数据,寻找并建立更为高效的数据特征表示方式,从而提高模型准确率,是目前机器学习在各领域应用中迫切需要解决的一个问题。
4 机器学习在蛋白质功能预测中的应用进展随着测序技术的发展,测序得到的蛋白质数量和功能已知的蛋白质数量之间的差距日益增大,为解决该问题,蛋白质功能注释算法(critical assessment of functional annotation, CAFA)全球挑战[51]提出了一种时滞性能评估方法,将来自于已发表的文章或数据库中已注释的蛋白质序列分别作为训练集和测试集,该方法可以提高功能预测模型算法的可靠性。当前大部分蛋白质功能预测的算法,包括基于序列比对的方法和基于机器学习的方法,都参照了CAFA的延时评估。目前,计算机和生物化工领域均处于蓬勃发展时期,如何将二者有效结合,利用计算机手段辅助生物化工领域开展研究工作也一直是研究热点之一,例如,生物酶法催化化学反应虽然具有绿色环保和专一性强等优点,但是特定功能酶的挖掘和功能鉴定的过程成本高昂,若能使用计算机方法辅助酶的高效挖掘和指导生化实验的开展必将事半功倍,也能有效解决未知功能蛋白序列数量与已获得功能注释的蛋白序列数量之间的巨大差距问题。
4.1 传统机器学习方法针对不同的数据集,机器学习预测蛋白质功能主要利用蛋白质序列和结构等数据作为输入并生成特征,通过利用不同的算法来完成模型的构建和优化,如KNN (k nearest neighbors)算法[52]、朴素贝叶斯(naive bayes, NB)[53-54]、SVM[55]和神经网络[56]等。只从序列提取特征是最简单的蛋白质功能类别分类模型[57],虽然蛋白质结构包含的信息比蛋白质序列更丰富,但是从目前的研究结果来看,从氨基酸序列直接预测蛋白质功能也可获得良好的准确率[58]。如表 2所示,Che等[59]从SwissProt数据库收集并去除高度相似序列后最终获得了59 764个蛋白序列组成的数据集,通过ACC控制算法提取特征后采用KNN算法构建模型用于酶功能的预测,结果表明该方法对于单功能酶分类准确率为94.1%,多功能酶分类准确率达到91.25%,并且将模型制作为了在线预测网站,大大提高了模型的实用性。Osman等[60]选择了PDB数据库中6个超家族的3 200种酶作为了数据集,采用混合GA-BP算法的神经网络模型对数据集中的2 000种酶进行了测试,最终该分类模型获得了72.94%的平均准确率。Mohammed等[61]从EXPASY酶数据库和SwissProt数据库分别收集了64 948个酶序列和128 292个非酶蛋白序列,利用Pfam、Superfamily和Prosite三个结构域数据库分别对这些序列的功能、结构和基序或活性位点区域进行特征提取,通过5种不同的机器学习算法(KNN、SVM、决策树、随机森林和朴素贝叶斯)进行模型训练,所获得模型ECemble的准确率在97%至99%之间,优于传统的BLAST和同类的开源方法EFICAz[71]。此外,作者还将ECemble应用于肠道宏基因组并从中预测了人类肠道微生物组编码的酶,有助于了解微生物编码酶在人类代谢系统健康功能中所起的作用。
| Algorithm | Database | Number of protein sequences/Structures | Accuracy (%) | References |
| ACC+KNN | SwissProt | 59 764 | 91.3 | [59] |
| GA-BP | PDB | 3 200 | 72.9 | [60] |
| KNN, SVM, DT, RF, NB | EXPASY and SwissProt | 193 240 | 97.0–99.0 | [61] |
| SVM | PDB | 492 | 35.0 | [62] |
| NB | PDB | 492 | 45.0 | [63] |
| SVM+KNN | PDB | 39 251 | 93.4 | [64] |
| SVM+KNN | PDB | 40 034 | 95.5 | [65] |
| RF | UniProtKB and SwissProt | 1 121 | 98.0 | [66] |
| SVM | PDB | 4 000 | 88.5 | [67] |
| CNN | SwissProt | 22 168 | 94.2 | [68] |
| CNN | SwissProt | 22 168 | 96.7 | [69] |
| PSSM+CNN | UniProt | 3 265 | 89.7 | [70] |
从氨基酸序列特征来预测蛋白质功能主要是基于同源性的原则,但是当一个蛋白没有功能已获得注释的相似蛋白序列时,仅仅利用序列来预测功能的方法就失效了。因此,2005年由Dobson和Doig[62]提出了从结构信息出发来预测蛋白质功能的方法,他们根据EC编号选择了6个酶分类中的492个蛋白结构数据,以二级结构含量和表面性质等简单晶体结构属性作为特征信息输入,最终获得的SVM模型准确率为35%。2006年,Borro等[63]在Dobson和Doig的研究基础上,使用了贝叶斯算法将准确率提高到了45%。2016年,Amidi等[64]将结构信息和氨基酸序列比对融合,使用SVM和KNN算法对来源于PDB数据库中39 251个蛋白质数据集进行训练,分类准确率达到了93.4%。2017年,Amidi等[65]结合了蛋白结构和氨基酸序列信息,通过SVM和最近邻算法构建了多酶功能预测模型,对PDB数据库中40 034种酶的EC编号第一个数字预测的准确率达到95.5%,这一结果也说明了在酶功能预测方面,蛋白质信息的组合可以提供更为准确的预测结果。
只以不同数据来源的序列或结构作为输入信息,模型输入信息过于单一,准确性不足,适用性也受到一定限制。在酶数据库中,通常会为酶序列添加功能注释标签以便分类和查阅,但是由于酶的种类和催化功能多样,利用机器学习对蛋白酶功能进行预测不是简单的单一标签分类问题。2014年,Nagao等[66]首次应用随机森林算法来预测每个同源超家族酶中EC编号的第4位数字,除了全长序列相似性外,还以活性位点残基、配体结合残基和保守残基之间的相似性作为输入特性,对同源超家族中的每一种酶的功能进行了预测。该模型从UniProtKB和SwissProt数据库创建数据集并通过蛋白质结构域注释数据库Gene3D进行注释分类,最终通过306个CATH同源超家族的1 121种酶进行模型测试表明该模型准确率达到了98%,还通过随机森林推定出来一些特异性残基,发现了在功能分化程度高的超家族中所含有的活性残基数量是最多的。整合蛋白质多种数据有利于对蛋白质功能进行预测,如Srivastava等[67]根据EC编号和酶名称对来源于PDB蛋白质数据库4 000条序列数据进行分类并分为训练集和测试集,通过SVM和随机森林算法结合一级结构、结构分子量、配体分子分子量和链长等7个特征来预测训练集蛋白的EC类别,结果表明SVM模型的总体准确率(88.49%)优于随机森林(53.9%)。在实际情况中,影响蛋白质功能的各种生物学和理化性质之间并不完全独立,还需要考虑各种特性组合对蛋白质注释的贡献。GOLabeler[72]是一个以序列作为输入的模型,整合了序列同源性、蛋白结构域、基序以及氨基酸理化性质等多种特征信息,使用5种不同分类器(BLAST-kNN、LR-3mer、LR-InterPro、LR-ProFET和朴素GO项频率计算)分别学习不同特征,然后再通过学习排序算法(learning to rank, LTR)根据不同特征的影响程度对5个分类器分别进行权重调整。在来源于CAFA1和CAFA2的数据集中,GOLabeler对于未知功能蛋白的GO术语项标签预测性能远优于其他参赛模型,且GOLabeler在CAFA3和CAFA-π挑战中也获得了最佳性能。Alperen等[73]通过现有的分类器集成模型利用序列相似性和理化性质等特征进行了训练,对858个EC酶的分类进行了预测,结果显示模型F1值均大于0.9,可以通过氨基酸序列很好地预测未知酶的功能。F1值(F1-score)是分类问题的重要衡量指标,是精确率(预测为正的样本中实际为正的比例)和召回率(实际为正的所有样本中预测为正的比例)的调和平均数。此外,蛋白质相互作用网络也已被越来越多的研究者作为蛋白特征来提升机器学习模型的性能,例如,FunCat是一个蛋白类别的逻辑回归器,是Ni等[74]以BioGrid数据库中含有5 386个注释蛋白质的118 363个酵母蛋白相互作用网络作为数据集,通过对蛋白质相互作用网络特征训练以及3倍交叉验证检验,最终成功预测了酵母的17个主要蛋白类别,为利用蛋白质相互作用网络拓扑结构之间的功能关联来表示蛋白质提供了思路。
4.2 深度学习方法深度学习相比传统机器学习能够更高效地处理复杂且高度非线性的大数据问题,CNN属于深度学习的一种模型,在各类研究领域已被广泛应用,其能够通过神经网络算法的强大拟合能力对训练数据的特征关系进行充分学习从而以高精度输出预测结果。对于蛋白质功能预测,以CNN为代表的深度学习相比于传统的机器学习算法通常能够获得更优的拟合结果。例如,Li等[68]开发出了一个全新的端到端特征提取和分类的深度学习模型DEEPre,以ENZYME数据库构建序列数据集,模型框架以原始序列编码为输入,根据分类结果从原始编码中提取卷积和顺序特征,直接提高了模型性能并成功通过预测酶EC编号来预测酶的功能。DEEPre通过SwissProt数据库分别收集22 168条酶和非酶序列作为验证测试集,对酶亚类预测准确率达到94.15%。在此基础上,Zou等[69]又提出了一种基于分层多标签深度学习的酶功能预测模型mlDEEPre,该模型首先预测给定的未知蛋白序列是单功能酶还是多功能酶,对于多功能酶又进一步预测其具体功能,准确率达到了96.7%。
GO术语[43]能够分别对蛋白质功能的几个层次进行准确描述,有助于理解蛋白质的分子或生化功能。目前,有着越来越多的深度学习方法利用GO术语来帮助预测蛋白质功能[75]。例如,DeepGO[76]是一个完全由数据驱动的、不依赖任何手动输入特征标签信息而构建的深度学习预测蛋白质功能模型,其数据集包括了60 710种来自SwissProt数据库的蛋白质序列,其中GO术语27 760个类别,涵盖了SwissProt中90%以上已注释的蛋白质序列。DeepGO使用了深度学习方法来学习2种对预测蛋白质功能有用的特征(蛋白质序列以及蛋白质在相互作用网络中的位置),通过CAFA建立的评估标准证明其与BLAST等传统方法相比有显著改进,并且在预测蛋白质的细胞定位方面表现出良好的预测性能。DeepGOplus[77]以来自CAFA3挑战的数据作为数据集,将基于序列相似性的预测与CNN模型结合,最终可以快速对任何蛋白质进行功能预测,并且每秒可以注释40个蛋白质。同时,由于DeepGOPlus对氨基酸序列的长度没有限制,因此可将其用于蛋白质功能的基因组规模注释。此外,除了选择处理性能更好的算法来构建模型,选取合适的特征对于机器学习模型预测性能也有重要影响,例如,与简单地基于3-mer序列特征提取的DeepGO相比,DeepFunc[78]使用了信息更丰富的序列特征,如与蛋白质链相关的结构域、家族和基序来编码蛋白质序列,这导致DeepFunc的预测性能优于DeepGO。此外,氨基酸序列位置评分矩阵(position specific scoring matrix, PSSM)在生物信息学领域中主要被应用于蛋白质之间的同源性评估,将其与深度学习相结合能够为蛋白质功能预测提供一种新的方法。例如,Le等[79]提出了一种依据PSSM和蛋白质生化特性预测转运蛋白中鸟苷三磷酸(guanosine triphosphate, GTP)结合位点的方法,从分子功能角度以95.6%的准确率识别转运蛋白质类别。此外,Le等[70]还从UniProt数据库中收集了682种SNARE蛋白和2 583种非SNARE蛋白序列,并应用BLAST去除相似度大于30%的冗余序列,然后将PSSM作为CNN框架的输入,最终在SNARE蛋白质的识别中达到了89.7%的准确率。
5 总结与展望通过人工智能策略,特别是机器学习对蛋白质功能进行预测的方法近年来极大地促进了蛋白质功能预测领域的发展。机器学习方法可以在没有任何生物学知识的背景下,通过对蛋白质序列与功能之间映射关系的学习及规律总结,直接推断未知功能蛋白质的生物学信息。但是酶的种类和催化机制的多样性也为该方法带来了重大挑战,因为一种机器学习算法很难涵盖所有的函数并解决相应的复杂关系问题,目前缺少普适性的模型能够预测所有蛋白的具体功能。同样,数据收集和特征处理需要严格的质量控制,用于模型测试的新数据收集往往是整个研究中工作量最为庞大且耗时耗力的,数据格式也难以标准化处理,不同来源的实验数据具有不同的可靠性,还需要考虑到数据之间的差异性。此外,各个家族之间的蛋白质数量差异巨大,一些家族甚至能达到上万条的注释数量,然而一些家族却只能达到几十条的少量注释数量,从机器学习的角度这不利于模型训练。同时,拥有少量数据量的家族在计算机领域被称为低样本量标签[80],如何对低样本量标签的准确注释是蛋白质功能预测研究领域的难点。因此,如何提高数据质量,对数据进行特征表示以获得精确度更高的计算模型,是利用机器学习预测蛋白质功能时需要重点解决的问题。
此外,自然语言处理(natural language processing, NLP)也是人工智能领域的重要分支之一,通过计算机对人类的语言进行处理、理解和运用,进而执行目标任务。蛋白质序列本质上和语言相似,字母能形成具有特定意义的单词和句子,同样,氨基酸之间的多种排列组合能够形成具有各种结构和功能的蛋白。因此,近几年NLP技术在蛋白质研究和设计领域也引起了关注[81],如ProGen[82]和ProtGPT2[83]通过利用NLP技术生成具有特定功能特性且接近天然结构能量的蛋白序列。未来,NLP技术有望被更广泛地应用于蛋白质功能预测领域,有助于蛋白质序列-结构-功能关系的进一步研究。
| [1] |
RÖTHLISBERGER D, KHERSONSKY O, WOLLACOTT AM, JIANG L, DECHANCIE J, BETKER J, GALLAHER JL, ALTHOFF EA, ZANGHELLINI A, DYM O, ALBECK S, HOUK KN, TAWFIK DS, BAKER D. Kemp elimination catalysts by computational enzyme design. Nature, 2008, 453(7192): 190-195. DOI:10.1038/nature06879
|
| [2] |
SKRLJ B, KONC J, KUNEJ T. Identification of sequence variants within experimentally validated protein interaction sites provides new insights into molecular mechanisms of disease development. Molecular Informatics, 2017, 36(9): 1700017. DOI:10.1002/minf.201700017
|
| [3] |
QIAO WL, AKHTER N, FANG XW, MAXIMOVA T, PLAKU E, SHEHU A. From mutations to mechanisms and dysfunction via computation and mining of protein energy landscapes. BMC Genomics, 2018, 19(7): 1-13.
|
| [4] |
FRASCA M, BIANCHI NC. Multitask protein function prediction through task dissimilarity. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2019, 16(5): 1550-1560. DOI:10.1109/TCBB.2017.2684127
|
| [5] |
CAO RZ, CHENG JL. Integrated protein function prediction by mining function associations, sequences, and protein-protein and gene-gene interaction networks. Methods, 2016, 93: 84-91. DOI:10.1016/j.ymeth.2015.09.011
|
| [6] |
SIMPSON LW, GOOD TA, LEACH JB. Protein folding and assembly in confined environments: implications for protein aggregation in hydrogels and tissues. Biotechnology Advances, 2020, 42: 107573. DOI:10.1016/j.biotechadv.2020.107573
|
| [7] |
CHANG YC, HU ZJ, RACHLIN J, ANTON BP, KASIF S, ROBERTS RJ, STEFFEN M. COMBREX-DB: an experiment centered database of protein function: knowledge, predictions and knowledge gaps. Nucleic Acids Research, 2016, 44(D1): D330-D335. DOI:10.1093/nar/gkv1324
|
| [8] |
AL-SHAHIB A, BREITLING R, GILBERT DR. Predicting protein function by machine learning on amino acid sequences-a critical evaluation. BMC Genomics, 2007, 8(1): 1-10. DOI:10.1186/1471-2164-8-1
|
| [9] |
BONETTA R, VALENTINO G. Machine learning techniques for protein function prediction. Proteins: Structure, Function, and Bioinformatics, 2020, 88(3): 397-413. DOI:10.1002/prot.25832
|
| [10] |
CONSORTIUM TU. UniProt: the universal protein knowledgebase. Nucleic Acids Research, 2017, 45(D1): D158-D169. DOI:10.1093/nar/gkw1099
|
| [11] |
DAS S, ORENGO CA. Protein function annotation using protein domain family resources. Methods, 2016, 93: 24-34. DOI:10.1016/j.ymeth.2015.09.029
|
| [12] |
郭芳, 张良, 冯旭东, 李春. 植物源UDP-糖基转移酶及其分子改造. 中国生物工程杂志, 2021, 41(9): 78-91. GUO F, ZHANG L, FENG XD, LI C. Plant-derived UDP-glycosyltransferase and its molecular modification. China Biotechnology, 2021, 41(9): 78-91 (in Chinese). |
| [13] |
WATSON JD, LASKOWSKI RA, THORNTON JM. Predicting protein function from sequence and structural data. Current Opinion in Structural Biology, 2005, 15(3): 275-284. DOI:10.1016/j.sbi.2005.04.003
|
| [14] |
CAMACHO C, COULOURIS G, AVAGYAN V, MA N, PAPADOPOULOS J, BEALER K, MADDEN TL. BLAST+: architecture and applications. BMC Bioinformatics, 2009, 10(1): 1-9. DOI:10.1186/1471-2105-10-1
|
| [15] |
POTTER SC, LUCIANI A, EDDY SR, PARK Y, LOPEZ R, FINN RD. HMMER web server: 2018 update. Nucleic Acids Research, 2018, 46(W1): W200-W204. DOI:10.1093/nar/gky448
|
| [16] |
LV ZB, AO CY, ZOU Q. Protein function prediction: from traditional classifier to deep learning. PROTEOMICS, 2019, 1900119. DOI:10.1002/pmic.201900119
|
| [17] |
THOMPSON JD, HIGGINS DG, GIBSON TJ. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Research, 1994, 22(22): 4673-4680. DOI:10.1093/nar/22.22.4673
|
| [18] |
秦晶晶, 孙春玉, 张美萍, 王义. 植物UDP-糖基转移酶分类、功能以及进化. 基因组学与应用生物学, 2018, 37(1): 440-450. QIN JJ, SUN CY, ZHANG MP, WANG Y. Classification, function and evolution of plant UDP-glycosyltransferase. Genomics and Applied Biology, 2018, 37(1): 440-450 (in Chinese). |
| [19] |
PUNTA M, OFRAN Y. The rough guide to in silico function prediction, or how to use sequence and structure information to predict protein function. PLoS Computational Biology, 2008, 4(10): e1000160. DOI:10.1371/journal.pcbi.1000160
|
| [20] |
ONO E, HOMMA Y, HORIKAWA M, KUNIKANE-DOI S, IMAI H, TAKAHASHI S, KAWAI Y, ISHIGURO M, FUKUI Y, NAKAYAMA T. Functional differentiation of the glycosyltransferases that contribute to the chemical diversity of bioactive flavonol glycosides in grapevines (Vitis vinifera). The Plant Cell, 2010, 22(8): 2856-2871. DOI:10.1105/tpc.110.074625
|
| [21] |
NOMURA Y, SEKI H, SUZUKI T, OHYAMA K, MIZUTANI M, KAKU T, TAMURA K, ONO E, HORIKAWA M, SUDO H, HAYASHI H, SAITO K, MURANAKA T. Functional specialization of UDP-glycosyltransferase 73P12 in licorice to produce a sweet triterpenoid saponin, glycyrrhizin. The Plant Journal, 2019, 99(6): 1127-1143. DOI:10.1111/tpj.14409
|
| [22] |
CHEN K, HU ZM, SONG W, WANG ZL, HE JB, SHI XM, CUI QH, QIAO X, YE M. Diversity of O-glycosyltransferases contributes to the biosynthesis of flavonoid and triterpenoid glycosides in Glycyrrhiza uralensis. ACS Synthetic Biology, 2019, 8(8): 1858-1866. DOI:10.1021/acssynbio.9b00171
|
| [23] |
HUANG Y, LI D, WANG JH, CAI Y, DAI ZB, JIANG D, LIU CS. GuUGT, a glycosyltransferase from Glycyrrhiza uralensis, exhibits glycyrrhetinic acid 3- and 30-O-glycosylation. Royal Society Open Science, 2019, 6(10): 191121. DOI:10.1098/rsos.191121
|
| [24] |
GILLE C, GOEDE A, PREIßNER R, ROTHER K, FRÖMMEL C. Conservation of substructures in proteins: interfaces of secondary structural elements in proteasomal subunits 1 1Edited by R. Huber. Journal of Molecular Biology, 2000, 299(4): 1147-1154. DOI:10.1006/jmbi.2000.3763
|
| [25] |
MESTROM, PRZYPIS, KOWALCZYKIEWICZ, POLLENDER, KUMPF, MARSDEN, BENTO, JARZĘBSKI, SZYMAŃSKA, CHRUŚCIEL, TISCHLER, SCHOEVAART, HANEFELD, HAGEDOORN. Leloir glycosyltransferases in applied biocatalysis: a multidisciplinary approach. International Journal of Molecular Sciences, 2019, 20(21): 5263. DOI:10.3390/ijms20215263
|
| [26] |
CONSORTIUM W, BURLEY SK, BERMAN HM, BHIKADIYA C, BI CX, CHEN L, DI COSTANZO L, CHRISTIE C, DUARTE JM, DUTTA S, FENG ZK, GHOSH S, GOODSELL DS, GREEN RK, GURANOVIC V, GUZENKO D, HUDSON BP, LIANG YH, LOWE R, PEISACH E, et al. Protein data bank: the single global archive for 3D macromolecular structure data. Nucleic Acids Research, 2019, 47(D1): D520-D528. DOI:10.1093/nar/gky949
|
| [27] |
YE Y, GODZIK A. FATCAT: a web server for flexible structure comparison and structure similarity searching. Nucleic Acids Research, 2004, 21(Web Server): W582-W585.
|
| [28] |
WANG S, MA JZ, PENG J, XU JB. Protein structure alignment beyond spatial proximity. Scientific Reports, 2013, 3: 1448. DOI:10.1038/srep01448
|
| [29] |
DAWSON NL, LEWIS TE, DAS S, LEES JG, LEE D, ASHFORD P, ORENGO CA, SILLITOE I. CATH: an expanded resource to predict protein function through structure and sequence. Nucleic Acids Research, 2017, 45(D1): D289-D295. DOI:10.1093/nar/gkw1098
|
| [30] |
ZONG GN, FEI S, LIU X, LI J, GAO YR, YANG X, WANG XQ, SHEN YQ. Crystal structures of rhamnosyltransferase UGT89C1 from Arabidopsis thaliana reveal the molecular basis of sugar donor specificity for UDP-β-l-rhamnose and rhamnosylation mechanism. The Plant Journal, 2019, 99(2): 257-269.
|
| [31] |
WANG ZL, GAO HM, WANG S, ZHANG M, CHEN K, ZHANG YQ, WANG HD, HAN BY, XU LL, SONG TQ, YUN CH, QIAO X, YE M. Dissection of the general two-step di- C-glycosylation pathway for the biosynthesis of (iso)schaftosides in higher plants. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(48): 30816-30823. DOI:10.1073/pnas.2012745117
|
| [32] |
LECUN Y, BOSER B, DENKER J, HENDERSON D, HOWARD R, HUBBARD W, JACKEL L. Handwritten digit recognition with a back-propagation network. Advances in neural information processing systems, 1989, 2.
|
| [33] |
GAWEHN E, HISS JA, BROWN JB, SCHNEIDER G. Advancing drug discovery via GPU-based deep learning. Expert Opinion on Drug Discovery, 2018, 13(7): 579-582. DOI:10.1080/17460441.2018.1465407
|
| [34] |
YANG X, WANG YF, BYRNE R, SCHNEIDER G, YANG SY. Concepts of artificial intelligence for computer-assisted drug discovery. Chemical Reviews, 2019, 119(18): 10520-10594. DOI:10.1021/acs.chemrev.8b00728
|
| [35] |
JUMPER J, EVANS R, PRITZEL A, GREEN T, FIGURNOV M, RONNEBERGER O, TUNYASUVUNAKOOL K, BATES R, ŽÍDEK A, POTAPENKO A, BRIDGLAND A, MEYER C, KOHL SAA, BALLARD AJ, COWIE A, ROMERA-PAREDES B, NIKOLOV S, JAIN R, ADLER J, BACK T, et al. Highly accurate protein structure prediction with AlphaFold. Nature, 2021, 596(7873): 583-589. DOI:10.1038/s41586-021-03819-2
|
| [36] |
TUNYASUVUNAKOOL K, ADLER J, WU Z, GREEN T, ZIELINSKI M, ŽÍDEK A, BRIDGLAND A, COWIE A, MEYER C, LAYDON A, VELANKAR S, KLEYWEGT GJ, BATEMAN A, EVANS R, PRITZEL A, FIGURNOV M, RONNEBERGER O, BATES R, KOHL SAA, POTAPENKO A, et al. Highly accurate protein structure prediction for the human proteome. Nature, 2021, 596(7873): 590-596. DOI:10.1038/s41586-021-03828-1
|
| [37] |
HUANG S, CAI N, PACHECO PP, NARRANDES S, WANG Y, XU W. Applications of support vector machine (SVM) learning in cancer genomics. Cancer Genomics & Proteomics, 2018, 15(1): 41-51.
|
| [38] |
WU YY, WANG GY. Machine learning based toxicity prediction: from chemical structural description to transcriptome analysis. International Journal of Molecular Sciences, 2018, 19(8): 2358. DOI:10.3390/ijms19082358
|
| [39] |
GATOS I, TSANTIS S, SPILIOPOULOS S, KARNABATIDIS D, THEOTOKAS I, ZOUMPOULIS P, LOUPAS T, HAZLE JD, KAGADIS GC. A machine-learning algorithm toward color analysis for chronic liver disease classification, employing ultrasound shear wave elastography. Ultrasound in Medicine & Biology, 2017, 43(9): 1797-1810.
|
| [40] |
MAZURENKO S, PROKOP Z, DAMBORSKY J. Machine learning in enzyme engineering. ACS Catalysis, 2020, 10(2): 1210-1223. DOI:10.1021/acscatal.9b04321
|
| [41] |
MITCHELL AL, ATTWOOD TK, BABBITT PC, BLUM M, BORK P, BRIDGE A, BROWN SD, CHANG HY, EL-GEBALI S, FRASER MI, GOUGH J, HAFT DR, HUANG HZ, LETUNIC I, LOPEZ R, LUCIANI A, MADEIRA F, MARCHLER-BAUER A, MI HY, NATALE DA, et al. InterPro in 2019: improving coverage, classification and access to protein sequence annotations. Nucleic Acids Research, 2019, 47(D1): D351-D360. DOI:10.1093/nar/gky1100
|
| [42] |
CONSORTIUM TU. UniProt: a worldwide hub of protein knowledge. Nucleic Acids Research, 2019, 47(D1): D506-D515. DOI:10.1093/nar/gky1049
|
| [43] |
ASHBURNER M, BALL CA, BLAKE JA, BOTSTEIN D, BUTLER H, CHERRY JM, DAVIS AP, DOLINSKI K, DWIGHT SS, EPPIG JT, HARRIS MA, HILL DP, ISSEL-TARVER L, KASARSKIS A, LEWIS S, MATESE JC, RICHARDSON JE, RINGWALD M, RUBIN GM, SHERLOCK G. Gene ontology: tool for the unification of biology. Nature Genetics, 2000, 25(1): 25-29. DOI:10.1038/75556
|
| [44] |
YOU ZH, LEI YK, ZHU L, XIA JF, WANG B. Prediction of protein-protein interactions from amino acid sequences with ensemble extreme learning machines and principal component analysis. BMC Bioinformatics, 2013, 14(8): 1-11.
|
| [45] |
PRICE MN, WETMORE KM, WATERS RJ, CALLAGHAN M, RAY J, LIU HL, KUEHL JV, MELNYK RA, LAMSON JS, SUH Y, CARLSON HK, ESQUIVEL Z, SADEESHKUMAR H, CHAKRABORTY R, ZANE GM, RUBIN BE, WALL JD, VISEL A, BRISTOW J, BLOW MJ, et al. Mutant phenotypes for thousands of bacterial genes of unknown function. Nature, 2018, 557(7706): 503-509. DOI:10.1038/s41586-018-0124-0
|
| [46] |
BILESCHI ML, BELANGER D, BRYANT DH, SANDERSON T, CARTER B, SCULLEY D, BATEMAN A, DEPRISTO MA, COLWELL LJ. Using deep learning to annotate the protein universe. Nature Biotechnology, 2022, 40(6): 932-937. DOI:10.1038/s41587-021-01179-w
|
| [47] |
CORRAL-CORRAL R, BELTRÁN J, BRIZUELA C, del RIO G. Systematic identification of machine-learning models aimed to classify critical residues for protein function from protein structure. Molecules, 2017, 22(10): 1673. DOI:10.3390/molecules22101673
|
| [48] |
GADO JE, HARRISON BE, SANDGREN M, STÅHLBERG J, BECKHAM GT, PAYNE CM. Machine learning reveals sequence-function relationships in family 7 glycoside hydrolases. Journal of Biological Chemistry, 2021, 297(2): 100931. DOI:10.1016/j.jbc.2021.100931
|
| [49] |
YANG M, FEHL C, LEES KV, LIM EK, OFFEN WA, DAVIES GJ, BOWLES DJ, DAVIDSON MG, ROBERTS SJ, DAVIS BG. Functional and informatics analysis enables glycosyltransferase activity prediction. Nature Chemical Biology, 2018, 14(12): 1109-1117. DOI:10.1038/s41589-018-0154-9
|
| [50] |
WOLPERT DH. The lack of a priori distinctions between learning algorithms. Neural Computation, 1996, 8(7): 1341-1390. DOI:10.1162/neco.1996.8.7.1341
|
| [51] |
ZHOU NH, JIANG YX, BERGQUIST T, LEE AJ, KACSOH B, CROCKER A, LEWIS KA, GEORGHIOU G, NGUYEN HN, HAMID MN, DAVIS L, DOGAN T, ATALAY V, RIFAIOGLU A, DALKIRAN A, ATALAY RC, ZHANG CX, HURTO RL, FREDDOLINO PL, ZHANG Y, et al. The CAFA challenge reports improved protein function prediction and new functional annotations for hundreds of genes through experimental screens. Genome Biology, 2019, 20(1): 244. DOI:10.1186/s13059-019-1835-8
|
| [52] |
LAN L, DJURIC N, GUO YH, VUCETIC S. MS-k NN: protein function prediction by integrating multiple data sources. BMC Bioinformatics, 2013, 14(3): 1-10.
|
| [53] |
GLIGORIJEVIĆ V, JANJIĆ V, PRŽULJ N. Integration of molecular network data reconstructs gene ontology. Bioinformatics, 2014, 30(17): i594-i600. DOI:10.1093/bioinformatics/btu470
|
| [54] |
HALPERIN I, GLAZER DS, WU S, ALTMAN RB. The FEATURE framework for protein function annotation: modeling new functions, improving performance, and extending to novel applications. BMC Genomics, 2008, 9(2): 1-14.
|
| [55] |
蔡从中, 韩连漪, 王万录, 陈宇综. 支持向量机程序SVMProt预测SARS病毒蛋白质的功能. 重庆大学学报(自然科学版), 2003, 26(9): 148-150. CAI CZ, HAN LY, WANG WL, CHEN YZ. Prediction of the function of SARS proteins by using a support vector machine program SVMProt. Journal of Chongqing University (Natural Science Edition), 2003, 26(9): 148-150 (in Chinese). |
| [56] |
VOLPATO V, ADELFIO A, POLLASTRI G. Accurate prediction of protein enzymatic class by N-to-1 neural networks. BMC Bioinformatics, 2013, 14(1): 1-7. DOI:10.1186/1471-2105-14-1
|
| [57] |
PAN YL, LIU DW, DENG L. Accurate prediction of functional effects for variants by combining gradient tree boosting with optimal neighborhood properties. PLoS One, 2017, 12(6): e0179314. DOI:10.1371/journal.pone.0179314
|
| [58] |
DEHZANGI A, LÓPEZ Y, LAL SP, TAHERZADEH G, MICHAELSON J, SATTAR A, TSUNODA T, SHARMA A. PSSM-Suc: accurately predicting succinylation using position specific scoring matrix into bigram for feature extraction. Journal of Theoretical Biology, 2017, 425: 97-102. DOI:10.1016/j.jtbi.2017.05.005
|
| [59] |
CHE YX, JU Y, XUAN P, LONG R, XING F. Identification of multi-functional enzyme with multi-label classifier. PLoS One, 2016, 11(4): e0153503. DOI:10.1371/journal.pone.0153503
|
| [60] |
OSMAN MH, LIONG C, HASHIM I. Hybrid learning algorithm in neural network system for enzyme classification. International Journal of Advances in Soft Computing and Its Applications, 2010, 2: 209-220.
|
| [61] |
MOHAMMED A, GUDA C. Application of a hierarchical enzyme classification method reveals the role of gut microbiome in human metabolism. BMC Genomics, 2015, 16(7): 1-19.
|
| [62] |
DOBSON PD, DOIG AJ. Predicting enzyme class from protein structure without alignments. Journal of Molecular Biology, 2005, 345(1): 187-199. DOI:10.1016/j.jmb.2004.10.024
|
| [63] |
BORRO LC, OLIVEIRA SR, YAMAGISHI ME, MANCINI AL, JARDINE JG, MAZONI I, SANTOS EH, HIGA RH, KUSER PR, NESHICH G. Predicting enzyme class from protein structure using Bayesian classification. Genetics and Molecular Research, 2006, 5(1): 193-202.
|
| [64] |
AMIDI A, AMIDI S, VLACHAKIS D, PARAGIOS N, ZACHARAKI EI. A machine learning methodology for enzyme functional classification ccombining structural and protein sequence descriptors[M]//Bioinformatics and Biomedical Engineering. Cham: Springer International Publishing, 2016: 728-738.
|
| [65] |
AMIDI S, AMIDI A, VLACHAKIS D, PARAGIOS N, ZACHARAKI EI. Automatic single- and multi-label enzymatic function prediction by machine learning. PeerJ, 2017, 5: e3095. DOI:10.7717/peerj.3095
|
| [66] |
NAGAO C, NAGANO N, MIZUGUCHI K. Prediction of detailed enzyme functions and identification of specificity determining residues by random forests. PLoS One, 2014, 9(1): e84623. DOI:10.1371/journal.pone.0084623
|
| [67] |
SRIVASTAVA A, MAHMOOD A, SRIVASTAVA R. A comparative analysis of SVM random forest methods for protein function prediction[C]//2017 International Conference on Current Trends in Computer, Electrical, Electronics and Communication (CTCEEC). September 8-9, Mysore, India, IEEE, 2018: 1008-1010.
|
| [68] |
LI Y, WANG S, UMAROV R, XIE BQ, FAN M, LI LH, GAO X. DEEPre: sequence-based enzyme EC number prediction by deep learning. Bioinformatics (Oxford, England), 2018, 34(5): 760-769.
|
| [69] |
ZOU Z, TIAN S, GAO X, LI Y. mlDEEPre: Multi-functional enzyme function prediction With hierarchical multi-label deep learning. Frontiers In Genetics, 2019, 9.
|
| [70] |
LE NQK, NGUYEN VN. SNARE-CNN: a 2D convolutional neural network architecture to identify SNARE proteins from high-throughput sequencing data. PeerJ Computer Science, 2019, 5: e177.
|
| [71] |
ARAKAKI AK, HUANG Y, SKOLNICK J. EFICAz2: enzyme function inference by a combined approach enhanced by machine learning. BMC Bioinformatics, 2009, 10(1): 1-15.
|
| [72] |
YOU RH, ZHANG ZH, XIONG Y, SUN FZ, MAMITSUKA H, ZHU SF. GOLabeler: improving sequence-based large-scale protein function prediction by learning to rank. Bioinformatics, 2018, 34(14): 2465-2473.
|
| [73] |
ALPEREN D, SUREYYA RA, JESUS MM, RENGUL CA, VOLKAN A, TUNCA D. ECPred: a tool for the prediction of the enzymatic functions of protein sequences based on the EC nomenclature. BMC Bioinformatics, 2018, 19(1): 334.
|
| [74] |
NI QS, WANG ZZ, HAN QJ, LI GG, WANG XM, WANG GY. Using logistic regression method to predict protein function from protein-protein interaction data[C]//2009 3rd International Conference on Bioinformatics and Biomedical Engineering. June 11-13, 2009, Beijing, China. IEEE, 2009: 1-4.
|
| [75] |
IDDO F. Automated protein function prediction: the genomic challenge. Briefings in Bioinformatics, 2006, 7(3): 225-242.
|
| [76] |
MAXAT K, ASIF KM, ROBERT H, JONATHAN W. DeepGO: predicting protein functions from sequence and interactions using a deep ontology-aware classifier. Bioinformatics (Oxford, England), 2018, 34(4): 660-668.
|
| [77] |
KULMANOV M, HOEHNDORF R. DeepGOPlus: improved protein function prediction from sequence. Bioinformatics, 2019, 36: 422-429.
|
| [78] |
ZHANG FH, SONG H, ZENG M, LI YH, KURGAN L, LI M. DeepFunc: a deep learning framework for accurate prediction of protein functions from protein sequences and interactions. Proteomics, 2019, 19(12): 1900019.
|
| [79] |
LE NQK, OU YY. Incorporating efficient radial basis function networks and significant amino acid pairs for predicting GTP binding sites in transport proteins. BMC Bioinformatics, 2016, 17(19): 183-192.
|
| [80] |
WENG HQ, JI SL, LIU CC, WANG T, HE QM, CHEN JH. Fast-RCM: fast tree-based unsupervised rare-class mining. IEEE Transactions on Cybernetics, 2021, 51(10): 5198-5211.
|
| [81] |
FERRUZ N, HÖCKER B. Controllable protein design with language models. Nature Machine Intelligence, 2022, 4(6): 521-532.
|
| [82] |
MADANI A, KRAUSE B, GREENE ER, SUBRAMANIAN S, MOHR BP, HOLTON JM, OLMOS JL, XIONG C, SUN ZZ, SOCHER R, FRASER JS, NAIK N. Large language models generate functional protein sequences across diverse families. Nature Biotechnology, 2023, 1-8.
|
| [83] |
FERRUZ N, SCHMIDT S, HÖCKER B. ProtGPT2 is a deep unsupervised language model for protein design. Nature Communications, 2022, 13: 4348.
|
 2023, Vol. 39
2023, Vol. 39