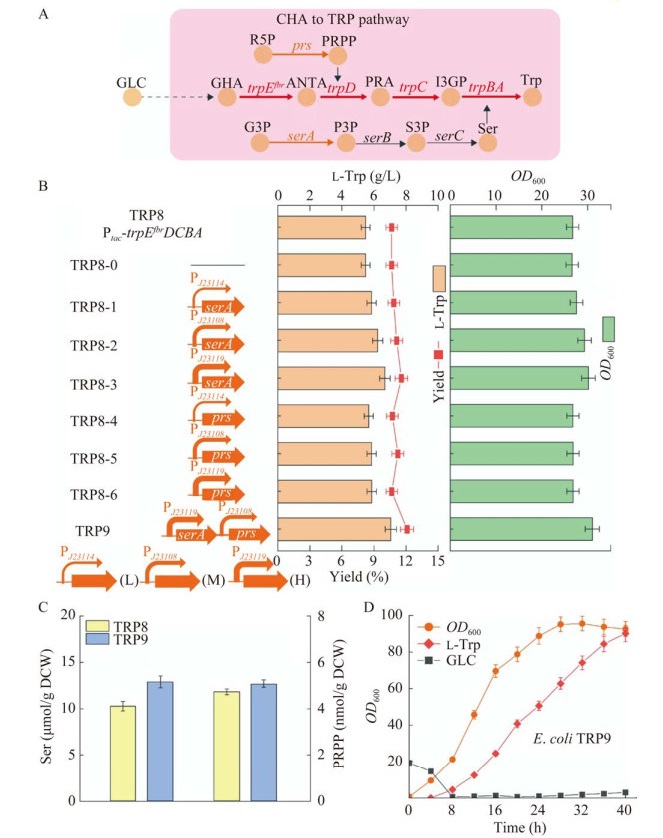
| [1] |
LIU S, XU JZ, ZHANG WG. Advances and prospects in metabolic engineering of Escherichia coli for l-tryptophan production. World Journal of Microbiology and Biotechnology, 2022, 38(2): 1-16.
|
|
| [2] |
IKEDA M. Amino acid production processes[M]//Microbial Production of l-Amino Acids. Berlin, Heidelberg: Springer Berlin Heidelberg, 2002: 1-35.
|
|
| [3] |
FANG MY, ZHANG C, YANG S, CUI JY, JIANG PX, LOU K, WACHI M, XING XH. High crude violacein production from glucose by Escherichia coli engineered with interactive control of tryptophan pathway and violacein biosynthetic pathway. Microbial Cell Factories, 2015, 14(1): 1-13. DOI:10.1186/s12934-014-0183-3
|
|
| [4] |
FANG MY, WANG TM, ZHANG C, BAI JL, ZHENG X, ZHAO XJ, LOU CB, XING XH. Intermediate-sensor assisted push-pull strategy and its application in heterologous deoxyviolacein production in Escherichia coli. Metabolic Engineering, 2016, 33: 41-51. DOI:10.1016/j.ymben.2015.10.006
|
|
| [5] |
IKEDA M. Towards bacterial strains overproducing l-tryptophan and other aromatics by metabolic engineering. Applied Microbiology and Biotechnology, 2006, 69(6): 615-626. DOI:10.1007/s00253-005-0252-y
|
|
| [6] |
ZHANG J, PETERSEN SD, RADIVOJEVIC T, RAMIREZ A, PÉREZ-MANRÍQUEZ A, ABELIUK E, SÁNCHEZ BJ, COSTELLO Z, CHEN Y, FERO MJ, MARTIN HG, NIELSEN J, KEASLING JD, JENSEN MK. Combining mechanistic and machine learning models for predictive engineering and optimization of tryptophan metabolism. Nature Communications, 2020, 11: 4880. DOI:10.1038/s41467-020-17910-1
|
|
| [7] |
NIU H, LI RR, LIANG QF, QI QS, LI Q, GU PF. Metabolic engineering for improving l-tryptophan production in Escherichia coli. Journal of Industrial Microbiology and Biotechnology, 2019, 46(1): 55-65. DOI:10.1007/s10295-018-2106-5
|
|
| [8] |
RODRIGUEZ A, MARTNEZ JA, FLORES N, ESCALANTE A, GOSSET G, BOLIVAR F. Engineering Escherichia coli to overproduce aromatic amino acids and derived compounds. Microbial Cell Factories, 2014, 13(1): 1-15. DOI:10.1186/1475-2859-13-1
|
|
| [9] |
CHEN L, ZENG AP. Rational design and metabolic analysis of Escherichia coli for effective production of l-tryptophan at high concentration. Applied Microbiology and Biotechnology, 2017, 101(2): 559-568. DOI:10.1007/s00253-016-7772-5
|
|
| [10] |
BERRY A. Improving production of aromatic compounds in Escherichia coli by metabolic engineering. Trends in Biotechnology, 1996, 14(7): 250-256. DOI:10.1016/0167-7799(96)10033-0
|
|
| [11] |
SHEN T, LIU Q, XIE XX, XU QY, CHEN N. Improved production of tryptophan in genetically engineered Escherichia coli with TktA and PpsA overexpression. Journal of Biomedicine and Biotechnology, 2012, 2012: 605219.
|
|
| [12] |
XU QY, BAI F, CHEN N, BAI G. Gene modification of the acetate biosynthesis pathway in Escherichia coli and implementation of the cell recycling technology to increase l-tryptophan production. PLoS One, 2017, 12(6): e0179240. DOI:10.1371/journal.pone.0179240
|
|
| [13] |
GUO L, DING S, LIU YD, GAO C, HU GP, SONG W, LIU J, CHEN XL, LIU LM. Enhancing tryptophan production by balancing precursors in Escherichia coli. Biotechnology and Bioengineering, 2022, 119(3): 983-993. DOI:10.1002/bit.28019
|
|
| [14] |
JIANG Y, CHEN B, DUAN CL, SUN BB, YANG JJ, YANG S. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system. Applied and Environmental Microbiology, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14
|
|
| [15] |
GIBSON DG, YOUNG L, CHUANG RY, VENTER JC, HUTCHISON CA Ⅲ, SMITH HO. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods, 2009, 6(5): 343-345. DOI:10.1038/nmeth.1318
|
|
| [16] |
CHEN Z, CHEN XJ, LI QY, ZHOU P, ZHAO ZJ, LI BG. Transcriptome analysis reveals potential mechanisms of l-serine production by Escherichia coli fermentation in different carbon-nitrogen ratio medium. Foods, 2022, 11(14): 2092. DOI:10.3390/foods11142092
|
|
| [17] |
DING DQ, BAI DY, LI JL, MAO ZT, ZHU YR, LIU P, LIN JP, MA HW, ZHANG DW. Analyzing the genetic characteristics of a tryptophan-overproducing Escherichia coli. Bioprocess and Biosystems Engineering, 2021, 44(8): 1685-1697. DOI:10.1007/s00449-021-02552-4
|
|
| [18] | |
|
| [19] |
NEUMANN P, WEIDNER A, PECH A, STUBBS MT, TITTMANN K. Structural basis for membrane binding and catalytic activation of the peripheral membrane enzyme pyruvate oxidase from Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(45): 17390-17395. DOI:10.1073/pnas.0805027105
|
|
| [20] |
WANG J, HUANG J, SHI JM, XU QY, XIE XX, CHEN N. Fermentation characterization of an l-tryptophan producing Escherichia coli strain with inactivated phosphotransacetylase. Annals of Microbiology, 2013, 63(4): 1219-1224. DOI:10.1007/s13213-012-0579-4
|
|
| [21] |
SAWERS G. A novel mechanism controls anaerobic and catabolite regulation of the Escherichia coli tdc operon. Molecular Microbiology, 2004, 39(5): 1285-1298. DOI:10.1111/j.1365-2958.2001.02316.x
|
|
| [22] |
LIU LN, DUAN XG, WU J. L-tryptophan production in Escherichia coli improved by weakening the pta-AckA pathway. PLoS One, 2016, 11(6): e0158200. DOI:10.1371/journal.pone.0158200
|
|
| [23] |
ZHU JF, SHIMIZU K. Effect of a single-gene knockout on the metabolic regulation in Escherichia coli for d-lactate production under microaerobic condition. Metabolic Engineering, 2005, 7(2): 104-115. DOI:10.1016/j.ymben.2004.10.004
|
|
| [24] |
DOWD SR, PRATT EA, SUN ZY, HO C. Nature and environment of the sulfhydryls of membrane-associated d-lactate dehydrogenase of Escherichia coli. Biochimica et Biophysica Acta (BBA)-Protein Structure and Molecular Enzymology, 1995, 1252(2): 278-283.
|
|
| [25] |
WANG J, SHEN XL, REY J, YUAN QP, YAN YJ. Recent advances in microbial production of aromatic natural products and their derivatives. Applied Microbiology and Biotechnology, 2018, 102(1): 47-61. DOI:10.1007/s00253-017-8599-4
|
|
| [26] |
李柱. 通过理性遗传改造大肠杆菌利用葡萄糖合成色氨酸的研究[D]. 天津: 天津大学硕士学位论文, 2020.
LI Z. Engineering Escherichia coli for production of tryptophan from glucose via rational genetic manipulations[D]. Tianjin: Master's Thesis of Tianjin University, 2020 (in Chinese).
|
|
| [27] |
JUMINAGA D, BAIDOO EEK, REDDING- JOHANSON AM, BATTH TS, BURD H, MUKHOPADHYAY A, PETZOLD CJ, KEASLING JD. Modular engineering of l-tyrosine production in Escherichia coli. Applied and Environmental Microbiology, 2012, 78(1): 89-98. DOI:10.1128/AEM.06017-11
|
|
| [28] |
GUO L, DIAO WW, GAO C, HU GP, DING Q, YE C, CHEN XL, LIU J, LIU LM. Engineering Escherichia coli life span for enhancing chemical production. Nature Catalysis, 2020, 3(3): 307-318. DOI:10.1038/s41929-019-0411-7
|
|
| [29] |
郭亮, 高聪, 柳亚迪, 陈修来, 刘立明. 大肠杆菌生产饲用氨基酸的研究进展. 合成生物学, 2021, 2(6): 964-981. GUO L, GAO C, LIU YD, CHEN XL, LIU LM. Advances in bioproduction of feed amino acid by Escherichia coli. Synthetic Biology Journal, 2021, 2(6): 964-981 (in Chinese).
|
|
| [30] |
MIGUEL S, GUO WH, FENG XY, SHAO ZY. Investigating strain dependency in the production of aromatic compounds in Saccharomyces cerevisiae. Biotechnology and Bioengineering, 2016, 113(12): 2676-2685. DOI:10.1002/bit.26037
|
|
| [31] |
GOSSET G. Improvement of Escherichia coli production strains by modification of the phosphoenolpyruvate: sugar phosphotransferase system. Microbial Cell Factories, 2005, 4(1): 1-11. DOI:10.1186/1475-2859-4-1
|
|
| [32] |
DODGE TC, GERSTNER JM. Optimization of the glucose feed rate profile for the production of tryptophan from recombinant E coli. Journal of Chemical Technology & Biotechnology, 2002, 77(11): 1238-1245.
|
|
| [33] |
LIU LN, CHEN S, WU J. Phosphoenolpyruvate: glucose phosphotransferase system modification increases the conversion rate during l-tryptophan production in Escherichia coli. Journal of Industrial Microbiology and Biotechnology, 2017, 44(10): 1385-1395. DOI:10.1007/s10295-017-1959-3
|
|
| [34] | |
|
| [35] |
XIONG B, ZHU YD, TIAN DG, JIANG S, FAN XG, MA Q, WU HY, XIE XX. Flux redistribution of central carbon metabolism for efficient production of l-tryptophan in Escherichia coli. Biotechnology and Bioengineering, 2021, 118(3): 1393-1404. DOI:10.1002/bit.27665
|
|
| [36] |
ITO J, YANOFSKY C. The nature of the anthranilic acid synthetase complex of Escherichia coli. Journal of Biological Chemistry, 1966, 241(17): 4112-4114. DOI:10.1016/S0021-9258(18)99820-0
|
|
| [37] |
CHEN YY, LIU YF, DING DQ, CONG LN, ZHANG DW. Rational design and analysis of an Escherichia coli strain for high-efficiency tryptophan production. Journal of Industrial Microbiology and Biotechnology, 2018, 45(5): 357-367. DOI:10.1007/s10295-018-2020-x
|
|
 2023, Vol. 39
2023, Vol. 39