中国科学院微生物研究所、中国微生物学会主办
文章信息
- 樊祥瑞, 王俊燕, 梁丽亚, 刘嵘明
- FAN Xiangrui, WANG Junyan, LIANG Liya, LIU Rongming
- 基于CRISPR/Cas系统的多重基因编辑与调控技术
- Multiplex gene editing and regulation techniques based on CRISPR/Cas system
- 生物工程学报, 2023, 39(6): 2449-2464
- Chinese Journal of Biotechnology, 2023, 39(6): 2449-2464
- 10.13345/j.cjb.220989
-
文章历史
- Received: December 9, 2022
- Accepted: March 29, 2023
- Published: April 13, 2023
CRISPR/Cas系统是存在于古细菌或细菌中特有的一种获得性免疫系统[1]。相较于锌指核酸酶(zinc-finger nucleases, ZFNs)[2]和转录激活样效应因子核酸酶(transcription activator-like effector nuclease, TALENs)[3]等传统的基因编辑方法,基于CRISPR/Cas系统的识别和剪切作用开发出的基因编辑工具能够使用较短的向导RNA (guide RNA, gRNA)序列实现精准定位,大大降低了设计难度和应用成本,并且已经在微生物、植物、动物及人体细胞内得到了广泛应用[4]。目前,基于CRISPR/Cas系统开发的技术和工具呈现井喷式发展趋势,尤其在基因编辑[5-6]、基因调控[7-8]、基因成像[9]和表观遗传[10]等领域相较于传统技术具有明显优势。
基于CRISPR/Cas系统开发的基因编辑与调控工具,主要是在Ⅱ类CRISPR-Cas系统基础上构建的,包括Cas9[11]、Cas12a/b/c[12]及Cas13a/b/c[13]等。不同于Ⅰ类CRISPR/Cas系统需要编码多个蛋白串联为多蛋白效应复合物发挥作用,Ⅱ类CRISPR/Cas系统只需要编码单个蛋白即可对外源基因或菌株自身特定位点进行剪切[14],这种剪切会导致DNA双链断裂(double-strand breaks, DSB),触发菌株自身易错修复,这种修复极易导致碱基的插入、缺失、倒置和替换,但可以通过同源片段的重组来实现精准修复[15]。目前,通过对CRISPR/Cas系统的优化和改造,已经开发出了一系列基因编辑与调控工具,如碱基编辑器(base editor, BE)[16-17]、先导编辑器(prime editor, PE)[18-19]、基于CRISPR/Cas系统的基因抑制(CRISPR interference, CRISPRi)[20]和基于CRISPR/Cas系统的基因激活(CRISPR activation, CRISPRa)[21]等。这些基于CRISPR/Cas系统的基因编辑与调控工具已经在合成生物学等领域得到了广泛应用,为生命科学、生物工程、生物医药和农业新物种培育领域带来了创造性的变革。然而,基于CRISPR/Cas系统开发的基因编辑与调控工具在多重基因编辑与调控方面还仍然存在着挑战,尤其是在提高基因编辑与调控的效率、特异性以及降低脱靶率等方面[22]。随着第三代测序技术和DNA合成技术的不断发展以及计算机辅助设计在生物学中的应用,多重基因编辑与调控的准确度和特异性不断提升。本文对基于CRISPR/Cas系统的多重基因编辑与调控技术的优化方法及相关进展进行了系统阐述。这些研究工作显著地扩展了CRISPR/Cas系统在多重基因编辑与调控方面的应用,并为CRISPR/Cas系统在多个领域的应用作出了重要贡献。
1 基于CRISPR/Cas系统的双链断裂的多重基因编辑技术 1.1 基于CRISPR/Cas系统的单细胞多位点编辑基于CRISPR/Cas系统的基因编辑过程中,DNA双链进行剪切后在自身或者外源DNA片段的作用下进行同源重组对DNA双链进行恢复。在真核生物方面,Bao等[23]在酿酒酵母(Saccharomyces cerevisiae)细胞中通过对Cas9蛋白的改造(D147Y和P411T)、质粒系统以及培养条件的优化建立了一种可实现高效同源整合CRISPR基因编辑技术(homology-integrated CRISPR, HI-CRISPR),此方法可以同时对3个基因进行敲除。该系统中多基因敲除效率与质粒拷贝数成正比,因此可以通过增加培养时间提高敲除效率。使用HI-CRISPR技术培养4 d后,成功实现了对3个基因的同时敲除,敲除效率分别为27%、75%和80%;使用同样的技术培养6 d后,其3个基因同时敲除的效率高达100%,是当前报道最高的基因敲除效率。Xie等[24]也开发了一种便捷的基因组编辑工具,利用植物中的转录机制与tRNA-gRNA阵列相结合开发了一种内源性RNA处理系统。通过将tRNA和gRNA串联形成紧凑的重复序列,利用植物中的RNase识别tRNA并进行切割,能够同时产生多个特异性靶向目标序列的gRNA,进而分别引导Cas9蛋白对靶DNA链进行切割,提供了一种在植物中进行多位点基因编辑的简单方法。其中,在水稻中可以同时靶向4个基因位点,染色体敲除效率为4%−20%。在原核生物方面,Jiang等[25]在大肠杆菌(Escherichia coli)中通过设计与构建双质粒系统,可实现同时对3个基因的敲除,并且使用温敏型复制子特性,利用控制温度的方法去除Cas质粒,使菌株不含有任何质粒,更有利于生产菌株的进一步改造;Zhang等[26]在酿酒酵母中利用tRNA阵列的方式,通过2轮迭代成功实现了8个位点的同时敲除。除此之外,还有很多团队在不同的物种中开发了单细胞多位点基因编辑技术,如乳酸克鲁维酵母(Kluyveromyces lactis)[27]、汉逊酵母(Ogataea polymorpha)[28]、斑马鱼[29]、谷氨酸棒状杆菌(Corynebacterium glutamicum)[30]、水稻[31]和人体细胞[32]等。但是多位点基因编辑效率在单细胞中随着编辑位点的增加而降低(表 1)。
| CRISPR/Cas systems | Number of the targets | Editing efficiency (%) | Organisms | References |
| Prokaryotes | ||||
| CRISPR/Cas9 | 4 loci | 40 | Escherichia coli | [33] |
| CRISPR/Cas12a | 3 loci | 20 | Escherichia coli | [34] |
| CRISPR/Cas9 | 3 loci | 100 | Escherichia coli | [25] |
| CRISPR/Cas12a | 2 loci | 75 | Streptomyces coelicolor | [35] |
| CRISPR/Cas9 | 3 loci | 23 | Corynebacterium glutamicum | [30] |
| Eukaryotes | ||||
| CRISPR/Cas9 | 5 loci | 33 | Zebra fish | [29] |
| Csy4 | 4 loci | 96 | Saccharomyces cerevisiae | [36] |
| CRISPR/Cas9 | 8 loci | 87 | Saccharomyces cerevisiae | [26] |
| CRISPR/Cas9 | 6 loci | 65 | Saccharomyces cerevisiae | [37] |
| CRISPR/Cas12a | 4 loci | 85 | Saccharomyces cerevisiae | [38] |
| CRISPR/Cas9 | 3 loci | 100 | Saccharomyces cerevisiae | [23] |
| CRISPR/Cas12a | 9 loci | < 54 | Rice | [31] |
| CRISPR/Cas9 | 3 loci | 30 | Ogataea polymorpha | [28] |
| CRISPR/Cas9 | 3 loci | 2 | Kluyveromyces lactis | [27] |
| CRISPR/Cas12a | 5 loci | 40 | HEK293T cells | [32] |
CRISPR相关转座酶(CRISPR-associated transposases, CASTs)[39]是CRISPR元件在进化过程中被Tn7转座子劫持,协同进化衍生出的一种转座酶,CASTs能够在RNA的引导下介导DNA转座并且不依赖同源重组,2019年Strecker等[40]利用来源于蓝藻的CASTs系统构建了一种基因插入工具,可以用于在大肠杆菌中进行基因整合。利用此技术,在大肠杆菌中选择了48个可插入位点,经过一轮基因编辑后在29个位点检测到了插入基因,基因整合效率达到60.4%。2020年,Zhang等[41-42]开发了基于CRISPR转座酶的多拷贝基因组整合工具(multicopy chromosomal integration using CRISPR-associated transposases, MUCICAT),此方法可利用单个crRNA靶向基因组上的重复序列,在大肠杆菌基因组中实现了10个基因拷贝的整合,此外,MUCICAT方法也可以使用crRNA阵列的方式进行多基因失活或DNA插入,成功实现了对大肠杆菌基因组上的11个基因进行改造,通过2轮改造使N-乙酰葡糖胺(N-acetylglucosamine, GlcNAc)产量提升至11.59 g/L。2021年,Yang等[43]利用半透明假交替单胞菌(Pseudoalteromonas translucida) KMM520的高活性I-F型CRISPR转座酶PtrCAST,与霍乱弧菌(Vibrio cholerae) Tn6677 I-F型CRISPR转座酶VchCAST组成正交CASTs,分别能以100%的效率将相应的目的基因插入大肠杆菌染色体上实现基因的多拷贝。
1.2 基于CRISPR/Cas系统的可追踪基因编辑技术Wang等[44]在2009年开发的多重自动基因编辑工具(multiplex automated genome engineering, MAGE)技术可以在细胞群中实现多重基因编辑,可以高效地进行基因组的整合或调控。MAGE技术主要用于原核生物的基因编辑,如Isaacs等[45]使用MAGE技术将大肠杆菌中的终止密码子进行替换。MAGE技术虽然可以实现在细胞群的高通量基因编辑,但是需要对筛选获得的单个细胞进行基因组测序才能对基因编辑进行追踪。可追踪的基因编辑技术可以实现只需要对分子条形码(barcode)进行短序列的高通量测序,即可实现性状和基因型的一一对应,极大地减少了在对目标菌株改造过程中物力和时间的投入。Warner等[46]在2010年开发的可追踪多元重组技术(trackable multiplex recombineering, TRMR),通过在靶向基因前插入上调及下调核糖体结合位点(ribosomebinding site, RBS)片段及用于追踪的分子条形码,进而实现对基因转录的调控,并结合文库高通量合成进而实现全基因组层面的基因转录调节,在经过特定筛选后,可通过高通量测序分子条形码追踪基因编辑情况。
Garst等[47]开发的基于CRISPR/Cas系统的可追踪基因编辑技术(CRISPR-enabled trackable genome engineering, CREATE)是一种基于CRISPR/Cas系统的高通量多重基因编辑技术(图 1)。CREATE为3质粒系统,其中2个质粒用于表达Cas9蛋白和λ-red蛋白,另一个用于表达CREATE盒[gRNA和同源臂(homology arm, HA)],多个功能单元协同作用实现精准定位的基因编辑和同源重组。其中,借助计算机辅助设计及DNA合成技术,可实现高通量合成CREATE盒,从而实现在细胞群层面针对多个靶向位点引入不同突变,并且将CREATE盒的特征序列设计成可以用于追踪基因编辑的DNA条形码。在基因编辑以及菌株筛选后,只需要对DNA条形码进行高通量测序就可追踪导入序列的信息,可以通过一次高通量测序得到细胞群中基因编辑引入突变的变化情况,大幅减少了追踪时间及成本,为细胞工厂的快速构建及筛选提供了一种强有力的工具。Garst等[47]利用CREATE技术成功提高了大肠杆菌对利福平的耐受性。随后Bassalo等[48]利用CREATE技术对赖氨酸代谢途径设计了16 300个同源片段,对赖氨酸的整体代谢过程进行了深入解析;Zheng等[49]利用CREATE和实验室自适应突变提高了大肠杆菌对糠醛的耐受性;Liu等[50-51]使用CREATE技术实现了对大肠杆菌全局调控网络的82个基因特定位点的饱和突变,使突变菌株对糠醛、苯乙烯、异丙醇和异丁醇耐受性得到了不同水平的提高,并将CREATE技术引入到了酿酒酵母(Saccharomyces cerevisiae)中,对酿酒酵母中的全局调控因子进行了多基因改造,提高了酿酒酵母在高葡萄糖和高乙醇条件下的耐受性。

|
| 图 1 CREATE技术的基本原理及实现流程 Fig. 1 Schematic diagram and workflow for CREATE strategy. |
| |
CREATE技术解决了多重基因编辑的难题,但因其无法对含有CREATE盒的质粒进行清除,故无法实现迭代基因编辑。针对以上问题,Liu等[52]设计构建了靶向CREATE盒质粒的gRNA元件并将其整合至Cas蛋白质粒中。在每轮基因编辑之后,通过阿拉伯糖诱导上述gRNA的转录,进而消除CREATE盒质粒。这一方法不仅成功实现了CREATE的迭代编辑还进一步提升了菌株性状的稳定性,此技术被称为迭代CRISPR支持可追踪的基因组工程(iterative CRISPR enabled trackable genome engineering, iCREATE)。为了验证iCREATE的基因编辑效率,使用iCREATE来提高大肠杆菌的3-羟基丙酸产量,一共进行了13轮iCREATE。在前7轮iCREATE中,敲除了3-羟基丙酸的竞争途径;然后又通过5轮iCREATE对3-羟基丙酸生成过程中的关键酶以及前体物质的生成量进行了优化;最后1轮使用iCREATE对大肠杆菌中的全局调控因子进行了优化,提高了大肠杆菌对3-羟基丙酸的耐受性。经过共13轮iCREATE改造后得到的菌株3-羟基丙酸的产量比出发菌株提高了57倍。Liu等[53]还将iCREATE用于木糖和葡萄糖的代谢调控,经过数轮iCREATE改造后的菌株成功提高了对含有木糖和葡萄糖的混合糖代谢速率以及对秸秆等水解液副产物的耐受性,最终得到一株可利用高糠醛和高乙酸秸秆水解液生产3-羟基丙酸(3-hydroxypropionic acid, 3-HP)的突变菌株,其3-羟基丙酸产量比出发菌株提高了7倍。
2 基于CRISPR/Cas系统的单链断裂的多重基因编辑技术 2.1 基于碱基编辑器的多重基因编辑Komo等[15]利用人工改造的Cas9蛋白先后开发了胞嘧啶碱基编辑器(cytosinebase editor, CBE)和腺嘌呤碱基编辑器(adenosinebase editor, ABE)[17]。碱基编辑器主要是由两部分组成的,sgRNA以及由人工改造只保留单链截切能力的Cas9 (D10A, nCas9)蛋白与胞嘧啶脱氨酶或者tRNA腺嘌呤脱氨酶组成的融合蛋白,通过sgRNA引导,人工改造的Cas9蛋白可以靶向目标DNA链,经过脱氨酶的作用实现了C-T或G-A碱基替换(图 2)。

|
| 图 2 CBE和ABE的工作原理 Fig. 2 Schematic diagrams of CBE and ABE. |
| |
Wang等[30]利用碱基编辑器nCas9 (D10A)-AID [活化诱导的胞苷脱氨酶(activation induced cytidine deaminase, AID)]开发了一种多位点自动化谷氨酸棒状杆菌碱基编辑方法(multiplex automated Corynebacterium glutamicum base editing method, MACBETH),在谷氨酸棒状杆菌中实现了单、双和三位点编辑,其编辑效率分别达到了100%、87.2%和23.3%。另外,通过对谷氨酸合成相关的3个基因(odhA、pyk和ldhA)进行组合建库,使得pyk和ldhA双失活菌株的谷氨酸产量提高了3倍。Tong等[54]利用不含DSB的单核苷酸碱基编辑系统(CRISPR-base editing system, CRISPR-BEST)。通过Csy4 (I-F型CRISPR相关核糖核酸内切酶Cas6)识别位点分离设计的3-spacer sgRNA阵列,该阵列能够同时靶向链霉菌中3个生物合成基因簇(biosynthetic gene clusters, BGCs)的关键基因,实现了对SCO5087、SCO3230和SCO5892这3个位点的同时编辑,其中,SCO5087和SCO5892的编辑效率达到了100%,但是SCO3230的编辑效率只有33%。Banno等[55]将尿嘧啶DNA糖化酶抑制剂(uracil glycosylase inhibitor, UGI)和LVA标签(序列为AANDENYALVA的蛋白质降解标签)与dCas-CDA (Petromyzon marinus cytosine deaminase PmCDA1, CDA)相结合,开发了碱基编辑器dCas-CDA-UL,通过将其与包含2组串联重复sgRNA表达单元的多重sgRNA表达质粒结合使用,实现了在大肠杆菌中同时编辑6个位点。
Davis等[56]通过tRNA-gRNA阵列开发了一种新的腺嘌呤碱基编辑器ABE8e,解决了需要通过2个腺病毒运输载体(adeno-associated virus, AAV)来运输胞嘧啶碱基编辑器的问题,通过将gRNA与改造的tRNA开发成紧凑串联结构,实现了在一个质粒上可以同时产生多个gRNA进行多基因编辑。基于内源性RNA处理系统,Yuan等[57]通过设计hCtRNA (75 nt人类半胱氨酸tRNA),开发基于串联组装tRNA-gRNA阵列的DAP架构(drive-and-process architectures),并在nCas9 (D10A)的N端融合TadA-8e (V106W)-1xUGI-hA3A (Y130F)脱氨模块,开发了基于nCas9的双脱氨酶碱基编辑器(A-to-G and C-to-T multiplex editor, ACME)。将2个16个基因座阵列与ACME相结合,在人体细胞中实现了多重碱基编辑(multiple base-editing, MBE),其碱基编辑位点最高可达到31个,平均编辑效率达到51.3%±11.5%。
2.2 基于先导编辑器的多重基因编辑先导编辑器(prime editor, PE)最初是由Anzalone等[58]开发的一种基因编辑工具(图 3)。PE可以实现DNA片段的插入、删除以及12种碱基替换(其中包括转换和颠换)。PE是由两部分组成的,一部分是pegRNA (prime editing guide RNA,在sgRNA3′末端添加一段RNA序列,该序列包含引物结合位点以及携带有目标编辑序列的逆转录模板),另一部分是融合蛋白[nCas9 (H840A)和逆转录酶两部分的融合蛋白]。其基本原理是nCas9 (H840A)在pegRNA的引导下进行靶DNA链的切割,pegRNA3′端的引物结合位点与断裂DNA单链的互补序列相结合,之后逆转录酶沿pegRNA上的逆转录模板进行转录,DNA双链经过编辑之后延伸出一段新合成的DNA单链,即携带目标突变的3′flap和不携带目标突变的5′flap,通过结构特异性内切酶切除以及修复之后最终实现精准编辑。

|
| 图 3 Prime editor的工作原理 Fig. 3 Schematic diagram of prime editor. |
| |
PE的靶向能力因gRNA的表达装置受到限制,其在多重基因编辑方面未取得较好的研究进展。tRNA-gRNA阵列的出现,为实现多基因编辑提供了良好的思路。Yuan等[57]将hCtRNA分别与缺口gRNA (nicking gRNA)和pegRNA组合形成实现单位点高效编辑的串联结构,通过在一个hCtRNA-M阵列上组装6个gRNA,每2个gRNA靶向一个基因,实现对HEK3、RNF2和FANCF在内的3个基因同时编辑。由于tRNA从重复表达单元中剪切下来时,tRNA序列的5′前导序列的一部分会保留在pegRNA序列的3′延伸序列,只有位于hCtRNA-M序列下游的gRNA碱基对对应的编辑位点FANCF表现出较高的编辑效率。为了提高HEK3和RNF2的编辑效率,以实现3个位点同时、高效的基因编辑。研究人员在5′前导序列和3′延伸序列之间插入了一段间隔序列。由于该间隔序列包含一部分终止子序列,从而阻碍了由于tRNA的5′前导序列残留引起的基因编辑效率降低的过程。通过上述改造,最终实现了3个基因位点的同时编辑,其编辑效率分别达到39.1%±0.4%、57.5%±2.3%及36.1%±2.1%。
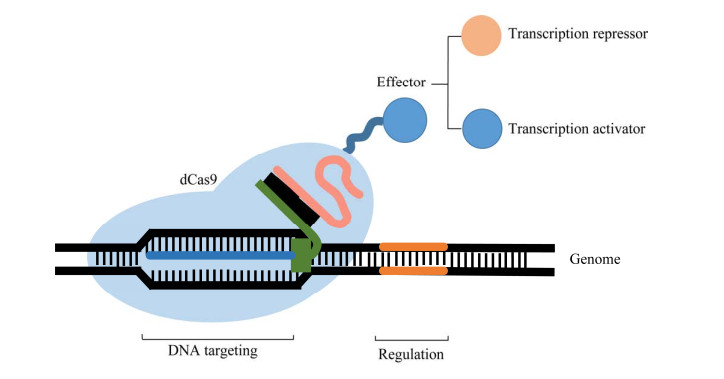
3 基于CRISPR/Cas系统的多重基因调控技术CRISPR激活[20](CRISPR activation, CRISPRa)和CRISPR抑制[21](CRISPR interference or inhibition, CRISPRa)是利用dCas (dead-Cas)蛋白(Cas蛋白的核酸酶结构域HNH和RuvC突变,失去了核酸酶的活性)和转录激活因子(如VP64)或阻遏结构域(如KRAB)融合(图 4),通过结构域的不同功能实现对基因的表达进行精准控制。基于CRISPR/Cas系统的基因调控技术CRISPRa和CRISPRi在起作用时不会引起双链或单链断裂,对宿主细胞毒性较小,其在转录调控方面具有较广的应用前景(表 2)。
|
| 图 4 CRISPRi和CRISPRa的工作原理 Fig. 4 Schematic diagram of CRISPRi and CRISPRa. |
| |
| CRISPR/Cas systems | Number of the genes | Regulation efficiencies (%) | Organisms | References |
| CRISPRi | ||||
| CRISPR/Cas9 | 9 | 85–100 | Escherichia coli | [59] |
| CRISPR/Cas9 | 3 | 75–95 | Yeast | [60] |
| CRISPR/Cas9 | 4 | 60–90 | Streptomyces | [61] |
| CRISPR/Cas9 | 6 | – | Synechocystis | [62] |
| CRISPR/Cas12a | 4 | – | Escherichia coli | [63] |
| CRISPR/Cas12a | 10 | 40–80 | Human | [64] |
| CRISPRa | ||||
| CRISPR/Cas9 | 4 | 55–85 | Mice | [65] |
| CRISPR/Cas9 | 3 | – | Escherichia coli | [66] |
| CRISPR/Cas9 | 3 | – | Saccharomyces cerevisiae | [67] |
| –: No relevant data. | ||||
在单个细胞多重基因抑制方面,Reis等[59]在大肠杆菌中通过设计互不重复的gRNA序列(extra-long sgRNA arrays, ELSAs),同时实现了对9个基因的抑制。在设计过程中,通过机器学习自动设计实现了gRNA的自动化设计,并设计了最大长度为22 bp的组成型启动子。通过自动化设计含有20个gRNA的ELSAs序列,实现了对6个基因的抑制(ackA, iclR, poxB, pta, sdhC, sdhD),提高了大肠杆菌琥珀酸中的产量;通过自动化设计含有15个gRNA的ELSAs序列,对9个有关氨基酸生物合成途径基因的抑制(hisD, proC, lysA, tyrA, aroF, pheA, leuA, ilvD, argH),实现了对目的菌株生长的抑制。在这两种多基因抑制过程中,每个基因都有1−2个gRNA的靶点,此设计可以很大程度上降低CRISPRi脱靶率,其基因抑制效率均大于85%。Mccarty等[60]在酵母中使用单个启动子对应12个gRNA,每4个gRNA靶向同一个基因,实现了对3个基因的抑制。
除Cas9外,CRISPR-Cas系统中Ⅱ类V-A型蛋白Cas12a,也被广泛应用于多重基因抑制。Zhang等[63]通过同时转录4个gRNA及表达dCas12a蛋白,实现了对malT-proP-degP-rseA这4个基因的抑制。Campa等[64]将Cas12a与Triplex序列以及20个gRNA序列相连(图 5),实现了同时对10个不同基因(RAB5A, RAB7A, PIK3C3, EEA1, RAB11A, RAB9A, CHRM3, PLCB1, PRKC1, FZD1)的抑制,其基因抑制效率范围为40%−80%。

|
| 图 5 20个sgRNA排列方式 Fig. 5 20 sgRNA arrangements. |
| |
在细胞群多重基因抑制方面,Wang等[68]通过设计2 281个gRNA,靶向44个已知表型的基因,通过观察各个基因的抑制效率,总结了gRNA的设计原则,包括可以使用15个gRNA同时靶向同一基因以实现基因的稳定抑制以及在开放阅读框(open reading frame, ORF)的前5%内尽可能多地选择sgRNA靶点等,并且以此规则建立了大肠杆菌的基因抑制的文库,包含55 671个sgRNA,实现了对大肠杆菌代谢关键基因的筛选。Rousset等[69]合成了7 177个sgRNA,通过对大肠杆菌中营养代谢限速酶的转录抑制,发现了对大肠杆菌代谢调控起到关键作用的基因,并对大肠杆菌的必需基因进行了解析。
3.2 基于CRISPR/Cas系统的多重基因激活在单细胞多位点激活方面,Cheng等[65]将dCas9与连续10个激活因子(VP16)偶联,成功实现了对小鼠内源基因的激活,并且可以利用不同的gRNA比例来调节基因的激活程度。Lian等[66]通过选择和优化Cas蛋白,开发出了基于CRISPR系统的同时可实现激活、抑制和删除的技术(CRISPR transcriptional activation, transcriptional interference, and gene deletion, CRISPR-AID),通过使用不同长度的gRNA设计或者使用3种不同Cas蛋白的共同表达,实现了在一个系统中同时实现基因的激活、敲除和插入,利用CRISPR-AID系统成功将β-胡萝卜素的产量较出发菌提高了2.8倍,之后,Lian等[67]将CRISPR-AID系统进行了优化并在酿酒酵母中建立了高通量的基因注释方法。
在细胞群基因激活方面,Schmidt等[70]研究T细胞中与信号因子调节相关的基因,将dCas9与激活因子偶联,并结合CRISPRa协同激活技术(CRISPRa synergistic activation mediator)[71]进行sgRNA的设计,成功对T细胞信号表达因子调节的相关基因进行了筛选及解析。
3.3 基于base editor的多重基因调控CRISPRa和CRISPRi在转录调控过程中需保持稳定才能发挥作用,但是在进行工业化生产过程中CRISPRa和CRISPRi稳定性容易受到发酵环境以及生产周期的影响。Wang等[72]开发了基于base editor的基因转录调控技术(base editor-targeted and template-free expression regulation, BETTER)。此技术是通过在靶基因前插入特定的转录或翻译元件,在gRNA的引导下,在插入元件的靶向位点产生多个碱基突变,从而构建突变文库,如使用胞嘧啶编辑器在插入的RBS序列中靶向编辑连续8个G的核心序列,从而构建不同强度的转录调控元件文库。他们将BETTER技术用于谷氨酸棒杆菌代谢木糖的调控,结合高通量筛选获得的突变菌株,其木糖吸收效率达到7.3 mmol/g CDW,高于谷氨酸棒杆菌已报道的最高效率;将BETTER用于番茄红素合成途径10个基因的组合调控,经过筛选获得的突变菌株,其番茄红素产率达到2.9 mg/g CDW,优于报道的谷氨酸棒杆菌产番茄红素的最高产量。
4 总结与展望本文主要针对基于CRISPR/Cas系统的多重基因编辑与调控技术进行了系统的总结与阐述,包括基于CRISPR/Cas系统的双链断裂的基因编辑技术、基于CRISPR/Cas系统的单链断裂的基因编辑技术以及基于CRISPR/Cas系统的多重基因调控技术。这些研究表明可以通过优化功能元件以及改造Cas蛋白实现更加高效和精准的多重基因编辑与调控,但是在高通量的多重基因编辑与调控方面依然有较大的挑战[73]。首先,在单个细胞的多位点基因编辑与调控方面,提高编辑或者调控位点的个数、提高特异性以及开发新的编辑与调控系统都是急需解决的问题。其次在细胞群的多重基因编辑与调控方面,gRNA文库的设计和高通量的筛选方法的不足同样也阻碍了多重基因编辑和调控技术的推广。
为解决以上问题,研究人员正在进行不断的尝试,尤其是近年来计算机辅助设计及深度学习在合成生物学方面的广泛应用,在多重基因编辑与调控方面已经取得了一定的成就[74]。iCREATE技术[52]利用计算机辅助设计用于分子条形码和基因文库的设计;ELSAs序列[59]用深度学习实现自动化设计等。另外,通过在不同微生物中实现新的CRISPR/Cas系统的应用以及对现有CRISPR/Cas系统的优化也在不断取得进展。Schuler等[75]提出的转座子编辑器,其使用的IscB转座子蛋白的体积仅为Cas9蛋白的1/3。该研究揭示了IscB和Cas9核糖核蛋白之间的结构和机制相似性,通过替换开发出了“史上最小Cas9”,为开发更小、更强大、更活跃的新一代基因组编辑工具提供了新起点。2020年诺贝尔化学奖得主Jennifer A. Doudna教授团队[76]在噬菌体中发现了多类CRISPR/Cas系统,其中一些来自噬菌体的Casλ酶(属于Ⅴ型,仅700多个氨基酸大小)能够编辑植物和人类细胞基因组,并且兼顾了小型化和高编辑效率。未来多种改造方法、改造方式的快速发展以及采用系统性的改造策略将会进一步提升CRISPR/Cas系统工具的特异性和安全性。
总而言之,CRISPR/Cas系统的发现以及功能性开发,是基因编辑、基因调控等领域的革命性突变,是更好地认识世界和改造世界的一种有力工具,将会极大促进生命科学、生物工程以及农作物育种等领域的发展。
| [1] |
GASIUNAS G, BARRANGOU R, HORVATH P, SIKSNYS V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39): E2579-E2586.
|
| [2] |
CARROLL D. Genome engineering with zinc-finger nucleases. Genetics, 2011, 188(4): 773-782. DOI:10.1534/genetics.111.131433
|
| [3] |
NEMUDRYI AA, VALETDINOVA KR, MEDVEDEV SP, ZAKIAN SM. TALEN and CRISPR/Cas genome editing systems: tools of discovery. Acta Naturae, 2014, 6(3): 19-40. DOI:10.32607/20758251-2014-6-3-19-40
|
| [4] |
GAJ T, GERSBACH CA, BARBAS CF, 3RD. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology, 2013, 31(7): 397-405. DOI:10.1016/j.tibtech.2013.04.004
|
| [5] |
WANG HX, LI M, LEE CM, CHAKRABORTY S, KIM HW, BAO G, LEONG KW. CRISPR/Cas9-based genome editing for disease modeling and therapy: challenges and opportunities for nonviral delivery. Chemical Reviews, 2017, 117(15): 9874-9906. DOI:10.1021/acs.chemrev.6b00799
|
| [6] |
JIANG W, BIKARD D, COX D, ZHANG F, MARRAFFINI LA. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508
|
| [7] |
QI L S, LARSON MH, GILBERT LA, DOUDNA JA, WEISSMAN JS, ARKIN AP, LIM WA. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022
|
| [8] |
GILBERT LA, LARSON MH, MORSUT L, LIU Z, BRAR GA, TORRES SE, STERN-GINOSSAR N, BRANDMAN O, WHITEHEAD EH, DOUDNA JA, LIM WA, WEISSMAN JS, QI LS. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 154(2): 442-451. DOI:10.1016/j.cell.2013.06.044
|
| [9] |
DREISSIG S, SCHIML S, SCHINDELE P, WEISS O, RUTTEN T, SCHUBERT V, GLADILIN E, METTE MF, PUCHTA H, HOUBEN A. Live-cell CRISPR imaging in plants reveals dynamic telomere movements. Plant Journal, 2017, 91(4): 565-573. DOI:10.1111/tpj.13601
|
| [10] |
KANG JG, PARK JS, KO JH, KIM YS. Regulation of gene expression by altered promoter methylation using a CRISPR/Cas9-mediated epigenetic editing system. Scientific Reports, 2019, 9(1): 1-12. DOI:10.1038/s41598-018-37186-2
|
| [11] |
MALI P, YANG LH, ESVELT KM, AACH J, GUELL M, DICARLO JE, NORVILLE JE, CHURCH GM. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033
|
| [12] |
SHMAKOV S, SMARGON A, SCOTT D, COX D, PYZOCHA N, YAN W, ABUDAYYEH OO, GOOTENBERG JS, MAKAROVA KS, WOLF YI, SEVERINOV K, ZHANG F, KOONIN EV. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology, 2017, 15(3): 169-182. DOI:10.1038/nrmicro.2016.184
|
| [13] |
BARRANGOU R, GERSBACH CA. Expanding the CRISPR toolbox: targeting RNA with Cas13b. Molecular Cell, 2017, 65(4): 582-584. DOI:10.1016/j.molcel.2017.02.002
|
| [14] |
CONG L, RAN FA, COX D, LIN SL, BARRETTO R, HABIB N, HSU PD, WU XB, JIANG WY, MARRAFFINI LA, ZHANG F. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143
|
| [15] |
KOMOR AC, KIM YB, PACKER MS, ZURIS JA, LIU DR. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946
|
| [16] |
REES HA, LIU DR. Base editing: precision chemistry on the genome and transcriptome of living cells. Nature Reviews Genetics, 2018, 19(12): 770-788. DOI:10.1038/s41576-018-0059-1
|
| [17] |
GAUDELLI NM, KOMOR AC, REES HA, PACKER MS, BADRAN AH, BRYSON DI, LIU DR. Programmable base editing of A. T to G. C in genomic DNA without DNA cleavage. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644
|
| [18] |
LIN Q, ZONG Y, XUE C, WANG S, JIN S, ZHU Z, WANG Y, ANZALONE AV, RAGURAM A, DOMAN JL, LIU DR, GAO C. Prime genome editing in rice and wheat. Nature Biotechnology, 2020, 38(5): 582-585. DOI:10.1038/s41587-020-0455-x
|
| [19] |
ANZALONE AV, KOBLAN LW, LIU DR. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nature Biotechnology, 2020, 38(7): 824-844. DOI:10.1038/s41587-020-0561-9
|
| [20] |
SANDER JD, JOUNG JK. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology, 2014, 32(4): 347-355. DOI:10.1038/nbt.2842
|
| [21] |
GILBERT LA, HORLBECK MA, ADAMSON B, VILLALTA JE, CHEN Y, WHITEHEAD EH, GUIMARAES C, PANNING B, PLOEGH HL, BASSIK MC, QI LS, KAMPMANN M, WEISSMAN JS. Genome-scale CRISPR-mediated control of gene repression and activation. Cell, 2014, 159(3): 647-661. DOI:10.1016/j.cell.2014.09.029
|
| [22] |
TSAI SQ, WYVEKENS N, KHAYTER C, FODEN JA, THAPAR V, REYON D, GOODWIN MJ, ARYEE MJ, JOUNG JK. Dimeric CRISPR RNA-guided Fokl nucleases for highly specific genome editing. Nature Biotechnology, 2014, 32(6): 569-576. DOI:10.1038/nbt.2908
|
| [23] |
BAO Z, XIAO H, LIANG J, ZHANG L, XIONG X, SUN N, SI T, ZHAO H. Homology-integrated CRISPR-Cas (HI-CRISPR) system for one-step multigene disruption in Saccharomyces cerevisiae. ACS Synthetic Biology, 2015, 4(5): 585-594. DOI:10.1021/sb500255k
|
| [24] |
XIE KB, MINKENBERG B, YANG YN. Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(11): 3570-3575. DOI:10.1073/pnas.1420294112
|
| [25] |
JIANG Y, CHEN B, DUAN CL, SUN BB, YANG JJ, YANG S. Multigene editing in the Escherichia coli genome via the CRISPR-Cas9 system. Applied and Environmental Microbiology, 2015, 81(7): 2506-2514. DOI:10.1128/AEM.04023-14
|
| [26] |
ZHANG YP, WANG J, WANG ZB, ZHANG YM, SHI SB, NIELSEN J, LIU ZH. A gRNA-tRNA array for CRISPR-Cas9 based rapid multiplexed genome editing in Saccharomyces cerevisiae. Nature Communications, 2019, 10(1): 1053. DOI:10.1038/s41467-019-09005-3
|
| [27] |
HORWITZ AA, WALTER JM, SCHUBERT MG, KUNG SH, HAWKINS K, PLATT DM, HERNDAY AD, MAHATDEJKUL-MEADOWS T, SZETO W, CHANDRAN SS, NEWMAN JD. Efficient multiplexed integration of synergistic alleles and metabolic pathways in yeasts via CRISPR-Cas. Cell Systems, 2015, 1(1): 88-96. DOI:10.1016/j.cels.2015.02.001
|
| [28] |
WANG LY, DENG AH, ZHANG Y, LIU SW, LIANG Y, BAI H, CUI D, QIU QD, SHANG XL, YANG Z, HE XP, WEN TY. Efficient CRISPR-Cas9 mediated multiplex genome editing in yeasts. Biotechnology for Biofuels, 2018, 11: 1-16. DOI:10.1186/s13068-017-1003-x
|
| [29] |
GUO SY, GAO G, ZHANG CZ, PENG G. Multiplexed genome editing for efficient phenotypic screening in Zebrafish. Veterinary Sciences, 2022, 9(2): 92. DOI:10.3390/vetsci9020092
|
| [30] |
WANG Y, LIU Y, LIU J, GUO YM, FAN LW, NI XM, ZHENG XM, WANG M, ZHENG P, SUN JB, MA YH. MACBETH: Multiplex automated Corynebacterium glutamicum base editing method. Metabolic Engineering, 2018, 47: 200-210. DOI:10.1016/j.ymben.2018.02.016
|
| [31] |
WANG MG, MAO YF, LU YM, WANG ZD, TAO XP, ZHU JK. Multiplex gene editing in rice with simplified CRISPR-Cpf1 and CRISPR-Cas9 systems. Journal of Integrative Plant Biology, 2018, 60(8): 626-631. DOI:10.1111/jipb.12667
|
| [32] |
CHEN FB, LIAN M, MA BX, GOU SX, LUO X, YANG KM, SHI H, XIE JK, GE WK, OUYANG Z, LAI CD, LI N, ZHANG QJ, JIN Q, LIANG YH, CHEN T, WANG JW, ZHAO XZ, LI L, YU MY, et al. Multiplexed base editing through Cas12a variant-mediated cytosine and adenine base editors. Communications Biology, 2022, 5(1): 1163. DOI:10.1038/s42003-022-04152-8
|
| [33] |
FENG X, ZHAO DD, ZHANG XL, DING X, BI CH. CRISPR/Cas9 assisted multiplex genome editing technique in Escherichia coli. Biotechnology Journal, 2018, 13(9): e1700604. DOI:10.1002/biot.201700604
|
| [34] |
AO X, YAO Y, LI T, YANG TT, DONG X, ZHENG ZT, CHEN GQ, WU Q, GUO YY. A multiplex genome editing method for Escherichia coli based on CRISPR-Cas12a. Frontiers in Microbiology, 2018, 9: 2307. DOI:10.3389/fmicb.2018.02307
|
| [35] |
LI L, WEI KK, ZHENG GS, LIU XC, CHEN SX, JIANG WH, LU YH. CRISPR-Cpf1-assisted multiplex genome editing and transcriptional repression in Streptomyces. Applied and Environmental Microbiology, 2018, 84(18): e00827-18.
|
| [36] |
FERREIRA R, SKREKAS C, NIELSEN J, DAVID F. Multiplexed CRISPR/Cas9 genome editing and gene regulation using Csy4 in Saccharomyces cerevisiae. Acs Synthetic Biology, 2018, 7(1): 10-15. DOI:10.1021/acssynbio.7b00259
|
| [37] |
MANS R, van ROSSUM HM, WIJSMAN M, BACKX A, KUIJPERS NGA, van den BROEK M, DARAN-LAPUJADE P, PRONK JT, van MARIS AJA, DARAN JMG. CRISPR/Cas9: a molecular Swiss army knife for simultaneous introduction of multiple genetic modifications in Saccharomyces cerevisiae. Fems Yeast Research, 2015, 15(2): fov004.
|
| [38] |
SWIAT MA, DASHKO S, DEN RIDDER M, WIJSMAN M, van der OOST J, DARAN JM, DARAN-LAPUJADE P. FnCpf1: a novel and efficient genome editing tool for Saccharomyces cerevisiae. Nucleic Acids Research, 2017, 45(21): 12585-12598. DOI:10.1093/nar/gkx1007
|
| [39] |
KLOMPE SE, VO PLH, HALPIN-HEALY TS, STERNBERG SH. Transposon-encoded CRISPR-Cas systems direct RNA-guided DNA integration. Nature, 2019, 571(7764): 219-225. DOI:10.1038/s41586-019-1323-z
|
| [40] |
STRECKER J, LADHA A, GARDNER Z, SCHMID-BURGK JL, MAKAROVA KS, KOONIN EV, ZHANG F. RNA-guided DNA insertion with CRISPR-associated transposases. Science, 2019, 365(6448): 48-53. DOI:10.1126/science.aax9181
|
| [41] |
ZHANG YW, SUN XM, WANG QZ, XU JQ, DONG F, YANG SQ, YANG JW, ZHANG ZX, QIAN Y, CHEN J, ZHANG J, LIU YM, TAO RS, JIANG Y, YANG JJ, YANG S. Multicopy chromosomal integration using CRISPR-associated transposases. Acs Synthetic Biology, 2020, 9(8): 1998-2008. DOI:10.1021/acssynbio.0c00073
|
| [42] |
ZHANG YW, YANG JW, YANG SQ, ZHANG JZ, CHEN J, TAO RS, JIANG Y, YANG JJ, YANG S. Programming cells by multicopy chromosomal integration using CRISPR-associated transposases. Crispr Journal, 2021, 4(3): 350-359. DOI:10.1089/crispr.2021.0018
|
| [43] |
YANG SQ, ZHANG YW, XU JQ, ZHANG J, ZHANG JZ, YANG JJ, JIANG Y, YANG S. Orthogonal CRISPR-associated transposases for parallel and multiplexed chromosomal integration. Nucleic Acids Research, 2021, 49(17): 10192-10202. DOI:10.1093/nar/gkab752
|
| [44] |
WANG HH, ISAACS FJ, CARR PA, SUN ZZ, XU G, FOREST CR, CHURCH GM. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460(7257): 894-U133. DOI:10.1038/nature08187
|
| [45] |
ISAACS FJ, CARR PA, WANG HH, LAJOIE MJ, STERLING B, KRAAL L, TOLONEN AC, GIANOULIS TA, GOODMAN DB, REPPAS NB, EMIG CJ, BANG D, HWANG SJ, JEWETT MC, JACOBSON JM, CHURCH GM. Precise manipulation of chromosomes in vivo enables genome-wide codon replacement. Science, 2011, 333(6040): 348-353. DOI:10.1126/science.1205822
|
| [46] |
WARNER JR, REEDER PJ, KARIMPOUR-FARD A, WOODRUFF LBA, GILL RT. Rapid profiling of a microbial genome using mixtures of barcoded oligonucleotides. Nature Biotechnology, 2010, 28(8): 856-U138. DOI:10.1038/nbt.1653
|
| [47] |
GARST AD, BASSALO MC, PINES G, LYNCH SA, HALWEG-EDWARDS AL, LIU R, LIANG L, WANG Z, ZEITOUN R, ALEXANDER WG, GILL RT. Genome-wide mapping of mutations at single-nucleotide resolution for protein, metabolic and genome engineering. Nature Biotechnology, 2017, 35(1): 48-55. DOI:10.1038/nbt.3718
|
| [48] |
BASSALO MC, GARST AD, CHOUDHURY A, GRAU WC, OH EJ, SPINDLER E, LIPSCOMB T, GILL RT. Deep scanning lysine metabolism in Escherichia coli. Molecular Systems Biology, 2018, 14(11): e8371. DOI:10.15252/msb.20188371
|
| [49] |
ZHENG Y, KONG S, LUO S, CHEN C, CUI Z, SUN X, CHEN T, WANG Z. Improving furfural tolerance of Escherichia coli by integrating adaptive laboratory evolution with CRISPR-enabled trackable genome engineering (CREATE). ACS Sustainable Chemistry & Engineering, 2022, 10(7): 2318-2330.
|
| [50] |
LIU R, LIANG L, FREED EF, CHOUDHURY A, ECKERT CA, GILL RT. Engineering regulatory networks for complex phenotypes in E. coli. Nature Communications, 2020, 11(1): 4050. DOI:10.1038/s41467-020-17721-4
|
| [51] |
LIU R, LIANG L, CHOUDHURY A, GARST AD, ECKERT CA, OH EJ, WINKLER J, GILL RT. Multiplex navigation of global regulatory networks (MINR) in yeast for improved ethanol tolerance and production. Metabolic Engineering, 2019, 51: 50-58. DOI:10.1016/j.ymben.2018.07.007
|
| [52] |
LIU R, LIANG L, CHOUDHURY A, BASSALO MC, GARST AD, TARASAVA K, GILL RT. Iterative genome editing of Escherichia coli for 3-hydroxypropionic acid production. Metabolic Engineering, 2018, 47: 303-313. DOI:10.1016/j.ymben.2018.04.007
|
| [53] |
LIU R, LIANG L, GARST A D, CHOUDHURY A, NOGUE V SI, BECKHAM GT, GILL RT. Directed combinatorial mutagenesis of Escherichia coli for complex phenotype engineering. Metabolic Engineering, 2018, 47: 10-20. DOI:10.1016/j.ymben.2018.02.007
|
| [54] |
TONG YJ, WHITFORD CM, ROBERTSEN HL, BLIN K, JORGENSEN TS, KLITGAARD AK, GREN T, JIANG XL, WEBER T, LEE SY. Highly efficient DSB-free base editing for streptomycetes with CRISPR-BEST. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(41): 20366-20375. DOI:10.1073/pnas.1913493116
|
| [55] |
BANNO S, NISHIDA K, ARAZOE T, MITSUNOBU H, KONDO A. Deaminase-mediated multiplex genome editing in Escherichia coli. Nature Microbiology, 2018, 3(4): 423-429. DOI:10.1038/s41564-017-0102-6
|
| [56] |
DAVIS JR, WANG X, WITTE IP, HUANG TP, LEVY JM, RAGURAM A, BANSKOTA S, SEIDAH NG, MUSUNURU K, LIU DR. Efficient in vivo base editing via single adeno-associated viruses with size-optimized genomes encoding compact adenine base editors. Nature Biomedical Engineering, 2022, 6(11): 1272-1283. DOI:10.1038/s41551-022-00911-4
|
| [57] |
YUAN QC, GAO X. Multiplex base- and prime-editing with drive-and-process CRISPR arrays. Nature Communications, 2022, 13(1): 2771. DOI:10.1038/s41467-022-30514-1
|
| [58] |
ANZALONE AV, RANDOLPH PB, DAVIS JR, SOUSA AA, KOBLAN LW, LEVY JM, CHEN PJ, WILSON C, NEWBY GA, RAGURAM A, LIU DR. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature, 2019, 576(7785): 149-157. DOI:10.1038/s41586-019-1711-4
|
| [59] |
REIS AC, HALPER SM, VEZEAU GE, CETNAR DP, HOSSAIN A, CLAUER PR, SALIS HM. Simultaneous repression of multiple bacterial genes using nonrepetitive extra-long sgRNA arrays. Nature Biotechnology, 2019, 37(11): 1294-1301. DOI:10.1038/s41587-019-0286-9
|
| [60] |
MCCARTY NS, SHAW WM, ELLIS T, LEDESMA-AMARO R. Rapid assembly of gRNA arrays via modular cloning in yeast. Acs Synthetic Biology, 2019, 8(4): 906-910. DOI:10.1021/acssynbio.9b00041
|
| [61] |
ZHAO YW, LI L, ZHENG GS, JIANG WH, DENG ZX, WANG ZJ, LU YH. CRISPR/dCas9-mediated multiplex gene repression in Streptomyces. Biotechnology Journal, 2018, 13(9): e1800121. DOI:10.1002/biot.201800121
|
| [62] |
KACZMARZYK D, CENGIC I, YAO L, HUDSON EP. Diversion of the long-chain acyl-ACP pool in Synechocystis to fatty alcohols through CRISPRi repression of the essential phosphate acyltransferase PlsX. Metabolic Engineering, 2018, 45: 59-66. DOI:10.1016/j.ymben.2017.11.014
|
| [63] |
ZHANG X, WANG J, CHENG Q, ZHENG X, ZHAO G, WANG J. Multiplex gene regulation by CRISPR-ddCpf1. Cell Discovery, 2017, 3: 17018.
|
| [64] |
CAMPA CC, WEISBACH NR, SANTINHA AJ, INCARNATO D, PLATT RJ. Multiplexed genome engineering by Cas12a and CRISPR arrays encoded on single transcripts. Nature Methods, 2019, 16(9): 887-893. DOI:10.1038/s41592-019-0508-6
|
| [65] |
CHENG AW, WANG H, YANG H, SHI L, KATZ Y, THEUNISSEN TW, RANGARAJAN S, SHIVALILA CS, DADON DB, JAENISCH R. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Research, 2013, 23(10): 1163-1171. DOI:10.1038/cr.2013.122
|
| [66] |
LIAN J, HAMEDIRAD M, HU S, ZHAO H. Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system. Nature Communications, 2017, 8(1): 1688. DOI:10.1038/s41467-017-01695-x
|
| [67] |
LIAN JZ, SCHULTZ C, CAO MF, HAMEDIRAD M, ZHAO HM. Multi-functional genome-wide CRISPR system for high throughput genotype-phenotype mapping. Nature Communications, 2019, 10(1): 5794. DOI:10.1038/s41467-019-13621-4
|
| [68] |
WANG T, GUAN C, GUO J, LIU B, WU Y, XIE Z, ZHANG C, XING XH. Pooled CRISPR interference screening enables genome-scale functional genomics study in bacteria with superior performance. Nature Communications, 2018, 9(1): 2475. DOI:10.1038/s41467-018-04899-x
|
| [69] |
ROUSSET F, CUI L, SIOUVE E, BECAVIN C, DEPARDIEU F, BIKARD D. Genome-wide CRISPR-dCas9 screens in E. coli identify essential genes and phage host factors. PLos Genetics, 2018, 14(11): e1007749. DOI:10.1371/journal.pgen.1007749
|
| [70] |
SCHMIDT R, STEINHART Z, LAYEGHI M, FREIMER JW, BUENO R, NGUYEN VQ, BLAESCHKE F, YE CJ, MARSON A. CRISPR activation and interference screens decode stimulation responses in primary human T cells. Science, 2022, 375(6580): eabj4008. DOI:10.1126/science.abj4008
|
| [71] |
SANSON KR, HANNA RE, HEGDE M, DONOVAN KF, STRAND C, SULLENDER ME, VAIMBERG EW, GOODALE A, ROOT DE, PICCIONI F, DOENCH JG. Optimized libraries for CRISPR-Cas9 genetic screens with multiple modalities. Nature Communications, 2018, 9(1): 5416. DOI:10.1038/s41467-018-07901-8
|
| [72] |
WANG Y, CHENG HJ, LIU Y, LIU Y, WEN X, ZHANG K, NI XM, GAO N, FAN LW, ZHANG ZH, LIU J, CHEN JZ, WANG LX, GUO YM, ZHENG P, WANG M, SUN JB, MA YH. In-situ generation of large numbers of genetic combinations for metabolic reprogramming via CRISPR-guided base editing. Nature Communications, 2021, 12(1): 678. DOI:10.1038/s41467-021-21003-y
|
| [73] |
LIU R, LIANG L, FREED EF, GILL RT. Directed evolution of CRISPR/Cas systems for precise gene editing. Trends in Biotechnology, 2021, 39(3): 262-273. DOI:10.1016/j.tibtech.2020.07.005
|
| [74] |
SAPOVAL N, AGHAZADEH A, NUTE MG, ANTUNES DA, BALAJI A, BARANIUK R, BARBERAN CJ, DANNENFELSER R, DUN C, EDRISI M, ELWORTH RAL, KILLE B, KYRILLIDIS A, NAKHLEH L, WOLFE CR, YAN Z, YAO V, TREANGEN TJ. Current progress and open challenges for applying deep learning across the biosciences. Nature Communications, 2022, 13(1): 1728. DOI:10.1038/s41467-022-29268-7
|
| [75] |
SCHULER G, HU CY, KE AL. Structural basis for RNA-guided DNA cleavage by IscB-omega RNA and mechanistic comparison with Cas9. Science, 2022, 376(6600): 1476-1481. DOI:10.1126/science.abq7220
|
| [76] |
AL-SHAYEB B, SKOPINTSEV P, SOCZEK KM, STAHL EC, LI Z, GROOVER E, SMOCK D, EGGERS AR, PAUSCH P, CRESS BF, HUANG CJ, STASKAWICZ B, SAVAGE DF, JACOBSEN SE, BANFIELD JF, DOUDNA JA. Diverse virus-encoded CRISPR-Cas systems include streamlined genome editors. Cell, 2022, 185(24): 4574-4586. DOI:10.1016/j.cell.2022.10.020
|
 2023, Vol. 39
2023, Vol. 39