中国科学院微生物研究所、中国微生物学会主办
文章信息
- 李杰奕, 佟函泽, 吴毅
- LI Jieyi, TONG Hanze, WU Yi
- 酵母基因组大尺度遗传操纵工具研究进展
- Tools for large-scale genetic manipulation of yeast genome
- 生物工程学报, 2023, 39(6): 2465-2484
- Chinese Journal of Biotechnology, 2023, 39(6): 2465-2484
- 10.13345/j.cjb.230278
-
文章历史
- Received: April 11, 2023
- Accepted: June 5, 2023
2. 天津大学 教育部合成生物学前沿科学中心和系统生物工程重点实验室, 天津 300072
2. Frontier Science Center for Synthetic Biology and Key Laboratory of Systems Bioengineering, Ministry of Education, Tianjin University, Tianjin 300072, China
酵母是人类研究和利用最受欢迎的模式生物之一。酵母具有相对简单的基因组结构以及遗传操纵便捷等优势,被广泛应用于细胞周期等基础生物学研究的各个领域[1]。1996年酿酒酵母(Saccharomyces cerevisiae)成为了第一个被完全测序的真核生物[2]。此外,大多数酵母的安全性都得到了美国食品和药物管理局的认可[3],部分工程酵母细胞工厂已被用于生产多种蛋白质[4]、药品[5]、生物燃料[6]、精细化学品[7]和其他增值产品[8]。遗传改造酵母基因组的能力推动了工业相关酵母菌株的研发,满足了日益增长的使用廉价原料进行化学生产的需求。
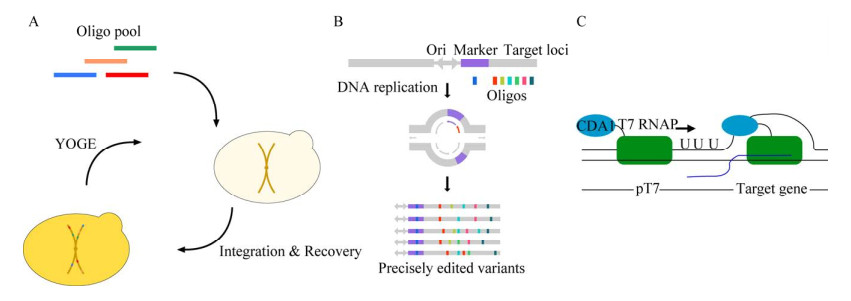
传统对于基因改造例如物理/化学诱变[9-10]、转座子突变[11]、易错PCR[12]等方式倾向于通过点突变的方式实现对基因的编辑,但点突变往往不足以实现某些复杂的基因调控,例如实现多基因共同表达、打造复杂的基因网络等。因此单个碱基或多个碱基随机性突变已无法适应当下对于菌株改造的需求,大尺度的基因组改造特别是10 kb以上的改造尺度以及多个基因靶点同时编辑改造就在基因组操纵领域显得尤为重要[13]。酵母基因组大尺度遗传操纵是一类利用位点特异性重组、规律成簇的间隔短回文重复序列(clustered regularly interspaced short palindromic repeats, CRISPR)等技术实现酵母基因组的大尺度编辑和改造的方法。其作用效果包括大片段DNA的敲除和整合、多靶点编辑、染色体核型改造等。通过该类工具可以实现对酵母细胞全基因组的编辑和调控,探究大尺度多基因变化与表型之间的关系。在本篇综述中,总结了最近开发的酵母高效大尺度基因组遗传操纵工具,根据其工作原理将这些工具分为重组酶介导的大尺度遗传操纵、核酸酶介导的大尺度遗传操纵、从头合成大片段DNA以及酵母其他大尺度操纵工具,并阐述了这些工具在产生大规模遗传变异中的效用,重点关注能够定位到单个菌株的理性改造工具(图 1)。
1 重组酶介导的大尺度遗传操纵位点特异性重组酶是一类酶,它们能够识别并结合到特定的DNA序列,并在该序列上发生DNA链切割和交换,从而实现基因组DNA的敲除、重组、倒置和置换等操作[18]。位点特异性重组系统因其具有高效精确的优点,在基因工程领域得到了广泛的应用[19]。重组酶按照其性质分类可分为酪氨酸重组酶和丝氨酸重组酶。在以往的研究中在酵母中建立的酪氨酸重组酶位点特异性重组系统有Cre/loxP、FLP/FRT、Vika/vox、Dre/rox和Rci/sfax101系统。此外ϕC31、ϕBT1、R4和BXB1等丝氨酸重组酶也在酵母中有应用(表 1)。
| Technical name | Technical features | Main application | References | |
| Cre/loxP | Recombinase recognition of loxP sites mediates gene inversion and deletion | Recovery of gene tags, exploration of gene and chromosome functions | [20-22] | |
| FLP/FRT | Recombinase recognition of FRT sites mediates gene inversion and deletion | Controls deletion of DNA fragments between recombination sites | [23] | |
| Vika/vox | Recombinase recognition of vox sites mediates gene inversion and deletion | Binding to SCRaMbLE controls deletion of DNA fragments between recombination sites | [24] | |
| Dre/rox | Recombinase recognition of Rox sites mediates gene inversion and deletion | Controls deletion of DNA fragments between recombination sites | [25-26] | |
| Rci/sfax101 | The recombinase recognizes the sfax site to mediate gene inversion with almost no deletion of the same site | Controls inversion of DNA fragments between recombination sites | [14] | |
| ϕC31 | The recombinase recognizes the corresponding attB and attP sites and controls the irreversible inversion and deletion of genes between the sites through the directionality of the sites | Deletion and inversion of DNA fragments between controlled recombination sites | [27] [28] |
|
| ϕBT1 | ||||
| R4 | ||||
| BXB1 | ||||
| SCRaMbLE | Heterozygous diploids and cross-species rearrangements | Rearrangement of synthetic and wild-type yeast haploids to obtain heterozygous diploid and cross-species diploid strains | Increased strain tolerance to caffeine and heat | [29] |
| Multiple rounds of iterative genome rearrangement | After one round of rearrangement, the high-yield spores were screened for the next round of rearrangement to quickly accumulate a large number of rearrangements | Increased carotenoid production | [30] | |
| Circular chromosomal rearrangement | Circular chromosome formation by telomere excision genomic rearrangement of synthetic circular chromosomes | Increased production of pro-deoxyviolet violacein, increased alkaline tolerance | [31] | |
| In vitro DNA rearrangement | Utilizing the interaction of Cre recombinase with DNA containing multiple loxPsym sites, efficient rearrangement of DNA can be achieved in test tubes | Exploration of gene function and interaction in β-carotene metabolic pathway | [32] | |
| Integrated genome rearrangement | Integrate exogenous metabolic pathways into yeast cells and optimize the chassis through rearrangement | Increases production of violacein and beta-carotene | [25] | |
位点特异性重组酶能够通过与重组位点结合切割和重组DNA链来实现基因组操纵。在天然酵母中,通过插入重组位点,位点特异性重组酶能够介导大片段DNA的删除、易位和倒置等操作,进而实现大尺度的基因组操纵。其中源于噬菌体P1的Cre/loxP系统(图 2A)在酵母中的应用十分广泛。Akada等[20]借助PCR引入Cre/loxP系统进行DNA片段的位点特异性删除。研究人员利用两端相重叠的引物,在PCR反应中扩增出目标基因的两侧,然后将其连接起来形成一个线性DNA分子。该分子经过酵母细胞质质粒的重组和重组酶的切割,可以实现无缝基因删除和标记回收从而开发出新的酵母标签循环系统。Delneri等[21]将2个loxP位点插入到不同的染色体上,然后使用Cre酵母转录激活因子诱导2个loxP序列的重组。由于这些loxP位点分别位于不同染色体上,它们的重组将导致2条染色体之间的DNA片段交换。借助Cre/loxP系统使得酿酒酵母产生了染色体间移位从而产生许多新的基因型和表型。然后,研究人员利用系统生物学的方法对这些基因型和表型进行了深入的分析,揭示酿酒酵母进化的关键因素和过程,从而探索酵母种间生殖隔离的影响因素。Naseeb等[22]为了探索基因顺序对表型、生殖隔离和基因表达的影响利用Cre/loxP系统构建了一个倒置和非倒置菌株的文库,产生了最大800 kb基因的倒位。其研究结果表明,大规模基因顺序的改变有一定可能性对酵母生长造成负面影响,并且一旦发生顺序变化,就会对转录组网络产生影响。
除了Cre/loxP系统外其他位点特异性重组系统也有在酵母中进行基因整合和敲除的例子。Park等[23]用于在酿酒酵母中利用FLP/FRT重组技术实现来删除特定的基因,建立了条件性基因删除系统。该系统在诱导表达9 h后基因切除效率可以达到96%。Karimova等[24]报道了一种新的高效、特异性的位点特异性的重组系统,称为Vika/vox系统。Vika重组酶是一种结构与Cre相似的蛋白质,能够识别和结合到Vox序列,并催化2个Vox序列之间的DNA重组。该团队使用Vika/vox系统成功地进行了基因插入、替换和删除等操作,并且尚未观察到不良的副作用或非特异性的DNA修饰。与Cre/loxP系统相比,Vika/vox系统在体外和体内都表现出更高的特异性、更高的重组效率和良好的温度适应性。Liu等[25]将Cre/loxP、Vika/vox、Dre/rox联用,可以在特定位点引入双链断裂,并在不同基因组区域之间重新组合这些片段,从而实现基因组重排。这种技术可以快速生成大量的遗传多样性,使得酿酒酵母细胞在不同条件下可以产生不同的代谢产物,从而更好地理解基因组的功能和相互作用。
DNA反转是一种基因重组的形式,它涉及基因组中的DNA片段在特定的位点上反转。这种反转通常由DNA酶催化完成,它们剪切DNA链并重新连接DNA分子的两端,使得DNA片段在基因组中的方向被反转。DNA反转是一种重要的基因重组过程,可以在调节基因表达、复制DNA序列、修复DNA损伤等方面发挥作用。Han等[14]基于细菌天然的Rci重组酶和sfxa101位点通过Rci重组酶的定向进化,筛选到特异性反转的Rci8突变体在酿酒酵母中构建位点特异性DNA反转系统,该系统只介导反向位点之间DNA片段的反转,而几乎不介导同向位点之间DNA片段的删除。基于Rci8/sfxa101在线性染色体上构建位点特异性DNA反转系统,发现该DNA反转系统在线性染色体上工作能够实现500 kb尺度的基因反转。Rci8/sfxa101系统的出现扩展了位点特异性重组系统在真核生物中的应用,为大尺度基因组遗传操纵提供了全新工具。
除了酿酒酵母外在非常规酵母中也有使用位点特异性重组酶进行基因敲除的例子。Fickers等[33]在解脂耶氏酵母(Yarrowia lipolytica)中同样构建了Cre/loxP系统,并使用该方法实现了解脂耶氏酵母多个基因的快速敲除。Song等[34]在多形汉逊酵母(Hansenula polymorpha)中利用Zeocin和MazF双筛选标记成功实现了对多形汉逊酵母基因的无痕敲除。Cre/loxP系统等重组酶介导的大尺度基因操纵工具在非常规酵母中的应用丰富了非常规酵母的靶向性基因操纵工具,使得靶向性基因敲除等操作更加便捷高效。
相比于酪氨酸重组酶,丝氨酸重组酶在酵母中的应用相对较少,可能是因为丝氨酸重组酶联会迅速,以至于联会复合体不稳定而难以检测[18],但是也有Thomason等[27]利用ϕC31在粟酒裂殖酵母(Schizosaccharomyces pombe)中进行基因的插入与整合,Xu等[28]在酿酒酵母中提供了ϕBT1、R4和BXB1等丝氨酸重组酶可供选择。这些丝氨酸重组酶也可以介导重组位点间DNA片段的删除。值得注意的是,上述位点特异性重组系统重组位点相互正交,为酵母基因组工程化改造提供了多样性选择。
1.2 重组酶介导的人工染色体大尺度遗传操纵基因组结构变异一直是一个具有挑战性的研究领域,近年来长读长测序技术的出现推动了这一领域的发展[35]。尽管如此,基因组结构变异文库的生成仍然十分困难。基因组的重排能产生数百kb甚至Mb尺度的染色体变化,包括大片段DNA的缺失、插入、重复、倒置和易位,人工合成染色体酿酒酵母的出现使得研究人员可以通过酵母基因组重排生成基因组结构变异文库。在合成酵母基因组计划(Sc2.0)中,研究人员在人工合成的酿酒酵母染色体非必需基因终止密码子后3 bp处添加了loxPsym位点[36]。通过Cre重组酶特异性识别并介导不同loxPsym位点之间进行重组(图 2B),建立了人工合成染色体酿酒酵母基因组重排(synthetic chromosome rearrangement and modification by loxP-mediated evolution, SCRaMbLE)[37]。相比于传统loxP位点loxPsym位点序列结构对称,2个loxPsym位点之间的DNA反转和删除理论概率相同[38]。在合成型酵母染色体上插入数百个loxPsym使得合成型酿酒酵母染色体具有重排的随机性和灵活性。通过SCRaMbLE系统可以实现全基因组范围内loxPsym位点间的DNA片段缺失、插入、重复、倒置和易位,这使得快速获得大量具有基因型和表型多样性的酵母菌株成为可能[39]。SCRaMbLE系统的建立使得酵母基因组大尺度改造能力迈上了新台阶。
在利用SCRaMbLE系统进行大尺度基因组改造过程中必需基因的删除会导致菌株的死亡。Shen等[29]通过将含有人工合成染色体的酵母单倍体与来自酵母菌株库的野生型酿酒酵母和野生型奇异酵母单倍体交配,获得了杂合二倍体和跨物种二倍体。这种重排方式被称为杂合二倍体与跨物种基因组重排,其优势在于能降低因大尺度改造而导致的菌株死亡。通过跨物种二倍体的基因组重排,研究人员成功获得咖啡因耐受性提升的重排菌株,并全基因组测序和验证实验定位到与咖啡因耐受性的提升有关基因。此外,杂合二倍体与跨物种基因组重排策略还成功得到了耐受42 ℃高温的酵母菌株yYW166。
多轮迭代重排能持续驱动酵母进化从而产生更大尺度的基因组变化。Jia等[30]将一轮重排后筛选得到的高产二倍体酵母的高产孢子与其他人工合成染色体的单倍体酵母交配,再进行下一轮基因组重排,这样的多轮循环可以快速积累大量重排。通过这种方式验证了YEL013W的缺失对于提升类胡萝卜素产量的有效性,最终使类胡萝卜素产量提升38.8倍。经过5轮迭代重排累积了多个染色体重排产生了超过500 kb的基因组变化使生产菌株快速进化。
拓扑结构变异影响2个loxP位点之间的空间位阻,与线性染色体相比,环形染色体在重排多样性方面可能会带来不一样的效果。Wang等[31]对环形合成型Ⅴ号染色体进行重排时发现与线性合成型Ⅴ号染色体相比,重排后的环状染色体出现大量的DNA片段复制现象。5轮迭代后,合成型环形染色体增加的长度多达原长的1倍以上。此外,合成型环形染色体在基因组重排过程中还产生了非整倍的菌株。借助合成型环形染色体的基因组重排技术可以有效扩大酵母染色体结构变异的规模和数量。其对含有合成型环形Ⅴ号染色体的紫色杆菌素前体物(purple bacillin precursor, PDV)生产菌进行多轮诱导重排,一共得到53个新的结构连接。将其中29个新的连接位点整合到酿酒酵母中使PDV产量增加了约3.48倍,表明环形染色体重排产生的全新结构变异与PDV的产量上升有关。
在SCRaMbLE系统中重组酶Cre的连续作用和泄漏表达可能会降低基因组的稳定性。在体外进行重组反应能够规避这一问题。此外,DNA文库的体外构建提供了一种研究基因型-表型关系的新思路,对于优化生物合成途径有重要意义[40]。Wu等[32]设计开发了一种体外DNA重排技术,利用Cre重组酶与包含多个loxPsym位点DNA的相互作用,可以在试管内实现DNA的高效重排。研究者采用纯化的Cre重组酶对编码多个loxPsym位点的DNA结构进行基于重排的优化。通过重排技术确定了94个独特的代谢通路结构,这些通路展现了crtI、crtE、crtYB和tHMG1在内的4个基因基于多个loxPsym位点发生的可测得的重排事件。将重排后质粒直接转化进酿酒酵母进行检测,得到了包括删除、反转和复制在内的17种独特的β-胡萝卜素代谢途径结构。最终使β-胡萝卜素产量增加了5.1倍,并发现了crtI基因是该代谢通路的关键基因,该基因的复制和反转都可以使β-胡萝卜素产量提高,为快速实现异源代谢通路的优化提供了一种有效方法。
除了利用酵母自身的基因进行重排外,将外源基因引入人工合成染色体酵母的重排过程可以实现外源代谢路径和底盘的组合优化。Liu等[25]开发了一种基于重组酶的整合基因组重排技术(称为“SCRaMbLE-in”)。在体外将调节元件、选定的重组酶和靶途径DNA混合后,调节元件可通过重组酶整合进靶重组位点上以产生一个组装代谢路径库。随后借助基因组重排技术将组装路径整合到人工合成染色体酵母基因组中,同时重组酶可诱导底盘细胞发生基因组重排。利用SCRaMbLE-in可以只通过两步反应,达到同时实现基因表达的多样化和底盘细胞的工程化的效果,快速优化宿主以更好地适应异源途径以及优化外源途径在细胞内表达,最终实现快速代谢工程。
2 核酸酶介导的大尺度遗传操纵基于核酸内切酶的基因编辑技术包括归巢核酸内切酶(meganucleases, MegNs)技术、锌指蛋白核酸酶(zinc finger nucleases, ZFNs)技术、转录激活因子样效应物核酸(transcription activator-like effector nucleases, TALENs)技术和CRISPR相关技术等[41-44]。通过对目标基因序列造成双链断裂(double strand break, DSB),进而对其进行定点基因组编辑。由于DSB在酵母中具有致命性,在完成双链断裂处的正确修复后细胞才能正常生长。因此这些方法理论上可用于无筛选标签修饰对目标位点进行精确改造,其中MegNs技术和CRISPR相关技术能够实现酵母基因组大尺度操纵(表 2)。
| Technical name | Maximum edit size | Technical features | Manipulation effect | Main application | References | |
| MegNs | 35 kb | Homing endonucleases introduce DSBs through target sequence targeting by recognizing specific DNA sequences | Accurate editing and high efficiency but lack of target sites | Multigene assembly, circular chromosome linearization | [45] | |
| CRISPR/Cas | Polygenic inactivation | 8 genes | Targeting multiple genes in tandem with tRNA sequences and multiple gRNAs to generate DSBs | Editing is precise and the application range is wide, but needs to construct multiple gRNA | Increase the production of free fatty acids | [46] |
| Integration of large fragments | 35 kb | Introduction of double-strand breaks on target chromosomes by the cas9 system facilitates multigene assembly and targeted integration | Editing is precise and the application range is wide | Genome integration of carotenoid biosynthesis pathway, glycolysis pathway, etc | [47] | |
| Multi-site mutation | Within 10 positions | Argeted by nCas9 at the locus of interest and gapped, Pol I-5M synthesizes new strands and cleaves displaced strands with low fidelity | Wide range of applications, good base diversity of editing sites, but narrow editing window | Increase the diversity of endogenous genes | [48] | |
| Loss of heterozygosity | 20 kb | Targeting a chromosome in heterozygous diploids using PAMs polymorphisms between two homologous chromosomes | Multi-site editing can be achieved but the editing efficiency is low | Obtain a large number of rearranged phenotypes | [49] | |
| Production of aneuploidy | 600 kb | Generation of DSBs mediated by CRISPR-targeted pericentromere elimination of entire chromosomes leading to aneuploidy | Large editing scale and wide application range | Synthetic X chromosome eliminated | [50] | |
| Karyotypic restructuring | 10 Mb | Reduction of the number of yeast chromosomes by CRISPR-mediated end-to-end chromosome fusion into chromosome loops | Large editing scale but high cost and time-consuming | Provides a potential research model for studying ring chromosome disorders | [51-52] | |
MegNs是具有较大识别位点特征的脱氧核糖核酸内切酶,其可识别超过14个碱基对的双链DNA序列,如酿酒酵母中的I-Sce I归巢核酸内切酶可以识别18 bp的序列[53]。为了实现对目标基因的编辑,通常需要将归巢核酸内切酶识别位点先引入宿主的染色体目标位置,随后MegNs可在基因组目标位点附近引入DSB,进而提高基因编辑效率。Kuijpers等[45]利用I-Sce I在目标染色体位点引入双链断裂,使超过35 kb基因片段的整合到目标位点并且整合效率非常高。Mitchell等[15]利用I-Sce I归巢核酸内切酶创造了能用于酿酒酵母的合成生物学装置“telomerator”,其中心是I-Sce I归巢核酸内切酶的识别位点,在完成切割后使识别位点两端端粒暴露出来(图 3A)。将这一装置插入到环形染色体特定位置可以将环状染色体线性化从而介导染色体核型改造,从而开发出来一种可以探测染色体端粒、着丝粒和复制起点等功能元件之间的空间关系的新工具。
|
| 图 3 核酸酶介导的大尺度遗传操纵[15] Fig. 3 Large-scale manipulation mediated by endonucleases[15]. A:Telomerator技术. B:CRISPR-Cas9介导的大规模操纵基因组的机制. CRISPR-Cas9可以通过HDR或NHEJ修复诱导各种重排(缺失、倒置、复制和易位) A: Telomerator technology. B: Mechanisms of CRISPR-Cas9-mediated large scale manipulation of the genome. Various rearrangements (deletion, inversion, duplication and translocation) can be induced by CRISPR-Cas9 through HDR or NHEJ repairing. |
| |
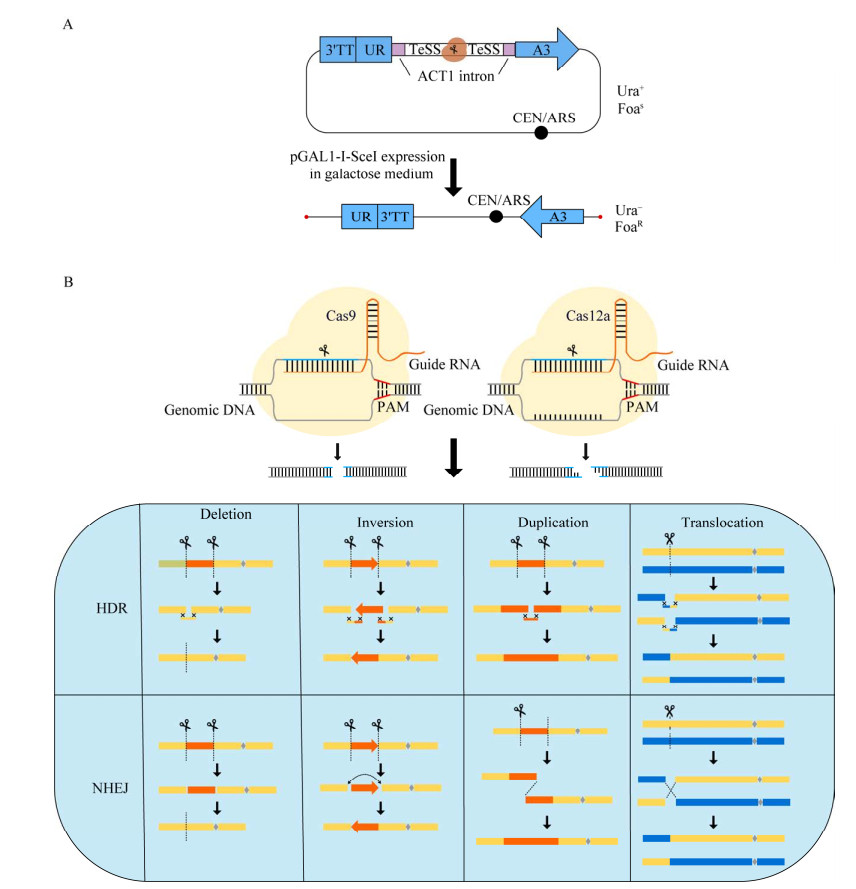
CRISPR/Cas基因编辑技术是近年兴起的由小分子RNA介导的一种靶向基因组编辑的新技术[54]。已成为诱导靶向基因编辑的一种简便而有效的方案。CRISPR/Cas系统是一种适应性免疫系统,源于细菌和古菌中用来抵御外来核酸的免疫防御机制[55]。目前,代谢工程中的大多数基因操作都可以利用CRISPR/Cas系统实现[56]。CRISPR/Cas系统通过工程化的单链引导RNA (single guide RNA, sgRNA)引导Cas核酸酶效应体形成复合物,从而切割前间隔序列邻近基序(protospacer adjacent motif, PAM)序列上游3 bp处的基因组,造成DSB[57]。当供体DNA存在与基因组DNA同源序列时,酿酒酵母便可通过DSB实现对基因组的特异性基因编辑。通过CRISPR/Cas可实现酵母大片段DNA的敲除和整合、多位点碱基突变、杂合性缺失、非整倍体的产生以及染色体核型改造(图 3B)。
利用CRISPR/Cas系统可以实现无筛选标签的情况对酿酒酵母基因和大片段DNA进行敲除[46]。Jakočiūnas等[58]开发了用于酿酒酵母基因组多基因敲除的CRISPR/Cas9系统,最多可以同时对5个不同的基因组位点进行敲除。通过共转化sgRNA质粒和相应的线性同源重组片段,单个基因至5个基因的编辑效率达到50%–100%,工程菌株甲羟戊酸产量比野生型菌株提高了41倍。Zhang等[59]将tRNA序列与多个gRNA串联构建了GTR-CRISPR (gRNA-tRNA-array CRISPR/Cas9)系统,同时敲除8个基因的效率达到87%,极大地提高了基因编辑效率。除了Cas9外,在酵母中利用Cas12a也可以实现大片段的删除。Li等[60]使用CRISPR/Cas12a (Cpf1)系统构建质粒以组成型表达CrRNA阵列从酿酒酵母染色体上删除了trm10和rex4这2个基因之间的38 kb的非必需大DNA片段,证明CRISPR/Cas12a (Cpf1)系统可用于酿酒酵母的基因组简化。
CRISPR/Cas系统能够产生靶位点的DSB结合酵母自身的同源重组能力而实现大片段DNA的整合,Eauclaire等[47]利用CRISPR/Cas9系统成功将17个编码β-胡萝卜素合成的基因片段一次性整合到酿酒酵母基因组中,相邻DNA片段仅有50 bp互补,证明了CRISPR/Cas9系统与同源重组相结合的有效性。Verwaal等借助CRISPR/Cas12a系统同时将3个类胡萝卜素基因表达框整合到3个不同的基因组位点[61]。此外,CRISPR/Cas系统还可以靶向整合至酿酒酵母体内的新染色体,Postma等[62-63]依靠酿酒酵母强大的同源重组能力从头组装了两条新染色体,并将新染色体作为靶向目标,一次组装了16个DNA片段约35 kb的糖酵解途径以及花青素的生物合成途径。比较I-Sce I,CRISPR/Cas系统在提高同源重组效率的同时能实现多位点无痕迹的基因组整合,应用更广泛[64]。
复杂代谢网络的建立往往需要多基因协同作用,因此多靶点编辑成为未来基因编辑领域重要发展方向。2020年,Tou等[48]在酿酒酵母中建立了yEvolvR (yeast EvolvR)系统,研究人员着眼于将CRISPR技术与高突变率DNA聚合酶结合,来实现基因组中特定位点的有序和定向的多靶点编辑。该系统通过gRNA定位靶基因,由nCas9蛋白在目标基因座上形成缺口,再由融合的高突变率DNA聚合酶PolI合成新链并切割置换的链,将目标位点的突变率提高了12 434倍。yEvolvR技术另一个优势是能够有效提高内源基因的多样性,当gRNA双重靶向URA3和canI的条件下,可还原URA3中的无义突变并使canI失活。
像基因组重排技术一样,CRISPR也生成染色体间重组事件,进而产生杂合性缺失。CRISPR介导的杂合性缺失事件需要在杂合子二倍体中诱导DSB。在Sadhu等[49]研究中,gRNAs被设计为仅靶向杂合二倍体中的一条染色体,使用两条同源染色体之间的PAMs多态性使得特定的DSB可以在不同的交换位点诱导不同的杂合性丢失事件。研究人员使用靶向两条酵母染色体VII之一的左臂的95个gRNA,获得了384个杂合性缺失系,并分析了12种不同条件下的生长率,可以很快将特定的遗传变异与特定的生长表型联系起来。
染色体层面的大尺度操纵使得人们对于染色体结构和功能的理解进一步加深,在酵母中借助Cas9可以介导非整倍体的产生和染色体核型改造。Xu等[50]通过CRISPR-Cas9,靶定着丝粒周围产生双链断裂,可以有效地消除整个酿酒酵母染色体。作为这种CRISPR-Cas9染色体驱动系统的概念验证实验,合成的酵母染色体X被完全消除,而带有绿色荧光蛋白基因或合成紫色杆菌素途径成分的对应野生型染色体X得以保留,为基因功能分析和遗传稳定性研究提供了新的工具和方法。Shao等和Luo等[51-52]将CRISPR-Cas9的应用扩展到酵母的核型改造。这些研究人员能够通过CRISPR介导的染色体端到端融合,将酵母染色体的数量从16条减少到2条,甚至1条。类似地,该单染色体酵母的核型被进一步改变,产生单个环状染色体。
除了上述酿酒酵母中的应用外,CRISPR/Cas9系统还能实现非常规酵母的基因整合、敲除和多靶点编辑等基因组改造。在巴斯德毕赤酵母(Pichia pastoris)中,Weninger等[65]使用不同的启动子组合对基因表达水平、Cas9蛋白质与RNA的比例和转录起始位点的选择等方面的优化,可以实现对多个基因座的同时编辑。使用这种优化方法,基于非同源末端修复引入插入缺失突变的效率接近100%。同时,CRISPR/Cas系统在乳酸克鲁维酵母(Kluyveromyces lactis)系统中也得到了开发和成功应用,研究者们使用CRISPR/Cas9系统成功地将6个目的基因整合到了3个靶向基因座上,从而使粘康酸合成通路的快速构建成为可能[66]。另外,在多形汉逊酵母中,Numamoto等利用CRISPR/Cas9系统进行多重基因组编辑,通过在启动子后添加靶向位点将多形酵母中的白藜芦醇生物合成途径基因整合到rDNA重复簇中。该研究结果表明,这种策略导致多拷贝表达的白藜芦醇产量为97 mg/L,比具有单一拷贝途径的基础菌株高出21倍[67]。
3 从头合成大片段DNA重组酶和核酸酶介导的大尺度遗传操纵工具倾向于在原有基因组基础上进行改造,这使得对于基因组的改造与操作受到了天然酵母基因组固有结构的限制。近年来,随着DNA合成技术和大片段组装技术不断发展使得从头合成大片段DNA成为对基因组结构和功能研究的全新方式[68]。从头合成大片段DNA提供了一种从零开始构建完全人工化基因组的可能性,研究人员可以实现定制化设计,包括精确的序列和结构调整,以及添加或删除特定基因等。从头合成大片段DNA帮助更好地理解基因组的结构和功能,为合成基因组学的深入开展提供借鉴与参考(表 3)。
| Technical name | Maximum edit size (kb) | Technical features | Advantage | Deficiency | Main application | References |
| Synthetic Saccharomyces cerevisiae chromosomes | 700 | Stepwise replacement of wild-type chromosomes with synthetic chromosomes by chemical synthesis and homologous recombination in yeast | Large-scale transformation | High cost and time-consuming | Rearrangement of artificial yeast chromosomes provides a rich yeast chassis | [69-76] |
| Artificially synthesized exogenous gene | 100 | DNA fragments and vectors use homologous recombination to form circular plasmids or integrate into the genome in Saccharomyces cerevisiae | Low cost, no additional auxiliary tools required | Self-recombination ability | Integrated pathways for xylose utilization and zeaxanthin biosynthesis | [77] |
| Artificial synthesis of yeast pan-genome | 200 | Collected 8 yeast pan-genome sequences to form a new chromosome | Large-scale transformation | High cost and time-consuming | Expanded range of available carbon sources | [78] |
人工合成酿酒酵母基因组计划(Sc2.0)是从头合成大片段DNA一个代表性例子。该计划旨在对酿酒酵母整个基因组进行重新设计与化学再造,围绕人工设计合成酿酒酵母长染色体及对生命系统的调控进行研究。该计划的重要目标是实现酿酒酵母12 mb的基因组人工全合成,这是人类第一次实现如此大规模的基因组的化学合成,真正意义上从读取自然生命信息发展到写出人工生命信息阶段,颠覆了现有生命科学研究的范式[79]。迄今为止,已成功设计与合成出8.5条完整的人工合成染色体(synI, synII, synIII, synIV, synV, synVII, synIXR, synX, synXII)[69-76]。
Sc2.0项目采用多重规则指导重新设计天然酵母基因组,目的是在保持与野生型菌株表型尽可能相似的前提下提高基因组稳定性和灵活性。该项目通过删除自然重复和不稳定的元素,如转座子、tRNA和亚端粒等,来改善基因组的稳定性。此外,将所有275个核tRNA基因从其天然基因座中移除,并重新定位在第17个核染色体上,有助于更深入地理解高度复杂遗传结构的构建和维护机制[40]。为增加基因组的灵活性,Sc2.0项目采用同义密码子重新编码和高频的同义密码子替换稀有密码子的方法。此外,在合成酿酒酵母基因组过程中,每个非必需基因下游3 bp位置会添加loxPsym位点[69],通过表达Cre重组酶,合成的染色体可以被随机“洗牌”,从而引起广泛的基因组改变。这一创新性设计极大拓展了对于酵母基因组改造的尺度,对于后续酵母基因组的研究和底盘菌的开发具有重要的贡献。
在完成人工酵母基因组的设计后,研究人员采用高通量DNA从头合成技术、大片段DNA组装技术和染色体替换与融合技术开始进行人工酵母染色体的构建。Boeke团队[69]开发了一种标准化逐步策略,通过相应的合成DNA大片段替换原生染色体片段并利用营养标签辅助细胞表型筛选,最终获得人工合成染色体。戴俊彪团队[76]通过染色体分级组装的策略,在多个单倍体菌株中完成了对染色体不同区域大片段DNA的逐步替换,并利用同源染色体交叉互换的特性整合合成序列,快速获得完整的人工合成染色体。完成对人工基因组的初步构建后,研究人员进行了基因组测试和纠错,通过全基因组测序确定基因型,利用高通量染色体构象捕获技术(high-through chromosome conformation capture, Hi-C)进行染色体构象捕获表征基因组三维结构。表型测试包括在不同生长条件下对合成酵母菌株进行测试,以及竞争性生长实验。在synX染色体的初始设计合成过程中出现表型缺陷,Wu等[75]利用标签系统和混菌策略创建了“混菌PCR标签定位法”以定位和修复缺陷靶点,并挖掘出未知的酵母生物学新知识。Xie等[72]在构建Ⅴ号染色体时发现了34处变异,需要进行修正、整合、共转换和CRISPR/Cas9编辑等共22步,最终分离出一株具有“完美设计序列”的真核酵母染色体的菌株,并在不同培养条件下表现出良好的适应性。Shen等[74]通过“I-Sce I切割修复”的方法实现了合成型Ⅱ号染色体的缺陷纠正。
在践行人工合成酿酒酵母基因组计划过程发现和解析了大量未知的现象和信息,这些新知识可以用来快速提升基因组合成的能力和揭示影响细胞功能的重要因素。Sc2.0为合成更大的功能基因组提供参考,突破了底盘基因组对于大尺度操纵的限制,通过对合成染色体的拼接、插入、删除等操作,可以实现酵母基因组的大尺度重组和重构,从而了解生物进化的机制和规律。这对于理解生命起源、生物多样性和生态系统的演化等方面具有重要意义。随着DNA合成与装配技术的不断发展,基因组学的研究已经从“格物致知”迈向“建物致知”,为生命科学领域的进一步研究提供了全新的视角。
3.2 外源基因的大片段组装酿酒酵母同源重组能力十分强大,因此依靠其本身的同源重组能力可以实现大片段外源基因的整合。Shao等[77]创建出名为DNA组装器的新的组装方法使得研究人员能够通过酿酒酵母中的体内同源重组在一步中组装整个生化途径。DNA组装器可以在质粒或酵母染色体上以高效率快速组装功能性D-木糖利用途径和玉米黄质生物合成途径,实现了约100 kb由8个基因组成的一步组装。成为构建合成生物学,代谢工程和功能基因组学研究生化途径的有力工具。Kutyna等[78]研究人员通过对酿酒酵母不同品系的基因组序列进行比较和分析,并利用多种基因工程技术和遗传学方法,合成了一条新的微型染色体。该染色体包含了来自不同酵母品系的基因信息,并展现出了丰富的表型多样性。此外新的染色体还可以通过诱导重排产生结构变异进一步增加菌株的遗传多样性。
4 酵母其他大尺度操纵工具除了上述酵母基因组编辑工具外,酵母其他大尺度操纵工具也能在酵母基因组多个位点进行编辑(表 4)。例如,可以利用生物体自身同源重组介导的修复机制引入突变位点,通过引入含碱基突变的寡核苷酸建立突变文库。Church课题组[80]开发了一种在酿酒酵母中由单链寡核苷酸介导重组的方法,称为酵母寡核苷酸介导的基因组工程(yeast oligo-mediated genome engineering, YOGE)。通过敲除酵母中错配修复相关基因mlhI和msh2,过表达DNA重组酶突变体Rad51 (K342E)和Rad54蛋白,并通过对寡核苷酸长度、寡核苷酸加入量以及转化效率优化后,在3种不同的酿酒酵母中成功对基因组进行了修饰(图 4A)。并且YOGE可以进行迭代操作,每轮可产生10个细胞的基因组文库,对基因组进行多重修复。与之类似的真核多重自动化基因组工程(eukaryotic multiplex automated genome engineering, eMAGE)可在不导致DSB的情况下对酿酒酵母的多基因实现精确编辑修饰(图 4B)。通过在酿酒酵母中引入与复制叉的后随链互补的寡核苷酸,实现了大于40%的序列修饰效率。多次循环同时靶向基因内的多个位点提高了工程菌株突变效率,该方法产生了近100万个突变体。对β-胡萝卜素途径的启动子、编码序列和终止子进行编辑,获得了β-胡萝卜素产量不同的工程菌株[16]。基于寡核苷酸的多位点编辑工具在编辑位点数量上拥有突出优势,但突变效率会随单链DNA寡核苷酸突变序列的增加而降低且靶序列需要紧密接近复制起点限制了其大规模应用。
| Technical name | Maximum edit size | Technical features | Advantage | Deficiency | Main application | References |
| YOGE | 10 kb | Increased oligonucleotide mismatch rate by knockout of related mismatch repair genes and overexpression of recombinase genes | Many editing sites | Editing is inefficient | New tool for yeast genome engineering developed | [80] |
| eMAGE | More than 60 sites | Synthesize mutant ssDNA with modified annealing protein, and then anneal and integrate the mutant ssDNA into the genome | Fast editing speed and many editing positions | The target sequence needs to be close to the replication origin | Carotene metabolic pathway optimization | [16] |
| TRIDENT | Within 20 sites | T7 RNA polymerase targets the target DNA, and the fusion protein composed of modified cytidine deaminase and adenosine deaminase mutates the target region | Precise editing, large editing window, and good base diversity at editing sites | It is necessary to insert a recognition site and a termination site before the target region | Enhancing yeast pyrimethamine resistance | [17] |
T7聚合酶来引导特定的碱基编辑酶(APOBEC)能够精确地编辑DNA序列,从而实现类似CRISPR的突变或修饰DNA效果。Cravens等[17]描述了通过T7 RNAP实现靶向体内多样化(targeted In vivo diversification enabled by T7 RNAP, TRIDENT),这是一个以高于自然基因组错误率100万倍的突变率对目标的基因进行靶向、持续和可诱导多样化的平台(图 4C)。TRIDENT通过与T7 RNA聚合酶融合,将诱变酶靶向精确的基因位点,在靶向T7启动子的突变后产生突变窗口。定位于脱氨酶驱动突变位点的DNA修复因子能调节其靶向区域的突变多样性,使得该平台能够产生所有4种DNA核苷酸的突变情况且突变速度较快,能够应用于提高酵母对于部分抗生素的耐药性。相比于上文提到的yEvolvR而言TRIDENT拥有更大的编辑窗口和特异性,而且不会引起细胞非靶向位置突变。但是,TRIDENT的使用需要在编辑位点前插入T7聚合酶识别位点,在编辑复杂基因区域时受限。
5 总结与展望基因组大尺度遗传操纵具有重要的理论和应用价值,为酵母基因组功能研究和生物制造等领域的开发提供了巨大的潜力。从理论角度来看,酵母基因组大尺度遗传操纵相关技术能够实现对基因组级别的遗传和表达调控,为揭示基因型与表型之间的关系和基因调控机制提供了全新的思路,丰富了生物学的基础研究工具。从应用角度来看,酵母基因组大尺度遗传操纵相关技术为酵母工业的遗传调控和优化提供了新方法,可以利用该类技术构建或筛选出更为适应不同工业环境的酵母菌株,提高酵母生产的效率和质量。同时,酵母基因组大尺度遗传操纵相关技术也为合成生物学和基因组工程等新兴领域的发展提供了重要的技术支撑。
在现有酵母基因组大尺度改造工具中,利用位点特异性重组系统和CRISPR相关技术进行基因组大片段敲除已经相对比较成熟,此外,利用酿酒酵母自身强大的同源重组能力可以实现外源基因和大片段DNA的组装以及整合,在酿酒酵母中借助MegNs和CRISPR等基因编辑工具可以进一步提高整合效率。在利用CRISPR技术进行多靶点编辑时gRNA数量的增加可以有效提高真核生物多靶点编辑的尺度,Church等[81]利用包含43个gRNA阵列的质粒成功编辑了哺乳动物细胞中的33个位点,证明了在哺乳动物细胞中重新编码和高度多重编辑的可行性。CRISPR等相关技术进行多靶点编辑未来发展前景良好。从头合成染色体可以实现染色体尺度的基因组操纵帮助我们更好地理解基因组结构和功能,人工合成染色体基因组重排可以产生大尺度基因组结构变异文库,但是从头合成大片段DNA较高的人力物力成本限制了其大规模使用。能够以较低成本高效地完成大片段DNA的合成将成为未来发展的重要发展方向。目前,酿酒酵母间已能够实现染色体水平的大片段DNA转移,Guo等[82]使用偏向性交配的方法实现了合成型染色体向野生型酵母的转移,但是利用酵母交配的方法转移目的DNA难以扩充至其他物种。酵母原生质体与细胞进行融合的方法可以将在酵母体内组装的Mb级别DNA片段递送至包括小鼠细胞在内的其他宿主,但其转移效率极低[83]。高效通用性强的大片段DNA转移技术的缺失是大片段DNA改造技术路线发展上的限制瓶颈之一。
综上所述,酵母基因组大尺度遗传操纵工具增强了酵母基因组可塑性和应用潜力,为酵母细胞研究提供了更为高效的手段。在未来的研究中,酵母基因组大尺度遗传操纵技术的进一步发展和完善将为酵母生物学的研究和应用提供更为广阔的空间。
| [1] |
NASMYTH K. A prize for proliferation. Cell, 2001, 107(6): 689-701. DOI:10.1016/S0092-8674(01)00604-3
|
| [2] |
GOFFEAU A, BARRELL BG, BUSSEY H, DAVIS RW, DUJON B, FELDMANN H, GALIBERT F, HOHEISEL JD, JACQ C, JOHNSTON M, LOUIS EJ, MEWES HW, MURAKAMI Y, PHILIPPSEN P, TETTELIN H, OLIVER SG. Life with 6 000 genes. Science, 1996, 274(5287): 546-567. DOI:10.1126/science.274.5287.546
|
| [3] |
POTVIN G, AHMAD A, ZHANG Z. Bioprocess engineering aspects of heterologous protein production in Pichia pastoris: a review. Biochemical Engineering Journal, 2012, 64: 91-105. DOI:10.1016/j.bej.2010.07.017
|
| [4] |
KRAINER FW, DIETZSCH C, HAJEK T, HERWIG C, SPADIUT O, GLIEDER A. Recombinant protein expression in Pichia pastoris strains with an engineered methanol utilization pathway. Microbial Cell Factories, 2012, 11: 22. DOI:10.1186/1475-2859-11-22
|
| [5] |
PADDON CJ, KEASLING JD. Semi-synthetic artemisinin: a model for the use of synthetic biology in pharmaceutical development. Nature Reviews Microbiology, 2014, 12(5): 355-367. DOI:10.1038/nrmicro3240
|
| [6] |
XU P, QIAO KJ, AHN WS, STEPHANOPOULOS G. Engineering Yarrowia lipolytica as a platform for synthesis of drop-in transportation fuels and oleochemicals. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(39): 10848-10853. DOI:10.1073/pnas.1607295113
|
| [7] |
XUE ZX, SHARPE PL, HONG SP, YADAV NS, XIE DM, SHORT DR, DAMUDE HG, RUPERT RA, SEIP JE, WANG J, POLLAK DW, BOSTICK MW, BOSAK MD, MACOOL DJ, HOLLERBACH DH, ZHANG HX, ARCILLA DM, BLEDSOE SA, CROKER K, MCCORD EF, et al. Production of omega-3 eicosapentaenoic acid by metabolic engineering of Yarrowia lipolytica. Nature Biotechnology, 2013, 31(8): 734-740. DOI:10.1038/nbt.2622
|
| [8] |
JIN J, WANG Y, YAO MD, GU XL, LI B, LIU H, DING MZ, XIAO WH, YUAN YJ. Astaxanthin overproduction in yeast by strain engineering and new gene target uncovering. Biotechnology for Biofuels, 2018, 11(1): 230. DOI:10.1186/s13068-018-1227-4
|
| [9] |
PATNAIK R. Engineering complex phenotypes in industrial strains. Biotechnology Progress, 2008, 24(1): 38-47. DOI:10.1021/bp0701214
|
| [10] |
GUO MR, CHENG SB, CHEN GG, CHEN JL. Improvement of lipid production in oleaginous yeast Rhodosporidium toruloides by ultraviolet mutagenesis. Engineering in Life Sciences, 2019, 19(8): 548-556. DOI:10.1002/elsc.201800203
|
| [11] |
KUMAR A, SERINGHAUS M, BIERY MC, SARNOVSKY RJ, UMANSKY L, PICCIRILLO S, HEIDTMAN M, CHEUNG KH, DOBRY CJ, GERSTEIN MB, CRAIG NL, SNYDER M. Large-scale mutagenesis of the yeast genome using a Tn7-derived multipurpose transposon. Genome Research, 2004, 14(10a): 1975-1986. DOI:10.1101/gr.2875304
|
| [12] |
CIRINO PC, MAYER KM, UMENO D. Generating mutant libraries using error-prone PCR. Methods in Molecular Biology, 2003, 231: 3.
|
| [13] |
YANG Z, BLENNER M. Genome editing systems across yeast species. Current Opinion in Biotechnology, 2020, 66: 255-266. DOI:10.1016/j.copbio.2020.08.011
|
| [14] |
HAN PY, MA Y, FU ZH, GUO Z, XIE JN, WU Y, YUAN YJ. A DNA inversion system in eukaryotes established via laboratory evolution. ACS Synthetic Biology, 2021, 10(9): 2222-2230. DOI:10.1021/acssynbio.1c00132
|
| [15] |
MITCHELL LA, BOEKE JD. Circular permutation of a synthetic eukaryotic chromosome with the telomerator. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(48): 17003-17010. DOI:10.1073/pnas.1414399111
|
| [16] |
BARBIERI EM, MUIR P, AKHUETIE-ONI BO, YELLMAN CM, ISAACS FJ. Precise editing at DNA replication forks enables multiplex genome engineering in eukaryotes. Cell, 2017, 171(6): 1453-1467.e13. DOI:10.1016/j.cell.2017.10.034
|
| [17] |
CRAVENS A, JAMIL OK, KONG DZ, SOCKOLOSKY JT, SMOLKE CD. Polymerase-guided base editing enables in vivo mutagenesis and rapid protein engineering. Nature Communications, 2021, 12: 1579. DOI:10.1038/s41467-021-21876-z
|
| [18] |
GRINDLEY NDF, WHITESON KL, RICE PA. Mechanisms of site-specific recombination. Annual Review of Biochemistry, 2006, 75: 567-605. DOI:10.1146/annurev.biochem.73.011303.073908
|
| [19] |
SAUER B. Site-specific recombination: developments and applications. Current Opinion in Biotechnology, 1994, 5(5): 521-527. DOI:10.1016/0958-1669(94)90068-X
|
| [20] |
AKADA R, KITAGAWA T, KANEKO S, TOYONAGA D, ITO S, KAKIHARA Y, HOSHIDA H, MORIMURA S, KONDO A, KIDA K. PCR-mediated seamless gene deletion and marker recycling in Saccharomyces cerevisiae. Yeast, 2006, 23(5): 399-405. DOI:10.1002/yea.1365
|
| [21] |
DELNERI D, COLSON I, GRAMMENOUDI S, ROBERTS IN, LOUIS EJ, OLIVER SG. Engineering evolution to study speciation in yeasts. Nature, 2003, 422(6927): 68-72. DOI:10.1038/nature01418
|
| [22] |
NASEEB S, CARTER Z, MINNIS D, DONALDSON I, ZEEF L, DELNERI D. Widespread impact of chromosomal inversions on gene expression uncovers robustness via phenotypic buffering. Molecular Biology and Evolution, 2016, 33(7): 1679-1696. DOI:10.1093/molbev/msw045
|
| [23] |
PARK YN, MASISON D, EISENBERG E, GREENE LE. Application of the FLP/FRT system for conditional gene deletion in yeast Saccharomyces cerevisiae. Yeast (Chichester, England), 2011, 28(9): 673-681. DOI:10.1002/yea.1895
|
| [24] |
KARIMOVA M, ABI-GHANEM J, BERGER N, SURENDRANATH V, TERESA PISABARRO M, BUCHHOLZ F. Vika/vox, a novel efficient and specific Cre/loxP-like site-specific recombination system. Nucleic Acids Research, 2013, 41(2): e37. DOI:10.1093/nar/gks1037
|
| [25] |
LIU W, LUO ZQ, WANG Y, PHAM NT, TUCK L, PÉREZ-PI I, LIU LY, SHEN Y, FRENCH C, AUER M, MARLES-WRIGHT J, DAI JB, CAI YZ. Rapid pathway prototyping and engineering using in vitro and in vivo synthetic genome SCRaMbLE-in methods. Nature Communications, 2018, 9(1): 1936. DOI:10.1038/s41467-018-04254-0
|
| [26] |
SAUER B, MCDERMOTT J. DNA recombination with a heterospecific Cre homolog identified from comparison of the pac-c1 regions of P1-related phages. Nucleic Acids Research, 2004, 32(20): 6086-6095. DOI:10.1093/nar/gkh941
|
| [27] |
THOMASON L, CALENDAR R, OW D. Gene insertion and replacement in Schizosaccharomyces pombe mediated by the Streptomyces bacteriophage φC31 site-specific recombination system. Molecular Genetics and Genomics, 2001, 265(6): 1031-1038. DOI:10.1007/s004380100498
|
| [28] |
XU ZY, BROWN WRA. Comparison and optimization of ten phage encoded serine integrases for genome engineering in Saccharomyces cerevisiae. BMC Biotechnology, 2016, 16(1): 1-10. DOI:10.1186/s12896-015-0230-0
|
| [29] |
SHEN MJ, WU Y, YANG K, LI YX, XU H, ZHANG HR, LI BZ, LI X, XIAO WH, ZHOU X, MITCHELL LA, BADER JS, YUAN YJ, BOEKE JD. Heterozygous diploid and interspecies SCRaMbLEing. Nature Communications, 2018, 9: 1934. DOI:10.1038/s41467-018-04157-0
|
| [30] |
JIA B, WU Y, LI BZ, MITCHELL LA, LIU H, PAN S, WANG J, ZHANG HR, JIA N, LI B, SHEN M, XIE ZX, LIU D, CAO YX, LI X, ZHOU X, QI H, BOEKE JD, YUAN YJ. Precise control of SCRaMbLE in synthetic haploid and diploid yeast. Nature Communications, 2018, 9: 1933. DOI:10.1038/s41467-018-03084-4
|
| [31] |
WANG J, XIE ZX, MA Y, CHEN XR, HUANG YQ, HE B, BIN J, LI BZ, YUAN YJ. Ring synthetic chromosome V SCRaMbLE. Nature Communications, 2018, 9: 3783. DOI:10.1038/s41467-018-06216-y
|
| [32] |
WU Y, ZHU RY, MITCHELL LA, MA L, LIU R, ZHAO M, JIA B, XU H, LI YX, YANG ZM, MA Y, LI X, LIU H, LIU D, XIAO WH, ZHOU X, LI BZ, YUAN YJ, BOEKE JD. In vitro DNA SCRaMbLE. Nature Communications, 2018, 9: 1935. DOI:10.1038/s41467-018-03743-6
|
| [33] |
FICKERS P, LE DALL MT, GAILLARDIN C, THONART P, NICAUD JM. New disruption cassettes for rapid gene disruption and marker rescue in the yeast Yarrowia lipolytica. Journal of Microbiological Methods, 2003, 55(3): 727-737. DOI:10.1016/j.mimet.2003.07.003
|
| [34] |
SONG PP, LIU S, GUO XN, BAI XJ, HE XP, ZHANG BR. Scarless gene deletion in methylotrophic Hansenula polymorpha by using mazF as counter-selectable marker. Analytical Biochemistry, 2015, 468: 66-74. DOI:10.1016/j.ab.2014.09.006
|
| [35] |
HO SS, URBAN AE, MILLS RE. Structural variation in the sequencing era. Nature Reviews Genetics, 2020, 21(3): 171-189. DOI:10.1038/s41576-019-0180-9
|
| [36] |
PRETORIUS IS, BOEKE JD. Yeast 2.0—connecting the dots in the construction of the world's first functional synthetic eukaryotic genome. FEMS Yeast Research, 2018, 18(4): foy032.
|
| [37] |
NAGY A. Cre recombinase: the universal reagent for genome tailoring. Genesis, 2000, 26(2): 99-109. DOI:10.1002/(SICI)1526-968X(200002)26:2<99::AID-GENE1>3.0.CO;2-B
|
| [38] |
HOESS RH, WIERZBICKI A, ABREMSKI K. The role of the loxP spacer region in P1 site-specific recombination. Nucleic Acids Research, 1986, 14(5): 2287-2300. DOI:10.1093/nar/14.5.2287
|
| [39] |
SHEN Y, STRACQUADANIO G, WANG Y, YANG K, MITCHELL LA, XUE YX, CAI YZ, CHEN T, DYMOND JS, KANG K, GONG JH, ZENG XF, ZHANG YF, LI YR, FENG Q, XU X, WANG J, WANG J, YANG HM, BOEKE JD, BADER JS. SCRaMbLE generates designed combinatorial stochastic diversity in synthetic chromosomes. Genome Research, 2016, 26(1): 36-49. DOI:10.1101/gr.193433.115
|
| [40] |
RICHARDSON SM, MITCHELL LA, STRACQUADANIO G, YANG K, DYMOND JS, DiCARLO JE, LEE D, LAI VICTOR HUANG C, CHANDRASEGARAN S, CAI YZ, BOEKE JD, BADER JS. Design of a synthetic yeast genome. Science, 2017, 355(6329): 1040-1044. DOI:10.1126/science.aaf4557
|
| [41] |
DAVID F, SIEWERS V. Advances in yeast genome engineering. FEMS Yeast Research, 2015, 15(1): 1-14. DOI:10.1093/femsyr/fou003
|
| [42] |
CARROLL D. Genome engineering with zinc-finger nucleases. Genetics, 2011, 188(4): 773-782. DOI:10.1534/genetics.111.131433
|
| [43] |
MUSSOLINO C, MORBITZER R, LÜTGE F, DANNEMANN N, LAHAYE T, CATHOMEN T. A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity. Nucleic Acids Research, 2011, 39(21): 9283-9293. DOI:10.1093/nar/gkr597
|
| [44] |
LIAN JZ, SCHULTZ C, CAO MF, HAMEDIRAD M, ZHAO HM. Multi-functional genome-wide CRISPR system for high throughput genotype-phenotype mapping. Nature Communications, 2019, 10: 5794. DOI:10.1038/s41467-019-13621-4
|
| [45] |
SOLIS-ESCALANTE D, van den BROEK M, KUIJPERS NGA, PRONK JT, BOLES E, DARAN JM, DARAN-LAPUJADE P. The genome sequence of the popular hexose-transport-deficient Saccharomyces cerevisiae strain EBY. VW4000 reveals LoxP/Cre-induced translocations and gene loss. FEMS Yeast Research, 2015, 15(2): fou004.
|
| [46] |
DICARLO JE, NORVILLE JE, MALI P, RIOS X, AACH J, CHURCH GM. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research, 2013, 41(7): 4336-4343. DOI:10.1093/nar/gkt135
|
| [47] |
EAUCLAIRE SF, ZHANG JZ, RIVERA CG, HUANG LL. Combinatorial metabolic pathway assembly in the yeast genome with RNA-guided Cas9. Journal of Industrial Microbiology & Biotechnology, 2016, 43(7): 1001-1015.
|
| [48] |
TOU CJ, SCHAFFER DV, DUEBER JE. Targeted diversification in the S. cerevisiae genome with CRISPR-guided DNA polymerase Ⅰ. ACS Synthetic Biology, 2020, 9(7): 1911-1916. DOI:10.1021/acssynbio.0c00149
|
| [49] |
SADHU MJ, BLOOM JS, DAY L, KRUGLYAK L. CRISPR-directed mitotic recombination enables genetic mapping without crosses. Science, 2016, 352(6289): 1113-1116. DOI:10.1126/science.aaf5124
|
| [50] |
XU H, HAN MZ, ZHOU SY, LI BZ, WU Y, YUAN YJ. Chromosome drives via CRISPR-Cas9 in yeast. Nature Communications, 2020, 11: 4344. DOI:10.1038/s41467-020-18222-0
|
| [51] |
LUO JC, SUN XJ, CORMACK BP, BOEKE JD. Karyotype engineering by chromosome fusion leads to reproductive isolation in yeast. Nature, 2018, 560(7718): 392-396. DOI:10.1038/s41586-018-0374-x
|
| [52] |
SHAO YY, LU N, WU ZF, CAI C, WANG SS, ZHANG LL, ZHOU F, XIAO SJ, LIU L, ZENG XF, ZHENG HJ, YANG C, ZHAO ZH, ZHAO GP, ZHOU JQ, XUE XL, QIN ZJ. Creating a functional single-chromosome yeast. Nature, 2018, 560(7718): 331-335. DOI:10.1038/s41586-018-0382-x
|
| [53] |
PERNER P. Mining sparse and big data by case-based reasoning. Procedia Computer Science, 2014, 35: 19-33. DOI:10.1016/j.procs.2014.08.081
|
| [54] |
CONG L, RAN FA, COX D, LIN SL, BARRETTO R, HABIB N, HSU PD, WU XB, JIANG WY, MARRAFFINI LA, ZHANG F. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143
|
| [55] |
ZHANG F. Development of CRISPR-Cas systems for genome editing and beyond. Quarterly Reviews of Biophysics, 2019, 52: e6. DOI:10.1017/S0033583519000052
|
| [56] |
WANG CL, LIWEI M, PARK JB, JEONG SH, WEI GY, WANG YJ, KIM SW. Microbial platform for terpenoid production: Escherichia coli and yeast. Frontiers in Microbiology, 2018, 9: 2460. DOI:10.3389/fmicb.2018.02460
|
| [57] |
DELTCHEVA E, CHYLINSKI K, SHARMA CM, GONZALES K, CHAO YJ, PIRZADA ZA, ECKERT MR, VOGEL J, CHARPENTIER E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886
|
| [58] |
JAKOČIŪNAS T, BONDE I, HERRGÅRD M, HARRISON SJ, KRISTENSEN M, PEDERSEN LE, JENSEN MK, KEASLING JD. Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae. Metabolic Engineering, 2015, 28: 213-222. DOI:10.1016/j.ymben.2015.01.008
|
| [59] |
ZHANG YP, WANG J, WANG ZB, ZHANG YM, SHI SB, NIELSEN J, LIU ZH. A gRNA-tRNA array for CRISPR-Cas9 based rapid multiplexed genome editing in Saccharomyces cerevisiae. Nature Communications, 2019, 10: 1053. DOI:10.1038/s41467-019-09005-3
|
| [60] |
LI ZH, LIU M, LYU XM, WANG FQ, WEI DZ. CRISPR/Cpf1 facilitated large fragment deletion in Saccharomyces cerevisiae. Journal of Basic Microbiology, 2018, 58(12): 1100-1104. DOI:10.1002/jobm.201800195
|
| [61] |
VERWAAL R, BUITING-WIESSENHAAN N, DALHUIJSEN S, ROUBOS JA. CRISPR/Cpf1 enables fast and simple genome editing of Saccharomyces cerevisiae. Yeast, 2018, 35(2): 201-211. DOI:10.1002/yea.3278
|
| [62] |
POSTMA ED, DASHKO S, van BREEMEN L, TAYLOR PARKINS SK, van den BROEK M, DARAN JM, DARAN-LAPUJADE P. A supernumerary designer chromosome for modular in vivo pathway assembly in Saccharomyces cerevisiae. Nucleic Acids Research, 2021, 49(3): 1769-1783. DOI:10.1093/nar/gkaa1167
|
| [63] |
POSTMA ED, HASSING EJ, MANGKUSAPUTRA V, GEELHOED J, dela TORRE P, van den BROEK M, MOOIMAN C, PABST M, DARAN JM, DARAN-LAPUJADE P. Modular, synthetic chromosomes as new tools for large scale engineering of metabolism. Metabolic Engineering, 2022, 72: 1-13. DOI:10.1016/j.ymben.2021.12.013
|
| [64] |
HUANG SC, GENG AL. High-copy genome integration of 2,3-butanediol biosynthesis pathway in Saccharomyces cerevisiae via in vivo DNA assembly and replicative CRISPR-Cas9 mediated delta integration. Journal of Biotechnology, 2020, 310: 13-20. DOI:10.1016/j.jbiotec.2020.01.014
|
| [65] |
WENINGER A, HATZL AM, SCHMID C, VOGL T, GLIEDER A. Combinatorial optimization of CRISPR/Cas9 expression enables precision genome engineering in the methylotrophic yeast Pichia pastoris. Journal of Biotechnology, 2016, 235: 139-149. DOI:10.1016/j.jbiotec.2016.03.027
|
| [66] |
HORWITZ AA, WALTER JM, SCHUBERT MG, KUNG SH, HAWKINS K, PLATT DM, HERNDAY AD, MAHATDEJKUL-MEADOWS T, SZETO W, CHANDRAN SS, NEWMAN JD. Efficient multiplexed integration of synergistic alleles and metabolic pathways in yeasts via CRISPR-cas. Cell Systems, 2015, 1(1): 88-96. DOI:10.1016/j.cels.2015.02.001
|
| [67] |
NUMAMOTO M, MAEKAWA H, KANEKO Y. Efficient genome editing by CRISPR/Cas9 with a tRNA-sgRNA fusion in the methylotrophic yeast Ogataea polymorpha. Journal of Bioscience and Bioengineering, 2017, 124(5): 487-492. DOI:10.1016/j.jbiosc.2017.06.001
|
| [68] |
GIBSON DG, GLASS JI, LARTIGUE C, NOSKOV VN, CHUANG RY, ALGIRE MA, BENDERS GA, MONTAGUE MG, MA L, MOODIE MM, MERRYMAN C, VASHEE S, KRISHNAKUMAR R, ASSAD-GARCIA N, ANDREWS-PFANNKOCH C, DENISOVA EA, YOUNG L, QI ZQ, SEGALL-SHAPIRO TH, CALVEY CH, et al. Creation of a bacterial cell controlled by a chemically synthesized genome. Science, 2010, 329(5987): 52-56. DOI:10.1126/science.1190719
|
| [69] |
DYMOND JS, RICHARDSON SM, COOMBES CE, BABATZ T, MULLER H, ANNALURU N, BLAKE WJ, SCHWERZMANN JW, DAI JB, LINDSTROM DL, BOEKE AC, GOTTSCHLING DE, CHANDRASEGARAN S, BADER JS, BOEKE JD. Synthetic chromosome arms function in yeast and generate phenotypic diversity by design. Nature, 2011, 477(7365): 471-476. DOI:10.1038/nature10403
|
| [70] |
ANNALURU N, MULLER H, MITCHELL LA, RAMALINGAM S, STRACQUADANIO G, RICHARDSON SM, DYMOND JS, KUANG Z, SCHEIFELE LZ, COOPER EM, CAI YZ, ZELLER K, AGMON N, HAN JS, HADJITHOMAS M, TULLMAN J, CARAVELLI K, CIRELLI K, GUO ZY, LONDON V, et al. Total synthesis of a functional designer eukaryotic chromosome. Science, 2014, 344(6179): 55-58. DOI:10.1126/science.1249252
|
| [71] |
ZHANG WM, LAZAR-STEFANITA L, YAMASHITA H, SHEN MJ, MITCHELL LA, KURASAWA H, HAASE MAB, SUN XJ, JIANG QW, LAUER SL, MCCULLOCH LH, ZHAO Y, ICHIKAWA DM, EASO N, LIN SJ, FANFANI V, CAMELLATO BR, ZHU YN, CAI JT, XU ZW, et al. Manipulating the 3D organization of the largest synthetic yeast chromosome. SSRN Electronic Journal, 2022, 2022.04. 09.487066.
|
| [72] |
XIE ZX, LI BZ, MITCHELL LA, WU Y, QI X, JIN Z, JIA B, WANG X, ZENG BX, LIU HM, WU XL, FENG Q, ZHANG WZ, LIU W, DING MZ, LI X, ZHAO GR, QIAO JJ, CHENG JS, ZHAO M, et al. Perfect designer chromosome V and behavior of a ring derivative. Science, 2017, 355(6329): eaaf4704. DOI:10.1126/science.aaf4704
|
| [73] |
MITCHELL LA, WANG A, STRACQUADANIO G, KUANG Z, WANG XY, YANG K, RICHARDSON S, MARTIN JA, ZHAO Y, WALKER R, LUO YS, DAI HJ, DONG K, TANG ZJ, YANG YL, CAI YZ, HEGUY A, UEBERHEIDE B, FENYÖ D, DAI JB, et al. Synthesis, debugging, and effects of synthetic chromosome consolidation: synVI and beyond. Science, 2017, 355(6329): eaaf4831. DOI:10.1126/science.aaf4831
|
| [74] |
SHEN Y, WANG Y, CHEN T, GAO F, GONG JH, ABRAMCZYK D, WALKER R, ZHAO HC, CHEN SH, LIU W, LUO YS, MÜLLER CA, PAUL-DUBOIS-TAINE A, ALVER B, STRACQUADANIO G, MITCHELL LA, LUO ZQ, FAN YQ, ZHOU BJ, WEN B, et al. Deep functional analysis of synII, a 770-kilobase synthetic yeast chromosome. Science, 2017, 355(6329): eaaf4791. DOI:10.1126/science.aaf4791
|
| [75] |
WU Y, LI BZ, ZHAO M, MITCHELL LA, XIE ZX, LIN QH, WANG X, XIAO WH, WANG Y, ZHOU X, LIU H, LI X, DING MZ, LIU D, ZHANG L, LIU BL, WU XL, LI FF, DONG XT, JIA B, et al. Bug mapping and fitness testing of chemically synthesized chromosome X. Science, 2017, 355(6329): eaaf4706. DOI:10.1126/science.aaf4706
|
| [76] |
ZHANG WM, ZHAO GH, LUO ZQ, LIN YC, WANG LH, GUO YK, WANG A, JIANG SY, JIANG QW, GONG JH, WANG Y, HOU S, HUANG J, LI TY, QIN YR, DONG JK, QIN Q, ZHANG JY, ZOU XZ, HE X, et al. Engineering the ribosomal DNA in a megabase synthetic chromosome. Science, 2017, 355(6329): eaaf3981. DOI:10.1126/science.aaf3981
|
| [77] |
SHAO ZY, ZHAO H, ZHAO HM. DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways. Nucleic Acids Research, 2009, 37(2): e16. DOI:10.1093/nar/gkn991
|
| [78] |
KUTYNA DR, ONETTO CA, WILLIAMS TC, GOOLD HD, PAULSEN IT, PRETORIUS IS, JOHNSON DL, BORNEMAN AR. Construction of a synthetic Saccharomyces cerevisiae pan-genome neo-chromosome. Nature Communications, 2022, 13(1): 3628. DOI:10.1038/s41467-022-31305-4
|
| [79] |
BOEKE JD, CHURCH G, HESSEL A, KELLEY NJ, ARKIN A, CAI YZ, CARLSON R, CHAKRAVARTI A, CORNISH VW, HOLT L, ISAACS FJ, KUIKEN T, LAJOIE M, LESSOR T, LUNSHOF J, MAURANO MT, MITCHELL LA, RINE J, ROSSER S, SANJANA NE, et al. The genome project-write. Science, 2016, 353(6295): 126-127. DOI:10.1126/science.aaf6850
|
| [80] |
DICARLO JE, CONLEY AJ, PENTTILÄ M, JÄNTTI J, WANG HH, CHURCH GM. Yeast oligo-mediated genome engineering (YOGE). ACS Synthetic Biology, 2013, 2(12): 741-749. DOI:10.1021/sb400117c
|
| [81] |
CHEN YT, HYSOLLI E, CHEN AL, CASPER S, LIU SL, YANG K, LIU CL, CHURCH G. Multiplex base editing to convert TAG into TAA codons in the human genome. Nature Communications, 2022, 13: 4482. DOI:10.1038/s41467-022-31927-8
|
| [82] |
GUO Z, YIN HY, MA L, LI JY, MA JJ, WU Y, YUAN YJ. Direct transfer and consolidation of synthetic yeast chromosomes by abortive mating and chromosome elimination. ACS Synthetic Biology, 2022, 11(10): 3264-3272. DOI:10.1021/acssynbio.2c00174
|
| [83] |
LI LP, BLANKENSTEIN T. Generation of transgenic mice with megabase-sized human yeast artificial chromosomes by yeast spheroplast-embryonic stem cell fusion. Nature Protocols, 2013, 8(8): 1567-1582. DOI:10.1038/nprot.2013.093
|
 2023, Vol. 39
2023, Vol. 39