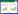中国科学院微生物研究所、中国微生物学会主办
文章信息
- 张娇, 陈江峰, 陈奕璇, 戴磊, 戴卓君
- ZHANG Jiao, CHEN Jiangfeng, CHEN Yixuan, DAI Lei, DAI Zhuojun
- 合成功能菌群的构建及其工程化应用
- Engineering microbial consortia through synthetic biology approach
- 生物工程学报, 2023, 39(5): 2517-2545
- Chinese Journal of Biotechnology, 2023, 39(5): 2517-2545
- 10.13345/j.cjb.230062
-
文章历史
- Received: January 31, 2023
- Accepted: June 21, 2023
合成功能菌群(synthetic microbial consortia)是指人工创建的2种或多种已知微生物在特定的可控环境条件中形成的共培养体系[1-2]。相比于单菌系统,合成功能菌群具有通讯(communication)及分工(division of labor)的特征:菌群成员可以通过信号分子交换、检测并相互响应等方式进行单向、双向甚至多向交流[3-4];菌群成员可以通过分工执行不同的任务,从而使整个菌群实现单一菌株无法完成的复杂功能[5-6]。这种成员之间的相互协调保证了群落结构和功能的稳定,降低了群体的代谢负担以及增加了对环境变化的适应性。在自然界中,天然的微生物群落在多个方面发挥着重要作用。例如,土壤中的微生物群落可以参与碳循环,并在固氮和分解有机物等环节发挥重要作用;肠道微生物群落在代谢营养物质和防止病原体入侵方面发挥着关键作用。因此,具备合成菌群并可以精准调控其组分的能力可以应用于包括生物制造、生物医学和生物修复在内的多个领域。
合成生物学的快速发展在医药、能源和环境等方面展现出了巨大的潜力。与传统生物研究相比,合成生物学依据工程设计原理构建或改造基本功能元件、基因线路和底盘细胞,并对生命的运行过程进行具有目的性的重新编程,实现丰富的、可设计的生物学功能[7-9]。将这种工程设计的原理引入菌群,获得结构明确的合成群落具有重要的意义。首先,相对于自然菌群,人工设计的群体结构简单,可以通过数学模型描述和验证,有利于对微生物群落组成、功能和互作的深入理解[10-11];同时,具备设计及构建合成功能菌群的能力也为其应用提供了前提,例如通过菌群成员分工实现高附加值天然产物的合成等。本文详细回顾了近年来合成功能菌群在设计原则、构建方法以及应用方面的诸多工作,并对未来的发展进行了展望。
1 合成功能菌群的构建微生物群落相比于单菌体系在功能上具备多种优势,通过设计及构建菌群中不同种群间的相互关系,使之构成稳定的群落结构是合成功能菌群构建中的常用思路。为了达成这一目的,首先需要理解多种群共存的方式。种群A与种群B之间能产生以下6种生态相互作用:偏利共生、偏害共生、互利共生、捕食、竞争和无利害共栖(图 1A)[12]。当微生物群落中种群数量增加时,群落复杂度也随着种群相互作用组合的增加和更高阶相互作用的出现而增加[13]。例如,第3个种群的加入会改变前2个种群相互关系的强度和种类,第4个种群的加入也会随之改变前3个种群的关系[14]。群落的稳定性依赖于其中种群关系的类型和强度,而通过数学模型可以用来预测群落的组成随着时间的变化[14-16]。下文总结了利用生态相互作用构建合成功能菌群的系列工作。
|
| 图 1 合成功能菌群的设计及构建[12, 18, 23, 31, 46] Fig. 1 Design and engineer synthetic microbial consortia[12, 18, 23, 31, 46]. A:不同种群间的相互作用. 种群A与种群B的6种生态相互作用:偏利共生、偏害共生、无利害共生、互利共生、竞争和捕食[12]. B:利用基因线路构建细菌间类似于捕食者-猎物的相互关系. 左图为捕食者-猎物关系概念图,包含1个捕食者种群Py和1个猎物种群Pd,当抗生素氯霉素(CM)存在时,Py通过降解CM保护Pd,相反地,Pd通过分泌乳球菌素A (lcnA)抑制Py生长;右图为Py和Pd的基因线路图,Py持续表达cat基因以降解环境中的CM,因而能够在CM环境中生存,同时表达荧光报告基因mCherry;Pd表达lcnA、lceA、lciA和lcmA,分泌乳球菌素A,同时也包含1个荧光报告基因yemGFP[18]. C:通过共生关系构建稳定的大肠杆菌-酿酵母菌群. Zhou等创建的共生微生物菌群以生产紫杉醇前体,其中,大肠杆菌消耗木糖并分泌乙酸盐抑制自身生长,而酿酒酵母利用乙酸盐作为唯一碳源,有效降低了乙酸盐浓度,让大肠杆菌得以存活[23]. D:2株菌表达正交性的同步裂解线路,当种群浓度过高时,同步裂解线路通过群体感应小分子诱导细胞自裂解,使2个种群能够维持稳定共存[31]. E:通过微胶囊的空间隔离构建稳定共存微生物群落. 每个微胶囊包裹工程化的微生物,将包含不同亚群的MSB进行组合,进一步构建多种群微生物群落[46] A: Different modes of interaction in two-strain consortia. Six pairwise social interactions: commensalism, amensalism, neutralism, cooperation, competition and predation[12]. B: Using gene circuits to build a predator-prey ecosystem. The consortium consists of a prey strain, Py, and a predator strain, Pd. In the presence of the antibiotic chloramphenicol (CM), Py confers a protection to Pd by degrading CM; In turn, Pd inhibits Py by producing lactococcin A (lcnA). Py carries a constitutively expressed gene, cat, allowing the deactivation of CM in the environment, and hence survives in the presence of CM. Py also contains the fluorescence reporter gene mCherry. Pd harbors four constitutively expressed genes, lcnA, lceA, lciA and lcmA, enabling the secretion of lcnA. Pd also contains the fluorescence reporter gene yemGFP[18]. C: A mutualistic Escherichia coli-S. cerevisiae consortium for production of oxygenated taxanes. E. coli grows on xylose and produces acetate, which is inhibitory to its own growth. Saccharomyces cerevisiae, on the other hand, uses acetate as the sole carbon source for growth, reducing the acetate concentration and allowing Escherichia coli to survive[23]. D: Two bacterial strains with orthogonal synchronized lysis circuits. Each strain is programmed to lyse upon reaching a critical population density by signal QS systems, maintaining a stable co-culture of two populations [31]. E: Engineering stably coexisting consortia by microbial swarmbot mediated spatial segregation strategy. Each microbial swarmbot (MSB) consists of a small population of engineered microbes encapsulated in polymeric microcapsules. The MSBs containing different subpopulations are then co-cultured to form the microbial swarmbot consortia (MSBC)[46]. |
| |
Balagaddé等[17]通过编辑大肠杆菌(Escherichia coli)的2个种群的群体感应(quorum sensing, QS),形成捕食者-猎物系统。其中1个大肠杆菌种群作为捕食者,另外1个大肠杆菌种群作为猎物。捕食者持续不断地表达自杀蛋白(CcdB),猎物通过产生能够使捕食者表达解毒蛋白(CcdA)的QS小分子使捕食者获益。当猎物的浓度降低时,捕食者会因为解毒剂的减少而开始死亡。同时,捕食者产生QS小分子从而促使猎物表达自杀蛋白(CcdB)。捕食者浓度降低时,猎物的密度会因为表达更少自杀蛋白而上升。作者创建了1个数学模型来预测系统可产生3种不同的种群动态模式:猎物主导、捕食者和猎物之间振荡以及捕食者主导,并通过实验验证了通过调控诱导剂浓度可使系统获得上述3种种群动态模式。
自此,研究者们建立了更多复杂的合成生态系统。例如,Liu等[18]建立了1个由环境因素,例如抗生素或入侵者调控的与捕食者-猎物相似的生态系统,该系统包括2株工程化的乳酸乳球菌(Lactococcus lactis),一个猎物菌Py和一个捕食者菌Pd (图 1B)。其中猎物菌Py能持续表达氯霉素乙酰转移酶以抑制氯霉素(chloramphenicol, CM)毒性,而捕食者菌Pd表达乳球菌素lcnA杀死Py。当环境中存在CM时,Py保护Pd,而Pd抑制Py生长,从而形成了捕食者-猎物关系;当环境中不存在CM时,Py不保护Pd,Pd仍然抑制Py生长,形成了Pd对Py的偏害共栖关系。由此,该系统的种间关系随着环境因素而改变。另外,文中测试了引入入侵者(与Py形成捕食-猎物关系;与Pd形成竞争关系)对系统动态的影响:当该入侵者不具有抗生素抗性时,捕食者和猎物展现出和没有入侵者时相似的动态行为,这时候入侵者只能短暂地繁殖然后种群密度逐步降低;当入侵者具备抗生素抗性时,捕食者和猎物的密度都会显著降低,而入侵者会繁殖壮大。上述2个例子利用群体感应、抗生素和基因线路等元素实现种群沟通及调控,这些模型有助于我们理解微生物在自然界的相互作用,同时具备预测复杂合成菌群行为的能力。
Nair等[19]对捕食者-猎物系统进行了更深入的研究,通过构建由黄色粘球菌(Myxococcus xanthus)作为捕食者,由大肠杆菌作为猎物的生态系统,发现种群间能相互适应并加速群落的共同进化,该研究表明,多能型掠食者性细菌是决定复杂微生物群落及其相互作用网络在自然环境中进化的关键因素。
1.1.2 共生关系另一种常用于构建稳定功能菌群结构的相互关系是共生。共生关系在自然界中无处不在。偏利共生指种间相互作用对一方有益而对另一方没有影响。在适当的条件下,偏利共生能够促进种群间共存,较“健壮”的种群支持适应度较低的种群存活以避免其死亡[20]。有趣的是,许多研究者发现大肠杆菌作为供养者的一方(大肠杆菌能依赖不同碳源生存),能和其他多个物种形成偏利共生关系[21]。鉴于大肠杆菌是微生物工程化常用的模式生物,这有利于研究者构建共生的工程化微生物群落,以改进生物被膜形成和代谢产物生产等。例如,Brenner等[22]构建了由2种大肠杆菌组成的菌群,其中第一个大肠杆菌种群可以形成生物被膜,但无法持续合成生存所需的关键代谢产物(二氨基乙酸和赖氨酸);只有当另一个大肠杆菌种群(不形成生物被膜)以高浓度存在时,释放出来的群体感应分子可以诱导第一个大肠杆菌种群合成自身生产代谢关键产物,才能维持生存并形成生物被膜。共生关系的另一种形式是互利共生,种间依赖彼此生存。例如,Zhou等[23]创建了1个共生微生物菌群以生产紫杉醇前体(图 1C),其中,大肠杆菌消耗木糖并分泌乙酸盐抑制自身生长,而酿酒酵母(Saccharomyces cerevisiae)利用乙酸盐作为唯一碳源,有效降低了乙酸盐浓度,让大肠杆菌得以存活。该研究利用每个物种的优势,将合成紫衫烷的代谢路径分割成不同部分,由大肠杆菌杆菌和酿酒酵母分别完成一部分,这种共生设计提高了共培养体系每个部分的稳定性,提高了产量并且降低了相比起竞争培养下产物的变异性。
相似地,Sanghak等[24]设计了1种共生体系以改进对CO的代谢转化。其中,粘液真杆菌(Eubacterium limosum)以CO作为碳源并将之转化为乙酸盐,当乙酸盐积累到一定浓度时,会对细胞产生负面作用,工程化的大肠杆菌能够将乙酸盐转化为有价值的化合物例如衣康酸或者羟基丙酸。该系统相比起单菌培养的粘液真杆菌,有更高效的CO转化能力和生化产物合成能力,该工作建立了1个能够利用工程化的共生菌株高产自然代谢产物的平台。另外,Kang等[25]设计了1个由利用藻酸盐的弧菌和产生3-羟丙酸(3-hydroxypropionic acid, 3-HP)的大肠杆菌组成的共生群落,用于直接将藻酸盐转化为3-HP。并在大肠杆菌中引入只在产生3-HP时才降解抗生素的基因线路,当添加抗生素作为选择压力时,菌群在48 h发酵过程中,成功适应环境并使3-HP产量提高了4.3倍。研究者们通过这些工作赋予合成菌群共生关系以改进代谢工程,提高了高附加值代谢产物的产量。
1.1.3 竞争关系竞争关系是自然界中最为常见的物种间相互作用,2个竞争相同营养物质以及生态位的菌种在缺少缓解机制的共培养体系中难以稳定共存。为了避免竞争性排除,不同物种通常占据自然界的不同生态位,这个过程称为生态位分化。生态位分化常用的一种机制是资源分配,即群落中的每一种成员利用不同的营养物质,以避免2个种群直接竞争。通过工程化改造使菌群成员正交性地利用至少一种初级代谢产物,即保证每个成员能用独有的方式获取营养物质(通常是碳源),能够实现微生物群落的资源分配[26]。例如,Eiteman等[27]建立了1个共培养的大肠杆菌体系,其中一种大肠杆菌利用葡萄糖,另一种利用木糖,通过共同代谢利用2种糖产生乳酸盐。类似地,许多工作将代谢工程线路植入共培养体系中,使菌株利用多种原料,例如葡萄糖和甘油等生产各种感兴趣的化合物[28-30]。
缓解竞争的其中一种方法是引入负反馈,使每个种群程序性控制种群数量。Scott等[31]利用正交性的同步裂解线路(synchronized lysis circuits, SLC)构建了2个能够维持稳定共生关系的大肠杆菌菌群(图 1D)。当种群浓度过高时,同步裂解线路通过QS小分子诱导细菌自裂解,从而引入负反馈调节。结果发现,生长速率较快的种群达到一定浓度时开始自裂解,降低了对生长速率较慢的种群的竞争压力,生长速率较慢的种群也能开始生长,直到浓度过高而开始自裂解。通过引入这种负反馈调节循环,每个种群有一定自限性,抵消了一部分竞争压力,达到共存。
1.1.4 高阶相互作用自然界中的微生物群落通常同时含有多种相互作用,而生物间的相互作用是物种共存和生物多样性维持的关键。通过设计多对和复杂的相互作用,有助于预测和编辑多样化的合成功能菌群[32]。Kong等[33]设计的合成微生物菌群包含了6对相互作用。例如,在2个共生的种群中,一个种群分泌信号分子乳酸链球菌肽,另一个种群表达由乳酸链球菌肽诱导的四环素抗性基因,使2个种群能在四环素环境中共生。另外,在2个具有竞争关系的种群中,2个种群分别产生能将对方杀死的毒素。基于此,Kong等利用数学模型指导设计了包含3种或4种种群的合成微生物群落,这些种群两两之间都具有相互作用,并进一步通过实验成功地建立了稳定的菌群。
对于一些群落,考虑高阶的相互作用(higher-order interactions, HOIs)对于预测群落动态,维持稳定共存和实现功能是至关重要的。HOIs是指3个以上种群共存时,种群间的非叠加的相互作用[34]。根据物种的数量和它们之间的相互作用,HOI网络可以是高度复杂的。因此,HOIs在群落中的作用以及其调节共存的程度往往是根据每个系统改变的[35]。Bairey等[14]发现,两两相互作用随着物种数量的增加而不稳定,四维的HOIs随着物种数量的减少而不稳定,三维HOIs稳定性不随群落大小而变化。在某些情况下,HOIs通过促进成员之间的共存使工程化微生物群落得以稳定存在。例如,在1个三成员群落中,第3个种群的加入能减弱2个敌对种群之间的负面相互作用。即使第3个物种与前2个物种拮抗,只要每个种群调节其余2个成员之间的抑制性相互作用,该群落也能保持稳定[36-38]。另外,新种群的加入也可能与现存群落产生协同作用,从而形成更具生产力的群落。如Embree等[39]发现,在他们构建的三、四和五成员群落中,氨基酸营养缺陷型在成员之间创造了复杂的相互依赖关系,促进了群落的稳定性和鲁棒性。HOIs能在多大程度上维持群落的共存,可能取决于网络结构和成员间的资源分配水平。其中,数学建模可以帮助预测HOIs对所讨论的群落的影响程度。Friedman等[40]利用计算和实验评估了这些相互作用对群落的预测能力。首先,他们通过实验确认了所有种群之间的两两相互作用:能够共存或1个种群会杀死另一个种群。利用这些结果,他们用计算机模型预测了所有三成员群落的行为模式。实验证明他们的模型正确预测了89.5%的群落的最终组成。接着,他们利用三成员群落的结果构建了7‒8个成员的群落的预测模型。当考虑三维HOIs时,他们的模型能够准确预测86%的最终群落组成,而当只考虑两两相互作用时,他们的模型只有62%准确率。该研究证明了利用计算模型预测微生物菌群时,考虑HOIs是必要的。
1.2 空间分隔和有序排布调控菌群结构在自然界中的微生物群落可以通过空间分隔实现共存[10, 41]。例如,土壤微生物通过形成几百微米的微菌落实现共存。空间分隔能够通过生态位分化减少种群对资源的竞争,确保适应度较低的成员的生存[32, 42-43]。因此,研究者尝试利用各种物理分隔技术,包括微流控装置、生物打印、模具/封装和其他制造技术,研究空间分隔如何影响多种群之间的相互作用和整个群落的功能。Kim等[44]设计微流控芯片获得了种群间的空间分隔,使种群之间保留适度距离,让具有不同生长速率的种群组成1个稳定的微生物群落。他们构建了1个由3种土壤微生物组成的共培养体系,包括棕色固氮菌(Azotobacter vinelandii, Av)、地衣芽孢杆菌(Bacillus licheniformis, Bl)和解凝乳类芽孢杆菌(Paenibacillus curdlanolyticus, Pc),这3个种群组成共生的关系,在群落中执行不同功能。其中,只有Av在有氧条件下用钼偶联固氮酶将气态氮固定到氨基酸中来提供氮源,只有Bl通过用β内酰胺酶降解青霉素G来降低抗生素压力,只有Pc提供碳能源,例如葡萄糖,使用纤维素酶裂解羧甲基纤维素。在这种共培养中,研究者发现无论营养物质丰富与否,群落都是不稳定的。在营养丰富的培养基中,Bl种群迅速增长,Av和Pc种群迅速下降到检测限以下。在营养贫瘠的培养基中,Av的种群数量增加,而Bl和Pc的种群数量减少。然而,当每个种群由物理分隔并仍能通过化学信号交流时,3个种群都能稳定增长。当分隔距离加长时,3个种群的浓度均下降,说明了种群需要适当的距离分隔以避免生长竞争,同时保持种群间的积极相互作用。微生物的空间分布通过距离和物理分隔调控了信号分子交换的频率,从而调控了种群间的竞争和积极相互作用关系。
Johnston等[12]利用水凝胶物理分隔了2种共生的种群:大肠杆菌和酵母,它们形成的共培养体系能够合成甜菜黄素。通过基因编辑,大肠杆菌产生l-dopa,而酵母能将l-dopa转化成甜菜黄素,通过水凝胶实现的物理分隔提高了甜菜黄素的产量。通过改变体系中凝胶的比例,也可以控制最终的种群组成。并且,该体系能够重复利用,并为菌群提供了长期保护。Dai等[45]利用聚合物微胶囊分隔大肠杆菌以控制菌群组成,分担脂肪酸合成的多酶代谢路径。控制蛋白比例对于复杂的胞外生物制品生产路径至关重要。在1个一锅式反应中,表达及纯化蛋白在时间及经济成本上优于每一种蛋白的单独表达及纯化,但在这种混合培养反应中,控制蛋白比例一直难以达成。为了解决这一问题,Dai等[45]直接通过控制微胶囊的数量控制菌群组成,从而控制了产出蛋白的比例。利用该微胶囊包裹微生物体系(microbial swarmbot, MSB),Wang等[46]后续通过将包含不同亚群的MSB进行组合(图 1E),进一步构建了多种跨物种的微生物群落(microbial swarmbot consortia, MSBC),由于MSB具有相对独立的生长空间和明确的承载上限,即使菌群中种群的分裂时间不匹配也可以平衡增长,这为跨物种微生物群落的稳定构建和精确调控提供了1种通用方法。相比于上文所采用的宏观水凝胶策略,微米级别的MSB及MSBC系统更利于种群间的传质(信号分子、代谢分子和生物大分子等),当然,水凝胶分隔体系还存在一定的局限性,例如该体系需要提前制备和物质传输速率较之混菌体系较慢,且胶囊存在一定程度的泄漏,后续还待开发更自主和稳固的空间分隔方式。
通过有序排布菌群结构可以调控菌群的共存及生物学功能。Gupta等[47]利用空间分隔和距离来调控基因表达,他们发展了1种微流控平台控制种群间的距离,使种群间仅通过可扩散性分子交流,证明了距离的增长降低了噪音从而增加细胞间振荡信号传导的可信度,同时降低了信号分子诱导的基因表达强度。Connel等[48]开发了1种微观3D打印技术,可以以空间分隔的方式排列多个细菌物种,形成了1个种群在另一个“核”种群周围形成“壳”的核壳嵌套结构,其中铜绿假单胞菌通过包围和屏蔽金黄色葡萄球菌免受β-内酰胺抗生素而产生抗生素耐药性,获得稳定的菌群共存。Tei等[49]提出了1种由硅胶模具内的区室分隔和通道组成的结构化细菌通讯设备,使细菌实现物理分隔,同时保证信号分子酰基高丝氨酸内酯(acyl homoserine lactone, AHL)能正常扩散,通过构建2株相互抑制基因表达的菌,他们发现改变中心区域连接通道的数量对2株菌基因表达以产生比对模式至关重要,该项工作证明空间分隔对种群的影响比生化参数等更为直接。
2 合成功能菌群的应用 2.1 生物合成随着合成生物学和代谢工程的发展,单菌的生物发酵已被广泛用于化学品的生物生产。但在合成长途径及复杂途径化学品时,由于过度的细胞资源消耗和压倒性的代谢负担通常会导致单个宿主菌株的生长受阻和/或生物合成性能变差[50]。此外,当生物合成途径所利用的部分或全部酶来自异源宿主时(如代谢途径同时含有原核酶和真核酶),单个宿主细胞很难为所有酶提供最佳环境[51]。为解决单菌生物生产遇到的困难,通过开发菌群共培养体系,利用菌群分工进行生物合成成为了一种替代方案[52]。相较于单菌培养,菌群共培养体系具有多种优势,包括减轻宿主代谢负担[53]、拓宽底物利用谱[54-55]、为不同的酶提供多样化的细胞环境[51]、对环境扰动具有更高的稳定性和鲁棒性[50]和可以合成单菌培养无法合成的化学品[56]。目前,微生物菌群已被用于生产生物燃料(如生物乙醇[57-58]、生物丁醇[55]和生物柴油[59-60])、大宗化学品(如乳酸[61-62]、乙酸[63])、复杂天然产物(如多酚[64]、生物碱[65]、聚酮[66]和萜烯[67])、高价值非天然化合物(如功能氮杂环[68])以及蛋白大分子[69]等,以下将详细介绍这些工作。
在生物燃料制备中,联合生物加工(consolidated bioprocessing, CBP)是在单一或组合微生物作用下,从木质纤维素直接生产生物燃料及化学品的方法。不过,工业生产的菌株往往难以降解木质纤维素,而具备木质纤维素降解能力的菌株又缺乏生物合成能力或遗传操作工具。利用合成生物学手段编辑菌株,使其既能分解木质纤维素又能合成目标产物是一个可行方案,但往往会造成单个菌株代谢负担重,进而影响目标产物的产量[70]。通过构建合成功能菌群,通过菌群中的成员实现底物分解以及产物合成的分工,是一个潜在的策略。例如,Jiang等[55]通过共培养热解糖热厌氧杆菌(Thermoanaerobacterium thermosaccharolyticum) M5和丙酮丁醇梭菌(Clostridium acetobutylicum) NJ4,使用这2种菌株进行劳动分工,从木聚糖中高效地生产生物丁醇。菌株M5可分泌木聚糖酶和木糖苷酶,具有高效的木聚糖降解能力。而搭档菌株NJ4的木糖利用能力可以缓解底物对菌株M5分泌的水解酶的抑制;同时,菌株NJ4也表现出优异的丁醇生产能力。经过工艺优化后,这2种菌株相互作用最终从70 g/L木聚糖中得到了13.28 g/L丁醇,这与菌株NJ4从70 g/L的木糖中产生的丁醇相似。此外,以未经处理的玉米芯作为底物时,该菌群通过CBP产生7.61 g/L丁醇。
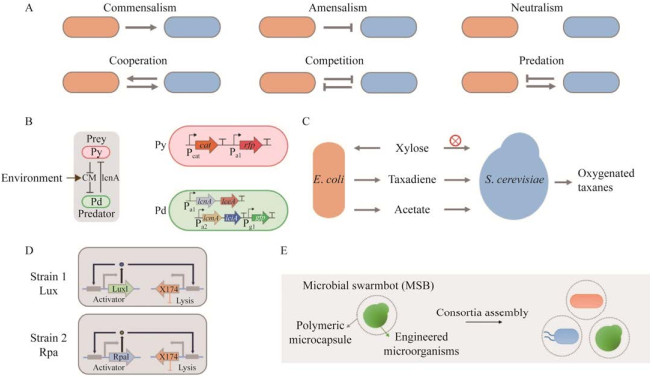
菌群共培养也被用来高效合成各种大宗化学品。如,Shahab等[62]制作了1个以氧气梯度为特征的膜曝气生物反应器(membrane-membrane- aerated reactor),培养3种不同的菌株,将纤维素和木糖转化为短链脂肪酸。好氧性真菌里氏木霉(Trichoderma reesei)作为生物膜生长在充氧空间生态位中,分泌纤维素分解酶来分解纤维素和木糖,兼性厌氧细菌戊糖乳杆菌(Lactobacillus penttosus)将中等大小的糖转化为乳酸,另一个消耗乳酸的专性厌氧细菌用于产物形成,如厌氧梭菌(Clostridium tyrobutyricum)将乳酸转化为丁酸。利用该系统对这3种菌株进行稳定的共培养,并且以纤维素和木糖的混合物作为模型底物,获得了每克碳水化合物0.35 g丁酸的产量,这一值略高于使用可溶性底物或添加商业纤维素分解酶获得的产量。而以榉木为底物时,达到相当于每吨原料196 kg丁酸的产量。此外,研究者也将C. tyrobutyricum与其他厌氧菌交换,也可产生其他短链脂肪酸,包括乙酸、丙酸、戊酸和己酸(图 2A)。
|
| 图 2 合成功能菌群的应用[62, 78, 90] Fig. 2 Applications of synthetic microbial consortia[62, 78, 90]. A: A three-member microbial consortium producing short-chain fatty acids from lignocellulose. A membrane-aerated bioreactor features an oxygen gradient (depicted at the bottom). Aerobic fungus Trichoderma reesei grows as a biofilm in the oxygen-replete spatial niche and secretes cellulolytic enzymes to break down cellulose and xylose to provide soluble sugars. Facultative anaerobic lactic acid bacterium Lactobacillus pentosus converts the soluble sugars to lactate. And then a lactate-consuming obligate anaerobic bacterium is for product formation, such as Clostridium tyrobutyricum, which converts lactic acid to butyric acid[62]. B: Neural-like computing in microbial consortia for 3×3 bit pattern classification. The pattern sets of 3×3-bit patterns include three categories, "z", "v", and "n". In simulated results, the weight W0 results in high enhanced yellow fluorescent protein (EYFP) levels in receivers for all patterns in "z", and leads to low EYFP output for "v" patterns and "n" patterns. The experimental results are consistent with the simulation results. The first four bars represent the "z" group producing high EYFP output, and the rest six bars represent the "n" group and "v" group producing low EYFP output. Meanwhile, the constitutively expressed blue fluorescent protein (BFP) expression levels are uniform across all patterns[78]. C: A co-culture of Ideonella sakaiensis and immobilized Geobacter sulfurreducens degrades PET to produced electricity and CO2 in a three-electrode bio-electrochemical system. In the reaction solution, I. sakaiensis ferments PET or EG into acetate and ethanol, and then, G. sulfurreducens biofilm attached on an inverse opal indium tin oxide (IO-ITO) electrode oxidizes acetate and transports metabolically generated electrons to a poised electrode for electricity production and releases CO2[90]. |
| |
也有研究者利用微生物菌群,通过微生物电合成(microbial electrosynthesis, MES)的手段,高效利用CO2合成乙酸。MES是电活性微生物从固态电极获得电子,利用电极提供的还原力催化CO2或有机物还原,以生成有价值的胞外多碳还原产物的过程[71-72]。MES作为一种绿色可持续的生物固碳技术,结合了电化学、工程学和微生物学的优势,受到越来越多的关注。除了反应器的设计,电极材料和介质组成等外,所使用的生物催化剂对MES的性能也有很大影响[71]。在一些研究中,以混菌作为生物催化剂,表现出了比纯种培养物作为生物催化剂更优的性能[63]。如,Roy等[63]从城市污水处理厂的厌氧污泥中通过生物富集实验获得了可固定CO2的混菌体系,主要包括醋酸杆菌属(Acetobacterium, 26%)、假单胞菌属(Pseudomonas, 12%)和硫螺旋菌属(Sulfurospirillum, 24%)。以未纯化的啤酒厂排放的工业CO2作为微生物电合成的CO2来源,以富集的混菌体系作为生物阴极,实现以0.26 g/(L∙d)的生产速率获得1.8 g/L的乙酸产量。优于以纯种扬氏梭菌(Clostridium ljungdahlii)作为生物阴极(扬氏梭菌可以利用H2作为能源,通过Wood-Ljungdahl途径固定CO2,是MES实验中常用的微生物催化剂)的乙酸产量[最终乙酸产量为1.14 g/L,生产速率为0.14 g/(L∙d)]。
菌群共培养也为非线性生物合成途径的模块化和平衡提供了一个新的视角。途径平衡是使用代谢工程方法进行微生物生物合成的常见和关键挑战。对于非线性生物合成途径,如分支-汇聚型途径,进行生物生产优化特别困难,因为它们需要所有互连的途径模块之间的微妙平衡。菌群共培养策略,可以通过灵活调整携带指定模块的共培养菌株的比例来协调各个途径模块,以达到途径平衡。迷迭香酸(rosmarinic acid, RA)是一种植物衍生的天然产物,属于多酚类化合物,其合成途径属于分支-汇聚型。咖啡酸(caffeic acid, CA)和丹参酸A (salvianic acid A, SAA),SAA是生成RA所需的2种平行前体,这2种芳香化合物以等摩尔比缩合酯化形成RA。CA和SAA都源自酪氨酸生物合成途径,彼此竞争来自同一上游路径的碳通量,在单菌中很难实现CA和SAA合成的微妙平衡。为此,Li等[64]将RA合成途径分成2个上游(CA和SAA)合成模块和1个下游(RA)合成模块,并分配到3株大肠杆菌中,开发了1个三菌株共培养体系。同时为了增加共培养体系的稳定性,研究者改造各模块菌株,使得CA和RA合成菌株优先利用葡萄糖生长,而SAA合成菌株利用木糖生长。通过对每一模块单独优化,并调节3个菌株的接种比例,RA产量达到172 mg/L,与传统的单菌培养策略相比,RA生物合成提高了8倍。
合成生物学的发展促进了细胞编辑能力的极大提升。通过人工设计复杂化合物的生物合成通路,可利用工程菌株合成自然界所不存在的非天然化合物。但是由于设计路线涉及多种异源途径,单个宿主细胞难以为所有酶提供最适环境;同时,大量生物合成基因的过度表达会消耗过多的细胞资源;在路径的不同部分之间也往往存在相互干扰,由此在单个菌株中进行复杂化合物的合成难以轻易克服上述技术障碍。而菌群间的分工合作为以上问题提供了1个解决思路。Feng等[68]在反合成分析的基础上,选择了11个基因进行系统地调节,设计了1条人工生物合成途径以直接从葡萄糖生产高附加值的非天然药物化合物——功能化含氮杂环,5-甲基-2-吡嗪羧酸(5-methyl-2-pyrazinecarboxylic acid, MPCA)以及其合成前体2, 5-二甲基吡嗪(2, 5-dimethylpyrazine, DMP)。由于该合成通路复杂,且酶来源于不同宿主,研究者合理设计了1个大肠杆菌菌群来完成此复杂任务。设计了3个合成模块来执行不同的生物功能,每个模块使用单独的大肠杆菌:1个模块通过将糖转化为所需的苏氨酸;1个模块通过将过量的苏氨酸转化为N-杂环骨架来产生N-杂环;以及1个模块用于吡嗪的最终区域选择性氧官能化。通过在不同阶段连续接种功能菌株,在空间和时间上调节生物转化。最终,使用一锅串联方案,所设计的大肠杆菌菌群成功将葡萄糖高水平转化为MPCA (6.2±0.1) g/L和其前体DMP (7.9±0.7) g/L。
除了合成化学品,微生物菌群还被用来实现多种蛋白质的分布式生产。例如,Villarreal等[69]用含15个或34个大肠杆菌的菌群共培养物共表达了34种原核翻译所涉及的关键蛋白质,其中包含3个起始因子、4个延伸因子、3个终止/释放因子和核糖体循环因子以及23个氨基酰基tRNA转移酶(aminoacyl tRNA transferase, AAT)。通过使用高效的His标签纯化方法成功地纯化了34种不同类型的蛋白质,这34种蛋白质构成了可以合成蛋白质的最小无细胞翻译机制。
迄今为止,通过模块化的菌群共培养策略实现分工合成化合物的应用越来越多,菌群分工为各种化合物的生物合成,特别是结构复杂和生物合成途径更长的复杂化合物的生物合成,提供了有效的方式。然而,尽管菌群分工合成化合物具有多种优势,但要实现这种方法的大规模工业化还需要付出很大的努力,主要挑战包括如何在共培养系统中保持组成菌株的共存,如何维持发酵条件(如pH值和温度),如何提高中间代谢物的交换水平,以及如何设计最佳途径等。不过随着合成生物学技术和基因编辑工具的进步,以及系统生物学的进步带来的计算工具的日益全面和预测能力的提升,未来,可以创建更强大和更稳健的菌群体系,从而在生物经济中生产更多高价值的化合物。
2.2 生物计算生物计算指用生物部件来完成计算。第一批合成生物学论文设计了拨动开关[8]、振荡器[7]和自动调节[73],这些线路可以用作设计计算机的基本组件[74]:内存、时钟和噪声滤波器。在过去20年的合成生物学研究中,工程微生物计算所需的工具得到了广泛的发展。然而,当前在单个菌株中实现的计算仍然相对较简单。进行复杂生物计算受到限制的主要原因在于目前对大规模遗传线路编程的能力仍非常有限,一方面,异源表达的基因线路在细胞内缺乏相互隔离或绝缘,信号通路经常会遭受意外串扰,并且随着遗传线路规模的增长,非正交相互作用也会显著增加,这使得扩展计算复杂性变得困难;另一方面,由于多个异源过程的表达,细胞往往受到高代谢负担的影响。因此,由于信号干扰和代谢负担等限制,利用单个菌株构建复杂遗传线路进行生物计算遇到了瓶颈。而以上限制可以通过构建分布式生物系统,使用多个菌株分布计算来克服,即通过构建菌群,将计算模块分布到不同的菌株单元中,可以减少宿主细胞执行的并行任务的数量,减轻代谢负担,同时在不同的菌株中划分独立的功能也可以防止串扰。
利用菌群进行分布式计算,1个成功的例子是构建模块化的逻辑线路,将复杂的逻辑功能分解为多个亚单元,每个亚单元都在相互通信的不同菌株中设计。Tamsir等[75]在大肠杆菌中构建不同版本的NOR、OR和NOT门(具有不同的输入和输出),后在琼脂表面上使用空间分离的菌落,其中每个菌落都包含1个逻辑门,菌落之间通过正交可扩散的群感效应分子联系,成功构建了所有16个双输入逻辑门。Regot等[76]也通过类似的方法构建了1个酵母菌群,基于酵母信息素(来自酿酒酵母和白色念珠菌的α因子)进行细胞通信,他们构造了所有双输入逻辑函数、多路复用器和带进位的1位加法器。Du等[77]通过设计sender菌株表达小分子,并能被对应receiver菌株感应,开发了6个正交性远超过传统群体感应系统的细胞-细胞通信信道。之后,他们同时使用多达4个正交的通信信道,构建了1个包含7个不同菌株细菌菌群,实现复杂的三输入AND-XOR逻辑门。具体而言,他们设计了7个菌株,每个菌株包含1个NOR门(细胞1–6)或1个Buffer门(细胞7),前3个NOR门(细胞1、细胞2和细胞3)组合成1个AND门电路,后3个NOR门(细胞4、细胞5和细胞6)和最后1个Buffer门(细胞7)组成XOR门电路。在琼脂平板上按照AND-XOR逻辑门的执行顺序,对7个菌株进行空间排列,这样的空间分隔避免了非预期的菌株间相互作用,同时处在邻近置的对应的sender/receiver细菌又可通过可以扩散的通讯分子进行细胞间的信号交流,最终成功实现了3输入、8输出的AND-XOR逻辑门。
除了利用菌群构建复杂逻辑计算,2021年,Li等[78]成功利用大肠杆菌细菌菌群,实现可用于模式分类的类神经计算。具体来说,研究者基于细菌群感效应,构建sender细菌和receiver细菌来进行决策。由化学诱导剂OC6的有无来表示输入模式,如“101”代表“高OC6、无OC6和高OC6”输入模式。OC6可以激活sender细菌产生群感效应分子OHC14。编辑不同的sender细菌,使其具有不同的启动子强度,以产生不同水平的OHC14,获得不同的权重。不同诱导剂输入模式激活1组sender细菌产生OCH14,获得OCH14的加权和。OHC14可扩散进receiver细菌,receiver细菌提供激活函数,将从sender细菌组产生的OCH14的加权和转换成不同的激活状态,从而可对输入模式进行分类。利用所构建菌群,成功对复杂的3×3位的输入模式进行模式分类。具体而言,设计“z” “v”和“n” 3组3×3位模式类别,每个模式类别由1个理想模式和9个噪音模式组成。每个噪音模式都与理想模式有一位翻转。选择了9组sender细菌,对应于每个模式中的9位和权重向量中的9个元素。当以
为了执行更大和更复杂的计算,自然界中的生命系统,比如大脑中庞大的神经网络,便是利用多细胞集合的能力,将计算需求分布至不同的细胞中。模拟自然系统,构建功能菌群分布计算,可降低每个计算单元的负担。如前所述,目前已有许多理论和实验的例子证明了以这种方式分配的好处。不过,目前所构建的可用于生物计算的功能菌群的信息处理能力与自然界中发现的微生物群落相比仍相形见绌。自然界的生物群落可以同时使用合作、互惠、竞争和共生等关系来驱动所需的群落结构以响应环境,这种结构可能会随着时间的推移而变化,而目前人工构建的功能菌群只在人类定义的有限计算范围内表现出色。参照自然,未来增强人工菌群功能的一个努力方向,即是通过提升对细胞间相互作用的调控能力,例如通过细胞群的空间模式[75]和/或正交通信通道[79-80]。
以往,受生物系统中观察到的不同行为的启发,许多计算机科研人员在研究计算范式时,以计算算法的形式试图模仿这种生物系统的固有优势来解决复杂的建模、仿真和优化问题,以此开发了许多算法,如遗传算法、蚁群优化和神经网络等[81-82]。如今,菌群的高度灵活和可重新配置的特性,有助于推动超越经典电子计算机的能力。在计算机科学中,计算机架构描述了系统不同组件之间的结构关系,并限制了算法在系统上的运行方式。由于物理差异,为某一特定计算机架构编译的程序可能无法在另一架构上执行。大多数电子计算机的架构在计算过程中是固定的,并被设计成在1组指令(代码)的指导下快速连续地执行原始的数字操作。虽然这种架构对于处理许多类型的任务来说是足够通用的,但它的静态架构使得它对大多数任务来说都不是最优的。而在菌群中,灵活性和最优性之间的这种严重权衡不需要存在,因为菌群可以动态地改变其物理组成和相互作用,以产生更好地匹配问题的“活架构”。并且菌群的这种重新配置甚至在计算过程中也是可能的,这提供了电子计算机无法比拟的控制和灵活性。利用这些独特的能力将为合成生物学家提供解决问题的新方法,并启发研究人员对新的计算机架构的思考[1, 83]。
2.3 生物降解菌群相较于单一菌株的优势在于菌群可以分工,这在污染物的生物降解中至关重要,因为污染物的降解通常需要多个步骤并涉及多个酶。此外,菌株分工也可以缓解降解产物的抑制作用,提高降解率。同时,通过构建菌群,不同菌株可以同时实现污染物的生物降解和生物转化。目前菌群已被用于降解多种污染物,包括多环芳烃(polycyclic aromatic hydrocarbons, PAHs)[84-85]、有机磷农药[86]、抗生素[87-88]以及聚对苯二甲酸乙二醇酯(polyethylene terephthalate, PET)[89-90]、聚乙烯polyethylene, PE)[91-92]、聚丙烯(polypropylene, PP)[93]、聚氯乙烯(polyvinyl chloride, PVC)[94]和聚氨酯(polyurethane, PU)[95]等塑料废物。
Zhang等[84]从PAHs污染土壤中富集了1个细菌菌群PDMC,主要包括鞘脂菌属(Sphingobium, 58.57%−72.40%)和假单胞菌属(Pseudomonas, 25.93%−39.75%),该菌群能够有效利用菲(phenanthrene, PHE)或二苯并噻吩(dibenzothiophene, DBT)作为唯一的碳源。当以PHE作为唯一碳源进行生长时,PDMC降解PHE的平均速率为6.67 mg/(L∙h),是目前已报道的降解速率最快的PHE降解菌群。同时,该PDMC菌群不仅可利用PHE或DBT作为其生长的唯一碳源,而且还可共同代谢至少9种其他PAHs或杂环衍生物,包括萘(naphthalene, NAP)、苊(acenaphthene, ACE)、蒽(anthracene, ANT)、芴(fluorene, FL)、荧蒽(fluoranthene, FLU)、苯并[a]蒽(benzo[a]anthracene, BaA)、二苯并呋喃(dibenzofuran, DBF)、咔唑(carbazole, CZ)和吲哚。当利用活性污泥反应器和工业废水模拟PDMC对PAHs混合污染水体的修复时,该菌群在5 d内去除了100%的PAHs。并且经过为期24 d的4轮循环反应,该菌群始终稳定具有优秀的降解能力。
2019年,Park等[91]从城市垃圾填埋场沉积物中分离出主要由芽孢杆菌属(Bacillus sp.)和类芽孢杆菌属(Paenibacillus sp.)组成的混合细菌菌群,可以加速微型PE的分解。该菌群可在以PE微塑料作为唯一碳源的液体培养基中生长,培养60 d后,可见细菌菌群在微塑料表面定植。同时,原始粗PE微塑料的平均粒径约为231 mm,与该菌群共培养60 d后,平均粒径降低至176 mm,减少22.8%,颗粒干重也降低了14.7%。而没有接种的对照组,平均粒径仅降低至225 mm,重量减少小于5% [先前的一项研究表明,塑料材料的风化和分解(即暴露在阳光和氧气中)会导致许多小颗粒变形[96]]。
2022年,Kalathil等[90]在生物电化学反应器中共培养大阪堺菌(Ideonella sakaiensis)和硫还原地杆菌(Geobacter sulfurreducens),可以降解PET,并产生电和CO2。大阪堺菌一直被认为是一种好氧菌,其在有氧条件下可以降解PET产生CO2和水,同时通过氧化磷酸化产生ATP。而该研究表明,大阪堺菌是一种兼性厌氧菌,其在无氧条件下可分解PET产生乙酸盐和乙醇。硫还原地杆菌在微生物燃料电池中具有高发电能力,其可氧化乙酸盐,并将代谢过程中产生的电子通过细胞外电子转移呼吸途径传输到电极,同时,硫还原地杆菌可以产生导电的蛋白质纳米线,可将代谢产生的电子传输到不溶性电子受体,如金属氧化物和金属电极。该生物电化学反应系统,在液体反应液中,大阪堺菌将PET发酵产生乙酸盐,随后附着在IO-ITO工作电极上形成生物膜的硫还原地杆菌则氧化乙酸盐产生CO2,并将电子转移到电极用于发电(图 2C)。该系统以PET作为唯一底物时,6 d可消耗23 mg PET膜,并显示出0.6 mA/cm2的最大电流密度。当将该PET转化共培养系统与1个O2还原阴极配对成双电极,可提供约0.4 V的估计电压,这相当于微生物燃料电池的功率密度约为0.2–0.3 mW/cm2[97]。
菌群在复杂环境中具有优越的降解策略,在生物降解方面表现出巨大潜力。不过,目前对于生物降解菌群的研究大多集中在从环境中分离的天然菌群上。近年来,模拟天然微生物群落构建完成复杂生物过程的人工菌群已成为合成生物学的重要研究方向,在代谢工程和合成生物学的帮助下,人工菌群逐渐显示出强大的降解潜力,为复杂底物的高效利用和环境的修复提供新的途径。
2.4 制备活体功能材料与传统材料相比,活体功能材料以活体细胞为结构单体组装材料。将编程后的工程活细胞组装和裁剪成具有生物系统特性的活体功能材料[98-99]。菌群在活体功能材料的制备上,也有诸多应用,例如菌群可调控生物被膜的制备。Dragoš等[100]借助枯草芽孢杆菌(Bacillus subtilis)在形成生物被膜时自发形成3个亚群,互相合作合成胞外多糖(exopolysaccharide, EPS)和结构蛋白TasA,然后形成细胞外间质,进而装成生物膜。之后,Drago等[100]又设计了1个两组分的微生物菌群,分别产生EPS和TasA,通过严格控制合成菌群中劳动分工使得2个种群之间以适当的比例接种时比野生型产生更高质量的生物膜。
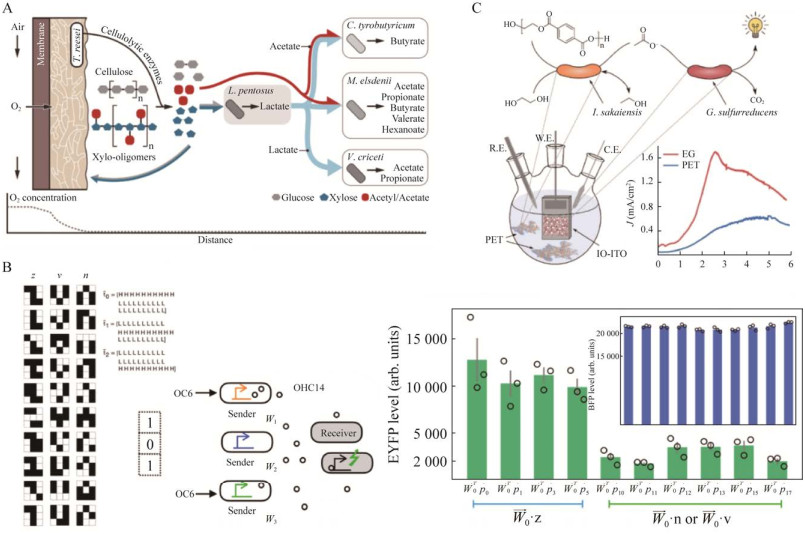
通过编辑菌群中不同细菌的相互作用,可制备快速自愈合的活体功能材料。如Chen等[101]改造大肠杆菌,使其在外膜表达抗原或者对应的抗体,将2种细菌混合后,细菌可通过抗原抗体相互作用产生粘合,从而形成可快速自修复的活体功能材料(LAMBA)。LAMBA相对于单种细菌具有更高的动态模量以及粘度,此菌群构建的LAMBA可直接作为3D打印材料构建宏观的二维或三维精细结构,也可用于构建微米级材料,如组装微米级纤维。同时,由于2种细菌表面抗原和抗体的非共价键的化学作用,该活体材料可以实现分钟级别自愈。研究者进一步将LAMBA与多种可穿戴器件组装在一起,如肌肉电信号传感器以及应变传感器,拓展了活体材料的构建框架和应用领域,尝试了IT与BT的融合(图 3A)。
|
| 图 3 菌群应用材料合成和能源制造[101, 111, 132] Fig. 3 Appling microbial consortia to material production and energy manufacturing[101, 111, 132]. A:可分钟自修复的活体功能材料传感器的制备. 依次通过细菌种群之间的工程粘附导致自主组装,LAMBA的3D打印,菌群分泌蛋白质组装和构建可自我修复的活体功能材料传感器[101]. B:菌群共培养使生物传感和代谢工程结合的例子. 利用微流控装置,在液滴中共培养传感器细胞(大肠杆菌)和生产细胞(酵母)。酵母产生的胞外对香豆酸被液滴捕获,并诱导共培养的大肠杆菌传感器细胞表达报告基因YFP. 随后,在第二微流控装置中,基于传感器细胞的荧光信号对液滴进行分选,以富集高产的酵母克隆[111]. C:大肠杆菌与希瓦氏菌共生关系模型. 葡萄糖被大肠杆菌发酵产生甲酸盐等代谢物,甲酸盐被希瓦氏菌作为电子供体吸收. 在MFC中,黄素介质分子被分泌以促进电子运动到外部电荷收集电极以获得能量. 黄素也可以被大肠杆菌吸收,以促进其在电极上的呼吸,因为大肠杆菌不分泌黄素[132] A: Preparation of engineered living material sensors that can be self-healing in minutes. The self-assembly of bacterial populations by engineered adhesion, the 3D printing of LAMBA, and microbial consortia secreting proteins are used to assemble and construct the self-healing living material sensors in order[101]. B: Microbial consortia enable the combination of biosensing and metabolic engineering. Sensor cells (Escherichia coli) and producer cells (yeast producers) are encapsulated in a droplet in a microfluidic device. Extracellular p-coumaric acid produced by the yeast cells is captured in the droplet and induces the expression of the reporter gene (YFP) in the coencapsulated E. coli biosensor cells. Droplets are subsequently sorted in a second microfluidic device based on the fluorescence signal from the biosensor cells to enrich for productive yeast clones[111]. C: Model of the mutualistic relationship between E. coli and Shewanella oneidensis. Glucose is fermented by E. coli to yield metabolites such as formate, which is taken up by S. oneidensis as electron donor. Flavin mediator molecules are secreted to facilitate electron movement to the external charge collecting electrode to derive energy in MFCs. Flavins can also be taken up by E. coli to facilitate its respiration on electrodes as E. coli does not secrete flavins[132]. |
| |
此外,菌群可以制备感知和响应环境的活体材料。如Gilbert等[102]描述了使用稳定共培养的酿酒酵母和产生细菌纤维素的驹形杆菌(Komagataeibacter rhaeticus)来制造功能性细菌纤维素为基础的生物材料。酵母菌株可将酶分泌到细菌纤维素中,调控细菌纤维素的物理性能并赋予其多种功能。再将工程酵母结合在纤维素基质,创造活体材料,达到可感受化学和光学刺激并作出反应,构建SCOBY系统。
相比于传统材料,利用生物组装材料,使材料具有信号处理、自组装以及自修复等生物动态特征的活体功能材料,在健康、医药和探测等一系列领域拥有巨大应用潜力。不过,当前的活体功能材料主要由单一物种细菌组装,使得活体材料的功能通常简单且单一。相比于同物种菌群,跨物种的合成功能菌群在活体材料领域具备重大潜力及优势。通过利用各物种的特性,可潜在地提升材料编程的多样性,实现功能的涌现。然而,目前多物种菌群的控制仍是一个难题。针对这一难题,基于生物材料介导的空间隔离机制,构建物理分隔的菌群,使得不同物种的微生物间具有相对独立的生长空间,但同时又可保证信号分子、代谢产物和蛋白等的自由交流,这将是制备跨物种菌群活体功能材料的可行方法之一。
2.5 生物传感微生物(如细菌、微藻、真菌、酵母和病毒)具有响应快速和便携性以及能够检测多种化学物质和低成本的优势,使得基于微生物制备生物传感器引起研究者的关注[103]。微生物生物传感器已被用于检测多种信号,如检测环境中的有机和无机污染物[104-107]、疾病生物标志物[108-109]以及生物制造过程中的产品形成等[110-111]。同时,也有研究者使用微生物菌群来制备生物传感器,以提高传感器性能,创建更复杂的传感系统。
微生物传感器可用于检测环境污染物,大多数检测环境污染物的微生物传感器都使用单一物种。然而,由于真实的废水样本是一个包含多种污染物的复杂污染系统,单一微生物具有对某些污染物特别敏感而对其他污染物没有反应的缺陷[112-114],因此单物种微生物传感器不足以客观地检测废水样本的总生物毒性。例如,大肠杆菌对重金属离子反应敏感,但对有机污染物,其急性毒性反应相对较低,而枯草芽孢杆菌与大肠杆菌的敏感性反应则刚好相反。因此,Gao等[104]为了实现对所有类型有毒物质的生物毒性的准确检测,通过共固定大肠杆菌、枯草芽孢杆菌和酿酒酵母组成的混合菌株,构建了用于多污染物毒理学分析的电化学生物传感器,以研究重金属离子、苯酚和农药等5种污染物的单一和联合毒性。结果表明,该混合微生物电化学传感器对5种单一毒物的灵敏度有所提高,可以有效地用于评估有毒混合物的联合效应,并反映真实废水样品的真实生物毒性,可弥补单菌株生物传感器检测范围受限的缺陷。
类似地,Yudina等[105]开发了1个基于稳定人工菌群的检测水样生化需氧量(biochemical oxygen demand, BOD)的生物传感器,相较于基于单一菌株的BOD生物传感器,该传感器可以显著扩大氧化底物的范围。BOD是指在一定条件下,微生物分解存在于水中的可生化降解有机物所进行的生物化学反应过程中所消耗的溶解氧的量,其是监测水环境纯度最广泛使用的指标之一[115]。对于饮用水和清洁水,BOD值不应超过2 mg/dm3;当BOD高于4 mg/dm3时,水被认为是污染的。Arlyapov等[116]共培养毕赤酵母(Pichia angusta)、食腺嘌呤芽生葡萄孢酵母(Arxula adenininovorans)和汉逊德巴利酵母(Debarymyces hansenii),并在此基础上用N-乙烯基吡咯烷酮改性的聚乙烯醇凝胶固定共培养物,以开发BOD生物传感器。相较于基于单一汉逊德巴利酵母制备的生物传感器,基于3种微生物共培养的生物传感器显著拓宽了生物受体氧化的底物的范围,该传感器能够在广泛的BOD范围(2.4–80.0 mg/dm3)内测定各种水样和发酵产物,与标准方法具有高度相关性(R=0.998 8),并且能稳定超过20 d。他们的结果证明了这种共培养物作为高性能生物传感器原型的生物元件的潜力,可广泛应用。
此外,也有研究人员将菌群与电子器件相结合,实现了生物电子与菌群的交互,从而实现了双向信息传递与交换。如Terrell等[117]开发了1个生物电子通信系统,通过氧化还原信号转导方式,实现在活细胞嵌入的生物电子接口和工程微生物菌群之间交换和传递信息。具体而言,首先,电极产生的氧化还原分子(过氧化氢)被用来为激活附着在电极上的工程大肠杆菌的基因表达,有效地创造了1个活的换能器电极。接着这些大肠杆菌解析电子信号,并产生群感效应分子AHL用于信号传导。AHL可被下游两类浮游细胞——验证细胞(verifier cells)和生物驱动细胞(actuator cells)感应,并激活下游浮游细胞,从而在生物学上传播信息。其中验证细胞实现对信号传播的验证,其感应AHL然后表达β-半乳糖苷酶,进而将4-氨基苯基-β-D-吡喃半乳糖苷(p-aminophenyl β-D-galactopyranoside, PAPG)分解成对氨基苯酚(p-aminophenol, PAP),PAP通过电化学氧化转化为电子输出传回电子装置,验证细胞通过直接电子反馈,实现生物信号传播的实时电子验证。AHL也诱导生物激活细胞合成和分泌粒细胞-巨噬细胞集落刺激因子(granulocyte- macrophage colony-stimulating factor, GM-CSF)和红色荧光蛋白,红色荧光蛋白可提供生物驱动细胞对AHL响应功能的光学报告。这项工作通过利用电化学编码和解码信息的氧化还原特异性生物处理,建立了跨越生物电子界面的双向(电子-生物-电子)通信平台,该通信平台的建立也体现了菌群的诸多优势,如信号传播、空间组织和跨种群的分工等。
菌群共培养可以使不同的应用结合,如代谢工程和生物传感。Siedler等[111]编辑生物传感器大肠杆菌,可响应对香豆素酸以产生荧光。之后,他们在液滴中共培养对香豆素酸生产酵母和此检测对香豆素酸的大肠杆菌传感器菌株,并在微流控装置中,通过大肠杆菌生物传感器的荧光信号,快速分选含有可产生大量胞外对香豆素酸的酵母的液滴。微生物菌群为扩大微生物生物传感器的能力和应用提供了一个平台(图 3B)。
目前,由于微生物生物传感器的优点和广泛的应用领域,人们对其愈发关注。利用菌群构建的传感器是提高微生物传感器性能的一种策略,通过包括具有不同代谢能力的不同物种,传感器可以检测和测量的底物范围更广;通过菌群间各成分的协同作用,可提高传感器灵敏度,增加信号输出;通过纳入多个物种,可对波动的环境条件具有更强的弹性和适应力;通过设计和构建组成的不同菌种,可以根据目标物质和环境定制传感器。虽然使用菌群可以提供优势,但具体实施仍存在挑战,如保持物种比例的稳定性、了解物种之间复杂的相互作用,以及为特定应用优化其性能。尽管如此,利用菌群的力量为加强微生物传感器和扩大其潜在应用带来了希望。
2.6 菌群医疗应用在医疗健康领域,菌群也有广泛的应用,由于肠道菌群调控以及粪菌移植方面已有诸多综述,此处我们不再着重讨论该部分内容。随着青霉素等抗生素的发现,人们一度认为细菌感染可以永远治愈。然而数十年来抗生素的滥用和误用已经激发了细菌的进化潜力,从而引发了具有多药耐药性的耐药菌株的产生[118]。一旦这些细菌结合成生物膜,它们的耐药性将更强。这是因为细胞外聚合物(extracellular polymeric substances, EPSs)是生物膜的主要成分,负责维持其结构和新陈代谢,其对细菌起到严格的保护作用,可防止抗菌药物的渗透。因此,迫切需要开发新的药物来解决日益严重的细菌感染问题。为了解决这一难题,Volzing等[119]设计了1个针对革兰氏阴性菌的菌群体系,其将构建好的2种抗菌肽的重组载体pMS-Alyteserin和pMS-A3APO分别转化至乳酸乳球菌,得到1个重组乳酸乳球菌菌群,该菌群诱导后可表达和分泌2种肽。来自该菌群的这2种抗菌肽对病原菌大肠杆菌和沙门氏菌具有较高的抗菌活性。但对乳酸乳球菌本身的抗菌活性明显较低。该菌群系统可作为乳酸菌生产和传递抗菌肽的模型,以杀伤革兰氏阴性致病菌。同时通过菌群制造多肽并将其运送到大肠杆菌或沙门氏菌感染的部位,可以避免以往多肽半衰期短的限制,以及多肽生产和纯化成本高的问题。这项工作证明了利用工程益生菌群递送抗菌肽作为一种补充抗生素治疗特定病原体的巨大潜力。
在另一方面,菌群也被用来消除代谢废物,改善机体内部环境,以达到治疗疾病的目的,如利用菌群治疗肝衰竭、肾衰竭和苯丙酮尿症[120-121]。肝衰竭是一种死亡率高达80%的致命疾病。治疗肝功能衰竭的唯一途径是侵入性手术,但它受限于有限的供体和高昂的费用。为解决这一困境,Zheng等[120]设计了1种具有种子形状、木质素包被和多孔结构的水凝胶微生态系统,该水凝胶模仿种子具有高肠道适应性的特性,通过封装微生物菌群引发的级联反应消除肝衰竭中的有害代谢物。原理是针对氨同化异常和肠道氧代谢异常,构建了1个由枯草杆菌和丁酸梭菌(Clostridium butyricum)组成的双组分人工微生物菌群,其中需氧枯草芽孢杆菌能够通过氧竞争消耗氧气来抑制病原菌的移位,同时微环境中氧的降低促进了厌氧菌——丁酸梭菌的氨耗能力,实现高效肠道氧消耗和随后的缺氧诱导的氨消除,达到治疗肝衰竭的作用。而同样影响着全球5亿患者的致命疾病肾衰竭,它伴随着更严重的有毒废物积累,包括尿素、肌酐、对甲酚硫酸盐、硫酸吲哚、吲哚衍生物和氧化三甲胺,肾衰竭患者通常需要透析以去除含氮废物并减轻肾脏负担。Zheng等[122]为解决这个问题,设计了1个可以在血液中的含氮废物通过肠道粘膜屏障扩散之前就将其代谢的微生物菌群,从而治疗肾衰竭。首先,研究人员从小鼠的粪便微生物群中分离出2株能够有效地将尿素(菌株1,属于大肠杆菌属)或肌酐(菌株2,属于芽孢杆菌属)转化为氨的细菌。同时,考虑到氨的高毒性,还分离到1株具有氨转化能力的肠杆菌(菌株3,肠杆菌属)。然后通过微流控技术,将3种细菌包裹在海藻酸钙微球中以实现空间共定位。最后在微球表面进一步聚合一层聚多巴胺,其可选择性地允许小分子含氮废物进入细菌微生态系统,以排除含氮大分子的过量消耗。最终,在肾脏损伤的动物中口服递送的该菌群可显著降低血液中尿素和肌酐的浓度。
人工菌群可降解的代谢废物种类可以通过进一步的菌株筛选以及合成生物学途径构建来扩大。同时,添加保护性益生菌,也可减轻机体损伤。通过结合不同类型疾病的特点以及治疗目的(如杀伤病原菌和/或清除代谢废物),未来可以进一步构建具有不同代谢特征和功能的菌群,实现个性化治疗。然而,在这个过程中,具体的治疗机制和安全性上仍有一些问题需要进一步研究与解决。目前基于菌群的治疗方法在治疗多种疾病方面仍有很大的发展空间。
2.7 构建微生物电化学系统现今电力作为世界最大的使用能源,每年需要消耗大量的不可再生资源,为了给发电中使用化石燃料提供一种替代方案,微生物电化学系统的研究与利用获得了与日俱增的关注。微生物电化学系统是1种利用微生物从生物有机材料中获取化学能以产生电能的新兴技术,其利用微生物的生物催化活性,从可生物降解的有机物质中获取电子,从而探索微生物细胞(电子供体)与电极表面(电子受体)之间的相互作用[123]。
微生物燃料电池(microbial fuel cell, MFC)是1种常见的微生物电化学系统,它利用微生物氧化有机底物将化学能转化为电能,可作用于废水修复、海水淡化和去除环境中的有毒化学物质[124-127]。在构建MFC时,阳极室中用作催化剂的微生物起着重要作用。目前,使用单一微生物或微生物菌群构建高性能的MFC均有例子[128-129]。不过利用菌群构建的MFC,对复杂环境适应能力强,有助于增强电化学活性等优势。如Venkataraman等[130]共培养铜绿假单胞菌(Pseudomonas aeruginosa)和产气肠杆菌(Enterobacter aerogenes)所制备的MFC的电流密度比这2种细菌中任何1种单独培养的电流密度至少增加14倍。主要机制是产气肠杆菌将葡萄糖主要发酵成2, 3-丁二醇,随后被铜绿假单胞菌消耗,该发酵产物2, 3-丁二醇刺激了铜绿假单胞菌和产氧肠杆菌之间的互利相互作用。Rosenbaum等[131]比较了希瓦氏菌在纯培养和与乳酸乳球菌共培养条件下的生物电化学性能,虽然单独的希瓦氏菌只能使用乳酸作为电子供体来产生电流,但共同培养能够将葡萄糖转化为电流,其库仑效率可达17%。为了进一步提升转化效率,由大肠杆菌和希瓦氏菌(Shewanella oneidensis)组成的MFC被构建。大肠杆菌发酵葡萄糖生成甲酸盐等代谢物,这些代谢物可被希瓦氏菌吸收为电子供体。希瓦氏菌分泌黄素可促进大肠杆菌在电极上的呼吸,它们的活性增加了MFC中从阴极到阳极的电流。同时,分泌出的黄素可促进电子向外部电荷收集电极运动,从而在MFC中获得能量[132](图 3C)。Qu等[133]构建了基于大肠杆菌和硫还原地杆菌菌群的MFC,他们证明大肠杆菌和硫还原地杆菌的共培养相对于硫还原地杆菌的纯培养,由于大肠杆菌消耗了泄漏到反应器中的氧气为硫还原地杆菌提供缺氧环境,系统性能得到了改善,将库仑效率提高到了约85%。
生物光伏(biophotovoltaics, BPV)是近年来MFC研究的一个较新的领域,其也被称为光微生物燃料电池(photosynthetic microbial fuel cell, PMFC)或微生物太阳能电池,是利用光合微生物(如蓝藻)将太阳能转化为电能的新兴技术[134-135]。与传统光伏技术相比,BPV以光合微生物作为光电转换材料,其无毒且可再生,更加环保,近年来受到越来越多的关注。但目前生物光伏发电最大的问题是,BPV系统的输出功率远落后于实际应用,主要原因是蓝藻等光合微生物虽然具有很高的光合效率,但产电活性很弱,难以从光合作用电子传递链(photosynthetic electron transfer chain, PETC)中提取电子到细胞外电极。近年来,有研究人员通过构建功能菌群的方式来解决这一难题[136-138]。如Zhu等[137]构建了由工程光合蓝藻和工程产电希瓦氏菌组成的功能菌群,其中蓝藻可吸收光能并固定二氧化碳来合成能量载体d-乳酸,而希瓦氏菌通过氧化d-乳酸进行产电,由此形成1条从光子到d-乳酸再到电能的定向电子流,完成从光能到化学能再到电能的能量转化过程。由该菌群构建的BPV系统可产生150 mW/m2的功率密度,并稳定维持了40多天。并且在该系统中,大约10.3%的总固定光能被分流到最终用于发电的能量载体中。该系统表明,具有定向电子流的合成菌群可以促进光合微生物向产电细菌的能量转移,从而实现更高的电子提取效率。之后为了进一步提高BPV性能,Zhu等[138]构建了1个四菌功能菌群,包含初级生产者(蓝藻)﹑初级分解者(大肠杆菌)和终端消费者(希瓦氏菌和地杆菌)。其中,工程化的蓝藻负责吸收光能并将电子储存为蔗糖,工程化的大肠杆菌负责将蔗糖降解为乳酸。随后,乳酸被希瓦氏菌和地杆菌完全氧化,生成的电子被转移到胞外电极上发电。在该四菌BPV系统中,蓝藻总固定光能的约70%被转化为电能,获得的最大功率密度为1 700 mW/m2。同时,为了克服蓝藻光合放氧与异养微生物厌氧产电之间存在的矛盾,研究人员开发了1种导电水凝胶来封装大肠杆菌、希瓦氏菌和地杆菌,以隔绝氧气。最后,将4种细菌组装成1个集成的BPV系统,实验表明,该系统可直接将光能转化为电能,并稳定运行1个月以上。该由功能菌群构建的BPV系统将生物光伏效率提高到新水平,表明应用功能菌群是提取光合电子以提高功率密度和延长BPV系统寿命的有效策略。
近年来,无论是以有机质作为生物体燃料的MFC,还是利用光合微生物将光能转化为电能的BPV,都有了很大的发展,有希望成为一种可持续和环保的能源技术。而在这2种技术中,功能菌群都展现了显著的提升系统性能的应用潜力。如在以微生物作为催化剂的MFC中,由单一微生物构建的MFC系统的发电能力和对复杂环境的适应能力要小于菌群构建的系统;同时,单一微生物对操作条件要求相对严格,只能使用选择性底物,而菌群更适合使用复杂底物。而BPV系统中,工程菌群也展现了促进光合微生物的电子输出、提高功率密度的能力。不过,目前这2种系统仍然由于输出功率较低、反应速度慢和对环境敏感,并且微生物菌群的类型对电化学特性也有很大影响等局限,其实际应用受限。尽管有这些限制,正在进行的研究和技术进步将继续解决这些问题,并探索提高微生物燃料电池的效率、可扩展性和实用性的方法。
3 总结与展望综上所述,本文对近年来合成功能菌群设计原则和构建方法以及应用等进行了回顾及总结。可以看到,随着合成生物学的快速发展,合成功能菌群领域也取得了诸多研究进展。而在未来,领域内仍然有诸多方向亟待解决及探索。
首先,仍然缺乏对菌群组分进行精准的时空调控的能力。通过设计基因线路编辑群落间相互作用是一种方法,但是通常受制于跨物种通讯工具的有限性[139]。因此,拓展跨物种间的通讯工具将对构建时空可调的跨物种菌群有着关键意义。即使存在着通信工具,调控细菌密度的模块(产生抑制的毒素等)通常会给菌群成员带来代谢负担。同时,随着群落中的微生物种类和数量、关系复杂性的上升,基于约束的代谢回路设计方式已经不再适用。构建物理分隔的菌群是一种替代方法,这种方法普适于所有种群。而在构建多组分菌群时,利用自动化(automation)产生大数据并结合机器学习(machine learning)将有助于弥补诸如计算能力和数学模型的缺失,获得复杂的合成功能菌群。
其次,目前的合成功能菌群依然主要利用模式生物,一个主要原因在于模式生物具备大量的已知元件及编辑方法。然而很多具备特定生物学功能的菌株为非模式生物,例如人体和动物的肠道微生物、植物根际微生物。针对非模式微生物的培养方法、基因编辑技术和功能基因组学都将极大拓展合成菌群的功能多样性[140-143]。
最后,目前构建的大部分合成功能菌群都只包含异养生物,为了维持菌群的代谢,需要持续不断地向系统提供营养。将光能自养生物纳入合成功能菌群可以利用生物体自身的光合作用系统将光能转换成化学能,为整个菌群提供能量,使得菌群不再需要依赖外源的营养补给[144-145]。目前已有一系列工作构建了光能自养的菌群,主要聚焦在蓝细菌以及大肠杆菌。通过分别改造蓝细菌及大肠杆菌,使蓝细菌可以分泌蔗糖,而大肠杆菌可以利用蔗糖生长。而在近期工作中,Li等[146]通过构建蓝细菌-需钠弧菌的功能自养菌群,实现了一系列化学品的负碳合成(carbon-negative biosynthesis)。而在未来,期待看到更加多样化、具有良好适配性的光能菌群,促进生物能源等一系列领域的可持续性发展。
 2023, Vol. 39
2023, Vol. 39