中国科学院微生物研究所、中国微生物学会主办
文章信息
- 薛珊, 王淑雅, 刘丽, 钟巧芳, 程在全, 肖素勤
- XUE Shan, WANG Shuya, LIU Li, ZHONG Qiaofang, CHENG Zaiquan, XIAO Suqin
- 基于CRISPR/Cas9的精准基因编辑技术研究进展
- Precision gene editing technologies based on CRISPR/Cas9: a review
- 生物工程学报, 2023, 39(7): 2566-2578
- Chinese Journal of Biotechnology, 2023, 39(7): 2566-2578
- 10.13345/j.cjb.220879
-
文章历史
- Received: November 4, 2022
- Accepted: February 7, 2023
- Published: February 10, 2023
2. 云南省农业科学院生物技术与种质资源研究所, 云南 昆明 650205
2. Biotechnology and Germplasm Resources Institute, Yunnan Academy of Agricultural Sciences, Kunming 650205, Yunnan, China
基因编辑技术近年来发展迅速,基因编辑的出现,让更加精准的基因调控成为现实,可以通过基因插入、基因敲除和碱基替换等对基因组靶位点进行一系列的人工修饰,以获得新的功能或表型。传统的基因编辑工具主要包括锌指核酸酶(zinc finger nucleases, ZFNs)[1]、转录激活-效应器核酸酶(transcription activator-like effector nucleases, TALENs)[2]和聚集的规则间隔短回文重复序列(clustered regularly interspaced short palindromic repeat, CRISPR)[3]。
ZFN和TALEN是第一代和第二代技术,它们都包含DNA识别结合域和DNA切割域[4]。在实际应用过程中,ZFN和TALEN基因编辑技术都需要在靶点两侧各设计一个锌指蛋白(ZFN)结构域或TAL效应子(tal effector, TALE)蛋白结构域,靶位点位于两个结合位点之间,两个特异蛋白分别结合到结合位点后,2个核酸内切酶Fok I相互作用形成二聚体,在靶序列处产生DNA双链断裂,最终实现基因编辑。但是,ZFN和TALEN基因编辑技术存在目标识别率低、成本高、脱靶率高、结构复杂及需要设计特定的蛋白质识别靶DNA等缺点。于是,研究者基于细菌和古生菌免疫防御机制开发了第三代基因编辑技术CRISPR/Cas基因编辑系统。CRISPR基因编辑系统依赖于2个关键组件:CRISPR相关Cas蛋白和单链向导RNA (small guide RNA, sgRNA)。Cas9是一种与sgRNA结合的核酸酶,通过sgRNA中存在的20 nt核苷酸序列激活Cas9并靶向特定基因组位点,称为原间隔区相邻基序或PAM位点(protospacer adjacent motif, PAM)。Cas9随后在靠近PAM位点切割双链DNA,造成DNA双链断裂(double-strand break, DSB),非同源末端连接(non-homologous end joining, NHEJ)对DSB的低保真度修复将在DSB位点随机引入碱基的插入/缺失,从而在靶位点产生突变[5-7]。但传统的CRISPR/Cas9编辑系统精准度不够,于是,基于CRISPR/Cas9系统的精准编辑技术应运而生。
1 单碱基编辑技术发展单碱基编辑技术可以应用于通过精确改变单个碱基以实现关键氨基酸改变的研究,除了可以通过引入终止密码子实现基因的功能缺失突变,还可以对一些启动子区的调控位点进行改变以实现对基因的表达调控。此编辑器的开发在遗传疾病的基因治疗、生物性状的改造等领域具有重要意义。
1.1 胞嘧啶碱基编辑系统2016年,哈佛大学David Liu团队[8]使用专门设计的核酸内切酶失活的Cas9蛋白(nuclease-dead Cas9, dCas9)开发出胞嘧啶碱基编辑器(cytosine base editors, CBE),通过胞嘧啶脱氨酶将胞嘧啶(cytosine, C)转化为尿嘧啶(uracil, U),在DNA复制和修复过程中又将U转化为胸腺嘧啶(thymine, T),从而实现C.G-T.A碱基对的自由转换。这种技术不需要产生DNA双链断裂就可以对基因组特定碱基进行高效替换。第一代胞嘧啶碱基编辑器CBE1由大鼠胞嘧啶脱氨酶(rAPOBEC1)和dCas9组成,CBE1在体外编辑效率可达到25%–40%,但在哺乳动物体内编辑效率较低,编辑效率最高只有7.7%。研究人员发现主要原因是细胞内存在尿嘧啶DNA糖基化酶(uracil DNA glycocasylase, UDG),该酶可以识别UG配对,并通过碱基切除修复途径(base-excision repair, BER)将UG配对修复为CG配对。因此在第二代胞嘧啶编辑器CBE2中,研究人员在CBE1基础上融合了尿嘧啶DNA糖基化酶抑制剂(uracil glycosylase inhibitor, UGI)。这样就可以抑制UDG的作用,使得CBE2编辑器在哺乳动物的编辑效率最高可达到20%。为了提高胞嘧啶碱基编辑器的编辑效率,研究人员研发了第三代胞嘧啶碱基编辑器CBE3,研究人员将CBE2的dCas9换成具有单链切割活性的Cas9蛋白(nicking Cas9, nCas9),可以特异性地在靶标链上产生一个切口,从而可以刺激细胞内的碱基错配修复途径(mis-match repair, MMR)以含有U的非靶标链作为模板进行修复,从而提高编辑效率,该编辑器编辑效率是CBE2的2–6倍。但是CBE3依旧存在一些问题,例如会将C转换为G或者A,于是研究人员在CBE3的基础上融合了第二个UGI,增强了对UDG的抑制作用。
在单碱基编辑技术中,开发更灵活的工具十分重要。例如,优化密码子序列和核定位序列等方法获得了编辑效率更高的BE4max,编辑效率增加了1.9倍[9]。2020年李大力团队[10]开发了超高活性胞嘧啶碱基编辑器hyCBE。该编辑器在CBE3的基础上,将编辑窗口内的胞嘧啶脱氨,可以提高靠近PAM序列的C-T的编辑效率。该碱基编辑新工具的开发,有望为基因治疗提供自主研发的新工具,提升遗传疾病基因疗法精准性和长效性。
1.2 腺嘌呤碱基编辑系统2017年,David Liu团队[11]还报道了腺嘌呤碱基编辑器(adenine base editor, ABE),它促进腺嘌呤(adenine, A)的突变,通过使用腺嘌呤脱氨酶将其转化为次黄嘌呤(inosine, I),I被聚合酶识别为鸟嘌呤(guanin, G),实现了A.T-G.C碱基对的转换。这些传统的ABE是将nCas9和腺嘌呤脱氨酶组成融合蛋白,由起导航作用的sgRNA引导至靶位点发挥碱基替换作用。然而,sgRNA对DNA序列的错误结合有一定的宽容性,可以在错误的基因位点形成脱靶编辑效应[12-13]。2022年中国科学院广州生物医药与健康研究院赖良学研究员与五邑大学副教授邹庆剑团队[14]合作,首次将腺苷脱氨酶与转录激活因子样效应子(transcription activator-like effectors, TALE)融合,开发了一种不会产生Cas9依赖性脱靶的新型碱基编辑系统TaC9-ABE。在TaC9-ABE碱基编辑系统中,nCas9与sgRNA连接,而腺嘌呤脱氨酶与另一个具有导航作用的TALE相连接。nCas9在sgRNA引导下,结合到目标DNA位点,打开并切割单链DNA。同时,腺嘌呤脱氨酶在TALE引导下结合到相同的靶位点,脱氨酶即可对靶向链DNA进行编辑,实现靶位点的A到G突变。如果nCas9被gRNA带到错误的位点,由于没有脱氨酶的存在,A到G的转换就不能发生。TaC9-ABE碱基编辑系统在保证高效碱基编辑的同时降低基因脱靶效应。
1.3 糖基化酶碱基编辑器2020年,中国科学院天津工业生物技术研究所张学礼和毕昌昊团队[15]构建了胞嘧啶脱氨酶-nCas9-Ung蛋白复合物,创建了一个新的糖基化酶碱基编辑器(glycosylase base editor, GBE),该系统将尿嘧啶糖基转移酶(uracil-N-glycosylase, UNG)带到由胞嘧啶脱氨酶形成的尿嘧啶碱基位点,在该位点脱去尿嘧啶,建立无嘌呤(apurinic)/无嘧啶(apyrimidinic)位点,DNA损伤位点诱导启动DNA修复,从而可以诱导嘧啶与嘌呤之间的碱基颠换。GBE作为新一代碱基编辑技术,摆脱了传统碱基编辑依赖多次DNA复制的缺点,直接将目标碱基特异性修改成目的碱基,进一步完善了碱基编辑系统。但是该编辑器依然存在编辑效率较低和靶向位点范围受限等问题。2022年张学礼和毕昌昊等[16]将转录激活因子VP64融合表达在GBE编辑器脱氨酶的氨基端(Vp64-APOBEC-nCas9-UNG),结果表明Vp64-APOBEC-nCas9-UNG的C-G编辑效率比GBE有显著提高。除此之外,研究人员分别融合表达了SunTag系统与SpRY-Cas9,构建了SunTag-GBEs和SpRY-GBEs。这两个系统在编辑效率和编辑范围方面都比GBE有较大提高。该碱基编辑技术的突破,进一步完善了碱基编辑系统,提高了我国生物技术能力,将为生物产业核心关键技术的自主创新发挥重要作用。
2 引导编辑技术发展CBE和ABE可用于执行4个碱基转换(即C-T、G-A、A-G、T-C)。然而仍然缺乏有效的工具来诱导其他8种可能的碱基转变(C-A、C-G、G-C、G-T、A-C、A-T、T-A、T-G)以及碱基的插入和删除[17-18]。2019年10月,David Liu团队[19]开发了引导编辑,可以高效地执行12个碱基置换,而无需依赖DSB和供体DNA。该技术使用与nCas9融合的逆转录酶和prime编辑扩展的gRNA即引导编辑向导RNA (prime editing gRNA, pegRNA),这可使分子剪刀更加稳定和精确。该技术通过在sgRNA的3′端引入引物结合位点序列(primer binding site, PBS)并将其与nCas9断裂的非靶标链结合,逆转录酶根据其携带的RT模板逆转录出相应的含有目的突变的单链DNA,该细胞通过DNA修复进一步将目的突变引入基因组[20-21]。该技术可以有效地产生精确的碱基置换、插入和缺失等变异,极大地扩大了基因组精准编辑的范围和能力。
为了分析植物基因组的功能,实现精确作物育种,张勇团队[22]在此基础上开发了3种版本的植物碱基编辑器,利用PE2、PE3和PE3b对10个水稻基因进行靶向编辑,最高编辑效率达到1.55%。但是基因编辑技术的应用必须考虑到脱靶的可能性。2021年4月,高彩霞团队[23]构建了适用于植物的引导编辑器(plant prime editor, PPE)系统,在植物细胞及植物个体两个水平上对引导编辑系统的脱靶效应进行了深入评估。该团队首先检测了植物细胞中PE的编辑错配耐受性,发现编辑频率受pegRNA引物结合位点和间隔区错配的数量和位置的影响。该团队还评估了12个pegRNA在179个预测的脱靶位点的活性,通过对使用PE编辑技术编辑的29株水稻全基因组测序数据进行评估,发现PE编辑不会导致单核苷酸变异(single nucleotide variation, SNV)或小的插入或缺失。成功获得了具有单碱基、多碱基的水稻突变体植株,效率高达21.8%,这是现有基因编辑系统无法实现的。
引导编辑在问世后得到了广泛的应用,然而却有一个瓶颈尚未突破,就是它只能对有限长度的基因进行删除。如果一段序列超过100个碱基,就会影响这套系统的编辑效率。2021年10月薛文团队[24]在引导编辑中使用了具有完整功能的Cas9蛋白,再将其与逆转录酶融合在一起,这样的融合蛋白一次能切开两条DNA链。同时额外加入了一条pegRNA,靶向DNA上互补的序列,而逆转录酶合成的两段新DNA则形成互补的黏性末端。当这两个末端相结合时,就可以将原本位于中间的大段DNA删除掉。2021年Jay Shendure团队[25]利用引导编辑最初使用的特殊Cas9蛋白,该蛋白每次能切开一条DNA,但在两条pegRNA的作用下,却能达到删除中间序列的效果。研究者在人类肾脏细胞中对该系统进行测试,发现该系统可删除20–10 000个碱基。这两个团队开发的引导编辑系统都可一次性精准切除至多10 000个碱基,这大大拓展了引导编辑的应用范围。
3 双碱基编辑技术单碱基编辑器只能催化单一碱基类型的转换,限制了其广泛应用。因此,开发一种新的碱基编辑技术,可以有效地同时产生两种不同的碱基突变,将极大地丰富碱基编辑工具。李大力团队[26]将胞嘧啶脱氨酶hAID-腺嘌呤脱氨酶-Cas9n (SpCas9 D10A突变体)融合在一起,开发出了一种新型双功能碱基编辑器:A & C-BEmax。该编辑器可以有效地在同一等位基因的靶序列上转换C-T和A-G。该研究测试了HEK293T细胞中的28个内源性靶点,结果显示A & C BEmax的C-T编辑窗口从C3–C10位扩展到C2–C17位,在C7–C17位的编辑活性是AID-BE4max的1.9–14倍[27]。Nozomu Yachie团队[28]将来源于七鳃鳗的胞嘧啶脱氨酶(PmCDA1)和鼠源的胞嘧啶脱氨酶(rAPOBEC1)分别和腺苷脱氨酶(TadA)融合到nCas9的C端和N端,开发了3种新的双碱基碱基编辑器Target-ACE/Target-ACEmax/ACBEmax。使用该系统,研究人员在哺乳动物细胞中实现了A/C的同时突变,平均编辑效率最高分别达C的50%和A的40%。中国科学院遗传与发育生物学研究所的高彩霞团队和李家洋团队[29]在nCas9的N端融合了胞嘧啶脱氨酶(APOBEC3A)和进化的tRNA腺嘌呤脱氨酶(ecTadA-ecTadA7.10),以这种方式构建了4种新型的双碱基编辑器(saturated targeted endogenous mutagenesis editor, STEME),称为STEME-1至STEME-4。这些编辑器具有在单个sgRNA的指导下诱导C-T和A-G靶位点同时突变的独特优势,并且该技术显著地提高了靶基因碱基突变的饱和程度和突变类型的多样性。
4 转座子类基因编辑基因编辑工具的功能通常是破坏双链或单链DNA。长期以来,人们一直希望开发一种不破坏DNA链的精确的编辑方法,转座子类基因编辑可以满足此需求。转座子(transposon),是基因组中一段可移动的DNA序列,可以通过自己编码的转座酶将自己从一个位置切割并插入或直接插入到一个新的位置。因此,研究人员试图利用转座子将所需DNA序列插入目标位点,构建转座子类基因编辑技术,通过该技术可以将目的序列直接引入并插入细胞基因组中,而不会造成细胞破坏。
4.1 INTEGRATE2019年6月,哥伦比亚大学Charles Gersbach团队[30]在霍乱弧菌(Vibrio cholerae)中发现了一种独特的转座因子后,构建了一种名为INTEGRATE (通过gRNA辅助靶向插入转座因子)的基因编辑工具。在这项技术中,大的基因片段可以插入基因组,而不会引入DNA断裂。Halpin-Healy等[31]发现转座子可以整合到细菌基因组的特定位点,而无需切割DNA。在该系统中,基因编辑工具能够将任何DNA序列插入细菌基因组中的任何位置。对编辑后的细菌进行测序证实,INTEGRATE实现了精确插入,在非目标位置没有额外的拷贝。与CRISPR类似,整合酶通过gRNA找到了合适的位点。
4.2 CAST2019年6月,张峰团队[32]从蓝藻(Scytonema hofmanni Ag.)中获得了转座酶,其中3个亚基与CRISPR效应蛋白Cas12k相关,该系统被称为CAST或CRISPR相关转座酶(CRISPR-associated transposase),其中Cas12k用于搜索基因组中的特定序列位点。转座酶将基因片段直接插入靶位点,因为不是同源重组,所以这一过程具有安全上的优势。该CAST系统将nCas9与单链DNA转座酶的TNPA偶联,然后检测大肠杆菌(Escherichia coli)基因组中的蛋白质复合物,这种复合物可以促进外源DNA位点的特异性整合。这表明可以使用转座子实现基因敲除。然而,单链DNA模板的制备和体内递送仍然存在困难。
该团队随后研究了不同的CAST基因和不同长度的tracRNA对CAST系统活性的影响。结果发现4种CAST基因对外源基因整合至关重要,216 nt的tracRNA足以产生外源基因的整合。此外,研究小组还证实,这种整合只发生一次,从而避免了多次基因插入。尽管尚未在细菌之外进行研究,但CAST仍有望成为下一代高效基因编辑系统[33]。
5 其他基因编辑技术的开发除了CRISPR/Cas9系统,研究人员还希望使用一些具有编辑或序列识别的新酶,为研究人员创造新的基因编辑技术。并且传统编辑技术在商业应用中使用时会收取高额专利许可费,如何扩大基因编辑工具的使用范围,避免高额许可费,也成为研究人员开发新的编辑工具的目的之一。
5.1 MAD7受生物多样性的启发,美国公司Incripta开发了一种新的CRISPR核酸内切酶,称为MADzymes[34]。与Cas9相比,MADzymes能够识别不同PAM序列并且脱靶率更低,最重要的是,MAD7核酸酶是第一种不受知识产权限制的新型核酸酶,可以促使CRISPR工具在学术和商业环境中广泛使用的MAD酶。2021年邱金龙团队[35]利用MAD7实现植物基因组编辑,并且发现该系统的编辑效率与在植物中应用较多的CRISPR/LbCas12a系统相差无几。除此之外,研究者还开发了一种MAD7新变体,大大拓宽了编辑范围。利用该系统在植物中实现了高效多基因组编辑,如在水稻再生植物中,利用该系统获得的突变体的比例最高可达65.6%。
5.2 CRISPR/CasΦ2020年7月,Jennifer Doudna团队[36]发现了一种新的超紧凑CRISPR/Cas系统,称为CRISPR/CasΦ。该系统由一个70 kDa的CasΦ蛋白和一个仅存在于巨大噬菌体中的CRISPR序列组成。与常规Cas蛋白相比,CasΦ异常微小,但功能完整,可以通过单个活性位点即可生成成熟的CRISPR RNA (crRNA)并切割外源性DNA。与最广泛使用的CRISPR/Cas9和CRISPR/Cas12a系统相比,CRISPR/CasΦ系统展示出了更广泛的靶识别基因序列的能力。CasΦ分子量仅为Cas9或Cas12a基因组编辑酶的一半[37],体积较小,再加上CasΦ系统能够将多种功能结合到单个蛋白质中,因此传递到细胞中更容易,这点是基因编辑的一个重大突破。研究人员将CasΦ作为核糖核蛋白(ribonucleoproteins, RNPs)递送到拟南芥(Arabidopsis)原生质体中以编辑拟南芥PDS3基因,测序结果发现CasΦ引入8–10 bp的缺失。与其他3种Cas酶相比,CasΦ仅需CasΦ中单个RuvC活性位点便能进行pre-crRNA处理与DNA切割。小体积的CasΦ再加上对PAM的需求较小,对于基于载体的递送和更广泛的靶向基因组序列都特别有利[38]。CasΦ展示的巨大基因编辑潜力,极大扩展了基因组编辑的工具箱。
5.3 SPG and SPRY哈佛医学院的Benjamin Kleinstiver在2020年3月表示,研究人员长期以来一直致力于优化Cas9,提高其与不同PAM序列的兼容性,希望有一天不再需要PAM序列。为此,Benjamin Kleinstiver团队[13]设计了不需要特定PAM就能结合和切割DNA的Cas9蛋白变体,命名为SpG和SpRY。通过在人类细胞的编辑测试,发现SpG在识别NGN方面比SpCas9和其他变体效率更高。进一步实验表明,SpG对NGG的编辑活性为51.2%,其他3类则达到了53.7%。该研究开发的Cas9突变体SpRY是目前与PAM序列最兼容的Cas9突变株,几乎完全消除了PAM序列的限制,大大提高了其在基因组中的编辑能力。由此产生的基因编辑系统将精确编辑扩展到几乎整个基因组[39-40]。
5.4 TALED虽然基因编辑技术在细胞核基因组取得了巨大成功,但科学家们在编辑具有线粒体基因组方面一直没有突破性成果。这主要是因为线粒体是能量代谢的重要细胞器,如果基因发生突变,就会导致身体出现与能量代谢相关的严重遗传疾病。可喜的是,韩国大田的基础科学研究所Jin-Soo Kim研究团队[41]开发了一种可编程工具,称为转录激活因子样效应子相关脱氨酶TALED。TALED是能够在线粒体中进行A到G碱基转换的碱基编辑器。这一发现是长达数十年治愈人类遗传疾病之旅的结晶,为治愈多种线粒体遗传病带来了新的生机。
5.5 CRISPRi和CRISPRa在全基因组范围内靶向基因调控是了解基因功能的有效方法,CRISPR/Cas9系统作为功能基因组筛选工具,可以帮助人们在全基因组范围内开展功能研究。近年来,CRISPR/Cas9工具箱已经大大扩展,新增了CRISPRi和CRISPRa这两种工具。
CRISPRi利用无酶活性的dCas9融合KRAB转录抑制结构域,dCas9-KRAB融合蛋白在gRNA指引下,结合到靶基因TSS位点,由此抑制目标DNA的转录,从而沉默靶基因的表达[42]。与传统的CRISPR/Cas9技术相比,CRISPRi无需切割DNA就能够更精确、有效的沉默基因。Bruce Conklin团队[43]同时用CRISPR/Cas9和CRISPRi技术对诱导性多能干细胞(induced pluripotent stem cell, iPSC)基因进行沉默,发现CRISPRi可以使细胞中95%以上的靶基因沉默,而采用CRISPR/Cas9技术沉默基因效率为60%–70%。并且CRISPRi在基因沉默过程中没有造成任何非预期的突变。CRISPRi的作用类似于RNAi,但是RNAi针对成熟的RNA,而CRISPRi则是在DNA水平阻止转录的起始(表 1)。因此,与RNAi相比,CRISPRi可靶向lncRNA、microRNA、反向转录产物、细胞核内的转录本等,是研究非蛋白编码基因的有力工具。因此CRISPRi系统非常高效且应用广泛。
| Difference | CRISPRi | RNAi |
| Target site | DNA transcription initiation site | RNA |
| Work area | Nucleus | Cytoplasm |
| Object | Multiple transcripts, including mRNA, non-coding RNA and microRNA | Mainly mRNA |
| Experimental operation | Relatively complex | Simple |
| Off-target | Low | Higher |
| Interference efficiency | High | Higher |
CRISPRa是研究人员研发作为基因过表达的新技术之一。它通过dCas9与不同的转录激活子结合发挥上调靶基因表达的作用。为了加强激活效应,研究人员通过改进融合蛋白募集多个转录激活因子或具有多个激活结构域,这些系统包括多拷贝数的VP16、VPR (VP64-p65-Rta)及Sun Tag (募集多个抗体-VP64复合物)[44-46]。Kampmann[47]对dCas9和sgRNA进行优化,然后用优化过的CRISPRa激活10个基因,发现这些基因的活性提升了好几倍,而且还发现离转录起始位点越近,激活的效果越强,并且发现融合多个转录激活因子的增强效果高于融合单个转录激活因子。与传统的过表达技术相比,CRISPRa通过激活内源性启动子高效转录,从而促进基因表达,不需要额外构建外源性表达元件,不受基因转录本大小的限制。CRISPRa与过表达技术对比如表 2所示。
| Name | CRISPRa | ORF/transgenic expression |
| Number of components | ≥2 | 1 |
| Extent of overexpression | Can achieve high overexpression by multiplexing sgRNAs or activator domains | Can achieve high overexpression |
| Specificity | Very high, but caveat of bidirectional promoters | Very high |
| Limitations for long ORFs | No | Yes |
| Differential overexpression of splice isoforms | No | Yes |
| Expression of variants not matching host cell genotype | No | Yes |
基因编辑自开发以来,发展迅猛。基因编辑技术不仅在微生物、植物和动物中应用广泛,而且还延伸到人类健康领域,尤其是近年来,基因编辑技术对人类基因治疗作出了积极贡献。例如,通过CRISPR/Cas9基因编辑纠正了地中海贫血突变的碱基,并利用编辑后的CAR-T细胞潜在地提高人体对血癌的抵抗力。除此之外,基因编辑技术也促进了农业育种的发展,在创造新的农艺性状和提高作物抗逆性和抗病性方面显示出巨大的潜力。尽管如此,基因编辑技术仍有几个障碍需要克服。
6.1 降低基因编辑脱靶效应不同的基因编辑工具都存在很高的脱靶问题,在经典的CRISPR/Cas9编辑中,sgRNA引导Cas9酶结合到特定位点进行切割。一般来说,sgRNA识别序列约为20 bp,但Cas9允许在一定的容错率内进行错配切割,例如,在目标序列外形成突变,或者sgRNA设计中的高GC含量导致碱基不匹配或偏离目标的编辑。同时,研究表明,使用质粒传递Cas9蛋白和sgRNA将导致长时间表达,这将大大增加脱靶的风险。为了解决该问题,用计算机来预测sgRNA的脱靶率,减少GC含量,并通过修改相关的Cas9蛋白缩短所用sgRNA的序列长度,这些都可以降低脱靶的风险。然而,这些工具受到PAM序列要求的限制,NHEJ修复大大增加了非目标编辑的可能性。因此,利用同源重组(homologous recombination, HR)的体内修复功能对DSB进行修复是提高基因编辑准确性的有效手段。
6.2 扩大编辑工具的可编辑范围由于Cas蛋白依赖PAM序列进行快速识别,因此PAM序列是CRISPR/Cas9裂解的必要条件。当研究人员不考虑PAM序列时,即使sgRNA与目标序列完全匹配,Cas蛋白也不会切割DNA。因此,PAM序列在CRISPR系统中具有重要意义。PAM序列越严格,编辑工具脱靶的风险越低,可设计的靶序列的数量将大大减少,因此,拓宽PAM序列、寻找新的Cas蛋白、设计新的Cas蛋白变体或制造新的工具尤为重要。
6.3 开放新编辑的酶工具版权目前,CRISPR基因编辑技术日趋成熟。然而,顶尖的研究团队拥有大量的技术开发版权。为了开发和应用相关技术,研究人员需要支付高昂的版权费,这可能给许多研究人员和研究机构带来不必要的经济负担,还会限制编辑工具的开发和应用。近年来,中国和美国已成为基因编辑技术研发领域最具竞争力的参与者。以美国Inscripta为首的新兴公司选择在某种程度上发布MAD7等关键工具的版权,因为这能使更多的研究人员参与到基因编辑的研究中来,加速编辑工具开发的进步,并扩大其技术的潜在应用。
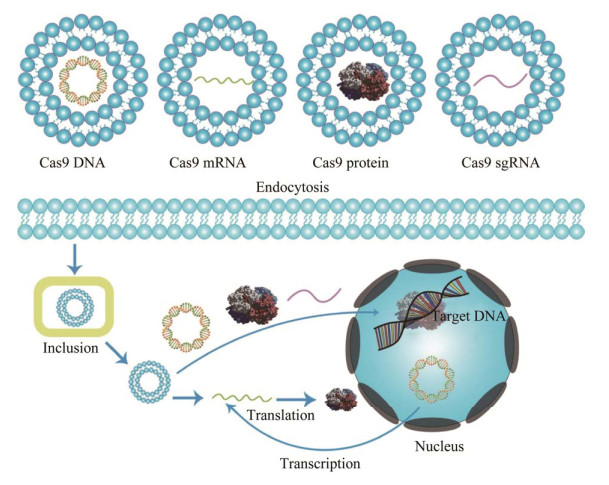
6.4 开发CRISPR/Cas9递送系统CRISPR/Cas9是目前应用最多的基因编辑工具之一,广泛应用于微生物、植物和动物中,但是目前限制该工具潜力发挥的最大障碍是如何高效将该系统递送到细胞中。目前,在靶细胞实现基因编辑的方式有3种(图 1):(1) 递送编码Cas9蛋白和sgRNA的质粒;(2) 递送Cas9 mRNA和sgRNA;(3) 递送Cas9蛋白和sgRNA[48]。CRISPR/Cas9的递送方式主要包括物理方法、病毒载体和非病毒载体。
其中物理方法使用最多的是粒子轰击[50],但该方法在体内较难使用[51]。病毒载体的转染效率较高,在体内外使用范围较广。病毒载体主要包括腺病毒(adenovirus, Adv or AV)、慢病毒(lentivirus, LV)和腺相关病毒(adeno-associated virus, AAV)。腺病毒载体系统一般以人类病毒作为载体,以人类细胞作为宿主,属于非整合型的病毒载体。腺病毒载体具有宿主范围广、对人致病性低、可以在增殖和非增殖细胞中感染和表达基因、无插入致突变性、能同时表达多个基因等优点。腺相关病毒载体具有安全性好,免疫原性低,能感染分裂细胞和非分裂细胞,能介导基因的长期稳定表达等优点。因此在神经系统的体内和体外研究中,腺相关病毒载体备受关注。目前病毒载体依旧常用于临床试验中,但是病毒载体在安全性上面有一定的潜在问题,如致癌风险大,且病毒载体的负载能力有限,例如AAV的负载能力只有4.5 kb,LV病毒负载能力约为8 kb,但是Cas9质粒一般大于或等于8 kb,所以很大程度上限制了病毒载体的应用范围,很难实现CRISPR/Cas9系统的高效递送[52]。
研究人员一直致力于研究新生物材料载体,这些新载体的研发基于纳米技术、生物材料、生物医学等多学科交叉,具有高基因编辑效率、高组织/细胞特异性、低免疫原性等优点。目前主要有脂质、聚合物、金纳米颗粒等非生物载体。这些非病毒载体包载能力较强、免疫原性低,在临床上发挥价值的潜能很大。
除了在医学临床上递送系统一直不断进步,在植物领域的递送系统也有较大发现,南方科技大学朱健康团队[53]开发了一种植物遗传修饰方法:CDB递送系统(cut-dip-budding, CDB),克服了传统技术由于植物组织培养过程带来的困难。CDB递送方法与现有的递送方法不同,没有基因型依赖性。同时,该技术能够将基因编辑工具递送到植物里并长出基因编辑修饰的植株。此外,随着对植物根蘖性机制研究的深入,未来有望通过CDB递送系统对大量的植物物种进行简便的遗传转化操作,实现遗传改良。
| [1] |
TOWNSEND JA, WRIGHT DA, WINFREY RJ, FU FL, MAEDER ML, JOUNG JK, VOYTAS DF. High-frequency modification of plant genes using engineered zinc-finger nucleases. Nature, 2009, 459(7245): 442-445. DOI:10.1038/nature07845
|
| [2] |
JOUNG JK, SANDER JD. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology, 2013, 14(1): 49-55. DOI:10.1038/nrm3486
|
| [3] |
JINEK M, CHYLINSKI K, FONFARA I, HAUER M, DOUDNA JA, CHARPENTIER E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829
|
| [4] |
ZHANG Y, MASSEL K, GODWIN ID, GAO CX. Applications and potential of genome editing in crop improvement. Genome Biology, 2018, 19(1): 1-11. DOI:10.1186/s13059-017-1381-1
|
| [5] |
ESVELT KM, MALI P, BRAFF JL, MOOSBURNER M, YAUNG SJ, CHURCH GM. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods, 2013, 10(11): 1116-1121. DOI:10.1038/nmeth.2681
|
| [6] |
DOETSCHMAN T, GEORGIEVA T. Gene editing with CRISPR/Cas9 RNA-directed nuclease. Circulation Research, 2017, 120(5): 876-894. DOI:10.1161/CIRCRESAHA.116.309727
|
| [7] |
YUMLU S, STUMM J, BASHIR S, DREYER AK, LISOWSKI P, DANNER E, KÜHN R. Gene editing and clonal isolation of human induced pluripotent stem cells using CRISPR/Cas9. Methods, 2017, 121/122: 29-44. DOI:10.1016/j.ymeth.2017.05.009
|
| [8] |
KOMOR AC, KIM YB, PACKER MS, ZURIS JA, LIU DR. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946
|
| [9] |
KOBLAN LW, DOMAN JL, WILSON C, LEVY JM, TAY T, NEWBY GA, MAIANTI JP, RAGURAM A, LIU DR. Improving cytidine and adenine base editors by expression optimization and ancestral reconstruction. Nature Biotechnology, 2018, 36(9): 843-846. DOI:10.1038/nbt.4172
|
| [10] |
ZHANG XH, CHEN L, ZHU BY, WANG LR, CHEN CY, HONG MJ, HUANG YF, LI HY, HAN HH, CAI BL, YU WS, YIN SM, YANG L, YANG ZZ, LIU MZ, ZHANG Y, MAO ZY, WU YX, LIU MY, LI DL. Increasing the efficiency and targeting range of cytidine base editors through fusion of a single-stranded DNA-binding protein domain. Nature Cell Biology, 2020, 22(6): 740-750. DOI:10.1038/s41556-020-0518-8
|
| [11] |
GAUDELLI NM, KOMOR AC, REES HA, PACKER MS, BADRAN AH, BRYSON DI, LIU DR. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644
|
| [12] |
JIANG TT, HENDERSON JM, COOTE K, CHENG Y, VALLEY HC, ZHANG XO, WANG Q, RHYM LH, CAO YY, NEWBY GA, BIHLER H, MENSE M, WENG ZP, ANDERSON DG, McCAFFREY AP, LIU DR, XUE W. Chemical modifications of adenine base editor mRNA and guide RNA expand its application scope. Nature Communications, 2020, 11: 1979. DOI:10.1038/s41467-020-15892-8
|
| [13] |
WALTON RT, CHRISTIE KA, WHITTAKER MN, KLEINSTIVER BP. Unconstrained genome targeting with near-PAMless engineered CRISPR-Cas9 variants. Science, 2020, 368(6488): 290-296. DOI:10.1126/science.aba8853
|
| [14] |
LIU Y, ZHOU JZ, LAN T, ZHOU XQ, YANG Y, LI C, ZHANG QJ, CHEN M, WEI S, ZHENG SW, CHENG LY, ZHENG YL, LAI LX, ZOU QJ. Elimination of Cas9-dependent off-targeting of adenine base editor by using TALE to separately guide deaminase to target sites. Cell Discovery, 2022, 8: 28.
|
| [15] |
ZHAO DD, LI J, LI SW, XIN XQ, HU MZ, PRICE MA, ROSSER SJ, BI CH, ZHANG XL. Glycosylase base editors enable C-to-A and C-to-G base changes. Nature Biotechnology, 2021, 39(1): 35-40. DOI:10.1038/s41587-020-0592-2
|
| [16] |
DONG XX, YANG C, MA ZZ, CHEN M, ZHANG XL, BI CH. Enhancing glycosylase base-editor activity by fusion to transactivation modules. Cell Reports, 2022, 40(3): 111090. DOI:10.1016/j.celrep.2022.111090
|
| [17] |
HUA K, JIANG YW, TAO XP, ZHU JK. Precision genome engineering in rice using prime editing system. Plant Biotechnology Journal, 2020, 18(11): 2167-2169. DOI:10.1111/pbi.13395
|
| [18] |
KIM HK, YU G, PARK J, MIN S, LEE S, YOON S, KIM HH. Predicting the efficiency of prime editing guide RNAs in human cells. Nature Biotechnology, 2021, 39(2): 198-206. DOI:10.1038/s41587-020-0677-y
|
| [19] |
ANZALONE AV, RANDOLPH PB, DAVIS JR, SOUSA AA, KOBLAN LW, LEVY JM, CHEN PJ, WILSON C, NEWBY GA, RAGURAM A, LIU DR. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature, 2019, 576(7785): 149-157. DOI:10.1038/s41586-019-1711-4
|
| [20] |
WEI Y, ZHANG XH, LI DL. The new favorite of gene editing technology-single base editors. Hereditas, 2017, 39(12): 1115-1121.
|
| [21] |
CHENG TL, LI S, YUAN B, WANG XL, ZHOU WH, QIU ZL. Expanding C-T base editing toolkit with diversified cytidine deaminases. Nature Communications, 2019, 10: 3612. DOI:10.1038/s41467-019-11562-6
|
| [22] |
TANG X, SRETENOVIC S, REN QR, JIA XY, LI MK, FAN TT, YIN DS, XIANG SY, GUO YC, LIU L, ZHENG XL, QI YP, ZHANG Y. Plant prime editors enable precise gene editing in rice cells. Molecular Plant, 2020, 13(5): 667-670. DOI:10.1016/j.molp.2020.03.010
|
| [23] |
JIN S, LIN QP, LUO YF, ZHU ZX, LIU GW, LI YJ, CHEN KL, QIU JL, GAO CX. Genome-wide specificity of prime editors in plants. Nature Biotechnology, 2021, 39(10): 1292-1299. DOI:10.1038/s41587-021-00891-x
|
| [24] |
JIANG TT, ZHANG XO, WENG ZP, XUE W. Deletion and replacement of long genomic sequences using prime editing. Nature Biotechnology, 2022, 40(2): 227-234. DOI:10.1038/s41587-021-01026-y
|
| [25] |
CHOI J, CHEN W, SUITER CC, LEE C, CHARDON FM, YANG W, LEITH A, DAZA RM, MARTIN B, SHENDURE J. Precise genomic deletions using paired prime editing. Nature Biotechnology, 2022, 40(2): 218-226. DOI:10.1038/s41587-021-01025-z
|
| [26] |
ZHANG XH, ZHU BY, CHEN L, XIE L, YU WS, WANG Y, LI LX, YIN SM, YANG L, HU HD, HAN HH, LI YM, WANG LR, CHEN G, MA XY, GENG HQ, HUANG WF, PANG XF, YANG ZZ, WU YX, et al. Dual base editor catalyzes both cytosine and adenine base conversions in human cells. Nature Biotechnology, 2020, 38(7): 856-860. DOI:10.1038/s41587-020-0527-y
|
| [27] |
KAPLANIS J, AKAWI N, GALLONE G, McRAE JF, PRIGMORE E, WRIGHT CF, FITZPATRICK DR, FIRTH HV, BARRETT JC, HURLES ME. Exome-wide assessment of the functional impact and pathogenicity of multinucleotide mutations. Genome Research, 2019, 29(7): 1047-1056. DOI:10.1101/gr.239756.118
|
| [28] |
SAKATA RC, ISHIGURO S, MORI H, TANAKA M, TATSUNO K, UEDA H, YAMAMOTO S, SEKI M, MASUYAMA N, NISHIDA K, NISHIMASU H, ARAKAWA K, KONDO A, NUREKI O, TOMITA M, ABURATANI H, YACHIE N. Publisher correction: base editors for simultaneous introduction of C-to-T and A-to-G mutations. Nature Biotechnology, 2020, 38(7): 901.
|
| [29] |
LI C, ZHANG R, MENG XB, CHEN S, ZONG Y, LU CJ, QIU JL, CHEN YH, LI JY, GAO CX. Targeted, random mutagenesis of plant genes with dual cytosine and adenine base editors. Nature Biotechnology, 2020, 38(7): 875-882. DOI:10.1038/s41587-019-0393-7
|
| [30] |
KWON JB, GERSBACH CA. Jumping at the chance for precise DNA integration. Nature Biotechnology, 2019, 37(9): 1004-1006. DOI:10.1038/s41587-019-0210-3
|
| [31] |
HALPIN-HEALY TS, KLOMPE SE, STERNBERG SH, FERNÁNDEZ IS. Structural basis of DNA targeting by a transposon-encoded CRISPR-Cas system. Nature, 2020, 577(7789): 271-274. DOI:10.1038/s41586-019-1849-0
|
| [32] |
STRECKER J, LADHA A, GARDNER Z, SCHMID-BURGK JL, MAKAROVA KS, KOONIN EV, ZHANG F. RNA-guided DNA insertion with CRISPR-associated transposases. Science, 2019, 365(6448): 48-53. DOI:10.1126/science.aax9181
|
| [33] |
STRECKER J, LADHA A, MAKAROVA KS, KOONIN EV, ZHANG F. Response to comment on "RNA-guided DNA insertion with CRISPR-associated transposases". Science, 2020, 368(6495): eabb2920. DOI:10.1126/science.abb2920
|
| [34] |
JARCZYNSKA ZD, RENDSVIG JKH, PAGELS N, VIANA VR, NØDVIG CS, KIRCHNER FH, STRUCKO T, NIELSEN ML, MORTENSEN UH. DIVERSIFY: a fungal multispecies gene expression platform. ACS Synthetic Biology, 2021, 10(3): 579-588. DOI:10.1021/acssynbio.0c00587
|
| [35] |
LIN QP, ZHU ZX, LIU GW, SUN C, LIN DX, XUE CX, LI SN, ZHANG DD, GAO CX, WANG YP, QIU JL. Genome editing in plants with MAD7 nuclease. Journal of Genetics and Genomics, 2021, 48(6): 444-451. DOI:10.1016/j.jgg.2021.04.003
|
| [36] |
PAUSCH P, AL-SHAYEB B, BISOM-RAPP E, TSUCHIDA CA, LI Z, CRESS BF, KNOTT GJ, JACOBSEN SE, BANFIELD JF, DOUDNA JA. CRISPR-CasΦ from huge phages is a hypercompact genome editor. Science, 2020, 369(6501): 333-337. DOI:10.1126/science.abb1400
|
| [37] |
ZHANG YX, REN QR, TANG X, LIU SS, MALZAHN AA, ZHOU JP, WANG JH, YIN DS, PAN CT, YUAN MZ, HUANG L, YANG H, ZHAO YX, FANG Q, ZHENG XL, TIAN L, CHENG YH, LE Y, McCOY B, FRANKLIN L, et al. Expanding the scope of plant genome engineering with Cas12a orthologs and highly multiplexable editing systems. Nature Communications, 2021, 12: 1944. DOI:10.1038/s41467-021-22330-w
|
| [38] |
AL-SHAYEB B, SACHDEVA R, CHEN LX, WARD F, MUNK P, DEVOTO A, CASTELLE CJ, OLM MR, BOUMA-GREGSON K, AMANO Y, HE C, MÉHEUST R, BROOKS B, THOMAS A, LAVY A, MATHEUS-CARNEVALI P, SUN C, GOLTSMAN DSA, BORTON MA, SHARRAR A, et al. Clades of huge phages from across earth's ecosystems. Nature, 2020, 578(7795): 425-431. DOI:10.1038/s41586-020-2007-4
|
| [39] |
REN J, MENG XB, HU FY, LIU Q, CAO YX, LI HY, YAN CJ, LI JY, WANG KJ, YU H, WANG C. Expanding the scope of genome editing with SpG and SpRY variants in rice. Science China Life Sciences, 2021, 64(10): 1784-1787. DOI:10.1007/s11427-020-1883-5
|
| [40] |
XU ZY, KUANG YJ, REN B, YAN DQ, YAN F, SPETZ C, SUN WX, WANG GR, ZHOU XP, ZHOU HB. SpRY greatly expands the genome editing scope in rice with highly flexible PAM recognition. Genome Biology, 2021, 22(1): 1-15. DOI:10.1186/s13059-020-02207-9
|
| [41] |
CHO SI, LEE S, MOK YG, LIM K, LEE J, LEE JM, CHUNG E, KIM JS. Targeted A-to-G base editing in human mitochondrial DNA with programmable deaminases. Cell, 2022, 185(10): 1764-1776.e12. DOI:10.1016/j.cell.2022.03.039
|
| [42] |
QI LS, LARSON MH, GILBERT LA, DOUDNA JA, WEISSMAN JS, ARKIN AP, LIM WA. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022
|
| [43] |
MANDEGAR MA, HUEBSCH N, FROLOV EB, SHIN E, TRUONG A, OLVERA MP, CHAN AH, MIYAOKA Y, HOLMES K, SPENCER CI, JUDGE LM, GORDON DE, ESKILDSEN TV, VILLALTA JE, HORLBECK MA, GILBERT LA, KROGAN NJ, SHEIKH SP, WEISSMAN JS, QI LS, et al. CRISPR interference efficiently induces specific and reversible gene silencing in human iPSCs. Cell Stem Cell, 2016, 18(4): 541-553. DOI:10.1016/j.stem.2016.01.022
|
| [44] |
TANENBAUM ME, GILBERT LA, QI LS, WEISSMAN JS, VALE RD. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell, 2014, 159(3): 635-646. DOI:10.1016/j.cell.2014.09.039
|
| [45] |
CHAVEZ A, SCHEIMAN J, VORA S, PRUITT BW, TUTTLE M, P R IYER E, LIN SL, KIANI S, GUZMAN CD, WIEGAND DJ, TER-OVANESYAN D, BRAFF JL, DAVIDSOHN N, HOUSDEN BE, PERRIMON N, WEISS R, AACH J, COLLINS JJ, CHURCH GM. Highly efficient Cas9-mediated transcriptional programming. Nature Methods, 2015, 12(4): 326-328. DOI:10.1038/nmeth.3312
|
| [46] |
KONERMANN S, BRIGHAM MD, TREVINO AE, JOUNG J, ABUDAYYEH OO, BARCENA C, HSU PD, HABIB N, GOOTENBERG JS, NISHIMASU H, NUREKI O, ZHANG F. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature, 2015, 517(7536): 583-588. DOI:10.1038/nature14136
|
| [47] |
KAMPMANN M. CRISPRi and CRISPRa screens in mammalian cells for precision biology and medicine. ACS Chemical Biology, 2018, 13(2): 406-416. DOI:10.1021/acschembio.7b00657
|
| [48] |
EOH J, GU L. Biomaterials as vectors for the delivery of CRISPR-Cas9. Biomaterials Science, 2019, 7(4): 1240-1261. DOI:10.1039/C8BM01310A
|
| [49] |
赵子璇, 李春辉, 周莉莉, 赵德尧, 翁郁华, 夏新华, 黄渊余. CRISPR/Cas系统递送技术及其应用研究进展. 生物化学与生物物理进展, 2020, 47(4): 286-299. ZHAO ZX, LI CH, ZHOU LL, ZHAO DY, WENG YH, XIA XH, HUANG YY. Delivery and application progresses of CRISPR/Cas gene editing system. Progress in Biochemistry and Biophysics, 2020, 47(4): 286-299 (in Chinese). DOI:10.16476/j.pibb.2019.0281 |
| [50] |
LAUSTSEN A, BAK RO. Electroporation-based CRISPR/Cas9 gene editing using Cas9 protein and chemically modified sgRNAs. Methods in Molecular Biology (Clifton, N J), 2019, 1961, 127-134.
|
| [51] |
WELLS DJ. Gene therapy progress and prospects: electroporation and other physical methods. Gene Therapy, 2004, 11(18): 1363-1369. DOI:10.1038/sj.gt.3302337
|
| [52] |
YIN H, KANASTY RL, ELTOUKHY AA, VEGAS AJ, DORKIN JR, ANDERSON DG. Non-viral vectors for gene-based therapy. Nature Reviews Genetics, 2014, 15(8): 541-555. DOI:10.1038/nrg3763
|
| [53] |
CAO XS, XIE HT, SONG ML, LU JH, MA P, HUANG BY, WANG MG, TIAN YF, CHEN F, PENG J, LANG ZB, LI GF, ZHU JK. Cut-dip-budding delivery system enables genetic modifications in plants without tissue culture. The Innovation, 2023, 4(1): 100345. DOI:10.1016/j.xinn.2022.100345
|
 2023, Vol. 39
2023, Vol. 39