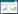中国科学院微生物研究所、中国微生物学会主办
文章信息
- 贺志敏, 马文瑞, 于丽平, 吕鹤书, 杨明峰
- HE Zhimin, MA Wenrui, YU Liping, LÜ Heshu, YANG Mingfeng
- 定点突变提高虎杖聚酮合酶的苯亚甲基丙酮合酶活性
- Site-directed mutagenesis enhances the activity of benzylidene acetone synthase of polyketide synthase from Polygonum cuspidatum
- 生物工程学报, 2023, 39(7): 2806-2817
- Chinese Journal of Biotechnology, 2023, 39(7): 2806-2817
- 10.13345/j.cjb.220815
-
文章历史
- Received: October 10, 2022
- Accepted: December 6, 2022
- Published: December 9, 2022
聚酮合酶(polyketide synthase, PKS)广泛存在于细菌、真菌及植物体内[1-2],参与催化合成聚酮类化合物(polyketide, PK)[3],这些化合物具有抗肿瘤、抗炎、抗氧化、心血管保护等多种功能[4-6]。PKS大体上可分为3类,其中I型PKS (type I polyketide synthase)和Ⅱ型PKS (type Ⅱ polyketide synthase)主要存在于微生物中;Ⅲ型PKS (type Ⅲ polyketide synthase)即查尔酮合成酶(chalcone synthase, CHS)家族,主要存在于植物中。CHS催化丙二酰辅酶A和4-香豆酰辅酶A通过Claisen缩合反应生成柚皮素查尔酮(naringenin chalcone, Nar),进而生成类黄酮化合物[7],而苯亚甲基丙酮合酶(benzylidene acetone synthase, BAS)使用和CHS相同的底物,催化合成苯亚甲基丙酮(benzalacetone, BA),进而生成一系列苯丁烷类化合物(图 1)。
|
| 图 1 虎杖黄酮类化合物合成途径[8] Fig. 1 Reactions underlying the conversion of 4-coumaroyl-CoA and malonyl-CoA to p-hydroxy benzalacetone by benzylidene acetone synthase (BAS), to naringenin chalcone by chalcone synthase (CHS), and to 4-CTAL by p-coumaroyl triacetic acid synthase (CTAS). Polyketide pyrones, BNY and CTAL, are derailment products of the CHS reactions in vitro when the reaction mixtures are acidified before extraction[8]. |
| |
自1999年Ferrer等解析了紫花苜蓿(Medicago sativa)的CHS晶体结构后,开始了对植物Ⅲ型聚酮合酶原子水平的研究[9]。研究结果表明,植物Ⅲ型PKS超家族成员的三维结构和催化机制极其相似,催化活性中心的Cys164、His303和Asn336 (紫花苜蓿CHS氨基酸序列顺序,其他下同)在所有的Ⅲ型PKS中都高度保守,构成了酶活性中心催化三联体;Phe215和Phe265起到“gatekeeper”的作用[10],与该酶活性有关的其他活性位点残基还有Thr132、Ser133、Glu192、Thr194、Thr197、Phe215、Phe265、Ser338和Pro375。这些氨基酸在CHS中高度保守,但在其他Ⅲ型PKS中被其他氨基酸所取代,正是这些特定位置的氨基酸决定了聚酮合酶的不同催化活性。定点突变技术是研究蛋白结构与功能的常用方法,Jez等将紫花苜蓿(M. sativa) MsCHS的197位、256位和338位突变成非洲菊(Gerbera hybrid) 2-吡喃酮合酶(2-pyrazone synthase, 2-PS)相对应的氨基酸(T197L、G256L、S338I)之后,突变体能利用小分子乙酰辅酶A为底物生成较查尔酮更小的吡喃酮[11]。这是对植物Ⅲ型PKS的首次定向突变[11]。
虎杖(Polygonum cuspidatum Sieb.et Zucc.)属于廖科(Polygonaceae)多年生草本植物,其虎杖苷、白藜芦醇、黄酮类等化合物含量丰富,具有多种药理活性,其根和茎可广泛入药[12]。已有研究表明,虎杖的药用价值与虎杖中所含有的黄酮类化合物相关[13]。PcPKS1是虎杖合成黄酮类化合物的关键限速酶,具有CHS和BAS双功能活性。目前已经从苔藓、蕨类、裸子植物和被子植物等多种植物中克隆了CHS基因序列[14]。由于聚酮合酶结构的复杂性,通过晶体结构和点突变等手段寻找关键活性位点,从而合理改造PKS,使之产生更有效的药用化合物成为一大研究方向。PKS基因结构多样,但对于虎杖PcPKS1的关键活性位点的研究相对较少。
本实验室在前期研究工作中,从虎杖中克隆到PcPKS1基因并表明PcPKS1具有CHS和BAS双功能酶特性[15]。经系统发育分析表明,PcPKS1是一个典型的CHS,而功能和酶学分析表明PcPKS1同时具有CHS和BAS催化活性[15]。本研究采用定点突变对PcPKS1进行分子修饰并测定修饰后功能变化,以深入了解关键氨基酸位点的改变对PcPKS1酶活性的变化,为进一步利用虎杖PcPKS1进行基因工程操作奠定基础。
1 材料与方法 1.1 材料原核表达载体pET30a及大肠杆菌(Escherichia coli) DH5α购自北京博迈德基因技术有限公司;大肠杆菌表达菌株BL21(DE3) pLysS购于北京全式金生物技术有限公司;重组质粒pET30a−PcPKS1由Guo等于2017年构建[16];Muta−directTM定点突变试剂盒购于北京赛百盛基因技术有限公司;引物合成及序列测序由生工生物工程(上海)股份有限公司完成。本研究所用引物如表 1所示。
| Mutation site | Primer name | Primer sequence (5ʹ→3ʹ) |
| T133LS134A | M13301 | CCATGTCATCATGTGCACACTCGCTGGTGTTGACATGC |
| M13302 | GCATGTCAACACCAGCGAGTGTGCACATGATGACATGG | |
| S339V | M33901 | CGACTACGGAAACATGGTGAGTGCGTGTGTGTTGTTC |
| M33902 | GAACAACACACACGCACTCACCATGTTTCCGTAGTCG | |
| The shaded part represents the codon corresponding to the mutated amino acid; The underline represents the mutated base. | ||
以pET30a−PcPKS1质粒为模板,50 μL PCR反应体系进行PCR扩增,PCR扩增程序为:95 ℃ 5 min;95 ℃ 1 min,56 ℃ 30 s,72 ℃ 30 s,共30个循环;72 ℃ 10 min。PCR反应结束后加入1 μL (10 U/μL) MutazymeTM酶消化甲基化质粒,热激法转入大肠杆菌DH5α感受态细胞,待平板长出单克隆之后,进行测序鉴定。将测序正确的质粒记为pET30a-T133LS134A、pET30a-F216V、pET30a-F266W、pET30a-S339V、pET30a-S339A、pET30a-S339F。
1.2.2 PcPKS1和突变体的原核表达与纯化将测序正确的2个重组质粒通过热激法转入大肠杆菌E. coli BL21(DE3) pLysS感受态细胞,于37 ℃倒置培养12−16 h,构成突变表达菌株。在重组表达菌株和突变表达菌株中加入0.5 mmol/L IPTG,25 ℃、180 r/min,大量诱导表达6 h,经超声破碎细胞后,采用镍螯合亲和层析纯化,使用批量法使重组蛋白与树脂在容器中通过摇动而充分结合,通过离心分离树脂,再装柱进行洗脱。经镍柱纯化后的样品含有较高的盐和咪唑,采用脱盐柱PD−10 (填料葡聚糖凝胶G−25)进行脱盐,十二烷基磺酸钠−聚丙烯酰胺凝胶电泳(SDS−PAGE)检测表达情况。
1.2.3 PcPKS1和突变体体外酶促反应与产物检测PcPKS1和突变体体外酶促反应体系(250 μL),以终浓度为25 μmmol/L 4−香豆酰辅酶A、65 μmol/L丙二酰辅酶A为底物,在不同pH的0.1 mol/L磷酸钾缓冲溶液(pH 6.0、6.5、7.0、7.5、8.0、8.5、9.0)中,加入2.0 μg纯化好的PcPKS1蛋白和突变体蛋白。整个反应体系于30 ℃恒温水浴锅中反应30 min后取出,加入终浓度5%的冰醋酸,然后用等体积250 μL的乙酸乙酯振荡萃取2次,10 000 r/min离心10 min,吸取上层有机相至新的离心管,利用氮吹仪吹干后,用50 μL 50% (体积分数)色谱甲醇水溶解。
酶促反应产物经SunfireTM C18反相柱(5.0 μm, 4.6 mm×150 mm)的Waters HPLC检测。流动相为甲醇(A)和水(B),流速为0.8 mL/min,梯度洗脱条件:10%–70% A 30 min、70% A 10 min。苯亚甲基丙酮的最大吸收波长为323 nm,柚皮素查尔酮的最大吸收波长为289 nm。
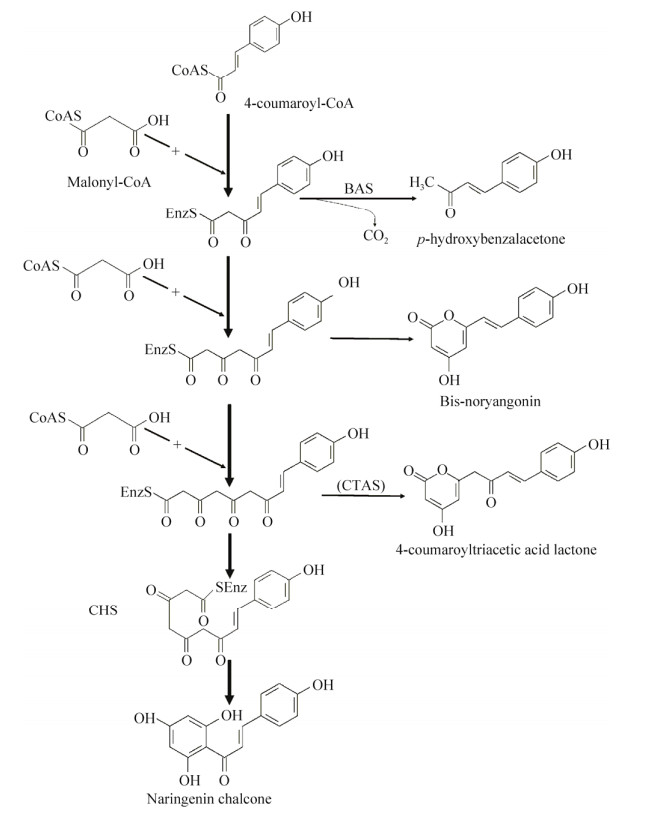
2 结果与分析 2.1 PcPKS1突变位点的选择PcPKS1是一个由1 182个碱基编码393个氨基酸的蛋白质酶,将PcPKS1与植物类型Ⅲ PKS超家族中不同种属的CHS、BAS重要氨基酸序列进行比较。发现存在多个氨基酸残基可能影响到其双功能酶特性,包括苏氨酸(Thr133)、丝氨酸(Ser134)、丝氨酸(Ser339) (图 2)。
|
| 图 2 六种植物类型Ⅲ PKS家族氨基酸序列比对 Fig. 2 Comparison of amino acid sequences of Ⅲ PKS family of six plant types. Ms_CHS2: Medicago sativa CHS2; Cs_CHS: Cucumis sativus CHS. PcPKS1, PcPKS2: Polygonum cuspidatum PcPKS1, PcPKS2; RiPKS4: Rubus idaeus RiPKS4; Rp_BAS: Rheum Palmatum BAS; Pc_STS: Polygonum cuspidatum STS. Black boxes mark the amino acid sites for mutation. |
| |
将测序结果与野生型PcPKS1基因比对,成功将PcPKS1的第133位苏氨酸突变成亮氨酸,第134位丝氨酸突变成丙氨酸,第339位丝氨酸突变成缬氨酸,构建了2个突变体T133LS134A和S339V。2个突变基因有关突变位置基因序列比对结果如图 3所示。

|
| 图 3 PcPKS1与突变体突变位置基因序列比对 Fig. 3 Nucleotide sequence alignment of PcPKS1 and mutant. |
| |
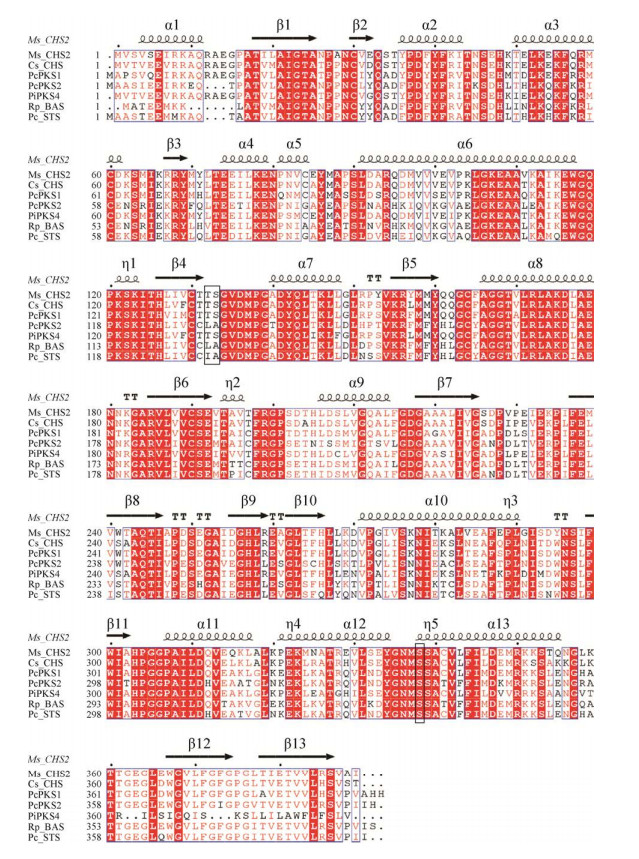
重组菌经0.5 mmol/L IPTG诱导大量表达后,成功表达出目的蛋白。收集破碎细胞上清液和沉淀,上清液中的蛋白含量明显多于沉淀中的。取上清液经Ni柱亲和层析纯化2次及PD-10柱脱盐,其中,PcPKS1得到43 kDa的单一目的条带(图 4)。突变体T133LS134A及S339V蛋白同样分离到43 kDa的可溶性蛋白,且表达量比野生型PcPKS1蛋白表达量高(图 5)。

|
| 图 4 虎杖PcPKS1蛋白表达与纯化 Fig. 4 Expression and purification of PcPKS1 from Polygonum cuspidatum. 1: Total protein; 2: Supernatant protein after ultrasonic centrifugation; 3: Precipitated protein after ultrasonic centrifugation; 4: Supernatant after incubation with Ni2+ resin once; 5: Supernatant after 2 incubations with Ni2+ resin; 6: Ni2+ resin elution collection solution; 7: PD-10 column protein elution collection solution. |
| |
|
| 图 5 突变体T133LS134A (A)及S339V (B)蛋白表达与纯化 Fig. 5 Expression and purification of T133LS134A (A) and S339V (B). 1: Total protein; 2: Supernatant protein after ultrasonic centrifugation; 3: Precipitated protein after ultrasonic centrifugation; 4: Supernatant after incubation with Ni2+ resin once; 5: Supernatant after incubation with Ni2+ resin twice; 6: Supernatant after incubation with Ni2+ resin for three times; 7: Ni2+ resin elution collection solution; 8: PD-10 column protein elution collection solution. |
| |
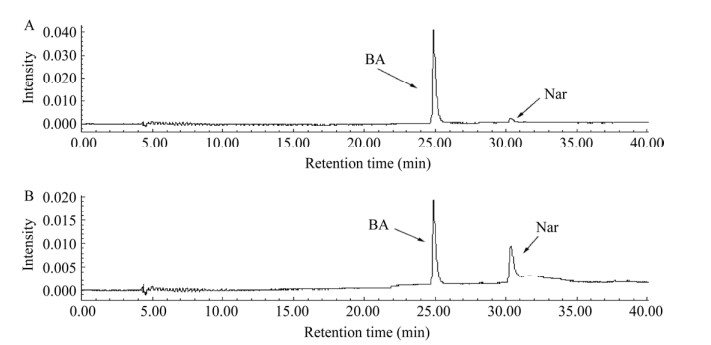
取苯亚甲基丙酮(benzalacetone, BA)及柚皮素查尔酮(naringenin chalcone, Nar)标准品混合液10 μL进行HPLC检测。运行结束后,HPLC图谱显示在25 min左右出现苯亚甲基丙酮吸收峰,且323 nm处为其最大吸收波长,在30 min左右出现柚皮素查尔酮吸收峰且289 nm为其最大吸收波长(图 6)。
|
| 图 6 苯亚甲基丙酮和柚皮素查尔酮标品HPLC色谱图 Fig. 6 HPLC elution profiles of benzylidene acetone and naringenin chalcone. A: 323 nm HPLC chromatogram. B: 289 nm HPLC chromatogram. |
| |
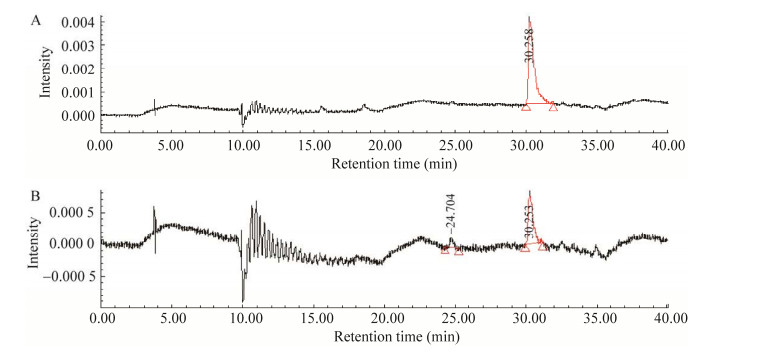
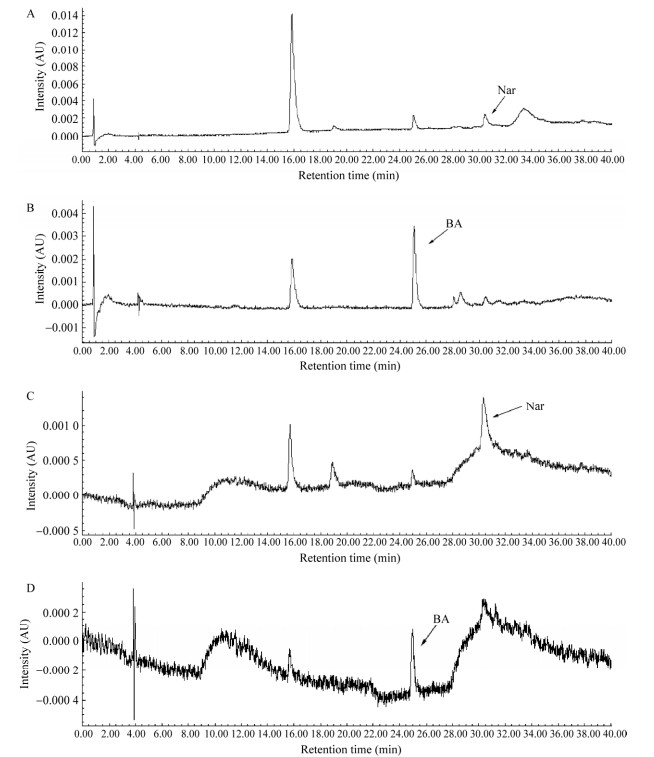
PcPKS1突变体体外酶促反应采用与野生型PcPKS1体外酶促反应相同配比的250 μL体系(表 2和图 7)。突变体T133LS134A在pH 7.0时柚皮素查尔酮合成活性显著低于野生型PcPKS1,在pH 9.0时,突变体T133LS134A以苯亚甲基丙酮为主产物,其活性显著高于野生型PcPKS1。突变体S339V在pH 7.0时出现柚皮素查尔酮吸收峰,活性高于突变体T133LS134A柚皮素查尔酮活性。在pH 9.0时能产生一定量的苯亚甲基丙酮,其活性介于野生型和突变体T133LS134A之间。值得注意的是,两个突变体的BAS活性均显著高于野生型(表 2和图 8)。
| Sample No. | pH 9.0, 323 nm, active relative area of BAS | pH 7.0, 289 nm, active relative area of CHS |
| Standard sample | 807±0.402 | 389±0.433 |
| PcPKS1 | 80±0.258 | 1 089±0.283 |
| T133LS134A | 498±0.374 | 99±0.345 |
| S339V | 453±0.451 | 521±0.412 |
|
| 图 7 PcPKS1体外酶促反应产物HPLC色谱图 Fig. 7 HPLC elution profiles of PcPKS1 fusion enzymes in vitro reaction product. A: HPLC result of enzymatic product at pH 7.0 and 289 nm. B: HPLC result of enzymatic product at pH 9.0 and 323 nm. |
| |
|
| 图 8 突变体T133LS134A和S339V体外酶促反应产物HPLC色谱图 Fig. 8 HPLC elution profiles of T133LS134A and S339V enzymes in vitro reaction product. A: HPLC result of T133LS134A enzymatic product at pH 7.0 and 289 nm. B: HPLC result of T133LS134A enzymatic product at pH 9.0 and 323 nm. C: HPLC results of S339V enzymatic products at pH 7.0 and 289 nm. D: HPLC results of S339V enzymatic products at pH 9.0 and 323 nm. |
| |
查尔酮合酶(chalcone synthase, CHS) (EC 2.3.1.74)超家族是聚酮合酶超家族中研究最早、最透彻的家族成员,1972年,Kreuzaler等首次在欧芹悬浮细胞培养物中检测到CHS的体外酶促活性[17]。研究显示几乎所有的CHS在标准体外试验中仅有CHS不具有BAS的活性[18]。在本实验室前期研究工作中,从蓼科植物虎杖中提取克隆出的PcPKS1具有CHS和BAS双功能酶特性[9]。PcPKS1经序列及系统发育分析表明是一个典型的CHS,而功能和酶学分析中PcPKS1呈现出同时具有CHS和BAS双功能催化活性。
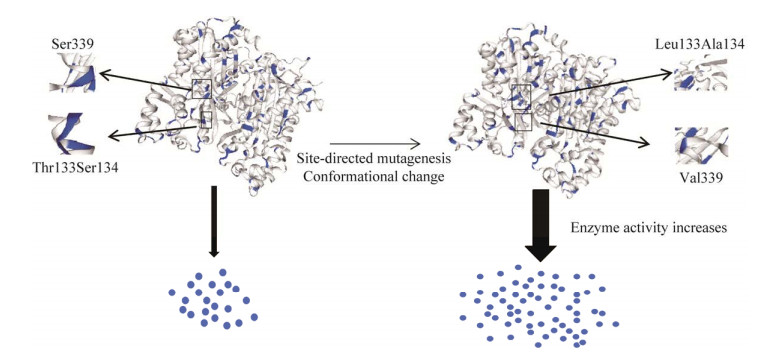
突变体T133LS134A和S339V与野生型PcPKS1相比,其活性位点的改变导致三维结构的改变,BAS酶活得到了显著提升(图 9)。其中,Ser134和Ser338是与CHS产物柚皮素查尔酮的香豆酰衍生部分接触的残基,Ter133是丙二酰衍生部分与活性腔接触的残基之一。这些位于活性腔周围的多个残基被认为对酶的底物和产物专一性具有重要作用。
|
| 图 9 PcPKS1与突变体三维结构模式图 Fig. 9 3D structure pattern diagram of PcPKS1 and mutant. |
| |
本研究定点突变虎杖PcPKS1基因的Thr133、Ser134、Ser339位点(图 10)。Thr132在植物Ⅲ型PKS中高度保守,2010年,Shimokawa等对掌叶大黄(Rheum palmatum) BAS的Leu132进行了一系列的定点突变(L132G、L132A、L132S、L132C、L132T、L132F、L132Y、L132W和L132P),结果显示,L132T突变恢复了RpBAS的查尔酮合酶活性[19]。本研究突变体T133LS134A仍具有双功能酶活性,在pH 7.0的条件下,其CHS活性显著低于野生型PcPKS1,这与掌叶大黄的研究结果一致,再次确认了该位点对于聚酮合酶的活性具有较大影响。2009年,Klundt等将金丝桃(Hypericum androsaemum)的二苯甲酮合酶(benzophenone synthase,BPS)活性口袋内的T135 (对应MsCHS中T132)突变为Leu,与本研究突变位点及突变氨基酸均一致,突变结果表明原本催化苯甲酰CoA与丙二酰CoA聚合形成二苯甲酮的BPS在功能上转变为苯基吡喃酮合酶活性,该位点的突变改变了野生型BPS的底物和产物特异性[20]。该研究也充分证明,单一活性位点氨基酸残基的改变完全可以彻底改变一种Ⅲ型PKS原有的活性而产生另一种全新的酶。
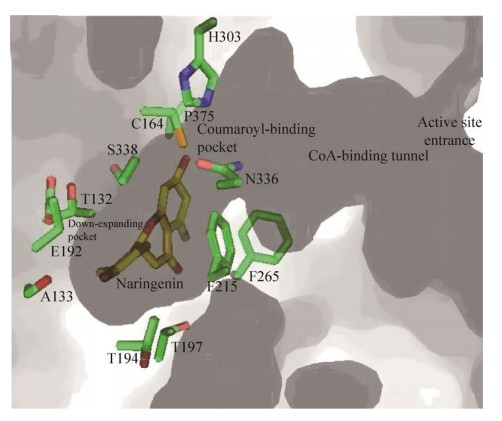
|
| 图 10 紫花苜蓿CHS2活性位点腔结构[22] Fig. 10 Enzyme reaction, overall structure and detailed active site cavity structure of Medicago sativa CHS2[22]. The side chains of Thr132, Ser133, Thr194, Thr197, Phe215, Gly256, Phe265, Ser338和Pro375 are shown as stick models with carbons in green; among them, T132 is Polygonum cuspidatum Thr133, A133 is P. cuspidatum Ser134, S338 means P. cuspidatum Ser339. 关键活性位点残基(Thr132、Ser133、Glu192、Thr194、Thr197、Phe215、Phe265、Ser338和Pro375),其中T132为虎杖Thr133、A133为虎杖Ser134、S338表示虎杖Ser339 |
| |
2007年,Abe等将掌叶大黄(R. palmatum) BAS的338位丝氨酸突变为缬氨酸(S338V)后,其BAS活性增加了2倍[21],与本研究中338位点的突变效果相同,表明338位点对于PcPKS1发挥BAS酶活性具有重要作用。推测S338位点更有利于底物与酶的结合,从而提高酶的催化效率。
PcPKS1在pH 7.0时仅具有CHS活性,在pH 9.0时同时具有BAS和CHS活性,表明PcPKS1相对于其他单一CHS而言具有更加复杂的空间结构来使其在不同生理状态下表现出不同的催化活性。突变体T133LS134A和S339V均具有双功能酶活性,但在pH 9.0时只体现苯亚甲基丙酮合酶活性,表明活性位点周围的单个或多个氨基酸均能对酶活性产生直接影响。这也反映了植物Ⅲ型聚酮合酶结构与功能之间的复杂性。
4 结论本研究以虎杖PcPKS1基因为入手点,对PcPKS1进行定点突变,最终成功构建了2个突变体。研究发现突变体T133LS134A和S339V在pH 7.0和pH 9.0的体外酶促条件下,具有一定的BAS和CHS双功能活性,同时两个突变体BAS活性均显著高于野生型PcPKS1酶活性。
| [1] |
BARTON SD, NAKANISHI K, METH-COHN O. Editors-in-chief[M]//Comprehensive Natural Products Chemistry. Amsterdam: Elsevier, 1999.
|
| [2] |
LIM Y, GO M, YEW W. Exploiting the biosynthetic potential of type Ⅲ polyketide synthases. Molecules, 2016, 21(6): 806. DOI:10.3390/molecules21060806
|
| [3] |
YI T, ZHANG H, CAI ZW. Analysis of Rhizoma Polygoni Cuspidati by HPLC and HPLC-ESI/MS. Phytochemical Analysis, 2007, 18(5): 387-392. DOI:10.1002/pca.993
|
| [4] |
马兰青, 师光禄, 叶和春, 刘本叶, 王有年. 植物类型Ⅲ聚酮合酶超家族基因结构、功能及代谢产物. 生物工程学报, 2010, 26(11): 1482-1492. MA LQ, SHI GL, YE HC, LIU BY, WANG YN. Plant-specific type Ⅲ polyketide synthase superfamily: gene structure, function and metabolistes. Chinese Journal of Biotechnology, 2010, 26(11): 1482-1492 (in Chinese). DOI:10.13345/j.cjb.2010.11.001 |
| [5] |
SHEN B. Polyketide biosynthesis beyond the type Ⅰ, Ⅱ and Ⅲ polyketide synthase paradigms. Current Opinion in Chemical Biology, 2003, 7(2): 285-295. DOI:10.1016/S1367-5931(03)00020-6
|
| [6] |
SCHRODER J. A family of plant-specific polyketide synthases: facts and predictions. Trends in Plant Science, 1997, 2(10): 373-378. DOI:10.1016/S1360-1385(97)87121-X
|
| [7] |
MOORE BS, HOPKE JN. Discovery of a new bacterial polyketide biosynthetic pathway. ChemBiochem a European Journal of Chemical Biology, 2001, 2(1): 35-38. DOI:10.1002/1439-7633(20010105)2:1<35::AID-CBIC35>3.0.CO;2-1
|
| [8] |
MA LQ, GUO YW, GAO DY, MA DM, WANG YN, LI GF, LIU BY, WANG H, YE HC. Identification of a Polygonum cuspidatum three-intron gene encoding a type Ⅲ polyketide synthase producing both naringenin and p-hydroxybenzalacetone. Planta, 2009, 229(5): 1077-1086. DOI:10.1007/s00425-009-0899-1
|
| [9] |
FERRER JL, JEZ JM, BOWMAN ME, DIXON RA, NOEL JP. Structure of chalcone synthase and the molecular basis of plant polyketide biosynthesis. Nature Structural Biology, 1999, 6(8): 775-784. DOI:10.1038/11553
|
| [10] |
AUSTIN MB, NOELL JP. The chalcone synthase superfamily of type Ⅲ polyketide synthases. Natural Product Reports, 2003, 20(1): 79-110. DOI:10.1039/b100917f
|
| [11] |
JEZ JM, AUSTIN MB, FERRER JL, BOWMAN ME, SCHRÖDER J, NOEL JP. Structural control of polyketide formation in plant-specific polyketide synthases. Chemistry & Biology, 2000, 7(12): 919-930.
|
| [12] |
胡春生, 吴祖泽, 张庆林. 聚酮类化合物异源表达研究进展. 生物技术通讯, 2013, 24(4): 573-578. HU CS, WU ZZ, ZHANG QL. Research progress in heterologous express of polyketides. Letters in Biotechnology, 2013, 24(4): 573-578 (in Chinese). |
| [13] |
MAIOLO SA, FAN PH, PBOBROVSKAYA L. Bioactive constituents from cinnamon, hemp seed and Polygonum cuspidatum protect against H2O2 but not rotenone toxicity in a cellular model of Parkinson's disease. Journal of Traditional Complementary Medicine, 2018, 8(3): 420-427. DOI:10.1016/j.jtcme.2017.11.001
|
| [14] |
REIMOLD U, KRÖGER M, KREUZALER F, HAHLBROCK K. Coding and 3′ non-coding nucleotide sequence of chalcone synthase mRNA and assignment of amino acid sequence of the enzyme. The EMBO Journal, 1983, 2(10): 1801-1805. DOI:10.1002/j.1460-2075.1983.tb01661.x
|
| [15] |
MA LQ, PANG XB, SHEN HY, PU GB, WANG HH, LEI CY, WANG H, LI GF, LIU BY, YE HC. A novel type Ⅲ polyketide synthase encoded by a three-intron gene from Polygonum cuspidatum. Planta, 2009, 229(3): 457-469. DOI:10.1007/s00425-008-0845-7
|
| [16] |
GUO HL, YANG YD, MA YD, LIU WB, FENG J, LUO ZQ, LU HS, LIU CM, YANG MF, WANG YN, MA LQ. A bifunctional type Ⅲ polyketide synthase from raspberry (Rubus idaeus L.) with both chalcone synthase and benzalacetone synthase activity. Journal of Plant Biochemistry and Biotechnology, 2017, 26(1): 80-90. DOI:10.1007/s13562-016-0365-7
|
| [17] |
KREUZALER F, HAHLBROCK K. Enzymatic synthesis of aromatic compounds in higher plants: formation of naringenin (5, 7, 4′-trihydroxyflavanone) from p-coumaroyl coenzyme A and malonyl coenzyme A. FEBS Letters, 1972, 28(1): 69-72. DOI:10.1016/0014-5793(72)80679-3
|
| [18] |
杨继, 顾红雅. 查尔酮合酶超家族(chalcone synthase superfamily)基因重复和分化的式样. 科学通报, 2006, 51(7): 745-749. YANG J, GU HY. Patterns of gene repetition and differentiation in chalcone synthase superfamily. Chinese Science Bulletin, 2006, 51(7): 745-749 (in Chinese). |
| [19] |
SHIMOKAWA Y, MORITA H, ABE I. Structure- based engineering of benzalacetone synthase. Bioorganic & Medicinal Chemistry Letters, 2010, 20(17): 5099-5103.
|
| [20] |
KLUNDT T, BOCOLA M, LUTGE M, BEUERLE T, LIU BY, BEERHUES L. A single amino acid substitution converts benzophenone synthase into phenylpyrone synthase. Journal of Biological Chemistry, 2009, 284(45): 30957-30964. DOI:10.1074/jbc.M109.038927
|
| [21] |
ABE T, MORITA H, NOMA H, KOHON T, NOGUCHI H, ABE I. Structure function analysis of benzalacetone synthase from Rheum palmatum. Bioorganic & Medicinal Chemistry Letters, 2007, 17(11): 3161-3166.
|
| [22] |
吕鹤书, 柳春梅, 路平, 师光禄, 马兰青, 王有年. 植物类型Ⅲ聚酮合酶超家族晶体结构与功能. 生物工程学报, 2012, 28(1): 1-14. LU HS, LIU CM, LU P, SHI GL, MA LQ, WANG YN. Plant-specific type Ⅲ polyketide synthases superfamily: crystal structures and functions. Chinese Journal of Biotechnology, 2012, 28(1): 1-14 (in Chinese). |
 2023, Vol. 39
2023, Vol. 39