广西大学生命科学与技术学院, 广西 南宁 530004
收稿日期:2022-11-09;接收日期:2023-01-09;网络出版时间:2023-05-25
基金项目:巴马县人才科技计划项目(202100180);广西研究生教育创新计划项目(YCSW2022028)
College of Life Science and Technology, Guangxi University, Nanning 530004, Guangxi, China
Received: November 9, 2022; Accepted: January 9, 2023; Published: May 25, 2023
Supported by: This work was supported by the Bama County Program for Talents in Science and Technology, Guangxi, China (202100180) and by the Innovation Projects of Guangxi Graduate Research (YCSW2022028)
红边龙血树(Dracaena marginata)隶属于百合科(Liliaceae)龙血树属(Dracaena),也称为千年木,原产于马达加斯加和毛里求斯,包含三色千年木、五彩千年木、七彩千年木等品种[1]。红边龙血树茎杆细长,一般在成熟后开始分枝,其顶部为一簇边缘呈紫红色的细狭叶片,整体姿态优雅美观,被广泛应用于园林园艺、城市景观美化与室内陈设栽培。红边龙血树具有较强适应性,特别是在季节性干旱及不规律降水环境下可长期生存,因而在世界各地都有规模化引进种植。目前,关于红边龙血树的研究多集中于室内外栽培和园艺研究相关,而对于红边龙血树的物种鉴定、遗传多样性和系统发育分析较少[2-4]。
叶绿体是植物最重要的细胞器之一,具有独立于核基因组之外、结构简单易于测序、在植物进化中高度保守等特点,能为植物的进化分类与资源利用提供可靠理论依据[5-6]。如基于叶绿体基因组可显著提高对栎属(Quercu)物种的辨别能力[7];叶绿体基因组分析发现扁果草(Isopyrum anemonoides)与东北扁果草(Isopyrum manshuricum)虽在形态上较为相似但应分别独立为一个属[8]等。同时,以叶绿体转化为主的叶绿体基因工程,在外源基因表达水平和转基因植物安全性有明显优势,并能互补核转化在控制转基因沉默和遗传稳定性方面的局限性[9],在黄花蒿和烟草中以叶绿体基因组为平台通过转基因工程已经实现了抗性性状的改良和产量的提升[10-11]。可见叶绿体基因组的解析对植物遗传、农艺等多方面研究有重要的贡献。
在龙血树属物种叶绿体基因组解析方面,2019年首次报道6个龙血树属物种的叶绿体基因组,通过叶绿体基因组序列可将不同的物种进行准确的识别和分类鉴定[12];随后,对加那利龙血树(Dracaena draco)叶绿体基因组的研究,揭示了其特殊的结构特征与在龙血树属内的进化地位[13];2022年通过对索科特拉龙血树(Dracaena cinnabari)和山樱花龙血树(Dracaena serrulata)叶绿体基因组的比较分析,揭示了二者的分子特征及独立于其他龙血树属植物的系统发育关系[14]。但由于龙血树属物种多样性较为丰富,对不同种类、形态特征和地理来源的龙血树物种研究较少,叶绿体基因组研究相对落后,截至目前仅有10种共12个龙血树叶绿体基因组完成测序,难以满足对龙血树属植物更深入的分子系统与基因工程研究需求,亟待补充与完善。
本研究首次对红边龙血树进行了叶绿体基因组测序分析,并与已报道的龙血树属物种叶绿体基因组进行了综合详细的比较,分析结果不仅有助于对红边龙血树进行分类鉴定和应用研究,对红边龙血树进行叶绿体基因组比较分析,同样丰富了龙血树属植物细胞器基因组信息库,以利于完善龙血树属起源与进化、品种改良以及种质资源开发利用的研究内容。
1 材料与方法
1.1 实验材料 本研究所用的红边龙血树种植于广西大学环境与资源微生物实验室苗圃(经度108.29,纬度22.85),供试材料生长健壮、长势良好。用于比较分析的近缘种叶绿体序列均从NCBI (https://www.ncbi.nlm.nih.gov/)的GenBank数据库下载,所属龙血树物种包括2个被鉴定为剑叶龙血树(Dracaena cochinchinensis, MF943127; MN200195),2个被鉴定为海南龙血树(Dracaena cambodiana, MH293451; MN200194)以及被鉴定为长花龙血树(Dracaena angustifolia, MN200193)、细枝龙血树(Dracaena elliptica, MN200196)、河口龙血树(Dracaena hokouensis, MN200197)、矮龙血树(Dracaena terniflora, MN200198)、加那利龙血树(MN990038)、山樱花龙血树(MT408026)、金心香龙血树(Dracaena fragrans, MW123093)、索科特拉龙血树(OK235335)的叶绿体序列,共12个。其中GenBank登录号为MF943127的剑叶龙血树经分析认为鉴定有误,并非剑叶龙血树,文中名称以一种龙血树(Dracaena sp. MF943127)表示。
1.2 方法
1.2.1 基因组提取与测序 剪取新鲜嫩叶,利用十六烷基三甲基溴化铵法(cetyltrimethylammonium bromide,CTAB)提取叶片总DNA,经质量检测合格后构建350 bp文库,利用二代Illumina HiSeq4000平台进行双末端测序,测序由深圳华大科技有限公司完成,下机后对原始数据过滤,共得到48.7 Gb clean reads。
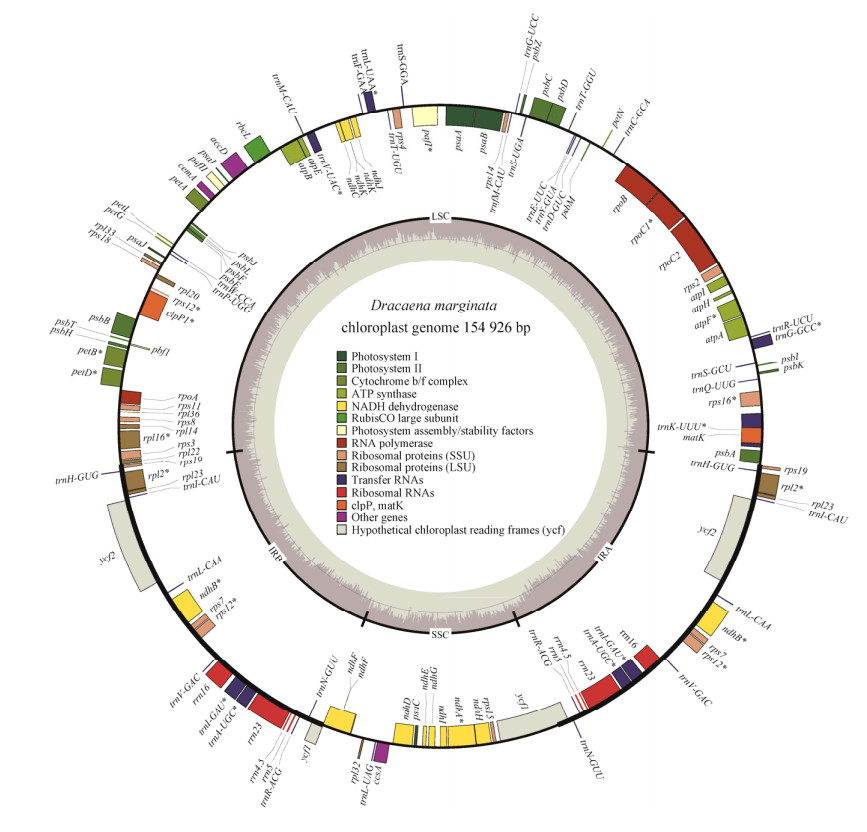
1.2.2 叶绿体基因组组装与注释 过滤后的数据使用GetOrganelle软件[15]进行从头组装,经手动检查校正获得一条环状叶绿体基因组序列。使用CPGAVAS2 (http://47.90.241.85:16019/analyzer/home/)在线注释工具进行在线注释[16],以海南龙血树叶绿体基因组(Genbank登录号MN200194)的注释信息作为参考,并进行多轮手动矫正获得了完整的注释信息,上传NCBI的GenBank数据库获得登录号为OP756062。叶绿体基因组图谱利用Chloroplot在线工具(https://irscope.shinyapps.io/Chloroplot/)绘制[17]。
1.2.3 叶绿体基因组特征分析 密码子偏好性分析使用Codon W (v1.4.2)软件对叶绿体基因组蛋白质编码区的密码子数量、密码子使用频率和同义密码子相对使用度(relative synonymous codon usage, RSCU)进行分析与统计。之后使用在线软件REPuter (设置参数:maximum computed repeats为1 000, minimal repeat size为30, hamming distance为3)对红边龙血树长重复序列进行分析[18],通过MISA软件(默认参数)进行简单重复序列(simple sequence repeat, SSR)位点分析[19]。进一步使用IRscope在线可视化工具,对龙血树属叶绿体基因组四分体边界进行比较分析[20]。
1.2.4 红边龙血树与其近缘物种系统发育分析 依据NCBI在线BLAST比对结果,下载万年青属的开口箭(Rohdea chinensis, MH356725)、弯蕊开口箭(Rohdea wattii, MW822041)、黄精属的卷叶黄精(Polygonatum cirrhifolium, MZ150834)、多花黄精(Polygonatum cyrtonema, MW248135)、长梗黄精(Polygonatum filipes, MZ150842),所下序列与龙血树属11个物种13个叶绿体共同使用Homblocks软件进行多序列比对[21],使用MEGA X软件(预测模型为“GTR+G+I”, bootstrap value 1 000)对龙血树属植物以及近缘种构建叶绿体基因组系统发育树[22]。
2 结果与分析
2.1 红边龙血树叶绿体序列特征分析 本研究所用的红边龙血树标本形态特征如图 1所示,其茎杆细长,茎秆和叶片边缘呈红色或紫色,剪取其健康的叶片,提取DNA进行全基因组测序。红边龙血树叶绿体基因组大小为154 926 bp,总的GC含量为37.54%,具有典型的四分体结构:2个反向重复区(inverted repeats, IR)将整个叶绿体基因组分隔为1个大单拷贝区(large single-copy, LSC)和1个小单拷贝区(small single-copy region, SSC)(图 2)。大单拷贝区和小单拷贝区长度分别为83 883 bp和18 515 bp,GC含量分别为35.54%和31.20%。2个反向重复区大小均为26 524 bp,GC含量为42.89%。
2.2 红边龙血树叶绿体基因类型分析 红边龙血树叶绿体基因组共注释出132个基因,包括86个编码蛋白质基因、38个转运RNA (transfer RNA, tRNA)基因和8个核糖体RNA (ribosomal RNA, rRNA)基因。进一步根据基因功能分为4类:光合作用基因(photosynthesis gene)、自我复制基因(self-replication)、其他基因(other genes)和未知功能基因(unkown genes)。其中,20个基因存在多个拷贝(表 1),23个基因含有内含子,包括pafI、clpP1、rps12基因有2个内含子(表 2)。
表 1 红边龙血树叶绿体基因组基因构成
Table 1 List of genes of Dracaena marginata chloroplast genome
| Category of genes |
Gene type |
Group of genes |
| Photosynthesis gene |
Rubisco large subunit |
rbcL |
| Subunits of ATP synthase |
atpA, atpB, atpE, atpF, atpH, atpI |
| Subunits of photosystem Ⅰ |
psaA, psaB, psaC, psaI, psaJ |
| Subunits of photosystem Ⅱ |
psbA, psbB, psbC, psbD, psbE, psbF, psbH, psbI, psbJ, psbK, psbL, psbM, psbT, psbZ |
| Subunits of NADH-dehydrogenase |
ndhA, ndhB*, ndhC, ndhD, ndhE, ndhF, ndhG, ndhH, ndhI, ndhJ, ndhK |
| Subunits of cytochrome b/f complex |
petA, petB, petD, petG, petL, petN |
| Self-replication |
DNA dependent RNA polymerase |
rpoA, rpoB, rpoC1, rpoC2 |
| Large subunit of ribosome |
rpl14, rpl16, rpl2*, rpl20, rpl22, rpl23*, rpl32, rpl33, rpl36 |
| Small subunit of ribosome |
rps11, rps12*, rps14, rps15, rps16, rps18, rps19*, rps2, rps3, rps4, rps7*, rps8 |
| Ribosomal RNA |
rrn16*, rrn23*, rrn4.5*, rrn5* |
| Transfer RNA |
trna-UGC*, trnC-GCA, trnD-GUC, trnE-UUC, trnF-GAA, trnG-GCC, trnG-UCC, trnH-GUG*, trnI-CAU*, trnI-GAU*, trnK-UUU, trnL-CAA*, trnL-UAA, trnL-UAG, trnM-CAU, trnN-GUU*, trnP-UGG, trnQ-UUG, trnR-ACG*, trnR-UCU, trnS-GCU, trnS-GGA, trnS-UGA, trnT-GGU, trnT-UGU, trnV-GAC*, trnV-UAC, trnW-CCA, trnY-GUA, trnfM-CAU |
| Other genes |
Subunit of acetyl-CoA-carboxylase |
accD |
| c-type cytochrom synthesis gene |
ccsA |
| Envelop membrane protein |
cemA |
| Maturase gene |
matK |
| Unkown genes |
Conserved open reading frames |
ycf1*, ycf2* |
| *: Two gene copies. |
表 2 红边龙血树叶绿体多外显子的基因信息
Table 2 Information of gene with multiple exons of chloroplast genome of Dracaena marginata
| Gene |
Strand |
Length of sequence (bp) |
| Exon Ⅰ |
Intron Ⅰ |
Exon Ⅱ |
Intron Ⅱ |
Exon Ⅲ |
| rps16 |
LSC |
35 |
868 |
218 |
| |
| atpF |
LSC |
143 |
828 |
404 |
| |
| rpoC1 |
LSC |
431 |
719 |
1 625 |
| |
| pafI |
LSC |
123 |
731 |
219 |
744 |
162 |
| rps12 |
LSC-TR |
25 |
543 |
231 |
– |
113 |
| clpP1 |
LSC |
68 |
813 |
290 |
645 |
224 |
| petB |
LSC |
5 |
763 |
641 |
| |
| petD |
LSC |
7 |
747 |
474 |
| |
| rpl16 |
LSC |
8 |
1 017 |
398 |
| |
| rpl2 |
IR |
390 |
653 |
430 |
| |
| ndhB |
IR |
776 |
700 |
755 |
| |
| rps12 |
LSC-TR |
113 |
– |
231 |
543 |
25 |
| ndhA |
SSC |
557 |
1 077 |
539 |
| |
| ndhB |
IR |
776 |
700 |
755 |
| |
| rpl2 |
IR |
390 |
653 |
430 |
| |
| trnK-UUU |
LSC |
36 |
2 567 |
39 |
| |
| trnG-GCC |
LSC |
22 |
717 |
36 |
| |
| trnL-UAA |
LSC |
16 |
545 |
49 |
| |
| trnV-UAC |
LSC |
38 |
587 |
36 |
| |
| trnI-GAU |
IR |
41 |
938 |
34 |
| |
| trnA-UGC |
IR |
37 |
817 |
34 |
| |
| trnA-UGC |
IR |
37 |
817 |
34 |
| |
| trnI-GAU |
IR |
41 |
938 |
34 |
| |
| –: Trans-splicing structure, without introns. |
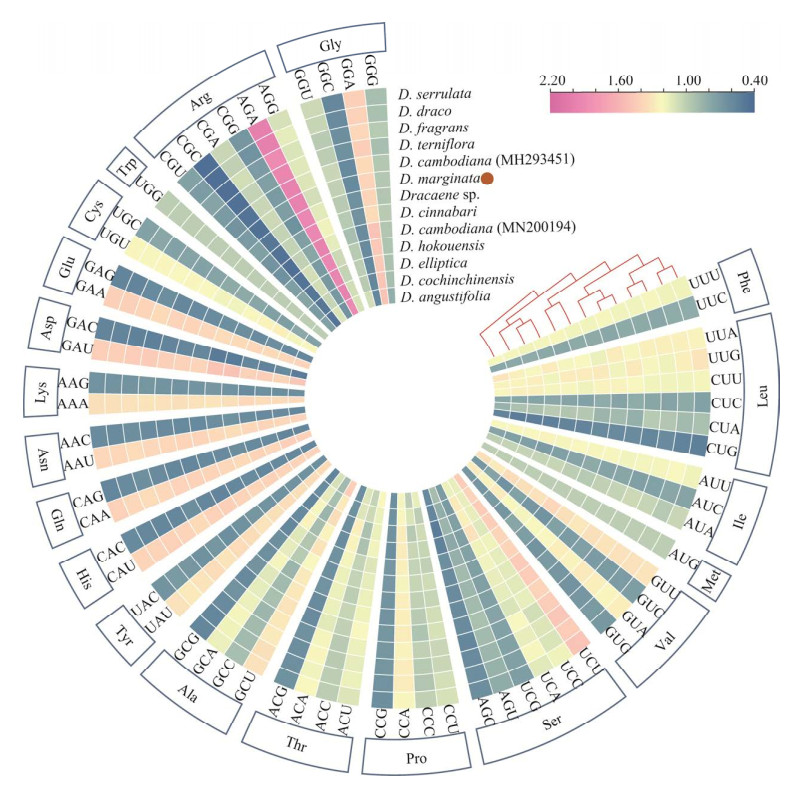
2.3 密码子偏好性分析 红边龙血树叶绿体基因组中有效密码子数(effective number of codon, ENC)为55.59,密码子的GC含量比例为38.6%,其中第3密码子是GC的比例为36%,密码子偏好性与龙血树(GenBank登录号MF943127)和海南龙血树最接近。在整个红边龙血树叶绿体基因组中,与其他龙血树叶绿体基因组一样,最常用到的密码子是编码苯丙氨酸(Phe)的AAA (数量为2 161),而编码精氨酸(Arg)的AGA具有最高相对同义密码子使用度(RSCU值为1.98),且也是唯一高频使用的密码子(图 3)。此外,还检测到33个RSCU值大于1.00的密码子(终止密码子除外),它们当中有27个以A或U结尾,仅有6个以C或G结尾,这一结果与龙血树属其他物种相似,表明在龙血树属的物种进化上叶绿体基因组密码子更倾向于以A或U结尾。
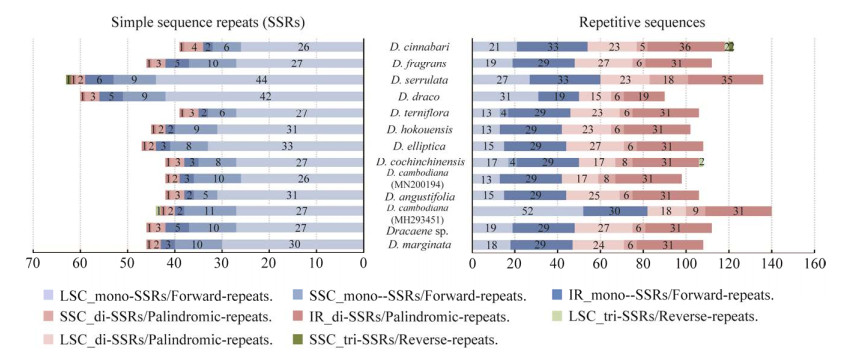
2.4 简单重复序列(SSR)与长重复序列分析 在红边龙血树叶绿体基因组中共预测到54个长重复序列(30–71 bp),包含23个正向重复序列(forward repeat, F)与31个回文重复序列(palindromic repeat, P),定位于LSC区、SSC区和IR区的占比分别为38.9%、5.6%和55.6% (图 4B)。在整个叶绿体基因组中未检测到反向重复序列(reverse repeat, R)与互补重复序列(comple-mentary repeat, C)。此外,还检测到46个简单重复序列(SSR),包含3个单碱基重复序列与3个双碱基重复序列(图 4A)。其中定位于LSC区的位点有30个(占比65.2%),定位于SSC区的位点有10个(占比21.7%),定位于IR区的位点有4个(占比8.7%)。在所有简单重复序列中,单碱基重复序列(mono-SSRs)均为A/T重复类型,其中,重复单元为A与重复单元为T的SSR位点均在重复频率为10时数目最多,均为13个。双碱基重复序列(di-SSRs)均为AT/TA重复类型,重复频率均为6,其中有一个TA重复位点位于LSC区,2个AT重复位点分别位于LSC区和SSC区。与其他龙血树叶绿体基因组相比,红边龙血树叶绿体基因组长重复序列的类型与分布与细枝龙血树、龙血树(GenBank登录号MF943127)、长花龙血树和金心香龙血树相似,短重复序列的类型和分布与河口龙血树、细枝龙血树和龙血树(GenBank登录号MF943127)相似。13个叶绿体基因组中,海南龙血树(GenBank登录号MH293451)检测出最多的长重复序列位点(数目为140),山樱花龙血树次之(数目为136),但检测出数目最多的简单重复序列位点(数目为63),与Ahmad等[14]的研究结果相似。
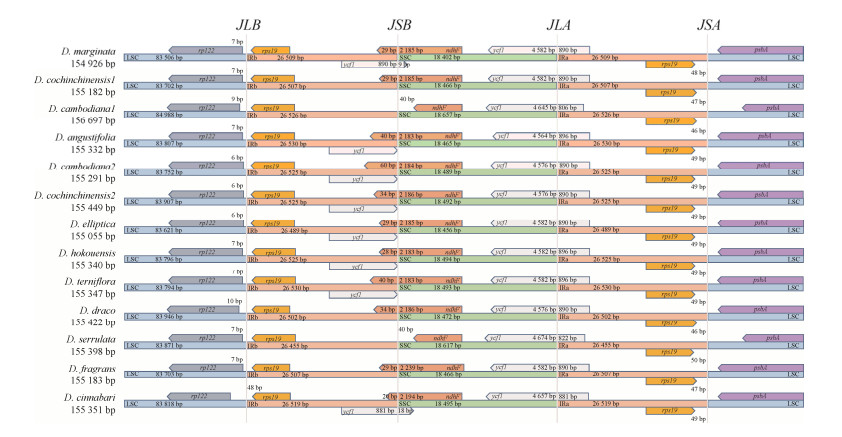
2.5 红边龙血树叶绿体IR区边界结构差异分析 IR区通常高度保守,对维持叶绿体基因组的稳定有着重要作用[23]。对叶绿体中IR区边界的结构分析发现13个龙血树叶绿体LSC区、SSC区、IR区大小分别为83 506–84 988 bp、18 402–18 657 bp、26 455–26 530 bp (图 5)。在13个龙血树叶绿体基因组中,红边龙血树的LSC区与SSC区最小。由两个IR区分隔单拷贝区所形成的边界上,rpl22与rps19基因分列在LSC/IRb (JLB)边界两侧,ycf1和ndhf基因分列或横跨于IRb/SSC (JSB)边界,ycf1基因横跨IRa/LSC (JSA)边界,rps19和psbA基因分列于SSC/Ira (JLA)边界。
红边龙血树JSB边界上的ycf1基因由IRb区跨越边界进入SSC区9个碱基,ndhf基因由SSC区跨越边界进入IRb区29个碱基(图 5),这与索科特拉龙血树JSB边界类型相似,但是JLB边界上的rpl22基因仅在索科特拉龙血树中发生了位移。红边龙血树的ndhF基因也跨越JSB边界,在IRb与SSC区长度分别为29 bp和2 185 bp,与龙血树(GenBank登录号MF943127)和细枝龙血树的跨越结果完全一样;ycf1基因跨越JLA边界在SSC与IRa区长度分别为4 582 bp和890 bp,与龙血树(GenBank登录号MF943127)、细枝龙血树和金心香龙血树的跨越结果完全一样。ycf1和psbA基因在海南龙血树(GenBank登录号MH293451)和山樱花龙血树JLA与JSA边界上有着相似的位移情况,体现出IRa区域向SSC区扩张但在LSC区方向上收缩,红边龙血树的JLA与JSA边界与除这二者外其他龙血树JLA与JSA边界相似。
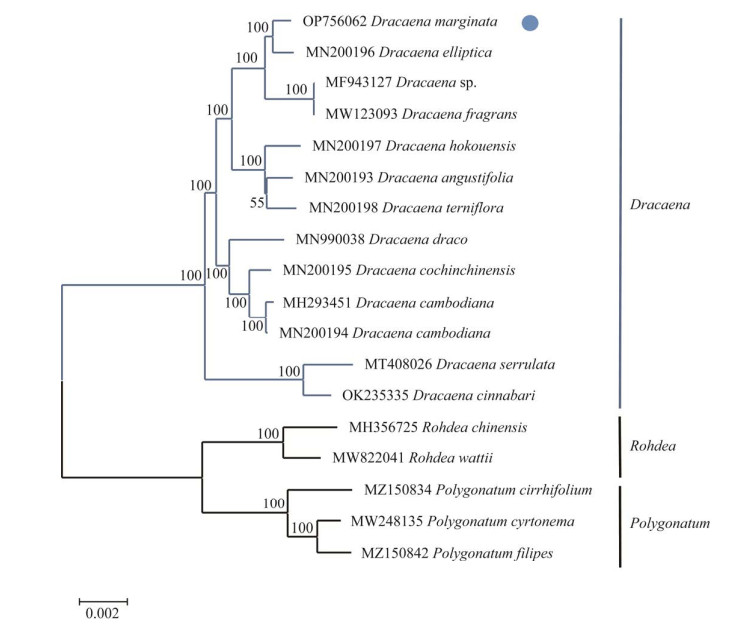
2.6 龙血树属植物系统发育分析 基于13个龙血树属植物与2个万年青属植物、3个芦笋属植物的叶绿体基因组构建系统发育树(图 6)。结果显示13个龙血树属植物均能很好地分类,聚为一个大的分支。红边龙血树与细枝龙血树聚为一类,两者亲缘关系最近,相似度达到99%。龙血树(GenBank登录号MF943127)和金心香龙血树聚为一类,又与红边龙血树聚为一支。索科特拉龙血树与山樱花龙血树独立于其他龙血树单独聚为一类,河口龙血树、矮龙血树、长花龙血树聚为一支,加那利龙血树与两个海南龙血树及剑叶龙血树(GenBank登录号为MN200195)聚为一支。
本研究构建的叶绿体基因组系统发育树拓扑结构与Ahmad等的结果一致[14],通过对龙血树形态比对,剑叶龙血树与红边、细枝、金心香龙血树在形态上具有较大差异,而与海南龙血树形态接近,历来分子研究也支持剑叶龙血树与海南龙血树有较近的亲缘关系,剑叶龙血树与海南龙血树可能是进化过程中的姐妹种。值得注意的是,其中两个被命名为剑叶龙血树的叶绿体序列(GenBank登录号分别为MF943127和MN200195)并未聚在一起,显示出较远亲缘关系,综合分析认为GenBank登录号为MF943127的物种不应该是剑叶龙血树,上传的物种名鉴定错误,造成了分类学的混乱,建议予以更正。
3 讨论与结论 叶绿体基因组为母系遗传,保守性较高,尤其在龙血树属物种间,叶绿体结构以及基因组成排列都高度保守。本研究首次组装获得了红边龙血树叶绿体基因组,全长154 926 bp,深入分析了其叶绿体基因组结构特征,注释得到132个功能基因,与龙血树属其他叶绿体基因组的基因数量和种类相近,表明龙血树属植物的叶绿体基因组总体比较保守。
红边龙血树叶绿体密码子偏好性较低,与其他龙血树叶绿体使用密码子的频率有较高一致性。在33个RSCU值大于1.00的密码子中,有27个第3碱基为A或U,表现出密码子以A或U结尾的偏好性。这种情况不仅出现在其他龙血树属物种中,在百合属[24]和丝兰属[25]等其他单子叶物种,山核桃属[26]、山茶属[27]和杨属[28]等双子叶物种中都有所体现,原因可能是由于叶绿体基因组高AT含量所致。
检测SSR位点时设置较高长度下限(单核苷酸到六核苷酸的最小重复值分别设置为10、6、5、5、5、5),虽然有较低检出率,但可有效减少无效检出[27]。在红边龙血树叶绿体基因组中共检测到52个SSR位点、108个长重复序列位点(重复两次),其中SSR位点大多位于LSC区,而长重复序列位点则大多位于IR区。虽然检测SSR位点时参数设置不同,但本文龙血树属叶绿体基因组中,例如剑叶龙血树(GenBank登录号MN200195)、加那利龙血树和索科特拉龙血树等,SSR位点类型与分布的研究结果与其他研究团队所报道结果相近[12-14],而红边龙血树与龙血树(GenBank登录号MF943127)、细枝龙血树、金心香龙血树有着相似的重复序列类型及分布情况。进一步对IR边界进行研究,同样发现红边龙血树的IR边界基因跨越信息与龙血树(GenBank登录号MF943127)、细枝龙血树、金心香龙血树的跨越结果基本一致,体现了红边龙血树叶绿体结构与龙血树(GenBank登录号MF943127)、细枝龙血树、金心香龙血树有着高度相似性。
对龙血树属、万年青属和黄精属植物叶绿体基因组的聚类分析表明,红边龙血树与细枝龙血树聚为一支,关系最近,这与从重复序列及IR边界角度分析得出的二者近缘结果相一致。本研究使用叶绿体全基因组构建系统发育树的结果,支持山樱花龙血树与索科特拉龙血树单独聚为一支,加那利龙血树与海南龙血树、剑叶龙血树(GenBank登录号MN200195)聚为一支[14],而与Lu等基于trnL-trnF、ndhF-repl32、trnQ-rpsl6等联合片段分析认为山樱花龙血树与加那利龙血树近缘的结果不相符[29]。基于叶绿体全基因组建树可以充分利用保守的基因组中零散分布的多样性碱基,给予树状结构更高的支持度,比基于少量DNA片段构建的系统发育树有更高可信度。在龙血树属物种之间,仅通过少量基因进行鉴定存在一定误差。
综上所述,本研究获得了红边龙血树叶绿体基因组的序列、基因构成、基因组结构等基本特征,开展了多物种的比较叶绿体基因组分析,并利用叶绿体基因组序列构建了红边龙血树的系统发育关系,为未来红边龙血树的物种鉴定、进化地位、基因组研究、叶绿体转基因工程等提供重要理论依据,同时也是对龙血树属植物细胞器的遗传多样性与综合开发利用研究的重要补充。
 2023, Vol. 39
2023, Vol. 39