中国科学院微生物研究所、中国微生物学会主办
文章信息
- 张晨曦, 刘鼎阔
- ZHANG Chenxi, LIU Dingkuo
- 细菌群集运动特性的研究进展
- Characterization of bacterial swarming motility: a review
- 生物工程学报, 2023, 39(8): 3188-3203
- Chinese Journal of Biotechnology, 2023, 39(8): 3188-3203
- 10.13345/j.cjb.220892
-
文章历史
- Received: November 9, 2022
- Accepted: February 1, 2023
2. 天津市生物饲料添加剂重点实验室, 天津 300383
2. Tianjin Key Laboratory of Biological Feed Additives, Tianjin 300383, China
细菌借助鞭毛、Ⅳ型菌毛(type Ⅳ pili, TFP)和表面活性剂在半固体培养基的表面协调性地迁移,这种运动模式称为群集运动(swarming motility)。除此之外,细菌运动还包括在液体环境中由鞭毛单独介导的游泳运动(swimming motility)和在固体表面上由Ⅳ型菌毛单独介导的蹭行运动(twitching motility)等。游泳运动和蹭行运动主要是细菌的个体行为,而群集运动是细菌的群体行为。群集运动对于细菌的生存具有重要意义,它可以帮助细菌开拓新的栖息地,也在细菌的表面不可逆黏附过程中发挥着重要作用[1]。早在1947年,Lominski等[2]就使用分离膜蛋白常用的洗涤剂抑制变形杆菌(Bacillus proteus)的群集运动,但由于受到其过程复杂性的影响,抑制效果并不明显,于是展开了对群集运动原因和过程的探索。群集运动是一个周期性的、定向的运动。通常群集运动是突然发生的,其表现为成群的微生物从菌落的接种点向外扩展并迁移相当远的距离,在适宜的条件下可完全覆盖培养基表面。细菌的群集运动常常造成医院内感染,例如与患者导尿管相关的细菌感染现象[3]。理解细菌群集运动的特性有助于探索这种集体行为的发生机制,从而为抑制微生物扩散提供新的方法,最终期望能改善对细菌感染的防治。
细菌对群集运动的适应性变化包括细胞伸长、鞭毛过表达、特殊定子蛋白的募集和表面活性剂的分泌等[3-6]。本文综述了群集运动背后的分子生物学机制以及生物物理性质的变化,讨论了环境因素对细菌群集运动的影响,总结和评述了细菌群集运动在生物技术方面的应用,并对该领域的发展和面临的挑战进行了展望。
1 细菌群集运动细菌群集运动是由鞭毛和Ⅳ型菌毛共同介导的群体运动行为。类似的群体运动也在其他生物中出现,比如昆虫的聚集,鸟群和鱼群的群体运动等[7-9]。群集运动涉及一些细胞过程,例如关键蛋白表达的变化、细胞间化学信号交流的变化以及机械性变化[3, 10-14],具体表现为细胞可以在群集运动前改变其自身的纵横比并生长出额外的鞭毛等。同时,菌毛蛋白的表达、胞内第二信使c-di-GMP的含量以及细菌群体感应系统等在分子生物学机制层面上能够对细菌群集运动产生一定的影响。群集运动的典型特征是稠密排列的细胞簇以连贯的旋涡和喷射的模式移动[15]。此外,与单分散游动的细菌不同,在群集运动时细菌与表面边界不断发生相互作用[16]。
在群集运动过程中,细菌利用鞭毛驱动自身向前或后退。例如,Wu等[17]发现,黄色粘球菌(Myxococcus xanthus)通过这种前进-后退式的周期性的方向逆转高效地从接种中心迁移到群集前缘的低密度区,从而降低局部竞争,获得更多的氧气和营养。不同菌种细胞具有不同的鞭毛特性,因而群集运动中细胞鞭毛的变化也因菌种不同而有差异。Harshey等研究发现在群集运动期间,大肠杆菌(Escherichia coli)和鼠伤寒杆菌(Salmonella typhimurium)细胞的鞭毛数量增加了1倍;而在铜绿假单胞菌(Pseudomonas aeruginosa)群集运动期间,细胞则产生了2个极性鞭毛[18-19]。群集运动除了与细胞自身特性有关外,也跟外界环境有着密切的关系。围绕着铜绿假单胞菌群集运动细胞外基质是一个水合环境,由多糖、生物表面活性剂、多肽和蛋白质组成。这个基质需要有足够的水分来保持鞭毛的正常运作,并为不断扩张的菌落提供足够的体积。有研究发现群集前缘卷须的形成需要鼠李糖脂,例如铜绿假单胞菌在群集运动时分泌鼠李糖脂表面活性剂,用于降低定殖表面液体层的表面张力,从而促进群集运动[20-22]。细菌群集运动伴随着大量毒力相关基因的过度表达,如编码Ⅲ型分泌系统及其效应物的基因,编码细胞外蛋白酶的基因,以及与铁运输相关的基因[23-24]。此外,与浮游细胞相比,群集运动细胞对多粘菌素B、庆大霉素和环丙沙星表现出适应性抗生素耐药性。综上可知,群集运动过程的复杂性导致临床上对其防治困难,对该运动模式的理解具有深远的理论研究及应用价值。
2 群集运动的分子生物学机制 2.1 鞭毛的作用细菌的鞭毛通过质子或钠离子的跨膜能驱动自身马达转动,鞭毛逆时针(counter clockwise, CCW)旋转成束,驱动细菌向前“奔跑”;鞭毛顺时针(clockwise, CW)旋转则解束,使细菌呈现“翻滚”状的运动模式,导致细菌运动的重新定向[25-28]。鞭毛旋转方向的周期性转换是发生群集运动的细菌群落快速定殖于表面所必需的,细菌能够通过这种方式促进表面润湿以克服表面附近的粘性阻力,从而实现群集的快速扩张[29-30]。在大肠杆菌中,鞭毛马达的转子作为一个转换器,由FliG、FliM和FliN蛋白组成,决定鞭毛的顺时针或逆时针旋转[31-32]。趋化信号分子磷酸-CheY通过与FliM N端附近的保守段结合,能够将鞭毛的逆时针旋转转换为顺时针旋转[33-34]。除此之外,鞭毛在细菌群集运动期间常常过表达,在宏观上表现为鞭毛密度的增加,以此来产生更高的细胞推进力[4]。Tuson等[3]测试了鞭毛密度和细胞速度之间的关系,他们在奇异变形杆菌(Proteus mirabilis)细胞中过量表达了鞭毛操纵子的主调节器FlhD4C2,发现鞭毛密度的增加导致了群集运动细胞速度的增加。
群集运动细胞会产生比浮游细胞更高的鞭毛扭矩,用于克服增强的粘性阻力。当外部负载增强时,鞭毛马达会大量招募定子单元,从而产生更高的扭矩[35]。人们认为大约有十几个MotAB鞭毛定子复合物通过MotB的C端肽聚糖结合域锚定在鞭毛马达周围的肽聚糖层上,形成一个跨膜质子或钠离子通道,将膜梯度与鞭毛扭矩的产生耦合起来。MotB蛋白由309个残基组成,包括1个小的N端细胞质片段(残基1–29)、1个单一的TM螺旋(残基30–50)和1个大的C端周质结构域(残基51–309) (图 1A)[26]。这些残基对扭矩形成的调节作用通常较为复杂。例如,残基51–72作为MotAB质子通道的塞子,能够在定子装配到马达前防止质子过早泄漏[37-38]。Castillo等[26]分析了MotB (Δ72–100)突变菌株鞭毛马达的扭矩-速度关系,实验结果表明,MotB的第72−100个残基是对活性定子数量的负载依赖性调控所必需的。Che等[39]将沙门氏菌MotB跨膜螺旋上的关键天冬氨酸残基(结合阳离子)突变为谷氨酸,在低负载下鞭毛马达的转速显著降低,在高负载下能够获得稳定的转速,而沙门氏菌野生型菌株鞭毛马达的转速在任何负载下都是稳定的,表明MotB (D33E)突变极大地改变了马达性能对外部负载变化的反应。
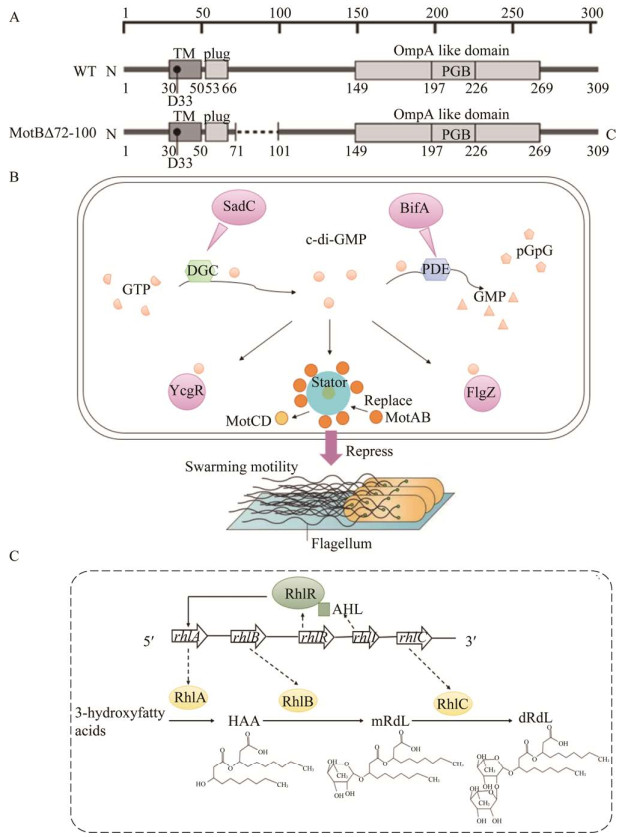
|
| 图 1 群集运动的分子生物学机制[26, 36] Fig. 1 The molecular biological mechanisms of swarming motility[26, 36]. A:MotB和MotB (Δ72−100)的主要结构. 沙门氏菌MotB有1个单一的跨膜结构域(“TM”,残基30−50)和1个包括OmpA样结构域(残基149−269)的周质区,该结构域有1个假定的肽聚糖结合(PGB)基序(残基197−226). B:C-di-GMP介导的细菌群集运动信号通路. C:AHLs信号分子调控铜绿假单胞菌鼠李糖脂生物合成途径(根据参考文献[36]修改) A: Primary structure of MotB and MotB (Δ72−100). Salmonella MotB contains 309 amino acids and has a single transmembrane domain ("TM", residues 30−50) and a periplasmic region including an OmpA-like domain (residues 149−269) with a putative peptidoglycanbinding (PGB) motif (residues 197−226). B: The bacterial swarming motility signaling pathway mediated by c-di-GMP. C: Rhamnolipid biosynthesis pathway in P. aeruginosa regulated by AHLs (modified from reference[36]). |
| |
Ⅳ型菌毛是细菌表面的动态丝状附件,通过伸出和缩回影响细菌与表面相关的行为。铜绿假单胞菌pilA、pilW、pilX或pilY1基因的突变菌株Ⅳ型菌毛表达量下降,与铜绿假单胞菌野生型(wild type, WT)菌株相比群集运动能力增强,而Ⅳ型菌毛收缩功能受损的pilH突变菌株表现出群集运动能力下降[40-43]。Anyan等[44]在此基础之上利用实验和计算模拟的结合研究了Ⅳ型菌毛在细菌群集运动过程中的作用机制,结果表明细胞间的Ⅳ型菌毛-Ⅳ型菌毛相互关联促进了细胞-细胞的相互作用,限制了单个细胞的移动,减缓了群集运动的速度。然而,Ⅳ型菌毛缺失的菌株无法改变其径向游动方向,容易进入有毒环境而导致细胞死亡,Ⅳ型菌毛完整的野生型菌株细胞能够停止向有毒化合物的方向游动,从而有助于其获得更好的生存环境。由此说明,Ⅳ型菌毛对细菌群集运动的影响具有一定的复杂性。
2.3 c-di-GMP对群集运动的调节细胞内的第二信使环二鸟苷酸(cyclic diguanosine monophosphate, c-di-GMP)是多种细菌的重要调节器。它参与调控细菌的多种功能,包括细胞分化、生物被膜的形成以及鞭毛与菌毛介导的细胞运动行为等[45-47]。细胞内c-di-GMP的稳态水平是由该分子的相对合成和降解速度控制的。二鸟苷酸环化酶(diguanylate cyclases, DGCs)作为一种c-di-GMP的合成酶,可以将2个三磷酸鸟苷(guanosine triphosphate, GTP)分子催化合成1个c-di-GMP,这种酶的结构特征是具有1个GGDEF结构域;而磷酸二酯酶(phosphodiesterases, PDEs)作为c-di-GMP的一种分解酶,可将c-di-GMP分解为不同的产物,该酶的结构特征是具有1个EAL或HD-GYP结构域。通常情况下,二鸟苷酸环化酶活性增强使得细胞内c-di-GMP水平升高,导致生物被膜形成能力的增强,抑制鞭毛和菌毛介导的运动能力;相反,磷酸二酯酶活性增加使得c-di-GMP水平下降,导致细菌生物被膜形成减少,促进鞭毛和菌毛介导的运动能力[48]。在铜绿假单胞菌中,突变具有PDE功能的BifA蛋白活性点位导致细胞中c-di-GMP的水平相对于野生型菌株明显增加,该突变菌株群集运动能力出现严重缺陷且能够形成更厚的生物被膜[49]。而具有DGC功能的SadC蛋白则相反,突变该蛋白的活性点位导致细胞内c-di-GMP的水平相对于野生型菌株下降,从而对群集运动以及生物被膜的形成分别具有促进和抑制的作用[50]。
c-di-GMP对于细胞群集运动行为的调控可以通过多种分子机制来实现。c-di-GMP可以通过调节鞭毛蛋白的表达进而调节细菌的群集运动。例如,在大肠杆菌和沙门氏菌中,YcgR与鞭毛转子蛋白FliG和FliM以一种依赖c-di-GMP的方式相互作用,与第二信使结合的YcgR改变了FliG C端结构域的方向,从而使转子-定子的界面被破坏,导致扭矩产生的效率降低,使鞭毛马达偏向于逆时针旋转[51]。MotCD是负责驱动铜绿假单胞菌群集运动的定子,MotAB定子无法支持群集运动,Kuchma等[52]提出,在c-di-GMP含量较高时,MotAB定子能够将MotCD从鞭毛马达上移开,从而影响马达功能,进而抑制细菌群集运动。此外,Baker等[35]的研究发现,当c-di-GMP含量升高时,铜绿假单胞菌群集运动被含有PilZ结构域的蛋白FlgZ和胞外多糖Pel的产生所抑制。研究证明FlgZ是c-di-GMP的受体,与c-di-GMP结合的FlgZ通过与MotCD定子蛋白的相互作用,进而阻碍群集运动。C-di-GMP对细菌群集运动的调节机制如(图 1B)所示。
2.4 群体感应对群集运动的调节群体感应(quorum sensing, QS)是指微生物群体在其生长过程中,由于群体密度的增加,信号分子的浓度也达到一定水平,通过包括受体蛋白在内相关蛋白的信号传递,影响特定基因的表达,调控微生物群体的生理特征,如生物被膜形成和其他群体行为等[53]。N-酰基高丝氨酸内酯类(acyl-homoserine lactones, AHLs)化合物是革兰氏阴性细菌的一类信号分子,革兰氏阳性细菌一般利用寡肽类分子(autoinducer-P, AI-P)作为信号分子,另外许多革兰氏阴性和阳性细菌都可以产生一种通用信号分子AI-2[54]。有些细菌利用两种甚至3种不同信号分子调节自身群体行为,说明群体感应机制极为复杂。
细菌的群集运动通常发生在细胞数密度较大的条件下。在此条件下,细胞的行为受到群体感应信号的调控,进而影响到细胞的群集运动。其中,受群体感应调控表达的表面活性分子对于细胞的群集运动有非常重要的影响。在铜绿假单胞菌中,这种表面活性分子是二鼠李糖-β-羟基烷酸-β-羟基烷酸酯(鼠李糖脂)。利用两种AHLs信号能够调节鼠李糖脂的合成,这两种AHLs分别与LuxR同源转录调控因子LasR和RhlR具有特异的亲和性。只有当丁酰基高丝氨酸内酯的浓度达到一定阈值时,RhlR才激活rhlA和rhlB基因的表达,进而促进鼠李糖脂的合成(图 1C)[36]。在水稻细菌性谷枯病菌(Burkholderia glumae)中同样发现群体感应调节鼠李糖脂的产生[21]。早期的研究表明[55],水稻细菌性谷枯病菌BGR1菌株的一个群体感应信号阴性突变菌株(tofI–)在28 ℃和37 ℃条件下都缺乏群集运动。外源性的辛酰基-l-高丝氨酸内酯的加入使tofI–突变菌株的群集运动表型恢复正常,且鼠李糖脂的产量恢复到野生型菌株水平。同时,将外源鼠李糖脂加入到培养基中也能够恢复水稻细菌性谷枯病菌tofI–突变菌株的群集运动,证明群体感应通过正向调控鼠李糖脂的合成进而正向调节水稻细菌性谷枯病菌的群集运动。
3 单细胞尺度上群集运动行为的表征细菌细胞通过在半固体表面进行的群集运动,其在表面的覆盖区域可以从最开始的接种点,发展为相对复杂的密度不均一的图案。按照发展的不同时间阶段,可以分为:低密度单细胞期(SC);单细胞和类竹筏状期(SC+R);高密度类竹筏状期(R);类竹筏状和生物被膜前体期(R+BP);生物被膜期(B) (图 2A (a-b)) [56]。细胞在不同的群集运动阶段具有不同的运动速度和密度等。细菌在单细胞期运动速度较高,但细胞密度最低;类竹筏状期的细胞运动速度与单细胞期相似,但细胞密度有所增加;生物被膜期的细胞运动速度最低,细胞密度达到最高(图 2A (c))[56]。在空间分布上,在菌落的群集运动中其边缘处的细菌与接种点中心的细菌相比,鞭毛密度增加,菌体更长,且排列成链[4]。这些结果表明,群集运动中不同位置处的细菌能够自主调节其生物物理特征,以适应不同的运动状态和模式。
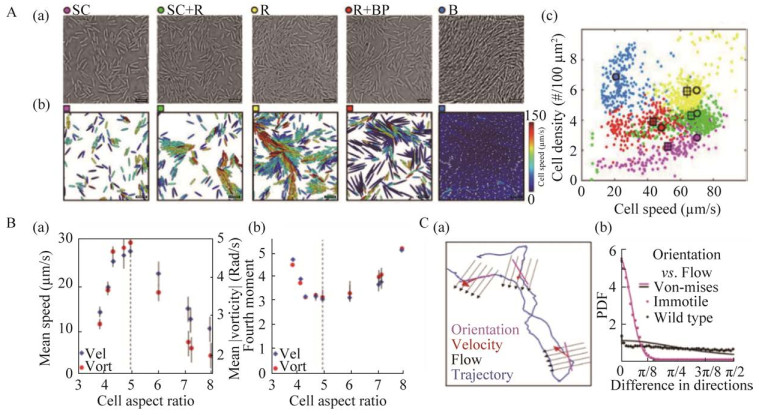
|
| 图 2 单细胞尺度上群集运动行为的表征[56-58] Fig. 2 Characterization of swarming behavior at the single-cell scale[56-58]. A:微观动力学角度下的群集运动阶段. a: 群集运动不同阶段的典型图像;b: 对于每个阶段,用从特定阶段提取的细胞形状、运动性和密度作为输入参数进行模拟(比例尺,10 µm);c: 群集运动不同阶段对应的细胞速度和细胞密度,圆圈代表实验值,方框代表模拟值. B:群集边缘的光流分析. a: 平均速度和平均绝对涡度与细胞长宽比的函数关系;b: 速度和涡度场的第四矩(峰度)与细胞长宽比的函数关系. C:单个细胞与集体流的方向差异. a: 单个细菌的轨迹示例(蓝色线),与集体流(黑色箭头)、细胞方向(粉色条)和细胞瞬时速度(红色箭头)的叠加;b: 同一时间、同一位置上细胞方向与流场之间夹角的分布. 静止的细胞倾向于与集体流对齐,而野生型菌株表现出较大的偏差. 冯·米塞斯分布与结果的拟合较差,这表明当细菌在活跃的群集内移动时,静止细胞和野生型菌株细胞之间具有显著差异,并揭示了细胞自我推进的重要性 A: The swarming phases from microscopic dynamics. a: Typical images for different phases of swarming; b: For each phase, simulations were run with the cell shape, motility, and density extracted from the particular phase as input parameters. (Scale bars, 10 µm); c: Cell speed and cell density corresponding to different phases of swarming, the circles represent experimental values and the boxes represent simulated values. B: Optical flow analyses of the swarm edge. a: The mean speed and mean absolute vorticity as a function of cell aspect ratio; b: The scaled fourth moment (kurtosis) of the velocity and vorticity fields as a function of the aspect ratio. C: Directional differences between individual cells and flow. a: An example trajectory of an individual bacteria (blue line), superimposed with the collective flow (black arrows), cell orientation (pink bar), and instantaneous cell velocity (red arrow); b: Distribution of angles between cell orientation and the flow vector field at the same position and time. Immotile cells tend to be aligned with the flow while WT exhibit large deviations. The von-Mises distribution fits the results poorly, which demonstrates the significant differences between immotile and WT cells while moving inside the active swarm, and exposes the complex importance of self-propulsion of the cells. |
| |
为了更好地理解细菌细胞的群集运动动力学,人们构建了细菌群集运动的动力学模型[59-60]。在这些模型中,细菌通常被视为是可以自主驱动的棒状物体,在群集运动过程中,细菌受到短程的空间力和长程的水动力共同作用,几何因素如细菌的长宽比往往起着重要作用[61]。一些群集运动的细菌在实际运动开始前会伸长,在一些对自主推进的棒状粒子的大量研究中证实[9, 15],伸长会促进排列,强烈影响其动力学特性。根据理论模型研究,长径比小的粒子由于颗粒间的弱排列相互作用,不能形成宏观有序相;相反,较长的粒子容易形成堵塞相(jamming phase),具有迁移率低的特点。Peruani等[9]将单个自驱动棒状粒子在成簇的生长过渡过程与粒子的长宽比变化联系起来,在固定的填充率(packing fraction) η下,由kc=C/η−1给出长宽比的过渡临界值kc,其中C为常数,约等于1.46。因此,在真实群集运动中,细菌的长宽比不能过大或过小。通常情况下,大多数群集物种的长宽比为5[10, 13]。Ilkanaiv等[57]将具有相同宽度但不同长度的野生型枯草芽孢杆菌(Bacillus subtilis)和8个不同突变株的菌落接种到琼脂平板上并允许其生长,研究了不同长度的细菌群集运动的集体动力学。实验表明,野生型菌株(长宽比为4.9)和具有相似长宽比的突变菌株运动最快。然而,较短和较长的细胞表现出反常的、非高斯统计的速度分布和自相关函数的非指数衰减,表明其群集运动能力较低(图 2B (a-b))[57]。更长的刚性菌如长宽比为20的C型类芽孢杆菌(Paenibacillus dendritiformis)不具有典型的旋涡和喷射状群集运动模式,而是以密集且细长的直线条移动[61]。然而,Tuson等[3]观察到长宽比同样为20的奇异变形杆菌出现了典型的旋涡和喷射现象。它与长宽比为5的细菌物种具有相似的群集运动模式。这是因为这些细胞在群集运动过程中可以弯曲,细胞比较柔软,不属于自驱动棒状杆范畴。
细菌单细胞运动对菌落整体群集运动能力的影响具有多重性。成簇的细胞在时间尺度上出现在细菌群集运动中的单细胞-类竹筏状过渡阶段,在空间分布上则存在于菌群集体的前缘[17]。在黄色粘球菌(Myxococcus xanthus)中,细菌通过周期性逆转其运动方向,增加了群集边缘的细菌外流,促进了菌落前缘的细胞排列,并产生平行细胞簇。由此可见,群集运动中的单细胞并不一定与群集整体具有同样的运动方向。Ryan等[58]也发现枯草芽孢杆菌野生型菌株群集运动中的细菌并不严格遵循集体流方向,可能垂直甚至逆着集体流游动。此外,在高倍放大下精确测量的细胞轴(细胞方向)与集体流之间的角度显示出很大的偏差(图 2C (a–b))[58]。因此,从活性物质的角度看,细胞的个体自我推进对整个群集运动的影响,不仅仅是简单地产生前进运动,可能与细胞在集体流之间移动的能力有关。Meacock等[62]利用单细胞追踪技术发现,在细胞的高密度情况下,当∆pilH与野生型菌株混合时,具有更高运动活性的铜绿假单胞菌∆pilH菌株会被缓慢移动的野生型细胞取代。根据液晶理论[63-64],具有不同方向的细胞在彼此相遇处形成拓扑缺陷,这样的点缺陷有2种不同的形状,一种是具有1个对称轴的+1/2拓扑电荷缺陷,呈“彗星”状,另一种是具有3个对称轴的−1/2拓扑电荷缺陷,呈“三叶草”状。2个具有+1/2拓扑电荷的“彗星”能够在足够小的间隔内合并,导致其中的细胞通过垂直站立的方式由二维空间逃离到三维空间。运动活性更高的∆pilH菌株与野生型菌株相比更易克服“彗星”间的排斥力,导致细胞更多地逃离到三维空间从而受困于营养匮乏的菌落内部。单个细胞运动速度较慢的菌落其活性不足以克服排斥力,因此能够避免局部受困从而实现快速扩张。
4 环境因素对群集运动的影响一些在表面生长的模型系统中的研究工作表明,琼脂平板表面的群集运动受到平板环境的影响[65]。以沙门氏菌为例,研究发现添加不同碳源可以观察到不同的群集模式,表明群集运动对不同的营养类别有特异性反应[66]。Shrout等[40]观察了铜绿假单胞菌野生型菌株和群体感应突变菌株在不同碳源上的群集运动直径,发现野生型菌株在以琥珀酸或谷氨酸为唯一碳源的培养基上生长时很容易群集,但在以葡萄糖为唯一碳源的培养基上的群集运动能力较差;群体感应突变株在琥珀酸条件下生长时群集能力有缺陷,其他2个测试碳源上的群集类似于野生型菌株,在谷氨酸上的群集直径较大,而在葡萄糖上的群集直径很小。随后,Jamieson等[67]在几种不同培养条件下检测了奇异贪噬菌(Variovorax paradoxus)的群集运动,当提供铵离子以及组氨酸、色氨酸或甘氨酸作为氮源时,观察到了快速的群集运动;当使用蛋氨酸、精氨酸或酪氨酸时,则观察到了较慢的群集运动。因此,不同碳源和氮源相对应的营养环境能够影响细菌的群集运动。然而,营养条件影响细菌群集运动的机理比较复杂,一些多肽或氨基酸可能在细菌中具有信号转导、参与调节细胞内第二信使的水平或生物表面活性剂的分泌等多种功能[68]。例如,Bernier等[69]利用先前在囊性纤维化痰中鉴定出的19种氨基酸作为营养源,发现精氨酸可以抑制铜绿假单胞菌的群集运动。二鸟苷酸环化酶(SadC和RoeA)双突变菌株的c-di-GMP合成能力不足,该突变株在含4.8 mmol/L精氨酸的培养基上呈现出野生型菌株的群集运动能力,同时在与群集运动实验所用相同培养基中的浮游细胞内检测到c-di-GMP明显增加了1.7倍,这些结果表明精氨酸的存在促进了c-di-GMP的合成,弥补了突变菌株细胞内c-di-GMP水平的不足。这些结果说明,氨基酸可以通过控制细胞内c-di-GMP水平,在一定程度上调节群集运动[69-70]。此外,Xavier等[71]研究发现鼠李糖脂的产生受基本培养基中氮元素限制的调节,证明氮元素能够影响鼠李糖脂表面活性剂的分泌。Kollaran等[72]发现,在蛋白胨生长培养基(peptone growth media, PGM)中培养的铜绿假单胞菌PA14菌株,其鼠李糖基转移酶链A (rhlA)的转录高于在细菌基础培养基(Luria Bertani, LB)中培养的PA14。综上所述,细菌周围营养条件也可以通过调节鼠李糖脂表面活性剂的合成进而调控群集运动的发生。
细菌周围环境中的水分含量对群集运动也会有着一定的影响,研究证明,细菌的群集能力以及在固体培养基表面产生液体膜的能力在很大程度上依赖于琼脂浓度。大多数细菌的群集运动发生在一个狭窄的琼脂浓度范围内,如铜绿假单胞菌群集运动发生所需的琼脂浓度在0.5%–1%之间,过少的水将导致不良的群集运动,而过多的水将可能允许细菌游泳运动(swimming motility)的发生[13, 20, 73]。有报道称,铜绿假单胞菌在硬琼脂上的rhlA表达量比在稍软琼脂上的表达量低,硬琼脂表面在非常接近于群集边缘处限制了群体感应的启动和鼠李糖脂的产生,在宏观上表现为边缘卷须形成的减少(图 3)[20]。另一方面,在创造群集运动所需要的流体环境时,群集菌落下方的琼脂和黏液基质之间的渗透活性的比率很重要[19]。Yang等[74]利用达西定律(Darcy’s law)分析了基质含水量对细菌群集运动的影响。根据该定律,流量与琼脂的渗透系数k成正比,增加琼脂的百分比降低了凝胶基质的渗透性,导致固定压差下的水流减少,水流的减少反过来又会使细菌的群集运动速度降低。
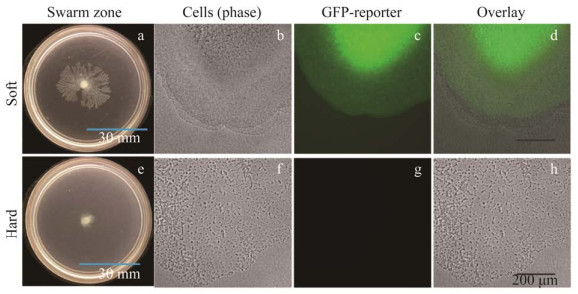
|
| 图 3 分别在软琼脂(0.4%)和硬(0.6%)琼脂上生长40 h后,有鼠李糖脂结合荧光指示的野生型铜绿假单胞菌群集运动[20] Fig. 3 Pseudomonas aeruginosa swarming after 40 hours for a rhamnolipid fluorescence reporter wild-type strain growing on soft (0.4%) and hard (0.6%) agar[20]. (a, e):整个群集菌落. (b, f):群集边缘的相差图像. (c, g):PrhlA: : gfp结合的荧光图像. (d, h):b/c的叠加图像与f/g的叠加图像,表明鼠李糖脂产生于邻近群集边缘处. 平板图像比例尺=30 mm;叠加图像比例尺=200 mm (a, e): Entire swarm colony. (b, f): Phase-contrast image of swarm edge. (c, g): Fluorescence of PrhlA: : gfp fusion. (d, h): Overlay of b+c and f+g showing proximity of rhamnolipid production to the swarm edge. Whole plate scale bar=30 mm; Overlay scale bar=200 mm. |
| |
基于细菌群集运动的这一特性,研究人员开发出用于疾病治疗的新手段。De等[75]研究了细菌群集运动与肠道应激(intestinal stress)之间的关系,他们将分离出的优势群集运动菌株接种到结肠炎小鼠体内,取其粪便进行16S rRNA测序、转录组分析并对微生物体外相互作用进行研究。结果发现从小鼠粪便中分离的肠杆菌(Enterobacter)群集菌株SM3,与群集运动缺陷菌株相比能够为肠道系统创造厌氧环境,从而可以富集具有修复炎症肠道屏障作用的有益厌氧菌(类杆菌S24-7家族),最终实现对肠道炎症的有效缓解。
此外,细菌群集运动的特性在农业方面也展现出潜在应用价值。枯草芽孢杆菌是植物生长-促进根际细菌(plant growth promoting rhizobacteria, PGPR)的一种,具有刺激植物生长和控制植物疾病的能力,通过高效定殖于植物根部发挥作用。Gao等[76]在番茄种子播种之前,用枯草芽孢杆菌SWR01菌株和群集运动缺陷型菌株处理番茄种子,2周后观察根尖的定殖效果,结果发现枯草芽孢杆菌群集运动缺陷会导致细菌在番茄根部的定殖效率大大降低。细菌的这一特性为促进PGPR根部定殖提供了新途径,可以通过对枯草芽孢杆菌群集运动相关基因进行过表达,增强其群集运动能力,实现细菌对农作物根部更加高效地定殖,进而促进农作物的生长发育。
细菌作为一种有效的生物修复剂,还可被应用于环境治理过程。Khan等[77]从工业废水中分离出一株高原芽孢杆菌(Bacillus altitudinis) MT422188菌株,研究发现由鞭毛介导的细菌群集运动和游泳运动行为在铜的存在下得到了促进,此外铜作为高原芽孢杆菌MT422188菌株的一种化学引诱剂,能够促使细菌向铜离子移动。高原芽孢杆菌MT422188菌株通过群集运动和游泳运动及其对铜的趋化性,可以实现在表面及液体环境中对重金属铜的生物捕获及清除。另一项研究报道了在镉存在的情况下耐金属贪铜菌(Cupriavidus metallidurans) CH34菌株表现出显著的群集运动和趋化行为,表明耐金属贪铜菌CH34菌株是金属镉生物修复的潜在候选者[78]。类似地,多氯联苯(polychlorinated biphenyls, PCB)对假单胞菌(Pseudomonas) B4菌株具有化学引诱作用,借助假单胞菌B4菌株高水平的群集运动能力可以实现对土壤污染物PCB的生物降解[79]。
截至目前,基于细菌群集运动这一特性的相关应用研究比较匮乏,主要集中在医疗、农业和环境污染控制等方面,细菌群集运动所具有的潜在应用价值还有待继续发掘。
6 结论与展望细菌群集运动是一个相对复杂的过程,与菌群种类和生长环境关系密切,其呈现出的表现形式也相对丰富。从分子生物学角度来看,鞭毛蛋白的表达量、胞内c-di-GMP的含量以及群体感应信号分子的合成等都会对群集运动产生一定的影响,在菌落中复杂地调控着细菌集体行为。在生物物理学方面,群集运动细胞与单个游泳运动细胞和生长细胞相比具有很大差异,其独特的物理性质表现有益于菌落整体的扩张。细菌从周围的生长环境中获取营养和水分,不同的碳源和氮源等营养因素会导致胞内c-di-GMP水平和表面活性剂鼠李糖脂产量的不同,进而影响细菌群集运动的能力。此外,环境中水分的含量也在一定程度上影响细菌的群集运动。然而,目前细菌生长所需的各种氨基酸等环境信号影响群集运动的具体分子机制尚不清楚,培养基中水分含量对群集运动所需鼠李糖脂表面活性剂分泌的作用机制也有待挖掘,基于群集运动特性的应用研究还有待开展。
在研究手段方面,目前对细菌群集运动的大部分研究还停留在定性分析的层面,未来需要利用高分辨率显微镜和单细胞追踪技术进一步对群集运动进行具有单细胞分辨率的定量研究[80-81]。近年来,细菌群集运动引起了国内研究学者的广泛关注。笔者团队发展了针对不同研究对象的细菌显微追踪技术,并结合先进的基因操控技术在单细胞水平上对细菌的群体行为比如生物被膜的形成以及细菌群集运动等开展了研究,加深了人们对生物群体行为的理解,2020年本实验室在单细胞水平上观察到黄色粘球菌对大肠杆菌的单体捕食行为,对单个细胞的长度、运动速度、偏转角度以及均方位移进行了测定,实现了对该种细菌运动行为的定量表征[82]。徐启旺课题组[83]以奇异变形杆菌为研究对象,结合生物波理论对运动活泼、具有潜在生长能力的细菌潜生体(cryptic growth cell, CGC),和运动能力弱或无、生长繁殖力旺盛的繁殖体(vegetative cell, VC)的研究来观察奇异变形杆菌迁徙生长过程中菌体形态、鞭毛数量、呼吸酶活性的变化,阐述了变形杆菌的周期性群集运动机制。吴艺林课题组[84]提出了一个噪声自推进粒子模型,他们认为群集运动鞭毛细菌中集体振荡运动的出现是一个强大的自组织过程,可能由局部相互作用介导。张何朋课题组[85]同时测量了粘质沙雷氏菌(Serratia marcescens)群集运动系统的方向场和速度场,利用实验数据确定了理论模型中的重要参数,将活性向列系统的复杂时空动力学通过微观模型定量再现。另外,通过不同学科的交叉,以自驱动胶体系统为模型来研究类群集运动行为,为进一步理解细菌细胞的群集运动提供了一个很好的思路和方向。在这个方向,国外已经有了大量的研究,比如Vicsek[86]模型、Boid模型和Couzin模型等[87]。但目前仍然缺乏可用于半固体表面上的多层高密度细菌运动行为方面的有效研究模型,这为未来活性物质理论模型的发展提供了新的方向。
随着研究技术的进步和相关模型的发展,我们希冀能够加深对细菌群集运动机制的理解,进而能够调控细菌的群集运动,为防治与其相关的细菌感染提供新的方法和手段。
| [1] |
MADUKOMA CS, LIANG PX, DIMKOVIKJ A, CHEN JX, LEE SW, CHEN DZ, SHROUT JD. Single cells exhibit differing behavioral phases during early stages of Pseudomonas aeruginosa swarming. Journal of Bacteriology, 2019, 201(19): e00184-19.
|
| [2] |
LOMINSKI I, LENDRUM AC. The mechanism of swarming of proteus. The Journal of Pathology and Bacteriology, 1947, 59(4): 688-691. DOI:10.1002/path.1700590423
|
| [3] |
TUSON HH, COPELAND MF, CAREY S, SACOTTE R, WEIBEL DB. Flagellum density regulates Proteus mirabilis swarmer cell motility in viscous environments. Journal of Bacteriology, 2013, 195(2): 368-377. DOI:10.1128/JB.01537-12
|
| [4] |
GILLIS A, DUPRES V, MAHILLON J, DUFRÊNE YF. Atomic force microscopy: a powerful tool for studying bacterial swarming motility. Micron, 2012, 43(12): 1304-1311. DOI:10.1016/j.micron.2012.01.014
|
| [5] |
CHAWLA R, FORD KM, LELE PP. Torque, but not FliL, regulates mechanosensitive flagellar motor-function. Scientific Reports, 2017, 7: 5565. DOI:10.1038/s41598-017-05521-8
|
| [6] |
KÖHLER T, CURTY LK, BARJA F, van DELDEN C, PECHÈRE JC. Swarming of Pseudomonas aeruginosa is dependent on cell-to-cell signaling and requires flagella and pili. Journal of Bacteriology, 2000, 182(21): 5990-5996. DOI:10.1128/JB.182.21.5990-5996.2000
|
| [7] |
COUZIN ID, KRAUSE J. Self-organization and collective behavior in vertebrates. Advances in the Study of Behavior, 2003, 32: 1-75.
|
| [8] |
TONER J, TU YH, RAMASWAMY S. Hydrodynamics and phases of flocks. Annals of Physics, 2005, 318(1): 170-244. DOI:10.1016/j.aop.2005.04.011
|
| [9] |
PERUANI F, DEUTSCH A, BÄR M. Nonequilibrium clustering of self-propelled rods. Physical Review E, 2006, 74(3): 030904. DOI:10.1103/PhysRevE.74.030904
|
| [10] |
HARSHEY RM. Bacterial motility on a surface: many ways to a common goal. Annual Review of Microbiology, 2003, 57: 249-273. DOI:10.1146/annurev.micro.57.030502.091014
|
| [11] |
KAISER D. Coupling cell movement to multicellular development in myxobacteria. Nature Reviews Microbiology, 2003, 1(1): 45-54. DOI:10.1038/nrmicro733
|
| [12] |
KAISER D. Bacterial swarming: a re-examination of cell-movement patterns. Current Biology, 2007, 17(14): R561-R570. DOI:10.1016/j.cub.2007.04.050
|
| [13] |
KEARNS DB. A field guide to bacterial swarming motility. Nature Reviews Microbiology, 2010, 8(9): 634-644. DOI:10.1038/nrmicro2405
|
| [14] |
ZHANG Y, DUCRET A, SHAEVITZ J, MIGNOT T. From individual cell motility to collective behaviors: insights from a prokaryote, Myxococcus xanthus. FEMS Microbiology Reviews, 2012, 36(1): 149-164. DOI:10.1111/j.1574-6976.2011.00307.x
|
| [15] |
WENSINK HH, DUNKEL J, HEIDENREICH S, DRESCHER K, GOLDSTEIN RE, LÖWEN H, YEOMANS JM. Meso-scale turbulence in living fluids. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(36): 14308-14313.
|
| [16] |
BE'ER A, HARSHEY RM. Collective motion of surfactant-producing bacteria imparts superdiffusivity to their upper surface. Biophysical Journal, 2011, 101(5): 1017-1024. DOI:10.1016/j.bpj.2011.07.019
|
| [17] |
WU YL, KAISER AD, JIANG Y, ALBER MS. Periodic reversal of direction allows Myxobacteria to swarm. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(4): 1222-1227.
|
| [18] |
HARSHEY RM, MATSUYAMA T. Dimorphic transition in Escherichia coli and Salmonella typhimurium: surface-induced differentiation into hyperflagellate swarmer cells. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(18): 8631-8635.
|
| [19] |
VERSTRAETEN N, BRAEKEN K, DEBKUMARI B, FAUVART M, FRANSAER J, VERMANT J, MICHIELS J. Living on a surface: swarming and biofilm formation. Trends in Microbiology, 2008, 16(10): 496-506. DOI:10.1016/j.tim.2008.07.004
|
| [20] |
NICKZAD A, LÉPINE F, DÉZIEL E. Quorum sensing controls swarming motility of Burkholderia glumae through regulation of rhamnolipids. PLoS One, 2015, 10(6): e0128509. DOI:10.1371/journal.pone.0128509
|
| [21] |
KAMATKAR NG, SHROUT JD. Surface hardness impairment of quorum sensing and swarming for Pseudomonas aeruginosa. PLoS One, 2011, 6(6): e20888. DOI:10.1371/journal.pone.0020888
|
| [22] |
CHUA SL, LIU Y, LI YY, TING HJ, KOHLI GS, CAI Z, SUWANCHAIKASEM P, GOH KKK, NG SP, TOLKER-NIELSEN T, YANG L, GIVSKOV M. Reduced intracellular c-di-GMP content increases expression of quorum sensing-regulated genes in Pseudomonas aeruginosa. Frontiers in Cellular and Infection Microbiology, 2017, 7: 451. DOI:10.3389/fcimb.2017.00451
|
| [23] |
WANG QF, FRYE JG, MCCLELLAND M, HARSHEY RM. Gene expression patterns during swarming in Salmonella typhimurium: genes specific to surface growth and putative new motility and pathogenicity genes. Molecular Microbiology, 2004, 52(1): 169-187. DOI:10.1111/j.1365-2958.2003.03977.x
|
| [24] |
OVERHAGE J, BAINS M, BRAZAS MD, HANCOCK REW. Swarming of Pseudomonas aeruginosa is a complex adaptation leading to increased production of virulence factors and antibiotic resistance. Journal of Bacteriology, 2008, 190(8): 2671-2679. DOI:10.1128/JB.01659-07
|
| [25] |
KOJIMA S, BLAIR DF. Solubilization and purification of the MotA/MotB complex of Escherichia coli. Biochemistry, 2004, 43(1): 26-34. DOI:10.1021/bi035405l
|
| [26] |
CASTILLO DJ, NAKAMURA S, MORIMOTO YV, CHE YS, KAMI-IKE N, KUDO S, MINAMINO T, NAMBA K. The C-terminal periplasmic domain of MotB is responsible for load-dependent control of the number of stators of the bacterial flagellar motor. B Biophysics, 2013, 9: 173-181. DOI:10.2142/biophysics.9.173
|
| [27] |
BERG HC. The rotary motor of bacterial flagella. Annual Review of Biochemistry, 2003, 72: 19-54. DOI:10.1146/annurev.biochem.72.121801.161737
|
| [28] |
COPELAND MF, FLICKINGER ST, TUSON HH, WEIBEL DB. Studying the dynamics of flagella in multicellular communities of Escherichia coli by using biarsenical dyes. Applied and Environmental Microbiology, 2010, 76(4): 1241-1250. DOI:10.1128/AEM.02153-09
|
| [29] |
WANG QF, SUZUKI A, MARICONDA S, PORWOLLIK S, HARSHEY RM. Sensing wetness: a new role for the bacterial flagellum. The EMBO Journal, 2005, 24(11): 2034-2042. DOI:10.1038/sj.emboj.7600668
|
| [30] |
MARICONDA S, WANG QF, HARSHEY RM. A mechanical role for the chemotaxis system in swarming motility. Molecular Microbiology, 2006, 60(6): 1590-1602. DOI:10.1111/j.1365-2958.2006.05208.x
|
| [31] |
KHAN IH, REESE TS, KHAN S. The cytoplasmic component of the bacterial flagellar motor. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(13): 5956-5960.
|
| [32] |
FRANCIS NR, SOSINSKY GE, THOMAS D, DEROSIER DJ. Isolation, characterization and structure of bacterial flagellar motors containing the switch complex. Journal of Molecular Biology, 1994, 235(4): 1261-1270. DOI:10.1006/jmbi.1994.1079
|
| [33] |
SARKAR MK, PAUL K, BLAIR D. Chemotaxis signaling protein CheY binds to the rotor protein FliN to control the direction of flagellar rotation in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(20): 9370-9375.
|
| [34] |
FORD KM, ANTANI JD, NAGARAJAN A, JOHNSON MM, LELE PP. Switching and torque generation in swarming E. coli. Frontiers in Microbiology, 2018, 9: 2197. DOI:10.3389/fmicb.2018.02197
|
| [35] |
BAKER AE, O'TOOLE GA. Bacteria, rev your engines: stator dynamics regulate flagellar motility. Journal of Bacteriology, 2017, 199(12): e00088.
|
| [36] |
段海荣, 魏赛金, 黎循航. 铜绿假单胞菌中鼠李糖脂生物合成的研究进展. 中国生物工程杂志, 2020, 40(9): 43-51. DUAN HR, WEI SJ, LI XH. Advances in rhamnolipid biosynthesis by Pseudomonas aeruginosa research. China Biotechnology, 2020, 40(9): 43-51 (in Chinese). DOI:10.13523/j.cb.2005024 |
| [37] |
HOSKING ER, VOGT C, BAKKER EP, MANSON MD. The Escherichia coli MotAB proton channel unplugged. Journal of Molecular Biology, 2006, 364(5): 921-937.
|
| [38] |
MORIMOTO YV, CHE YS, MINAMINO T, NAMBA K. Proton-conductivity assay of plugged and unplugged MotA/B proton channel by cytoplasmic pH luorin expressed in Salmonella. FEBS Letters, 2010, 584(6): 1268-1272. DOI:10.1016/j.febslet.2010.02.051
|
| [39] |
CHE YS, NAKAMURA S, MORIMOTO YV, KAMI-IKE N, NAMBA K, MINAMINO T. Load-sensitive coupling of proton translocation and torque generation in the bacterial flagellar motor. Molecular Microbiology, 2014, 91(1): 175-184. DOI:10.1111/mmi.12453
|
| [40] |
SHROUT JD, CHOPP DL, JUST CL, MORTEN H, MICHAEL G, PARSEK MR. The impact of quorum sensing and swarming motility on Pseudomonas aeruginosa biofilm formation is nutritionally conditional. Molecular Microbiology, 2006, 62(5): 1264-77. DOI:10.1111/j.1365-2958.2006.05421.x
|
| [41] |
KUCHMA SL, GRIFFIN EF, O'TOOLE GA. Minor pilins of the type Ⅳ pilus system participate in the negative regulation of swarming motility. Journal of Bacteriology, 2012, 194(19): 5388-5403. DOI:10.1128/JB.00899-12
|
| [42] |
KUCHMA SL, BALLOK AE, MERRITT JH, HAMMOND JH, LU W, RABINOWITZ JD, O'TOOLE GA. Cyclic-di-GMP-mediated repression of swarming motility by Pseudomonas aeruginosa: the pilY1 gene and its impact on surface-associated behaviors. Journal of Bacteriology, 2010, 192(12): 2950-2964. DOI:10.1128/JB.01642-09
|
| [43] |
YEUNG ATY, TORFS ECW, JAMSHIDI F, BAINS M, WIEGAND I, HANCOCK REW, OVERHAGE J. Swarming of Pseudomonas aeruginosa is controlled by a broad spectrum of transcriptional regulators, including MetR. Journal of Bacteriology, 2009, 191(18): 5592-5602. DOI:10.1128/JB.00157-09
|
| [44] |
ANYAN ME, AMIRI A, HARVEY CW, TIERRA G, MORALES-SOTO N, DRISCOLL CM, ALBER MS, SHROUT JD. Type Ⅳ pili interactions promote intercellular association and moderate swarming of Pseudomonas aeruginosa. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(50): 18013-18018.
|
| [45] |
BAKER AE, DIEPOLD A, KUCHMA SL, SCOTT JE, HA DG, ORAZI G, ARMITAGE JP, O'TOOLE GA. PilZ domain protein FlgZ mediates cyclic di-GMP-dependent swarming motility control in Pseudomonas aeruginosa. Journal of Bacteriology, 2016, 198(13): 1837-1846. DOI:10.1128/JB.00196-16
|
| [46] |
VALENTINI M, FILLOUX A. Biofilms and cyclic di-GMP (c-di-GMP) signaling: lessons from Pseudomonas aeruginosa and other bacteria. Journal of Biological Chemistry, 2016, 291(24): 12547-12555. DOI:10.1074/jbc.R115.711507
|
| [47] |
VALENTINI M, FILLOUX A. Multiple roles of c-di-GMP signaling in bacterial pathogenesis. Annual Review of Microbiology, 2019, 73: 387-406. DOI:10.1146/annurev-micro-020518-115555
|
| [48] |
KRASTEVA PV, GIGLIO KM, SONDERMANN H. Sensing the messenger: the diverse ways that bacteria signal through c-di-GMP. Protein Science, 2012, 21(7): 929-948. DOI:10.1002/pro.2093
|
| [49] |
KUCHMA SL, BROTHERS KM, MERRITT JH, LIBERATI NT, AUSUBEL FM, O'TOOLE GA. BifA, a cyclic-di-GMP phosphodiesterase, inversely regulates biofilm formation and swarming motility by Pseudomonas aeruginosa PA14. Journal of Bacteriology, 2007, 189(22): 8165-8178. DOI:10.1128/JB.00586-07
|
| [50] |
MERRITT JH, BROTHERS KM, KUCHMA SL, O'TOOLE GA. SadC reciprocally influences biofilm formation and swarming motility via modulation of exopolysaccharide production and flagellar function. Journal of Bacteriology, 2007, 189(22): 8154-8164. DOI:10.1128/JB.00585-07
|
| [51] |
PAUL K, NIETO V, CARLQUIST WC, BLAIR DF, HARSHEY RM. The c-di-GMP binding protein YcgR controls flagellar motor direction and speed to affect chemotaxis by a "backstop brake" mechanism. Molecular Cell, 2010, 38(1): 128-139. DOI:10.1016/j.molcel.2010.03.001
|
| [52] |
KUCHMA SL, DELALEZ NJ, FILKINS LM, SNAVELY EA, ARMITAGE JP, O'TOOLE GA. Cyclic di-GMP-mediated repression of swarming motility by Pseudomonas aeruginosa PA14 requires the MotAB stator. Journal of Bacteriology, 2015, 197(3): 420-430. DOI:10.1128/JB.02130-14
|
| [53] |
KIM W, SURETTE MG. Coordinated regulation of two independent cell-cell signaling systems and swarmer differentiation in Salmonella enterica serovar typhimurium. Journal of Bacteriology, 2006, 188(2): 431-440. DOI:10.1128/JB.188.2.431-440.2006
|
| [54] |
XAVIER KB, BASSLER BL. LuxS quorum sensing: more than just a numbers game. Current Opinion in Microbiology, 2003, 6(2): 191-197. DOI:10.1016/S1369-5274(03)00028-6
|
| [55] |
KIM J, KANG Y, CHOI O, JEONG Y, JEONG JE, LIM JY, KIM M, MOON JS, SUGA H, HWANG I. Regulation of polar flagellum genes is mediated by quorum sensing and FlhDC in Burkholderia glumae. Molecular Microbiology, 2007, 64(1): 165-179. DOI:10.1111/j.1365-2958.2007.05646.x
|
| [56] |
JECKEL H, JELLI E, HARTMANN R, SINGH PK, MOK R, TOTZ JF, VIDAKOVIC L, ECKHARDT B, DUNKEL J, DRESCHER K. Learning the space-time phase diagram of bacterial swarm expansion. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(5): 1489-1494.
|
| [57] |
ILKANAIV B, KEARNS DB, ARIEL G, BE'ER A. Effect of cell aspect ratio on swarming bacteria. Physical Review Letters, 2017, 118(15): 158002. DOI:10.1103/PhysRevLett.118.158002
|
| [58] |
RYAN SD, ARIEL G, BE'ER A. Anomalous fluctuations in the orientation and velocity of swarming bacteria. Biophysical Journal, 2016, 111(1): 247-255. DOI:10.1016/j.bpj.2016.05.043
|
| [59] |
RAMASWAMY S. The mechanics and statistics of active matter. Annual Review of Condensed Matter Physics, 2010, 1: 323-345. DOI:10.1146/annurev-conmatphys-070909-104101
|
| [60] |
BAR M, GROSSMANN R, HEIDENREICH S, PERUANI F. Self-propelled rods: insights and perspectives for active matter. Annual Review of Condensed Matter Physics, 2020, 11(1): 441-466. DOI:10.1146/annurev-conmatphys-031119-050611
|
| [61] |
BE'ER A, STRAIN SK, HERNÁNDEZ RA, BEN-JACOB E, FLORIN EL. Periodic reversals in Paenibacillus dendritiformis swarming. Journal of Bacteriology, 2013, 195(12): 2709-2717. DOI:10.1128/JB.00080-13
|
| [62] |
MEACOCK OJ, DOOSTMOHAMMADI A, FOSTER KR, YEOMANS JM, DURHAM WM. Bacteria solve the problem of crowding by moving slowly. Nature Physics, 2021, 17(2): 205-210. DOI:10.1038/s41567-020-01070-6
|
| [63] |
GIOMI L, BOWICK MJ, MISHRA P, SKNEPNEK R, CRISTINA MARCHETTI M. Defect dynamics in active nematics. Philosophical Transactions of the Royal Society A: Mathematical, Physical and Engineering Sciences, 2014, 372(2029): 20130365. DOI:10.1098/rsta.2013.0365
|
| [64] |
DOOSTMOHAMMADI A, IGNÉS-MULLOL J, YEOMANS J, SAGUÉS F. Active nematics. Nature Communications, 2018, 9(1): 3246. DOI:10.1038/s41467-018-05666-8
|
| [65] |
RICE SA, KOH KS, QUECK SY, LABBATE M, LAM KW, KJELLEBERG S. Biofilm formation and sloughing in Serratia marcescens are controlled by quorum sensing and nutrient cues. Journal of Bacteriology, 2005, 187(10): 3477-3485. DOI:10.1128/JB.187.10.3477-3485.2005
|
| [66] |
KIM W, SURETTE MG. Prevalence of surface swarming behavior in Salmonella. Journal of Bacteriology, 2005, 187(18): 6580-6583. DOI:10.1128/JB.187.18.6580-6583.2005
|
| [67] |
JAMIESON WD, PEHL MJ, GREGORY GA, ORWIN PM. Coordinated surface activities in Variovorax paradoxus EPS. BMC Microbiology, 2009, 9(1): 1-18.
|
| [68] |
FRASER GM, HUGHES C. Swarming motility. Current Opinion in Microbiology, 1999, 2(6): 630-635. DOI:10.1016/S1369-5274(99)00033-8
|
| [69] |
BERNIER SP, HA DG, KHAN W, MERRITT JH, O'TOOLE GA. Modulation of Pseudomonas aeruginosa surface-associated group behaviors by individual amino acids through c-di-GMP signaling. Research in Microbiology, 2011, 162(7): 680-688. DOI:10.1016/j.resmic.2011.04.014
|
| [70] |
PALMER KL, AYE LM, WHITELEY M. Nutritional cues control Pseudomonas aeruginosa multicellular behavior in cystic fibrosis sputum. Journal of Bacteriology, 2007, 189(22): 8079-8087. DOI:10.1128/JB.01138-07
|
| [71] |
XAVIER JB, KIM W, FOSTER KR. A molecular mechanism that stabilizes cooperative secretions in Pseudomonas aeruginosa. Molecular Microbiology, 2011, 79(1): 166-179. DOI:10.1111/j.1365-2958.2010.07436.x
|
| [72] |
KOLLARAN AM, JOGE S, KOTIAN HS, BADAL D, PRAKASH D, MISHRA A, VARMA M, SINGH V. Context-specific requirement of forty-four two-component loci in Pseudomonas aeruginosa swarming. iScience, 2019, 13: 305-317.
|
| [73] |
DECHESNE A, SMETS BF. Pseudomonad swarming motility is restricted to a narrow range of high matric water potentials. Applied and Environmental Microbiology, 2012, 78(8): 2936-2940.
|
| [74] |
YANG A, TANG WS, SI TY, TANG JX. Influence of physical effects on the swarming motility of Pseudomonas aeruginosa. Biophysical Journal, 2017, 112(7): 1462-1471.
|
| [75] |
DE A, CHEN WJ, LI H, WRIGHT JR, LAMENDELLA R, LUKIN DJ, SZYMCZAK WA, SUN K, KELLY L, GHOSH S, KEARNS DB, HE Z, JOBIN C, LUO XP, BYJU A, CHATTERJEE S, YEOH BS, VIJAY-KUMAR M, TANG JX, PRAJAPATI M, et al. Bacterial swarmers enriched during intestinal stress ameliorate damage. Gastroenterology, 2021, 161(1): 211-224.
|
| [76] |
GAO SF, WU HJ, YU XF, QIAN LM, GAO XW. Swarming motility plays the major role in migration during tomato root colonization by Bacillus subtilis SWR01. Biological Control, 2016, 98: 11-17.
|
| [77] |
KHAN M, KAMRAN M, KADI RH, HASSAN MM, ELHAKEM A, ALHAITHLOUL HAS, SOLIMAN MH, MUMTAZ MZ, ASHRAF M, SHAMIM S. Harnessing the potential of Bacillus altitudinis MT422188 for copper bioremediation. Frontiers in Microbiology, 2022, 13: 878000.
|
| [78] |
SHAMIM S, REHMAN A, QAZI MH. Swimming, swarming, twitching, and chemotactic responses of Cupriavidus metallidurans CH34 and Pseudomonas putida Mt2 in the presence of cadmium. Archives of Environmental Contamination and Toxicology, 2014, 66(3): 407-414.
|
| [79] |
GORDILLO F, CHÁVEZ FP, JEREZ CA. Motility and chemotaxis of Pseudomonas sp. B4 towards polychlorobiphenyls and chlorobenzoates. FEMS Microbiology Ecology, 2007, 60(2): 322-328.
|
| [80] |
ZHAO K, TSENG BS, BECKERMAN B, JIN F, GIBIANSKY ML, HARRISON JJ, LUIJTEN E, PARSEK MR, WONG GCL. Psl trails guide exploration and microcolony formation in Pseudomonas aeruginosa biofilms. Nature, 2013, 497(7449): 388-391.
|
| [81] |
张文超, 张静超, 赵坤. 细菌显微追踪技术在生物被膜中的应用. 生物工程学报, 2017, 33(9): 1411-1432. ZHANG WC, ZHANG JC, ZHAO K. Application of bacterial tracking techniques in biofilms. Chinese Journal of Biotechnology, 2017, 33(9): 1411-1432 (in Chinese). |
| [82] |
ZHANG WC, WANG Y, LU HN, LIU Q, WANG CD, HU W, ZHAO K. Dynamics of solitary predation by Myxococcus xanthus on Escherichia coli observed at the single-cell level. Applied and Environmental Microbiology, 2020, 86(3): e02286-19.
|
| [83] |
贾雄飞, 刘俊康, 徐启旺. 奇异变形杆菌周期性群集运动的研究. 第三军医大学学报, 2005, 27(9): 868-870. JIA XF, LIU JK, XU QW. Periodic variety in swarming motility of Proteus mirabilis. Acta Academiae Medicinae Militaris Tertiae, 2005, 27(9): 868-870 (in Chinese). |
| [84] |
CHEN C, LIU S, SHI XQ, CHATÉ H, WU YL. Weak synchronization and large-scale collective oscillation in dense bacterial suspensions. Nature, 2017, 542(7640): 210-214.
|
| [85] |
LI H, SHI XQ, HUANG MJ, CHEN X, XIAO MF, LIU CL, CHATÉ H, ZHANG HP. Data-driven quantitative modeling of bacterial active nematics. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(3): 777-785.
|
| [86] |
VICSEK T, CZIRÓK A, BEN-JACOB E, COHEN I, SHOCHET O. Novel type of phase transition in a system of self-driven particles. Physical Review Letters, 1995, 75(6): 1226-1229.
|
| [87] |
COUZIN ID, KRAUSE J, JAMES R, RUXTON GD, FRANKS NR. Collective memory and spatial sorting in animal groups. Journal of Theoretical Biology, 2002, 218(1): 1-11.
|
 2023, Vol. 39
2023, Vol. 39




