中国科学院微生物研究所、中国微生物学会主办
文章信息
- 葛祥宇, 史社坡, 王娟
- GE Xiangyu, SHI Shepo, WANG Juan
- 新型免疫抑制剂brasilicardin A的研究进展
- Advances of the novel immunosuppressant brasilicardin A
- 生物工程学报, 2023, 39(9): 3605-3614
- Chinese Journal of Biotechnology, 2023, 39(9): 3605-3614
- 10.13345/j.cjb.221006
-
文章历史
- Received: December 15, 2022
- Accepted: April 7, 2023
- Published: April 19, 2023
免疫抑制剂是一类可抑制机体异常免疫反应的药物,在器官移植排斥反应的预防和治疗、自身免疫性疾病的治疗中有着极其重要的作用。仅在器官移植方面,预计到2023年,全球器官移植免疫抑制剂市场将达到34亿美元,且全球自身免疫疾病治疗药物也处于稳定增长的趋势,预计到2025年将达到1 522亿美元。因此随着器官移植技术的不断发展以及自身免疫性疾病发病率的增长,免疫抑制剂的需求量将迅猛增长。
目前市场上主要的免疫抑制剂环孢菌素A (cyclosporin A)[1]、他克莫司(tacrolimus)[2]、雷帕霉素(rapamycin)[3]和咪唑立宾(mizoribine)[4]等均是来自于真菌或放线菌的代谢产物,微生物来源的免疫抑制剂在临床上占有重要地位。免疫抑制剂,不管是用于器官排斥反应,还是自身免疫性疾病的治疗,都需要终身用药,因此,其安全性是患者能否长期坚持用药的关键。但当前上市的各种免疫抑制剂均存在不同程度的不良反应,例如他克莫司的抗排斥作用较强,可能引起神经系统损伤、肾功能异常、发生糖尿病和各种感染特别是病毒感染的几率明显增加;环孢菌素A的肝肾毒性较大,神经病变、血压升高、牙龈增生和多毛的不良反应比较多。因此,寻找新的高效低毒的免疫抑制剂一直都是全球药物研发的热点领域。
诺卡菌属(Nocardia)是一群需氧性放线菌,其中星形诺卡菌(Nocardia asteroides)、巴西诺卡菌(Nocardia brasiliensis)和豚鼠诺卡菌(Nocardia cavae) 3种菌致病性最强。但近年来研究发现诺卡菌除致病性外,可产生多种催化功能的酶和结构新颖、生物活性高的小分子,具有巨大的制药潜力[5-9]。例如从多株巴西诺卡菌中分离得到具有抗菌作用的蒽醌类化合物SO-75R1和细胞毒性活性的蒽醌brasiliquinones A–C、吲哚生物碱brasilidine A[10-12],值得注意的是,Kobayashi课题组从一名患肺诺卡菌病的临床病人体内分离得到的巴西诺卡菌N. brasiliensis IFM 0406,除了致病性以外,从中发现的二萜糖苷类化合物brasilicardin A (BraA) (图 1)具有显著免疫抑制作用(IC50=0.057 μg/mL)[13-14]。鉴于BraA显著的免疫抑制活性及新颖的作用机制,BraA类似物的分离鉴定、衍生化合成和全合成引起全球关注。但BraA及其类似物的高效获得还存在一定的瓶颈,本文综述了BraA的分子特征、药理活性、作用机制、化学合成和生物合成基因簇,同时也阐述了近年来在异源宿主中获得的BraA类似物和衍生化方面的研究进展,增加人们对该新型强免疫抑制剂临床应用价值的理解,以期为BraA及其类似物的规模化生产提供参考。
|
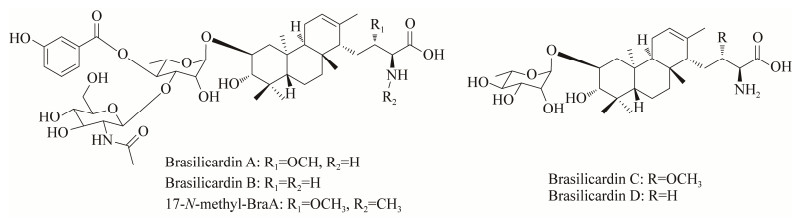
| 图 1 Brasilicardin A及其类似物结构 Fig. 1 Chemical structures of brasilicardins. |
| |
BraA化合物干粉呈白色结晶态,[α]D30+15.0 (c 0.5, MeOH)。分子式为C45H68N2O16,分子量为892。可溶于水、甲醇和乙醇,不溶于氯仿、乙酸乙酯、丙酮和乙醚。紫外光谱中,BraA的甲醇溶液在212、239和300 nm处显示最大吸收。该化合物是一种二萜糖苷类化合物,分子结构由罕见的反式/顺式/反式全氢菲母核与鼠李糖、N-乙酰氨基葡萄糖、氨基酸部分和3-羟基苯甲酯组成(图 1)[15]。
BraA类似物的分离鉴定也备受关注,Komatsu等[16]从N. brasiliensis IFM 0406分离鉴定brasilicardin B (BraB)、brasilicardin C (BraC)和brasilicardin D (BraD)这3个类似物(图 1)。从结构上来看,BraB比BraA在氨基酸侧链上少一个甲氧基,BraC比BraA少了N-乙酰氨基葡萄糖和3-羟基苯甲酯;BraD比BraB少了N-乙酰氨基葡萄糖和3-羟基苯甲酯。
2 BraA药理活性及作用机制 2.1 BraA的免疫抑制活性及作用机制利用小鼠混合淋巴细胞反应实验模型进行免疫抑制活性评价,BraA具有显著免疫抑制作用(IC50=0.057 μg/mL)。且研究发现BraA发挥免疫抑制活性的作用机制与现有临床常用的免疫抑制剂不同,BraA通过抑制氨基酸转运体L系统的转运进而影响T-淋巴细胞对氨基酸的摄入而发挥免疫抑制作用[17]。相比目前已知的免疫抑制剂环孢菌素A、子囊霉素和他克莫司等,BraA在小鼠混合淋巴细胞反应中显示低毒、高效的优势[14, 18-20]。
对BraB、BraC和BraD进行免疫抑制活性评价发现BraB、BraC具有一定的免疫抑制活性,而BraD并无此活性,提示了氨基酸侧链部分上的甲氧基及N-乙酰氨基葡萄糖和3-羟基苯甲酯对免疫抑制活性的重要性[16]。Komatsu等[21]基于BraA的衍生化修饰发现,17-N-甲基-BraA具有比BraA更强的免疫抑制活性。因此,BraA及类似物作为新型的免疫抑制剂,极具开发潜力,如能阐明该类结构的构效关系,将成为全球免疫抑制剂新领域。
2.2 BraA的抗肿瘤活性及作用机制Komaki等[22]在对BraA进行免疫抑制活性评价时,也发现其抗肿瘤活性,尤其对小鼠白细胞P388和P388/ADR细胞系表现显著活性,其IC50分别为0.220 μg/mL和0.078 μg/mL (阳性药阿霉素相应的IC50分别为0.05 μg/mL和1.00 μg/mL)。在此基础上还发现,BraA可以显著提高P388白血病小鼠的存活时间。初步研究显示BraA可在体外诱导P388细胞停滞在G1期从而发挥其抗肿瘤作用。
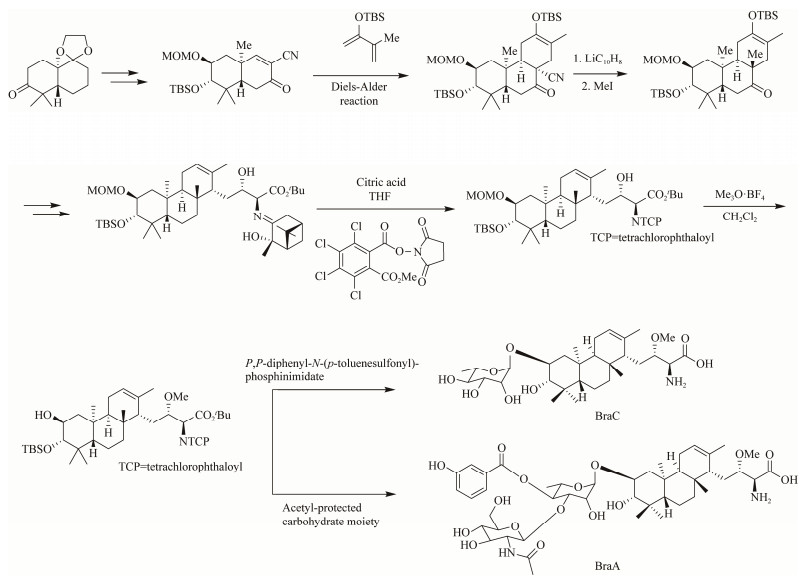
3 BraA的化学合成BraA类成分在N. brasiliensis IFM 0406中的产率极低,近百升的培养液中才能得到毫克级样品,而且N. brasiliensis IFM 0406为致病菌,大量的发酵培养势必对环境带来安全隐患。利用原菌种进行BraA及其衍生物的大规模制备,其挑战性很大,因此BraA的全合成一直受到高度关注。因BraA类成分具有独特的反式/顺式/反式稠环饱和菲及鼠李糖、葡萄糖胺等糖基化结构,化学合成BraA及其结构多样的类似物也极具挑战性。研究人员主要针对合成路线中独特二萜母核、糖苷配基等关键问题开展研究。
2003年,Coltart等[23]率先通过Wieland-Miescher酮与二烯进行Diels-Alder加成反应,再经过甲基化、脱氧的步骤,最终合成反式/顺式/反式稠环饱和菲。Jung等[24]于2013年构建了二烯/烯等当量的Diels-Alder环合反应,并成功将该反应应用到BraA的C环的合成中从而成功合成了二萜母核。
2011年Jung等[25]以鼠李糖和葡糖胺为底物,通过三氟甲磺酸三甲基硅酯介导的糖基化等9步反应,得到乙酰基保护的二糖酰亚胺酯,再以简易获得的胆固醇为苷元,通过以三氟甲磺酸三甲基硅酯介导的糖苷化而获得BraA类似物。2015年Jung等[26]从2, 7-二甲氧基萘出发,经过15步反应合成了以萘环代替反式/顺式/反式稠环饱和菲的BraA类似物,也为BraA的全合成过程中基团保护等提供参考。
在几次BraA部分合成的实例后,Anada等[27]以全氢萘酮转换的亲双烯体含氰基烯酮为底物,通过Diels-Alder加成/还原角甲基化、选择性脱保护等29步,最终实现BraA、BraC的全合成,虽然产率分别仅有2.1%和1.4%,但这是首次BraA的全合成(图 2)。
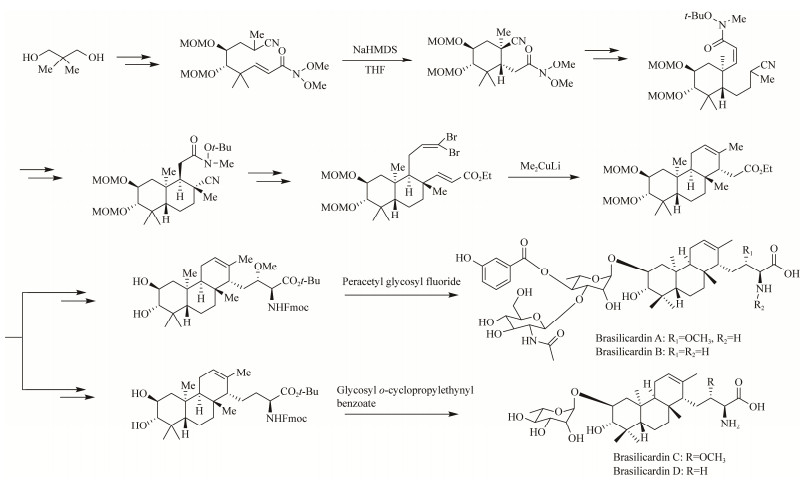
近来Yoshimura等[28]开发了一种立体选择性合成方法,基于Michael加成的立体选择性构建三元二萜母环,然后在碳环骨架上立体选择性引入氨基酸片段、以糖基化合物作为糖供体进行区域和立体选择性糖基化的分子内共轭加成反应等39步反应最终实现BraA、BraC的全合成(图 3)及BraB、BraD的首次全合成。不久之后,该课题组统一建立了brasilicardins这类特殊结构母核合成的方法,再完成糖基化和氨基酸的取代,有助于该类化合物的结构多样性[29]。
此外,Eitel等[30]以甲醇为媒介,建立特异水解BraA合成过程中全部乙酰基保护的二糖链上3-羟基苯甲酯的乙酰基的方法,使得brasilicardins合成更容易。总的来说,由于BraA及类似物的化学合成步骤多、反应条件苛刻、最终产率低(约2%)等因素,无法高效合成BraA及其类似物以用于深入研究和药物开发利用。
4 BraA生物合成机制及应用近年来,随着生物信息学的快速发展,越来越多的天然产物及相关化合物生物合成途径得以解析。鉴于BraA显著的免疫抑制活性,生物学家们在阐明BraA的生物合成机制方面也做出了不断的努力。
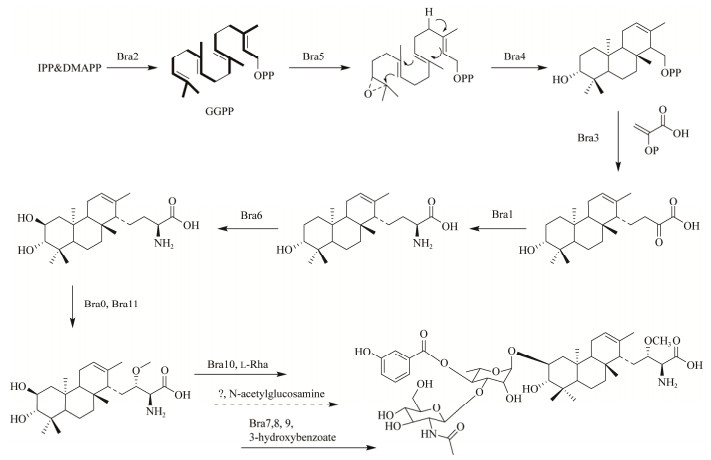
4.1 BraA的生物合成机制Kobayashi课题组通过同位素标记实验,率先证明BraA三环二萜母核在N. brasiliensis IFM 0406体内是由2-C-甲基-d-赤藓糖醇-4-磷酸(2-methyl-d-erythritol-4-phosphate, MEP)途径而来[31]。在此基础上,Dairi等[32]以合成二萜通用前体所需的香叶基香叶基焦磷酸合酶(geranylgeranyl pyrophosphate synthase, GGPPS)为探针,鉴定了与BraA生物合成相关的基因簇,包括鲨烯环氧化酶(bra5)和二萜环合酶(bra4)等在内的共11个基因(bra1–11),且基因敲除实验证实该段DNA序列确实是BraA的生物合成基因簇,并初步提出BraA的生物合成途径(图 4),但合成BraA结构中的鼠李糖和葡萄糖胺等相关基因目前尚不明确。
细菌二萜合酶(bacterial terpene synthases, TS)因结构不同而催化多样的环化反应,根据碳正离子中间体的形成方式,分为Ⅰ型合酶和Ⅱ型合酶2个亚型。Ⅰ型合酶催化香叶基香叶基焦磷酸二磷酸基团的异裂而获得碳正离子进而发生环合;Ⅱ型合酶通过氧环结构或碳碳双键质子化产生碳正离子,同时保持完整的二磷酸部分。典型Ⅰ型和Ⅱ型合酶分别具有特征结构域DDxxD、NSE/DTE和DxDD[33-35]。而BraA基因簇的环合酶Bra4属于非典型Ⅱ型合酶,与链霉菌(Streptomyces sp.) Tü6071中合成二萜phenalinolactone A的二萜合酶类似,含有E/DSAE/N结构域[36],其以环氧香叶基香叶基焦磷酸二磷酸为底物催化特殊的环化反应(图 4)。这也恰恰与Dairi课题组研究结果一致,Bra4不能接受香叶基香叶基焦磷酸为底物进行环合[32]。正是二萜合酶不同的结构和催化机制,决定了产物萜类化合物的结构多样性和活性多样性。
4.2 BraA的外源表达为顺利实现BraA的外源表达,Buchmann等[37]对产BraA原始致病菌株进行全基因组测序,有利于其在亲缘外源菌株中实现合成。2018年,Stegmann课题组率先将BraA生物合成基因簇以及来源于白色链霉菌(Streptomyces albus) J1074的合成l-鼠李糖的相关模块同时引入亲缘放线菌拟无枝酸菌(Amycolatopsis japonicum)构建生物工程菌,成功实现BraC在工程菌中的合成(302 mg/L)。同年该课题组还发现了具有催化羟基化反应的双加氧酶基因bra0,进一步完善了BraA生物合成基因簇[38-39]。Wolański等[40]还发现了除bra12以外LysR型的转录调控因子,实现BraA类似物外源表达的负向调控。BraA完整的生物合成基因簇至今仍未阐明,所幸的是,合成BraA三环二萜骨架相关的基因等已经得到验证,这为利用基因簇在其他同源微生物中外源合成BraA类似物提供重要的生物信息学基础。
4.3 BraA及类似物的联合合成随着测序技术的不断升级、生物信息学的发展,部分天然产物生物合成途径被阐明,这为利用合成生物学策略构建工程菌,并实现利用工程菌定向生产目标化合物提供了必要条件。然而,大多数天然产物的生物合成途径未知或没有完全阐明;因此,仅应用微生物合成策略来合成特定化合物仍具有挑战性。工程菌发酵与化学合成的联合应用,可以实现目标化合物的快速大量合成。例如先经过工程菌合成抗疟疾药物青蒿素的关键前体青蒿酸(产量高达25 g/L)和抗癌药物紫杉醇的前体紫杉二烯(产量为1 g/L)[41-42],再结合简单的化学合成,最终实现目标天然产物的规模化制备。
2021年Botas等[43]报道了一种微生物和化学联合合成BraA的方法[44],避免了从原始致病菌株中提取的安全问题。该课题组从70株外源表达菌株筛选到最适菌株,通过培养条件和菌株等的优化,实现了BraC和BraE产量分别为1 669 mg/L和926 mg/L的高效定向合成。然后以BraE为苷元通过简单而直接的5步合成路线,实现了BraA的快速合成。整个半合成产率为7%,而中间体重复利用的情况下,产率可达16%。这个新颖、经济可行的合成途径为BraA及类似物的获得及构效研究奠定了基础。
5 总结与展望免疫抑制剂是当前临床急需的药物,是国际创新药物发现的热点。BraA为新近发现的新型强免疫抑制剂,本文总结了BraA的结构性质、药理活性、化学合成方法、生物合成机制及应用,但目前BraA来源受限,且发现的BraA类似物数量很少,其免疫抑制作用机制尚未完全阐明,所以应继续从天然产物中寻找BraA类似物、对BraA进行结构修饰或全合成BraA类似物,结合免疫抑制活性筛选和安全性评价,以期发现具有临床应用价值的免疫抑制剂。
基因组挖掘是随着高通量测序技术和天然产物合成机制研究而发展起来的一种新的天然产物发现策略,通过微生物的基因簇信息预测其合成特定天然产物的潜能,继而进行微生物培养、代谢产物分离和结构鉴定,最终得到预测的化合物类型。近年来,利用基因组挖掘发现结构新颖、活性好的小分子已经成为国内外的研究热点,例如链霉菌天然产物holomycin、pyridomycin、chaxamycins A–D等的发现,平板素(platencin)、平板霉素(platensimycin)高产菌株的筛选[45-48]。因此,利用已知的BraA生物合成基因簇对相关的公开基因组数据进行挖掘,极有可能在同属或亲缘关系较近的菌株中发现其结构功能相似的基因簇,从而获得BraA类似物[49-50]。本课题组根据已经阐明的BraA三环二萜骨架合成的相关基因信息,对NCBI数据库中微生物基因组数据进行分析挖掘,共筛选到7株含有BraA相似基因簇的候选菌,并结合初步液相色谱质谱联用(liquid chromatograph-mass spectrometer, LC-MS)分析,发现多株非致病菌具有合成BraA类似物的潜能。
此外,基于目标分子生物合成机制的组合合成及合成生物学研究在药物研发和生产中具有非常重要的作用。一方面,在微生物体内针对生物合成模块进行合理的遗传操作或重组,建立结构复杂多样的天然产物类似物库,为发现更具有临床应用价值的药物先导化合物提供崭新的途径。另一方面,通过从非致病菌中靶向分离及在阐明活性分子生物合成机制基础上的组合合成,将会发现结构新颖多样的BraA类似物,解决强免疫抑制剂BraA及其类似物难以获得的瓶颈问题。本课题组将基因线路的模块化操作,从基因水平将不同菌株相关基因簇中合成母核模块及后修饰酶模块重组合,结合LC-MS靶向分离,获得多个BraA类似物,目前相关研究正在进一步完善中。本研究不仅为结构多样BraA类似物的组合合成提供指导,而且为新型免疫抑制剂BraA及其类似物工程菌的构建和规模化制备奠定基础,以期为BraA及其类似物的高效生产提供参考。
| [1] |
SURVASE SA, KAGLIWAL LD, ANNAPURE US, SINGHAL RS. Cyclosporin A—a review on fermentative production, downstream processing and pharmacological applications. Biotechnology Advances, 2011, 29(4): 418-435. DOI:10.1016/j.biotechadv.2011.03.004
|
| [2] |
ONG SC, GASTON RS. Thirty years of tacrolimus in clinical practice. Transplantation, 2021, 105(3): 484-495. DOI:10.1097/TP.0000000000003350
|
| [3] |
GANESH SK, SUBATHRA DEVI C. Molecular and therapeutic insights of rapamycin: a multi-faceted drug from Streptomyces hygroscopicus. Molecular Biology Reports, 2023, 50(4): 1-19.
|
| [4] |
YOKOTA S. Mizoribine: mode of action and effects in clinical use. Pediatrics International, 2002, 44(2): 196-198. DOI:10.1046/j.1328-8067.2002.01536.x
|
| [5] |
LUO Q, HIESSL S, POEHLEIN A, DANIEL R, STEINBÜCHEL A. Insights into the microbial degradation of rubber and gutta-percha by analysis of the complete genome of Nocardia nova SH22a. Applied and Environmental Microbiology, 2014, 80(13): 3895-3907. DOI:10.1128/AEM.00473-14
|
| [6] |
ENGELBRECHT A, SAAD H, GROSS H, KAYSSER L. Natural products from Nocardia and their role in pathogenicity. Microbial Physiology, 2021, 31(3): 217-232. DOI:10.1159/000516864
|
| [7] |
CHENG ZH, YU BY, CORDELL GA, QIU SX. Biotransformation of quinovic acid glycosides by microbes: direct conversion of the ursane to the oleanane triterpene skeleton by Nocardia sp. NRRL 5646. Organic Letters, 2004, 6(18): 3163-3165. DOI:10.1021/ol048787b
|
| [8] |
DHAKAL D, CHAUDHARY AK, POKHREL A, JHA AK, DARSANDHARI S, SHRESTHA B, SOHNG JK. Underpinning the secondary metabolites from Nocardia spp.. Journal of Biomolecule Reconstruction, 2013, 10(1): 9-17.
|
| [9] |
DHAKAL D, RAYAMAJHI V, MISHRA R, SOHNG JK. Bioactive molecules from Nocardia: diversity, bioactivities and biosynthesis. Journal of Industrial Microbiology and Biotechnology, 2019, 46(3/4): 385-407.
|
| [10] |
MIKAMI Y, YAZAWA K, OHASHI S, MAEDA A, AKAO M, ISHIBASHI M, KOBAYASHI J, YAMAZAKI C. SO-75R1, a new mutactimycin derivative produced by Nocardia brasiliensis. The Journal of Antibiotics, 1992, 45(6): 995-997. DOI:10.7164/antibiotics.45.995
|
| [11] |
TSUDA M, SATO H, TANAKA Y, YAZAWA K, MIKAMI Y, SASAKI T, KOBAYASHI J. Brasiliquinones A–C, new cytotoxic benz[α]anthraquinones with an ethyl group at C-3 from actinomycete Nocardia brasiliensis. Journal of the Chemical Society, Perkin Transactions 1, 1996, 15: 1773-1775.
|
| [12] |
KOBAYASHI J, TSUDA M, NEMOTO A, TANAKA Y, YAZAWA K, MIKAMI Y. Brasilidine A, a new cytotoxic isonitrile-containing indole alkaloid from the actinomycete Nocardia brasiliensis. Journal of Natural Products, 1997, 60(7): 719-720. DOI:10.1021/np970132e
|
| [13] |
KOMAKI H, NEMOTO A, TANAKA Y, YAZAWA K, TOJO T, TAKAGI H, KADOWAKI K, MIKAMI Y, SHIGEMORI H, KOBAYASHI J. Brasilicardin A; a new terpenoid antibiotic produced by Nocardia brasiliensis. Actinomycetologica, 1998, 12(2): 92-96. DOI:10.3209/saj.12_92
|
| [14] |
SHIGEMORI H, KOMAKI H, YAZAWA K, MIKAMI Y, NEMOTO A, TANAKA Y, SASAKI T, IN Y, ISHIDA T, KOBAYASHI J. Brasilicardin A. A novel tricyclic metabolite with potent immunosuppressive activity from actinomycete Nocardia brasiliensis. The Journal of Organic Chemistry, 1998, 63(20): 6900-6904. DOI:10.1021/jo9807114
|
| [15] |
KOMAKI H, NEMOTO A, TANAKA Y, TAKAGI H, YAZAWA K, MIKAMI Y, SHIGEMORI H, KOBAYASHI J, ANDO A, NAGATA Y. Brasilicardin A, a new terpenoid antibiotic from pathogenic Nocardia brasiliensis: fermentation, isolation and biological activity. The Journal of Antibiotics, 1999, 52(1): 13-19. DOI:10.7164/antibiotics.52.13
|
| [16] |
KOMATSU K, TSUDA M, SHIRO M, TANAKA Y, MIKAMI Y, KOBAYASHI J. Brasilicardins B–D, new tricyclic terpernoids from actinomycete Nocardia brasiliensis. Bioorganic & Medicinal Chemistry, 2004, 12(21): 5545-5551.
|
| [17] |
USUI T, NAGUMO Y, WATANABE A, KUBOTA T, KOMATSU K, KOBAYASHI J, OSADA H. Brasilicardin A, a natural immunosuppressant, targets amino acid transport system L. Chemistry & Biology, 2006, 13(11): 1153-1160.
|
| [18] |
RÜHLMANN A, NORDHEIM A. Effects of the immunosuppressive drugs CsA and FK506 on intracellular signalling and gene regulation. Immunobiology, 1997, 198(1/2/3): 192-206.
|
| [19] |
MILLER LW. Cardiovascular toxicities of immunosuppressive agents. American Journal of Transplantation, 2002, 2(9): 807-818. DOI:10.1034/j.1600-6143.2002.20902.x
|
| [20] |
HALLORAN PF. Immunosuppressive drugs for kidney transplantation. New England Journal of Medicine, 2004, 351(26): 2715-2729. DOI:10.1056/NEJMra033540
|
| [21] |
KOMATSU K, TSUDA M, TANAKA Y, MIKAMI Y, KOBAYASHI J. SAR studies of brasilicardin A for immunosuppressive and cytotoxic activities. Bioorganic & Medicinal Chemistry, 2005, 13(5): 1507-1513.
|
| [22] |
KOMAKI H, TANAKA Y, YAZAWA K, TAKAGI H, ANDO A, NAGATA Y, MIKAMI Y. Antitumor activity of brasilicardin A, a novel terpenoid antibiotic from Nocardia brasiliensis. The Journal of Antibiotics, 2000, 53(1): 75-77. DOI:10.7164/antibiotics.53.75
|
| [23] |
COLTART DM, DANISHEFSKY SJ. Novel synthetic approach to the 8, 10-dimethyl anti-syn-anti-perhydrophenanthrene skeleton. Organic Letters, 2003, 5(8): 1289-1292. DOI:10.1021/ol034213f
|
| [24] |
JUNG ME, PEREZ F, REGAN CF, YI SW, PERRON Q. Se-phenyl prop-2-eneselenoate: an ethylene equivalent for Diels-alder reactions. Angewandte Chemie International Edition, 2013, 52(7): 2060-2062. DOI:10.1002/anie.201208294
|
| [25] |
JUNG ME, KOCH P. An efficient synthesis of the protected carbohydrate moiety of brasilicardin A. Organic Letters, 2011, 13(14): 3710-3713. DOI:10.1021/ol2013704
|
| [26] |
JUNG ME, CHAMBERLAIN BT, KOCH P, NIAZI KR. Synthesis and bioactivity of a brasilicardin A analogue featuring a simplified core. Organic Letters, 2015, 17(14): 3608-3611.
|
| [27] |
ANADA M, HANARI T, KAKITA K, KUROSAKI Y, KATSUSE K, SUNADOI Y, JINUSHI Y, TAKEDA K, MATSUNAGA S, HASHIMOTO S. Total synthesis of brasilicardins A and C. Organic Letters, 2017, 19(20): 5581-5584.
|
| [28] |
YOSHIMURA F, ITOH R, TORIZUKA M, MORI G, TANINO K. Asymmetric total synthesis of brasilicardins. Angewandte Chemie International Edition, 2018, 57(52): 17161-17167.
|
| [29] |
YOSHIMURA F, ITOH R, TORIZUKA M, MORI G, TANINO K. Chemical synthesis of brasilicardins. Journal of Synthetic Organic Chemistry, Japan, 2020, 78(11): 1085-1093.
|
| [30] |
EITEL M, ZINAD DS, SCHOLLMEYER D, GROSS H, KOCH P. Selective mono-de-O-acetylation of the per-O-acetylated brasilicardin carbohydrate side chain. Carbohydrate Research, 2021, 504: 108312.
|
| [31] |
SHIGEMORI H, KOMAKI H, YAZAWA K, MIKAMI Y, NEMOTO A, TANAKA Y, KOBAYASHI J. Biosynthesis of diterpenoid moiety of brasilicardin A via non-mevalonate pathway in Nocardia brasiliensis. Tetrahedron Letters, 1999, 40(23): 4353-4354.
|
| [32] |
HAYASHI Y, MATSUURA N, TOSHIMA H, ITOH N, ISHIKAWA J, MIKAMI Y, DAIRI T. Cloning of the gene cluster responsible for the biosynthesis of brasilicardin A, a unique diterpenoid. The Journal of Antibiotics, 2008, 61(3): 164-174.
|
| [33] |
SMANSKI MJ, PETERSON RM, HUANG SX, SHEN B. Bacterial diterpene synthases: new opportunities for mechanistic enzymology and engineered biosynthesis. Current Opinion in Chemical Biology, 2012, 16(1/2): 132-141.
|
| [34] |
DICKSCHAT JS. Bacterial terpene cyclases. Natural Product Reports, 2016, 33(1): 87-110.
|
| [35] |
DONG LB, RUDOLF JD, DENG MR, YAN XH, SHEN B. Discovery of the tiancilactone antibiotics by genome mining of atypical bacterial type Ⅱ diterpene synthases. ChemBioChem, 2018, 19(16): 1727-1733.
|
| [36] |
DÜRR C, SCHNELL HJ, LUZHETSKYY A, MURILLO R, WEBER M, WELZEL K, VENTE A, BECHTHOLD A. Biosynthesis of the terpene phenalinolactone in Streptomyces sp. Tü6071: analysis of the gene cluster and generation of derivatives. Chemistry & Biology, 2006, 13(4): 365-377.
|
| [37] |
BUCHMANN A, EITEL M, KOCH P, SCHWARZ PN, STEGMANN E, WOHLLEBEN W, WOLAŃSKI M, KRAWIEC M, ZAKRZEWSKA-CZERWIŃSKA J, MÉNDEZ C, BOTAS A, ELENA NÚÑEZ L, MORÍS F, CORTÉS J, GROSS H. High-quality draft genome sequence of the actinobacterium Nocardia terpenica IFM 0406, producer of the immunosuppressant brasilicardins, using illumina and PacBio technologies. Genome Announcements, 2016, 4(6): e01391-16.
|
| [38] |
SCHWARZ PN, BUCHMANN A, ROLLER L, KULIK A, GROSS H, WOHLLEBEN W, STEGMANN E. The immunosuppressant brasilicardin: determination of the biosynthetic gene cluster in the heterologous host Amycolatopsis japonicum. Biotechnology Journal, 2018, 13(2): 1700527-1700538.
|
| [39] |
SCHWARZ PN, ROLLER L, KULIK A, WOHLLEBEN W, STEGMANN E. Engineering metabolic pathways in Amycolatopsis japonicum for the optimization of the precursor supply for heterologous brasilicardin congeners production. Synthetic and Systems Biotechnology, 2018, 3(1): 56-63.
|
| [40] |
WOLAŃSKI M, KRAWIEC M, SCHWARZ PN, STEGMANN E, WOHLLEBEN W, BUCHMANN A, GROSS H, EITEL M, KOCH P, BOTAS A, MÉNDEZ C, ELENA NÚÑEZ L, MORÍS F, CORTÉS J, ZAKRZEWSKA-CZERWIŃSKA J. A novel LysR-type regulator negatively affects biosynthesis of the immunosuppressant brasilicardin. Engineering in Life Sciences, 2021, 21(1/2): 4-18.
|
| [41] |
PADDON CJ, WESTFALL PJ, PITERA DJ, BENJAMIN K, FISHER K, MCPHEE D, LEAVELL MD, TAI A, MAIN A, ENG D, POLICHUK DR, TEOH KH, REED DW, TREYNOR T, LENIHAN J, FLECK M, BAJAD S, DANG G, DENGROVE D, DIOLA D, et al. High-level semi-synthetic production of the potent antimalarial artemisinin. Nature, 2013, 496(7446): 528-532.
|
| [42] |
AJIKUMAR PK, XIAO WH, TYO KEJ, WANG Y, SIMEON F, LEONARD E, MUCHA O, PHON TH, PFEIFER B, STEPHANOPOULOS G. Isoprenoid pathway optimization for taxol precursor overproduction in Escherichia coli. Science, 2010, 330(6000): 70-74.
|
| [43] |
BOTAS A, EITEL M, SCHWARZ PN, BUCHMANN A, COSTALES P, ELENA NÚÑEZ L, CORTÉS J, MORÍS F, KRAWIEC M, WOLAŃSKI M, GUST B, RODRIGUEZ M, FISCHER WN, JANDELEIT B, ZAKRZEWSKA-CZERWIŃSKA J, WOHLLEBEN W, STEGMANN E, KOCH P, MÉNDEZ C, GROSS H. Genetic engineering in combination with semi-synthesis leads to a new route for Gram-scale production of the immunosuppressive natural product brasilicardin A. Angewandte Chemie International Edition, 2021, 60(24): 13536-13541.
|
| [44] |
ZHANG H, TANG X. Combining microbial and chemical syntheses for the production of complex natural products. Chinese Journal of Natural Medicines, 2022, 20(10): 729-736.
|
| [45] |
LI B, WALSH CT. Identification of the gene cluster for the dithiolopyrrolone antibiotic holomycin in Streptomyces clavuligerus. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(46): 19731-19735.
|
| [46] |
NIKOLOULI K, MOSSIALOS D. Bioactive compounds synthesized by non-ribosomal peptide synthetases and type-Ⅰ polyketide synthases discovered through genome-mining and metagenomics. Biotechnology Letters, 2012, 34(8): 1393-1403.
|
| [47] |
SCHEFFLER RJ, COLMER S, TYNAN H, DEMAIN AL, GULLO VP. Antimicrobials, drug discovery, and genome mining. Applied Microbiology and Biotechnology, 2013, 97(3): 969-978.
|
| [48] |
HINDRA, HUANG T, YANG D, RUDOLF JD, XIE P, XIE G, TENG Q, LOHMAN JR, ZHU X, HUANG Y, ZHAO LX, JIANG Y, DUAN Y, SHEN B. Strain prioritization for natural product discovery by a high-throughput real-time PCR method. Journal of Natural Products, 2014, 77(10): 2296-2303.
|
| [49] |
CHAVALI AK, RHEE SY. Bioinformatics tools for the identification of gene clusters that biosynthesize specialized metabolites. Briefings in Bioinformatics, 2018, 19(5): 1022-1034.
|
| [50] |
CHOUDOIR MJ PEPE-RANNEY C, BUCKLEY DH. Diversification of secondary metabolite biosynthetic gene clusters coincides with lineage divergence in Streptomyces. Antibiotics, 2018, 7(1): 12.
|
 2023, Vol. 39
2023, Vol. 39