中国科学院微生物研究所、中国微生物学会主办
文章信息
- 郭士琳, 李常昆, 吕得玚, 陈骐
- GUO Shilin, LI Changkun, LV Deyang, CHEN Qi
- 基于环状RNA开发核酸药物研究进展
- Research advances in nucleic acid drugs developed based on circular RNAs
- 生物工程学报, 2024, 40(5): 1421-1430
- Chinese Journal of Biotechnology, 2024, 40(5): 1421-1430
- 10.13345/j.cjb.230810
-
文章历史
- Received: November 28, 2023
- Accepted: February 12, 2024
- Published: February 27, 2024
2. 福建省天然免疫生物学重点实验室, 福建 福州 350117
2. Fujian Key Laboratory of Innate Immune Biology, Fuzhou 350117, Fujian, China
环状RNA (circular RNA, circRNA)是在转录过程中或转录后通过反向剪接形成的一类共价闭合的单链RNA,广泛存在于多种生物体中,具有多种生物学功能,并与多种疾病的发生发展有关。环状RNA最早发现于植物类病毒中,是一种具有高热稳定性、协同性和自互补性的天然棒状结构大分子[1]。1979年,有学者首次通过电子显微镜观察到真核细胞中有环状RNA存在[2]。此后,多种环状RNA转录本相继在人源细胞、小鼠细胞和果蝇细胞中被发现[3-5]。随着高通量测序技术的迅猛发展,Salzman等发现环状RNA在真核细胞中普遍存在并且进化保守[6]。2013年,Nature杂志重磅发文,揭示了环状RNA在生物体内重要的调节功能[7-8],环状RNA的相关研究随之引起了广泛关注。环状RNA虽然缺乏真核生物翻译所需要的帽子结构,但是当添加内部核糖体进入位点(internal ribosome entry site, IRES)序列或者使用N6-甲基腺嘌呤修饰核苷酸合成的环状RNA也具有编码与表达蛋白的能力[9-10]。随着体外环化技术的发展,比mRNA多方面更具优势的环状RNA已成为核酸药物领域的新秀。
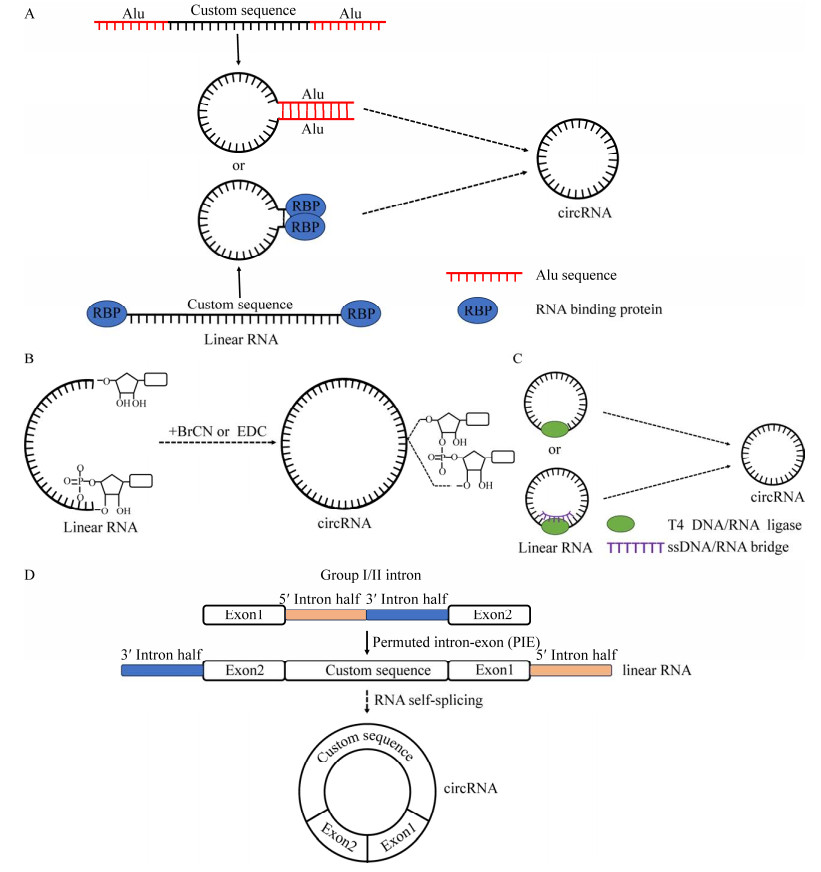
1 环状RNA合成工艺环状RNA在体内合成的方式主要是依赖Alu反向互补序列和RNA结合蛋白的反向剪接作用[11-13]。环状RNA的体外合成是通过体外转录获得线性RNA前体,随后通过多种方法使得RNA环化。目前RNA环化的方式主要有化学合成法、酶促连接法以及借助内含子与外显子重排(permuted intron-exon, PIE)策略等,每种方法各有优劣,示意图见图 1。
|
| 图 1 4种RNA环化方式 Fig. 1 Synthesis of circRNA by four major ways. A:体内合成法. B:化学法. C:酶促连接法. D:PIE法 A: In vivo RNA circularization. B: In vitro RNA circularization by chemical strategies. C: Circularization by T4 DNA/RNA ligases. D: Circularization by PIE. |
| |
化学法合成环状RNA是指利用溴化氰或1-乙基-(3-二甲基氨基丙基)碳酰二亚胺[1-(3-dimethylaminopropyl)-3-ethylcarbodiimide hydrochloride, EDC]在体外合成环状RNA,但因其连接效率低、容载片段短、毒性较大等缺点,已基本不采取这种策略[14]。
酶促连接是借助T4 DNA/RNA连接酶将线性RNA游离的两端连接起来。因为T4 DNA/RNA连接酶只能识别线性RNA分子5′端的单磷酸基团[15-17],在体外转录产生线性RNA后,还需使用焦磷酸酶或限制性内切酶处理后才可连接。在不使用夹板序列辅助连接时,会出现RNA分子间连接的副产物,而使用夹板序列辅助连接时,在连接位点处往往会出现碱基残留,其连接效率也会受夹板序列的变化而改变[16]。T4连接酶环化策略因为相对低效的连接效率和比较昂贵的成本,很难实现工业化制备。
PIE策略是基于内含子的自剪接作用,重排RNA序列使其自剪接环化,又可细分为基于Ⅰ类内含子的PIE策略和基于Ⅱ类内含子的PIE策略。此方案不需要在反应体系中添加蛋白酶,但是需要提供鸟苷酸为Ⅰ类内含子的自剪接反应提供底物,以及较高温度的反应体系[18-19]。遗憾的是此种方法合成的环状RNA也会存在几十甚至上百个碱基的外显子序列残留,有研究表明残留的外源序列可能会引起生物体内的免疫反应[20]。2022年,苏州科锐迈德生物医药科技有限公司在基于Ⅰ类内含子的PIE策略的基础上研发了Clean-PIE成环策略,该策略的巧妙之处是可以通过筛选蛋白编码区或者IRES序列找到最优的成环位点,实现了无外源序列引入的环化方案,而且环化效率也更高[21]。而基于Ⅱ类内含子的PIE策略中经过筛选优化后的Ⅱ类内含子可以使RNA高效自剪接成环且无外显子序列残留,而且该反应无需额外提供鸟苷酸和高温亦可进行[22-23],这种方案简化了环状RNA的成环反应过程以及原料使用,降低了工业成本,是理想的工业级别的环状RNA体外合成方案。
2 环状RNA纯化方案体外环化反应大多需要添加金属离子及升高温度来提升反应效率,这也会导致反应液中的RNA骨架发生随机的非特异性切割[24],从而产生副产物开环RNA和弥散产物。目前大规模纯化环状RNA的方式多采用蛋白酶处理与高效液相色谱技术(high performance liquid chromatography, HPLC)联用的方法。RNase R来源于大肠杆菌RNR超家族,可以从3′端到5′端方向切割降解RNA,能够降解几乎所有的线性RNA分子,但不易降解环状RNA、套索结构或3′末端少于7 nt的双链RNA分子[25]。磷酸酶可以消除体外转录后线性RNA分子5′端的焦磷酸基团。使用RNase R和磷酸酶处理环化反应后的RNA混合物,能有效除去混合物中的线性RNA,再使用高效液相色谱仪辅以排阻色谱柱可以将RNA碎片、开环RNA、双链RNA、残留蛋白等杂质和副产物与环状RNA分离[19, 26-27],获得的环状RNA,可通过冷冻干燥或沉淀法浓缩保存。
3 环状RNA鉴定方式环状RNA与其线性形式相对分子质量、碱基分布等基本一致,仅空间结构有所差别。环状RNA的形成主要是通过使用发散引物(divergent primers)进行RT-PCR反应后使用Sanger测序加以鉴定[7, 19];而RNase R和RNase H的消化反应,可以用来评估形成的环状RNA的纯度[19]。通过分析Urea PAGE凝胶电泳中线性RNA带和环状RNA带的灰度值,可以基本确定该反应的连接效率[28]。有研究表明,ThermoFisher Scientific公司推出的E-Gel EX电泳系统中,环状RNA的迁移速率低于前体RNA以及开环RNA,但是迁移速率也会受上样缓冲液、染料、电压等因素影响,因此还需要联合Northern blotting、RNase酶切反应和高效液相色谱分析做确认分析[29]。
4 环状RNA可表达蛋白在真核生物中,mRNA 5′端甲基化的帽子结构被认为是翻译起始必要的元件[30],环状RNA与mRNA相比,虽然缺少翻译所需要的帽子结构,但在1995年有学者发现通过添加某些病毒的IRES序列可以介导环状RNA翻译蛋白[9]。在2017年,另有学者发现使用N6-甲基腺嘌呤修饰核苷酸的环状RNA也可以结合核糖体从而驱动环状RNA的翻译[10]。但是有学者发现使用N1-甲基假尿苷替换尿嘧啶会抑制Ⅰ类内含子的剪切活性,阻断环化反应的发生,也会使环状RNA丧失蛋白表达的能力[31]。2018年Wesselhoeft等[19]在体外合成的环状RNA可以在真核细胞中高效表达蛋白,且表达量和表达周期均优于相同序列的线性RNA。2022年,北京大学的魏文胜教授团队首次研发出针对新冠肺炎病毒的环状RNA疫苗,该环状RNA在动物体内表达新冠抗原可以有效引起细胞免疫及体液免疫,从而为宿主提供广谱保护;且保存在4、25、37 ℃的环境下,环状RNA疫苗表达抗原蛋白的能力均优于同类mRNA疫苗[26],这也进一步表明了环状RNA的稳定性要优于线性RNA,基于环状RNA开发的疫苗相比mRNA疫苗在运输费用和储藏费用上都有着极大优势。同年,斯坦福大学的Chen等[32]通过优化环状RNA的拓扑结构、非编码区、内部核糖体进入位点序列和核酸适配体等,极大提高了环状RNA在真核细胞中表达蛋白的效率,为环状RNA核酸药物替代蛋白药物提供了可能。
5 生物信息学的作用稳定的二级结构会提升RNA分子在胞质中的稳定性[15],与mRNA相似,环状RNA的碱基序列和二级结构也会影响编码蛋白的能力,因此可以通过优化RNA序列来增强环状RNA的稳定性、成环效率与表达蛋白的效率。2020年,百度团队针对mRNA折叠稳定性和密码子两个方面,对mRNA序列最小自由能(minimum free energy, MFE)和密码子适应指数(codon adaptation index, CAI)进行优化,仅需几分钟就可以预测到既稳定又能够高效翻译的mRNA序列[33]。2023年7月,中国药科大学张亮教授和Coderna.AI创始人黄亮教授在“LinearDesign”的基础上,进一步推出了针对环状RNA结构预测与序列设计算法平台“circDesign”,该算法除了对MFE和CAI进行优化外,还评估了开放阅读框对IRES折叠的影响。动物实验表明,基于该算法设计的两款环状RNA疫苗在小鼠体内的稳定性和翻译效率均有提升[34]。
IRES序列是环状RNA翻译蛋白质的关键调节元件,此前研究的IRES序列多是从病毒基因组中发掘[9, 32]。2023年,浙江大学智能创新药物研究院周展教授团队研发了一个专门用于预测环状RNA的IRES序列工具“DeepCIP”[35],这将更好地帮助我们研究环状RNA的编码潜力以及提升环状RNA药物的设计能力。这些生物信息学软件和工具对环状RNA核酸药物开发都起到重要的推动作用,对挖掘药物潜力、节约药物研发时间、降低开发成本、提高药物开发成功率等都有重要意义。
6 环状RNA的安全性环状RNA安全性一直存在着争议。2017年,Chen等[36]最早报道使用Ⅰ类内含子环化,RNase R和磷酸酶纯化的环状RNA会引发强烈的天然免疫反应。2019年,Wesselhoeft等[36]通过RNase R处理、磷酸酶处理及HPLC纯化得到的同样是基于Ⅰ类内含子体外合成环状RNA却不会触发机体产生天然免疫反应。同年,Chen等[37]又发文称外源的环状RNA进入哺乳动物体内会引发天然免疫反应,使用N6-甲基腺嘌呤修饰核苷酸会降低环状RNA的免疫原性。2022年,中国科学院分子细胞科学卓越创新中心的陈玲玲教授团队报道通过Ⅰ类内含子自我剪接产生的环状RNA因其引入外源序列或产生双链RNA结构具有免疫原性,但是通过T4连接酶产生的环状RNA不具有免疫原性[20]。另外,体外转录产物通常包含部分不完整转录物及双链RNA。当双链RNA进入细胞时,受体细胞会将其感知为入侵病毒而引起细胞因子风暴,这也是环状RNA引起免疫原性的另一重要原因。对此,Moderna公司研究团队设计开发了一种新型T7 RNA聚合酶,相比野生型T7 RNA聚合酶,突变体可以简化体外转录的生产过程,在不降低产量的情况下,显著减少免疫刺激性副产物双链RNA的产生,进一步降低了环状RNA药物激发免疫的可能[38]。
此外,2022年,苏州大学支巧明团队发现由内源基因衍生的环状RNA与miRNA结合后会异常激活下游通路而导致炎症加重,并且可能引起癌症发生[39]。无独有偶,2023年6月,澳大利亚弗林德斯大学的Qadir等[40]发现部分内源环状RNA可能会与染色体结合形成circR-loops结构导致转录暂停、染色质易位和DNA修复异常,从而参与癌症的发生。因此,环状RNA的临床应用可能存在安全风险。
7 环状RNA药物的递送递送系统是保证核酸药物有效性和安全性的关键,其在保护RNA结构、增强靶向能力、热稳定性和减少不良反应等方面起到重要作用。最早各种病毒载体被应用于在生物体内递送核酸药物,但因其潜在的生物安全问题且难以大规模制备等因素,现已不再是核酸药物体内递送的首选[41]。相比之下,类病毒载体因其毒性低、免疫反应低、载体容量大、材料来源广泛、易于大量制备且所携带的基因不整合至宿主细胞基因组等优势在递送领域受到强烈关注[42-43]。在目前进入临床阶段的药物管线中,脂质纳米颗粒(lipid nanoparticle, LNP)是核酸药物最常应用的递送包裹材料。脂质纳米颗粒对大多数细胞几乎没有毒性,但在LNP配方中使用聚乙二醇可能导致过敏反应[44-45]。另外临床研究表明LNP具有肝外组织靶向传递的特异性[46-47],尽管有学者已研发出能够靶向其他组织的新型LNP[48-49],依然迫切需要能够将环状RNA靶向递送且无毒副作用的新型载体。
在此背景下,细胞来源的囊泡结构,如外泌体(exosomes),因其良好的生物相容性、细胞特异性和跨越生理屏障功能,在核酸药物递送领域受到越来越多的关注。近年,东南大学姚红红教授团队发现从外泌体中分离得到的EV-circSCMH1能够有效促进缺血性脑卒中模型小鼠与恒河猕猴脑血管修复[50-51]。虽然外泌体目前还不能大规模应用于体外转录环状RNA的递送,但其在递送领域仍有巨大潜力。
8 环状RNA药物的应用现状和未来趋势环状RNA相对于线性RNA的显著稳定性和低免疫原性,其翻译蛋白质和提供调节功能的能力使这种分子具有巨大的潜在应用价值。2018年,Wesselhoeft等[19]通过优化PIE系统和筛选IRES序列,实现了环状RNA在哺乳动物细胞内高效稳定表达蛋白质。该文的通讯作者Anderson教授也基于该项技术创立了世界首家利用环状RNA开发新疗法的公司Orna Therapeutics,并在第25届美国基因与细胞治疗学会年会上公布了其已经成功构建了能够编码杜氏肌营养不良症(Duchenne muscular dystrophy, DMD)所缺乏的肌营养不良蛋白的长达12 000 nt的超长环状RNA及可以在体内编码抗原嵌合受体的环状RNA,为实现生物体内原位CAR-T细胞疗法提供了可能性。2022年,北京大学魏文胜团队制备得到的环状RNA可以表达针对SARS-CoV-2的中和抗体并可以在小鼠和恒河猴中引发特异性免疫反应[26]。2022年,清华大学林欣和喻国灿团队设计了编码肿瘤抗原的环状RNA疫苗,在动物模型中有优秀的抗肿瘤疗效[52]。同年,汉诺威医学院Lu等[53]发文称人工制备的环状RNA模拟物Circ-INSR可预防和逆转阿霉素介导的心肌细胞死亡,并改善心脏功能。
环状RNA作为一种新兴生物学分子,在核酸药物领域已经崭露头角,除了直接发挥环状RNA的调节作用,还可长效表达蛋白,替代蛋白药物,并且在RNA疫苗、体内原位CAR-T细胞疗法等领域也有建树。体外合成环状RNA技术将成为科研与工业领域的热门,目前国内外已建立了Orna Therapeutics、Chimerna Therapeutics、Laronde、Circio、Circode (环码生物)、Therorna (圆因生物)、吉赛生物、科锐迈德、耀海生物等程序化环状RNA药物研发平台。随着研发模式和工程体系的完善,待解决安全性及靶向递送的难题后,环状RNA会在核酸药物领域发挥更大作用。
9 讨论与展望核酸药物因其开发周期短、药物成本低、药效显著成为临床用药和新药研发的热点。其中,环状RNA作为一类携带遗传信息的生物大分子,既可以发挥其内在功能调控转录与基因表达,也可以通过表达蛋白来治疗因某些蛋白质功能丧失而引起的疾病。环状RNA的高稳定性、低免疫原性、便于大规模合成和纯化、不易在细胞中引起基因突变等优点,使其具有巨大的医学应用潜力。与mRNA药物相比,环状RNA药物有着诸多优势:一方面,环状RNA是共价闭合的环状结构,没有游离的末端,因此对RNA外切酶的耐受程度更高,使其稳定性更高,其半衰期及蛋白表达周期也更长,一次给药即可发挥长效作用;另一方面,体外合成环状RNA不需要加帽酶、加尾及使用假尿苷修饰即可表达蛋白且免疫原性较低,其药物成本也相对低廉,环状RNA与mRNA药物的对比情况见表 1。近年来,随着研究的深入,环状RNA也暴露出一些问题:(1) 部分内源环状RNA因含有与基因组DNA高度互补的序列,会与染色体结合导致DNA双链打开,单链DNA更易发生断裂。基因组DNA断裂后在环状RNA的干扰下会产生错误修复,导致产生致癌基因及染色体易位的情况。体外构建环状RNA药物也应充分考虑该潜在风险。(2) 环状RNA由于其独特的空间结构,并非所有蛋白序列都可以很好地在细胞中得到表达。为适应蛋白翻译需要,需要对其IRES序列进行高通量筛选,导致药物开发难度增加。(3) 在合成环状RNA的过程中,机械应力和金属离子可能导致RNA环状结构开环,降低了目的产物得率,增加了纯化难度及引发免疫的可能性。(4) 目前大规模纯化环状RNA主要是通过液相色谱层析技术,在规模和纯度上具有一定的局限性。(5) 目前国际上关于环状RNA还没有统一的质量标准,主要参考mRNA药物的标准进行评估,不利于相关药物研发。(6) 环状RNA的递送主要仍依托于LNP,具有一定的局限性,而细胞外囊泡等新型递送载体仍需深入研究。
| Item | circRNA | mRNA | References |
| Preparation | IVT, circularization | IVT, cap and poly(A) tail design | [20] |
| Purification | RNase R digestion, SEC-HPLC, IP-RP-HPLC | Affinity purification (oligo-dT resin), ion exchange chromatography | [26, 54] |
| Capping | Not required | Required | [26, 55] |
| Pseudouridine | Not required | Required | [26, 55] |
| Codon optimizations | Required | Required | [33] |
| Half-life | 108−144 h | 36−48 h | [19, 26, 31] |
| Storage | RNase free, non-cryopreservation or under fewer freezing condition | RNase free, ultra-cold condition | [26] |
| Delivery | LNP, exosomes | LNP, exosomes | [42, 51] |
综上所述,环状RNA作为一种新型的生物学分子,具有成为新一代核酸药物的潜力,但仍旧有许多挑战需要解决。继续深入研究,对于加快环状RNA的应用具有重要作用。
| [1] |
SANGER HL, KLOTZ G, RIESNER D, GROSS HJ, KLEINSCHMIDT AK. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proceedings of the National Academy of Sciences of the United States of America, 1976, 73(11): 3852-3856.
|
| [2] |
HSU MT, COCA-PRADOS M. Electron microscopic evidence for the circular form of RNA in the cytoplasm of eukaryotic cells[J]. Nature, 1979, 280: 339-340. DOI:10.1038/280339a0
|
| [3] |
NIGRO JM, CHO KR, FEARON ER, KERN SE, RUPPERT JM, OLINER JD, KINZLER KW, VOGELSTEIN B. Scrambled exons[J]. Cell, 1991, 64(3): 607-613. DOI:10.1016/0092-8674(91)90244-S
|
| [4] |
CAPEL B, SWAIN A, NICOLIS S, HACKER A, WALTER M, KOOPMAN P, GOODFELLOW P, LOVELL-BADGE R. Circular transcripts of the testis-determining gene Sry in adult mouse testis[J]. Cell, 1993, 73(5): 1019-1030. DOI:10.1016/0092-8674(93)90279-Y
|
| [5] |
KOZAK M. An analysis of 5′-noncoding sequences from 699 vertebrate messenger RNAs[J]. Nucleic Acids Research, 1987, 15(20): 8125-8148. DOI:10.1093/nar/15.20.8125
|
| [6] |
SALZMAN J, GAWAD C, WANG PL, LACAYO N, BROWN PO. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J]. PLoS One, 2012, 7(2): e30733. DOI:10.1371/journal.pone.0030733
|
| [7] |
MEMCZAK S, JENS M, ELEFSINIOTI A, TORTI F, KRUEGER J, RYBAK A, MAIER L, MACKOWIAK SD, GREGERSEN LH, MUNSCHAUER M, LOEWER A, ZIEBOLD U, LANDTHALER M, KOCKS C, Le NOBLE F, RAJEWSKY N. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495: 333-338. DOI:10.1038/nature11928
|
| [8] |
HANSEN TB, JENSEN TI, CLAUSEN BH, BRAMSEN JB, FINSEN B, DAMGAARD CK, KJEMS J. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495: 384-388. DOI:10.1038/nature11993
|
| [9] |
CHEN CY, SARNOW P. Initiation of protein synthesis by the eukaryotic translational apparatus on circular RNAs[J]. Science, 1995, 268(5209): 415-417. DOI:10.1126/science.7536344
|
| [10] |
YANG Y, FAN XJ, MAO MW, SONG XW, WU P, ZHANG Y, JIN YF, YANG Y, CHEN LL, WANG Y, WONG CC, XIAO XS, WANG ZF. Extensive translation of circular RNAs driven by N6-methyladenosine[J]. Cell Research, 2017, 27(5): 626-641. DOI:10.1038/cr.2017.31
|
| [11] |
WANG Y, WANG ZF. Efficient backsplicing produces translatable circular mRNAs[J]. RNA, 2015, 21(2): 172-179. DOI:10.1261/rna.048272.114
|
| [12] |
JECK WR, SORRENTINO JA, WANG K, SLEVIN MK, BURD CE, LIU JZ, MARZLUFF WF, SHARPLESS NE. Circular RNAs are abundant, conserved, and associated with ALU repeats[J]. RNA, 2013, 19(2): 141-157. DOI:10.1261/rna.035667.112
|
| [13] |
CONN SJ, PILLMAN KA, TOUBIA J, CONN VM, SALMANIDIS M, PHILLIPS CA, ROSLAN S, SCHREIBER AW, GREGORY PA, GOODALL GJ. The RNA binding protein quaking regulates formation of circRNAs[J]. Cell, 2015, 160(6): 1125-1134. DOI:10.1016/j.cell.2015.02.014
|
| [14] |
DOLINNAYA NG, SOKOLOVA NI, ASHIRBEKOVA DT, SHABAROVA ZA. The use of BrCN for assembling modified DNA duplexes and DNA-RNA hybrids; comparison with water-soluble carbodiimide[J]. Nucleic Acids Research, 1991, 19(11): 3067-3072. DOI:10.1093/nar/19.11.3067
|
| [15] |
LEPPEK K, BYEON GW, KLADWANG W, WAYMENT-STEELE HK, KERR CH, XU AF, KIM DS, TOPKAR VV, CHOE C, ROTHSCHILD D, TIU GC, WELLINGTON-OGURI R, FUJII K, SHARMA E, WATKINS AM, NICOL JJ, ROMANO J, TUNGUZ B, DIAZ F, CAI H, et al. Combinatorial optimization of mRNA structure, stability, and translation for RNA-based therapeutics[J]. Nature Communications, 2022, 13: 1536. DOI:10.1038/s41467-022-28776-w
|
| [16] |
MOORE MJ, QUERY CC. Joining of RNAs by splinted ligation[J]. Methods in Enzymology, 2000, 317: 109-123.
|
| [17] |
MOORE MJ. Joining RNA molecules with T4 DNA ligase[J]. Methods in Molecular Biology, 1999, 118: 11-19.
|
| [18] |
KRUGER K, GRABOWSKI PJ, ZAUG AJ, SANDS J, GOTTSCHLING DE, CECH TR. Self-splicing RNA: autoexcision and autocyclization of the ribosomal RNA intervening sequence of tetrahymena[J]. Cell, 1982, 31(1): 147-157. DOI:10.1016/0092-8674(82)90414-7
|
| [19] |
WESSELHOEFT RA, KOWALSKI PS, ANDERSON DG. Engineering circular RNA for potent and stable translation in eukaryotic cells[J]. Nature Communications, 2018, 9: 2629. DOI:10.1038/s41467-018-05096-6
|
| [20] |
LIU CX, GUO SK, NAN F, XU YF, YANG L, CHEN LL. RNA circles with minimized immunogenicity as potent PKR inhibitors[J]. Molecular Cell, 2022, 82(2): 420-434.e6. DOI:10.1016/j.molcel.2021.11.019
|
| [21] |
QIU Z, HOU Q, ZHAO Y, ZHU J, ZHA M, LI D, LI Y, LIU C, LI N, CAO Y, YANG J, SUN Z, ZUO C. Clean-PIE: a novel strategy for efficiently constructing precise circRNA with thoroughly minimized immunogenicity to direct potent and durable protein expression[EB/OL]. [2023-11-15] https://www.biorxiv.org/content/10.1101/2022.06.20.496777v2.
|
| [22] |
MIKHEEVA S, HAKIM-ZARGAR M, CARLSON D, JARRELL K. Use of an engineered ribozyme to produce a circular human exon[J]. Nucleic Acids Research, 1997, 25(24): 5085-5094. DOI:10.1093/nar/25.24.5085
|
| [23] |
PETKOVIC S, MÜLLER S. RNA circularization strategies in vivo and in vitro[J]. Nucleic Acids Research, 2015, 43(4): 2454-2465. DOI:10.1093/nar/gkv045
|
| [24] |
LI YF, BREAKER RR. Kinetics of RNA degradation by specific base catalysis of transesterification involving the 2′-hydroxyl group[J]. Journal of the American Chemical Society, 1999, 121(23): 5364-5372. DOI:10.1021/ja990592p
|
| [25] |
CHENG ZF, DEUTSCHER MP. Purification and characterization of the Escherichia coli exoribonuclease RNase R. Comparison with RNase Ⅱ[J]. The Journal of Biological Chemistry, 2002, 277(24): 21624-21629. DOI:10.1074/jbc.M202942200
|
| [26] |
QU L, YI ZY, SHEN Y, LIN LR, CHEN F, XU YY, WU ZG, TANG HX, ZHANG XX, TIAN F, WANG CH, XIAO X, DONG XJ, GUO L, LU SY, YANG CY, TANG C, YANG Y, YU WH, WANG JB, et al. Circular RNA vaccines against SARS-CoV-2 and emerging variants[J]. Cell, 2022, 185(10): 1728-1744.e16. DOI:10.1016/j.cell.2022.03.044
|
| [27] |
YANG JL, ZHU JF, SUN JJ, CHEN YY, DU YR, TAN YL, WU LP, ZHAI MT, WEI LX, LI N, HUANG K, HOU QB, TONG ZB, BECHTHOLD A, TIAN H, SUN ZH, ZUO CJ. Intratumoral delivered novel circular mRNA encoding cytokines for immune modulation and cancer therapy[J]. Molecular Therapy-Nucleic Acids, 2022, 30: 184-197. DOI:10.1016/j.omtn.2022.09.010
|
| [28] |
BEADUDRY D, PERREAULT JP. An efficient strategy for the synthesis of circular RNA molecules[J]. Nucleic Acids Research, 1995, 23(15): 3064-3066. DOI:10.1093/nar/23.15.3064
|
| [29] |
ABE BT, WESSELHOEFT RA, CHEN R, ANDERSON DG, CHANG HY. Circular RNA migration in agarose gel electrophoresis[J]. Molecular Cell, 2022, 82(9): 1768-1777.e3. DOI:10.1016/j.molcel.2022.03.008
|
| [30] |
MUTTACH F, MUTHMANN N, RENTMEISTER A. Synthetic mRNA capping[J]. Beilstein Journal of Organic Chemistry, 2017, 13: 2819-2832. DOI:10.3762/bjoc.13.274
|
| [31] |
WESSELHOEFT RA, KOWALSKI PS, PARKER-HALE FC, HUANG YX, BISARIA N, ANDERSON DG. RNA circularization diminishes immunogenicity and can extend translation duration in vivo[J]. Molecular Cell, 2019, 74(3): 508-520.e4. DOI:10.1016/j.molcel.2019.02.015
|
| [32] |
CHEN R, WANG SK, BELK JA, AMAYA L, LI ZJ, CARDENAS A, ABE BT, CHEN CK, WENDER PA, CHANG HY. Engineering circular RNA for enhanced protein production[J]. Nature Biotechnology, 2023, 41: 262-272. DOI:10.1038/s41587-022-01393-0
|
| [33] |
ZHANG H, ZHANG L, LIN A, XU CC, LI ZY, LIU KB, LIU BX, MA XP, ZHAO FF, JIANG HL, CHEN CX, SHEN HF, LI HW, MATHEWS DH, ZHANG YJ, HUANG L. Algorithm for optimized mRNA design improves stability and immunogenicity[J]. Nature, 2023, 621: 396-403. DOI:10.1038/s41586-023-06127-z
|
| [34] |
XU C, ZHANG L, WANG W, TANG Y, WANG Q, AN J, XU H, GE Y, ZHU H, WANG H, LI B, WANG X, QIU X, SHEN M, CHEN G, SHEN H, HUANG L, LI H. Improving the circularization efficiency, stability and translatability of circular RNA by circDesign[EB/OL]. [2023-11-15] https://www.biorxiv.org/content/10.1101/2023.07.09.548293v2.
|
| [35] |
ZHOU YX, WU JC, YAO SH, XU YL, ZHAO WB, TONG YG, ZHOU Z. DeepCIP: a multimodal deep learning method for the prediction of internal ribosome entry sites of circRNAs[J]. Computers in Biology and Medicine, 2023, 164: 107288. DOI:10.1016/j.compbiomed.2023.107288
|
| [36] |
CHEN YG, KIM MV, CHEN XQ, BATISTA PJ, AOYAMA S, WILUSZ JE, IWASAKI A, CHANG HY. Sensing self and foreign circular RNAs by intron identity[J]. Molecular Cell, 2017, 67(2): 228-238.e5. DOI:10.1016/j.molcel.2017.05.022
|
| [37] |
CHEN YG, CHEN R, AHMAD S, VERMA R, KASTURI SP, AMAYA L, BROUGHTON JP, KIM J, CADENA C, PULENDRAN B, HUR S, CHANG HY. N6-methyladenosine modification controls circular RNA immunity[J]. Molecular Cell, 2019, 76(1): 96-109.e9. DOI:10.1016/j.molcel.2019.07.016
|
| [38] |
DOUSIS A, RAVICHANDRAN K, HOBERT EM, MOORE MJ, RABIDEAU AE. An engineered T7 RNA polymerase that produces mRNA free of immunostimulatory byproducts[J]. Nature Biotechnology, 2023, 41: 560-568. DOI:10.1038/s41587-022-01525-6
|
| [39] |
WAN DW, WANG ST, XU ZH, ZAN XQ, LIU F, HAN Y, JIANG M, WU AR, ZHI QM. PRKAR2A-derived circular RNAs promote the malignant transformation of colitis and distinguish patients with colitis-associated colorectal cancer[J]. Clinical and Translational Medicine, 2022, 12(2): e683. DOI:10.1002/ctm2.683
|
| [40] |
QADIR J, WEN SY, YUAN H, YANG BB. CircRNAs regulate the crosstalk between inflammation and tumorigenesis: the bilateral association and molecular mechanisms[J]. Molecular Therapy, 2023, 31(6): 1514-1532. DOI:10.1016/j.ymthe.2022.12.005
|
| [41] |
YIN H, KANASTY RL, ELTOUKHY AA, VEGAS AJ, DORKIN JR, ANDERSON DG. Non-viral vectors for gene-based therapy[J]. Nature Reviews Genetics, 2014, 15: 541-555. DOI:10.1038/nrg3763
|
| [42] |
LING SK, YANG SQ, HU XD, YIN D, DAI Y, QIAN XQ, WANG DW, PAN XY, HONG JX, SUN XD, YANG H, PALUDAN SR, CAI YJ. Lentiviral delivery of co-packaged Cas9 mRNA and a Vegfa-targeting guide RNA prevents wet age-related macular degeneration in mice[J]. Nature Biomedical Engineering, 2021, 5: 144-156. DOI:10.1038/s41551-020-00656-y
|
| [43] |
YIN D, LING SK, WANG DW, DAI Y, JIANG H, ZHOU XJ, PALUDAN SR, HONG JX, CAI YJ. Targeting herpes simplex virus with CRISPR-Cas9 cures herpetic stromal keratitis in mice[J]. Nature Biotechnology, 2021, 39: 567-577. DOI:10.1038/s41587-020-00781-8
|
| [44] |
ZHU ZY, GAO P, HU Y, WANG JY, WANG HJ, YANG JF, HUANG LF, JI C, NI YH, FANG L. PEGylated versus non-PEGylated drugs: a cross-sectional analysis of adverse events in the FDA Adverse Event Reporting System (FAERS) database[J]. International Journal of Clinical Pharmacology and Therapeutics, 2020, 58(6): 332-342. DOI:10.5414/CP203735
|
| [45] |
CASTELLS MC, PHILLIPS EJ. Maintaining safety with SARS-CoV-2 vaccines[J]. The New England Journal of Medicine, 2021, 384(7): 643-649. DOI:10.1056/NEJMra2035343
|
| [46] |
AKINC A, QUERBES W, DE S, QIN JE, FRANK-KAMENETSKY M, JAYAPRAKASH KN, JAYARAMAN M, RAJEEV KG, CANTLEY WL, DORKIN JR, BUTLER JS, QIN LL, RACIE T, SPRAGUE A, FAVA E, ZEIGERER A, HOPE MJ, ZERIAL M, SAH DW, FITZGERALD K, et al. Targeted delivery of RNAi therapeutics with endogenous and exogenous ligand-based mechanisms[J]. Molecular Therapy, 2010, 18(7): 1357-1364. DOI:10.1038/mt.2010.85
|
| [47] |
DILLIARD SA, SIEGWART DJ. Passive, active and endogenous organ-targeted lipid and polymer nanoparticles for delivery of genetic drugs[J]. Nature Reviews Materials, 2023, 8: 282-300. DOI:10.1038/s41578-022-00529-7
|
| [48] |
CHENG Q, WEI T, FARBIAK L, JOHNSON LT, DILLIARD SA, SIEGWART DJ. Selective organ targeting (SORT) nanoparticles for tissue-specific mRNA delivery and CRISPR-Cas gene editing[J]. Nature Nanotechnology, 2020, 15(4): 313-320. DOI:10.1038/s41565-020-0669-6
|
| [49] |
QIU M, TANG Y, CHEN JJ, MURIPH R, YE ZF, HUANG CF, EVANS J, HENSKE EP, XU QB. Lung-selective mRNA delivery of synthetic lipid nanoparticles for the treatment of pulmonary lymphangioleiomyomatosis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(8): e2116271119.
|
| [50] |
YANG L, HAN B, ZHANG ZT, WANG SG, BAI Y, ZHANG Y, TANG Y, DU LL, XU L, WU FF, ZUO L, CHEN XF, LIN Y, LIU KZ, YE QQ, CHEN BL, LI B, TANG TC, WANG Y, SHEN L, et al. Extracellular vesicle-mediated delivery of circular RNA SCMH1 promotes functional recovery in rodent and nonhuman primate ischemic stroke models[J]. Circulation, 2020, 142(6): 556-574. DOI:10.1161/CIRCULATIONAHA.120.045765
|
| [51] |
LI B, XI W, BAI Y, LIU X, ZHANG Y, LI L, BIAN L, LIU CC, TANG Y, SHEN L, YANG L, GU XC, XIE J, ZHOU ZQ, WANG Y, YU XY, WANG JH, CHAO J, HAN B, YAO HH. FTO-dependent m6A modification of Plpp3 in circSCMH1-regulated vascular repair and functional recovery following stroke[J]. Nature Communications, 2023, 14: 489. DOI:10.1038/s41467-023-36008-y
|
| [52] |
LI HJ, PENG K, YANG K, MA WB, QI SL, YU XY, HE J, LIN X, YU GC. Circular RNA cancer vaccines drive immunity in hard-to-treat malignancies[J]. Theranostics, 2022, 12(14): 6422-6436. DOI:10.7150/thno.77350
|
| [53] |
LU DC, CHATTERJEE S, XIAO K, RIEDEL I, HUANG CK, COSTA A, CUSHMAN S, NEUFELDT D, RODE L, SCHMIDT A, JUCHEM M, LEONARDY J, BÜCHLER G, BLUME J, GERN OL, KALINKE U, WEN TAN WL, FOO R, VINK A, van LAAKE LW, et al. A circular RNA derived from the insulin receptor locus protects against doxorubicin-induced cardiotoxicity[J]. European Heart Journal, 2022, 43(42): 4496-4511. DOI:10.1093/eurheartj/ehac337
|
| [54] |
LEE KH, KIM S, SONG J, HAN SR, KIM JH, LEE SW. Efficient circular RNA engineering by end-to-end self-targeting and splicing reaction using Tetrahymena group Ⅰ intron ribozyme[J]. Molecular Therapy-Nucleic Acids, 2023, 33: 587-598. DOI:10.1016/j.omtn.2023.07.034
|
| [55] |
STAROSTINA EV, SHARABRIN SV, ANTROPOV DN, STEPANOV GA, SHEVELEV GY, LEMZA AE, RUDOMETOV AP, BORGOYAKOVA MB, RUDOMETOVA NB, MARCHENKO VY, DANILCHENKO NV, CHIKAEV AN, BAZHAN SI, ILYICHEV AA, KARPENKO LI. Construction and immunogenicity of modified mRNA-vaccine variants encoding influenza virus antigens[J]. Vaccines, 2021, 9(5): 452. DOI:10.3390/vaccines9050452
|
 2024, Vol. 40
2024, Vol. 40