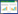中国科学院微生物研究所、中国微生物学会主办
文章信息
- 许雪晨, 王浩淼, 陈修来, 吴静, 高聪, 宋伟, 魏婉清, 刘佳, 柳亚迪, 刘立明
- XU Xuechen, WANG Haomiao, CHEN Xiulai, WU Jing, GAO Cong, SONG Wei, WEI Wanqing, LIU Jia, LIU Yadi, LIU Liming
- 代谢工程改造大肠杆菌底物利用途径促进L-赖氨酸生产
- Metabolic engineering of the substrate utilization pathway in Escherichia coli increases L-lysine production
- 生物工程学报, 2024, 40(8): 2513-2527
- Chinese Journal of Biotechnology, 2024, 40(8): 2513-2527
- 10.13345/j.cjb.240049
-
文章历史
- Received: January 17, 2024
- Accepted: March 16, 2024
2. 江南大学 生命科学与健康工程学院, 江苏 无锡 214122
2. School of Life Sciences and Health Engineering, Jiangnan University, Wuxi 214122, Jiangsu, China
L-赖氨酸作为重要的工业原料,广泛应用于食品、医药、饲料等领域[1]。近年来,随着应用研究的不断深入,对于L-赖氨酸的需求量以每年10%−15%的增长率持续增长,全球L-赖氨酸的市场规模已超过300万t[2]。为了满足全球市场对L-赖氨酸日益增长的需求,提高L-赖氨酸的生产能力成为了目前的研究热点。发酵法生产L-赖氨酸的菌种主要包括大肠杆菌(Escherichia coli)[3-4]、谷氨酸棒状杆菌(Corynebacterium glutamicum)[5-6]、黄色短杆菌(Brevibacterium flavum)[7]等。其中,大肠杆菌由于其遗传背景清晰、基因编辑手段丰富、生长周期短等特点,已成为生产L-赖氨酸的主要菌种。然而,利用大肠杆菌生产L-赖氨酸仍然需要进一步强化代谢路径以提高L-赖氨酸的底物转化率。研究人员为了进一步提高大肠杆菌中L-赖氨酸的底物转化率,开发了一系列代谢工程策略:(1) 强化L-赖氨酸合成路径基因。通过过表达L-赖氨酸合成路径上的天冬氨酸激酶基因lysC、天冬氨酸半醛脱氢酶基因asd、二氢吡啶二羧酸还原酶基因dapB等路径酶基因,使L-赖氨酸产量达到125.6 g/L[8]。(2) 增强前体草酰乙酸、天冬氨酸合成基因。通过敲除丙酮酸激酶基因pykF,使天冬氨酸积累量达到6.95 mmol/L,同时过表达磷酸烯醇式丙酮酸羧化酶基因ppc,增强草酰乙酸合成,最终使L-赖氨酸产量提高了22.22%[9-10]。(3) 关键酶蛋白质工程改造。为了解除L-赖氨酸对天冬氨酸激酶LysC的反馈抑制,通过过表达抗反馈抑制突变体LysCT344M,使L-赖氨酸产量提高至126.5 g/L,生产强度达到3.14 g/(L·h)[8, 11]。(4) 辅因子工程。通过过表达E. coli吡啶核苷酸磷酸转移酶编码基因pntAB,提高NADPH含量,将L-赖氨酸产量提高至134.9 g/L,转化率45.4%[3, 6]。以上研究策略虽然在改善底物转化率的同时显著提高了大肠杆菌的L-赖氨酸产量,但大肠杆菌仍存在对底物的摄取能力不足、底物利用不充分等问题[12],限制了转化率的进一步提高。
为此,本研究测定了E. coli XC1对不同底物的利用能力,随后,通过敲除全局调控因子基因mlc[13]、表达α-磷酸-葡萄糖苷酶基因malA和丙酮酸依赖型麦芽糖磷酸转移酶亚基基因malP,得到了E. coli XC3菌株,显著提高了L-赖氨酸生产菌株对二糖、三糖底物的利用效率。在此基础上,进一步改造XC3氮代谢路径,并引入硝酸盐同化路径,构建了L-赖氨酸生产菌E. coli XC4,显著提高了L-赖氨酸生产菌的氮源利用效率。最终,通过优化发酵过程中残糖浓度和碳氮比,使E. coli XC4菌株L-赖氨酸产量达到204.00 g/L,转化率达到72.32%,生产强度达到5.67 g/(L·h),大幅提高了L-赖氨酸生产菌株的生产性能,为L-赖氨酸的工业化生产奠定了基础。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒和引物| Plasmids and strains | Relevant characteristics | Sources |
| Plasmids | ||
| pTargetF | sgRNA | Lab store |
| pCas9 | araBAD promoter, KanR | Lab store |
| pEM | pBR322 ori, PT5, AmpR | Lab store |
| pEM-gdhA | pBR322 ori, PT5, AmpR, gdhA | This study |
| pEM-glnA | pBR322 ori, PT5, AmpR, glnA | This study |
| pEM-amtB | pBR322 ori, PT5, AmpR, amtB | This study |
| pEM-gltBD | pBR322 ori, PT5, AmpR, gltBD | This study |
| pET28a | pBR322 ori, PT7, KanR | This study |
| pET28a-1 | pBR322 ori, PT7, KanR, KoNasAC | This study |
| pET28a-2 | pBR322 ori, PT7, KanR, PpNasB | This study |
| pET28a-3 | pBR322 ori, PT7, KanR, RcNasAC | This study |
| pET28a-4 | pBR322 ori, PT7, KanR, BsNasBC | This study |
| pET28a-5 | pBR322 ori, PT7, KanR, PsNasC | This study |
| pET28a-6 | pBR322 ori, PT7, KanR, BmNasBC | This study |
| pET28a-7 | pBR322 ori, PT7, KanR, RpNasA | This study |
| pET28a-8 | pBR322 ori, PT7, KanR, MgNasA | This study |
| pET28a-9 | pBR322 ori, PT7, KanR, AvNasC | This study |
| pET28a-10 | pBR322 ori, PT7, KanR, KoNirD | This study |
| pET28a-11 | pBR322 ori, PT7, KanR, PpNirBD | This study |
| pET28a-12 | pBR322 ori, PT7, KanR, RcNasB | This study |
| pET28a-13 | pBR322 ori, PT7, KanR, EcNirBD | This study |
| pET28a-14 | pBR322 ori, PT7, KanR, BsNasDE | This study |
| pET28a-15 | pBR322 ori, PT7, KanR, PsNirBD | This study |
| pET28a-16 | pBR322 ori, PT7, KanR, BmNasDE | This study |
| pET28a-17 | pBR322 ori, PT7, KanR, PdNasBG | This study |
| pET28a-18 | pBR322 ori, PT7, KanR, KpNasD | This study |
| Strains | ||
| Escherichia coli JM109 | General cloning host | TaKaRa Bio |
| E. coli M2019435 | Derivative of E. coli MG1655 for producing L-lysine | Lab store |
| E. coli XC1 | E. coli M2019435 aspC-lysC-asd-dapA-dapB | This study |
| E. coli XC2 | E. coli XC1 Δmlc | This study |
| E. coli XC3 | E. coli XC2 ΔcadA: : malAP | This study |
| E. coli XC4 | E. coli XC3 ΔgntT: : gdhA | This study |
| E. coli XC3-1 | E. coli XC3-pEM-amtB | This study |
| E. coli XC3-2 | E. coli XC3-pEM-gdhA | This study |
| E. coli XC3-3 | E. coli XC3-pEM-gltBD | This study |
| E. coli XC3-4 | E. coli XC3-pEM-glnA | This study |
| Primers | Sequences (5′→3′) | Size (bp) |
| mlc-up-F | CCTTCCTGAATACCGACAACCATCAAC | 27 |
| mlc-up-R | TGCTTCACCATAGCCTACAGAAGCATAACTTAGACTTTCAAGGT | 44 |
| mlc-down-F | TGAAAGTCTAAGTTATGCTTCTGTAGGCTATGGTGAAGCACTTC | 44 |
| mlc-down-R | CATGAACCGCTTATGGCGATGAT | 23 |
| mlc-N20-F | GGCGGAAACCAGACCGATGAGTTTTAGAGCTAGAAATAGCAAG | 43 |
| mlc-N20-R | ACTAGTATTATACCTAGGACTGAGCTAGCTGTCATCGGTCTGGTTTCCGCC | 51 |
| cadA-up-F | TGGGGCTGCGATAGAACCG | 19 |
| cadA-up-R | CCCATGTGTTGGGAGGGGCCTGGCTTGCCACTTCCCTTTTTTGC | 44 |
| cadA-down-F | GGCCCCTCCCAACACATGGGAC | 22 |
| cadA-down-R | GGCTCCTGGATGATGTTGGTAGG | 23 |
| cadA-N20-F | GGATCTTCACGATACAGAGAGTTTTAGAGCTAGAAATAGCAAG | 43 |
| cadA-N20-R | TCTCTGTATCGTGAAGATCCACTAGTATTATACCTAGGACTGAGCTAGCTG | 51 |
| malA-1 | TGAGCGTCTAGAGCCCCTG | 19 |
| malA-2 | AAATGGGGGAATTTCATTTATGATATGCGGGTTGGGCTTTG | 41 |
| malP-1 | TTTCACACAAGGAGACTGCCATGATGCAAAAAATTCAGCGCTTTGGAA | 48 |
| malP-2 | TCCCGAAAAGGAGGGCAGTCTTTATTGATTCAATATTTTTTCCACACGCTCCCG | 54 |
| nirBD-F | GCACTAGTAAAGAGGAGAAAGGATCCATGAGCAAAGTCAGACTCGCA | 47 |
| nirBD-R | CCTTTCTCCTCTTTACTAGTGCTAGCATTATACCTA | 36 |
| BsnasB-F | GTGGACAGCAAATGGGTCGCGGATCCATGAAGAAACAAAGATTAGTGTTAGCGGGA | 56 |
| BsnasB-R | GACGGGCGCATATAAGAAAGGATGACTCGAGCACCACCACCACCACCACTG | 51 |
| BsnasC-F | TGCCGCGCGGCAGCCATATGATGACTGAACGACTGCTTAGATATTTCCG | 49 |
| BsansC-R | TGGATCCTTTCTCCTCTTTAATAAGCTTTTAAATAGGTATGATTCGGACGGCGC | 54 |
| gdhA-F | CACCATCACCATCACGGATCCATGGATCAGACATATTCTCTGGAGTCATTCC | 52 |
| gdhA-R | AGGTCGACCCGGGGTACCGAGCTCTTAAATCACACCCTGCGCCAGC | 46 |
| glnA-F | GGTGCCGCGCGGCAGCCATATG ATGTCCGCTGAACACGTACTG | 43 |
| glnA-R | TTAGACGCTGTAGTACAGCTCAAACTCTCTCGAGCACCACCACCACCA | 48 |
| amtB-F | TCACCATCACCATCACGGATCCATGAAGATAGCGACGATAAAAACTGGGC | 50 |
| amtB-R | TGCAGGTCGACCCGGGGTACCGAGCTCTTACGCGTTATAGGCATTCTCGCC | 51 |
| gltBD-F | GCATCACCATCACCATCACGGATCCATGTTGTACGATAAATCCCTTGAGAGGG | 53 |
| gltBD-R | AGGTCGACCCGGGGTACCGAGCTCTTAAACTTCCAGCCAGTTCATAATACCGT | 53 |
| gntT-up-F | GTTACGCCATTCTGCAATTTGGC | 23 |
| gntT-up-R | GATTTACCTGGCCTTTCATTTGTTATGG | 28 |
| gntT-down-F | AAATGAAAGGCCAGGTAAATCTAACACTGACTGCCGGATGCG | 42 |
| gntT-down-R | CTGGGAGTGCGGGGATCTAA | 20 |
| gntT-N20-F | AGAGTACTTTAACCTGACTATGTTTTAGAGCTAGAAATAGCAAG | 44 |
| gntT-N20-R | ATAGTCAGGTTAAAGTACTCTACTAGTATTATACCTAGGACTGAGCTAGCTG | 52 |
实验中所用的酶制剂均购自宝生物工程(大连)有限公司;实验中所用的试剂盒、一步同源重组酶购自南京诺唯赞生物科技股份有限公司;PCR引物由苏州金唯智生物科技有限公司合成;其他试剂均购自国药集团化学试剂有限公司。5 L全自动发酵罐购自迪必尔生物工程(上海)有限公司。
1.1.3 培养基LB培养基(g/L):酵母粉5.00,蛋白胨10.00,NaCl 5.00。
发酵培养基:葡萄糖20.00 g/L,甜菜糖蜜30.00 g/L,玉米浆干粉10.00 g/L,甜菜碱3.00 g/L,MgSO4 1.50 g/L,FeSO4 20.00 mg/L,苏氨酸450.00 mg/L,氯化钾1.00 g/L,甲硫氨酸400.00 mg/L,生物素0.60 mg/L,使用氨水调节pH至7.20,121 ℃灭菌15 min。
1.2 方法 1.2.1 重组菌株构建基因敲除与整合采用CRISPR-Cas9技术,重组质粒的构建采用一步同源重组法进行,基因gdhA、glnA、gltBD、amtB、nirB、nirD以E. coli MG1655基因组为模板,通过PCR扩增获得。来源于枯草芽孢杆菌(Bacillus subtilis)的基因nasC、nasB、malA、malP以B. subtilis 168基因组为模板,通过PCR扩增获得。本研究所用其他基因均由苏州金唯智生物科技有限公司进行密码子优化及合成。
1.2.2 培养方法种子培养:于保藏甘油管中吸取200 μL菌液,移入装有50 mL LB培养基的三角瓶中,37 ℃、200 r/min条件下培养8 h。
摇瓶发酵:将发酵培养基中初始糖浓度调整至50 g/L,以15%接种量将种子培养液接种于装有70 mL发酵培养基的500 mL三角瓶中,37 ℃、190 r/min培养36 h,发酵过程中每4 h用氨水调整pH。
发酵罐发酵:采用5 L全自动搅拌发酵罐进行发酵,发酵初始装液量为2 L,将种子液以15%接种量(体积分数)接种至发酵培养基中,发酵温度为37 ℃,调整初始转速400 r/min,通入100%无菌干燥空气进行发酵,过程中通过调整搅拌转速及通气速率维持溶解氧浓度30%−40%,通过流加氨水将pH维持在6.70−6.80之间,通过调整硫酸铵流加速率控制氨氮浓度,通过调整葡萄糖流加速率控制残糖浓度,共发酵36 h。
1.2.3 分析检测方法转化率计算:转化率=总产酸(g)/总耗糖(g)×100%=[产酸浓度(g/L)×下罐体积(L)]/总耗糖(g)×100%。
L-赖氨酸检测:取发酵液1 mL,12 000 r/min离心2 min后取上清稀释至合理倍数,使用乙氧基亚甲基丙二酸二乙酯(diethyl ethoxymethylene malonate, DEEMM)衍生L-赖氨酸,衍生体系包括4.5 μL DEEMM、70.5 μL蒸馏水、150 μL甲醇、450 μL硼酸缓冲液。衍生后混合物经0.22 μm过滤,使用HPLC系统进行分析,使用Agilent-C18色谱柱(250 mm×4.6 mm, 5 μm),柱温35 ℃,流速1 mL/min,流动相由100%乙腈(A)和pH 4.80的25 mmol/L乙酸钠(B)组成。
细胞浓度检测:取细胞培养液,稀释至OD562为0.20−0.80之间,通过紫外分光光度计检测其在波长562 nm下吸光度。
糖浓度检测:葡萄糖采用SBA-40E生物传感分析仪检测(深圳市希尔曼生物技术有限公司),麦芽糖、潘糖、异麦芽糖采用ICS-3000离子色谱仪检测。NH4+采用E-10离子分析仪检测。
有机酸检测:HPLC法检测乙酸、乳酸、琥珀酸、酮戊二酸、柠檬酸,取发酵液2 mL,12 000 r/min离心10 min,收集上清液,使用Aminex HPX-87H (300 mm×7.8 mm, 9 μm)色谱柱进行检测,检测波长210 nm,柱温52 ℃,流动相为5 mmol/L稀硫酸。
2 结果与分析 2.1 L-赖氨酸生产菌XC1底物利用效率评估为了得到L-赖氨酸高产菌株,以实验室保藏的L-赖氨酸生产菌株E. coli M2019435为底盘,借助CRISPR基因编辑技术在基因组上增加aspC、lysC、asd、dapA、dapB的拷贝数,增强L-赖氨酸合成路径,得到突变菌株XC1。如图 1E所示,其在5 L发酵罐中菌体浓度、L-赖氨酸产量、产率和生产强度为19.20 g/L、145.00 g/L、60.25%和4.03 g/(L·h)。
|
| 图 1 不同碳氮源对菌株XC1生长性能和L-赖氨酸产量的影响 Fig. 1 Effect of different carbon and nitrogen sources on the growth performance and L-lysine production of strain XC1. A: Dry cell weight of XC1 on different carbon sources. B: Dry cell weight of XC1 on different nitrogen sources. C: L-lysine production of XC1 on different carbon sources. D: L-lysine production of XC1 on different nitrogen sources. E: Production performance of the XC1 in 5L fermentor. A:XC1菌株在不同碳源上的生长情况. B:XC1菌株在不同氮源上的生长情况. C:XC1菌株在不同碳源上的L-赖氨酸生产情况. D:XC1菌株在不同氮源上的L-赖氨酸生产情况. E:5 L发酵罐中XC1菌株的生产指标 |
| |
如表 3所示,工业葡萄糖原料除了含有97.498%葡萄糖外,还含有一定的麦芽糖、潘糖、异麦芽糖等二糖和三糖成分。甜菜糖蜜中则含有约50%的蔗糖及约1%的果糖[14]。以不同二糖和三糖为唯一碳源,菌株XC1的生长性能和L-赖氨酸产量如图 1A、1C所示。菌株XC1在2.00 g/L葡萄糖、蔗糖、麦芽糖、潘糖、异麦芽糖、果糖为唯一碳源时的最大细胞干重(dry cell weight, DCW)分别为0.91、0.38、0.15、0.05、0.05、0.35 g/L;L-赖氨酸产量分别为1.17、0.75、0.33、0、0、0.32 g/L。这一结果表明:(1) 菌株XC1都能利用二糖和三糖进行生长;(2) 但与以葡萄糖为碳源进行比较,以蔗糖、麦芽糖、果糖为碳源时菌株XC1生长性能分别降低了58.20%、83.50%、61.50%,而L-赖氨酸产量分别降低了35.80%、71.70%和72.60%,表明菌株XC1利用蔗糖、麦芽糖、果糖的能力相对较差;(3) 以潘糖、异麦芽糖为碳源时,菌株XC1生长停滞,几乎不产L-赖氨酸,表明菌株XC1不能利用异麦芽糖和潘糖。
| Component | Proportion (%) |
| Glucose | 97.498 |
| Maltose | 0.939 |
| Panose | 0.550 |
| Isomaltose | 0.944 |
| Other | 0.069 |
菌株XC1对不同氮源的利用能力如图 1B、1D所示,在分别以5.00 g/L尿嘧啶、(NH4)2SO4、NaNO2、NaNO3、尿素为唯一氮源时,菌株XC1的DCW分别为0.56、0.45、0.28、0.24、0.15 g/L;L-赖氨酸产量分别为1.17、3.78、0.43、0.46、0.32 g/L。其中,以(NH4)2SO4为氮源时,菌株XC1的DCW分别比以尿嘧啶、NaNO2、NaNO3、尿素为氮源低24.44%、高96.00%、高133.33%和高273.33%,L-赖氨酸产量高223.08%、778.34%、720.32%和1 071.25%。这一结果表明,菌株XC1对NH4+形式的氮源具有较高的利用效率,而对NO2−和NO3−形式的氮源利用效率较差。
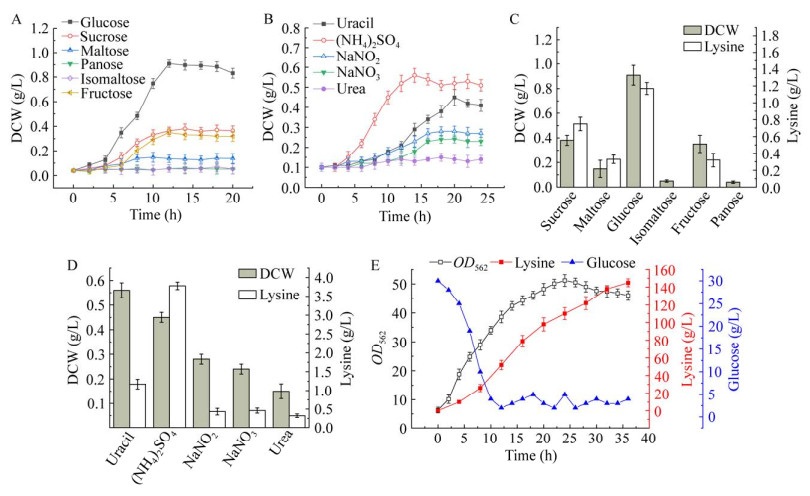
2.2 代谢工程改造菌株XC1提高碳源利用效率为了提高菌株XC1对麦芽糖的利用能力,通过敲除转录抑制因子基因mlc,解除mlc对麦芽糖操纵子的阻遏调控[15],获得工程菌株XC2 (XC1-Δmlc)。如图 2A、2D所示,与菌株XC1相比,工程菌株XC2最大DCW、麦芽糖消耗速率比菌株XC1分别提高了73.33% (0.15 g/L提高到0.26 g/L)和4.43倍[15.50 mg/(L·h)提高到68.75 mg/(L·h)]。为了进一步提高菌株对异麦芽糖及潘糖的利用,在工程菌株XC2的赖氨酸脱羧酶2的cadA位点上整合表达来源于B. subtilis的α-磷酸-葡萄糖苷酶基因malA和丙酮酸依赖型麦芽糖磷酸转移酶亚基基因malP[16],获得菌株XC3 (XC2-ΔcadA: : malAP)。在以异麦芽糖为唯一碳源时(图 2B、2E),工程菌株XC3最大DCW和异麦芽糖消耗速率分别提高到0.18 g/L和28.75 mg/(L·h)。以潘糖为唯一碳源时(图 2C、2F),工程菌株XC3最大DCW和潘糖消耗速率分别提高到0.13 g/L和15.04 mg/(L·h)。将工程菌株XC3于5 L发酵罐中进行L-赖氨酸发酵生产,结果如图 2G所示,工程菌株XC3的L-赖氨酸产量、转化率和生产强度为160.00 g/L、63.78%和4.44 g/(L·h),比菌株XC1提高了10.34%、5.86%和10.17%。上述结果表明,通过改造菌株XC1的碳底物利用途径,L-赖氨酸生产菌株对麦芽糖、异麦芽糖、潘糖等二糖和三糖的利用效率显著提高,同时也提高了L-赖氨酸糖酸转化率和生产强度。
|
| 图 2 不同菌株对麦芽糖、异麦芽糖、潘糖的利用能力比较 Fig. 2 Comparison of the utilization abilities of maltose, isomaltose and panose by different strains. A: Growth of XC1 and XC2 on maltose culture medium. B: Growth of XC1, XC2 and XC3 on isomaltose culture medium. C: Growth of XC1, XC2 and XC3 on panose culture medium. D: Maltose consumption rate of recombinant strains. E: Isomaltose consumption rate of recombinant strains. F: Panose consumption rate of recombinant strains. G: Comparison of L-lysine production and yield in recombinant strains. A:XC1和XC2菌株在麦芽糖培养基上的生长情况. B:XC1、XC2和XC3菌株在异麦芽糖培养基上的生长情况. C:XC1、XC2和XC3菌株在潘糖培养基上的生长情况. D:各重组菌株的麦芽糖消耗速率. E:各重组菌株的异麦芽糖消耗速率. F:各重组菌株的潘糖消耗速率. G:重组菌株间L-赖氨酸产量及转化率比较 |
| |
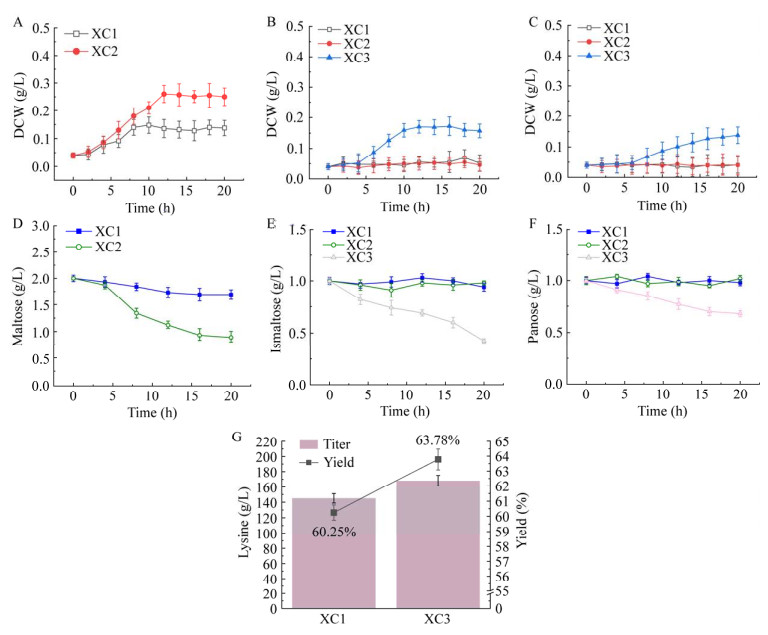
L-赖氨酸分子结构中含有2个氨基,每生成1 mol赖氨酸需要2 mol NH4+参与[3],因此,氮源供应对进一步提高L-赖氨酸合成效率具有重要意义。为了提高工程菌株XC3对NH4+的利用效率,在菌株XC3中分别过表达铵转运蛋白AmtB (amtB编码)、谷氨酸脱氢酶(glutamate dehydrogenase, GDH) (gdhA编码)、谷氨酸合成酶GOGAT (gltBD编码)和谷氨酰胺合成酶(glutamine synthetase, GS) (glnA编码)[17-19],得到重组菌株XC3-1、XC3-2、XC3-3、XC3-4,实验结果如图 3B所示:(1) 在菌株XC3中过表达4种氮代谢基因均能提升菌株生长性能,其中菌株XC3-2 (过表达gdhA)的DCW达到13.20 g/L,比菌株XC3、XC3-1 (过表达amtB)、XC3-3 (过表达gltBD)和XC3-4 (过表达glnA)分别提高17.80%、17.60%、11.40%和6.40%;(2) 摇瓶发酵中菌株XC3-2 L-赖氨酸产量达到38.20 g/L,比菌株XC3、XC3-1、XC3-3和XC3-4分别提高了47.50%、25.00%、48.02%和10.96%。这一结果表明,通过强化谷氨酸脱氢酶增强NH4+进入大肠杆菌代谢路径的效率,能有效地提高菌株的生长性能和L-赖氨酸合成能力。
|
| 图 3 强化无机氮源利用路径的构建与评估 Fig. 3 Contruction and evaluation for enhancing the utilization of inorganic nitrogen sources. A: Schematic diagram of nitrate assimilation pathway. B: Growth and production situation of recombinant strains. C: Comparison of nitrate reductase activities. D: Comparison of nitrite reductase activities. E: Comparison of NH4+ production by various enzyme combinations. F: Comparison of growth of XC4 in culture media with different concentrations of nitrate. N.D. represents not detected. A:硝酸盐同化路径示意图. B:重组菌株生产性能比较. C:硝酸盐还原酶酶活比较. D:亚硝酸盐还原酶酶活比较. E:不同硝酸盐同化路径酶组合的NH4+生产情况. F:XC4菌株在不同浓度硝酸盐中的生长情况 |
| |
甜菜糖蜜作为L-赖氨酸发酵的重要原料,含有3%−6%硝酸盐,是潜在的可利用氮源[20]。为了提高L-赖氨酸生产菌株对硝酸盐的利用率,在菌株XC3-2中构建硝酸盐同化路径[21],促使硝酸盐经亚硝酸盐转化成NH4+而被菌株所利用(图 3A)。这一硝酸盐利用途径需要硝酸盐还原酶(nitrate reductase, NAS)和亚硝酸盐还原酶(nitrite reductase, NIR)[22],借助BRENDA数据库(https://www.brenda-enzymes.org/)和KEGG数据库(https://www.kegg.jp/),发现来源于B. subtilis的BsNasBC和E. coli的EcNirBD比酶活最高,为2.160 U/mg和3.540 U/mg (表 4、表 5,图 3C、3D)。将催化两步反应的酶进行两两组合,通过测定NH4+生成量评估催化能力。结果表明,BsNasBC和EcNirBD与2 mmol/L NaNO3共反应,共生成215 μmol/L NH4+ (图 3E),分别比其他组合高19.40%、667.00%、438.00%、467.00%、448.00%和382.00%,是构建硝酸盐同化路径的最优组合。将BsNasBC、EcNirBD导入XC3-2菌株,得到突变菌株XC4,检测菌株在0、10、25、50、100 mmol/L NaNO3为唯一氮源时的生长情况,结果如图 3F所示。菌株XC4能够在10、25和50 mmol/L的硝酸盐培养基上生长,最大DCW分别为0.57、0.55和0.37 g/L;但无法在0 mmol/L以及100 mmol/L的硝酸盐培养基上进行生长。上述结果表明,成功构建了一株氮源利用效率提高且能利用硝酸盐为氮源的L-赖氨酸生产菌株XC4。通过初始添加甜菜糖蜜及流加硫酸铵的方式供给氮源,在5 L发酵罐上评估了菌株XC4的生产性能,L-赖氨酸产量、产率和生产强度分别达到175.00 g/L、68.32%和4.86 g/(L·h),比出发菌株XC1提高了20.69%、13.39%和20.60%。
| Designation | Expression status | Specific activity (U/mg) |
| KoNasAC | Insoluble | Inactive |
| PpNasB | Insoluble | 0.051 |
| RcNasAC | Insoluble | 0.015 |
| BsNasBC | Soluble | 2.160 |
| PsNasC | Soluble | 0.170 |
| BmNasBC | Soluble | 0.080 |
| RpNasA | Non-expression | Inactive |
| MgNasA | Non-expression | Inactive |
| AvNasC | Non-expression | Inactive |
| Designation | Expression status | Specific activity (U/(mg) |
| KoNirD | Insoluble | Inactive |
| PpNirBD | Insoluble | Inactive |
| RpNirD | Insoluble | Inactive |
| RcNasB | Insoluble | 0.072 |
| EcNirBD | Soluble | 3.540 |
| BsNasDE | Soluble | 1.350 |
| PsNirBD | Soluble | 0.012 |
| BmNasDE | Soluble | 0.027 |
| PdNasBG | Non-expression | Inactive |
| KpNasD | Non-expression | Inactive |
发酵过程中残糖浓度对发酵过程中副产物的生成具有重要影响,残糖过高会导致乳酸、乙酸等副产物积累,从而降低转化率;残糖过低,则会导致细胞生长受限,降低生产强度[23]。在5 L发酵罐上研究了不同残糖浓度(10.00、5.00、2.00、0.50 g/L)对菌株XC4发酵生产L-赖氨酸的影响,结果如表 6、图 4所示。将残糖控制在0.50 g/L时,L-赖氨酸产量达到188.00 g/L,比控制在10.00、5.00、2.00 g/L分别提高了7.43%、9.30%、6.21%。转化率达到69.44%,比控制在10.00、5.00、2.00 g/L分别提高了7.73%、5.84%、1.64%。而此时发酵液中的副产物乙酸、柠檬酸、丙氨酸、酮戊二酸、乳酸等显著降低。从上述结果可知,控制残糖处于0.50 g/L时,菌株的L-赖氨酸生产效率最高。
| Parameter | Glucose (g/L) | (D/A–1)×100% | (D/B–1)×100% | (D/C–1)×100% | |||
| 10.00 (A) |
5.00 (B) |
2.00 (C) |
0.50 (D) |
||||
| Culture time (h) | 36.00 | 36.00 | 36.00 | 36.00 | 0.00 | 0.00 | 0.00 |
| Maximum DCW (g/L) | 19.30 | 20.00 | 18.80 | 18.40 | −4.66 | −8.00 | −2.12 |
| Titer of lysine (g/L) | 175.00 | 172.00 | 177.00 | 188.00 | 7.42 | 9.30 | 6.21 |
| Yield of lysine on glucose (%) | 64.46 | 65.61 | 68.32 | 69.44 | 1.70 | 0.52 | 1.02 |
| Productivity (g/(L·h)) | 4.86 | 4.78 | 4.92 | 5.22 | 8.50 | 13.80 | 10.30 |
| Titer of lysine on DCW | 9.07 | 8.60 | 9.41 | 10.22 | 13.70 | 23.80 | 12.60 |
| Consumption of glucose (g) | 684.00 | 655.00 | 647.00 | 676.00 | −1.16 | 3.20 | 4.48 |
| Consumption of glucose on DCW | 35.44 | 32.75 | 34.41 | 36.70 | 3.55 | 12.06 | 6.65 |
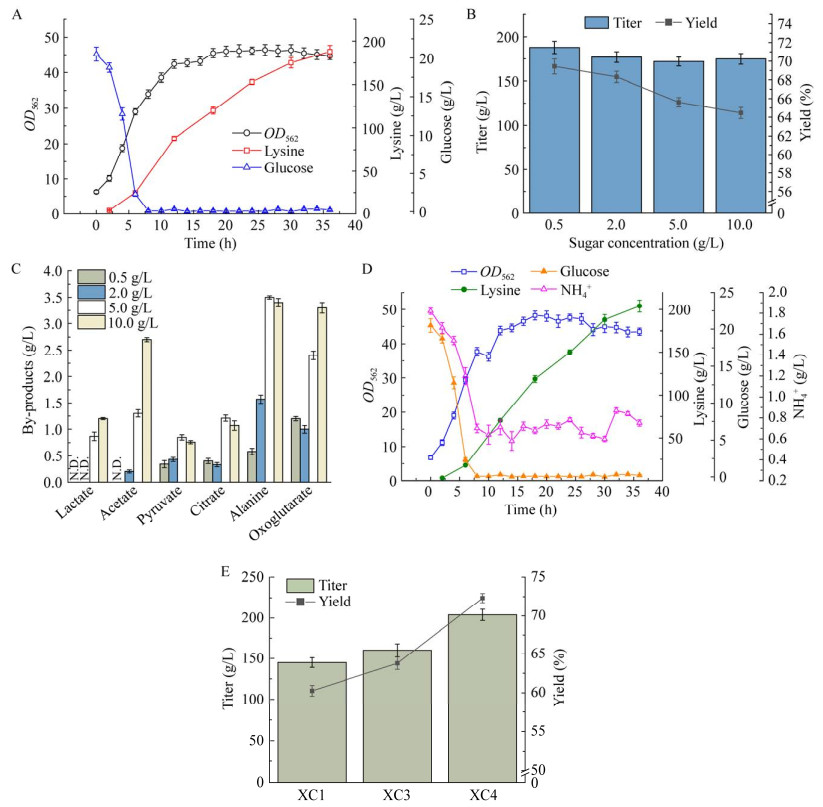
|
| 图 4 XC4菌株在5 L发酵罐中的生长和L-赖氨酸生产情况 Fig. 4 Growth characteristics and L-lysine production of strain XC4 in a 5 L fermenter. A: Fermentation process curve of strain XC4 in a 5 L fermentation tank. B: Comparison of yield and titer of XC4 under different sugar concentrations. C: Accumulation of by-products of XC4 under different sugar concentrations. D: Fermentation process curve of optimized XC4. E: Maximum titer and yield of each recombinant strain. N.D. represents not detected. A:XC4在5 L发酵罐中的发酵过程曲线. B:XC4在不同糖浓度下的L-赖氨酸产量和转化率比较. C:XC4在不同糖浓度下的副产物积累情况. D:碳氮比优化后的XC4发酵过程曲线. E:各重组菌株最大产量及转化率 |
| |
L-赖氨酸(C6H14N2O2)合成不同于丙氨酸、苏氨酸、色氨酸等氨基酸的合成:每合成1分子L-赖氨酸需要1分子葡萄糖及2分子NH4+参与,这一特点表明控制发酵液中碳氮比十分重要,因此,在确定最优糖浓度0.50 g/L的基础上,通过维持不同氨氮浓度(0.25−0.50、0.50−1.00、1.00−1.50、1.50−2.00 g/L),使发酵过程中碳氮比分别处于2:1−1:1、1:1−1:2、1:2−1:3、1:3−1:4,研究了不同碳氮比对菌株XC4生产L-赖氨酸的影响,结果如图 4C−4E和表 7所示:(1) 控制碳氮比1:1−1:2之间时,L-赖氨酸产量由188.00 g/L提高至204.00 g/L,提高了8.51%。(2) 转化率由69.44%提高至72.32%,提高了4.18%。(3) 单位细胞L-赖氨酸生产能力提高至11.20,提高了9.80%。(4) 生产强度由5.22 g/(L·h)提高至5.67 g/(L·h),提高了8.62%。(5) 乳酸、乙酸、丙酮酸、柠檬酸、酮戊二酸等积累量均降至1.50 g/L以下。上述结果表明,通过优化补料策略维持碳氮比在1:1−1:2之间,实现碳氮源的适量供应,能够有效提升XC4菌株的L-赖氨酸产量及转化率,降低副产物积累,从而提高L-赖氨酸生产效率。
| Parameter | Carbon nitrogen ratio | (B/A–1)×100% | (B/C–1)×100% | (B/D–1)×100% | |||
| 2:1−1:1 (A) | 1:1−1:2 (B) | 1:2−1:3 (C) | 1:3−1:4 (D) | ||||
| Maximum DCW (g/L) | 19.80 | 18.20 | 18.40 | 21.10 | −7.07 | −1.09 | −12.70 |
| Titer of lysine (g/L) | 175.00 | 204.00 | 188.00 | 170.00 | 16.50 | 8.51 | 20.00 |
| Yield of lysine on glucose (%) | 68.71 | 72.32 | 69.44 | 66.11 | 5.28 | 4.17 | 9.42 |
| Productivity (g/(L·h)) | 4.86 | 5.67 | 5.22 | 4.72 | 16.67 | 8.62 | 20.12 |
| Titer of lysine on DCW | 8.83 | 11.20 | 10.20 | 8.05 | 26.80 | 9.80 | 39.13 |
大肠杆菌利用碳源底物的途径主要有:(1) 磷酸转移酶系统(phosphotransferase system, PTS)将葡萄糖转运至胞内进行利用。(2) 通过激活麦芽糖操纵子(malATQ)表达,将麦芽糖转运至胞内并进一步分解为单糖进行利用。但上述2个碳源利用途径都受到全局调控因子Mlc调控[13],PTS系统葡萄糖转运酶EIICB在转运葡萄糖时会螯合Mlc蛋白而失活,抑制葡萄糖转运效率[14];同时,Mlc还会通过结合麦芽糖操纵子转录激活因子MalT,抑制麦芽糖操纵子的表达[15],限制大肠杆菌对麦芽糖的高效利用。本研究以实验室保藏菌株为底盘细胞,首次在L-赖氨酸生产菌株中敲除了转录调控因子基因mlc,弱化Mlc蛋白对EIIBC和麦芽糖操纵子的阻遏作用,得到的突变菌株E. coli XC2麦芽糖消耗速率提高了4.43倍。进一步通过引入来源于B. subtilis的malAP[24],强化E. coli XC2对异麦芽糖、潘糖等三糖的利用,得到了菌株E. coli XC3,E. coli XC3 L-赖氨酸产量提升至160.00 g/L,转化率提升至63.78%。
L-赖氨酸和L-精氨酸的高效合成都需要充足的氮源供应。在L-精氨酸高产菌株构建中,通过代谢工程改造氮源供应是一个有效的策略,如在L-精氨酸生产菌C. glutamicum SDNN403中过表达谷氨酰胺合成酶基因glnA使精氨酸产量提高了41.50%[25];在C. crenatum中强化铵转运蛋白基因amtB表达,将精氨酸产量提高了35.14%[26]。在本研究中,为了进一步提高L-赖氨酸生产菌的氮代谢路径效率,通过过表达谷氨酸脱氢酶基因gdhA,得到了E. coliXC3-2,此菌株在摇瓶发酵上的L-赖氨酸产量比出发菌株提高了55.10%。为了提高E. coli XC3-2对其他氮源(硝态氮)的利用,首次在大肠杆菌中构建了硝酸盐同化路径[27-28],得到菌株E. coli XC4能够利用NO3−、NO2−为氮源,最终,在5 L发酵罐上经过残糖浓度优化和碳氮比优化,E. coli XC4生产强度达到5.67 g/(L·h),L-赖氨酸产量达204.00 g/L,糖酸转化率达到72.32%。
虽然通过改造碳源、氮源代谢路径得到的E. coli XC4菌株相比菌株XC1的L-赖氨酸产量和转化率分别提高了40.69%和20.03%,但仍与理论转化率(81.00%)存在较大差距。因此,在传统代谢工程改造的基础上,亟须开发全新策略,例如:(1) 构建谷氨酸棒杆菌-大肠杆菌混合发酵体系以蔗糖为碳源生产L-赖氨酸[29];(2) 通过DNA复制工程加速DNA突变频率,缩短实验室适应性进化周期[30-31],同时结合高通量菌株筛选方法[32-33],提高高产菌株筛选效率;(3) 针对发酵后期发酵液中渗透压高、产物毒性强等问题,开发抗逆元件提高菌株抗逆性能[34],都能进一步提高L-赖氨酸的产量和转化率。
| [1] |
WENDISCH VF. Metabolic engineering advances and prospects for amino acid production[J]. Metabolic Engineering, 2020, 58: 17-34. DOI:10.1016/j.ymben.2019.03.008
|
| [2] |
DO CARMO FÉLIX FK, LETTI LAJ, de MELO PEREIRA GV, BONFIM PGB, SOCCOL VT, SOCCOL CR. L-lysine production improvement: a review of the state of the art and patent landscape focusing on strain deavelopment and fermentation technologies[J]. Critical Reviews in Biotechnology, 2019, 39(8): 1031-1055. DOI:10.1080/07388551.2019.1663149
|
| [3] |
YING HX, HE X, LI Y, CHEN KQ, OUYANG PK. Optimization of culture conditions for enhanced lysine production using engineered Escherichia coli[J]. Applied Biochemistry and Biotechnology, 2014, 172(8): 3835-3843. DOI:10.1007/s12010-014-0820-7
|
| [4] |
YE C, LUO QL, GUO L, GAO C, XU N, ZHANG L, LIU LM, CHEN XL. Improving lysine production through construction of an Escherichia coli enzyme-constrained model[J]. Biotechnology and Bioengineering, 2020, 117(11): 3533-3544. DOI:10.1002/bit.27485
|
| [5] |
LIU J, OU Y, XU JZ, RAO ZM, ZHANG WG. L-lysine production by systems metabolic engineering of an NADPH auto-regulated Corynebacterium glutamicum[J]. Bioresource Technology, 2023, 387: 129701. DOI:10.1016/j.biortech.2023.129701
|
| [6] |
WU WJ, ZHANG Y, LIU DH, CHEN Z. Efficient mining of natural NADH-utilizing dehydrogenases enables systematic cofactor engineering of lysine synthesis pathway of Corynebacterium glutamicum[J]. Metabolic Engineering, 2019, 52: 77-86. DOI:10.1016/j.ymben.2018.11.006
|
| [7] |
KAJSIKOVA M, KAJSIK M, DRAHOVSKA H, BUKOVSKA G. Complete genome sequence of the industrial L-lysine production strain [Brevibacterium] flavum CCM 251[J]. Biologia, 2022, 77(5): 1423-1428. DOI:10.1007/s11756-022-01021-x
|
| [8] |
XU JZ, HAN M, REN XD, ZHANG WG. Modification of aspartokinase Ⅲ and dihydrodipicolinate synthetase increases the production of L-lysine in Escherichia coli[J]. Biochemical Engineering Journal, 2016, 114: 79-86. DOI:10.1016/j.bej.2016.06.025
|
| [9] |
PIAO XY, WANG L, LIN BX, CHEN H, LIU WF, TAO Y. Metabolic engineering of Escherichia coli for production of L-aspartate and its derivative β-alanine with high stoichiometric yield[J]. Metabolic Engineering, 2019, 54: 244-254. DOI:10.1016/j.ymben.2019.04.012
|
| [10] |
郭亮, 高聪, 柳亚迪, 陈修来, 刘立明. 大肠杆菌生产饲用氨基酸的研究进展[J]. 合成生物学, 2021, 2(6): 964-981. GUO L, GAO C, LIU YD, CHEN XL, LIU LM. Advances in bioproduction of feed amino acid by Escherichia coli[J]. Synthetic Biology Journal, 2021, 2(6): 964-981 (in Chinese). |
| [11] |
KIKUCHI Y, KOJIMA H, TANAKA T. Mutational analysis of the feedback sites of lysine-sensitive aspartokinase of Escherichia coli[J]. FEMS Microbiology Letters, 1999, 173(1): 211-215. DOI:10.1111/j.1574-6968.1999.tb13504.x
|
| [12] |
D'ESTE M, ALVARADO-MORALES M, ANGELIDAKI I. Amino acids production focusing on fermentation technologies-a review[J]. Biotechnology Advances, 2018, 36(1): 14-25. DOI:10.1016/j.biotechadv.2017.09.001
|
| [13] |
KIM SY, NAM TW, SHIN D, KOO BM, SEOK YJ, RYU S. Purification of Mlc and analysis of its effects on the pts expression in Escherichia coli[J]. The Journal of Biological Chemistry, 1999, 274(36): 25398-25402. DOI:10.1074/jbc.274.36.25398
|
| [14] |
ROCHA S, MARZIALETTI T, KOPP M, CEA M. Reaction mechanism of the microwave-assisted synthesis of 5-hydroxymethylfurfural from sucrose in sugar beet molasses[J]. Catalysts, 2021, 11(12): 1458. DOI:10.3390/catal11121458
|
| [15] |
LENGSFELD C, SCHÖNERT S, DIPPEL R, BOOS W. Glucose- and glucokinase-controlled mal gene expression in Escherichia coli[J]. Journal of Bacteriology, 2009, 191(3): 701-712. DOI:10.1128/JB.00767-08
|
| [16] |
YAMAMOTO H, SERIZAWA M, THOMPSON J, SEKIGUCHI J. Regulation of the glv operon in Bacillus subtilis: YfiA (GlvR) is a positive regulator of the operon that is repressed through CcpA and cre[J]. Journal of Bacteriology, 2001, 183(17): 5110-5121. DOI:10.1128/JB.183.17.5110-5121.2001
|
| [17] |
MIYAKOSHI M. Multilayered regulation of amino acid metabolism in Escherichia coli[J]. Current Opinion in Microbiology, 2024, 77: 102406. DOI:10.1016/j.mib.2023.102406
|
| [18] |
YANG ZM, HAN YL, MA Y, CHEN QH, ZHAN YH, LU W, CAI L, HOU MS, CHEN SF, YAN YL, LIN M. Global investigation of an engineered nitrogen-fixing Escherichia coli strain reveals regulatory coupling between host and heterologous nitrogen-fixation genes[J]. Scientific Reports, 2018, 8: 10928. DOI:10.1038/s41598-018-29204-0
|
| [19] |
KUMAR R, SHIMIZU K. Metabolic regulation of Escherichia coli and its gdhA, glnL, gltB, gdhD mutants under different carbon and nitrogen limitations in the continuous culture[J]. Microbial Cell Factories, 2010, 9: 8. DOI:10.1186/1475-2859-9-8
|
| [20] |
BIRKE RR, CAUSON T, TURETSCHEK R, EMERSTORFER F, KARNER T, DOMIG KJ, HANN S. Requirements for accurate quantification of nitrate and nitrite in molasses: insights from an interlaboratory comparison[J]. Food Control, 2022, 134: 108712.
|
| [21] |
SHI WW, LU W, LIU QL, ZHI YE, ZHOU P. The identification of the nitrate assimilation related genes in the novel Bacillus megaterium NCT-2 accounts for its ability to use nitrate as its only source of nitrogen[J]. Functional & Integrative Genomics, 2014, 14(1): 219-227.
|
| [22] |
CHEN X, LIU CM, ZHU BL, WEI WX, SHENG R. The contribution of nitrate dissimilation to nitrate consumption in narG- and napA-containing nitrate reducers with various oxygen and nitrate supplies[J]. Microbiology Spectrum, 2022, 10(6): e0069522. DOI:10.1128/spectrum.00695-22
|
| [23] |
XU JZ, YU HB, HAN M, LIU LM, ZHANG WG. Metabolic engineering of glucose uptake systems in Corynebacterium glutamicum for improving the efficiency of L-lysine production[J]. Journal of Industrial Microbiology & Biotechnology, 2019, 46(7): 937-949.
|
| [24] |
ABE K, KURODA A, TAKESHITA R. Engineering of Escherichia coli to facilitate efficient utilization of isomaltose and panose in industrial glucose feedstock[J]. Applied Microbiology and Biotechnology, 2017, 101(5): 2057-2066. DOI:10.1007/s00253-016-8037-z
|
| [25] |
GUO J, MAN ZW, RAO ZM, XU MJ, YANG TW, ZHANG X, XU ZH. Improvement of the ammonia assimilation for enhancing L-arginine production of Corynebacterium crenatum[J]. Journal of Industrial Microbiology & Biotechnology, 2017, 44(3): 443-451.
|
| [26] |
XU MJ, LI J, SHU QF, TANG M, ZHANG X, YANG TW, XU ZH, RAO ZM. Enhancement of L-arginine production by increasing ammonium uptake in an AmtR-deficient Corynebacterium crenatum mutant[J]. Journal of Industrial Microbiology & Biotechnology, 2019, 46(8): 1155-1166.
|
| [27] |
LUQUE-ALMAGRO VM, GATES AJ, MORENO-VIVIÁN C, FERGUSON SJ, RICHARDSON DJ, ROLDÁN MD. Bacterial nitrate assimilation: gene distribution and regulation[J]. Biochemical Society Transactions, 2011, 39(6): 1838-1843. DOI:10.1042/BST20110688
|
| [28] |
SPARACINO-WATKINS C, STOLZ JF, BASU P. Nitrate and periplasmic nitrate reductases[J]. Chemical Society Reviews, 2014, 43(2): 676-706. DOI:10.1039/C3CS60249D
|
| [29] |
SGOBBA E, STUMPF AK, VORTMANN M, JAGMANN N, KREHENBRINK M, DIRKS-HOFMEISTER ME, MOERSCHBACHER B, PHILIPP B, WENDISCH VF. Synthetic Escherichia coli-Corynebacterium glutamicum consortia for L-lysine production from starch and sucrose[J]. Bioresource Technology, 2018, 260: 302-310. DOI:10.1016/j.biortech.2018.03.113
|
| [30] |
WANG XW, LI QG, SUN CM, CAI Z, ZHENG XM, GUO X, NI XM, ZHOU WJ, GUO YM, ZHENG P, CHEN N, SUN JB, LI Y, MA YH. GREACE-assisted adaptive laboratory evolution in endpoint fermentation broth enhances lysine production by Escherichia coli[J]. Microbial Cell Factories, 2019, 18(1): 106. DOI:10.1186/s12934-019-1153-6
|
| [31] |
刘佳, 郭亮, 罗秋玲, 陈修来, 高聪, 宋伟, 刘立明. 细胞寿命在大肠杆菌细胞工厂构建中的应用[J]. 生物工程学报, 2021, 37(4): 1277-1286. LIU J, GUO L, LUO QL, CHEN XL, GAO C, SONG W, LIU LM. Application of chronological lifespan in the construction cell factories[J]. Chinese Journal of Biotechnology, 2021, 37(4): 1277-1286 (in Chinese). DOI:10.13345/j.cjb.200430 |
| [32] |
WANG Y, LI QG, ZHENG P, GUO YM, WANG LX, ZHANG TC, SUN JB, MA YH. Evolving the L-lysine high-producing strain of Escherichia coli using a newly developed high-throughput screening method[J]. Journal of Industrial Microbiology and Biotechnology, 2016, 43(9): 1227-1286. DOI:10.1007/s10295-016-1803-1
|
| [33] |
丁爽, 陈修来, 高聪, 宋伟, 吴静, 魏婉清, 刘佳, 刘立明. 模块化工程改造大肠杆菌生产L-色氨酸[J]. 生物工程学报, 2023, 39(6): 2359-2374. DING S, CHEN XL, GAO C, SONG W, WU J, WEI WQ, LIU J, LIU LM. Modular engineering of Escherichia coli for high-level production of L-tryptophan[J]. Chinese Journal of Biotechnology, 2023, 39(6): 2359-2374 (in Chinese). DOI:10.13345/j.cjb.221005 |
| [34] |
SÉVIN DC, SAUER U. Ubiquinone accumulation improves osmotic-stress tolerance in Escherichia coli[J]. Nature Chemical Biology, 2014, 10: 266-272. DOI:10.1038/nchembio.1437
|
 2024, Vol. 40
2024, Vol. 40