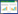中国科学院微生物研究所、中国微生物学会主办
文章信息
- 蒲春香, 李金龙, 龚大春, 罗华军, 张大伟
- PU Chunxiang, LI Jinlong, GONG Dachun, LUO Huajun, ZHANG Dawei
- 维生素生物合成途径中酶的代谢与功能的探索
- Enzyme metabolism and functions in vitamin biosynthesis pathways
- 生物工程学报, 2024, 40(8): 2570-2603
- Chinese Journal of Biotechnology, 2024, 40(8): 2570-2603
- 10.13345/j.cjb.240293
-
文章历史
- Received: April 2, 2024
- Accepted: July 16, 2024
- Published: July 16, 2024
2. 中国科学院天津工业生物技术研究所, 天津 300308;
3. 中国科学院大学, 北京 100049
2. Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
维生素是一类正常代谢所必需的有机化合物,在食品中微量存在,缺乏这些营养物质可能会导致代谢功能障碍[1]。例如维生素A与失明相关,维生素B1与脚气病、维生素B3与糙皮病、维生素B6与贫血、维生素C与坏血病以及维生素D与佝偻病相关,某些维生素缺乏症还可能对生命构成严重威胁[2-3]。维生素主要分为水溶性维生素和脂溶性维生素两大类。水溶性维生素包括维生素B和维生素C,脂溶性维生素包括维生素A、D、E和K。脂溶性维生素与脂肪有关并与膳食脂肪一起吸收,且与脂肪吸收具有相同的机制[4]。虽然许多植物和微生物可以自行合成维生素,但是动物通常无法自行产生足够的维生素来满足其需求[5]。人类和其他动物需要通过饮食或补充剂获取足够的维生素,以维持最佳的健康状态[6]。
维生素的生产主要为化学合成(VB6、VC等)和生物发酵(主要为结构相对复杂的品种,如VB12、VB2等)方法。化学合成法通常原材料价格高,容易产生有害废物且废物处理成本高昂,生物发酵法则因其低成本、低能耗、环保安全和废物易于回收等优势而备受关注。随着合成生物学的发展,生物发酵法已经获得研究人员和市场的广泛认可,并逐渐在工业中应用,以提高各种维生素的产量,比如维生素B2,在枯草芽孢杆菌中,通过对核黄素生物合成基因进行过表达,改善嘌呤从头合成以及戊糖供应,同时降低关键酶RibC的活性,从而降低VB2的消耗,增加VB2的积累,最终使核黄素产量提升至26 g/L[7]。另外在枯草芽孢杆菌的基础上通过过表达ilvBHCD、panBCDE、serA和glyA以及编码协助甘氨酸裂解循环酶的基因生产泛酸(维生素B5),最佳菌株在48 h补料发酵过程中产量可达82−86 g/L[8]。然而,与氨基酸等大宗化学品相比(如L-赖氨酸:213.0 g/L[9];L-脯氨酸:142.4 g/L[10];L-谷氨酸:136.3 g/L[11]),维生素的生物发酵产量仍然相对较低,尤其是一些高度复杂的分子,比如VB12,目前工业发酵水平仅为百毫克每升。除VB2、VB5和VC等少数维生素产品能够达到几十克每升的发酵水平外,其他维生素产品的发酵水平目前仍然处于毫克每升或几克每升,这严重限制了维生素生物工业化应用的进程[12]。
在生物发酵过程中,酶是一类绿色、安全可持续的生物催化剂,它在生物催化和微生物发酵过程中发挥着关键作用。酶在维生素的生物生产过程中同样起着催化和促进各种生化反应的重要作用。例如在生物催化方面,维生素A的商品形式通常是维生素A的酯类衍生物,为了增加其稳定性,工业上一般会将其制备成维生素A丙酸酯、维生素A棕榈酸酯、维生素A亚油酸酯、维生素A乳酸酯等形式;除维生素A棕榈酸酯和维生素A乳酸酯外,上述维生素A衍生物通常采用化学法合成,而化学合成中前体棕榈酰氯的合成需要使用有毒试剂,会对环境造成一定的破坏[13-14]。浙江大学于洪巍团队筛选出来源于大肠杆菌(Escherichia coli)的酯酶BioH,利用该酯酶先催化维生素A醋酸酯和低级醇生成维生素A醇,再催化维生素A醇与棕榈酸酯反应合成维生素A棕榈酸酯,反应总收率可达95%;此外,通过固定化技术实现酯酶的重复使用,显著降低维生素A棕榈酸酯的生产成本,此方法成功应用于工业生产[5, 13, 15]。而在微生物发酵方面,Liu等[16]通过蛋白质工程手段对维生素B6途径上多个酶进行筛选改造,单个酶的改造可以使产量提升近20倍,同时组合使用多模块迭代优化的代谢工程手段将维生素B6的产量从几毫克每升提高到1.4 g/L的发酵水平,为当时报道最高的维生素B6的生物发酵产量。由此可见,选择更优秀的酶作为生物催化剂,可以更好地加速底物转化为所需产品。然而,维生素的生物合成途径通常具有复杂性和多样性,涉及多种酶的催化作用和多个中间产物的转化,比如维生素B12的合成需要至少36个酶的有效配合,且中间产物难以检测,这为研究人员在阐明维生素合成途径及其相关酶的机制和进行改造带来了巨大挑战[17-18],尤其是维生素生产相关酶的动力学等关键性质研究相对较少。然而,随着合成生物技术、计算生物学技术和多学科交叉的发展,这一情况正在逐渐改变。
本文对维生素生产途径中相关酶的动力学性质等研究进行了整理归纳,从酶的代谢、研究手段和应用等方面进行综述,预测了所有涉及的酶在常见菌种中的动力学性质,综合评估了这些酶的动力学催化水平,并比较了糖酵解途径(glycolytic pathway),又称EMP途径,与维生素途径和不同维生素产品途径间的酶学性质差异,对可能的限速步骤进行讨论。通过理解两种途径中酶活性的进化适应,可以更好地平衡维生素合成途径中的酶活性,优化代谢流,避免代谢物的积累,为后续维生素的生产合成以及其他代谢产品的合成提供参考。
1 维生素的生物合成途径及其酶的改造维生素的生物合成是一个高度复杂的过程,酶在其中扮演着关键的角色(图 1)。这些酶表现出特异性、多样性以及复杂的调控机制,确保了维生素合成过程的高效性和精确性。
|
| 图 1 维生素代谢途径总览 Fig. 1 Overview of vitamin metabolism pathways. Different colors represent representative biosynthetic pathways of different vitamins, and the numerical values on the lines indicate the quantity of enzymes required for each step. Shared pathways such as EMP, TCA, and PPP are depicted by black lines, while lines without numerical values indicate that only one enzyme is needed to complete that step. Pathway data is sourced from the KEGG website[22]. DHBP: 5-amino-6-(1-D-ribitylamino)uracil; DRL: 6,7-dimethyl-8-(D-ribityl)lumazine; Arp: L-3,4-dihydroxybutan-2-one 4-phosphate; ABEE: 4-aminobenzoate; PRPP: 5-phospho-alpha-D-ribose 1-diphosphate; AIR: aminoimidazole ribotide; HMP-PP: 4-amino-5-hydroxymethyl-2-methylpyrimidine diphosphate; cThz-p: 2-(2-carboxy-4-methylthiazol-5-yl)ethyl phosphate; DOXP: 1-deoxy-D-xylulose 5-phosphate; IPP: isopentenyl diphosphate; GGPP: geranylgeranyl diphosphate; 7-DHC: 7-dehydrocholesterol; MGGBQ: 6-geranylgeranyl-2-methylbenzene-1,4-diol; DHNA: 1,4-dihydroxy-2-naphthoate; HepPP: all-trans-polyprenyl diphosphate; DMK-7: demethylmenaquinol; 5-ALA: 5-aminolevulinate; UDPG: UDP-glucose; GDD: GDP-mannose; KAPA: 8-amino-7-oxononanoate. |
| |
酶作为生物体内的催化剂,在几乎所有生物化学反应中都发挥着关键作用。酶能够显著提高化学反应的速率,使得在生物体内的温度和压力条件下便可以完成在化学反应中只能高温高压才能实现的过程。此外,酶的高度底物特异性确保了生物化学反应的精确性和效率。在维生素的生物合成过程中,酶同样扮演着至关重要的角色。每一种维生素的合成都涉及一系列酶的催化反应,这些反应将简单的前体分子转化为复杂的维生素分子。这些酶的活性和特性直接影响了维生素合成的效率和产量。因此,深入理解这些酶的动力学性质,研究如何通过生物工程技术改造这些酶以优化维生素的生产,是突破维生素生物合成瓶颈的重要方向。本文将详细探讨各种维生素合成途径中所涉及的酶的数量、种类、关键酶及其改造应用研究。
1.1 水溶性维生素 1.1.1 维生素B1维生素B1,也称为硫胺素,是B族维生素中首个被发现的成员,焦磷酸硫胺素(thiamine pyrophosphate, TPP)是硫胺素的活性形式。维生素B1通常被用于治疗硫胺素缺乏症并预防其相关疾病,包括脚气病、科尔萨科夫综合征和精神类疾病[19-20]。此外,维生素B1也广泛被用作食品工业的添加剂,并在化妆品和医疗保健行业中作为抗氧化剂大量使用[21]。VB1的生物合成主要通过戊糖磷酸途径生成5-磷酸-α-D-核糖-1-二磷酸(5-phospho-alpha-D-ribose 1-diphosphate, PRPP),并通过两种酶的催化生成氨基咪唑核糖(aminoimidazole ribotide, AIR),再通过两种酶生成4-氨基-5-羟甲基-2-甲基嘧啶二磷酸(4-amino-5-hydroxymethyl-2-methylpyrimidine diphosphate, HMP-PP),最后与2-(2-羧基-4-甲基噻唑-5-基)乙基磷酸酯[2-(2-carboxy-4-methylthiazol-5-yl)ethyl phosphate, cThz-P]在两种酶的共同作用下生成硫胺素单磷酸(thiamine phosphate, TMP)。此外,HMP-PP的合成不仅依赖于戊糖磷酸途径,也可以通过磷酸吡哆醛(pyridoxal phosphate, PLP)在两种酶的作用下生成(图 1,浅绿色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素B1的已知合成途径涉及不同生物体对酶的需求,具体为:原核生物合成需9种酶,古细菌合成需5种,植物合成需6种,其他途径需7种,补救途径需4种酶,因此维生素B1合成共涉及16种不同的酶。Schyns等[23]通过遗传突变和转录组分析的方法,将4种突变(thiL、thiN、yuaJ和ykoD)结合到单一菌株中得到菌株TH95,在10 L规模的发酵条件下,硫胺素产物产量为1 270 µg/L(细胞外)和120 µg/L (细胞内),总计1 390 µg/L。维生素B1的生物合成途径受多种生物合成酶的调控,其中THIC、THI1、TH1、TH2和TPK是该途径中的关键酶。而thiC和thiH基因参与铁硫簇的代谢,受到S-腺苷甲硫氨酸(S-adenosylmethionine, SAM)代谢物的抑制,ThiC酶的催化活性非常低,kcat值为0.14 min–1,因此成为主要的代谢瓶颈之一[12]。
目前,其中9种酶的动力学性质已得到研究,4种酶已进行改造研究。例如,甘氨酸氧化酶(EC: 1.4.3.19)可以催化甘氨酸被氧气氧化生成亚甲基甘氨酸,该产物可以作为维生素B1合成途径上的前体,其中对枯草芽孢杆菌来源的酶研究得最多。Rosini等[24]为了探究该酶应用于生物传感器中小的非手性氨基酸偏好性的选择问题,通过半理性设计,对底物活性腔周围的11个氨基酸位点分别进行了饱和突变,并将效果好的突变进行叠加,其中突变体H244K/M261R,对甘氨酸的催化活性增加了5.4倍。Tatsumi等[25]为了研究该酶在血液中的稳定性问题,首先将溶剂可及性最高的半胱氨酸245突变为丝氨酸,然后对辅因子周围的氨基酸进行保守突变,以维持蛋白三维结构,最后构建了三突变体T42A/C245S/L301V,使得kcat/Km值提升了5倍,并且在350 d内,酶活性仍然维持在较高水平;该研究提供了一种快速和简便的人血浆中甘氨酸检测方法,可用于生理学研究和临床应用。甘氨酸是维生素B1合成途径的中间产物,该研究对于监测体内维生素B1合成状况也具有重要指导意义。硫胺素二磷酸激酶(thiamine pyrophosphokinase, hTPK1)可以催化硫胺素转化为其活性形式焦磷酸硫胺素,是催化形成活性VB1的最后一步。Onozuka等[26]利用保守序列比对以及同源建模的方法,对D71、D73、D100、D133、Q96、T99和R131的位置进行定点突变;结果表明,Q96E突变可以增加酶对硫胺素的亲和力,D133N导致硫胺素的kcat/Km值显著降低,而T99A和R131G的突变导致ATP的kcat/Km值显著降低。该研究系统性地评估了hTPK1的催化机制,并识别到可能参与底物识别的关键氨基酸残基。
1.1.2 维生素B2维生素B2又称核黄素,是一种重要的微量营养素,作为辅酶黄素单核苷酸(riboflavin 5'-phosphate, FMN)和黄素腺嘌呤二核苷酸(flavin-adenine dinucleotide, FAD)的直接前体,在所有活细胞的生化反应中发挥作用[27-28]。这些辅酶参与碳水化合物、脂质、酮体和蛋白质的代谢,为生物体提供大量能量[29]。维生素B2的合成起始于两个不同的代谢途径。首先,戊糖磷酸途径中的D-核糖-5-磷酸(D-ribulose 5-phosphate, R5P)通过1个酶促反应转化为2-羟基-3-氧代丁基磷酸酯(2-hydroxy-3-oxobutyl phosphate, DHBP)。接着,嘌呤途径中的5ʹ-三磷酸鸟苷(guanosine 5ʹ-triphosphate, GTP)经过4个酶的作用,转化为5-氨基-(1-D-核糖基氨基)尿嘧啶(5-amino-6-(1-D-ribitylamino)uracil, ARP)。最终,DHBP和ARP通过另外2个酶的催化反应,合成维生素B2 (图 1,绿色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素B2的生物合成是一个多样化的过程,它在植物、细菌和某些真菌中通过不同的代谢途径进行。每种途径都涉及9种酶,而整个合成过程,总共涉及11种不同的酶,其中包括将维生素B2转化为其活性形式FAD所需的酶。目前,除了Li等[30]在2005年的专利申请公开的核黄素生产菌株可以达到26.8 g/L外,目前文献报道的最高产量为Xu等[31]使用基于液滴微流体筛选从菌株S1的随机突变文库中得到的核黄素生产菌U3,产量可达到24.3 g/L。在核黄素的生物合成途径中,GTP环化水解酶Ⅱ (由ribA编码)被识别为限速酶,因为它在合成途径中具有最高的酶成本[32],并且Burgess等[33]曾发现rib操纵子基因的过表达可以增加核黄素的产量。
在维生素B2合成过程中,已有6种酶的动力学特性得到深入研究,4种酶经过改造以提高其性能。例如,GTP环化酶Ⅱ (EC: 3.5.4.25)是核黄素合成的第一步,将GTP分解为甲酸、核糖基嘧啶化合物以及磷酸。Lehmann等[34]通过非理性设计,利用易错PCR技术生成了一个突变的枯草芽孢杆菌(Bacillus subtilis) ribA基因库,同时对突变体蛋白的GTP环化酶Ⅱ活性和GTP底物的Km值进行了测量;并发现其中一种突变体携带7个点突变(Y210C/A290T/Q293R/A296T/K322R/F339Y/M361I: Construct E),GTP环化酶Ⅱ活性增加了2倍,但底物亲和力Km值增加了4倍;随后通过stopped-flow单转换实验在严格单转换条件下(酶过量),野生型和Construct E的吸收曲线相似;但在预稳态条件下(底物过量),Construct E产生产物的初始速率明显高于野生型;这表明突变影响的是产物释放步骤而非产物形成步骤,在细胞内GTP浓度远高于Km值时,突变导致的底物吸收速率降低可以忽略不计,GTP环化酶的活性和结合能力是将更多底物引流进入VB2合成途径的关键点。
在核黄素合成途径中,倒数第二步涉及的酶是6,7-二甲基-8-核黄素合酶(EC: 2.5.1.78),该酶在枯草芽孢杆菌中是一种由60个亚基形成的二十面体衣壳结构,内部包裹着一个同源三聚体的核黄素合酶单元。这一步反应将5-氨基-6-(1-D-核糖基氨基)尿嘧啶和L-3,4-二羟基丁酮-2-酮-4-磷酸转化为6,7-二甲基-8-(D-核糖基)光氨酮、水分子和磷酸根离子。Fischer等[35]通过半理性设计方法,研究了暴露在活性中心表面的氨基酸残基对催化性能的影响,发现与酶底物5-氨基-6-核糖基氨基-2,4(1H, 3H)-嘧啶二酮和3,4-二羟基-2-丁酮四磷酸直接接触的极性氨基酸残基在不影响催化性能的前提下能够被替换;然而,与3,4-二羟基-2-丁酮四磷酸的磷酸盐基团形成盐桥的R127以及被认为在质子转移反应中起到协助作用的H88残基的替换使催化活性降低了约一个数量级,说明该酶的催化作用主要通过建立两个底物之间的有利拓扑关系来实现,而酸碱催化只起到次要作用。
除了参与维生素B2合成的酶以外,对其继续转化酶的研究也至关重要。Serrano等[36]报道了一种原核生物的双功能FAD合成酶(fatty acid desaturase, FADS),它由两个模块组成:C-末端模块具有ATP: 核黄素激酶(riboflavin kinase, RFK)活性,N-末端模块具有ATP: FMN腺苷酸转移酶(FAD synthetase, FMNAT)活性,在核黄素合成途径中RFK和FMNAT这两种酶分别催化核黄素转换为活性的核黄素-5-磷酸(riboflavin-5-phosphate, FMN),以及FMN转换为FAD的过程;通过半理性设计的方法,研究人员引入点突变来分析盐桥对配体结合和催化活性的影响,结果表明这些突变对FMNAT模块的配体结合和催化性质几乎没有影响,但显著降低了RFK活性的催化效率;R66的替代还改变了单体和寡聚体之间的比例,并修改了单分子方法观察到的四聚体结构。这些酶的研究对于在合成中如何更大程度积累核黄素产品具有重要的参考价值。
1.1.3 维生素B3维生素B3,也称为烟酸(niacin),是一种水溶性维生素,也是维生素B复合体的一部分。它在人体内起着重要的生理功能。同时也是辅酶烟酸腺嘌呤二核苷酸和烟酸腺嘌呤二核苷酸磷酸的组成部分,参与细胞内能量代谢[12, 37]。这些辅酶在葡萄糖、脂肪和蛋白质的代谢中发挥重要作用,促进食物转化为能量。维生素B3的生物合成可以根据关键前体喹啉酸盐(quinolinate)的不同合成方法分成以下两条路径:第一条是由天冬氨酸出发需要2种酶合成quinolinate,最后再经过4种酶合成VB3;另外一条途径是由分支酸(chorismate)出发,合成quinolinate需要11种酶,然后再通过与上一条路径完全相同的4种酶合成VB3 (图 1,红色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素B3的生物合成主要通过两条途径进行:首先是天冬氨酸途径,这一途径需要8种酶的参与;其次是分支酸途径,同样需要8种酶。尽管两种途径各自涉及8种酶,但在整个维生素B3合成过程中,实际上共有10种不同的酶参与。Belenky等[38]通过调节烟酸的运输来平衡胞内NAD+的代谢生长,并发现在酵母细胞中添加0.005 mol/L烟酸能够促进8 mg/L的烟酰胺核糖核苷的生成。除传统发酵法外,Wang等[39]使用工程化的大肠杆菌菌株BAG表达来自红球菌属(Rhodococcus rhodochrous) J1的高分子量腈水合酶来合成维生素B3 (尼克酰胺),以3-氰基吡啶为底物,在高密度的BAG中发酵,60 min内可使烟酰胺的产量达到508 g/L。程琳等[40]先在大肠杆菌中异源表达了来自松噬几丁质菌(Chitinophaga pinensis)的烟酰胺磷酸核糖转移酶(nampt),构建了NMN的补救合成途径,通过敲除NMN下游代谢分解途径基因pncC、ushA以及调控NAD+衍生物基因nadR,所获得的重组菌摇瓶水平产量达到60 mg/L,是初始产量的4.95倍,进一步在5 L罐上进行发酵试验,产量最高达到390.1 mg/L。
目前,已有9种酶进行了动力学性质研究,7种酶进行了改造研究。例如,烟酸核苷酸腺苷酸转移酶(nicotinate-nucleotide adenylyltransferase, NadD)能够催化烟酸单核苷酸(nicotinic acid mononucleotide, NaMN)和烟酰胺单核苷酸(nicotinamide mononucleotide, NMN)与ATP的反应,将它们腺苷化形成尼古丁酸腺苷二核苷酸(nicotinic acid adenosine dinucleotide, NaAD)和烟酰胺腺嘌呤二核苷酸(NAD+),是维生素B3生产过程中的中间步骤。Wang等[41]为了提高酰胺化途径的活性,对NadD进行工程改造,使其更倾向于与NMN结合;通过半理性设计的方法,对活性口袋周围的氨基酸残基8个双位点进行饱和突变体文库构建,通过酶偶联比色法筛选出2个对NMN具有较高偏好性的变体,分别命名为11B4 (Y84V/Y118D)和16D8 (A86W/Y118N);在存在NMN的情况下,这两个变体都能够使大肠杆菌生长,而无需外源性NMN,并且在稳定期内细胞中的NAD(H)水平高于对照菌株,两个突变体相对于NMN分别增加了100倍和600倍;研究通过蛋白工程方法改变NadD的底物特异性,成功建立了酰胺途径完成NAD+生物合成的新途径,为细胞内NAD+水平的调控提供了新的途径,对操纵细胞代谢过程具有重要意义。
NAD+糖水解酶(EC: 3.2.2.6)是一个多功能酶,可以催化NAD+的水解反应,将NAD+分解为烟酰胺(nicotinamide)和腺苷二磷酸核糖(ADP-ribose),可用于生成维生素B3的合成途径。为了研究牛CD38/NAD+糖水解酶的催化机制,Kuhn等[42]通过半理性设计的方法,对活性中心周围的氨基酸进行了定点突变,获得了一个关键的活性位点残基E218,突变导致催化活性显著下降,E138和D147通过基态不稳定化和溶剂化作用在水解位点附近也起到重要作用,W118和W181这些非极性残基的突变可能会影响过渡态的稳定性,这些残基的突变,会一定程度上导致催化效率的下降;这些发现对于理解CD38酶家族的催化机制以及相关生物学过程具有重要意义。此外,对维生素B3的生物合成起到指导和启示作用,也能为相关疾病的治疗和药物开发提供新的思路。
1.1.4 维生素B5维生素B5,又称泛酸(pantoate),是一种人体必需的微量元素[43]。它是辅酶A (CoA)和酰基载体蛋白(acyl carrier protein, ACP)的重要前体,参与人类和动物的多种生物代谢[44-45]。若摄入不足,可能引发记忆力减退、肾上腺功能衰竭及关节炎等病症[46-47]。VB5的合成首先需要丙酮酸经过4种酶的催化得到缬氨酸(valine),在此基础上通过3种酶的催化得到泛酸,并与β-alaine在1种酶的催化作用下得到。其中丙氨酸(β-alaine)可以由以下两个来源得到:第一个来源是通过莽草酸途径得到精氨酸,在此基础上通过一种酶的催化即可得到;另一条途径是通过精氨酸(arginine)和脯氨酸(proline)的代谢途径和反应得到精胺(spermine),最后再经过2种酶催化即可得到(图 2,浅紫色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
基于β-丙氨酸合成前体的差异,维生素B5的生物合成途径主要分为两大类:第一类途径起始于天冬氨酸,涉及5种酶的催化;第二类途径以精胺为起点,需经过6种酶的作用,综合这两种途径,维生素B5的合成总共涉及7种不同的酶。Tigu等[48]通过在大肠杆菌中表达6种不同来源的泛酸合成酶,发现来自谷氨酸棒杆菌的泛酸合成酶具有最高的比活力和转换数;通过在发酵对数早期添加底物(D-泛解酸和β-丙氨酸),在32 h内产生97.1 g/L的D-泛酸,转化率达到99.1%,生产率达到3.0 g/L,是目前报道的最高生产强度。Guo等[49]通过筛选关键基因、构建异源途径、调整基因拷贝数、敲除内源旁路基因、平衡NADPH的利用等方法构建的酵母菌株DPA171在优化的进料批次发酵条件下产生了4.1 g/L的D-泛酸,这是迄今为止在酿酒酵母(Saccharomyces cerevisiae)中报道的最高产量。Dibble等[50]发现泛酸激酶2 (PANK2)催化维生素B5的第一步磷酸化反应,生成4’-磷酸泛酸(4’-phosphopantothenate),是CoA合成的速率决定步骤;研究表明,PANK2的活性可能受到细胞内CoA水平的反馈抑制调节:当细胞内CoA水平充足时,PANK2的活性可能降低,从而减少CoA的合成。因此,PANK2被认为是维生素B5合成途径中的关键限速酶,能够调节和维持细胞内CoA水平的稳定。
所有参与维生素B5合成的酶都已经完成动力学特性的研究,并且有5种酶已进行过改造研究。例如,L-天冬氨酸-α-脱羧酶(L-aspartic acid-α-decarboxylase, ADC)是途径中的第1个酶,ADC可以将L-天冬氨酸通过脱羧反应转化为β-丙氨酸。而β-丙氨酸是维生素B5合成过程中的重要前体,Pei等[51]先是对3种来源(大肠杆菌、谷氨酸棒杆菌、枯草杆菌)的ADC进行了实验表征,发现枯草来源的ADC相较于其他两种来源显示出更高的特异活性和热稳定性,并专门针对枯草来源的ADC通过非理性设计的方法开发了一种高通量的β-丙氨酸荧光测定法;通过对4 000种突变体的筛选,获得了2种高特异性和高催化稳定性的变体,特异性和催化稳定性分别提升18%−22%和29%−64%;最佳突变体在8 h内的L-天冬氨酸转化产生的β-丙氨酸比野生型高出50%,显示出在工业上利用生物法生产β-丙氨酸的巨大潜力。
维生素B5合成的最后一个酶,泛解酸β-丙氨酸连接酶(AMP形成)又称泛酸合成酶(pantothenate synthetase, EC: 6.3.2.1)催化泛酸和丙氨酸与ATP的反应,生成泛酸、AMP和焦磷酸二酯。Jonczyk等[52]对不同来源的酶(拟南芥和大肠杆菌)进行了异构效应差异研究;结果表明虽然它们序列上存在显著的相似性,并且都是二聚体,但拟南芥来源表现出了明显的异构效应,而大肠杆菌来源则几乎没有异构行为;为了验证这一差异,研究人员通过半理性设计的方法对拟南芥泛酸合成酶的亚单位接触区域进行了突变,以达到弱化或改变其异构特性,但不影响酶的二聚形态的目的;结果发现,这些突变导致了拟南芥来源酶的异构效应明显减弱或改变,并且发现拟南芥来源酶在低泛酸水平下具有更高的催化效率,这可能与其异构效应相关;相比之下,大肠杆菌来源酶在不同泛酸浓度下的催化行为基本上没有变化。这些实验证据表明,选择和维持拟南芥泛酸合成酶中特定亚单位的接触区域,导致了与异构体互作相关的功能优化。这一结果支持了植物中泛酸生物合成的异构体行为可能在亚细胞分布上提供了选择性优势的假设。
1.1.5 维生素B6维生素B6存在6种可相互转化的吡啶化合物形式,包括吡哆醇(pyridoxine, PN)、吡哆胺(pyridoxamine, PM)、吡哆醛(pyridoxal, PL)、吡哆醇5-磷酸(pyridoxine 5’-phosphate, PNP)、吡哆胺5-磷酸(pyridoxamine 5’-phosphate, PMP)和吡哆醛5-磷酸(pyridoxal 5’-phosphate, PLP)[53-55],为多种生化功能所必需,其中大多数与氨基酸生物合成和降解有关,维生素B6参与调节情绪、睡眠和食欲(与血清素的合成有关)以及血压和心率(与多巴胺的合成有关),其缺乏可能会导致抑郁症、癫痫等神经精神性疾病[56-58]。维生素B6的主要商品化形式为PN,可以在体内通过转化形成PLP,从而在生物体中作为多种蛋白质和酶的辅助因子发挥作用。已报道了两种VB6从头合成途径:1‑脱氧木酮糖5‑磷酸(1-deoxy-D-xylulose 5-phosphate, DXP)依赖性途径和DXP非依赖性途径。DXP依赖性途径可以通过D-赤藓糖-4-磷酸(D-erythrose 4-phosphate, E4P)经过7种酶的催化得到PLP;而DXP非依赖性途径则是通过谷氨酰胺(L-glutamine)、核糖5-磷酸(ribulose-5p, R5P)以及甘油醛3-磷酸(glyceraldenhyde-3p, G3P)在一种酶的催化下得到(图 1,棕色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
大肠杆菌等多数革兰氏阴性菌主要通过DXP依赖途径进行合成,该途径共需要7种酶,某些真菌和植物采用DXP非依赖途径合成PLP,此途径通过R5P、G3P和谷氨酰胺在单一酶催化下合成,维生素B6合成的途径中涉及的酶类总计8种,Tian等[59]通过转录组和代谢组学分析,研究了吡哆醇与氨基酸以及TCA循环的关系,通过发酵优化琥珀酸、氨基酸和碳氮比,在分批补料发酵中使PN产量达到1.95 g/L。除此之外,Wang等[60]利用伤寒沙门氏菌酸性磷酸酶和大肠杆菌吡哆醇氧化酶作为途径酶,以PN和焦磷酸盐(pyrophosphate, PPi)为底物合成PLP;在双酶催化下,最优菌株PLP的产量最高达到了15 g/L以上,产率为1.02 g/(L·h),这是目前报道过的最高生产率。Hoshino等[61]通过在野生型苜蓿中华根瘤(Sinorhizobium meliloti) IFO14782中过表达磷酸吡哆醇合成酶(pyridoxine 5-phosphate synthase, PdxJ),7 d内产生362 mg/L的吡哆醇,PdxJ酶的活性较低(kcat=0.07 s–1),是维生素B6生物合成途径中的限速步骤,其催化的反应生成的中间产物4-磷酸羟基-苏氨酸(4-phosphohydroxy-L-threonine, 4HTP)具有细胞毒性,因此成为维生素B6生物合成的主要瓶颈。
维生素B6合成途径中已有8种酶完成动力学性质研究,其中3种酶已经进行改造研究。例如,吡哆醇5ʹ-磷酸合酶(EC: 2.6.99.2, PdxJ)催化1-脱氧-D-木醛糖-5-磷酸(1-deoxy-D-xylulose 5-phosphate)与3-氨基-2-氧丙基磷酸(3-amino-2-oxopropyl phosphate)的成环反应,是DXP依赖途径进入维生素B6 (PNP形式)补救路线的最后一个酶,也是VB6合成途径中的主要限速酶之一。为了更好地发挥PdxJ酶拉动上游产物往下游产品的推动作用,Liu等[16]利用基于计算的迭代饱和突变策略,对PdxJ酶进行多轮的迭代设计与组合,单点、双点和三点突变的最高产量逐步提升,直到第4轮最优组合产量不再提升时终止迭代,构建了200多个突变的实体库,最终获得的三点突变体E104T/I218L/G194C (LL239)的菌株产量比对照菌株提高18倍;除此之外,研究人员还继续对途径中的其他酶进行筛选设计,并通过代谢迭代优化,将产量从几毫克每升提升到1.4 g/L;该研究通过系统性地对DXP依赖途径上几乎所有酶进行优化设计,为维生素B6的生物合成开辟了新的思路,也为其他产品的生物催化提供了可行的指导方案。
吡哆醛5'-磷酸氧化酶(EC: 1.4.3.5)催化吡哆醛5'-磷酸与氧气发生反应,生成过氧化氢和吡哆醛5'-磷酸,是维生素B6 (PLP形式)合成过程中的最后一步。Gu等[62]探讨了赖氨酸乙酰化对大肠杆菌吡哆醛5'-磷酸氧化酶(EcPNPOx)活性的调节作用,使用质谱技术筛选了大肠杆菌的蛋白质乙酰化修饰,并鉴定出乙酰化的EcPNPOx蛋白中的两个赖氨酸残基,并在此基础上通过半理性设计对K72进行定点突变分析,研究证明乙酰化的赖氨酸残基在蛋白质的酶活性和酶学特性中起着重要作用;此外,还发现在体外条件下,EcPNPOx可以被乙酰磷酸非酶类乙酰化,而CobB是一种蛋白质去乙酰化酶,也称为乙酰赖氨酸酶,它在细胞中起到去除蛋白质上的乙酰基修饰的作用,并可以被CobB蛋白进行去乙酰化修饰;该研究还比较了乙酰化和去乙酰化的EcPNPOx的酶活性,结果显示乙酰化会导致其酶活性的降低,而CobB蛋白的去乙酰化修饰可以恢复其酶活性。
吡哆醛5ʹ-磷酸合酶(兼具谷氨酰胺水解功能,EC: 4.3.3.6),是DXP非依赖途径所需要的酶,包括Pdx1和Pdx2两个亚基。Pdx2的主要功能是水解谷氨酰胺产生游离的氨基。Pdx1具有合成功能,可以利用R5P和G3P以及Pdx2提供的氨基,直接合成维生素B6 (PLP形式)。Moccand等[63]利用半理性设计的方法,通过酶活测定、酶的动力学性质和突变研究发现,在维生素B6的合成过程中,Pdx1的亚基与三糖底物结合并参与反应的后半部分,这个部分负责将氨与吡哆醛磷酸结合,形成活性的PLP,而被发现的这个催化位置又被称为P2位点;并通过突变和活性分析证明了P2位点对PLP合成的催化作用,特别是对G3P的结合和催化;同时研究还发现产物的释放还需要由依赖吡哆醛磷酸的酶而触发;此外还证明了位于Pdx1 C端的一个精氨酸残基(R288)在调节该复杂蛋白机制的协同作用中起着重要的作用;该项研究阐明了DXP非依赖途径的维生素B6生物合成的多个机制,为深入理解这一过程提供了证据,对维生素B6的生物合成过程具有重要意义,同时为开发针对依赖此途径的病原体的药物提供了潜在的治疗机制。
1.1.6 维生素B7维生素B7,也称为生物素,作为各种羧化酶和脱羧酶的辅因子,在多种生化功能中发挥着关键作用,包括碳水化合物、脂肪酸和氨基酸的代谢[64-65],因此,生物素在饮食中具有至关重要的作用。生物素依赖性羧化酶如丙酮酸羧化酶(pyruvate carboxylase, PC)、3-甲基巴豆酰辅酶A (3-methylcrotonyl-coA carboxylase, MCC)和丙酰辅酶A羧化酶(propionyl-coA carboxylase, PCC)功能障碍与危及人类生命的疾病有关[66]。VB7的生物合成需要从乙酰辅酶A经过4个酶催化到长链酰基辅酶ACP,然后经过两个酶催化得到8-氨基-7-氧代壬酸酯[(8S)-8-amino-7-oxononanoic acid, KAPA],这里8-氨基-7-氧代壬酸酯还可以通过由庚二酸(pimelate)被两种酶催化所得到,最后再经过3种酶催化生成VB7 (图 1,浅红色,国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素B7的生物合成可通过以下4种主要途径实现:第一种为经由BioF为起始的途径,该途径需要5种酶;第二种为途径BioU的途径,需要4种酶;第三种为BioI途径,需要5种酶;第四种为BioW途径,需要5种酶。这些途径中涉及的酶类总计有9种,早在1995年Masuda等[67]通过构建和优化重组菌株以及调整培养条件等方式将生物素产量提升到600 mg/L。Clack等[68]通过化学诱变实验,降低了易变假单胞菌(Pseudomonas mutabilis)中生物素的负反馈调节,并通过引入表达载体和优化发酵条件来增加生物素产量,最后使其产量达到15 g/L,尽管专利中报道的产量较高,但缺乏后续研究。在B. subtilis中,生物素合成酶(BioC、BioB、BioA、BioK)受到S-腺苷-L-甲硫氨酸(SAM)副产物的强烈抑制[69]。这些酶对SAM的需求量很高,尤其是BioB,其kcat值仅仅为0.12 min–1[70]。
在维生素B7的合成过程中,有5种酶已完成动力学特性研究,2种酶进行过改造研究。例如,腺苷甲硫氨酸-8-氨基-7-氧代壬酸转氨酶又称7,8-二氨基壬酸合酶(7,8-diaminopelargonic acid (DAPA) synthase, EC: 2.6.1.62),通过催化作用将S-腺苷基-L-甲硫氨酸的一个氨基基团转移给8-氨基-7-氧代壬酸,形成S-腺苷基-4-甲硫-2-酮丁酸(S-adenosyl-4-methylthio-2-oxobutanoate)和7,8-氨基壬酸(DAPA),是VB7合成途径的倒数第3个酶。为了探究其活性中心催化残基的催化机制,Sandmark等[71]通过多序列比对方法找出保守残基,并进行突变研究;结果表明,与野生型酶相比,突变体Y17F和Y144F与底物DAPA的反应速率分别降低了约99.92%和99.97%,但是突变体的晶体结构中活性位点处并没有显示显著变化;这两个氨基酸残基可能在识别DAPA底物和催化反应中发挥作用而不影响结构;与前两个突变不同,D147N突变后并没有检测到催化活性,并且活性位点的结构发生了显著变化,表明该残基在结构上起着重要的作用;在研究的4个氨基酸残基中,只有Y144和R253在DAPA合酶的氨基酸序列中严格保守,证明催化过程中对催化作用至关重要的活性位点氨基酸残基不一定保守,表明在进化过程中,同一酶可能会出现不同的高效催化解决方案。
脱硫生物素合酶(DTBS, EC: 6.3.3.3)催化ATP、7,8-氨基壬酸(DAPA)和CO2,生成ADP、磷酸和脱硫生物素,这是生物素合成途径上的倒数第2步。Salaemae等[72]研究发现,MtDTBS (结核分枝杆菌来源)不仅可以利用ATP、CTP、GTP、ITP、TTP或UTP,且具有相似的Km值和kcat值,与其他DTBS同源物种主要通过嘌呤碱基和关键天冬氨酸的羧基酰胺侧链之间的氢键结合而优先结合ATP的情况不同,为了探究其中差异,作者通过对大肠杆菌DTBS中的D175进行了半理性设计,发现了175调控的门控作用,排除了除ATP外的其他核苷酸三磷酸的结合。这项研究为MtDTBS具有广泛的核苷酸特异性提供了证据,也为ATP的特异性结合提供了设计思路。
1.1.7 维生素B9维生素B9,亦称叶酸,属于水溶性B族维生素[73]。叶酸普遍以聚谷氨酸形式存在,其生物活性形式为四氢叶酸[74],在某些器官和骨髓的细胞分裂和创造过程中发挥重要作用。叶酸的一个关键功能是有助于子宫内胎儿的脊髓和神经系统的形成。与其他B族维生素相似,叶酸有助于促进体内能量的生成[73]。VB9的生物合成需要通过磷酸戊糖途径生成PRPP,再通过3种酶催化得到GTP,然后通过多种酶催化得到关键前体7,8-二氢蝶酸(dihydropteroate) (此处的前体也可以通过分支酸由两种酶催化得来),然后通过一种酶生成叶酸的活性形式四氢叶酸(图 1,灰色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素B9的生物合成主要通过三种途径进行:第一种途径起始于GTP,涉及10种酶;第二种途径由ribA和trpF基因介导,涉及9种酶;第三种途径由磷酸核糖焦磷酸合酶(PTPS)介导,涉及7种酶,总计涉及14种不同的酶。其中酵母菌(Ascoidea gossypii)是一种丝状真菌,其天然菌种可合成40 μg/L的叶酸,经代谢工程处理后可达6 595 μg/L,是当时报道的最高值[75]。此外,Schmitz等[76]发现一个名为“Power-to-Protein”的两阶段技术,该技术利用可再生电力和二氧化碳来生产微生物蛋白的同时可以生产6.7 mg/100 g生物质,实现了零或负的净碳排放。Blancquaert等[77]通过过表达限速酶氨基脱氧柯里斯酸合成酶(aminodeoxychorismate synthase, ADCS)和GTP环化水解酶Ⅰ(GTPCHI),成功将水稻中的叶酸含量提高了150倍,并增强了其在储存期间的稳定性。
目前,其中10种酶已完成动力学特性研究,4种酶进行过改造。例如,二氢叶酸还原酶(EC: 1.5.1.3, dihydrofolate reductase, DHFR),该酶利用NADPH催化7,8-二氢叶酸(DHF)的立体选择性还原,将其转化为5,6,7,8-四氢叶酸(THF),是维生素B9合成过程中的最后一个酶。为了探究大肠杆菌DHFR中不同环结构(loops)之间的相互作用,以及环结构中残基的删除对酶结构稳定性和催化活性的影响。Horiuchi等[78]通过半理性设计的方法,选择了4个不同环中被认为在催化中具有重要调节作用的残基R52 (αC-βC环)、G67 (βC-βD)、G121 (βF-βG)和A145 (βG-βH),进行单点删除突变体的构建;发现除αC-βC环中的52号突变体缺失没有降低酶的折叠自由能外,其余单残基缺失突变体显示出明显的稳定性降低;此外,双缺失突变体的研究表明αC-βC环与βF-βG环和βG-βH环在稳定性方面存在非加性耦合;对αC-βC环或βC-βD环进行单个缺失会导致酶活性显著降低,证明这些环与活性位点附近的残基之间存在耦合;最终证明这四个环在功能上的相互依赖性,为理解该酶的催化机理提供了实验基础。
7,8-二氢蝶啶醛缩酶(EC: 4.1.2.25, 7,8-dihydropteridine aldehyde dehydratase, DHNP aldolase),该酶催化7,8-二氢蝶啶(DHNP)为6-(羟甲基)-7,8-二氢蝶啶(HP),是维生素B9合成过程中重要的中间步骤。Wang等[79]通过半理性设计的方法,对来自甲烷古菌(Methanocaldococcus jannaschii)的DHNP aldolase进行了研究;结果显示,M. jannaschii的DHNP aldolase在70 ℃下具有高催化能力(kcat/Km值大于105 L/(mol·s)),但在室温下的催化速率为0.07 L/(mol·s)与细菌的DHNP aldolase相当;研究还推测了可能参与催化的关键氨基酸残基,包括E25、H35、Q61和Y78,并通过点突变验证了这些残基对催化活性的重要性;研究提出,在缺乏催化碱基基团赖氨酸的情况下,水分子可能作为碱基体,严格保守的H35和Q61残基用于调节水分子的碱性;此外,Y78被证明对催化过程很重要,Y78F的突变使得kcat/Km值降低了2个数量级;另外,E25在底物结合中发挥重要作用,E25Q的突变导致Km值增加超过25倍;研究揭示了古细菌DHNP aldolase独特的结构和催化机制,不仅丰富了对DHNP aldolase进化轨迹的认识,也为维生素B9生物合成路径的研究提供了参考。
1.1.8 维生素B12维生素B12,也称作钴胺素,是唯一含有金属元素的维生素,属于一类含钴的复杂有机化合物[80],是目前已知的最大且结构最复杂的维生素分子。维生素B12缺乏可导致骨髓增生异常前期综合征,特征为环形铁粒幼细胞形成增多[81],以及恶性贫血、记忆力减退、精神忧郁等疾病。VB12的生物合成首先从2-oxoglutarate出发,通过7个酶的催化,得到尿卟啉原Ⅲ (urogenⅢ),在1种酶的催化下得到前咕啉-2 (precorin2),然后通过10种或11种酶的催化得到钴(Ⅱ)铁血红素a, c二酰胺[Cob(Ⅱ)yrimate a, c-diamide],再通过多种酶的催化得到腺苷基钴胺酸-GDP (adenosylcobinamide-GDP),最后与VB2的代谢产物α-核黄素核糖苷(alpha-ribazole)在一种酶的催化下得到VB12 (图 1,橙色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素B12的生物合成可以分为有氧途径和厌氧途径两种形式,两种途径的主要区别在于Cob(Ⅱ)yrinate diamide前体的合成过程中酶的需求不同,厌氧途径涉及13种酶,而有氧途径涉及11种酶,两种途径在后续合成阶段均需8种酶,总体来看,维生素B12的合成过程涉及31种不同的酶。Liu等[82]利用16S rRNA和全基因组系统发育树分析以及平均核苷酸同一性比较,确认了高产VB12的S305菌株为Ensifer adhaerens;通过转录组测序和KEGG卟啉代谢途径的富集分析,发现了S305菌株中多个基因的显著上调或下调,特别是cobA和cobT基因的过表达与VB12产量增加相关,将cobA和cobT基因整合到S305菌株中后,其VB12产量从176.6 mg/L提高到245.6 mg/L。Kang等[17]通过敲除或部分敲除cysG基因来构建不同的大肠杆菌菌株,以减少内源途径对合成途径的干扰。例如,HBA2菌株(ΔcysG)相比HBA1菌株(未敲除cysG)在合成HBA时显示出更高的产量,这表明通过减少CysG酶的活性,可以提高维生素B12合成途径中HBA的合成效率。
维生素B12生物合成过程中的28种酶已进行动力学特性研究,11种酶经过改造研究。例如,烟酸-核苷酸-二甲基苯并咪唑磷酸核糖基转移酶(nicotinate-nucleotide-dimethylbenzimidazole phosphoribosyltransferase, CobT),该酶通过与底物NaMN前体或NAD+发生反应,将辅酶B12的配体基团1,4-二甲氧基苯(5,6-dimethylbenzimidazole, DMB)活化为α-核糖基苯并咪唑磷酸(α-ribazole-phosphate, α-RP)或α-5,6-二甲基苯并咪唑腺苷酸二核苷酸(α-DMB adenine dinucleotide, α-DAD),这些活化产物是辅酶B12生物合成途径中后续反应所需的中间产物。为了深入了解CobT酶在VB12生物合成中的功能,Claas等[83]通过突变分析发现了一类无法在基因内互补实验中恢复VB12生物合成的CobT突变体(称为M类);为解析M类CobT突变体导致功能丧失的原因,作者结合野生型CobT酶的结构数据,确定了3个M类CobT突变体,并进行了体内和体外的功能分析;结构引导的定点突变实验结果表明,G257D和G171D突变体对5,6-二甲基苯并咪唑(DMB)的亲和力降低,或者DMB进入活性位点的通道受到限制;C160A突变使酶的催化效率降低了80%,最终证明报道的M类CobT突变体在基因内互补实验中无法互补的原因可能是因为蛋白质不稳定,或者两个突变的CobT蛋白质形成二聚体,抑或是由于残余活性非常低。该研究为设计和开发抗菌药物以及改良工业生产中的VB12生物合成过程提供了理论基础和指导。
咕啉类腺苷转移酶(corrinoid adenosyltransferase, CobA)的催化分为3个步骤:(1) 辅基腺苷化的第一步是通过电子转移将辅基中的Co(Ⅲ)还原为Co(Ⅱ)。在存在二氢黄素核苷酸或还原的黄素蛋白的情况下,这个步骤可以非酶催化地发生;(2) Co(Ⅱ)被辅基腺苷转移酶结合,从而使辅基的下位配体被芳香残基所取代。与自由化合物相比,四配位的Co(Ⅱ)中间体的还原电位提高了约250 mV,使其达到生理范围。然后,从自由的二氢黄素或还原的黄素蛋白的辅基中发生第二次单电子转移,将其还原为Co(Ⅰ);(3) Co(Ⅰ)对ATP的腺苷基进行亲核攻击,导致脱氧腺苷基的转移,同时将钴原子氧化为Co(Ⅲ)态。反应涉及2个ATP、2个二氢卟吩酸氨基甲酰胺[Cob(Ⅱ)yrinate a, c diamide]和还原型NADPH-血红素蛋白还原酶(reduced NADPH-hemoprotein reductase),是合成维生素B12的重要步骤。Moore等[84]通过对沙门氏菌属肠杆菌(Salmonella enterica)中CobA酶与ATP、四配位cobalamin和五配位cobalamin形成的复合物的晶体结构进行分析,首次提供了cob(Ⅱ)alamin存在于CobA活性位点的晶体学证据;该结构揭示了一个机制,其中该酶采用闭合构象,并且两个氨基酸残基F91和W93与cobalamin的核苷酸配体DMB相互作用,从而生成临时的四配位cobalamin,这对于adoCbl Co−C键的形成至关重要;通过对F91和W93的体内和体外突变分析验证了活性位点中较大疏水侧链残基的重要作用。这项研究为理解酶催化的cobalamin生物合成和相关代谢途径的反应机制提供了重要见解。
1.1.9 维生素C维生素C,也称为L-抗坏血酸(L-ascorbic acid, LAA),是体内多种酶反应的重要辅助因子[85],也是一种有效的抗氧化剂。维生素C的缺乏会导致免疫力受损,从而更容易受到疾病的感染,甚至可能会导致坏血病等严重的疾病[86]。维生素C的合成有以下两条路径:第一条可以通过葡萄糖-6-磷酸(glucose-6p),在4种酶的催化作用下生成UDP-葡萄糖(UDP-glucose),并在此基础上通过7种酶的催化作用生成维生素C;第二条则是通过果糖-6-磷酸(fructose-6p)在3种酶的催化下生成鸟苷二磷酸-D-甘露糖(GDP-D-mannose, GDD),最后在5种酶的催化下生成维生素C (图 1,黄绿色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素C的生物合成途径包括植物和动物途径,分别涉及5种和7种酶,总计12种不同的酶。高媛等[87]在5 L生物反应器中利用E. coli菌株过表达功能性的山梨糖/山梨酮脱氢酶,并优化了催化条件,以重组表达吡咯喹啉醌(pyrroloquinoline quinone, PQQ),他们采用全细胞催化法生产2-酮戊二酸(2-ketoglutaric acid, 2-KGA),以L-山梨糖为底物,产量达到了72.4 g/L,底物转化率为71.2%,生产强度为1.57 g/(L·h)。Sauer等[88]通过过表达酿酒酵母中的D-阿拉伯糖脱氢酶和D-阿拉伯糖酸内酯氧化酶,成功提升了维生素C的产量。
在维生素C的合成研究中,已有10种酶的动力学特性得到详细的研究,6种酶经过改造以提高其效率或性能。例如,UDP-葡萄糖6-脱氢酶(UDP-glucose 6-dehydrogenase, UGDH),该酶催化UDP-葡萄糖与水分子以及2分子NAD+发生反应,生成UDP-葡萄糖醛酸、2分子NADH和2分子氢离子,是合成维生素C过程中的重要前体物质。为了深入了解UGDH的结构与功能之间的关系,并探索不同构象对其活性和细胞功能的影响,Hyde等[89]通过半理性设计的方法引入了突变体,获得了除天然四聚体形式外的二聚体(T325D)和六聚体(T325A)的形式,并对其活性和细胞功能进行了评估;作者发现,只有动态的UGDH六聚体能够支持细胞的正常功能,而二聚体形式的UGDH在体外和体内的活性都降低。此外,研究还发现,通过稳定UGDH的六聚体结构可以调控其活性,这可能为癌症和其他疾病的治疗提供了新的选择。这些研究都为深入了解UGDH的聚合形式提供了可靠的基础。
L-半乳糖内酯脱氢酶(EC: 1.3.2.3, galactose dehydrogenase, GALDH),该酶催化L-半乳糖酮-1,4-内酯的氧化反应,将其转化为L-抗坏血酸,在植物中,这是维生素C合成的最后一步。为了研究GALDH的催化机制,Leferink等[90]通过位点导向的突变实验,阐明了该酶活性中心两个关键氨基酸残基的作用,揭示了该酶的催化机制,并提供了维生素C生物合成途径的证据;研究发现,R388对稳定FAD辅因子的阴离子形式非常关键,替换精氨酸388会大大降低GALDH与亚硫酸盐形成加合物的能力,表明精氨酸388靠近FAD的异卟啉环可稳定FAD的阴离子半氧化还原态;此外,E386参与底物的结合和正确的催化配位。用天冬氨酸取代谷氨酸386后,GALDH对L-半乳糖-γ-内酯的专一性降低,对L-古洛糖-γ-内酯的催化效率提高。该研究揭示了GALDH催化机理中两个氨基酸残基的重要作用,为维生素C的生物合成路径提供了证据,这项研究有助于理解维生素C在植物线粒体功能和抗氧化系统中的重要作用。
1.2 脂溶性维生素 1.2.1 维生素A维生素A是一种重要的脂溶性维生素,也被称为视黄醇。它在人体中发挥着多种关键的生理功能。维生素A有两种存在形式:预维生素A(如β-胡萝卜素)和活性形式的维生素A(视黄醇)。维生素A对于人类和动物是必需的营养素,它们主要通过饮食摄取。维生素A的来源分为两类:一是动物源食品,这些食品中含有直接可用的视黄醇或其酯化形式,或是可以转化为视黄醇的类胡萝卜素;二是植物源食品,尤其是富含类胡萝卜素的水果和蔬菜,这些类胡萝卜素在体内可被转化为活性形式的维生素A。维生素A的合成过程首先涉及两个关键前体:丙酮酸(pyruvate)和D-葡萄糖-3-磷酸(D-glyceraldehyde-3-phosphate)。在1-脱氧-D-木糖醇-5-磷酸合酶(1-deoxy-D-xylulose 5-phosphate synthase, DOXP synthase)的作用下,这两个分子通过缩合反应形成中间产物1-脱氧-D-木糖醇-5-磷酸(1-deoxy-D-xylulose 5-phosphate, DOXP)。接着,DOXP经过一系列反应转化为长链异戊烯二磷酸,再经过另外10种酶的催化反应,最终生成β-胡萝卜素(图 1,紫红色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。值得注意的是,β-胡萝卜素具有两种异构形式:全反式和顺式。胡萝卜素的两种异构体,即全反式和部分反式异构体,它们可以通过同一种酶——β-胡萝卜素合酶(β-carotene synthase)催化完成。β-胡萝卜素合酶通过特定的反应机制,将戊二烯基二萜二磷酸(geranylgeranyl diphosphate, GGPP)的碳链逐渐延长并形成双键,从而合成β-胡萝卜素的结构。在这个合成过程中,β-胡萝卜素合酶并不直接控制异构体的形成,而是通过反应条件、底物浓度和环境因素等来调节异构体的比例[91]。
维生素A的生物合成涉及6种酶。Larroude等[92]在解脂耶氏酵母(Yarrowia lipolytica)中过表达异源胡萝卜素合成酶,从而高效产生β-胡萝卜素;经过筛选最佳启动子组合得到的工程菌株在发酵过程中产量为1.5 g/L;通过优化发酵条件,并采用补料分批发酵方式,进一步提高了β-胡萝卜素的产量,达到6.5 g/L。Yoon等[93]通过基因表达差异、酶活性比较、代谢途径优化、温度敏感性的实验证明了CrtE、CrtB和CrtI为类胡萝卜素途径中的限速酶,其活性和表达水平直接影响了整个途径的代谢通量和最终产物的产量,通过优化CrtE、CrtB和CrtI的表达和功能,可以提高类胡萝卜素的生物合成效率。
目前关于维生素A合成所涉及的酶动力学特性研究较为有限,其中3种酶的动力学性质已被研究,仅1种酶进行过改造研究。例如,ζ-胡萝卜素去饱和酶(EC: 1.3.5.6, CrtI)催化7,9,9-tricis-neurosporene的脱氢反应,为了揭示类胡萝卜素生物合成路径进化过程中的CrtI酶功能改变机制,Stickforth等[94]发现在凝胶柱状菌(Rubrivivax gelatinosus)等紫色细菌中,该酶能够同时催化三步和四步脱饱和反应,产生神经孢烯和番茄红素,从而在类胡萝卜素合成途径中形成双重分支;研究人员通过非理性设计的方法构建了两个不同的突变库来筛选突变体,最后发现L208氨基酸的蜕变对酶的功能、特异性和选择性起着关键的作用,208位氨基酸位于跨膜区的高度亲疏水环境中,根据topological模型预测,该区域可能靠近活性中心,并与底物产生亲水性相互作用;对208位氨基酸的不同替换(丙氨酸和苯丙氨酸)导致CrtI对神经孢氮素的亲和力发生显著改变,这与产物分布的改变一致。这项研究首先满足了对指定产物的CrtI突变体的获得需求,同时从蛋白功能演化和分子设计的角度深入研究了单点突变对酶催化特性改变的原理,为类胡萝卜素和维生素A的生物合成调控提供了参考。
1.2.2 维生素D维生素D是一种脂溶性维生素,对促进人体钙和磷的吸收及利用至关重要,并有助于维持血钙和血磷的正常水平,有助于骨骼的形成、和发育。维生素D还参与调节骨骼中钙和磷的平衡,促进成骨细胞的活动,具有抗佝偻病的活性。其中,胆钙化醇(维生素D3)和麦角钙化醇(维生素D2)是维生素D的两种主要形式[95]。维生素D3主要在阳光的作用下在人体皮肤中合成,而维生素D2则需要完全从外部获取。维生素D3的合成首先经过丙酮酸和D-葡萄糖-3-磷酸通过DOXP合酶的催化发生缩合反应,形成DOXP。接着,经过6种酶的催化反应,形成(S)-2,3-epoxysqualene。然后,通过另外12种酶的作用,从7-脱氢胆固醇(7-dehydrocholesterol, 7-DHC)生成维生素D3。此外,通过紫外线光子触发的光化学反应,将7-DHC通过3种酶的转化,形成前维生素D3。最终,维生素D3被运输至肝脏,由CYP2R1酶催化转化为25-羟基维生素D3 [25(OH)D3],这是维生素D3的主要循环形式,并被进一步运输至肾脏及其他组织以供利用[96-97]。维生素D2的合成前体是麦角固醇[98],与维生素D3一样需要经过中间体(S)-2,3-epoxysqualene,随后经过15种酶生成,这一过程不需要中间体7-DHC (图 1,蓝绿色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素D的生物合成分为维生素D2和维生素D3两种形式,分别涉及18种和16种酶,且最终均需光照以转化为其活性形式,此过程中总共涉及23种不同的酶。Tan等[99]通过优化麦角甾醇的发酵参数,发现溶解氧可作为有效的酵母补料分批发酵的控制参数。当溶解氧控制在(12±1)%时,采用脉冲补料法,麦角甾醇的总收率可达1 160 mg/L。
在维生素D合成途径中,已有7种酶的动力学特性研究完成,同时有2种酶进行过改造研究。例如,甾醇24-C-甲基转移酶(EC: 2.1.1.41, 24-SMT)催化S-腺苷甲硫氨酸(S-adenosyl-L-methionine, SAM)和酵母甾醇(zymosterol)之间的化学反应。在这个过程中,SAM作为甲基供体,将甲基基团转移给酵母甾醇,形成S-腺苷L-同型半胱氨酸(S-adenosyl-L-homocysteine, SAH)和胆甾固醇(fecosterol)。Liu等[100]对布氏锥虫(Trypanosoma brucei)来源的SMT (TbSMT)进行了研究,并揭示了其催化机制及与其他甾醇C-24-甲基化酶的差异;通过探讨该酶催化的多种麦角甾烷类化合物的生成途径,并通过突变实验和分子模拟揭示了关键酪氨酸残基(Y66)在催化反应中的作用,研究结果表明,该酶与其他已知的24-甲基转移酶具有不同的活性位点结构和更大的底物适应范围,并推测TbSMT酶可能是一种原始类型的24-甲基转移酶。该研究为开发寄生虫特异性抑制剂奠定了基础,也拓展了对甾醇C-24-甲基化酶家族的认识。
维生素D3羟化酶(EC: 1.14.14.24, Vdh)催化维生素D3转化为25-羟基维生素D3 (calcidiol,维生素D3的活性形式),这是一种血浆中最丰富的维生素D形式。Fujii等[101]从放线菌(Pseudonocardia autotrophica)中分离获得一种可以水解维生素D3的新酶——维生素D3羟化酶,并对其生化性质进行了研究;研究人员首先确定了该酶的氨基酸序列并克隆了编码该酶的基因vdh,并表征了Vdh的酶活性参数,发现其对维生素D3的25位羟基化活性高于当时已报道的其他细胞色素P450酶;随后通过非理性设计的方法(随机突变+饱和突变)获得了4个单点最优的突变(T70R、V156S、E216A和E384R),并进行组合评估和氨基酸替换,最终得到Vdh-K1突变体(T70R/V156L/E216M/E384R);Vdh-K1突变体的羟化活性达到野生型的21.6倍,Vmax值提高6倍以上,达到(19 823±307) mmol/(min·mol),Km值上升至(32.3±1.5) μmol/L。该研究为维生素D3活性代谢产物的微生物转化合成提供了新的见解。
1.2.3 维生素E维生素E包括8种异构体分子,统称生育酚或生育三烯酚,仅由光合生物如植物和藻类合成[102]。维生素E缺乏可损害T细胞和B细胞的免疫功能[98]。生育酚有4中不同的化合物形式,分别称为α、β、γ和δ生育酚[12]。它在人体内不能合成,因此只能通过饮食方式进行补充。尽管植物中的维生素E以生育酚混合物形式存在,但α-生育酚因在人体内具有优先保留和分布的特性,展现出最高的生物活性[103]。α-生育酚和β-生育酚都可以经过D-赤藓糖-4-磷酸(E4P)和磷酸烯醇式丙酮酸(phosphoenolpyruvate, PEP)在多个酶的催化下生成分支酸(chorismate),随后通过5个酶的催化生成邻苯二酚(homogentisate),邻苯二酚与GGPP共同生成6-香叶基-2-甲基苯-1,4-二醇(2-methyl-3-gallol-1,4-benzoquinone, MGGBQ),最后α-生育酚需要经过3种酶进行合成,而β-生育酚只需要2种酶催化即可生成。此外,α-生育酚还有另外一条合成路径,Homogentisate并非与GGPP共同生成,而是与GGPP的下一步酶催化形成叶酸单甲酯二磷酸(phytyl-pp)。phytyl-pp与其他化合物共同生成2-甲基-6-叶酸单甲酯喹啉(2-methyl-6-phytylquimol),并通过2种或3种酶的催化作用最终形成α-生育酚(图 1,黄色;国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素E的生物合成主要分为α-生育酚(alpha-tocopherol)和β-生育酚(beta-tocopherol)的合成,其中α-生育酚需要4种酶进行合成,β-生育酚需要3种酶进行合成。Shen等[104]通过将来自光合生物的异源基因与内源性莽草酸途径和甲羟戊酸途径相结合,构建了生产生育三烯醇的酿酒酵母;经过识别和消除代谢瓶颈并增加前体供应,并设计了一个冷冲击触发温度控制系统,有效地控制了两阶段发酵,最终生产出320 mg/L的生育三烯醇。在7种相关酶类中,编号为EC: 2.5.1.116、EC: 2.1.1.95和EC: 2.5.1.115的3种酶已完成动力学性质研究,然而目前还没有酶进行过动力学性质改造。这可能与目前维生素E的生产方式主要以化学法和植物油提取法为主,且生物发酵法进展缓慢有关。但是,通过对生产酶进行组合优化,利用生物法合成其关键前体法尼烯已经有了巨大突破,可实现通过生物+化学半合成方法进行维生素E的生产[105-106]。因此,未来完全有可能通过生物催化的方式进行维生素E的生产,并且在这些途径中所涉及酶的改造上,仍有很大的提升空间。
1.2.4 维生素K维生素K是一种脂溶性维生素,对维持骨骼健康、血管钙化调控及血液凝固等生理功能至关重要[107]。维生素K主要有3种生物活性形式,其中2种是从天然来源获得的维生素K1 (叶绿醌/植酮二酮)和维生素K2 (甲基萘醌,MK7),另一种是由实验室合成的维生素K3 (甲萘醌)。维生素K有两种膳食来源:植物来源和内源性来源。叶绿醌是维生素K的主要膳食来源,存在于所有光合植物中[108-109],甲基萘醌主要由肠道微生物群中存在的不同细菌合成。人体每日所需的维生素K约有50%来源于内源性合成。肠杆菌、韦荣球菌、肠球菌、脆弱拟杆菌和其他类杆菌是合成MK的主要微生物[110]。根据与甲基萘醌环连接的类异戊二烯单元的数量,可分为14种同工型[111]。其中,甲萘醌-7 (menaquinone-7, MK7)是最有效的维生素K亚型,在循环中具有很长的半衰期。此外,MK7存在全顺式、全反式和顺反式形式,但只有全反式形式才具有生物活性。维生素K1 (VK1)的合成需要在E4P和PEP在多个酶的催化下生成chorismate。随后,chorismate经过7种酶的催化作用生成1,4-二羟基-2-萘酸(1,4-dihydroxy-2-naphthoate, DHNA)。最后,DHNA与叶酸单甲酯二磷酸(phytyl-pp)在3种酶的催化下合成VK1。VK2的合成与VK1的生产过程类似,只在生成DHNA之后,需要经过2种酶的催化作用将DHNA与聚异戊二烯磷酸(polyprenyl-PP)合并即形成VK2 (图 1,蓝色;附图见国家微生物科学数据中心,登录号:SUB1720761294457)。
维生素K的生物合成包括维生素K1和维生素K2两种形式,分别涉及11种和9种酶,总计13种不同的酶。Xu等[112]通过改造异戊烯焦磷酸(isopentenyl diphosphate, IPP)代谢途径,并应用遗传编码的双输入多输出(tunable input multi-output, TIMO)遗传回路来动态控制代谢通量,成功地在枯草杆菌(Bacillus subtilis)中提高了萜类化合物生物合成效率,在50 L生物反应器中,MK-7的产量达到1 549.6 mg/L。Kong等[113]发现过表达E. coli中的MenA、MenB、MenC、MenD、MenE、MenF或UbiE限速酶时,MK-8含量可增加至原有的2倍。
在维生素K合成途径中,6种酶的动力学特性已得到研究,5种酶已进行过改造。例如,2-琥珀酰-5-烯醇丙酮酰-6-羟基-3-环己烯-1-羧酸合酶(EC: 2.2.1.9, MenD)是一种依赖硫胺素二磷酸(thiamine diphosphate, ThDP)的酶,催化α-酮戊二酸和异恶唑酸甲酯转化为2-琥珀酰基-5-烯酮丙酸-6-羟基-3-环己烯-1-羧酸(2-succinyl-5-enolpyruvyl-6-hydroxy-3-cyclohexene-1-carboxylic acid, SEPHCHC),是维生素K2合成途径中的第2个酶。为了阐明MenD酶催化反应中两个高度保守的精氨酸残基(R395和R413)的作用机理,解析它们在调节该酶催化中硫胺二磷酸中间体的构象和稳定性方面的分子机制,Qin等[114]通过半理性设计方法,利用定点突变、酶动力学实验和蛋白质晶体学等手段对大肠杆菌来源的MenD酶进行了深入研究;结果表明,这些保守残基通过离子型氢键作用定向中间体中的琥珀酰基团,从而使其取得活性构象;当这种相互作用被适度削弱时,产生的突变蛋白仍具有催化活性,但琥珀酰基团在后脱羧中间体中可以取得活性或非活性构象;当这种氢键相互作用被强烈削弱时,琥珀酰基团相对于天然中间体反转了180°,导致反应中心的立体化学发生改变,从而失去催化活性,该研究深入阐明了MenD这类新型ThDP依赖性酶的催化机制,为开发其抑制剂奠定了理论基础。此外,由于MenD在多种致病菌的维生素K生物合成中不可或缺,这项研究也为开发新型抗生素提供了潜在靶点。同时,该研究有助于理解ThDP依赖型酶的催化机制,并为其他酶的工程应用提供理论指导。
邻琥珀酰苯甲酸酯-CoA连接酶(EC: 6.2.1.26, MenE),通过两步催化将o-succinylbenzoyl (OSB)催化成OSB-CoA。在第一步的腺苷酸化反应中,OSB底物与ATP反应形成腺苷酸中间体OSB-AMP;随后,在第二步的硫酯化反应中,该腺苷酸中间体与辅酶A反应形成OSB-CoA,这是维生素K合成过程中重要的中间产物。为了探究腺苷酸化酶类酶中腺苷酸化活性位点的形成机制,特别是在枯草芽孢杆菌中OSB-CoA合酶的作用机制,Chen等[115]通过对磷酸盖帽(P-loop)区域的丝氨酸/苏氨酸残基(T152, T155, T156)进行丙氨酸突变,并对OSB-CoA合酶的晶体结构进行研究,发现ATP与腺苷酸化酶之间的相互作用模式;研究人员还对多个保守氨基酸进行突变研究,其中两个保守的甘氨酸残基(G154, G157)被替换成脯氨酸,研究引入了立体位阻对P-loop结构和活性的影响,连接两个结构域的精氨酸残基R382被替换成丙氨酸或赖氨酸,研究结构域整体特征与ATP相互作用对活性的贡献。结果表明,将甘氨酸突变成脯氨酸对活性影响较小,说明小的立体位阻对活性影响不大,主要会通过氢键作用影响P-loop构象的变化;P-loop区的苏氨酸突变为丙氨酸后,kcat值降低2个数量级,Km值不同程度升高,说明这些残基不仅参与ATP结合,也影响腺苷化反应;R382的突变导致对ATP和OSB都有类似kcat和kcat/Km值的下降,但对CoA-SH的Km值升高更明显,说明该残基影响两个步骤的催化;这些发现对于理解腺苷酸化酶的催化机制以及对维生素K生物合成的作用具有重要意义[115]。该研究为开发新的抗菌剂,尤其是针对依赖维生素K的微生物病原体感染的治疗提供了潜在的目标和策略。
1,4-二羟基-2-萘酰辅酶A合酶(EC: 4.1.3.36, MenB)催化OSB-CoA发生分子内克莱森缩合反应,形成1,4-二羟基-2-萘酰辅酶A (1,4-dihydroxy-2-naphthoyl-CoA)。Li等[116]详细阐述了目前对MenB反应机制理解中存在的困惑,包括质子的抽离、碳碳键的形成以及四面体中间体的稳定等问题;主要原因在于目前MenB晶体结构的活性部位结序混乱,无法观察底物与酶的相互作用;为了解决这个问题,研究人员设计合成了一个OSB-NCoA的底物类似物,其中硫酯键被氨基取代,使反应过程停滞在质子抽离这一步;随后的结构生物学研究发现,这个底物类似物能使MenB的活性部位结构完整有序,研究人员得以首次观察到底物与酶完整的交互作用模式,通过结合位点导向突变和底物类似物的研究,提出了一个详细的MenB催化机制:该机制中,两个保守的酪氨酸残基(Y97, Y258)通过与底物的羧基进行质子转移而起到关键作用,不仅可以促进碳负离子的形成,也可以稳定了碳碳键形成后生成的四面体中间体。这项研究利用稳定的底物类似物成功地解析了MenB催化机制中多个重要问题,为维生素K的生物合成过程提供了详细的分子级理解,也为酶超家族内通过类似Claisen缩合反应的酶的研究提供了思路。
目前已知的维生素代表性高产量的菌种实例如表 1所示,维生素产品途径中酶的改造实例如表 2所示。
| Vitamins | Species | Method | Yield | References |
| VB1 | E. coli | ΔthiL, ΔthiN, ΔykoD and ΔyuaJ | 1.27 mg/L (extracellular) and 0.12 mg/L (intracellular) | [22] |
| VB2 | B. subtilis | Random mutations | 24.30 g/L | [31] |
| VB3 | E. coli | Expression of high-molecular-weight nitrile hydratase from Rhodococcus rhodochrous J1 | 508.00 g/L | [39] |
| VB5 | E. coli | Expression of six PS coding genes | 97.10 g/L | [48] |
| VB6 | E. coli | StAPase and EcPNPO were used as pathway enzymes, and pyridoxine (PN) and pyrophosphate (PPi) were used as substrates to synthesize PLP | 15.00 g/L | [60] |
| VB7 | Pseudomonas mutabilis | Expression vectors were introduced and fermentation conditions were optimized | 15.00 g/L | [68] |
| VB9 | Ascoidea gossypii | Metabolic engineering | 6.60 mg/L | [75] |
| VB12 | Ensifer adhaerens | cobAOE and cobTOE | 245.60 mg/L | [82] |
| VC | E. coli | Overexpression of functional sorbitose/sorbitone dehydrogenase | 72.40 g/L | [87] |
| VA | Yarrowia lipolytica | Overexpression of heterocarotene synthetase | 6.50 g/L | [92] |
| VD | Ergosterol | Optimization of fermentation parameters | 1.16 g/L | [99] |
| VE | Saccharomyces cerevisiae | Metabolic optimization, design of temperature control system | 0.32 g/L | [104] |
| VK | B. subtilis | A genetically encoded dual-input, multiple-output (TIMO) genetic circuit is applied to dynamically control metabolic flux | 1.55 g/L | [112] |
| OE: Represents gene overexpression. | ||||
| Vitamins | Total | R_num | M_num | EC number | Species | Method | Domain | Objectives | References |
| VB1 | 16 | 9 | 4 | 1.4.3.19 | B. subtilis | Saturation mutation | Active center | specificity | [24] |
| 1.4.3.19 | B. subtilis | Mutant screening | Active center | stability | [25] | ||||
| 2.7.6.2 | Human | Site-directed mutagenesis | Active center | mechanism | [26] | ||||
| VB2 | 11 | 6 | 4 | 3.5.4.25 | B. subtilis | Random screening | Whole protein | activity | [34] |
| 2.5.1.78 | B. subtilis | Site-directed mutagenesis | Active center | activity | [35] | ||||
| 2.7.1.26 2.7.7.2 |
Corynebacterium ammoniagenes | Site-directed mutagenesis | Active center | mechanism | [36] | ||||
| VB3 | 10 | 9 | 7 | 2.7.7.18 | E. coli | Saturation mutation | Active center | activity | [41] |
| 3.2.2.6 | Cow | Site-directed mutagenesis | Active center | mechanism | [42] | ||||
| VB5 | 7 | 7 | 5 | 4.1.1.11 | B. subtilis | Error-prone PCR | Whole protein | activity/stability | [51] |
| 6.3.2.1 | Arabidopsis thaliana and E. coli | Sequence comparison | Subunit contact area | mechanism | [52] | ||||
| VB6 | 8 | 8 | 3 | 2.6.99.2 | E. coli | Computational design | Active center | activity/yield | [16] |
| 1.4.3.5 | E. coli | Site-directed mutagenesis | Active center | mechanism | [62] | ||||
| 4.3.3.6 | B. subtilis | Site-directed mutagenesis | Active center | mechanism | [63] | ||||
| VB7 | 9 | 5 | 2 | 2.6.1.62 | E. coli | Sequence comparison | Active center | mechanism | [71] |
| 6.3.3.3 | E. coli | Site-directed mutagenesis | Active center | specificity | [72] | ||||
| VB9 | 10 | 10 | 4 | 1.5.1.3 | E. coli | Site-directed mutagenesis | Distal residue | activity/stability | [78] |
| 4.1.2.25 | Archaea | Site-directed mutagenesis | Active center | mechanism | [79] | ||||
| VB12 | 31 | 28 | 11 | 2.4.2.21 | M. jannaschii | Site-directed mutagenesis | Active center | mechanism | [83] |
| 2.5.1.17 | S. enterica | Site-directed mutagenesis | Active center | mechanism | [84] | ||||
| VC | 12 | 10 | 6 | 1.1.1.22 | Human | Site-directed mutagenesis | Active center | mechanism | [89] |
| 1.3.2.3 | A. thaliana | Site-directed mutagenesis | Active center | mechanism | [90] | ||||
| VA | 6 | 3 | 1 | 1.3.5.6 | R. gelatinosus | Random screening | Whole protein | mechanism/specificity | [94] |
| VD | 13 | 7 | 2 | 2.1.1.41 | T. brucei | Site-directed mutagenesis | Active center | mechanism | [100] |
| 1.14.14.24 | P. autotrophica | Random mutation/saturation mutation | Whole protein | activity | [101] | ||||
| VK | 13 | 6 | 5 | 2.2.1.9 | E. coli | Site-directed mutagenesis | Active center | mechanism | [114] |
| 6.2.1.26 | B. subtilis | Site-directed mutagenesis | Active center | mechanism | [115] | ||||
| 4.1.3.36 | E. coli | Site-directed mutagenesis | Active center | mechanism | [116] | ||||
| Total: The total number of enzymes in all synthetic pathways of a product, referencing pathway information from the KEGG database. R_num: The number of enzymes within the pathway that have been studied for their kinetics. M_num: The number of enzymes that have been subject to research on the modification of enzymatic parameters. The statistical data on enzyme kinetics studies are sourced from the BRENDA database. | |||||||||
前文详细描述了维生素的生物合成途径以及其中一部分已被改造的酶的动力学性质。然而,这些相关酶的动力学数据尚不足以覆盖整条途径,且已知数据的研究大都来源于多个物种,无法形成对某个物种的整体评价,也并不能很好地反映途径间的关系。因此,为了深入理解影响维生素生产的要素,并探索如何通过科学有效的手段进一步优化这些宝贵的生物合成途径,本课题组对这些途径中的酶学性质进行了一系列更为细致的分析比较。包括对现有真实数据的进一步挖掘和使用先进的深度学习算法对所有酶进行预测和分析,旨在识别出不同产品合成途径中的潜在瓶颈酶和高效催化步骤以及高低产产品间酶学性质的差异等信息,为进一步的代谢工程和合成生物学应用提供指导。通过揭示维生素生产相关酶的动力学参数(如kcat和Km)与代谢途径的调控关系,识别并优化代谢途径中的瓶颈酶,提升其表达量或活性,从而增加整体产量;其次,动力学参数有助于指导调节酶的相对表达量、平衡代谢流,避免中间产物积累和反馈抑制;最终,研究酶的动力学参数有助于筛选催化效率高且底物亲和力适宜的酶,进而通过定向进化或遗传改造提升维生素合成效率。
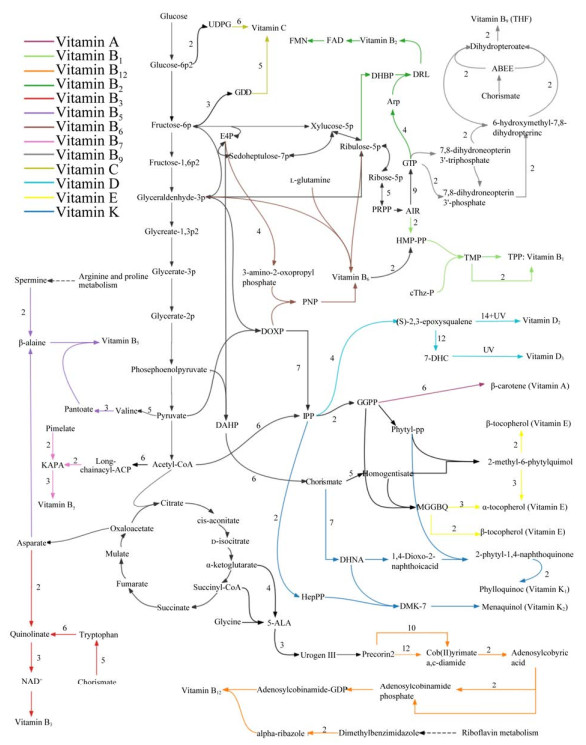
首先,基于KEGG数据库中维生素合成途径的185种对应酶,从BRENDA数据库[117]中收集了其所对应的所有动力学数据(包括kcat、Km和kcat/Km)。包含突变体和不同实验条件的数据共约5 917条,其中kcat有1 640条,Km有3 321条,kcat/Km有956条(国家微生物科学数据中心,登录号:SUB1720761294457)。为了排除突变数据在不同产品中的差异研究,影响对途径酶天然催化能力的判断,仅对野生型数据(多个条件取最高值)进行进一步分析。野生型数据分析显示,维生素合成的途径中虽然已经有约390条kcat数据,但这些数据的来源高达117个物种,仅涉及90种酶;Km数据1 126条,涉及263个物种,118个酶;kcat/Km数据209条,涉及80个物种,61个酶(图 2)。这些酶的kcat数值普遍较低,主要集中在个位数左右,Km数值也相对较低,大概集中在0.01–1 mmol/L之间。为了更好地了解维生素途径中相关酶的动力学数据所处的催化水平,将其与维持细胞代谢的主途径,例如EMP途径进行对比(野生型数据包括kcat 158条,Km 568条,kcat/Km 79条,223个物种和19种酶),发现kcat值有显著差异,而Km值则相近。EMP途径中kcat值主要集中在百位数左右,比维生素途径酶的整体数值高1–2个数量级(图 2)。这一现象可能是与EMP作为基本代谢途径,需要高效酶以迅速供应能量与代谢中间体来满足细胞环境的快速变化;而维生素在生物体内的需求量较小,进化可能倾向于选择较低催化效率的酶,以防过量合成导致的代谢负担或潜在毒性有关。
|
| 图 2 维生素途径和EMP途径中已报道的酶的野生型数据比较 Fig. 2 Comparison of reported wild-type data for enzymes in the vitamin pathway and the EMP pathway. A: Gaussian distribution graphs of real wild-type data for all enzymes in the vitamin pathway in terms of kcat, Km, and kcat/Km. B: Gaussian distribution graphs of real wild-type data for all enzymes in the EMP pathway in terms of kcat, Km, and kcat/Km. The x-axis represents the log values of the real data, and the y-axis represents the density values of the Gaussian distribution. |
| |
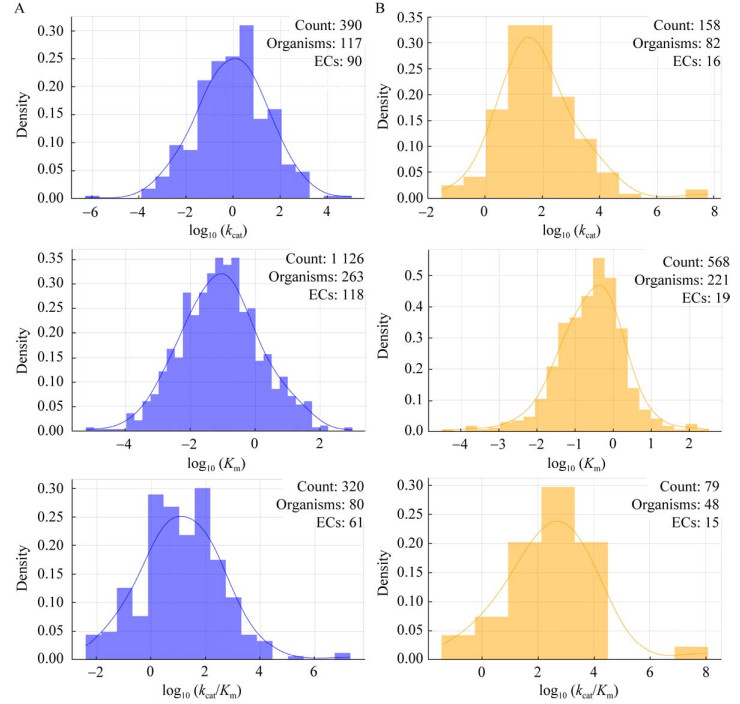
此外,不同维生素之间的动力学特性存在显著差异。比如,维生素B12和维生素B9的kcat值的分布比维生素B3和维生素C低一个数量级左右;但维生素B12和维生素B9在Km上的分布比维生素B3和维生素C也会低一个数量级左右(图 3)。因此,在合成特定生物产品的过程中,必须基于各物种酶的特性及其需求,调整生物工程策略,尤其是在产量逐步提升的过程中,由于胞内的通量提升,底物的相对浓度较高,可能Km已经不再是限制催化的瓶颈[34],反而需要更加关注kcat,加快产品转化的速率,或者通过代谢工程手段提升相关酶的表达水平等。
|
| 图 3 不同维生素产品途径酶之间野生型的已报道数据比较 Fig. 3 Comparison of reported wild-type data among enzymes in different vitamin production pathways. A: Gaussian distribution graph of reported wild-type kcat data among enzymes in 13 vitamin synthesis pathways. B: Gaussian distribution graph of reported wild-type Km data among enzymes in 13 vitamin synthesis pathways. The x-axis represents the log values of the real data, and the y-axis represents the count values of the Gaussian distribution. The top right corner of each subplot indicates the total number of data points in the pathway, the number of species involved, and the number of enzymes. |
| |
进一步分析Km数据(数据量最大)可以看出,平均每物种涉及的反应数量不超过5条,由于每个酶可能还会有多个底物存在的情况,所以每个物种涉及的酶的平均数据会更少。统计的13种维生素合成途径酶的平均数大于14个,所以对于已有研究的单一物种中单个途径酶的研究平均数不足1/3。这对于判断整个物种中维生素途径的代谢强弱和关键限速酶的确定带来了很大的挑战。随着深度学习等方法的发展,利用已知数据建立的模型,可以对未知反应进行预测,比如Li等[118]开发的DLKcat方法和Yu等[119]开发的UniKP方法,在酶反应动力学属性的预测上都达到了很高的水准。尤其是UniKP方法在kcat、Km和kcat/Km数据上都表现出色。因此,本研究采用UniKP方法对特定的常见物种(例如大肠杆菌和枯草芽孢杆菌)中所有反应进行酶学数据预测分析。另外,由于上述物种无法天然合成维生素A、D和E,还选择了一些其他来源的物种作为预测目标。
通过获取已报道或预测的酶学数据,可以根据kcat值和Km值识别瓶颈步骤,推断代谢优化改造的首选目标;根据kcat值和Km值多方位评估酶对底物的催化能力和亲和力,指导底物浓度的需求供给;对比不同物种中同一途径酶动力学参数以指导底盘选择等。与已报道的多个物种的数据相比,枯草芽孢杆菌和大肠杆菌来源的预测数据在kcat上更加集中,大多在1–100 s−1之间,而真实值由于物种较多,大多在0.001–1 000 s−1之间。Km以及kcat/Km值也是如此,Km在预测值中大都集中在0.01–1 mmol/L之间,而真实值会在0.0001–100 mmol/L之间;kcat/Km在预测值中大都集中在1–1 000 L/(mmol·s),真实值会在0.1–10 000 L/(mmol·s)之间(图 4)。因此,物种间的同工酶会存在很大的差异,例如,预测分析显示,在枯草芽孢杆菌中维生素B5合成相关酶的kcat值普遍高于大肠杆菌,但两者的Km值相近,因此,枯草芽孢杆菌在天然情况下可能比大肠杆菌在生产维生素B5的能力上更具优势,而且目前通过代谢工程改造手段得到的维生素B5的从头发酵菌株,枯草芽孢杆菌的产量也比大肠杆菌来源的高(枯草芽孢杆菌最高产量为86 g/L[43],大肠杆菌最高产量为28.5 g/L[45])。需要说明的是,它们的代谢改造手段和基因并不一致,但都没有对途径酶进行过进化改造,或许进一步的改造可以使其产量更高。同样,维生素B1也存在这样的现象,在枯草芽孢杆菌中的整体kcat水平比大肠杆菌来源的酶要高,但Km值却很类似(图 4)。因此,枯草芽孢杆菌在天然状态下可能比大肠杆菌在生产维生素B1方面更具催化优势。此外,维生素B6的途径酶在枯草芽孢杆菌和大肠杆菌中的差异较大,但这个差别主要源于途径的差异,枯草芽孢杆菌为DXP非依赖途径只需要一种酶,而大肠杆菌为DXP依赖途径,需要多种酶来完成。因此在底盘的选择中除了考虑酶活外,还要考虑合成路径的优化,以及在另外一个系统中构建新的途径需要克服表达/系统适配等问题。
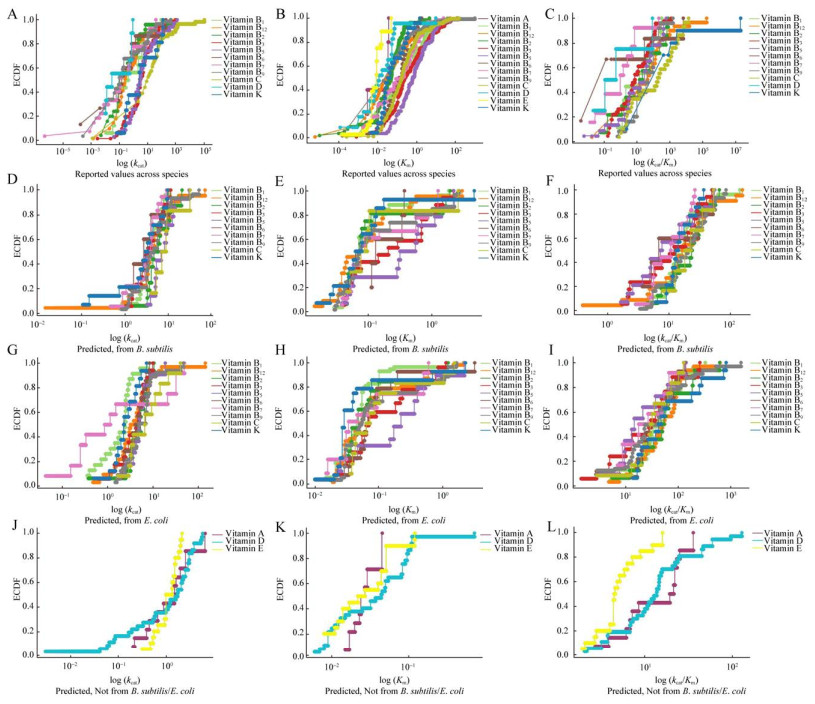
|
| 图 4 不同维生素产品代谢途径中酶动力学数据的经验累积分布函数图 Fig. 4 Empirical Cumulative Distribution Function (ECDF) graphs of enzyme kinetics data in different vitamin product metabolic pathways. A, B, C represent the ECDF graphs for reported log values across species of wild-type data for kcat, Km, and kcat/Km, respectively. D, E, F represent the ECDF graphs for predicted log values from Bacillus subtilis for kcat, Km, and kcat/Km, respectively. G, H, I represent the ECDF graphs for predicted log values from Escherichia coli for kcat, Km, and kcat/Km, respectively. J, K, L represent the ECDF graphs for predicted log values not sourced from Bacillus subtilis or Escherichia coli for kcat, Km, and kcat/Km, respectively. |
| |
除了相同途径在不同物种间的差异外,本身途径的不同酶在活性上也存在很大的差异。比如在预测结果中,枯草芽孢杆菌的维生素B12途径酶最高的kcat值为143.525 s−1,而最低的kcat值为0.477 s−1,相差将近3个数量级,其他途径上最高和最低kcat值的差异也在1个数量级以上,大肠杆菌中也是如此(国家微生物科学数据中心,登录号:SUB1720761294457),这个步骤很可能成为整条途径的瓶颈,需要通过蛋白工程手段提高其催化效率或者增加酶的表达水平。因此,如何调控这些酶,使其能够以最优的比例,有序且不积累中间产物的情况下顺利地通过整个催化途径,尤其是不积累一些具有反馈抑制/毒性的中间产物,是至关重要的。如前文所述,在生产维生素E的前体物质法尼希的时候,刘天罡等[105]通过优化甲羟戊酸途径(MVA途径)中各个酶的最佳比例,在大肠杆菌中达到1 g/L的产量,实现了巨大突破。除了不同酶活性间的差异外,甚至同一个酶中不同底物的活性也会有很大差异,比如,来自大肠杆菌维生素B7途径的DAPA synthase (EC: 2.6.1.62),能够催化DAPA的转氨反应,对底物DAPA和SAM的kcat预测值分别为0.337 s−1和0.043 s−1 (真实值分别为0.790 s−1和0.016 s−1),相差一个数量级之多,也就是说在真实反应的环境下,需要SAM的含量远高于其Km值(预测值:0.068 mmol/L,真实值:0.000 2 mmol/L[120]或者0.3 mmol/L[72]),且有效结合到催化口袋时,才能使反应趋向一个更高的底物kcat反应效率。因为酶对SAM的催化效率更低,需要底物多次进出活性口袋或者多次调整构象才能有效反应。因此,SAM的供应在这一步甚至整个途径的代谢流中都有至关重要的作用。同样,来自枯草芽孢杆菌维生素B9途径的7,8-二氢叶酸合成酶(7,8-dihydropteroate: L-glutamate ligase, DHPS),在ATP的供能下,完成7,8-二氢叶酸和L-谷氨酸的连接反应,其预测的kcat值分别为1.048 s−1和5.696 s−1,相差5倍之多,类似这样的情况还有很多(附图见国家微生物科学数据中心,登录号:SUB1720761294457)。因此,在代谢节点的流量控制和底物反应的类型上还需要进行特异性的调控,以达到最优的比例和顺序,提升最终产量。
通过对维生素合成途径中酶学性质的深入分析和比较,可以揭示不同维生素合成途径间及不同物种相同途径中酶活性的差异,还能识别潜在的瓶颈酶和高效催化步骤。这些数据强调了在设计代谢工程和合成生物学策略时,需要考虑到酶的动力学性质、途径中的代谢流量控制以及底物供应的平衡。应用先进的深度学习算法,例如DLKcat和UniKP,可以对酶的动力学性质进行预测,这些结果不仅可以补充实验数据的不足,也能够指导未来的实验设计。随着对这些复杂代谢途径理解的深入,研究人员将能更有效地优化维生素生产过程,提升产量并降低成本,实现更可持续和高效的生物合成途径。
3 总结与展望维生素的生物合成途径展示了生物体内复杂而精细的代谢网络,涉及多种酶的协同作用以及多条代谢途径的交叉和整合。虽然各维生素之间在前体供应上存在一定的相关性,但从维生素B1的硫胺素合成到维生素K的甲基萘醌合成,每一种维生素的生产都依赖于特定的生化反应和酶催化步骤,造就了各自独特的生理作用和合成途径。这些途径不仅反映了生物体对环境适应和生存的需求,也揭示了生命过程中能量转换和物质循环的基本规律,这也说明了需要多种维生素共存的原因。
从已报道的维生素途径酶的研究中可以看出,尽管在某些维生素中酶的相关研究已经取得了一些进展,但仍有大量酶未经过详细的酶学属性研究,改造研究的比例更是少之又少。这可能和维生素生物合成途径的复杂性有关,因为即使对单个酶进行了有效的改造,也可能需要进一步优化整个途径中的其他酶和代谢流,才能实现维生素生产的显著提升。这需要对整个生物合成途径有深入的理解和全面的调控策略。此外,在许多情况下,对特定酶的研究可能并不是直接针对维生素生产而发起的,而是为了理解这些酶在更广泛的生物化学过程,尤其是医学研究中的作用。这些酶不仅仅是维生素生产所需,而且由于与人体内代谢途径的差异,还可能会作为潜在治疗某些细菌感染的特异性靶蛋白或者检测手段的载体。这些研究扩展了对维生素生物合成途径的理解,并揭示了这些途径中的酶在医学和生物技术领域具有更广泛的应用潜力,特别是在抗微生物药物的发现和疾病诊断技术的开发中,这些酶作为潜在的靶标或工具,为研究提供了新的策略和方法。因此,深入研究这些酶的功能和机制,不仅对提高维生素生产的效率和产量具有重要意义,也可能对开发新的治疗方法和诊断技术提供关键的科学基础。这进一步强调了对维生素生物合成途径及其酶学性质进行细致研究的重要性,既有助于推动生物制造领域的发展,也可能为医学研究带来新的突破。
基于目前报道的数据,进一步分析酶学属性发现,EMP途径中酶的野生型kcat值普遍比维生素合成途径中酶的野生型kcat值高出1−2个数量级,而Km值分布相似。表明EMP途径中的酶催化效率远高于维生素合成途径中的酶。这可能反映了生物体对于不同途径的不同代谢需求,即EMP途径作为基础代谢途径,其酶的高效性有助于快速提供能量和代谢中间体,而维生素合成途径可能不需要如此高的催化效率。另一方面,Km值分布相似说明,尽管催化效率不同,但这些酶对其底物的亲和力大致相似。这可能意味着,尽管EMP途径需要更高效的催化来满足能量和代谢中间体的快速生成,两个途径的酶对底物的亲和力被保持在一个相对稳定的水平,以保证代谢流的平衡和细胞内底物的有效利用。这种差异可能反映了生物在进化过程中对不同代谢途径的适应性调整。此外,EMP途径是生物体获取能量的主要途径之一,对于所有生物来说都是至关重要的,因此其相关酶的高效率可能是为了保证在各种环境条件下都能有效地进行能量转换。而维生素合成途径虽然对维持生物体的正常生理功能也很重要,但可能面临的选择压力较小,或者其更侧重于合成速率的精细调控而非最大速率。因此,了解不同途径中酶的催化效率和底物亲和力可以帮助我们更好地设计和优化代谢途径,以提高目标化合物的生产效率。
另外,不同物种和不同途径中的酶在催化效率(kcat)、底物亲和力(Km)以及这两者的比值(kcat/Km)上存在的差异对于设计和优化微生物生产维生素等代谢产品的策略也至关重要。上述研究表明,即使是相同的维生素合成途径,不同的宿主微生物(如枯草芽孢杆菌和大肠杆菌)在生产能力上可能会有显著差异,某些物种可能因为其途径酶的天然活性更高而显示出催化优势。这强调了在代谢工程中优化整个途径中酶的表达比例和活性的重要性,以避免中间产物的积累和反馈抑制。同时,同一酶对不同底物的活性可能存在显著差异,这为代谢流的控制和优化带来了挑战。随着深度学习等方法的发展,通过已知数据建立的模型能够对未知反应的酶活进行高准确度的预测,为未知或未充分研究的物种/途径的工程改造提供了有力工具。这些研究可以指导如何根据物种中各个酶的特性和需求来调整工程改造的策略,以达到高效生产维生素产品和其他代谢产品的目的。
| [1] |
MARSHALL CW. Vitamins and minerals: help or harm[J]. Clinical Nutrition Insight, 1987, 13(7): 6.
|
| [2] |
Del Mondo, Angelo, Arianna Smerilli, Elisabet Sané, Clementina Sansone, Christophe Brunet. Challenging microalgal vitamins for human health[J]. Microbial Cell Factories, 2020, 1-23.
|
| [3] |
ASENSI-FABADO MA, MUNNÉ-BOSCH S. Vitamins in plants: occurrence, biosynthesis and antioxidant function[J]. Trends in Plant Science, 2010, 15(10): 582-592. DOI:10.1016/j.tplants.2010.07.003
|
| [4] |
McDOWELL LR. Vitamins in Animal and Human Nutrition[M]. 3rd edition. New York: John Wiley & Sons Inc., 2000: 3-14.
|
| [5] |
CAPONE K, SENTONGO T. The ABCs of nutrient deficiencies and toxicities[J]. Pediatric Annals, 2019, 48(11): e434-e440.
|
| [6] |
KONINGS EJM. Committee on food nutrition. Water-soluble vitamins[J]. Journal of AOAC International, 2003, 86(1): 147-149.
|
| [7] |
SCHWECHHEIMER SK, PARK EY, REVUELTA JL, BECKER J, WITTMANN C. Biotechnology of riboflavin[J]. Applied Microbiology and Biotechnology, 2016, 100(5): 2107-2119. DOI:10.1007/s00253-015-7256-z
|
| [8] |
HOHMANN HP, VANDIJL JM, KRISHNAPPA L, PRáGAI Z. Host Organisms: Bacillus subtilis[M]. 1st edtion. Industrial Biotechnology: Microorganisms, 2017: 221-297.
|
| [9] |
LI H, CHEN JJ, LI XY, GAN J, LIU HZ, JIAN Z, XU S, ZHANG AL, LI GL, CHEN KQ. Artificial neural network and genetic algorithm coupled fermentation kinetics to regulate L-lysine fermentation[J]. Bioresource Technology, 2024, 393: 130151. DOI:10.1016/j.biortech.2023.130151
|
| [10] |
LIU J, LIU MS, SHI T, SUN GN, GAO N, ZHAO XJ, GUO X, NI XM, YUAN QQ, FENG JH, LIU ZM, GUO YM, CHEN JZ, WANG Y, ZHENG P, SUN JB. CRISPR-assisted rational flux-tuning and arrayed CRISPRi screening of an L-proline exporter for L-proline hyperproduction[J]. Nature Communications, 2022, 13: 891. DOI:10.1038/s41467-022-28501-7
|
| [11] |
刘佳峰, 乔郅钠, 赵有玺, 徐美娟, 张显, 杨套伟, 饶志明. 理性代谢工程改造促进谷氨酸棒杆菌高效合成L-谷氨酸[J]. 生物工程学报, 2023, 39(8): 3273-3289. LIU JF, QIAO ZN, ZHAO YX, XU MJ, ZHANG X, YANG TW, RAO ZM. Rational metabolic engineering of Corynebacterium glutamicum for efficient synthesis of L-glutamate[J]. Chinese Journal of Biotechnology, 2023, 39(8): 3273-3289 (in Chinese). DOI:10.13345/j.cjb.230018 |
| [12] |
WANG YY, LIU LX, JIN ZX, ZHANG DW. Microbial cell factories for green production of vitamins[J]. Frontiers in Bioengineering and Biotechnology, 2021, 9: 661562. DOI:10.3389/fbioe.2021.661562
|
| [13] |
王盼盼, 于洪巍. 酶催化在维生素及其衍生物制备中的应用[J]. 合成生物学, 2022, 3(3): 500-515. WANG PP, YU HW. Application of enzyme catalysis in the preparation of vitamins and their derivatives[J]. Synthetic Biology Journal, 2022, 3(3): 500-515 (in Chinese). |
| [14] |
沈润溥, 皮士卿, 谢斌, 黄红军, 陈新志. 维生素A衍生物合成工艺的改进[J]. 应用化学, 2003, 20(12): 1211-1213. SHEN RP, PI SQ, XIE B, HUANG HJ, CHEN XZ. An improved method for synthesis of vitamin A derivatives via wittig-horner reaction[J]. Chinese Journal of Applied Chemistry, 2003, 20(12): 1211-1213 (in Chinese). DOI:10.3969/j.issn.1000-0518.2003.12.021 |
| [15] |
ZHOU YT, WANG HY, ZHOU JM, QIU SH, CAI TT, LI HQ, SHEN ZY, Vitamin A and its multi-effects on pancreas: recent advances and prospects[J]. Frontiers in Endocrinology 12 (2021): 620941.
|
| [16] |
LIU LX, LI JL, GAI YM, TIAN ZZ, WANG YY, WANG TH, LIU P, YUAN QQ, MA HW, LEE SY, ZHANG DW. Protein engineering and iterative multimodule optimization for vitamin B6 production in Escherichia coli[J]. Nature Communications, 2023, 14: 5304. DOI:10.1038/s41467-023-40928-0
|
| [17] |
KANG Q, FANG H, XIANG MJ, XIAO KX, JIANG PT, YOU C, LEE SY, ZHANG DW. A synthetic cell-free 36-enzyme reaction system for vitamin B12 production[J]. Nature Communications, 2023, 14: 5177. DOI:10.1038/s41467-023-40932-4
|
| [18] |
FANG H, LI D, KANG J, JIANG PT, SUN JB, ZHANG DW. Metabolic engineering of Escherichia coli for de novo biosynthesis of vitamin B12[J]. Nature Communications, 2018, 9: 4917. DOI:10.1038/s41467-018-07412-6
|
| [19] |
LENNARZ WJ, LANE MD. Encyclopedia of Biological Chemistry[M]. 2nd edition. Beijing: Science Press, 2006: 27-33.
|
| [20] |
PARKHOMENKO Y, VOVK A, PROTASOVA Z. Vitamin B1 and the pyruvate dehydrogenase complex[M]//Molecular Nutrition. Amsterdam: Elsevier, 2020: 185-206.
|
| [21] |
DiNICOLANTONIO JJ, LIU J, O'KEEFE JH. Thiamine and cardiovascular disease: a literature review[J]. Progress in Cardiovascular Diseases, 2018, 61(1): 27-32. DOI:10.1016/j.pcad.2018.01.009
|
| [22] |
KANEHISA M, GOTO S. KEGG: kyoto encyclopedia of genes and genomes[J]. Nucleic Acids Research, 2000, 28(1): 27-30. DOI:10.1093/nar/28.1.27
|
| [23] |
SCHYNS G, POTOT S, GENG Y, BARBOSA TM, HENRIQUES A, PERKINS JB. Isolation and characterization of new thiamine-deregulated mutants of Bacillus subtilis[J]. Journal of Bacteriology, 2005, 187(23): 8127-8136. DOI:10.1128/JB.187.23.8127-8136.2005
|
| [24] |
ROSINI E, PIUBELLI L, MOLLA G, FRATTINI L, VALENTINO M, VARRIALE A, D'AURIA S, POLLEGIONI L. Novel biosensors based on optimized glycine oxidase[J]. The FEBS Journal, 2014, 281(15): 3460-3472. DOI:10.1111/febs.12873
|
| [25] |
TATSUMI M, HOSHINO W, KODAMA Y, UEATRONGCHIT T, TAKAHASHI K, YAMAGUCHI H, TAGAMI U, MIYANO H, ASANO Y, MIZUKOSHI T. Development of a rapid and simple glycine analysis method using a stable glycine oxidase mutant[J]. Analytical Biochemistry, 2019, 587: 113447. DOI:10.1016/j.ab.2019.113447
|
| [26] |
ONOZUKA M, NOSAKA K. Steady-state kinetics and mutational studies of recombinant human thiamin pyrophosphokinase[J]. Journal of Nutritional Science and Vitaminology, 2003, 49(3): 156-162. DOI:10.3177/jnsv.49.156
|
| [27] |
BALASUBRAMANIAM S, CHRISTODOULOU J, RAHMAN S. Disorders of riboflavin metabolism[J]. Journal of Inherited Metabolic Disease, 2019, 42(4): 608-619. DOI:10.1002/jimd.12058
|
| [28] |
ANDREIEVA Y, PETROVSKA Y, LYZAK O, LIU W, KANG YQ, DMYTRUK K, SIBIRNY A. Role of the regulatory genes SEF1, VMA1 and SFU1 in riboflavin synthesis in the flavinogenic yeast Candida famata (Candida flareri)[J]. Yeast, 2020, 37(9/10): 497-504.
|
| [29] |
AVERIANOVA LA, BALABANOVA LA, SON OM, PODVOLOTSKAYA AB, TEKUTYEVA LA. Production of vitamin B2 (riboflavin) by microorganisms: an overview[J]. Frontiers in Bioengineering and Biotechnology, 2020, 8: 570828. DOI:10.3389/fbioe.2020.570828
|
| [30] |
李光镐, 朴英薰, 韩锺权, 朴长熙, 李庚翰. 生产核黄素的微生物和利用其生产核黄素的方法: CN03143072. X[P]. 2005-11-02. LI GH, PIAO YX, HAN ZQ, PIAO CX, LI GH. Microorganisms for Producing Riboflavin and Methods for Using Them: CN03143072. X[P]. 2005-11-02. |
| [31] |
XU F, LIU C, XIA MM, LI SX, TU R, WANG SJ, JIN HX, ZHANG DW. Characterization of a riboflavin-producing mutant of Bacillus subtilis isolated by droplet-based microfluidics screening[J]. Microorganisms, 2023, 11(4): 1070. DOI:10.3390/microorganisms11041070
|
| [32] |
WU K, MAO ZT, MAO YF, NIU JH, CAI JY, YUAN QQ, YUN LL, LIAO XP, WANG ZW, MA HW. ecBSU1: a genome-scale enzyme-constrained model of Bacillus subtilis based on the ECMpy workflow[J]. Microorganisms, 2023, 11(1): 178. DOI:10.3390/microorganisms11010178
|
| [33] |
Burgess C, O'connell-Motherway M, Sybesma W, Hugenholtz J, van Sinderen D. Riboflavin production in lactococcus lactis: potential for in situ production of vitamin-enriched foods[J]. Applied and Environ mental Microbiology, 2004, 70(10): 5769-5777. DOI:10.1128/AEM.70.10.5769-5777.2004
|
| [34] |
LEHMANN M, DEGEN S, HOHMANN HP, WYSS M, BACHER A, SCHRAMEK N. Biosynthesis of riboflavin. Screening for an improved GTP cyclohydrolase Ⅱ mutant[J]. The FEBS Journal, 2009, 276(15): 4119-4129. DOI:10.1111/j.1742-4658.2009.07118.x
|
| [35] |
FISCHER M, HAASE I, KIS K, MEINING W, LADENSTEIN R, CUSHMAN M, SCHRAMEK N, HUBER R, BACHER A. Enzyme catalysis via control of activation entropy: site-directed mutagenesis of 6, 7-dimethyl-8-ribityllumazine synthase[J]. Journal of Molecular Biology, 2003, 326(3): 783-793. DOI:10.1016/S0022-2836(02)01473-0
|
| [36] |
SERRANO A, SEBASTIÁN M, ARILLA-LUNA S, BAQUEDANO S, PALLARÉS MC, LOSTAO A, HERGUEDAS B, VELÁZQUEZ-CAMPOY A, MARTÍNEZ-JÚLVEZ M, MEDINA M. Quaternary organization in a bifunctional prokaryotic FAD synthetase: involvement of an arginine at its adenylyltransferase module on the riboflavin kinase activity[J]. Biochimica et Biophysica Acta, 2015, 1854(8): 897-906.
|
| [37] |
TANNOUS C, BOOZ GW, ALTARA R, MUHIEDDINE DH, MERICSKAY M, REFAAT MM, ZOUEIN FA. Nicotinamide adenine dinucleotide: Biosynthesis, consumption and therapeutic role in cardiac diseases[J]. Acta Physiologica, 2021, 231(3): e13551. DOI:10.1111/apha.13551
|
| [38] |
BELENKY P, STEBBINS R, BOGAN KL, EVANS CR, BRENNER C. Nrt1 and Tna1-independent export of NAD+ precursor vitamins promotes NAD+ homeostasis and allows engineering of vitamin production[J]. PloS One, 2011, 6(5): e19710. DOI:10.1371/journal.pone.0019710
|
| [39] |
WANG Z, LIU ZM, CUI WJ, ZHOU ZM. Establishment of bioprocess for synthesis of nicotinamide by recombinant Escherichia coli expressing high-molecular-mass nitrile hydratase[J]. Applied Biochemistry and Biotechnology, 2017, 182(4): 1458-1466. DOI:10.1007/s12010-017-2410-y
|
| [40] |
程琳, 龚劲松, Michael HALL, 苏畅, 徐国强, 许正宏, 史劲松. 大肠杆菌合成烟酰胺单核苷酸途径构建及发酵工艺研究[J]. 食品与发酵工业, 2023, 49(24): 8-14. CHENG L, GONG JS, HALL M, SU C, XU GQ, XU ZH, SHI JS. Pathway construction and fermentation process of synthesis of nicotinamide mononucleotide in Escherichia coli[J]. Food and Fermentation Industry, 2023, 49(24): 8-14 (in Chinese). |
| [41] |
WANG XY, ZHOU YJ, WANG L, LIU WJ, LIU YX, PENG C, ZHAO ZK. Engineering Escherichia coli nicotinic acid mononucleotide adenylyltransferase for fully active amidated NAD biosynthesis[J]. Applied and Environmental Microbiology, 2017, 83(13): e00692-e00617.
|
| [42] |
KUHN I, KELLENBERGER E, CAKIR-KIEFER C, MULLER-STEFFNER H, SCHUBER F. Probing the catalytic mechanism of bovine CD38/NAD+ glycohydrolase by site directed mutagenesis of key active site residues[J]. Biochimica et Biophysica Acta, 2014, 1844(7): 1317-1331.
|
| [43] |
ZHAO K, TANG H, ZHANG B, ZOU SP, LIU ZQ, ZHENG YG. Microbial production of vitamin B5: current status and prospects[J]. Critical Reviews in Biotechnology, 2023, 43(8): 1172-1192. DOI:10.1080/07388551.2022.2104690
|
| [44] |
LEONARDI R, JACKOWSKI S. Biosynthesis of pantothenic acid and coenzyme A[J]. EcoSal Plus, 2007, 2(2): 10.1128/ecosalplus. 3.6.3.4.
|
| [45] |
ZHANG B, ZHANG XM, WANG W, LIU ZQ, ZHENG YG. Metabolic engineering of Escherichia coli for dpantothenic acid production[J]. Food Chemistry, 2019, 294: 267-275. DOI:10.1016/j.foodchem.2019.05.044
|
| [46] |
LEE JH, AHN SY, LEE HA, WON KS, CHANG HW, OH JS, KIM HW. Dietary intake of pantothenic acid is associated with cerebral amyloid burden in patients with cognitive impairment[J]. Food & Nutrition Research, 2018, 62: 62.
|
| [47] |
ZENG L, GUO LL, WANG ZX, XU XX, SONG SS, XU LG, KUANG H, LI AK, XU CL. Immunoassays for the rapid detection of pantothenic acid in pharmaceutical and food products[J]. Food Chemistry, 2021, 348: 129114. DOI:10.1016/j.foodchem.2021.129114
|
| [48] |
TIGU, FiITSUM, ZHANG JL, LIU GX, CAI Z, LI Y. A highly active pantothenate synthetase from Corynebacterium glutamicum enables the production of D-pantothenic acid with high productivity[J]. Applied microbiology and biotechnology, 2018, 102(14): 6039-6046. DOI:10.1007/s00253-018-9017-2
|
| [49] |
GUO JX, SUN XX, YUAN YJ, CHEN QT, OU ZT, DENG ZX, MA T, LIU TG. Metabolic engineering of Saccharomyces cerevisiae for vitamin B5 production[J]. Journal of Agricultural and Food Chemistry, 2023, 71(19): 7408-7417. DOI:10.1021/acs.jafc.3c01082
|
| [50] |
DIBBLE CC, BARRITT SA, PERRY GE, LIEN EC, GECK RC, DUBOIS-COYNE SE, BARTEE D, ZENGEYA TT, COHEN EB, YUAN M, HOPKINS BD, MEIER JL, CLOHESSY JG, ASARA JM, CANTLEY LC, TOKER A. PI3K drives the de novo synthesis of coenzyme A from vitamin B5[J]. Nature, 2022, 608: 192-198. DOI:10.1038/s41586-022-04984-8
|
| [51] |
PEI WL, ZHANG JL, DENG SY, TIGU F, LI YX, LI Q, CAI Z, LI Y. Molecular engineering of L-aspartate-α-decarboxylase for improved activity and catalytic stability[J]. Applied Microbiology and Biotechnology, 2017, 101(15): 6015-6021. DOI:10.1007/s00253-017-8337-y
|
| [52] |
JONCZYK R, GENSCHEL U. Molecular adaptation and allostery in plant pantothenate synthetases[J]. The Journal of Biological Chemistry, 2006, 281(49): 37435-37446. DOI:10.1074/jbc.M607895200
|
| [53] |
FITZPATRICK TB, AMRHEIN N, KAPPES B, MACHEROUX P, TEWS I, RASCHLE T. Two independent routes of de novo vitamin B6 biosynthesis: not that different after all[J]. The Biochemical Journal, 2007, 407(1): 1-13. DOI:10.1042/BJ20070765
|
| [54] |
PARRA M, STAHL S, HELLMANN H. Vitamin B₆ and its role in cell metabolism and physiology[J]. Cells, 2018, 7(7): 84. DOI:10.3390/cells7070084
|
| [55] |
TRAMONTI A, NARDELLA C, Di SALVO ML, BARILE A, D'ALESSIO F, de CRÉCY-LAGARD V, CONTESTABILE R. Knowns and unknowns of vitamin B6 metabolism in Escherichia coli[J]. EcoSal Plus, 2021, 9(2): eESP-0004-2021. DOI:10.1128/ecosalplus.ESP-0004-2021
|
| [56] |
HVAS AM, JUUL S, BECH P, NEXØ E. Vitamin B6 level is associated with symptoms of depression[J]. Psychotherapy and Psychosomatics, 2004, 73(6): 340-343. DOI:10.1159/000080386
|
| [57] |
HOFFMANN GF, SCHMITT B, WINDFUHR M, WAGNER N, STREHL H, BAGCI S, FRANZ AR, MILLS PB, CLAYTON PT, BAUMGARTNER MR, STEINMANN B, BAST T, WOLF NI, ZSCHOCKE J. Pyridoxal 5'-phosphate may be curative in early-onset epileptic encephalopathy[J]. Journal of Inherited Metabolic Disease, 2007, 30(1): 96-99. DOI:10.1007/s10545-006-0508-4
|
| [58] |
HELLMANN H, MOONEY S. Vitamin B6: a molecule for human health?[J]. Molecules, 2010, 15(1): 442-459. DOI:10.3390/molecules15010442
|
| [59] |
TIAN ZZ, LIU LX, WU LJ, YANG ZX, ZHANG YH, DU LP, ZHANG DW. Enhancement of vitamin B6 production driven by omics analysis combined with fermentation optimization[J]. Microbial Cell Factories, 2024, 23(1): 137. DOI:10.1186/s12934-024-02405-1
|
| [60] |
WANG J, XU X, WEI WQ, SONG W, WEN J, HU GP, LI XM, GAO C, CHEN XL, LIU LM, WU J. Rational design of Salmonella typhi acid phosphatase for efficient production of pyridoxal 5ʹ-phosphate[J]. Journal of Agricultural and Food Chemistry, 2024.
|
| [61] |
HOSHINO T, ICHIKAWA K, TAZOE M. Dna encoding favin-adenine-dinucleotide-dependent-D-erythronate-4-phosphate-de-hydrogenase, pdxr, and microbial production of vitamin B6[J]. Google Patents. 2008
|
| [62] |
GU J, CHEN YY, GUO HS, SUN ML, YANG MK, WANG XD, ZHANG XE, DENG JY. Lysine acetylation regulates the activity of Escherichia coli pyridoxine 5ʹ-phosphate oxidase[J]. Acta Biochimica et Biophysica Sinica, 2017, 49(2): 186-192. DOI:10.1093/abbs/gmw129
|
| [63] |
MOCCAND C, KAUFMANN M, FITZPATRICK TB. It takes two to tango: defining an essential second active site in pyridoxal 5ʹ-phosphate synthase[J]. PloS One, 2011, 6(1): e16042. DOI:10.1371/journal.pone.0016042
|
| [64] |
ERB TJ. Carboxylases in natural and synthetic microbial pathways[J]. Applied and Environmental Microbiology, 2011, 77(24): 8466-8477. DOI:10.1128/AEM.05702-11
|
| [65] |
TONG L. Structure and function of biotin-dependent carboxylases[J]. Cellular and Molecular life Sciences, 2013, 70(5): 863-891. DOI:10.1007/s00018-012-1096-0
|
| [66] |
SCOTT W C. Literature review of both classic and novel roles of biotin (vitamin B7) in cellular processes[J]. UTSC's Journal of Natural Sciences, 2020, 1(1): 45-51.
|
| [67] |
MASUDA M, TAKAHASHI K, SAKURAI N, YANAGIYA K, KOMATSUBARA S, TOSA T. Further improvement of D-biotin production by a recombinant strain of Serratia marcescens[J]. Process Biochemistry, 1995, 30(6): 553-562.
|
| [68] |
CLACK BA, YOUNGBLOOD AB. Nucleic acid for biotin production: US7423136[P]. 2008-09-09.
|
| [69] |
LIN S, HANSON RE, CRONAN JE. Biotin synthesis begins by hijacking the fatty acid synthetic pathway[J]. Nature Chemical Biology, 2010, 6: 682-688. DOI:10.1038/nchembio.420
|
| [70] |
van ARSDELL SW, PERKINS JB, YOCUM RR, LUAN LD, HOWITT CL, PRASAD CHATTERJEE N, PERO JG. Removing a bottleneck in the Bacillus subtilis biotin pathway: BioA utilizes lysine rather than S-adenosylmethionine as the amino donor in the KAPA-to-DAPA reaction[J]. Biotechnology and Bioengineering, 2005, 91(1): 75-83. DOI:10.1002/bit.20488
|
| [71] |
SANDMARK J, ELIOT AC, FAMM K, SCHNEIDER G, KIRSCH JF. Conserved and nonconserved residues in the substrate binding site of 7,8-diaminopelargonic acid synthase from Escherichia coli are essential for catalysis[J]. Biochemistry, 2004, 43(5): 1213-1222. DOI:10.1021/bi0358059
|
| [72] |
SALAEMAE W, YAP MY, WEGENER KL, BOOKER GW, WILCE MCJ, POLYAK SW. Nucleotide triphosphate promiscuity in Mycobacterium tuberculosis dethiobiotin synthetase[J]. Tuberculosis, 2015, 95(3): 259-266. DOI:10.1016/j.tube.2015.02.046
|
| [73] |
MIRONENKO A, ELISEEVA T. Vitamin B9–description, benefits, effects on the body and best sources[J]. Journal of Healthy Nutrition and Dietetics, 2019, 4(10): 88-100.
|
| [74] |
MYSZCZYSZYN A, KRAJEWSKI R, OSTAPÓW M, HIRNLE L. Folic acid-role in the body, recommendations and clinical significance[J]. Nursing in the 21st Century, 2019, 18(1): 50-59.
|
| [75] |
SERRANO-AMATRIAIN C, LEDESMA-AMARO R, LÓPEZ-NICOLÁS R, ROS G, JIMÉNEZ A, REVUELTA JL. Folic acid production by engineered ashbya gossypii[J]. Metabolic Engineering, 2016, 38: 473-482. DOI:10.1016/j.ymben.2016.10.011
|
| [76] |
SCHMITZ LM, KREITLI N, OBERMAIER L, WEBER N, RYCHLIK M, ANGENENT LT. Power-to-vitamins: producing folate (vitamin B9) from renewable electric power and CO2 with a microbial protein system[J]. bioRxiv, 2024. DOI: 10.1101/2024.02.22.581687.
|
| [77] |
BLANCQUAERT D, van DAELE J, STROBBE S, KIEKENS F, STOROZHENKO S, de STEUR H, GELLYNCK X, LAMBERT W, STOVE C, van der STRAETEN D. Improving folate (vitamin B9) stability in biofortified rice through metabolic engineering[J]. Nature Biotechnology, 2015, 33: 1076-1078. DOI:10.1038/nbt.3358
|
| [78] |
HORIUCHI Y, OHMAE E, TATE SI, GEKKO K. Coupling effects of distal loops on structural stability and enzymatic activity of Escherichia coli dihydrofolate reductase revealed by deletion mutants[J]. Biochimica et Biophysica Acta, 2010, 1804(4): 846-855.
|
| [79] |
WANG Y, XU HM, GROCHOWSKI LL, WHITE RH. Biochemical characterization of a dihydroneopterin aldolase used for methanopterin biosynthesis in methanogens[J]. Journal of Bacteriology, 2014, 196(17): 3191-3198. DOI:10.1128/JB.01812-14
|
| [80] |
STABLER SP. Vitamin B12[M]//Present Knowledge in Nutrition. Amsterdam: Elsevier, 2020: 257-271.
|
| [81] |
OSMAN D, COOKE A, YOUNG TR, DEERY E, ROBINSON NJ, WARREN MJ. The requirement for cobalt in vitamin B12: a paradigm for protein metalation[J]. Biochimica et Biophysica Acta Molecular Cell Research, 2021, 1868(1): 118896. DOI:10.1016/j.bbamcr.2020.118896
|
| [82] |
LIU YH, HUANG W, WANG Q, MA CL, CHANG YY, SU JY. Research on the targeted improvement of the yield of a new VB12-producing strain, Ensifer adhaerens S305, based on genomic and transcriptomic analysis[J]. BMC Biotechnology, 2023, 23(1): 53. DOI:10.1186/s12896-023-00824-3
|
| [83] |
CLAAS KR, PARRISH JR, MAGGIO-HALL LA, ESCALANTE-SEMERENA JC. Functional analysis of the nicotinate mononucleotide: 5,6-dimethylbenzimidazole phosphoribosyltransferase (CobT) enzyme, involved in the late steps of coenzyme B12 biosynthesis in Salmonella enterica[J]. Journal of Bacteriology, 2010, 192(1): 145-154. DOI:10.1128/JB.01159-09
|
| [84] |
MOORE TC, NEWMISTER SA, RAYMENT I, ESCALANTE-SEMERENA JC. Structural insights into the mechanism of four-coordinate Cob(Ⅱ)alamin formation in the active site of the Salmonella enterica ATP: co(Ⅰ)rrinoid adenosyltransferase enzyme: critical role of residues Phe91 and Trp93[J]. Biochemistry, 2012, 51(48): 9647-9657. DOI:10.1021/bi301378d
|
| [85] |
KAWAHORI K, KONDO Y, YUAN XM, KAWASAKI Y, HANZAWA N, TSUJIMOTO K, WADA F, KOHDA T, ISHIGAMI A, YAMADA T, OGAWA Y, HASHIMOTO K. Ascorbic acid during the suckling period is required for proper DNA demethylation in the liver[J]. Scientific Reports, 2020, 10: 21228. DOI:10.1038/s41598-020-77962-7
|
| [86] |
PACKER L. Vitamin C in Health and Disease[M]. Boca Raton: CRC Press, 1997: 341-367.
|
| [87] |
高媛, 曾伟主, 周景文, 陈坚. 普通生酮基古龙酸菌中山梨糖脱氢酶和山梨酮脱氢酶的纯化及酶学性质分析[J]. 微生物学报, 2017, 57(10): 1546-1554. GAO Y, ZENG WZ, ZHOU JW, CHEN J. Purification and characterization of L-sorbose dehydrogenase and L-sorbosone dehydrogenase from Ketogulonicigenium vulgare WSH-001[J]. Acta Microbiologica Sinica, 2017, 57(10): 1546-1554 (in Chinese). |
| [88] |
SAUER M, BRANDUARDI P, VALLI M, PORRO D. Production of L-ascorbic acid by metabolically engineered Saccharomyces cerevisiae and Zygosaccharomyces bailii[J]. Applied and Environmental Microbiology, 2004, 70(10): 6086-6091. DOI:10.1128/AEM.70.10.6086-6091.2004
|
| [89] |
HYDE AS, THELEN AM, BARYCKI JJ, SIMPSON MA. UDP-glucose dehydrogenase activity and optimal downstream cellular function require dynamic reorganization at the dimer-dimer subunit interfaces[J]. Journal of Biological Chemistry, 2013, 288(49): 35049-35057. DOI:10.1074/jbc.M113.519090
|
| [90] |
LEFERINK NGH, JOSE MDF, van den BERG WAM, van BERKEL WJH. Functional assignment of Glu386 and Arg388 in the active site of L-galactono-γ-lactone dehydrogenase[J]. FEBS letters, 2009, 583(19): 3199-3203. DOI:10.1016/j.febslet.2009.09.004
|
| [91] |
WISE LA, WESSELINK AK, BETHEA TN, BRASKY TM, WEGIENKA G, HARMON Q, BLOCK T, BAIRD DD. Intake of lycopene and other carotenoids and incidence of uterine leiomyomata: a prospective ultrasound study[J]. Journal of the Academy of Nutrition and Dietetics, 2021, 121(1): 92-104. DOI:10.1016/j.jand.2020.08.013
|
| [92] |
LARROUDE M, CELINSKA E, BACK A, THOMAS S, NICAUD JM, LEDESMA-AMARO R. A synthetic biology approach to transform Yarrowia Lipolytica into a competitive biotechnological producer of β-carotene[J]. Biotechnology and Bioengineering, 2018, 115(2): 464-472. DOI:10.1002/bit.26473
|
| [93] |
YOON SH, KIM JE, LEE SH, PARK HM, CHOI MS, KIM JY, LEE SH, SHIN YC, KEASLING JD, KIM SW. Engineering the lycopene synthetic pathway in E. coli by comparison of the carotenoid genes of Pantoea agglomerans and Pantoea ananatis[J]. Applied Microbiology and Biotechnology, 2007, 74(1): 131-139. DOI:10.1007/s00253-006-0623-z
|
| [94] |
STICKFORTH P, SANDMANN G. Structural and kinetics properties of a mutated phytoene desaturase from Rubrivivax gelatinosus with modified product specificity[J]. Archives of Biochemistry and Biophysics, 2011, 505(1): 118-122. DOI:10.1016/j.abb.2010.09.021
|
| [95] |
HERRMANN M. Assessing vitamin D metabolism: four decades of experience[J]. Clinical Chemistry and Laboratory Medicine (CCLM), 2023, 61(5): 880-894. DOI:10.1515/cclm-2022-1267
|
| [96] |
DELRUE C, SPEECKAERT MM. Vitamin D and vitamin D-binding protein in health and disease[J]. International Journal of Molecular Sciences, 2023, 24(5): 4642. DOI:10.3390/ijms24054642
|
| [97] |
ISMAILOVA A, WHITE JH. Vitamin D, infections and immunity[J]. Reviews in Endocrine and Metabolic Disorders, 2022, 23(2): 265-277. DOI:10.1007/s11154-021-09679-5
|
| [98] |
MARUCCI L, BARBERIS M, KARR J, RAY O, RACE PR, de SOUZA ANDRADE M, GRIERSON C, HOFFMANN SA, LANDON S, RECH E, REES-GARBUTT J, SEABROOK R, SHAW W, WOODS C. Computer-aided whole-cell design: taking a holistic approach by integrating synthetic with systems biology[J]. Frontiers in Bioengineering and Biotechnology, 2020, 8: 942. DOI:10.3389/fbioe.2020.00942
|
| [99] |
TAN TW, ZHANG M, GAO H. Ergosterol production by fed-batch fermentation of Saccharomyces cerevisiae[J]. Enzyme and Microbial Technology, 2003, 33(4): 366-370. DOI:10.1016/S0141-0229(03)00132-7
|
| [100] |
LIU JL, GANAPATHY K, WYWIAL E, BUJNICKI JM, NWOGWUGWU CA, NES WD. Effect of substrate features and mutagenesis of active site tyrosine residues on the reaction course catalysed by Trypanosoma brucei sterol C-24-methyltransferase[J]. The Biochemical Journal, 2011, 439(3): 413-422. DOI:10.1042/BJ20110865
|
| [101] |
FUJII Y, KABUMOTO H, NISHIMURA K, FUJII T, YANAI S, TAKEDA K, TAMURA N, ARISAWA A, TAMURA T. Purification, characterization, and directed evolution study of a vitamin D3 hydroxylase from Pseudonocardia autotrophica[J]. Biochemical and Biophysical Research Communications, 2009, 385(2): 170-175. DOI:10.1016/j.bbrc.2009.05.033
|
| [102] |
MUTHULAKSHMI MV, SRINIVASAN A, SRIVASTAVA S. Antioxidant green factories: toward sustainable production of vitamin E in plant in vitro cultures[J]. ACS Omega, 2023, 8(4): 3586-3605. DOI:10.1021/acsomega.2c05819
|
| [103] |
PEH HY, TAN WS, LIAO WP, WONG WS. Vitamin E therapy beyond cancer: tocopherol versus tocotrienol[J]. Pharmacology & Therapeutics, 2016, 162: 152-169.
|
| [104] |
SHEN B, ZHOU PP, JIAO X, YAO Z, YE LD, YU HW. Fermentative production of Vitamin E tocotrienols in Saccharomyces cerevisiae under cold-shock-triggered temperature control[J]. Nature Communications, 2020, 11: 5155. DOI:10.1038/s41467-020-18958-9
|
| [105] |
刘天罡, 朱发银, 邓子新. 一种生产法尼烯的菌株及其应用: CN103243065A[P]. 2013-08-14. LIU TG, ZHU FY, DENG ZX. A strain for producing farnesene and its application: CN103243065A[P]. 2013-08-14. |
| [106] |
马田, 邓子新, 刘天罡. 维生素E的"前世"和"今生"[J]. 合成生物学, 2020, 1(2): 174-186. MA T, DENG ZX, LIU TG. The past and present of vitamin E[J]. Synthetic Biology Journal, 2020, 1(2): 174-186 (in Chinese). |
| [107] |
DAS AK, GHOSH S, SIL PC. Vitamin K[M]//Antioxidants Effects in Health. Amsterdam: Elsevier, 2022: 561-582.
|
| [108] |
LAIZÉ V, VIEGAS CSB, PRICE PA, CANCELA ML. Identification of an osteocalcin isoform in fish with a large acidic prodomain[J]. The Journal of Biological Chemistry, 2006, 281(22): 15037-15043. DOI:10.1074/jbc.M600373200
|
| [109] |
BOOTH SL. Vitamin K: food composition and dietary intakes[J]. Food & Nutrition Research, 2012, 56: 56.
|
| [110] |
DUDA-CHODAK A, TARKO T, SATORA P, SROKA P. Interaction of dietary compounds, especially polyphenols, with the intestinal microbiota: a review[J]. European Journal of Nutrition, 2015, 54(3): 325-341. DOI:10.1007/s00394-015-0852-y
|
| [111] |
HWANG S, CORDOVA B, ABDO M, PFEIFFER F, MAUPIN-FURLOW JA. ThiN as a versatile domain of transcriptional repressors and catalytic enzymes of thiamine biosynthesis[J]. Journal of Bacteriology, 2017, 199(7): e00810-e00816.
|
| [112] |
XU XH, LV XQ, CUI SX, LIU YF, XIA HZ, LI JH, DU GC, LI ZF, LEDESMA-AMARO R, CHEN J, LIU L. Remodeling isoprene pyrophosphate metabolism for promoting terpenoids bioproduction[J]. Engineering, 2023, 28: 166-178. DOI:10.1016/j.eng.2023.03.019
|
| [113] |
KONG MK, LEE PC. Metabolic engineering of menaquinone-8 pathway of Escherichia coli as a microbial platform for vitamin K production[J]. Biotechnology and Bioengineering, 2011, 108(8): 1997-2002. DOI:10.1002/bit.23142
|
| [114] |
QIN MM, SONG HG, DAI X, CHEN YZ, GUO ZH. Two active site arginines are critical determinants of substrate binding and catalysis in MenD: a thiamine-dependent enzyme in menaquinone biosynthesis[J]. The Biochemical Journal, 2018, 475(22): 3651-3667. DOI:10.1042/BCJ20180548
|
| [115] |
CHEN YZ, SUN YR, SONG HG, GUO ZH. Structural basis for the ATP-dependent configuration of adenylation active site in Bacillus subtilis o-succinylbenzoyl-CoA synthetase[J]. The Journal of Biological Chemistry, 2015, 290(39): 23971-23983. DOI:10.1074/jbc.M115.676304
|
| [116] |
LI HJ, LI XK, LIU NN, ZHANG HN, TRUGLIO JJ, MISHRA S, KISKER C, GARCIA-DIAZ M, TONGE PJ. Mechanism of the intramolecular Claisen condensation reaction catalyzed by MenB, a crotonase superfamily member[J]. Biochemistry, 2011, 50(44): 9532-9544. DOI:10.1021/bi200877x
|
| [117] |
CHANG A, JESKE L, ULBRICH S, HOFMANN J, KOBLITZ J, SCHOMBURG I, NEUMANN-SCHAAL M, JAHN D, SCHOMBURG D. BRENDA, the ELIXIR core data resource in 2021: new developments and updates[J]. Nucleic Acids Research, 2021, 49(D1): D498-D508. DOI:10.1093/nar/gkaa1025
|
| [118] |
LI F, YUAN L, LU H, LI G, CHEN Y, ENGQVIST M, EDUARD J, NIELSEN J. Deep learning-based kcat prediction enables improved enzyme-constrained model reconstruction[J]. Nature Catalysis, 2022, 5(8): 662-672. DOI:10.1038/s41929-022-00798-z
|
| [119] |
YU H, DENG H, HE J, KEASLING JD, LUO X. UniKP: a unified framework for the prediction of enzyme kinetic parameters[J]. Nature Communications, 2024, 14(1): 8211.
|
| [120] |
EISENBERG MA, STONER GL. [60]7,8-Diaminopelargonic acid aminotransferase[M]//Methods in Enzymology. Amsterdam: Elsevier, 1979: 342-347.
|
 2024, Vol. 40
2024, Vol. 40