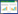中国科学院微生物研究所、中国微生物学会主办
文章信息
- 张静, 元跃, 王智文, 陈涛
- ZHANG Jing, YUAN Yue, WANG Zhiwen, CHEN Tao
- 基于工程化盐单胞菌TDZI-08一锅法合成衣康酸
- One-pot synthesis of itaconic acid by engineered Halomonas bluephagenesis TDZI-08
- 生物工程学报, 2024, 40(8): 2666-2677
- Chinese Journal of Biotechnology, 2024, 40(8): 2666-2677
- 10.13345/j.cjb.240071
-
文章历史
- Received: January 25, 2024
- Accepted: March 25, 2024
- Published: March 28, 2024
2. 菏泽学院 农业与生物工程学院, 山东 菏泽 274000
2. College of Agricultural and Biological Engineering, Heze University, Heze 274000, Shandong, China
衣康酸(itaconic acid, IA),也被称为亚甲基丁二酸,是美国能源部公布的12种高附加值平台化合物之一,在纤维、树脂和橡胶的生产中起着重要作用[1-2]。衣康酸可通过化学法和微生物发酵法合成[2-3],其中微生物发酵法是工业生产衣康酸的主要方法。生物合成途径是从三羧酸循环(tricarboxylic acid cycle, TCA)中间产物柠檬酸出发,经顺乌头酸酶(aconitase, ACN)催化转化为顺乌头酸,顺乌头酸再由顺乌头酸脱羧酶(cis-aconitate decarboxylase, CAD)催化成为衣康酸。目前工业上常用的生产菌株有丝状真菌土曲霉(Aspergillus terreus)和玉米黑粉菌(Ustilago maydis),最高产量分别可达160 g/L和220 g/L衣康酸[4-5]。然而,丝状真菌具有生长速度慢、生产效率低、需氧量高、发酵重现性差和遗传背景复杂等缺点,制约了衣康酸产量和得率的进一步提高。近年来,多种异源宿主如黑曲霉(Aspergillus niger)[6-7]、大肠杆菌(Escherichia coli)[8-9]、谷氨酸棒状杆菌(Corynebacterium glutamicum)[10-11]和解脂耶氏酵母(Yarrowia lipolytica)[12-13]等也被改造并利用发酵法合成衣康酸,但其产量(最高54.6 g/L[12])远低于天然生产宿主。
全细胞催化是利用微生物细胞作为催化剂进行底物转化的过程,相较于微生物发酵法,具有条件易控、反应高效、产物易于分离和细胞可回收利用等优点[14]。近年来,全细胞催化已应用于衣康酸的合成。通过对大肠杆菌[15-19]、希瓦氏菌(Shewanella livingstonensis)和盐单胞菌(Halomonas bluephagenesis)等进行代谢工程改造,并利用工程化菌株细胞作为催化剂,将廉价底物柠檬酸转化为衣康酸(表 1)。盐单胞菌催化系统是本课题组前期构建的,通过在盐单胞菌TD1.0中引入衣康酸生物合成的必需基因(CAD编码基因cadA和ACN编码基因acn)、共表达分子伴侣蛋白基因groESL、增加限速酶基因acn拷贝数和弱化竞争途径基因表达,获得了工程菌株TDZI-08 (图 1)。在优化条件下,TDZI-08细胞催化柠檬酸最高产生63.60 g/L衣康酸[20]。尽管该盐单胞菌催化系统的衣康酸产量和效率低于大肠杆菌催化系统(表 1),但由于盐单胞菌具有嗜盐嗜碱的特性,在工业应用时可进行开放式不灭菌连续培养,有利于简化生产工艺和降低生产成本[21]。因此,对盐单胞菌TDZI-08催化柠檬酸生产衣康酸工艺进行深入研究有望为其工业化生产提供实验基础。
| Strains | Strategies | Process | Titer (g/L) | Yield (g/g)a | References |
| Escherichia coli Rosetta(DE3) | Self-assembled reaction of aconitase (ACO) and CAD via protein-peptide interaction | Two-step cell catalysis |
8.70 | 0.10 | [15] |
| E. coli JY002 | Overexpression of acn and multiple cadA | 41.60 | 0.48 | [16] | |
| E. coli BL-CAR470E-DS/A-CS | Optimize CAD and synthesizing protein scaffolds to co-localize ACO and CAD | Two-step cell catalysis | 51.79 | − | [17] |
| E. coli AtCg | Overexpression of cadA and acnA under dual T7 promoters; optimization parameter and medium | Two-step cell catalysis | 67.00 | 0.35 | [18] |
| E. coli Lemo21(DE3) | Overexpression of acn and cadA; cold treatment for 24 h; integration of groESL to chromosome | Two-step cell catalysis | 98.17 | 0.51 | [19] |
| H. bluephagenesis TDZI-08 | Expression of cadA and acn; co-expression of molecular chaperone GroESL; increasing the copy number of the acn; downregulation of isocitrate dehydrogenase encoding gene icd; optimization the reaction conditions | Two-step cell catalysis | 63.60 | 0.63 | [20] |
| H. bluephagenesis TDZI-08 | Process optimization | One-pot synthesis | 40.50 | 0.68 | This study |
| a: The yield was calculated according to the following formula: concentration of IA produced (g/L)/concentration of citric acid consumed (g/L). | |||||
|
| 图 1 盐单胞菌TDZI-08以柠檬酸为底物合成衣康酸路径图 Fig. 1 Pathway diagram of the synthesis of IA using citric acid as substrate in Halomonas bluephagenesis TDZI-08. |
| |
利用工程菌株TDZI-08两步法催化合成衣康酸时,需要先在丰富培养基中制备细胞,然后经过细胞收集、浓缩和洗涤后再转移到反应体系中进行催化,繁琐的工艺不利于后续的工业化应用[20]。为了简化工艺,本研究基于工程菌株TDZI-08开发了一锅法合成衣康酸工艺,通过排查阻碍因素、优化碳源、氮源、诱导剂添加时间和表面活性剂添加量等,实现了细胞生长、诱导和催化合成的一体化,在开放式不灭菌条件下通过一锅法可高效合成衣康酸,从而简化了工艺流程,有利于提高经济效益,对衣康酸的产业化发展具有重要参考价值。
1 材料与方法 1.1 菌株本实验所使用的菌株是前期本课题组构建的工程化盐单胞菌H. bluephagenesis TDZI-08[20]。
1.2 培养基LB培养基(g/L):酵母抽提物5.0,胰蛋白胨10.0,氯化钠10.0。
60-LB培养基(g/L):酵母抽提物5.0,胰蛋白胨10.0,氯化钠60.0。
60-MM培养基(g/L):氯化钠60.0,酵母提取物5.0,Na2HPO4∙12H2O 9.7,KH2PO4 1.5,MgSO4 0.2,NH4Cl 1.0,溶液Ⅰ 10.0 mL,溶液Ⅱ 1.0 mL,根据实验需求添加适量柠檬酸钠或葡萄糖酸钠为碳源,采用5.0 mol/L NaOH溶液调pH至8.5−9.0。
溶液Ⅰ (g/L):柠檬酸铁铵5.0,CaCl2 2.0,用1.0 mol/L HCl溶液配制。
溶液Ⅱ (mg/L):ZnSO4∙7H2O 100.0,CoCl2∙6H2O 100.0,H3BO3 300.0,CuSO4∙5H2O 10.0,MnCl2∙4H2O 30.0,NiCl2∙6H2O 20.0,NaMoO4∙2H2O 30.0。
MM培养基(g/L):在60-MM基础上去除60.0 g/L氯化钠并调整碳源和氮源得到,柠檬酸钠和葡萄糖酸钠混合物(总钠离子物质的量浓度不低于0.68 mol/L以维持盐单胞菌生长渗透压)为碳源,酵母提取物5.0+氯化铵1.0或酵母提取物2.5+玉米浆干粉2.5+氯化铵5.0为氮源,其余组分不变。
培养基使用前添加50.0 μg/mL壮观霉素和25.0 μg/mL氯霉素。
1.3 一锅法合成衣康酸方法取−80 ℃保藏的TDZI-08甘油管液体,在含抗生素的60-LB平板上划线,37 ℃静置培养24 h;挑取单菌落接种于含抗生素的60-LB液体培养基中,37 ℃、200 r/min培养12 h获得一级种子液;取一级种子液按1% (体积分数)接种量转接至含抗生素的60-LB液体培养基中,37 ℃、200 r/min培养10−12 h获得二级种子液;将二级种子液按10% (体积分数)接种量转接至含有200 mL 60-MM液体培养基的2 L带挡板摇瓶中,37 ℃、200 r/min培养;当细胞OD600为3时,加入终浓度为1 mmol/L异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside, IPTG),30 ℃、200 r/min诱导培养10 h;诱导结束后,使用柠檬酸溶液(500 g/L)下调并维持pH至6.3 (同时也起到补加底物柠檬酸的作用),并添加表面活性剂曲拉通X-100 (Triton X-100)以增加细胞膜通透性促进反应进行[20],在30 ℃、200 r/min条件下进行催化反应。其中培养基的碳源、氮源、诱导剂添加时间以及表面活性剂添加量按2.3所示进行优化。
1.4 5 L发酵罐开放式一锅法合成衣康酸二级种子液制备操作同1.3所述,将二级种子液按10% (体积分数)接种量转接至含有2 L 60-MM液体培养基的5 L发酵罐(上海百仑生物科技有限公司)中,37 ℃、500 r/min培养,关联转速(500–800 r/min)和溶氧,维持溶氧在30%左右;培养基、补料液和发酵罐体均不灭菌,接种和补料在开放式条件下操作;当细胞OD600增长至15时,加入终浓度为1 mmol/L的IPTG,转至30 ℃诱导培养10 h;诱导结束后,将转速下调至500 r/min,断开通气,使用柠檬酸溶液(500 g/L)下调并维持pH至6.3,添加0.03% Triton X-100,进行催化反应,当衣康酸产量不再增加时停止反应。
1.5 细胞生物量和反应物浓度的测定细胞生物量使用600 nm下的吸光值(OD600)表示,取培养液适当稀释后,用紫外分光光度计TU-1801 (北京普析通用仪器有限责任公司)测定600 nm波长下的吸光度。反应物柠檬酸、衣康酸、顺乌头酸和葡萄糖酸钠的含量采用高效液相色谱(high performance liquid chromatography, HPLC)测定,检测条件为:HPLC系统(Agilent Technologies公司),分离柱为Aminex HPX-87H色谱柱(300 mm×7.8 mm, 9 μm, Bio-Rad公司),流动相为0.5 mmol/L H2SO4,流速为0.4 mL/min,柱温为60 ℃,柠檬酸、衣康酸和顺乌头酸采用紫外检测器(波长为210 nm)检测,葡萄糖酸钠采用示差检测器检测。
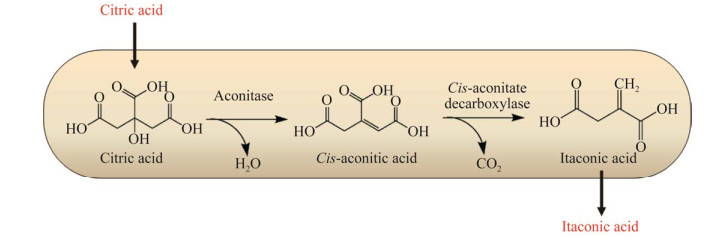
2 结果与分析 2.1 一锅法合成衣康酸初步测试为了实现利用菌株TDZI-08进行一锅法合成衣康酸,在60-MM培养基中添加32 g/L的二水合柠檬酸三钠(相当于21 g/L柠檬酸)作为碳源进行测试。接种后,分别在细胞密度(OD600)达到1和3时,添加IPTG进行诱导培养,48 h后取样检测细胞生物量、柠檬酸残余量和衣康酸产量,结果如图 2所示。在OD600为1时加入IPTG导致细胞生长和代谢迟缓,最高细胞密度(OD600)为1.73,且仅消耗了4.30 g/L柠檬酸,几乎没有衣康酸生成(图 2);当OD600为3时加IPTG诱导培养,细胞生长有明显改善,细胞OD600最高为8.04,消耗了14.10 g/L柠檬酸,但衣康酸产量非常低(0.81 g/L) (图 2)。结果表明一锅法合成衣康酸的效率很低,为提高其合成效率,需要对影响因素进行排查和优化。
|
| 图 2 不同细胞密度时IPTG诱导对衣康酸合成的影响 Fig. 2 Effect of IPTG induction at different cell density on IA synthesis. |
| |
前期研究发现,在盐单胞菌细胞催化合成衣康酸的过程中,维持反应体系中pH在6.0–6.5和添加表面活性剂Triton X-100是衣康酸高效合成的必要条件[20]。然而,由于60-MM培养基的pH约为9.0且不含表面活性剂,可能会影响一锅法合成衣康酸的效率。使用初步测试的条件培养和诱导细胞(生长和诱导阶段),48 h后使用1 mol/L HCl将培养液pH下调至6.5,并补加0.5% Triton X-100,以转变为适合细胞催化的条件(催化阶段),但衣康酸产量仍没有增加,这表明还有其他影响因素。
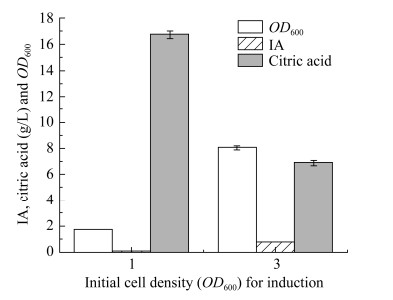
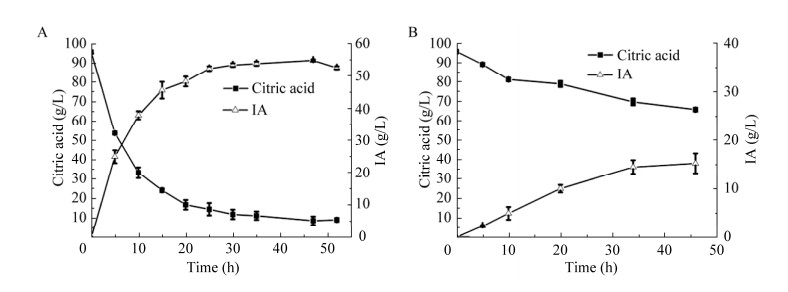
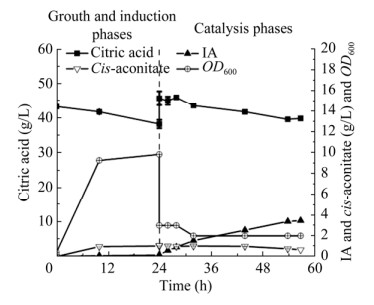
前期两步细胞催化时发现,在反应体系中添加高浓度氯化钠会显著降低衣康酸的合成效率。以96 g/L柠檬酸为底物,当催化体系中不含1 mol/L氯化钠时,反应正常,催化47 h产生55 g/L衣康酸(图 3A);当催化体系中含1 mol/L氯化钠时,柠檬酸消耗明显减慢,催化45 h仅产生15 g/L衣康酸(图 3B)。由于60-MM培养基中含60 g/L氯化钠(相当于1 mol/L氯化钠)以维持盐单胞菌生长所需的渗透压,推测可能是高浓度氯化钠的存在影响了一锅法合成衣康酸的效率。盐单胞菌能在高盐环境下生存,高浓度钠离子是其正常生长所依赖的[22]。为了排除氯化钠对衣康酸合成的影响,使用柠檬酸钠盐替代氯化钠以维持盐单胞菌的正常生长。盐单胞菌生长最适氯化钠浓度为40–80 g/L[23],初步选择40 g/L氯化钠对应的钠离子浓度(0.68 mol/L)以确定柠檬酸钠盐的初始添加量,将60-MM培养基调整为MM,即用67 g/L二水合柠檬酸三钠(含0.68 mol/L钠离子)替换60 g/L氯化钠,其余成分不变。以MM为培养基进行三阶段培养:(1) 生长阶段。培养基pH用5 mol/L NaOH调至9.0,在37 ℃、200 r/min下培养;(2) 诱导阶段。细胞OD600增长至3时,加入IPTG,30 ℃、200 r/min诱导培养至24 h,pH维持在8.5−9.0;(3) 催化阶段。添加0.5% Triton X-100,并使用柠檬酸溶液(500 g/L)将pH下调至6.3,于30 ℃、200 r/min下进行细胞催化,其间使用柠檬酸溶液(500 g/L)维持控制pH在6.3左右;取样测定柠檬酸、衣康酸和顺乌头酸浓度以及细胞OD600,结果如图 4所示。细胞诱导培养后(24 h时)的最高OD600为9.2,转为催化阶段后,OD600快速降至3左右,到催化结束时降至2左右;细胞OD600下降的很大原因是Triton X-100的添加导致细胞裂解,因为一定浓度的Triton X-100可以破坏细胞膜的结构,使细胞膜中的脂质层发生溶解导致细胞破碎;生长和诱导阶段几乎没有衣康酸产生(< 0.1 g/L),调节pH为6.3后,衣康酸快速积累,最高产生了3.50 g/L衣康酸。以上结果表明经过去除氯化钠、调控pH和添加表面活性剂后,可实现一锅法合成衣康酸。
|
| 图 3 高浓度氯化钠对细胞催化柠檬酸合成衣康酸效率的影响 Fig. 3 Effect of high concentration sodium chloride on the efficiency of cell-catalyzed IA synthesis. A: Catalytic system without 1 mol/L sodium chloride. B: Catalytic system containing 1 mol/L sodium chloride. A:催化体系中不含1 mol/L氯化钠. B:催化体系中含1 mol/L氯化钠 |
| |
|
| 图 4 一锅法合成衣康酸相关参数 Fig. 4 Characterization of one-pot synthesis of IA. |
| |
虽然初步确定了一锅法合成衣康酸的工艺,但与两步细胞催化法相比,衣康酸的产量仍较低,推测是因为一锅法培养时获得的细胞量(最高OD600仅9.2)太低所致。为了增加细胞量,对培养基中的碳源、氮源和IPTG添加时间进行优化。此外,对表面活性剂的添加量也进行了优化。
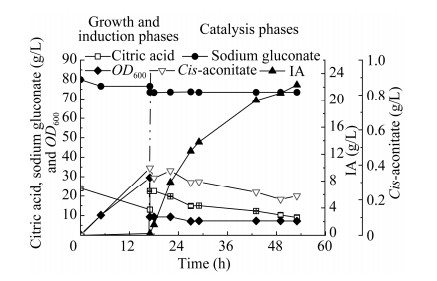
2.3.1 优化碳源和氮源为了改善盐单胞菌的生长,将培养基中的渗透压提高,即将总钠离子浓度提高至0.77 mol/L (相当于45 g/L氯化钠所含的钠离子浓度)。在改造盐单胞菌合成其他产品的研究中,发现使用80 g/L葡萄糖酸钠为碳源,2.5 g/L酵母提取物和2.5 g/L玉米浆干粉为氮源时,盐单胞菌生长良好;此外,本课题组的研究发现,将氯化铵浓度提高至5 g/L可以显著促进盐单胞菌的生长(相关数据尚未发表)。因此,采用80 g/L葡萄糖酸钠和40 g/L二水合柠檬酸三钠(总钠离子浓度为0.77 mol/L)混合物为碳源,同时添加2.5 g/L酵母提取物、2.5 g/L玉米浆干粉和5 g/L氯化铵为氮源,培养盐单胞菌TDZI-08并测试其细胞生长和衣康酸产生情况。结果表明,改变碳源和氮源条件后,细胞生长和衣康酸的合成都有明显改善(图 5)。在生长阶段,生物量迅速增长,培养17 h时OD600达到30;在17 h时添加0.5% Triton X-100并流加柠檬酸溶液(500 g/L)下调pH至6.3转为催化阶段,OD600降至10 (这是由于表面活性剂Triton X-100导致细胞裂解所致);在催化阶段衣康酸快速合成,经过36 h的催化产生了22.33 g/L衣康酸(图 5)。
|
| 图 5 改变碳源和氮源对底物消耗、细胞生长和衣康酸产量的影响 Fig. 5 Substrate consumption, cell growth, and IA production after changing carbon and nitrogen sources. |
| |
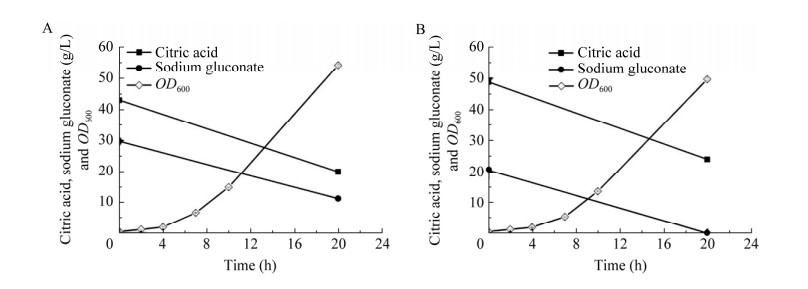
然而,在上述条件下葡萄糖酸钠仅在生长阶段有少量消耗,催化阶段不能用于衣康酸的合成,导致葡萄糖酸钠有大量残留(60−70 g/L) (图 5)。这既造成了底物的浪费也增加了产物分离难度,需要降低混合物中葡萄糖酸钠的添加量。因此,测试了两种碳源在不同配比条件下的细胞生长和底物消耗情况:(1) 30 g/L葡萄糖酸钠+67 g/L二水合柠檬酸三钠;(2) 20 g/L葡萄糖酸钠+75 g/L二水合柠檬酸三钠。结果如图 6所示,配比1最高细胞OD600 (OD600为54)虽然略高于配比2 (OD600为50),但诱导培养后配比1中葡萄糖酸钠剩余约11 g/L,而配比2中葡萄糖酸钠能够被完全消耗,从而更有利于后续的分离纯化。综合考虑,优选配比2作为MM培养基的碳源。
|
| 图 6 不同碳源配比下底物消耗和细胞生长 Fig. 6 Substrate consumption and cell growth under different carbon source ratios. A: 30 g/L sodium gluconate+67 g/L trisodium citrate dihydrate. B: 20 g/L sodium gluconate+75 g/L trisodium citrate dihydrate. A:30 g/L葡萄糖酸钠+67 g/L二水合柠檬酸三钠. B:20 g/L葡萄糖酸钠+75 g/L二水合柠檬酸三钠 |
| |
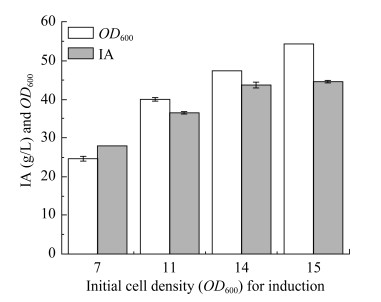
前期研究表明适当推迟IPTG添加有助于增加诱导培养的细胞量[20, 24]。以20 g/L葡萄糖酸钠+75 g/L二水合柠檬酸三钠为碳源,2.5 g/L酵母提取物、2.5 g/L玉米浆干粉和5 g/L氯化铵为氮源培养盐单胞菌TDZI-08,当细胞OD600分别增长至7、11、14和15时,添加IPTG并诱导培养10 h,然后添加0.5% Triton X-100并流加柠檬酸溶液(500 g/L)下调pH至6.3,转为催化阶段并反应36 h。诱导后的最高OD600和衣康酸产量如图 7所示。当细胞OD600分别为7、11、14和15时添加IPTG,诱导培养获得的最高细胞OD600分别为25、40、47和54;整个过程中初始添加的20 g/L葡萄糖酸钠均被完全消耗,此外还分别消耗了56.67、76.27、94.96、100.90 g/L柠檬酸;催化36 h的衣康酸产量分别为28.07、36.51、43.84、44.77 g/L,衣康酸总得率分别为0.376、0.387、0.388、0.380 g/g (葡萄糖酸+柠檬酸)。以上结果表明适当推迟IPTG添加有利于改善细胞生长和衣康酸合成,选择细胞OD600为15时作为IPTG的添加时间点。
|
| 图 7 IPTG诱导时的初始细胞密度(OD600)对细胞生长和衣康酸产量的影响 Fig. 7 Effect of initial cell density (OD600) induced by IPTG on cell growth and IA production. |
| |
为减少表面活性剂的使用,对Triton X-100添加量进行优化。以20 g/L葡萄糖酸钠+75 g/L二水合柠檬酸三钠为碳源,2.5 g/L酵母提取物、2.5 g/L玉米浆干粉和5 g/L氯化铵为氮源,培养盐单胞菌TDZI-08,当细胞OD600增长至15时添加IPTG并诱导培养10 h,然后分别添加0.01%、0.02%、0.03%和0.05% Triton X-100并补加柠檬酸溶液(500 g/L)下调pH至6.3转为催化阶段,当衣康酸产量不再增加时终止反应,结果如图 8所示。当Triton X-100添加量为0.03%和0.05%时,衣康酸最高产量分别为43.12 g/L (图 8C)和44.80 g/L (图 8D),与添加量0.5%时无差别(图 5);但当Triton X-100添加量降为0.02%时,只产生了33.89 g/L衣康酸(图 8B),继续降至0.01%时,产生的衣康酸仅11.79 g/L (图 8A)。因此,为保证催化的正常进行,Triton X-100添加量选定为0.03%。
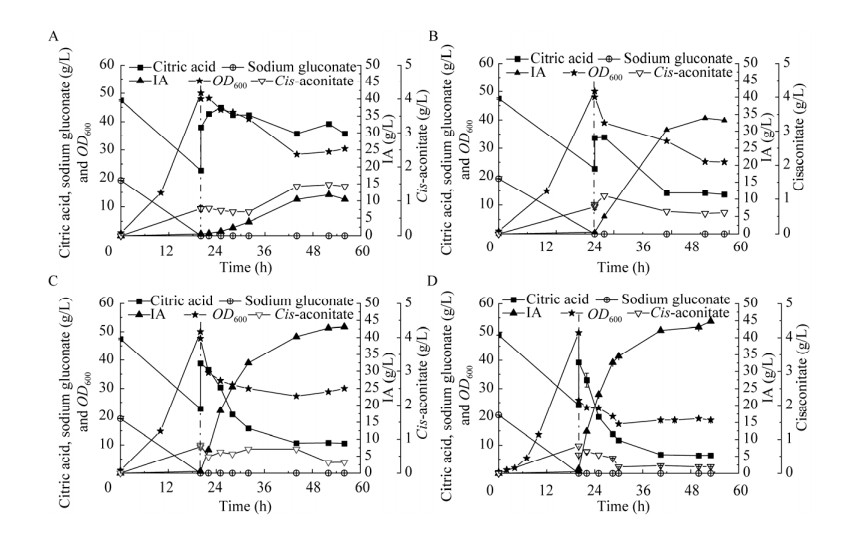
|
| 图 8 不同Triton X-100添加量对衣康酸合成的影响 Fig. 8 Effect of different Triton X-100 addition amounts on IA production. A: 0.01% Triton X-100. B: 0.02% Triton X-100. C: 0.03% Triton X-100. D: 0.05% Triton X-100. |
| |
至此,确定了一锅法合成衣康酸工艺:使用MM培养基(20 g/L葡萄糖酸钠+75 g/L二水合柠檬酸三钠为碳源,2.5 g/L酵母提取物+2.5 g/L玉米浆干粉+5 g/L氯化铵为氮源)进行三阶段培养。(1) 生长阶段:培养基pH用5 mol/L NaOH调至9.0,37 ℃、200 r/min培养10 h,此时细胞OD600约15;(2) 诱导阶段:细胞OD600为15时,加入IPTG,30 ℃、200 r/min诱导培养,pH维持在8.5–9.0,培养10 h,转为催化阶段;(3) 催化阶段:使用柠檬酸溶液(500 g/L)将pH下调并维持在6.3,添加0.03% Triton X-100,在30 ℃、200 r/min条件下进行催化。
根据上述条件,TDZI-08在摇瓶中采用一锅法催化反应36 h合成43.12 g/L衣康酸(体积200 mL),生长阶段消耗了20.0 g/L葡萄糖酸钠(实测17.3 g/L葡萄糖酸)和24.75 g/L柠檬酸,共计3.46 g葡萄糖酸和4.95 g柠檬酸,催化阶段共消耗13.68 g柠檬酸,催化阶段的衣康酸得率为0.63 g/g柠檬酸,衣康酸总得率为0.39 g/g (柠檬酸+葡萄糖酸);反应体系中只有衣康酸、柠檬酸和少量中间产物顺乌头酸(< 0.20 g/L),相对纯净,便于下游分离;整个过程无需细胞收集、浓缩和洗涤操作,简单便捷。
2.4 开放式不灭菌条件下在5 L发酵罐中一锅法合成衣康酸在5 L发酵罐中进行开放式不灭菌一锅法合成衣康酸。初始体积为2 L,细胞OD600增加至15时加IPTG并诱导培养10 h,然后转为催化阶段并到反应结束,周期时长为56 h,过程中补料液的流加使最终体积增加至2.04 L,期间取样检测细胞OD600、底物消耗及衣康酸产量,结果如图 9所示。在开放式不灭菌条件下,诱导培养后的细胞最大OD600为41 (23 h),添加0.03% Triton X-100并流加柠檬酸溶液(500 g/L)转为催化阶段后,OD600降至30左右,到催化结束时OD600降低至15左右;生长阶段(前23 h)葡萄糖酸钠(实测17.90 g/L葡萄糖酸)完全耗尽,柠檬酸(实测46.69 g/L)消耗了18.71 g/L (剩余27.98 g/L),生长阶段共计消耗了35.80 g葡萄糖酸和37.42 g柠檬酸;培养23 h时流加柠檬酸溶液(500 g/L)下调pH并维持至6.3转为催化阶段,到催化结束共流加了160 mL柠檬酸溶液(共计80 g柠檬酸),最后反应体系中剩余7.21 g/L柠檬酸,催化阶段共计消耗了121.25 g柠檬酸;催化阶段衣康酸快速积累,反应33 h产生了40.50 g/L衣康酸(共计82.62 g衣康酸),此阶段衣康酸产率为1.23 g/(L∙h),得率达到了理论值(0.68 g/g柠檬酸);整个过程的衣康酸总得率为0.42 g/g (柠檬酸+葡萄糖酸)。
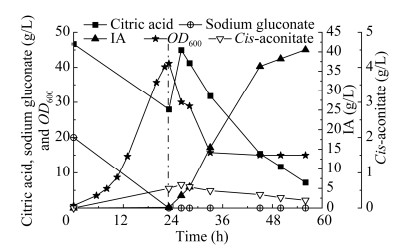
|
| 图 9 开放式不灭菌条件下在5 L发酵罐中一锅法合成衣康酸 Fig. 9 One-pot synthesis of IA in a 5 L fermentation tank under open and unsterile conditions. |
| |
本研究基于工程化盐单胞菌TDZI-08,通过排查影响因素、优化细胞培养和表面活性剂添加量等途径,开发了一锅法催化柠檬酸合成衣康酸工艺。在摇瓶中TDZI-08可利用一锅法合成43.12 g/L衣康酸,催化阶段的衣康酸得率为0.63 g/g柠檬酸,衣康酸总得率为0.39 g/g (柠檬酸+葡萄糖酸) (图 8C);在5 L发酵罐中进行开放式不灭菌一锅法合成,产生了40.50 g/L衣康酸,催化阶段的衣康酸得率达到了理论值(约为0.68 g/g柠檬酸),总得率为0.42 g/g (柠檬酸+葡萄糖酸) (图 9)。与前期的两步细胞催化法相比,本研究中的一锅法合成过程无须细胞收集、浓缩和洗涤操作,工艺流程更加简单;与传统大肠杆菌催化系统相比,盐单胞菌催化系统无须灭菌和无菌操作,生产成本更加低廉。然而,在发酵罐中一锅法合成衣康酸时OD600出现两次明显的下降,第一次是转为催化阶段时(23 h),OD600从41快速降至30 (图 9),这可能是因为表面活性剂Triton X-100的添加导致细胞裂解;第二次是在28 h时发酵罐内溶氧从之前的30%上升至70%,OD600从29 (28 h)降至15左右(33 h) (图 9),这可能与催化环境(包括溶氧和酸性环境)不利以及长时间搅拌导致细胞衰亡有关。细胞OD600的下降反映了细胞裂解的增加,导致更多的衣康酸合成酶释放至胞外,这会降低酶的稳定性和催化活性,从而减弱衣康酸的合成效率。因此,后续需对条件进行优化和调整,以进一步提高一锅法合成衣康酸的水平。
近年来随着研究的深入,以大肠杆菌为底盘的催化系统在衣康酸合成方面取得了显著进展。例如,2022年,Hsiang等[18]在大肠杆菌BL21中过表达cad和acn构建了工程菌E. coli AtCg,经过优化培养基、细胞量、反应时间和反应pH等参数后,利用两步细胞催化法以柠檬酸为底物合成了67.00 g/L衣康酸,产率为8.375 g/(L∙h) (表 1);2023年,Diankristanti等[19]在大肠杆菌Lemo21(DE3)基因组上整合表达分子伴侣基因groESL,并引入表达cadA和acnA的质粒,构建获得的菌株L21:: 7G经−80 ℃低温处理24 h后用于细胞催化,最高产生了98.17 g/L衣康酸,产率为9.82 g/(L∙h),且经过4轮重复催化合成的衣康酸产量仍保持在86.9% (表 1)。相比之下,以盐单胞菌为底盘的催化系统在衣康酸产量(40.50 g/L)和产率[1.23 g/(L∙h)]方面仍存在较大差距。为了提高盐单胞菌底盘的催化效率,可以进一步优化盐单胞菌工程菌株中衣康酸合成关键酶在转录(主要从启动子工程、密码子优化和优化蛋白表达系统[25]等方面展开)和翻译层面(主要对翻译起始区包括核糖体结合序列、核糖体结合序列与翻译起始位点之间的连接区域以及基因编码序列的前5个密码子进行优化[26])的表达,或利用蛋白质工程技术改善衣康酸合成关键酶的活性和稳定性,以期达到更高的衣康酸产量和产率。
致谢: 感谢清华大学陈国强教授提供盐单胞菌出发菌株和工具质粒。
| [1] |
OKABE M, LIES D, KANAMASA S, PARK EY. Biotechnological production of itaconic acid and its biosynthesis in Aspergillus terreus[J]. Applied Microbiology and Biotechnology, 2009, 84(4): 597-606.
|
| [2] |
KUENZ A, KRULL S. Biotechnological production of itaconic acid—things you have to know[J]. Applied Microbiology and Biotechnology, 2018, 102(9): 3901-3914.
|
| [3] |
PFEIFER VF, VOJNOVICH C, HEGER EN. Itaconic acid by fermentation with Aspergillus terreus[J]. Industrial & Engineering Chemistry, 1952, 44(12): 2975-2980.
|
| [4] |
KRULL S, HEVEKERL A, KUENZ A, PRÜßE U. Process development of itaconic acid production by a natural wild type strain of Aspergillus terreus to reach industrially relevant final titers[J]. Applied Microbiology and Biotechnology, 2017, 101(10): 4063-4072.
|
| [5] |
HOSSEINPOUR TEHRANI H, BECKER J, BATOR I, SAUR K, MEYER S, RODRIGUES LÓIA AC, BLANK LM, WIERCKX N. Integrated strain- and process design enable production of 220 g/L itaconic acid with Ustilago maydis[J]. Biotechnology for Biofuels, 2019, 12(1): 263.
|
| [6] |
HOSSAIN AH, LI A, BRICKWEDDE A, WILMS L, CASPERS M, OVERKAMP K, PUNT PJ. Rewiring a secondary metabolite pathway towards itaconic acid production in Aspergillus niger[J]. Microbial Cell Factories, 2016, 15(1): 130.
|
| [7] |
van der STRAAT L, VERNOOIJ M, LAMMERS M, van den BERG W, SCHONEWILLE T, CORDEWENER J, van der MEER I, KOOPS A, de GRAAFF LH. Expression of the Aspergillus terreus itaconic acid biosynthesis cluster in Aspergillus niger[J]. Microbial Cell Factories, 2014, 13: 11.
|
| [8] |
HARDER BJ, BETTENBROCK K, KLAMT S. Temperature-dependent dynamic control of the TCA cycle increases volumetric productivity of itaconic acid production by Escherichia coli[J]. Biotechnology and Bioengineering, 2018, 115(1): 156-164.
|
| [9] |
HARDER BJ, BETTENBROCK K, KLAMT S. Model-based metabolic engineering enables high yield itaconic acid production by Escherichia coli[J]. Metabolic Engineering, 2016, 38: 29-37.
|
| [10] |
MERKEL M, KIEFER D, SCHMOLLACK M, BLOMBACH B, LILGE L, HENKEL M, HAUSMANN R. Acetate-based production of itaconic acid with Corynebacterium glutamicum using an integrated pH-coupled feeding control[J]. Bioresource Technology, 2022, 351: 126994.
|
| [11] |
OTTEN A, BROCKER M, BOTT M. Metabolic engineering of Corynebacterium glutamicum for the production of itaconate[J]. Metabolic Engineering, 2015, 30: 156-165.
|
| [12] |
RONG LX, MIAO L, WANG SH, WANG YP, LIU SQ, LU ZH, ZHAO BX, ZHANG CY, XIAO DG, PUSHPANATHAN K, WONG A, YU AQ. Engineering Yarrowia lipolytica to produce itaconic acid from waste cooking oil[J]. Frontiers in Bioengineering and Biotechnology, 2022, 10: 888869.
|
| [13] |
ZHAO C, CUI ZY, ZHAO XY, ZHANG JX, ZHANG LH, TIAN YJ, QI QS, LIU JJ. Enhanced itaconic acid production in Yarrowia lipolytica via heterologous expression of a mitochondrial transporter MTT[J]. Applied Microbiology and Biotechnology, 2019, 103(5): 2181-2192.
|
| [14] |
de CARVALHO CC. Whole cell biocatalysts: essential workers from nature to the industry[J]. Microbial Biotechnology, 2017, 10(2): 250-263.
|
| [15] |
YANG ZW, GAO X, XIE H, WANG FQ, REN YH, WEI DZ. Enhanced itaconic acid production by self-assembly of two biosynthetic enzymes in Escherichia coli[J]. Biotechnology and Bioengineering, 2017, 114(2): 457-462.
|
| [16] |
KIM J, SEO HM, BHATIA SK, SONG HS, KIM JH, JEON JM, CHOI KY, KIM W, YOON JJ, KIM YG, YANG YH. Production of itaconate by whole-cell bioconversion of citrate mediated by expression of multiple cis-aconitate decarboxylase (cadA) genes in Escherichia coli[J]. Scientific Reports, 2017, 7: 39768.
|
| [17] |
FENG J, LI CQ, HE H, XU S, WANG X, CHEN KQ. Construction of cell factory through combinatorial metabolic engineering for efficient production of itaconic acid[J]. Microbial Cell Factories, 2022, 21(1): 275.
|
| [18] |
HSIANG CC, DIANKRISTANTI PA, TAN SI, KE YC, CHEN YC, EFFENDI SSW, NG IS. Tailoring key enzymes for renewable and high-level itaconic acid production using genetic Escherichia coli via whole-cell bioconversion[J]. Enzyme and Microbial Technology, 2022, 160: 110087.
|
| [19] |
DIANKRISTANTI PA, NG IS. Microbial itaconic acid bioproduction towards sustainable development: insights, challenges, and prospects[J]. Bioresource Technology, 2023, 384: 129280.
|
| [20] |
ZHANG J, JIN B, HONG KQ, LV Y, WANG ZW, CHEN T. Cell catalysis of citrate to itaconate by engineered Halomonas bluephagenesis[J]. ACS Synthetic Biology, 2021, 10(11): 3017-3027.
|
| [21] |
TAN D, WU Q, CHEN JC, CHEN GQ. Engineering Halomonas TD01 for the low-cost production of polyhydroxyalkanoates[J]. Metabolic Engineering, 2014, 26: 34-47.
|
| [22] |
王伟伟, 唐鸿志, 许平. 嗜盐菌耐盐机制相关基因的研究进展[J]. 微生物学通报, 2015, 42(3): 550-558. WANG WW, TANG HZ, XU P. Salt-tolerance related genes in halophilic bacteria and archaea[J]. Microbiology China, 2015, 42(3): 550-558 (in Chinese). |
| [23] |
陈国强, 薛源生, 谭丹, 吴琼. 一株盐单胞菌及其应用: CN102120973A[P]. 2011-07-13. CHEN G, XUE YS, TAN D, WU Q. Halomonas strain and application thereof: CN102120973A[P]. 2011-07-13 (in Chinese). |
| [24] |
OLAOFE OA, BURTON SG, COWAN DA, HARRISON STL. Improving the production of a thermostable amidase through optimising IPTG induction in a highly dense culture of recombinant Escherichia coli[J]. Biochemical Engineering Journal, 2010, 52(1): 19-24.
|
| [25] |
CHEN JP, GONG JS, SU C, LI H, XU ZH, SHI JS. Improving the soluble expression of difficult-to-express proteins in prokaryotic expression system via protein engineering and synthetic biology strategies[J]. Metabolic Engineering, 2023, 78: 99-114.
|
| [26] |
RENNIG M, MUNDHADA H, WORDOFA GG, GERNGROSS D, WULFF T, WORBERG A, NIELSEN AT, NØRHOLM MHH. Industrializing a bacterial strain for L-serine production through translation initiation optimization[J]. ACS Synthetic Biology, 2019, 8(10): 2347-2358.
|
 2024, Vol. 40
2024, Vol. 40