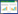中国科学院微生物研究所、中国微生物学会主办
文章信息
- 郑棚, 闫更轩, 王丽敏, 张云志, 陶勇, 于波
- ZHENG Peng, YAN Gengxuan, WANG Limin, ZHANG Yunzhi, TAO Yong, YU Bo
- 丙酸的合成生物制造
- Biomanufacturing of propionic acid
- 生物工程学报, 2024, 40(8): 2678-2694
- Chinese Journal of Biotechnology, 2024, 40(8): 2678-2694
- 10.13345/j.cjb.240097
-
文章历史
- Received: February 3, 2024
- Accepted: April 22, 2024
2. 黑龙江省科学院微生物研究所, 黑龙江 哈尔滨 150010
2. Institute of Microbiology, Heilongjiang Academy of Sciences, Harbin 150010, Heilongjiang, China
丙酸是一种重要的平台化学品,可作为广谱的微生物抗菌剂[1-2]、防腐剂[3]、人造香料和药物前体等[4]。工业生产丙酸主要是通过化学合成,以石油等化工产品为原料,经过加温、加压催化合成丙酸。合成方法包括轻油低碳烃氧化法、乙烯羰基化法(Reppe法)、乙醇羰基化法(Larson法)、丙醛氧化法、丙烯腈法和丙烯酸加氢法等[5]。
由于石油原料的不可再生性,开发基于生物基原料的丙酸合成方法受到广泛关注[6]。微生物发酵法是最常用的生物合成丙酸方法,以丙酸杆菌属(Propionibacterium)为主要生产菌株,利用葡萄糖、甘油、乳酸等生物基碳源为底物,通过厌氧发酵工艺合成丙酸[6]。丙酸的生物合成途径分为琥珀酸途径、丙烯酸途径和丙二醇途径,其中琥珀酸途径是丙酸杆菌最常见的发酵途径。在丙酸杆菌中,丙酸的合成主要由糖酵解、磷酸戊糖途径、三羧酸循环(tricarboxylic acid cycle, TCA)和Wood-Werkman循环结合完成,主要副产物为乙酸和琥珀酸。虽然丙酸生物发酵的研究和工业生产具有悠久的历史,但利用丙酸杆菌生产丙酸依然存在以下几个技术问题:(1) 发酵终产物丙酸对发酵菌株有着很强的抑制作用,造成丙酸产量难以达到理想水平;(2) 丙酸杆菌是一种厌氧菌,在厌氧发酵过程中,由于能量供应不足,细菌的生长速度较慢,因此导致发酵周期较长,最终影响了丙酸的生产强度;(3) 丙酸发酵液中含有乳酸、乙酸、琥珀酸等副产物,造成终产物纯度不高。受制于目前丙酸杆菌缺乏高效的遗传操作工具,通过基因工程改造丙酸杆菌提升丙酸产量的研究进展缓慢。
近年来,随着合成生物学技术的发展,国内外学者从异源宿主重构发酵合成途径和构建新催化合成途径等方面开展了丙酸合成研究,推动了丙酸生物合成技术的发展。通过对自然界中存在的丙酸代谢途径进行研究,识别并选取关键途径,并将这些途径重构到选定的宿主微生物中(例如大肠杆菌和酵母),实现了丙酸的高效生产。在这个过程中,优化代谢流通向丙酸生产,减少副产物的生成,是提高丙酸产量和生产效率的关键。合成生物学在丙酸生物合成技术的发展中有着举足轻重的作用,它不仅推动了丙酸生产途径的创新和优化,还为实现更加环保和可持续的化工生产提供了新的策略和方法。本文聚焦丙酸合成途径的设计,从丙酸杆菌的代谢工程改造、非天然宿主异源途径重构及假单胞菌生物转化合成3个方面综述了丙酸合成生物制造的研究进展。
1 代谢工程改造丙酸杆菌发酵合成丙酸利用微生物发酵生产丙酸的研究可追溯至20世纪初[7]。早期用于发酵合成丙酸的菌种全部来源于自然界,包括丙酸杆菌[8]、同型丙酸梭菌[9]、反刍硒菌[10]、环状微孔菌[11]以及新丙酸梭菌等[12]。丙酸杆菌属是目前最为熟知的丙酸生产菌属,其体内存在天然的丙酸合成途径Wood-Werkman循环(图 1)[13]。此外,丙酸杆菌能够利用多种碳源合成丙酸,包括葡萄糖、木糖、甘油、乳酸和纤维素水解物等,可降低丙酸合成的原料成本[7]。然而,丙酸杆菌属生产丙酸也具有发酵时间长、菌株生长慢、产物浓度低、副产物乙酸和琥珀酸多等问题,导致生产成本较高[14]。为了提高微生物生产丙酸的经济性和可持续性,大量研究聚焦于改造丙酸生产菌株,提升丙酸的产量和转化率。表 1总结了目前改造丙酸杆菌发酵合成丙酸的技术进展。
|
| 图 1 Wood-Werkman循环合成丙酸途径 Fig. 1 Schematic diagram of the synthesis of propionic acid through the Wood-Werkman cycle pathway. 1: MethylmalonyL-CoA carboxyltransferase; 2: Pyruvate carboxylase; 3: Malate dehydrogenase; 4: Fumarase; 5: Succinate dehydrogenase; 6: PropionyL-CoA: succinate CoA transferase; 7: MethylmalonyL-CoA mutase; 8: MethylmalonyL-CoA epimerase; 9: Pyruvate dehydrogenase; 10: Phosphotransacetylase. PEP: Phosphoenolpyruvate. 1:甲基丙二酰辅酶A羧化转移酶;2:丙酮酸羧化酶;3:苹果酸脱氢酶;4:延胡索酸酶;5:琥珀酸脱氢酶;6:丙酰辅酶A: 琥珀酸辅酶A转移酶;7:甲基丙二酰辅酶A变异酶;8:甲基丙二酰辅酶A差向异构酶;9:丙酮酸脱氢酶;10:磷酸乙酰转移酶 |
| |
| Strain | Genotype | Process | Substrate | Titer (g/L) |
Yield (g/g) |
Productivity (g/(L·h)) |
Purity (%) |
References |
| Propionibacterium acidipropionici | Wild type | Fed-batch | Glycerol | 22.60 | 0.45 | 0.03 | 96.0 | [15] |
| Propionibacterium acidipropionici | Wild type | Fed-batch | Glucose | 16.00 | 0.22 | 0.02 | 90.0 | [16] |
| Propionibacterium acidipropionici | Deletion of ack | Batcha | Glycerol | 106.00 | 0.53 | 0.04 | 86.0 | [17] |
| Propionibacterium acidipropionici | Wild type | Batch | Lactate | 15.10 | 0.44 | 0.12 | 73.0 | [8] |
| Propionibacterium acidipropionici | Wild type | Co-fermentation | Glycerol+ Glucose | 29.20 | 0.57 | 0.15 | 91.6 | [18] |
| Propionibacterium acidipropionici | Wild type | Fermentation | Glucose | 53.10 | 0.53 | 0.32 | 63.6 | [19] |
| Propionibacterium acidipropionici | Wild type | Sequential batch |
Glycerol | 27.30 | 0.63 | 1.42 | 80.0 | [20] |
| Propionibacterium acidipropionici | Wild type | Fed-batch | Corn stover hydrolysate | 64.70 | 0.50 | 0.77 | 70.0 | [21] |
| Propionibacterium acidipropionici | Wild type | Sequential batch |
Soy molasses | 33.00 | 0.42 | 0.81 | 63.5 | [22] |
| Propionibacterium freudenreichii | Wild type | Fed-batcha | Glucose | 67.00 | 0.43 | 0.14 | 78.0 | [23] |
| Propionibacterium freudenreichii | Wild type | Fed-batcha | Cane molasses | 92.00 | 0.46 | 0.36 | 80.0 | [24] |
| Propionibacterium jensenii ATCC 4868 | Overexpression of gldA |
Fed-batchb | Glycerol | 39.50 | N/A | 0.18 | N/A | [25] |
| Propionibacterium freudenreichii | Overexpression of CoA transferase |
Batch | Glycerol | 35.00 | 0.62 | 0.41 | N/A | [26] |
| Propionibacterium freudenreichii | Overexpression of ppc |
Batch | Glucose | 20.00 | 0.51 | 0.15 | N/A | [27] |
| Propionibacterium acidipropionici NRRL B-3569 |
Wild type | Continuous | Glycerol | 13.90 | 0.57 | 0.77 | N/A | [28] |
| Propionibacterium freudenreichii C.I.P. 59.32 | Wild type | Batch | Glucose | 21.80 | 0.31 | 0.57 | N/A | [29] |
| Propionibacterium freudenreichii DSM 4902 | Wild type | Batch | Sorghum bagasse hydrolysate | 22.00 | 0.45 | 0.17 | N/A | [30] |
| a: Fed-batch fermentation in fibrous-bed bioreactor; b: Fed-batch fermentation by engineered strain with an oxidoreduction potentiaL-shift control strategy. N/A: The data could not be obtained from the reference. | ||||||||
利用廉价生物质可显著降低丙酸发酵生产成本,并提高生物过程的可持续性[31]。丙酸杆菌已被证实可高效利用纤维素,且预处理过程中产生的某些抑制剂和纤维素水解产物还可促进丙酸杆菌的生长[32]。将菌株性能强化和廉价生物质利用相结合,会进一步提高丙酸杆菌厌氧发酵丙酸的经济性。Wei等[33]在不能利用木糖的费氏丙酸杆菌中过表达了酸性丙酸杆菌中木糖异构酶(xylose isomerase, xylA)、木糖转运蛋白(xylose transporter, xylT)和木糖激酶(xylokinase, xylB)等木糖分解代谢途径基因,使其即使在葡萄糖存在的情况下也能有效地利用木糖,而不会引起葡萄糖诱导的碳分解代谢抑制,经过改造的费氏丙酸杆菌以木质纤维素为原料生产丙酸时,产量可达到13.50 g/L,生产率为0.14 g/(L·h),转化率为0.41 g/g。除纤维素外,粗甘油也可作为丙酸工业生产的一种原料。虽然目前甘油发酵合成丙酸的产率较低,但通过与葡萄糖共发酵可以克服上述问题,Zhang等[17]改造的工程菌在分批补料发酵模式下,利用甘油和葡萄糖共发酵合成丙酸,产量提高到71.00 g/L,转化率为0.44 g/g。因此,未来利用廉价而丰富的甘油和纤维素水解产物进行组合发酵,将显著降低丙酸的发酵成本[31]。
1.2 基因工程改造增强菌株的丙酸合成能力除了原料成本外,微生物自身发酵性能也是决定丙酸发酵经济性的重要因素。Luna-Flores等[34]将两个野生型丙酸菌株进行基因组重组,重组后菌株的丙酸产量比野生型菌株提高25.0%。Baur等[35]在糖丁酸梭状芽胞杆菌(Clostridium saccharoperbutylacetonicum)中重构丙烯酸酯途径来合成丙酸,该途径包含8步酶反应,包括丙酸辅酶A转移酶、乳酸辅酶A脱水酶、丙烯酰辅酶A还原酶和D-乳酸脱氢酶,但改造后的工程菌株仅能产生0.7 mmol/L的丙酸。Baur等[35]认为是代谢通量的不平衡导致丙烯酰辅酶A在细胞中积累,从而使得丙酸的产量较低;为了实现胞内丙烯酰辅酶A的代谢平衡,研究人员重新优化了丙酸合成操纵子的表达,使得丙酸的浓度增加了近4倍;该研究首次报道了利用重组梭状菌株生产丙酸的研究,为生物基丙酸的生产开辟了一条新的途径。Liu等[36]通过研究发现了詹氏丙酸杆菌代谢甘油合成丙酸的关键限速步骤;甘油首先经甘油脱氢酶(glycerol dehydrogenase, GDH)转化为二羟丙酮,二羟丙酮进一步转化为磷酸二羟丙酮和丙酮酸,在厌氧条件下丙酮酸通过苹果酸脱氢酶(malate dehydrogenase, MDH)转化为草酰乙酸,然后转化为苹果酸,在富马酸水合酶(fumarate hydratase, FUM)的催化下继续转化为富马酸;富马酸通过琥珀酰辅酶A、甲基丙二酰辅酶A、丙酰辅酶A等多个中间产物转化为丙酸;在整个代谢反应中GDH、MDH和FUM均为限速酶,因此研究人员通过过表达3个关键酶提高了丙酸产量,其中GDH和MDH的共表达使丙酸浓度从(26.95±1.21) g/L增加到(39.43±1.90) g/L。Ammar等[27]在弗氏丙酸杆菌中表达了大肠杆菌来源的磷酸烯醇丙酮酸羧化酶(phosphoenolpyruvate carboxylase, PPC),与野生型相比,过表达PPC的菌株生长速度更快,丙酸的产量显著提升。
1.3 增强菌株的酸胁迫抗性丙酸生产是一个典型的产物抑制过程,因此提升菌株对丙酸的耐受性是提高丙酸合成效率的关键。Guan等发现了2个关键的抗酸因子,分别是精氨酸脱氨酶和谷氨酸脱羧酶系统,它们通过维持细胞内pH稳态来保护丙酸杆菌免受丙酸胁迫[37-38]。Guan等[39]尝试通过改造这些元件来提高詹氏丙酸杆菌ATCC 4868的耐酸性和丙酸产量;将5个编码精氨酸脱氨酶和谷氨酸脱羧酶系统的基因(arcA、arcC、gadB、gdh和ybaS)在詹氏丙酸杆菌中过表达,经测试后5种酶活性比野生型高26.7%−489.0%。虽然工程菌株的生长速率降低,但是丙酸产量显著增加。过表达谷氨酸脱羧酶基因gadB对丙酸抗性和产量的提高最为有效;工程菌株对丙酸的抗性比野生型菌株提高了10倍以上,丙酸的产量和转化率分别达到10.81 g/L和0.56 g/g甘油,与野生菌株相比分别提高了22.0%和21.7%[39]。
丙酸杆菌的丙酸合成与细胞生长部分相关,因此细胞高密度培养也是解决丙酸产量低的有效策略。纤维床生物反应器能够维持较高的细胞密度与活力。此外,细胞的固定化也可以减少丙酸对细胞的酸胁迫。Zhang等[17]首先敲除了产丙酸丙酸杆菌(Propionibacterium acidipropionici)的乙酸激酶,然后将工程菌固定于纤维床生物反应中;在纤维床生物反应器中驯化3个月后,工程菌对丙酸耐受的浓度提升到100.00 g/L,远高于野生菌耐受的最高浓度(约71.00 g/L);同时为了解析耐酸机制和影响因素,对驯化菌株的细胞形态、蛋白质表达谱、H+-ATP酶活性等进行了表征,探究了丙酸对菌株生长的抑制机理;研究结果表明,从纤维床生物反应器中获得的适应性进化突变株具有更佳的棒状形态和更高的H+-ATP酶活性,赋予了菌株更强的耐酸能力。
1.4 降低发酵副产物,提升丙酸合成性能丙酸在合成的同时会产生乙酸和琥珀酸等副产物,使转化率降低。Navone等[40]为了提高能量供给和抑制乙酸形成,在费氏丙酸杆菌亚种Propionibacterium shermanii中过表达戊糖-磷酸途径的葡萄糖-6-磷酸-1-脱氢酶和6-磷酸葡萄糖醇内酯酶,来调控氧化还原平衡,提升丙酸的合成能力;结果表明,在指数生长阶段,工程菌株产生的丙酸与乙酸的比值增加了4倍,丙酸的生产速率提高了13.0%,而琥珀酸的含量没有显著增加;进一步过表达ATP依赖性磷酸烯醇丙酮酸羧激酶和甲基丙二酰辅酶A脱羧酶来提高丙酸生产的能力,延长了菌株指数生长阶段,为丙酸生物合成过程中减少副产物形成提供了新思路。Wang等[26]在P. shermanii中过表达了琥珀酸辅酶A转移酶来研究菌株利用葡萄糖、甘油及其混合物为碳源发酵生产丙酸的影响;研究结果表明,工程菌的丙酸产量显著增加,转化率由原来的0.56 g/g增加到0.62 g/g,丙酸生产速率由原来的0.28 g/(L·h)增加到0.41 g/(L·h),提高了46.0%;在以葡萄糖和甘油为混合碳源的发酵中,丙酸与乙酸的比值提高了50.0%,丙酸与琥珀酸的比值也提高了23.0%,这表明工程菌产生的副产物乙酸和琥珀酸也显著降低;代谢通量分析结果表明,琥珀酸辅酶A转移酶的过表达能够将更多的碳通量转向丙酸,并且与葡萄糖利用相比,工程菌株更倾向利用甘油作为碳源。
综上所述,通过理性设计可以增强丙酸合成,并减少最终产物的抑制。同时,使用廉价可再生生物原料可以提高丙酸杆菌丙酸发酵的经济性。虽然通过菌株改造、生物工艺开发和廉价碳源利用能够一定程度上克服丙酸发酵的缺点,但是丙酸杆菌较低的生长速率和较长发酵周期仍然是限制因素。因此,为了进一步提升丙酸合成效率,需要构建性能更优的合成菌株。
2 代谢工程改造非天然宿主发酵生产丙酸除了改造丙酸杆菌,近年来,也有部分报道改造非天然宿主发酵生产丙酸的案例(表 2),本章节主要介绍在大肠杆菌和酿酒酵母宿主导入异源丙酸合成路径的代谢工程进展。
| Strain | Genotype | Substrate | Titer (g/L) |
Molar yield (%) | References |
| Escherichia coli | BW25113-∆ldhA, Ptrc: : sbm, ΔgldAΔdhaKΔptsI | Glycerol | 11.00 | 50.0 | [13] |
| E. coli | MG1655seq+/pBRPBAD and RBS‐optimized version of the Wood-Werkman cycle | Glucose | 0.62 | − | [41] |
| E. coli | MG1655-ΔldhA ΔpflB ΔptsI Ppck-galP-pckaΔducBΔducC | Glucose | 4.95 | 49.0 | [42] |
| E. coli | BW25113-∆ldhA, Ptrc: : sbmΔsdhΔiclR | Glucose | 30.90 | 49.7 | [43] |
| Saccharomyces cerevisiae | PPYK1: : PQCR10, Y22-tdcB-kivD-aldH, YCplac33, PPGK1-ThrA-TCYC1, PADH1-ThrB-TRPS2, PTDH3-ThrC-TADH1, PPGK1-HOM3-TCYC1, PADH1-ppc-TRPS2, PTDH3-aspC-TADH1 | Glucose | 1.05 | − | [44] |
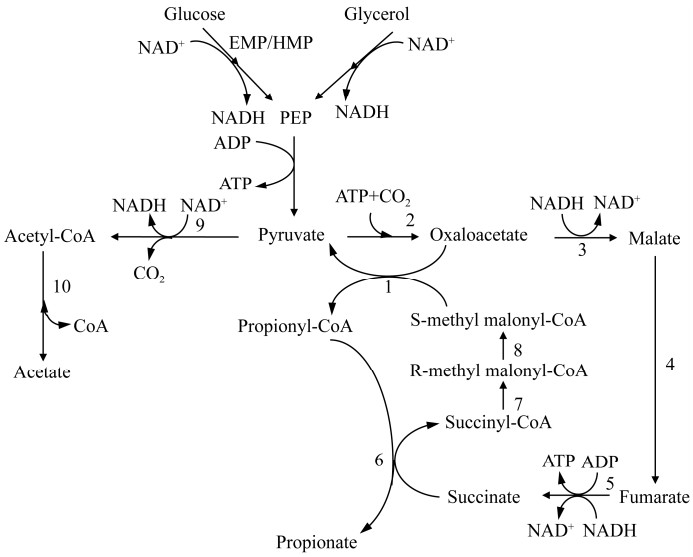
大肠杆菌是工业生产中最重要的微生物底盘,具有生长繁殖速度快、培养基需求简单、遗传操作成熟和易于生产放大等优势。近年来,也有部分研究人员尝试采用大肠杆菌系统开展丙酸合成途径的改造和优化。由于大肠杆菌缺乏催化丙酮酸异化为丙酸的天然酶,因此普遍认为野生大肠杆菌不具有合成丙酸的能力。Stine等[45]在前期开发了一个生物网络集成计算资源管理器(bionetwork integrated compute explorer, BNICE)的计算程序,它可以根据生化反应数据库的广义反应规则来预测混杂酶的活性并设计合成途径。天然途径合成丙酸会产生副产物乳酸和琥珀酸,而通过BNICE对丙酮酸合成丙酸的新途径进行预测,获得了5个不会有副产物生成的新途径,同时大多数途径中包括中间产物丙烯酸。Stine等[45]重点探究了丙烯酸还原为丙酸的酶催化过程,最终通过实验验证了来自酿酒酵母的Oye2p酶可以完成这一反应;通过验证发现,在大肠杆菌中过表达oye2p基因后,丙烯酸可被还原为丙酸;然而,利用oye2p还原丙烯酸生产丙酸的产率仅为0.17%,相对于多数已知丙酸合成途径的效率存在不足,仍需要开发更有效率的预测工具来为大肠杆菌的丙酸合成途径设计提供新的思路。Kandasamy等[46]首次通过在大肠杆菌内引入丙烯酸酯途径,实现了D-乳酸到丙酸的转化;这一途径由7个基因组成,分别编码丙酸辅酶A转移酶、乳酸辅酶A脱水酶和丙烯酰辅酶A还原酶,经改造后的大肠杆菌能够以葡萄糖为底物发酵合成丙酸,但产量远低于丙酸杆菌,仅为0.30 g/L。也有研究人员将Wood-Werkman循环在大肠杆菌中异源表达,经过多轮优化后仅合成了0.70 g/L的丙酸[41]。近年来发现大肠杆菌中存在一类新的由琥珀酸转化为丙酰辅酶A的代谢途径,该途径通过睡美人变位酶操纵子(sleeping beauty mutase, sbm)进行调控,其在天然大肠杆菌中处于休眠状态。如图 2所示,sbm操纵子由sbmA、ygfD、ygfG和ygfH这4个基因组成,除ygfD的功能尚不明确外,其他基因分别编码sbm途径的关键酶。目前,活化sbm操纵子并加强sbm代谢途径已成为构建产丙酸大肠杆菌的主要方法。Gonzalez等[41]通过活化sbm操纵子实现了琥珀酰辅酶A到丙酰辅酶A的转化,随后表达外源的双功能辅酶A脱氢酶,将丙酰辅酶A转化为丙醇和丙酸。随后研究发现,在葡萄糖为唯一碳源的条件下,简单活化sbm操纵子并不能使大肠杆菌生产丙酸,而在大肠杆菌中表达甲基丙二酰辅酶A差向异构酶基因后,可实现利用葡萄糖生产丙酸[13]。Akawi等[42]敲除了参与甘油异化途径的甘油-3-磷酸脱氢酶与ATP依赖性甘油激酶,工程菌株的丙酸产量达到11.00 g/L。其他研究人员通过增强sbm循环前体供应、启动子优化、引入外源甲基丙二酰辅酶A差向异构酶等多种代谢途径改良策略,改造后大肠杆菌的丙酸产量达到4.95 g/L,葡萄糖到丙酸的转化率达到0.49 mol/mol[36]。Miscevic等[43]发现还原型TCA循环的琥珀酸到琥珀酰辅酶A的转化效率是强化sbm途径通量并提高丙酸生物合成的关键;通过阻断氧化型TCA循环和解除乙醛酸分流,构建的菌株发酵产生丙酸的浓度达到30.90 g/L,这也是目前报道的大肠杆菌发酵合成丙酸的最高产量。
|
| 图 2 依赖于sbm操纵子的大肠杆菌丙酸合成途径构建 Fig. 2 Construction of propionic acid synthesis pathway in Escherichia coli based on sbm operon. |
| |
由于丙酸对微生物的强抑制活性,丙酸积累会严重影响菌株生长,因此提高菌株对酸胁迫的抗性,增强其在酸性环境下的生存能力与产酸效率,已成为丙酸生产亟须解决的难点。虽然大肠杆菌的生长率高,但是耐酸性差,导致大肠杆菌工程菌株丙酸产率不高。为了克服上述问题,研究人员也尝试在其他底盘细胞中重构丙酸合成途径并取得了一定进展。酿酒酵母通常被认为是生物安全菌株,且对发酵抑制成分和pH有较强的耐受性,被广泛用于生物技术工业生产[47-49]。Ding等[44]强化了酿酒酵母L-苏氨酸合成和整合L-苏氨酸到丙酸的合成途径;构建的工程菌株在摇瓶中利用葡萄糖好氧发酵产生0.21 g/L丙酸盐;随后强化途径基因、消除竞争途径、弱化丙酮酸激酶和过表达磷酸烯醇式丙酮酸羧化酶,将碳流引入丙酸合成途径从而提高产量;在分批补料发酵工艺中,丙酸产量达到1.05 g/L。上述结果显示酿酒酵母有望成为丙酸好氧发酵生产的潜在宿主。
3 途径设计与优化实现丙酸的生物转化合成丙酸是一种强生长抑制剂,在低至1% (质量体积比)的浓度下能够将丙酸杆菌生长速率降低50%[50],这对丙酸发酵生产菌株提出了巨大挑战[51]。如前文所述,尽管在酿酒酵母异源重构丙酸发酵合成路径,但产量仍然非常低。除发酵法生产丙酸外,生物催化法也是一个有前景的技术方向。采用生物催化过程,避免丙酸合成对菌体生长的抑制,可以有效提高丙酸生物合成的效率。恶臭假单胞菌以其对一系列化学胁迫的显著耐受性而闻名,并已广泛用于有毒底物的生物转化[52-53],例如将呋喃化合物转化为糠酸等[54]。恶臭假单胞菌KT2440不含内毒素和抗性基因,已被证实为生物安全的菌株[55]。与大肠杆菌相比,恶臭假单胞菌由于其代谢多样性、低营养需求、较好的抗逆性及生物安全性而成为构建丙酸生物转化底盘细胞的更优选择。本章节主要综述了改造恶臭假单胞菌KT2440催化L-苏氨酸和1, 2-丙二醇等底物合成丙酸的研究进展(表 3)。
| Strain | Genotype | Substrate | Titer (g/L) |
Molar yield (%) | Productivity (g/(L·h)) |
References |
| Pseudomonas putida PS10 | ΔprpC, ΔltaE: : Plac-tdcBC, ΔltaE, ΔbkdR: : P119-bkd, ΔltaE: : Plac-ilvA, ΔlacI: : Plac-HiYciA, ΔprpE | L-threonine | 50.3 | 99.8 | 0.60 | [56] |
| P. putida PS34 | PS10ΔtesBΔpp_4975ΔlacI: : Plac-kivD; PUCP18-kiv > D-RBS25 | L-threonine | 62.0 | > 98.0 | 1.07 | [57] |
| P. putida TVG02 | PS10-Δ3458Δ2213Δ4487Δ4702 Δ5331Δ4975ΔvdlDΔ3807Δ1466 ΔtesBΔtesAΔ1218Δ2308ΔpaaY ΔpaaIΔ1980Δ2050Δ0262Δ4180−4181 Δ4105Δ0254Δ0301ΔLacI: : PBAD-KPpdu |
1, 2-propanediol | 46.5 | > 99.0 | 1.55 | [58] |
| P. putida EM42 | ΔprpC: ΔprpE pPROBE_LvaR/PlvaA_yciA_ygfH |
Levulinic acid | 26.8 | 83.0 | 0.30 | [59] |
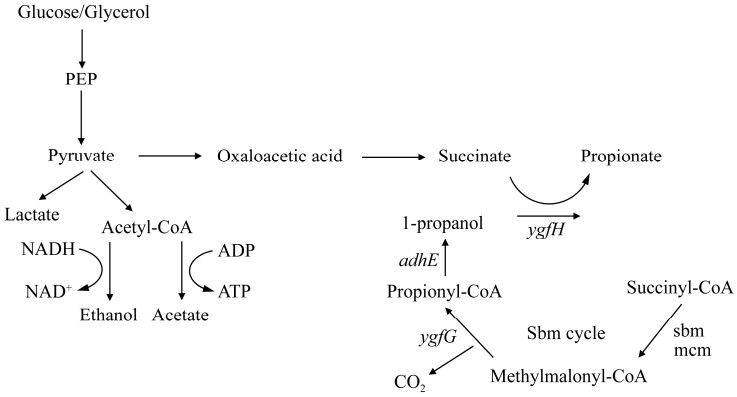
L-苏氨酸是大宗氨基酸,目前国内的年产能超百万吨。以L-苏氨酸为原料生产丙酸具有原料成本优势和经济性。大肠杆菌和假单胞菌等体内存在天然的L-苏氨酸降解合成丙酸的途径。如图 3所示,L-苏氨酸经过脱氨酶作用合成2-酮丁酸,进一步在裂解酶或支链酮酸脱氢酶的催化下合成丙酰辅酶A,随后被转化为丙酸。不同的是,大肠杆菌的L-苏氨酸降解途径产生甲酸[60],而恶臭假单胞菌KT2440降解L-苏氨酸合成丙酸的过程中,CO2为唯一副产物[57]。由于甲酸与丙酸分离纯化困难,甲酸的存在会造成终产物丙酸纯度不高。因此,改造假单胞菌的L-苏氨酸降解途径可以合成高纯度的生物基丙酸。
|
| 图 3 大肠杆菌和假单胞菌L-苏氨酸降解途径的比较 Fig. 3 The differences of L-threonine degradation pathways in Escherichia coli and Pseudomonas putida. |
| |
Mu等[57]通过改造恶臭假单胞菌KT2440的L-苏氨酸降解途径,率先实现转化L-苏氨酸合成丙酸;在恶臭假单胞菌菌株KT2440中,分别敲除分支代谢途径的甲基柠檬酸合酶和L-苏氨酸醛缩酶,阻断催化L-苏氨酸裂解为甘氨酸和乙醛,构建的初代菌株实现了丙酸的积累;接下来将大肠杆菌来源的L-苏氨酸通透酶(TdcB)整合到菌株基因组以加强L-苏氨酸的内运速率,随后将第二步反应中支链α-酮酸脱氢酶复合物的天然启动子更换为强启动子J23119,提高2-酮丁酸脱羧反应活性;进一步导入大肠杆菌来源的脱氨酶和异源表达来自流感嗜血杆菌的HiYciA硫酯酶显著增强了丙酸的合成能力;最后敲除丙酰辅酶A合成酶PrpE,构建的工程菌PS10在48 h内将400 mmol/L L-苏氨酸转化为399 mmol/L丙酸,摩尔转化率达到99.8%。
值得注意的是,在上述假单胞菌菌株的改造过程中,中间菌株PS06可以催化L-苏氨酸合成较高浓度的丙酸盐,但该菌株并未进行硫脂酶的强化[57],这证明KT2440菌株体内存在天然催化丙酰辅酶A水解的酶。Ma等[61]通过对KT2440菌株体内的13个基因注释的硫酯酶进行测试,开展系统的基因迭代缺失实验,成功地发现了KT2440体内负责丙酸生产的主要硫酯酶,特别是缺失硫酯酶PP_4975显著降低了丙酸产量,而经基因回补后恢复了性能;与来自流感嗜血杆菌的硫酯酶HiYciA相比,硫酯酶PP_4975在体外显示出更快的底物转化速率;该研究完善了恶臭假单胞菌KT2440的L-苏氨酸代谢过程,也为进一步改进菌株丙酸合成效率提供了新靶点。另一方面,为了提高假单胞菌的丙酸耐受性,研究人员通过比较转录组分析确定了恶臭假单胞菌KT2440的丙酸耐受关键因子,通过实验证实易化超家族(major facilitator superfamily, MFS)转运蛋白基因簇PP_1271、PP_1272和PP_1273参与恶臭假单胞菌KT2440菌株的丙酸耐受性[62]。过表达PP_1271−1273基因簇提高了菌株对丙酸的耐受性,并提升了丙酸的合成速率,该结果为恶臭假单胞菌丙酸胁迫响应研究提供了新的见解,并为开发丙酸催化合成高效菌株奠定了分子基础。
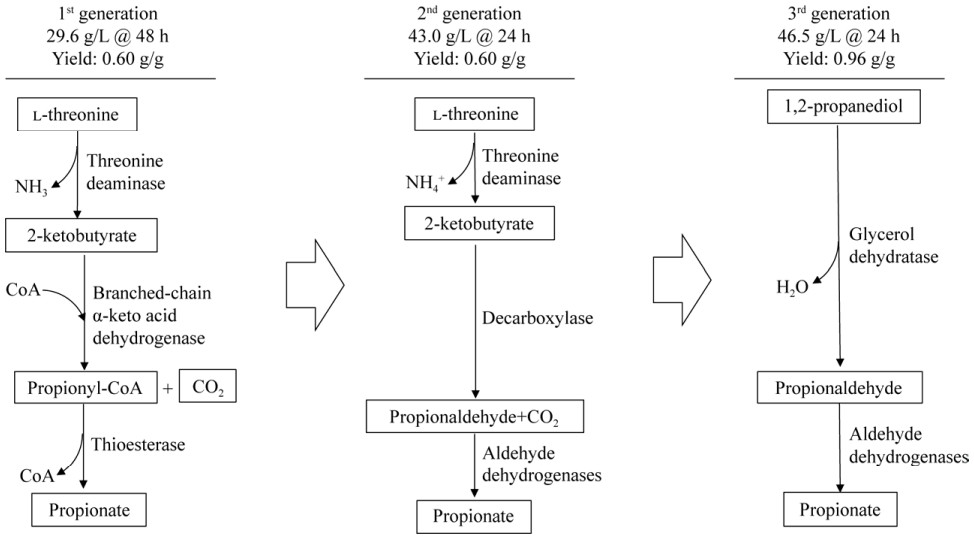
3.1.2 L-苏氨酸合成高纯度丙酸的人工途径设计与优化上述改造实现了L-苏氨酸到丙酸的高效生物转化,证实恶臭假单胞菌KT2440是有潜力的丙酸催化合成的底盘细胞。虽然上述催化工艺可以产生29.6 g/L的丙酸,L-苏氨酸的摩尔转化率达到99.8%,但生产周期需要48 h,效率仍有待进一步提升[62]。在假单胞菌降解L-苏氨酸的天然途径中,催化裂解2-酮丁酸的支链α-酮酸脱氢酶复合物(BKD,由bkdAa、bkdAb、bkdB和lpdV编码)是由3种催化组分组成的多酶复合物:二磷酸硫胺素依赖性脱羧酶/脱氢酶(E1)、硫辛酰转酰酶(E2)和二氢硫辛酰胺脱氢酶(E3)。E3与丙酮酸脱氢酶共享相同亚基,BKD的巨大复杂结构通常会阻碍其过表达[63]。此外,该L-苏氨酸降解途径需要辅酶A参与。虽然途径中辅酶A可以在最后一步实现自循环,但作为细胞代谢中的重要辅因子之一,辅酶A胞内浓度很难提升到较高水平以满足丙酸工业合成的效率[64-65]。为了进一步提高丙酸生产效率,应设计一条不涉及辅酶A参与的丙酸合成途径。研究报道甜瓜的丙酮酸脱羧酶可以裂解2-酮丁酸形成丙醛和CO2,而丙醛可以被生物氧化为丙酸[66]。根据这些数据,本课题组提出了从L-苏氨酸生产丙酸的新途径:即L-苏氨酸首先被脱除氨基生成2-酮丁酸,然后通过脱羧酶丙醛和CO2,然后氧化成丙酸。这种不依赖辅酶A且仅CO2作为副产物的途径可以促进从L-苏氨酸以高生产率生产高纯度丙酸。基于上述设计思路,本课题组在改造天然L-苏氨酸降解途径合成丙酸的底盘菌株基础上,导入乳酸乳球菌来源的2-酮异戊酸脱羧酶,实现了2-酮丁酸的高效裂解,构建的工程菌株可在24 h内催化600 mmol/L L-苏氨酸产生580 mmol/L (43.0 g/L)的丙酸[57]。此外,在补料分批模式中实现了62.0 g/L丙酸的高滴度,生产速率达到1.07 g/(L·h),对L-苏氨酸的摩尔转化率 > 0.98[57]。进一步建立了直接利用L-苏氨酸发酵液转化合成丙酸的新工艺,避免了L-苏氨酸的纯化,可大幅降低丙酸的生产成本。
值得注意的是,上述改造虽然实现了假单胞菌从L-苏氨酸到丙酸的高效生产,但并未强化丙醛氧化成丙酸的步骤,仍不清楚其体内氧化丙醛形成丙酸的内源酶。恶臭假单胞菌KT2440拥有较大的基因组,进化出复杂调控系统以适应各种环境胁迫。本课题组通过全基因组筛选,发现KT2440体内含有32个醛脱氢酶,这解释了KT2440对丙醛表现出超氧化活性的原因[59]。然而,迄今为止,只有少数几种醛脱氢酶被定性,对这32个注释的醛脱氢酶进行详细分析,通过测量在丙醛胁迫条件下的各个醛脱氢酶基因转录水平变化,结合相关基因的缺失和回补实验,以及在大肠杆菌异源表达醛脱氢酶基因等实验过程,确定了至少8个醛脱氢酶对丙醛具有高氧化能力,在分子水平上揭示了恶臭假单胞菌KT2440的强氧化能力,并再次证实了它是一种优良的有毒化合物生产宿主[67-69]。
3.2 1, 2-丙二醇转化路径合成丙酸的途径设计与优化虽然利用L-苏氨酸催化合成生物基丙酸具有经济性,但该途径的理论质量得率较低。L-苏氨酸经过2个连续的脱氨和脱羧反应后,丙酸的理论得率仅为0.62 g/g[57]。除了L-苏氨酸之外,1, 2-丙二醇也是一种廉价的大宗化学品。这两种化合物都是三碳原子骨架,从1, 2-丙二醇得到丙酸的理论产率可达0.97 g/g,在该反应中仅损失2个氢原子且无任何副产物,因此利用1, 2-丙二醇生产丙酸的途径具有高原子经济性的优势;目前已经实现利用甘油氢化还原或葡萄糖裂解合成生物基1, 2-丙二醇的工业化生产[70]。因此,利用生物基1, 2-丙二醇原料可以满足生物基丙酸的合成需求。我们进一步设计了1, 2-丙二醇合成丙酸的生物路径,即通过甘油脱水酶将1, 2-丙二醇脱水获得丙醛,然后利用恶臭假单胞菌KT2440自身超强氧化能力将丙醛氧化为丙酸;在设计的新途径中,仅需要表达外源甘油脱水酶,这将显著减轻工程菌株的代谢负担[58]。研究人员首先筛选了不同来源的甘油脱水酶,最终选定了来自肺炎克雷伯菌(Klebsiella pneumoniae)的甘油脱水酶(KPpdu);当前期采用强启动子驱动KPpdu在恶臭假单胞菌中表达时,菌株总是产生等量的丙酸和副产物1-丙醇[58]。生物体对醛类物质的生物解毒过程是催化合成有机醇或有机酸化合物;研究人员推测是甘油脱水酶活性过高导致丙醛积累,激发菌株对醛类物质的解毒机制,从而产生副产物1-丙醇;积累的丙醛易挥发,从而造成转化率偏低;通过精细调控甘油脱水酶KPpdu的表达,实现了1, 2-丙二醇脱水反应与胞内氧化能力相平衡;改造后的工程菌株可在24 h内将400 mmol/L 1, 2-丙二醇完全转化为丙酸,且无明显的丙醛或1-丙醇的积累[58]。这一结果表明,甘油脱水酶基因的表达水平对该途径至关重要。此外,丙醛还原为1-丙醇需要辅酶NADH的参与。恶臭假单胞菌KT2440菌株具有强的有氧呼吸系统,且具有抵抗不同应激的高缓冲能力[71]。在好氧条件下,细胞内NADH可以迅速被氧化为NAD+,这也有助于消除副产物1-丙醇的合成[47]。在进一步的分批转化工艺中,丙酸的产量达到46.5 g/L,生产速率和质量得率分别为1.55 g/(L·h)和0.96 g/g[57]。需要注意的是,甘油脱水酶的活性需要维生素B12维持。据报道,恶臭假单胞菌KT2440可以产生微量的维生素B12,但浓度太低(< 50 μg/L),无法支持甘油脱水酶的活化[72]。因此,进一步改造恶臭假单胞菌菌株以增加维生素B12的细胞内供应是菌株改进的方向。虽然目前的1, 2-丙二醇路线合成丙酸的滴度略低于L-苏氨酸路线,但实现了0.96 g/g的高质量产率,也显示出较好的工业应用潜力,后续可以通过进一步优化菌株以及生物转化过程来提高滴度[58]。
综上所述,在丙酸的生物合成制造技术发展过程中,第一代技术改造假单胞菌天然途径,实现转化L-苏氨酸高效合成丙酸;第二代技术人工设计转化路径,突破了天然途径的辅酶A限制,实现了转化L-苏氨酸合成丙酸技术的跃升。考虑到L-苏氨酸为四碳氨基酸,合成三碳的丙酸需要经过脱氨和脱羧步骤,原子经济性不高,研究人员进一步设计了以1, 2-丙二醇为原料的第三代丙酸生物催化合成新技术并实现了高得率的转化过程(图 4)。
|
| 图 4 生物催化合成丙酸技术水平的迭代提升 Fig. 4 Schematic diagram of the synthesis of propionic acid via biotransformation route. |
| |
除了本课题组开展的L-苏氨酸和1, 2-丙二醇为底物转化合成丙酸的研究外,Tiwari等[59]也阐述了改造恶臭假单胞菌EM42利用生物质衍生的乙酰丙酸生产丙酸。恶臭假单胞菌EM42是KT2440菌株的基因组删减菌株,具有更快的生长速率。乙酰丙酸是一种γ-酮酸(C5)的平台化合物,可以通过酸催化脱水和水解木质纤维素糖而获得,也是一种廉价的生物基原料。研究人员首先删除恶臭假单胞菌EM42的甲基柠檬酸合酶(metheyL-citrate synthase, PrpC)和丙酰辅酶A合酶(propionyl CoA synthase, PrpE)基因,随后,采用LA诱导型表达系统表达来自流感嗜血杆菌的硫酯酶(YciA)和大肠杆菌的来源的琥珀酸辅酶A转移酶(succinyL-CoA transferase, YgfH),在摇瓶培养条件下,丙酸产量提高了10倍;进一步优化发酵工艺,改造的恶臭假单胞菌在5 L发酵罐中,补料分批发酵生产丙酸的滴度和合成速率分别为26.80 g/L和0.30 g/(L·h);虽然该研究也取得了较好的结果,但仅实现了83.0%的摩尔转化率,还需进一步改造提升经济性,同时乙酰丙酸到丙酸的理论质量得率仅为0.64 g/g,与L-苏氨酸路线一样存在理论得率低的缺陷[59]。
4 结论与展望丙酸是应用非常广泛的食品添加剂、人工香料和防腐剂。作为重要的工业产品,丙酸在涂料、化妆品、药物和农药等方面也有着广泛应用。产物生产速率过低和下游提取成本高是目前限制丙酸生物发酵过程经济性的主要因素。由于丙酸对微生物的强抑制活性,丙酸会严重影响菌株生长,因此提高菌株对酸胁迫的抗性,增强其在酸性环境下的生存能力与产酸效率,已成为技术提升亟须解决的问题。研究人员通过开发生物催化工艺,实现了高纯度丙酸的生物合成。随着合成生物学的发展,有机酸分子转运蛋白的挖掘和非传统耐酸底盘细胞工厂的构建也将是实现丙酸高效发酵生产和提高经济性的有效手段。例如,库德里阿兹氏毕赤酵母(Pichia kudriavzevii)是一种优秀的耐酸酵母,可以在pH低于2.0的恶劣环境中生长。与丝状真菌相比,库德里阿兹氏毕赤酵母具有更快的生长速度、液体培养过程中更好的细胞形态均一性和更简单的遗传操作等优势,可以作为有机酸生产的优良宿主[73]。近期,Zhang等[74]改造了库德里阿兹氏毕赤酵母菌株E1实现了无中和剂的L-乳酸的低pH值发酵生产。该研究思路也为工程化耐酸菌株绿色生产高纯度生物基丙酸提供了可能。
综上所述,代谢工程和生物催化在生产丙酸等化学品方面的应用展现出了巨大的潜力。随着合成生物学和人工智能技术的不断进步,通过合成途径优化和菌株抗性工程改造能够极大拓展丙酸合成底盘细胞的性能,大幅提升丙酸发酵技术水平,更广泛地应用于生物制造。目前,丙酸的生物合成主要依赖于少数几种微生物宿主,如丙酸杆菌、大肠杆菌和酵母。未来可以探索和开发更多种类的微生物宿主,特别是那些具有独特代谢特性、原料利用能力强、耐受高浓度产品的非传统底盘微生物。通过对这些微生物的代谢途径和基因组进行深入研究和优化,可以实现更高效、更具成本效益的丙酸生产。同时,许多丙酸生产过程依赖于糖或其他传统农业原料作为碳源,因此需要拓展其他原料来源,包括使用非食用生物质(如农业废弃物、木质纤维素)和二氧化碳等可再生资源,这不仅可以降低生产成本,还有助于减少对食品供应的竞争和环境影响,对提高丙酸发酵的经济可行性和可持续性具有重要意义。
| [1] |
HUANG CB, ALIMOVA Y, MYERS TM, EBERSOLE JL. Short- and medium-chain fatty acids exhibit antimicrobial activity for oral microorganisms[J]. Archives of Oral Biology, 2011, 56(7): 650-654. DOI:10.1016/j.archoralbio.2011.01.011
|
| [2] |
RIVERO S, GIANNUZZI L, GARCÍA MA, PINOTTI A. Controlled delivery of propionic acid from chitosan films for pastry dough conservation[J]. Journal of Food Engineering, 2013, 116(2): 524-531. DOI:10.1016/j.jfoodeng.2012.12.025
|
| [3] |
SABRA W, DIETZ D, ZENG AP. Substrate-limited co-culture for efficient production of propionic acid from flour hydrolysate[J]. Applied Microbiology and Biotechnology, 2013, 97(13): 5771-5777. DOI:10.1007/s00253-013-4913-y
|
| [4] |
SHAMS S, FOLEY KA, KAVALIERS M, MacFABE DF, OSSENKOPP KP. Systemic treatment with the enteric bacterial metabolic product propionic acid results in reduction of social behavior in juvenile rats: contribution to a rodent model of autism spectrum disorder[J]. Developmental Psychobiology, 2019, 61(5): 688-699. DOI:10.1002/dev.21825
|
| [5] |
EŞ I, KHANEGHAH AM, HASHEMI SMB, KOUBAA M. Current advances in biological production of propionic acid[J]. Biotechnology Letters, 2017, 39(5): 635-645. DOI:10.1007/s10529-017-2293-6
|
| [6] |
VIDRA A, NÉMETH Á. Bio-produced propionic acid: a review[J]. Periodica Polytechnica Chemical Engineering, 2017, 62(1): 57-67. DOI:10.3311/PPch.10805
|
| [7] |
PIWOWAREK K, LIPIŃSKA E, HAĆ-SZYMAŃCZUK E, KIELISZEK M, ŚCIBISZ I. Propionibacterium spp. : source of propionic acid, vitamin B12, and other metabolites important for the industry[J]. Applied Microbiology and Biotechnology, 2018, 102(2): 515-538. DOI:10.1007/s00253-017-8616-7
|
| [8] |
CORAL J, KARP SG, PORTO de SOUZA VANDENBERGHE L, PARADA JL, PANDEY A, SOCCOL CR. Batch fermentation model of propionic acid production by Propionibacterium acidipropionici in different carbon sources[J]. Applied Biochemistry and Biotechnology, 2008, 151(2): 333-341.
|
| [9] |
SEELIGER S, JANSSEN PH, SCHINK B. Energetics and kinetics of lactate fermentation to acetate and propionate via methylmalonyL-CoA or acrylyL-CoA[J]. FEMS Microbiology Letters, 2002, 211(1): 65-70. DOI:10.1111/j.1574-6968.2002.tb11204.x
|
| [10] |
EATON DC, GABELMAN A. Fed-batch and continuous fermentation of Selenomonas ruminantium for natural propionic, acetic and succinic acids[J]. Journal of Industrial Microbiology, 1995, 15(1): 32-38. DOI:10.1007/BF01570010
|
| [11] |
SELDER L, SABRA W, JÜRGENSEN N, LAKSHMANAN A, ZENG AP. Co-cultures with integrated in situ product removal for lactate-based propionic acid production[J]. Bioprocess and Biosystems Engineering, 2020, 43(6): 1027-1035. DOI:10.1007/s00449-020-02300-0
|
| [12] |
ATO M, ISHII M, IGARASHI Y. Enrichment of amino acid-oxidizing, acetate-reducing bacteria[J]. Journal of Bioscience and Bioengineering, 2014, 118(2): 160-165. DOI:10.1016/j.jbiosc.2014.02.003
|
| [13] |
GONZALEZ-GARCIA RA, McCUBBIN T, WILLE A, PLAN M, NIELSEN LK, MARCELLIN E. Awakening sleeping beauty: production of propionic acid in Escherichia coli through the sbm operon requires the activity of a methylmalonyL-CoA epimerase[J]. Microbial Cell Factories, 2017, 16(1): 121. DOI:10.1186/s12934-017-0735-4
|
| [14] |
COLLOGRAI KC, Da COSTA AC, IENCZAK JL. Fermentation strategies to improve propionic acid production with propionibacterium ssp. : a review[J]. Critical Reviews in Biotechnology, 2022, 42(8): 1157-1179. DOI:10.1080/07388551.2021.1995695
|
| [15] |
CHEN Y, WANG T, SHEN N, ZHANG F, ZENG RJ. High-purity propionate production from glycerol in mixed culture fermentation[J]. Bioresource Technology, 2016, 219: 659-667. DOI:10.1016/j.biortech.2016.08.026
|
| [16] |
CHEN Y, SHEN N, WANG T, ZHANG F, ZENG RJ. Ammonium level induces high purity propionate production in mixed culture glucose fermentation[J]. RSC Advances, 2017, 7(1): 518-525. DOI:10.1039/C6RA25926J
|
| [17] |
ZHANG A, YANG ST. Engineering Propionibacterium acidipropionici for enhanced propionic acid tolerance and fermentation[J]. Biotechnology and Bioengineering, 2009, 104(4): 766-773. DOI:10.1002/bit.22437
|
| [18] |
LIU Y, ZHANG YG, ZHANG RB, ZHANG F, ZHU JH. Glycerol/glucose co-fermentation: one more proficient process to produce propionic acid by Propionibacterium acidipropionici[J]. Current Microbiology, 2011, 62(1): 152-158. DOI:10.1007/s00284-010-9683-5
|
| [19] |
STOWERS CC, COX BM, RODRIGUEZ BA. Development of an industrializable fermentation process for propionic acid production[J]. Journal of Industrial Microbiology & Biotechnology, 2014, 41(5): 837-852.
|
| [20] |
DISHISHA T, STÅHL Å, LUNDMARK S, HATTI-KAUL R. An economical biorefinery process for propionic acid production from glycerol and potato juice using high cell density fermentation[J]. Bioresource Technology, 2013, 135: 504-512. DOI:10.1016/j.biortech.2012.08.098
|
| [21] |
WANG XQ, SALVACHÚA D, NOGUÉ VSI, MICHENER WE, BRATIS AD, DORGAN JR, BECKHAM GT. Propionic acid production from corn stover hydrolysate by Propionibacterium acidipropionici[J]. Biotechnology for Biofuels, 2017, 10: 200. DOI:10.1186/s13068-017-0884-z
|
| [22] |
YANG H, WANG ZQ, LIN M, YANG ST. Propionic acid production from soy molasses by Propionibacterium acidipropionici: fermentation kinetics and economic analysis[J]. Bioresource Technology, 2018, 250: 1-9. DOI:10.1016/j.biortech.2017.11.016
|
| [23] |
FENG XH, CHEN F, XU H, WU B, YAO J, YING HJ, OUYANG PK. Propionic acid fermentation by Propionibacterium freudenreichii CCTCC M207015 in a multi-point fibrous-bed bioreactor[J]. Bioprocess and Biosystems Engineering, 2010, 33(9): 1077-1085. DOI:10.1007/s00449-010-0433-7
|
| [24] |
FENG XH, CHEN F, XU H, WU B, LI H, LI S, OUYANG PK. Green and economical production of propionic acid by Propionibacterium freudenreichii CCTCC M207015 in plant fibrous-bed bioreactor[J]. Bioresource Technology, 2011, 102(10): 6141-6146. DOI:10.1016/j.biortech.2011.02.087
|
| [25] |
ZHUGE X, LI JH, SHIN HD, LIU L, DU GC, CHEN J. Improved propionic acid production with metabolically engineered Propionibacterium jensenii by an oxidoreduction potentiaL-shift control strategy[J]. Bioresource Technology, 2015, 175: 606-612. DOI:10.1016/j.biortech.2014.10.038
|
| [26] |
WANG ZQ, AMMAR EM, ZHANG A, WANG LQ, LIN M, YANG ST. Engineering Propionibacterium freudenreichii subsp. shermanii for enhanced propionic acid fermentation: effects of overexpressing propionyL-CoA: succinate CoA transferase[J]. Metabolic Engineering, 2015, 27: 46-56. DOI:10.1016/j.ymben.2014.10.005
|
| [27] |
AMMAR EM, JIN Y, WANG ZQ, YANG ST. Metabolic engineering of Propionibacterium freudenreichii: effect of expressing phosphoenolpyruvate carboxylase on propionic acid production[J]. Applied Microbiology and Biotechnology, 2014, 98(18): 7761-7772. DOI:10.1007/s00253-014-5836-y
|
| [28] |
WALLENIUS J, PAHIMANOLIS N, ZOPPE J, KILPELÄINEN P, MASTER E, ILVESNIEMI H, SEPPÄLÄ J, EERIKÄINEN T, OJAMO H. Continuous propionic acid production with Propionibacterium acidipropionici immobilized in a novel xylan hydrogel matrix[J]. Bioresource Technology, 2015, 197: 1-6. DOI:10.1016/j.biortech.2015.08.037
|
| [29] |
BELGRANO FDS, VERÇOZA BRF, RODRIGUES JCF, HATTI‐KAUL R, PEREIRA N. EPS production by Propionibacterium freudenreichii facilitates its immobilization for propionic acid production[J]. Journal of Applied Microbiology, 2018, 125(2): 480-489. DOI:10.1111/jam.13895
|
| [30] |
AMMAR EM, MARTIN J, BRABO-CATALA L, PHILIPPIDIS GP. Propionic acid production by Propionibacterium freudenreichii using sweet sorghum bagasse hydrolysate[J]. Applied Microbiology and Biotechnology, 2020, 104(22): 9619-9629. DOI:10.1007/s00253-020-10953-w
|
| [31] |
AMMAR EM, PHILIPPIDIS GP. Fermentative production of propionic acid: prospects and limitations of microorganisms and substrates[J]. Applied Microbiology and Biotechnology, 2021, 105(16): 6199-6213.
|
| [32] |
RAMSAY JA, ALY HASSAN MC, RAMSAY BA. Biological conversion of hemicellulose to propionic acid[J]. Enzyme and Microbial Technology, 1998, 22(4): 292-295. DOI:10.1016/S0141-0229(97)00196-8
|
| [33] |
WEI PL, LIN M, WANG ZQ, FU HX, YANG H, JIANG WY, YANG ST. Metabolic engineering of Propionibacterium freudenreichii subsp. shermanii for xylose fermentation[J]. Bioresource Technology, 2016, 219: 91-97. DOI:10.1016/j.biortech.2016.07.056
|
| [34] |
LUNA-FLORES CH, PALFREYMAN RW, KRÖMER JO, NIELSEN LK, MARCELLIN E. Improved production of propionic acid using genome shuffling[J]. Biotechnology Journal, 2017, 12(2). DOI:10.1002/biot.201600120
|
| [35] |
BAUR T, WENTZEL A, DÜRRE P. Production of propionate using metabolically engineered strains of Clostridium saccharoperbutylacetonicum[J]. Applied Microbiology and Biotechnology, 2022, 106(22): 7547-7562. DOI:10.1007/s00253-022-12210-8
|
| [36] |
LIU L, ZHUGE X, SHIN HD, CHEN RR, LI JH, DU GC, CHEN J. Improved production of propionic acid in Propionibacterium jensenii via combinational overexpression of glycerol dehydrogenase and malate dehydrogenase from Klebsiella pneumoniae[J]. Applied and Environmental Microbiology, 2015, 81(7): 2256-2264. DOI:10.1128/AEM.03572-14
|
| [37] |
GUAN NZ, LIU L, SHIN HD, CHEN RR, ZHANG J, LI JH, DU GC, SHI ZP, CHEN J. Systems-level understanding of how Propionibacterium acidipropionici respond to propionic acid stress at the microenvironment levels: mechanism and application[J]. Journal of Biotechnology, 2013, 167(1): 56-63. DOI:10.1016/j.jbiotec.2013.06.008
|
| [38] |
GUAN NZ, SHIN HD, CHEN RR, LI JH, LIU L, DU GC, CHEN J. Understanding of how Propionibacterium acidipropionici respond to propionic acid stress at the level of proteomics[J]. Scientific Reports, 2014, 4: 6951. DOI:10.1038/srep06951
|
| [39] |
GUAN NZ, LI JH, SHIN HD, DU GC, CHEN J, LIU L. Metabolic engineering of acid resistance elements to improve acid resistance and propionic acid production of Propionibacterium jensenii[J]. Biotechnology and Bioengineering, 2016, 113(6): 1294-1304. DOI:10.1002/bit.25902
|
| [40] |
NAVONE L, MCCUBBIN T, GONZALEZ-GARCIA RA, NIELSEN LK, MARCELLIN E. Genome-scale model guided design of Propionibacterium for enhanced propionic acid production[J]. Metabolic Engineering Communications, 2018, 6: 1-12. DOI:10.1016/j.meteno.2017.11.001
|
| [41] |
GONZALEZ-GARCIA RA, McCUBBIN T, TURNER MS, NIELSEN LK, MARCELLIN E. Engineering Escherichia coli for propionic acid production through the Wood-Werkman cycle[J]. Biotechnology and Bioengineering, 2020, 117(1): 167-183. DOI:10.1002/bit.27182
|
| [42] |
AKAWI L, SRIRANGAN K, LIU XJ, MOO-YOUNG M, CHOU CP. Engineering Escherichia coli for high-level production of propionate[J]. Journal of Industrial Microbiology & Biotechnology, 2015, 42(7): 1057-1072.
|
| [43] |
MISCEVIC D, MAO JY, MOO-YOUNG M, CHOU CH P. High-level heterologous production of propionate in engineered Escherichia coli[J]. Biotechnology and Bioengineering, 2020, 117(5): 1304-1315. DOI:10.1002/bit.27276
|
| [44] |
DING WT, MENG QY, DONG GL, QI NL, ZHAO HM, SHI SB. Metabolic engineering of threonine catabolism enables Saccharomyces cerevisiae to produce propionate under aerobic conditions[J]. Biotechnology Journal, 2022, 17(3): e2100579. DOI:10.1002/biot.202100579
|
| [45] |
STINE A, ZHANG MM, RO S, CLENDENNEN S, SHELTON MC, TYO KEJ, BROADBELT LJ. Exploring de novo metabolic pathways from pyruvate to propionic acid[J]. Biotechnology Progress, 2016, 32(2): 303-311. DOI:10.1002/btpr.2233
|
| [46] |
KANDASAMY V, VAIDYANATHAN H, DJURDJEVIC I, JAYAMANI E, RAMACHANDRAN KB, BUCKEL W, JAYARAMAN G, RAMALINGAM S. Engineering Escherichia coli with acrylate pathway genes for propionic acid synthesis and its impact on mixed-acid fermentation[J]. Applied Microbiology and Biotechnology, 2013, 97(3): 1191-1200. DOI:10.1007/s00253-012-4274-y
|
| [47] |
HONG KK, NIELSEN J. Metabolic engineering of Saccharomyces cerevisiae: a key cell factory platform for future biorefineries[J]. Cellular and Molecular Life Sciences, 2012, 69(16): 2671-2690. DOI:10.1007/s00018-012-0945-1
|
| [48] |
ZHOU SF, LAMA SM, SANKARANARAYANAN M, PARK S. Metabolic engineering of Pseudomonas denitrificans for the 1, 3-propanediol production from glycerol[J]. Bioresource Technology, 2019, 292: 121933. DOI:10.1016/j.biortech.2019.121933
|
| [49] |
LIAN JZ, MISHRA S, ZHAO HM. Recent advances in metabolic engineering of Saccharomyces cerevisiae: new tools and their applications[J]. Metabolic Engineering, 2018, 50: 85-108. DOI:10.1016/j.ymben.2018.04.011
|
| [50] |
BLANC P, GOMA G. Kinetics of inhibition in propionic acid fermentation[J]. Bioprocess Engineering, 1987, 2(4): 175-179. DOI:10.1007/BF00387325
|
| [51] |
POBLETE-CASTRO I, BECKER J, DOHNT K, DOS SANTOS VM, WITTMANN C. Industrial biotechnology of Pseudomonas putida and related species[J]. Applied Microbiology and Biotechnology, 2012, 93(6): 2279-2290. DOI:10.1007/s00253-012-3928-0
|
| [52] |
CALERO P, NIKEL PI. Chasing bacterial chassis for metabolic engineering: a perspective review from classical to non-traditional microorganisms[J]. Microbial Biotechnology, 2019, 12(1): 98-124. DOI:10.1111/1751-7915.13292
|
| [53] |
KOOPMAN F, WIERCKX N, de WINDE JH, RUIJSSENAARS HJ. Efficient whole-cell biotransformation of 5-(hydroxymethyl) furfural into FDCA, 2, 5-furandicarboxylic acid[J]. Bioresource Technology, 2010, 101(16): 6291-6296. DOI:10.1016/j.biortech.2010.03.050
|
| [54] |
GUARNIERI MT, ANN FRANDEN M, JOHNSON CW, BECKHAM GT. Conversion and assimilation of furfural and 5-(hydroxymethyl) furfural by Pseudomonas putida KT2440[J]. Metabolic Engineering Communications, 2017, 4: 22-28. DOI:10.1016/j.meteno.2017.02.001
|
| [55] |
KAMPERS LFC, VOLKERS RJM, MARTINS DOS SANTOS VAP. Pseudomonas putida KT2440 is HV1 certified, not GRAS[J]. Microbial Biotechnology, 2019, 12(5): 845-848. DOI:10.1111/1751-7915.13443
|
| [56] |
MA C, MU QX, WANG L, SHI YN, ZHU LF, ZHANG SS, XUE YF, TAO Y, MA YH, YU B. Bio-production of high-purity propionate by engineering L-threonine degradation pathway in Pseudomonas putida[J]. Applied Microbiology and Biotechnology, 2020, 104(12): 5303-5313. DOI:10.1007/s00253-020-10619-7
|
| [57] |
MU QX, SHI YN, LI RS, MA C, TAO Y, YU B. Production of propionate by a sequential fermentation-biotransformation process via L-threonine[J]. Journal of Agricultural and Food Chemistry, 2021, 69(46): 13895-13903. DOI:10.1021/acs.jafc.1c05248
|
| [58] |
SHI YN, LI RS, ZHENG J, XUE YB, TAO Y, YU B. High-yield production of propionate from 1, 2-propanediol by engineered Pseudomonas putida KT2440, a robust strain with highly oxidative capacity[J]. Journal of Agricultural and Food Chemistry, 2022, 70(51): 16263-16272. DOI:10.1021/acs.jafc.2c06405
|
| [59] |
TIWARI R, SATHESH-PRABU C, LEE SK. Bioproduction of propionic acid using levulinic acid by engineered Pseudomonas putida[J]. Frontiers in Bioengineering and Biotechnology, 2022, 10: 939248. DOI:10.3389/fbioe.2022.939248
|
| [60] |
HESSLINGER C, FAIRHURST SA, SAWERS G. Novel keto acid formate-lyase and propionate kinase enzymes are components of an anaerobic pathway in Escherichia coli that degrades L-threonine to propionate[J]. Molecular Microbiology, 1998, 27(2): 477-492. DOI:10.1046/j.1365-2958.1998.00696.x
|
| [61] |
MA C, SHI YN, MU QX, LI RS, XUE YF, YU B. Unravelling the thioesterases responsible for propionate formation in engineered Pseudomonas putida KT2440[J]. Microbial Biotechnology, 2021, 14(3): 1237-1242. DOI:10.1111/1751-7915.13804
|
| [62] |
MA C, MU QX, XUE YB, XUE YF, YU B, MA YH. One major facilitator superfamily transporter is responsible for propionic acid tolerance in Pseudomonas putida KT2440[J]. Microbial Biotechnology, 2021, 14(2): 386-391. DOI:10.1111/1751-7915.13597
|
| [63] |
CUI QQ, ZHOU FL, LIU WF, TAO Y. Avermectin biosynthesis: stable functional expression of branched chain α-keto acid dehydrogenase complex from Streptomyces avermitilis in Escherichia coli by selectively regulating individual subunit gene expression[J]. Biotechnology Letters, 2017, 39(10): 1567-1574. DOI:10.1007/s10529-017-2389-z
|
| [64] |
RATHNASINGH C, RAJ SM, LEE YJ, CATHERINE C, ASHOK S, PARK S. Production of 3-hydroxypropionic acid via malonyL-CoA pathway using recombinant Escherichia coli strains[J]. Journal of Biotechnology, 2012, 157(4): 633-640. DOI:10.1016/j.jbiotec.2011.06.008
|
| [65] |
YANG YP, LIN YH, LI LY, LINHARDT RJ, YAN YJ. Regulating malonyL-CoA metabolism via synthetic antisense RNAs for enhanced biosynthesis of natural products[J]. Metabolic Engineering, 2015, 29: 217-226. DOI:10.1016/j.ymben.2015.03.018
|
| [66] |
WANG MM, ZHANG L, BOO KH, PARK E, DRAKAKAKI G, ZAKHAROV F. PDC1, a pyruvate/α-ketoacid decarboxylase, is involved in acetaldehyde, propanal and pentanal biosynthesis in melon (Cucumis melo L.) fruit[J]. The Plant Journal: for Cell and Molecular Biology, 2019, 98(1): 112-125. DOI:10.1111/tpj.14204
|
| [67] |
YU SQ, PLAN MR, WINTER G, KRÖMER JO. Metabolic engineering of Pseudomonas putida KT2440 for the production of para-hydroxy benzoic acid[J]. Frontiers in Bioengineering and Biotechnology, 2016, 4: 90.
|
| [68] |
BENTLEY GJ, NARAYANAN N, JHA RK, SALVACHÚA D, ELMORE JR, PEABODY GL, BLACK BA, RAMIREZ K, de CAPITE A, MICHENER WE, WERNER AZ, KLINGEMAN DM, SCHINDEL HS, NELSON R, FOUST L, GUSS AM, DALE T, JOHNSON CW, BECKHAM GT. Engineering glucose metabolism for enhanced muconic acid production in Pseudomonas putida KT2440[J]. Metabolic Engineering, 2020, 59: 64-75. DOI:10.1016/j.ymben.2020.01.001
|
| [69] |
NIU W, WILLETT H, MUELLER J, HE XY, KRAMER L, MA B, GUO JT. Direct biosynthesis of adipic acid from lignin-derived aromatics using engineered Pseudomonas putida KT2440[J]. Metabolic Engineering, 2020, 59: 151-161. DOI:10.1016/j.ymben.2020.02.006
|
| [70] |
MARINAS A, BRUIJNINCX P, FTOUNI J, URBANO FJ, PINEL C. Sustainability metrics for a fossiL- and renewable-based route for 1, 2-propanediol production: a comparison[J]. Catalysis Today, 2015, 239: 31-37. DOI:10.1016/j.cattod.2014.02.048
|
| [71] |
EBERT BE, KURTH F, GRUND M, BLANK LM, SCHMID A. Response of Pseudomonas putida KT2440 to increased NADH and ATP demand[J]. Applied and Environmental Microbiology, 2011, 77(18): 6597-6605. DOI:10.1128/AEM.05588-11
|
| [72] |
CAMERON B, BRIGGS K, PRIDMORE S, BREFORT G, CROUZET J. Cloning and analysis of genes involved in coenzyme B12 biosynthesis in Pseudomonas denitrificans[J]. Journal of Bacteriology, 1989, 171(1): 547-557. DOI:10.1128/jb.171.1.547-557.1989
|
| [73] |
XI YY, XU HT, ZHAN T, QIN Y, FAN FY, ZHANG XL. Metabolic engineering of the acid-tolerant yeast Pichia kudriavzevii for efficient L-malic acid production at low pH[J]. Metabolic Engineering, 2023, 75: 170-180. DOI:10.1016/j.ymben.2022.12.007
|
| [74] |
ZHANG B, LI RQ, YU LH, WU CC, LIU Z, BAI FY, YU B, WANG LM. L-lactic acid production via sustainable neutralizer-free route by engineering acid-tolerant yeast Pichia kudriavzevii[J]. Journal of Agricultural and Food Chemistry, 2023, 71(29): 11131-11140. DOI:10.1021/acs.jafc.3c03163
|
 2024, Vol. 40
2024, Vol. 40