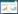中国科学院微生物研究所、中国微生物学会主办
文章信息
- 李佳然, 陈秀兰, 杨福全
- LI Jiaran, CHEN Xiulan, YANG Fuquan
- 磷酸化蛋白质组学研究中磷酸化肽段富集与分离方法研究进展
- Advances in the methods of phosphopeptide enrichment and separation in phosphoproteomic research
- 生物工程学报, 2022, 38(10): 3648-3658
- Chinese Journal of Biotechnology, 2022, 38(10): 3648-3658
- 10.13345/j.cjb.220599
-
文章历史
- Received: July 29, 2022
- Accepted: September 6, 2022
2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
磷酸化是细胞内最常见和最重要的蛋白质翻译后修饰之一。蛋白质的磷酸化和去磷酸化作为一种“分子开关”[1],通过影响蛋白质的结构、定位和功能,在细胞的信号转导、转录翻译、代谢和发育等生命过程中发挥着重要作用[2]。在真核细胞中,大约1/3的蛋白质会被磷酸化[3],磷酸化主要发生在丝氨酸(Ser, S)、苏氨酸(Thr, T) 和酪氨酸(Tyr, Y) 的侧链羟基上,称为O-磷酸化,这也是磷酸化研究的常见类型。尽管磷酸化蛋白的数目相对较多,但是磷酸化蛋白在细胞内是低丰度和亚化学计量的[4],大量非磷酸化蛋白的存在会对磷酸化蛋白的鉴定造成一定的挑战。为了提高磷酸化蛋白组的覆盖范围,发展高重复性和特异性的富集方法显得尤为重要。近年来不断有各种磷酸化富集方法的报道,使得磷酸化蛋白质和磷酸化位点的鉴定数目有了大幅度的提高。本文将综述亲和色谱法、免疫沉淀法、化学衍生法、基于色谱的方法和其他新发展方法等磷酸化肽段富集方法(图 1A),介绍不同磷酸化肽段富集方法的原理、优缺点、选择性(指富集材料对不同肽段的富集选择性,如对磷酸化肽段和非磷酸化肽段的选择性,对单磷酸化肽段和多磷酸化肽段的选择性,对丝氨酸、苏氨酸和酪氨酸磷酸化肽段的选择性) 及一些优化与改进策略,随后介绍磷酸化肽段富集和预分方法的不同组合策略。
|
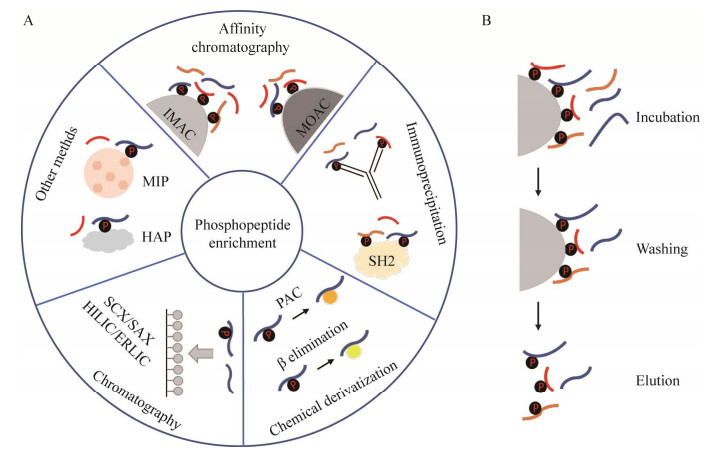
| 图 1 磷酸化肽段富集策略 Fig. 1 Strategies for enrichment of phosphopeptides. A:磷酸化肽段富集方法;B:基于亲和色谱的磷酸化肽段富集方法的主要步骤 (A) Phosphopeptide enrichment methods. (B) Main steps for affinity-based phosphopeptide enrichment methods. |
| |
近年来,基于亲和色谱的固定化金属离子亲和色谱(immobilized metal ion affinity chromatography, IMAC) 和金属氧化物亲和色谱(metal oxide affinity chromatography, MOAC) 的方法快速发展。亲和色谱富集磷酸化肽段的过程主要涉及3个步骤(图 1B):(1) 孵育:磷酸化蛋白质/肽段上带负电的磷酸基团与金属离子或者金属氧化物结合;(2) 清洗:除去结合的非磷酸化肽段;(3) 洗脱:释放与金属材料结合的磷酸化肽段。
IMAC利用固定化的带正电荷的金属阳离子与带负电荷的磷酸基团之间的亲和力富集磷酸化肽段,这些金属离子通过螯合配体以非共价相互作用固定在支持基质(如磁珠) 上。传统的螯合配体,如次氮基三乙酸(nitrilotriacetic acid, NTA) 或亚氨基二乙酸(iminodiacetic acid, IDA),与金属离子之间的特异性不高,因为每种金属离子只能与1个NTA或IDA配体配位,这可能会导致富集时磷酸化肽段丢失。Zhou等[5-6]报道了一种基于磷酸盐的新型高效螯合配体,与NTA或IDA不同,每个金属离子可以与多个磷酸盐基团结合,从而显著增加特异性富集磷酸化肽段的能力。固定化的金属离子也会影响IMAC的选择性,Jiang等[7]系统比较了IMAC中常用的8种不同金属离子富集磷酸化肽段的能力,发现高价金属离子Nb5+、Ti4+和Zr4+对磷酸化肽段表现出更好的选择性,因为它们与磷酸盐和羧酸基团的配位能力更强。其中,Ti4+-IMAC因其优越的性能已成为一种流行的磷酸化肽段富集方法,Yao等[8]将单分散Ti4+-IMAC微球用于HeLa细胞磷酸化肽段富集,最终鉴定出超过4 700种磷酸化肽段,特异性高达99%。然而,IMAC的性能容易受到生物样品中的许多分子,包括盐、去垢剂和其他小分子化合物的影响[9]。除了固体基质载体,Iliuk等[10]还开发了一种名为PolyMAC (polymer-based metal ion affinity capture) 的金属离子(Ti4+和Fe3+) 可溶性纳米聚合物,它由金属离子和醛两种侧基组成,可用于富集磷酸化肽段。PolyMAC-磷酸化肽段复合物可以通过共价偶联固体支持物,如琼脂糖凝胶,从溶液中分离出来。与固相IMAC相比,PolyMAC可以在均相水溶液中富集磷酸化肽段,从而克服了固相结合的异质性,具有螯合时间快、可重复性高的特点。
MOAC利用金属氧化物与磷酸基团之间的亲和力富集磷酸化肽段,这些金属氧化物大多数是两性的,在酸性条件下结合磷酸化肽段,碱性条件下释放磷酸化肽段。MOAC基质本身由金属氧化物或氢氧化物组成[11],不需要螯合基团固定金属离子。二氧化钛(TiO2) 是首个用于磷酸化肽段富集的金属氧化物[12],近年来也发现许多其他的氧化物在一定程度上对磷酸化肽段表现出特异性的亲和能力,如ZrO2[13]、SiO2[14]、Al(OH)3[15]。Leitner等[16]用不同复杂性的样品,包括合成肽段混合物到全细胞裂解物,评估了包含常用的TiO2、ZrO2等在内的7种金属氧化物的磷酸化肽段富集效率,发现亲和材料的性能主要与氧化物的等电点(isoelectric point, IEP) 相关,富集磷酸化肽段数量最多的材料(TiO2、ZrO2和In2O3) 的IEP约为6。与IMAC相比,MOAC中的金属阳离子与相邻的氧阴离子可以形成稳定的化学键,因此,MOAC对盐、去垢剂和不同溶剂有更强的耐受性,且具有更好的选择性和灵敏度[17]。
虽然IMAC和MOAC方法富集磷酸化肽段的特异性较高,但是IMAC和MOAC对不同类型的磷酸化肽段的富集具有不同的选择性,IMAC主要富集多磷酸化肽段,而MOAC主要富集单磷酸化肽段[18]。为了富集到更多的单磷酸化肽段和多磷酸化肽段,Thingholm等[19]首次将IMAC和MOAC结合,发展了IMAC顺序洗脱方法(sequential elution from IMAC, SIMAC),通过SIMAC方法富集后鉴定到的磷酸化位点数是单独使用TiO2法富集鉴定到的2倍。
除IMAC和MOAC外,Phos-tag (1, 3-bis [bis(pyridine-2-ylmethyl)-amino]propan-2-olato dizinc(Ⅱ)) 也是一种基于亲和相互作用的富集方法。Phos-tag是一种琼脂糖偶联双核锌复合物[20],可以在中性条件下富集磷酸化肽段。Phos-tag与磷酸基团的相互作用类似于IMAC,都是基于磷酸基团与金属离子之间的亲和力。Koike团队[21-22]将Phos-tag用于SDS-PAGE,成功分离到大分子量的磷酸化蛋白。
1.2 免疫沉淀法亲和色谱法对丝氨酸和苏氨酸磷酸化肽段的富集是有效的,但是酪氨酸磷酸化通常以较低的丰度发生,大约只占整体磷酸化事件的0.5%[23],常规富集方法对磷酸化酪氨酸无特异性,需要开发基于抗原抗体亲和相互作用的免疫沉淀富集磷酸化肽段的方法。目前市售的抗体对酪氨酸磷酸化的特异性比较高,对丝氨酸/苏氨酸磷酸化肽段的富集存在结合效率以及特异性的问题,因为与磷酸化丝氨酸和苏氨酸的脂肪侧链相比,磷酸化酪氨酸的芳香环更富电子,因此磷酸化酪氨酸有更多的机会与抗体结合位点结合,从而产生更强的特异性结合。此外,由于大多数抗体是磷酸氨基酸特异性的[11],一种抗体只能结合一种磷酸化的氨基酸,无法用于大规模的磷酸化蛋白组的研究。一些研究使用了丝氨酸和苏氨酸共有基序产生的抗体[24-25],但是由于这些抗体不能以相同的效率结合丝氨酸/苏氨酸位点,因此这种方法的产量比较低。此外,高昂的抗体成本进一步限制了其更广泛的使用。
为了解决酪氨酸磷酸化的亲和特异性问题,SH2 (Src homology) 结构域,一种存在于许多信号蛋白中的约100个氨基酸的保守结构域[26],被用来富集酪氨酸磷酸化肽段。SH2结构域可识别由酪氨酸磷酸化位点后加3−5个C端残基组成的短磷酸化肽段序列[27],但是天然的SH2结构域对酪氨酸磷酸化的亲和力非常有限[28]。Kaneko等[29]报道了具有三点突变体(T8V; S10A; K15L) 的高亲和力sSH2 (superbinder SH2) 结构域。与天然SH2相比,sSH2对磷酸化肽段的亲和力明显增强。Bian等[30]发展了使用sSH2结构域作为亲和试剂,通过亲和纯化质谱鉴定酪氨酸磷酸化蛋白质组的方法,并在9种人类细胞系中鉴定到约20 000条酪氨酸磷酸化肽段和大于10 000个酪氨酸磷酸化位点。这个方法的缺点在于使用sSH2亲和试剂进行富集时,需要用竞争性洗脱剂生物素-pYEEI (pTyr-Glu-Glu-Ile) 洗脱磷酸化肽段[30],而-pYEEI需要在LC-MS/MS分析之前被去除以免影响质谱的鉴定,额外的纯化步骤会导致样品的损失[31]。本文没有再更多地介绍基于SH2超亲体的酪氨酸磷酸化蛋白质组技术的应用,门丽影等[32]已经做了很好的综述。总之,这个酪氨酸磷酸化肽段的富集方法还需要进一步的优化。
1.3 化学衍生法化学衍生法富集磷酸化肽段的基本思想是用另外可以被特异性靶向的化学基团取代磷酸基团,然后基于引入的亲和基团提取衍生后的磷酸化肽段。比较典型的化学衍生法是β消去法和氨基磷酸酯化学法(phosphoramidate chemistry, PAC)。
β消去法通常与Michael加成反应相结合。在碱性条件下,磷酸化丝氨酸和苏氨酸去磷酸化并分别转化为脱氢丙氨酸和β-甲基脱氢丙氨酸,通过加入亲核试剂如硫醇试剂,肽段中产生的二硫醇可以作为生物素标签的交联剂[33]或用于直接亲和纯化[34]。由于酪氨酸的磷酸基团无法在碱性条件下发生消去反应,因此这种方法只能用来富集丝氨酸和苏氨酸磷酸化肽段。此外,β消去反应的发生可能会增加未磷酸化的丝氨酸和苏氨酸残基脱水的风险[34],导致副反应的产生而增加样品的复杂性。Nika等[35]在一组合成的磷酸化肽段中研究了β消去法的各种反应条件及实验参数,增加了磷酸化位点的鉴定数目,但这仅限于简单的蛋白样品。
PAC是另一典型的化学衍生法,在某些条件下,如水溶性碳二亚胺催化,磷酸化氨基酸的磷酸基团与酰胺基团反应生成氨基磷酸酯,经还原后可偶联到碘乙酰修饰的玻璃微珠上[36-37],被富集的磷酸基团在酸性条件下可被释放。与β消去法不同的是该方法也可富集含酪氨酸磷酸化肽段,且富集过程中磷酸基团不会被去除,便于检测确切的磷酸化位点。Zhou等[36]的工作证明了基于PAC法富集研究酵母磷酸化的可行性。Tao等[37]将磷酸化肽段与胺功能化的树枝状聚合物偶联,从而可以通过超滤富集磷酸化肽段。但是,该反应过程涉及一系列化学反应,因此一些副反应无法避免,增加了样品损失。
总之,由于上述缺点,目前化学衍生方法较少地被用于磷酸化肽段的富集。
1.4 基于色谱的方法基于色谱的方法也被用于富集磷酸化肽段,如强阳/阴离子交换色谱[38-39] (strong cation/anion exchange chromatography, SCX/SAX)、亲水相互作用色谱[40] (hydrophilic interaction liquid chromatography, HILIC)、静电排斥液相作用色谱[41] (electrostatic repulsion liquid interaction chromatography, ERLIC)。SCX/SAX富集磷酸化肽段是基于带有不同电荷的酶切肽段与阴/阳离子固定相之间的相互作用。在使用SCX富集磷酸化肽段时,反应在强酸性条件下进行,胰蛋白酶切割产生的赖氨酸和精氨酸残基完全质子化,因此大多数非磷酸化肽段带有2个正电荷,而磷酸基团为去质子化的状态,只有1个磷酸化位点的肽段会带有1个正电荷,具有2个磷酸化位点的肽段不带电荷,所以多磷酸化肽段首先被洗脱,然后是与固定相作用较弱的单磷酸化肽段,最后是非磷酸化肽段。而使用SAX富集磷酸化肽段时,反应在中性或碱性条件下进行,洗脱顺序与SCX相反。HILIC基于亲水性分离肽段,极性较强的肽段后被洗脱出来,因此磷酸化肽段在梯度末端被富集,但是研究表明HILIC对多磷酸化肽段的分辨率比单磷酸化肽段要低,因为多磷酸化肽段在HILIC高浓度的有机溶剂情况下溶解性较差[42-43]。ERLIC基于亲水相互作用和静电相互作用富集磷酸化肽段,是一种混合模式的色谱。Alpert[41]发现将ERLIC与SAX进行比较,ERLIC更适合从复杂混合物中鉴定胰蛋白酶磷酸化肽段。但是,这些基于色谱的方法富集磷酸化肽段特异性不高,现在更多地作为肽段的预分手段,将复杂的样品初步地分离得到若干个性质较为相似的组分,以提高后续磷酸化肽段的富集效率和磷酸化肽段的鉴定数目(后续第3节进行了详细介绍)。
1.5 其他方法除了上述方法之外,分子印迹聚合物(molecularly imprinted polymers, MIP) 和羟磷灰石层析(hydroxyapatite chromatography, HAP) 的富集方法不断发展。MIP是一种由分子印迹技术产生的仿生“合成抗体”或“塑料抗体”,具有类似天然酶和抗体的亲和力和特异性,能够识别并结合特定的目标分子。Emgenbroich等[44]报道了第一个合成的磷酸化酪氨酸选择性印迹聚合物,能够在大量非磷酸化肽段存在的情况下捕获酪氨酸磷酸化肽段。Helling等[45]对该系统进一步优化和验证,成功提取了牛胎素消化液中的酪氨酸磷酸化肽段。HAP是结晶磷酸钙(Ca10(PO4)6(OH)2) 的一种形式,常被用于分离各种蛋白质和核酸。Krenkova等[46]和Pinto等[47]发现磷酸化蛋白比未磷酸化蛋白更容易与HAP结合,因此提出了将HAP用于磷酸化肽段富集。Mamone等[48]用基于HAP的富集策略,成功从标准肽段混合物中富集出磷酸化肽段,并将单磷酸化肽段和多磷酸化肽段分离。与TiO2相比,HAP对多磷酸化肽段具有更高的亲和力。
鉴于磷酸化蛋白质组的复杂性以及不同富集方法对不同类型的磷酸化肽段的偏向性,目前没有一种方法可以富集到所有的磷酸化肽段,因此组合使用多种磷酸化肽段富集方法可以提高磷酸化肽段的鉴定效率和磷酸化蛋白质组的覆盖率。
2 富集方法的优化与改进在磷酸化肽段富集过程中,富集特异性会受到多种因素的影响。下述以常用的IMAC和MOAC富集方法为例阐述磷酸化肽段富集方法的优化与改进。
2.1 基于IMAC的优化与改进由于磷酸化肽段与IMAC之间的相互作用是路易斯酸碱反应[49],因此pH在富集过程中是重要的考虑因素。磷酸化肽段富集中存在非特异性结合的原因是含有酸性氨基酸(谷氨酸和天冬氨酸) 的非磷酸化肽段与金属离子材料结合,主要是氨基酸γ-基团上的羧酸基团与金属离子材料之间的亲和力引起的,类似于磷酸盐对金属离子的亲和力[11]。为了减少含酸性氨基酸的非磷酸化肽段与IMAC非特异性结合,富集时需要使用有机酸,如乙酸[50]或三氟乙酸(trifluoroacetic acid, TFA)[51],调节上样缓冲液pH值位于酸性氨基酸和磷酸盐的pKa值之间,即pH 2.0–2.5,此时酸性氨基酸被质子化,羧基的负电荷被掩盖不能再与带正电的金属离子结合[11],而大多数磷酸盐处于非质子化的状态,仍能与金属离子材料结合。除pH外,Ye等[52]发现较高浓度的乙腈(acetonitrile, ACN) 有利于磷酸盐去质子化和酸性氨基酸质子化,进一步减少了非特异性结合。因此,利用IMAC富集磷酸化肽段时,上样缓冲液中含有高浓度的ACN和TFA,如50% ACN和0.1% TFA。
2.2 基于MOAC的优化与改进与IMAC类似,MOAC的上样缓冲液需要使用有机酸进行酸化,使酸性氨基酸质子化和磷酸基团去质子化,然而缓冲液酸化不足以降低非磷酸化肽段的非特异性结合。研究发现在上样缓冲液中加入一些含羧酸或羟基官能团的,能够与非磷酸化肽段竞争TiO2的结合位点的试剂(non-phosphopeptide excluder),如谷氨酸[53]、乳酸[54]、乙醇酸[9]、柠檬酸[55]和2, 5-二羟基苯甲酸(2, 5-dihydroxybenzoic, DHB)[56],它们可以有效地减少非磷酸化肽段与TiO2的结合,同时不会对磷酸化肽段的结合造成影响。Larsen等[56]报道TiO2上最优的磷酸盐结合位点的配位几何结构不同于取代羧酸的最佳结合位点的配位几何结构,羧酸以螯合双齿方式与TiO2结合,而磷酸盐以桥联双齿的方式与TiO2结合。我们最近的工作[57]系统比较了上样缓冲液中谷氨酸、乳酸、乙醇酸和DHB这4种常用的non-phosphopeptide excluders对TiO2方法富集磷酸化肽段的影响,发现不同方法的富集特异性不同,含有乳酸的磷酸化肽段富集方法表现出最好的特异性及选择性,能鉴定到较多数目的磷酸化肽段。此外,TiO2珠子与肽段的比值也会影响富集特异性[58],因此富集方法应该根据不同样品进行调整与优化,以得到最佳的富集效率,富集得到更多的磷酸化肽段。
3 磷酸化肽段富集/预分方法研究进展为了提高磷酸化蛋白组的覆盖深度,往往需要将磷酸化肽段富集和预分相结合。较为传统的策略是先预分再富集(图 2A),即在肽段水平用SCX/SAX、HILIC或高pH反相色谱(high pH reverse-phase liquid chromatography, HpH-RP) 等色谱方法将酶切肽段预分成多个组分[59],然后对单个组分或合并组分分别进行磷酸化肽段富集。这种策略可以显著降低样品复杂程度,有效提高鉴定率。但是这种策略耗时耗力,而且需要较高的样品起始量。此外,由于预分后各个组分肽段含量不一样,很难准确地添加特定富集磷酸化肽段所需的材料。

|
| 图 2 磷酸化肽段先预分后富集(A) 和先富集后预分(B) 流程 Fig. 2 Workflow for the strategy of "fractionation before enrichment" (A) and "fractionation after enrichment" (B). |
| |
最近几年,另外一种先富集后预分的策略慢慢发展成熟(图 2B)。这种策略先利用TiO2或者IMAC方法从复杂样品中富集磷酸化肽段,再利用HILIC或者HpH-RP等色谱方法将富集到的磷酸化肽段预分成多个组分。Yue等[60]和Engholm-Keller等[43]用这种方式,采用HILIC方法预分磷酸化肽段,大大提高了磷酸化肽段的鉴定数目,而且大大减少了样品处理时间和样品损失。这种先富集后预分的策略对样品起始量要求低,而且过程更简单,但是有可能因为样品复杂程度的问题导致富集的特异性降低。我们最近的工作[57]综合评价了不同的磷酸化肽段富集和分离方法,采取先富集磷酸化肽段再预分的策略,即以乳酸作为non-phosphopeptide excluder进行TiO2富集磷酸化肽段后,用三乙胺(triethylamine, TEA) 或氨水(NH3·H2O)作为HpH-RP预分的分离试剂,使用StageTip进行HpH-RP预分,结果显示基于TEA的HpH-RP预分策略鉴定到的总磷酸化肽段数目多于基于氨水的HpH-RP预分策略,且基于TEA的方法在鉴定多磷酸化肽段(包括双磷酸化肽段和三磷酸化肽段) 时具有明显的优势。最终采用基于TEA的HpH-RP预分方法,在1 mg 293T细胞的肽段中鉴定到超过30 000条磷酸化肽段和约20 000个磷酸化位点。该研究为选择合适的磷酸化肽段富集方法提供了依据,并提供了一个高效可重复的大规模磷酸化蛋白质组学分析工作流程。
除了上述先预分后富集和先富集后预分的策略,Ren等[61]进行了富集和预分一体化的尝试,报道了一种TiO2串联分离(TiO2 with tandem fractionation, TAFT) 的方法,将用于TiO2富集的StageTip和HpH-RP的StageTip串联,能够在3 h内完成磷酸化蛋白组样品的制备。他们利用该方法富集HeLa细胞和HepG2.2.15细胞中的磷酸化肽段,分别在1 mg起始量蛋白中鉴定到超过14 000条磷酸化肽段。该方法具有较高的特异性和重现性,能够以低成本的高通量方式进行快速深入的磷酸化蛋白组分析。
4 总结总之,随着近年来磷酸化肽段富集技术的不断发展和质谱仪器分辨率灵敏度的不断提高,极大地消除了磷酸化蛋白组研究的瓶颈,但是截至目前,没有一种磷酸化富集方法能够完全描绘磷酸化蛋白组[18]。因此,尚不清楚现有富集方法获得的磷酸化肽段是否反映了生物体的真实情况。为了得到更为可靠的分析结果,最好能够得到覆盖深度更高的磷酸化肽段。此外,这些磷酸化肽段富集技术具有不同的特异性,为了克服因方法选择偏差造成的结果偏倚,最好根据具体的研究问题选择最适的富集策略,或者结合不同的富集方法,以提高磷酸化蛋白质组的覆盖范围。
| [1] |
Olsen JV, Blagoev B, Gnad F, et al. Global, in vivo, and site-specific phosphorylation dynamics in signaling networks. Cell, 2006, 127(3): 635-648. DOI:10.1016/j.cell.2006.09.026
|
| [2] |
Hunter T. Signaling—2000 and beyond. Cell, 2000, 100(1): 113-127. DOI:10.1016/S0092-8674(00)81688-8
|
| [3] |
Mann M, Ong SE, Grønborg M, et al. Analysis of protein phosphorylation using mass spectrometry: deciphering the phosphoproteome. Trends Biotechnol, 2002, 20(6): 261-268. DOI:10.1016/S0167-7799(02)01944-3
|
| [4] |
Aebersold R, Goodlett DR. Mass spectrometry in proteomics. Chem Rev, 2001, 101(2): 269-295. DOI:10.1021/cr990076h
|
| [5] |
Zhou HJ, Xu SY, Ye ML, et al. Zirconium phosphonate-modified porous silicon for highly specific capture of phosphopeptides and MALDI-TOF MS analysis. J Proteome Res, 2006, 5(9): 2431-2437. DOI:10.1021/pr060162f
|
| [6] |
Zhou H, Ye M, Dong J, et al. Robust phosphoproteome enrichment using monodisperse microsphere–based immobilized titanium (Ⅳ) ion affinity chromatography. Nat Protoc, 2013, 8(3): 461-480. DOI:10.1038/nprot.2013.010
|
| [7] |
Jiang JB, Sun XN, Li Y, et al. Facile synthesis of Fe3O4@PDA core-shell microspheres functionalized with various metal ions: a systematic comparison of commonly-used metal ions for IMAC enrichment. Talanta, 2018, 178: 600-607. DOI:10.1016/j.talanta.2017.09.071
|
| [8] |
Yao YT, Dong J, Dong MM, et al. An immobilized titanium (Ⅳ) ion affinity chromatography adsorbent for solid phase extraction of phosphopeptides for phosphoproteome analysis. J Chromatogr A, 2017, 1498: 22-28.
|
| [9] |
Jensen SS, Larsen MR. Evaluation of the impact of some experimental procedures on different phosphopeptide enrichment techniques. Rapid Commun Mass Spectrom, 2007, 21(22): 3635-3645. DOI:10.1002/rcm.3254
|
| [10] |
Iliuk AB, Martin VA, Alicie BM, et al. In-depth analyses of kinase-dependent tyrosine phosphoproteomes based on metal ion-functionalized soluble nanopolymers. Mol Cell Proteomics, 2010, 9(10): 2162-2172. DOI:10.1074/mcp.M110.000091
|
| [11] |
Fíla J, Honys D. Enrichment techniques employed in phosphoproteomics. Amino Acids, 2012, 43(3): 1025-1047. DOI:10.1007/s00726-011-1111-z
|
| [12] |
Pinkse MWH, Uitto PM, Hilhorst MJ, et al. Selective isolation at the femtomole level of phosphopeptides from proteolytic digests using 2D-NanoLC-ESI-MS/MS and titanium oxide precolumns. Anal Chem, 2004, 76(14): 3935-3943. DOI:10.1021/ac0498617
|
| [13] |
Wang XM, Guo ZY, Zhang Y, et al. ZrO2 doped magnetic mesoporous polyimide for the efficient enrichment of phosphopeptides. Talanta, 2018, 188: 385-392. DOI:10.1016/j.talanta.2018.05.069
|
| [14] |
Xiao RL, Pan YN, Li J, et al. Layer-by-layer assembled magnetic bimetallic metal-organic framework composite for global phosphopeptide enrichment. J Chromatogr A, 2019, 1601: 45-52.
|
| [15] |
Wolschin F, Wienkoop S, Weckwerth W. Enrichment of phosphorylated proteins and peptides from complex mixtures using metal oxide/hydroxide affinity chromatography (MOAC). Proteomics, 2005, 5(17): 4389-4397. DOI:10.1002/pmic.200402049
|
| [16] |
Leitner A, Sakeye M, Zimmerli CE, et al. Insights into chemoselectivity principles in metal oxide affinity chromatography using tailored nanocast metal oxide microspheres and mass spectrometry-based phosphoproteomics. Analyst, 2017, 142(11): 1993-2003. DOI:10.1039/C7AN00570A
|
| [17] |
Aryal UK, Ross ARS. Enrichment and analysis of phosphopeptides under different experimental conditions using titanium dioxide affinity chromatography and mass spectrometry. Rapid Commun Mass Spectrom, 2010, 24(2): 219-231. DOI:10.1002/rcm.4377
|
| [18] |
Bodenmiller B, Mueller LN, Mueller M, et al. Reproducible isolation of distinct, overlapping segments of the phosphoproteome. Nat Methods, 2007, 4(3): 231-237. DOI:10.1038/nmeth1005
|
| [19] |
Thingholm TE, Jensen ON, Robinson PJ, et al. SIMAC (sequential elution from IMAC), a phosphoproteomics strategy for the rapid separation of monophosphorylated from multiply phosphorylated peptides. Mol Cell Proteomics, 2008, 7(4): 661-671. DOI:10.1074/mcp.M700362-MCP200
|
| [20] |
Kinoshita E, Yamada A, Takeda H, et al. Novel immobilized zinc(Ⅱ) affinity chromatography for phosphopeptides and phosphorylated proteins. J Sep Sci, 2005, 28(2): 155-162. DOI:10.1002/jssc.200401833
|
| [21] |
Kinoshita-Kikuta E, Aoki Y, Kinoshita E, et al. Label-free kinase profiling using phosphate affinity polyacrylamide gel electrophoresis. Mol Cell Proteomics, 2007, 6(2): 356-366. DOI:10.1074/mcp.T600044-MCP200
|
| [22] |
Kinoshita E, Kinoshita-Kikuta E, Koike T. Separation and detection of large phosphoproteins using Phos-tag SDS-PAGE. Nat Protoc, 2009, 4(10): 1513-1521. DOI:10.1038/nprot.2009.154
|
| [23] |
Hunter T, Sefton BM. Transforming gene product of Rous sarcoma virus phosphorylates tyrosine. PNAS, 1980, 77(3): 1311-1315. DOI:10.1073/pnas.77.3.1311
|
| [24] |
Matsuoka S, Ballif BA, Smogorzewska A, et al. ATM and ATR substrate analysis reveals extensive protein networks responsive to DNA damage. Science, 2007, 316(5828): 1160-1166. DOI:10.1126/science.1140321
|
| [25] |
Grønborg M, Kristiansen TZ, Stensballe A, et al. A mass spectrometry-based proteomic approach for identification of serine/threonine-phosphorylated proteins by enrichment with phospho-specific antibodies: identification of a novel protein, frigg, as a protein kinase A substrate. Mol Cell Proteomics, 2002, 1(7): 517-527. DOI:10.1074/mcp.M200010-MCP200
|
| [26] |
Waksman G. Binding of a high affinity phosphotyrosyl peptide to the Src SH2 domain: crystal structures of the complexed and peptide-free forms. Cell, 1993, 72(5): 779-790. DOI:10.1016/0092-8674(93)90405-F
|
| [27] |
Low TY, Mohtar MA, Lee PY, et al. Widening the bottleneck of phosphoproteomics: evolving strategies for phosphopeptide enrichment. Mass Spectrom Rev, 2021, 40(4): 309-333. DOI:10.1002/mas.21636
|
| [28] |
Ladbury JE, Arold ST. Energetics of Src homology domain interactions in receptor tyrosine kinase-mediated signaling. Methods Enzymol, 2011, 488: 147-183.
|
| [29] |
Kaneko T, Huang HM, Cao X, et al. Superbinder SH2 domains act as antagonists of cell signaling. Sci Signal, 2012, 5(243): ra68.
|
| [30] |
Bian YY, Li L, Dong MM, et al. Ultra-deep tyrosine phosphoproteomics enabled by a phosphotyrosine superbinder. Nat Chem Biol, 2016, 12(11): 959-966. DOI:10.1038/nchembio.2178
|
| [31] |
Dong MM, Bian YY, Wang Y, et al. Sensitive, robust, and cost-effective approach for tyrosine phosphoproteome analysis. Anal Chem, 2017, 89(17): 9307-9314. DOI:10.1021/acs.analchem.7b02078
|
| [32] |
门丽影, 徐锋, 徐平. 基于SH2超亲体的微量样品酪氨酸磷酸化蛋白质组学技术研究进展及应用. 生物工程学报, 2021, 37(7): 2334-2341. Men LY, Xu F, Xu P. Advances and application of enrichment technology in SH2 superbinder-based tyrosine phosphoproteomics. Chin J Biotech, 2021, 37(7): 2334-2341 (in Chinese). |
| [33] |
Oda Y, Nagasu T, Chait BT. Enrichment analysis of phosphorylated proteins as a tool for probing the phosphoproteome. Nat Biotechnol, 2001, 19(4): 379-382. DOI:10.1038/86783
|
| [34] |
McLachlin DT, Chait BT. Improved beta-elimination- based affinity purification strategy for enrichment of phosphopeptides. Anal Chem, 2003, 75(24): 6826-6836. DOI:10.1021/ac034989u
|
| [35] |
Nika H, Lee J, Willis IM, et al. Phosphopeptide characterization by mass spectrometry using reversed-phase supports for solid-phase β-elimination/Michael addition. J Biomol Tech, 2012, 23(2): 51-68. DOI:10.7171/jbt.2012-2302-002
|
| [36] |
Zhou H, Watts JD, Aebersold R. A systematic approach to the analysis of protein phosphorylation. Nat Biotechnol, 2001, 19(4): 375-378. DOI:10.1038/86777
|
| [37] |
Tao WA, Wollscheid B, O'Brien R, et al. Quantitative phosphoproteome analysis using a dendrimer conjugation chemistry and tandem mass spectrometry. Nat Methods, 2005, 2(8): 591-598. DOI:10.1038/nmeth776
|
| [38] |
Beausoleil SA, Jedrychowski M, Schwartz D, et al. Large-scale characterization of HeLa cell nuclear phosphoproteins. PNAS, 2004, 101(33): 12130-12135. DOI:10.1073/pnas.0404720101
|
| [39] |
Zhang KL. From purification of large amounts of phospho-compounds (nucleotides) to enrichment of phospho-peptides using anion-exchanging resin. Anal Biochem, 2006, 357(2): 225-231. DOI:10.1016/j.ab.2006.07.001
|
| [40] |
McNulty DE, Annan RS. Hydrophilic interaction chromatography reduces the complexity of the phosphoproteome and improves global phosphopeptide isolation and detection. Mol Cell Proteomics, 2008, 7(5): 971-980. DOI:10.1074/mcp.M700543-MCP200
|
| [41] |
Alpert AJ. Electrostatic repulsion hydrophilic interaction chromatography for isocratic separation of charged solutes and selective isolation of phosphopeptides. Anal Chem, 2008, 80(1): 62-76. DOI:10.1021/ac070997p
|
| [42] |
Engholm-Keller K, Hansen TA, Palmisano G, et al. Multidimensional strategy for sensitive phosphoproteomics incorporating protein prefractionation combined with SIMAC, HILIC, and TiO(2) chromatography applied to proximal EGF signaling. J Proteome Res, 2011, 10(12): 5383-5397. DOI:10.1021/pr200641x
|
| [43] |
Engholm-Keller K, Birck P, Størling J, et al. TiSH—a robust and sensitive global phosphoproteomics strategy employing a combination of TiO2, SIMAC, and HILIC. J Proteom, 2012, 75(18): 5749-5761. DOI:10.1016/j.jprot.2012.08.007
|
| [44] |
Emgenbroich M, Borrelli C, Shinde S, et al. A phosphotyrosine-imprinted polymer receptor for the recognition of tyrosine phosphorylated peptides. Chemistry, 2008, 14(31): 9516-9529. DOI:10.1002/chem.200801046
|
| [45] |
Helling S, Shinde S, Brosseron F, et al. Ultratrace enrichment of tyrosine phosphorylated peptides on an imprinted polymer. Anal Chem, 2011, 83(5): 1862-1865. DOI:10.1021/ac103086v
|
| [46] |
Krenkova J, Lacher NA, Svec F. Control of selectivity via nanochemistry: monolithic capillary column containing hydroxyapatite nanoparticles for separation of proteins and enrichment of phosphopeptides. Anal Chem, 2010, 82(19): 8335-8341. DOI:10.1021/ac1018815
|
| [47] |
Pinto G, Caira S, Cuollo M, et al. Hydroxyapatite as a concentrating probe for phosphoproteomic analyses. J Chromatogr B, 2010, 878(28): 2669-2678. DOI:10.1016/j.jchromb.2010.07.024
|
| [48] |
Mamone G, Picariello G, Ferranti P, et al. Hydroxyapatite affinity chromatography for the highly selective enrichment of mono- and multi-phosphorylated peptides in phosphoproteome analysis. Proteomics, 2010, 10(3): 380-393. DOI:10.1002/pmic.200800710
|
| [49] |
Qiu W, Evans CA, Landels A, et al. Phosphopeptide enrichment for phosphoproteomic analysis-a tutorial and review of novel materials. Anal Chim Acta, 2020, 1129: 158-180.
|
| [50] |
Posewitz MC, Tempst P. Immobilized gallium(Ⅲ) affinity chromatography of phosphopeptides. Anal Chem, 1999, 71(14): 2883-2892. DOI:10.1021/ac981409y
|
| [51] |
Kokubu M, Ishihama Y, Sato T, et al. Specificity of immobilized metal affinity-based IMAC/C18 tip enrichment of phosphopeptides for protein phosphorylation analysis. Anal Chem, 2005, 77(16): 5144-5154. DOI:10.1021/ac050404f
|
| [52] |
Ye JY, Zhang XM, Young C, et al. Optimized IMAC-IMAC protocol for phosphopeptide recovery from complex biological samples. J Proteome Res, 2010, 9(7): 3561-3573. DOI:10.1021/pr100075x
|
| [53] |
Wu J, Shakey Q, Liu W, et al. Global profiling of phosphopeptides by titania affinity enrichment. J Proteome Res, 2007, 6(12): 4684-4689. DOI:10.1021/pr070481m
|
| [54] |
Sugiyama N, Masuda T, Shinoda K, et al. Phosphopeptide enrichment by aliphatic hydroxy acid-modified metal oxide chromatography for nano-LC-MS/MS in proteomics applications. Mol Cell Proteomics, 2007, 6(6): 1103-1109. DOI:10.1074/mcp.T600060-MCP200
|
| [55] |
Winter D, Seidler J, Ziv Y, et al. Citrate boosts the performance of phosphopeptide analysis by UPLC-ESI-MS/MS. J Proteome Res, 2009, 8(1): 418-424. DOI:10.1021/pr800304n
|
| [56] |
Larsen MR, Thingholm TE, Jensen ON, et al. Highly selective enrichment of phosphorylated peptides from peptide mixtures using titanium dioxide microcolumns. Mol Cell Proteomics, 2005, 4(7): 873-886. DOI:10.1074/mcp.T500007-MCP200
|
| [57] |
Li JR, Wang JF, Yan YM, et al. Comprehensive evaluation of different TiO2-based phosphopeptide enrichment and fractionation methods for phosphoproteomics. Cells, 2022, 11(13): 2047. DOI:10.3390/cells11132047
|
| [58] |
Li QR, Ning ZB, Tang JS, et al. Effect of peptide-to-TiO2 beads ratio on phosphopeptide enrichment selectivity. J Proteome Res, 2009, 8(11): 5375-5381. DOI:10.1021/pr900659n
|
| [59] |
Zarei M, Sprenger A, Metzger F, et al. Comparison of ERLIC-TiO2, HILIC-TiO2, and SCX-TiO2 for global phosphoproteomics approaches. J Proteome Res, 2011, 10(8): 3474-3483. DOI:10.1021/pr200092z
|
| [60] |
Yue XS, Hummon AB. Combination of multistep IMAC enrichment with high-pH reverse phase separation for in-depth phosphoproteomic profiling. J Proteome Res, 2013, 12(9): 4176-4186. DOI:10.1021/pr4005234
|
| [61] |
Ren L, Li CY, Shao WL, et al. TiO2 with tandem fractionation (TAFT): an approach for rapid, deep, reproducible, and high-throughput phosphoproteome analysis. J Proteome Res, 2018, 17(1): 710-721. DOI:10.1021/acs.jproteome.7b00520
|
 2022, Vol. 38
2022, Vol. 38