1. 浙江万里学院 生物与环境学院, 浙江 宁波 315100;
2. 江西省林业科学院, 江西 南昌 330033;
3. 宁波市林场, 浙江 宁波 315100
收稿日期:2022-03-23;接收日期:2022-06-21
基金项目:宁波市科技创新2025现代种业重大专项项目(2021Z005);浙江省重点研发计划(2021C02053);浙江省“生物工程”一流学科学生创新项目(CX2021047)
作者简介:吴月燕 浙江万里学院二级教授、研究生导师。主要从事园林园艺植物生理与分子生物学、植物生理生态等的教学和科研工作,为国家教育部和浙江省高等学校教学指导委员会委员;先后主持或参加了国家、省、市科研项目20余项,以第一负责人获得省部级、市级科技进步奖10余项;带领的科研团队培育葡萄和杜鹃花新品种42个,以第一发明人获得国家发明专利、新型专利及软件著作权10余项.
1. College of Biological and Environmental Sciences, Zhejiang Wanli University, Ningbo 315100, Zhejiang, China;
2. Jiangxi Academy of Forestry, Nanchang 330033, Jiangxi, China;
3. Ningbo Forest Farm, Ningbo 315100, Zhejiang, China
Received: March 23, 2022; Accepted: June 21, 2022
Supported by: Ningbo Science and Technology Innovation 2025 Major Special Project of Modern Seed Industry (2021Z005); Key Research and Development Project in Zhejiang Province, China (2021C02053); "Bioengineering" First Class Student Innovation Project in Zhejiang Province, China (CX2021047)
杜鹃花属(Rhododendron) 是杜鹃花科(Ericaceae) 中最大的属,是中国最大的木本被子植物属,该属广泛分布于整个北半球,从热带东南亚到澳大利亚东北部[1]。全世界共有杜鹃花属1 000余种,我国有丰富的杜鹃花资源,是世界杜鹃分布中心,约有600余种,其中有540余种为我国南方及西南地区特有[1-2]。杜鹃花种类丰富,按是否具有香气可分为有香型和无香型杜鹃,其中有香型杜鹃品种甚少。云锦杜鹃为有香型杜鹃,是中国特有的杜鹃属物种。云锦杜鹃(Rhododendron fortunei) 是杜鹃花科,杜鹃花属[3],常绿杜鹃亚属植物,云锦杜鹃花大、粉红色,开花时具有芳香[4],广泛分布于海拔620−2 000 m山脊向阳处或林下[5-6],具有较高的园林观赏价值。诺娃杜鹃(Rhododendron ‘Nova Zembla’) 是杜鹃花科,杜鹃花属,常绿杜鹃亚属植物,诺娃杜鹃花朵为宽漏斗形且花瓣呈深玫红色,最上一枚花瓣具有深褐红色斑点,开花时无香味散发,属于无香型杜鹃,它可耐受寒冷–28 ℃,可种植于高海拔地区,是目前高山杜鹃畅销的品种之一。与花色、花型等其他花朵性状的研究相比,对于花香的研究相对滞后,直到近几年才逐渐深入。当前,对植物花香物质合成途径及关键基因研究已成为热点[7]。
花香是植物的重要性状,在植物生长发育及进化中起着非常重要的作用,如保护植物自身免受病原体、寄生虫和草食动物的伤害,还可吸引授粉者和种子传播者[8]。植物花香由一系列低分子量的挥发性化合物组成,在植物与生物和非生物因素相互作用下产生,穿过细胞膜并释放到周围环境中[9]。植物不同器官均可释放挥发性有机化合物,迄今为止,已鉴定出1 700多种花香挥发性有机化合物,合成途径有萜类、苯丙类/苯类和脂肪酸类生物合成途径[10]。
萜类化合物,也被称为萜烯或类异戊二烯,萜类化合物是植物产生的挥发性化合物中含量最多且种类最丰富的化学物质[11],与参与初级代谢的少量萜类相比,大多数萜类为次生代谢物,虽然它们不参与植物的基本生长发育,但它们仍具有重要作用,它们是芳香化合物主要成分也是药理化合物,且能参与植物的防御反应[12]。近年来,本研究团队测定了云锦杜鹃花瓣香气物质成分,分析结果得出云锦杜鹃特征香气成分有苯环型化合物、萜类化合物和醇类化合物,其中苯环型化合物相对含量最高,萜类化合物种类最多[13]。萜烯合成酶(terpene synthases, TPSs) 是萜类化合物合成的关键酶,可催化不同的前体物质最终生成不同的萜类化合物。虽然萜类化合物的种类众多且数量庞大,但它们都由二甲基烯丙基二磷酸(dimethylallyl pyrophosphate, DMAPP) 和异戊烯基二磷酸(isopentenyl pyrophosphate, IPP) 经一系列催化过程而形成[14]。DMAPP与IPP在戊烯基转移酶的催化下缩合产生直接前体香叶酰二磷酸(geranyl pyrophosphate, GPP)、法呢基二磷酸(farnesyl pyrophosphate, FPP) 和香叶基香叶基焦磷酸(geranylgeranylpyrophosphate, GGPP)。随后,TPSs催化前体形成多种萜类化合物,包括半萜(C5)、单萜(C10)、倍半萜(C15)和二萜(C20)[12]。TPS基因家族系统分为7个亚家族,分别为TPS-a、TPS-b、TPS-c、TPS-d、TPS-e/f、TPS-g和TPS-h。其中TPS-a、TPS-b和TPS-g存在于被子植物中,TPS-a主要合成倍半萜合酶,TPS-b和TPS-g主要合成单萜合酶;TPS-c与TPS-e/f密切相关,主要合成二萜合酶,TPS-c存在于裸子植物中,TPS-e/f存在于维管植物中[14-15]。近年来已在多种植物中鉴定出TPS基因家族成员,如小麦[16]、拟南芥[17]和葡萄[18]等,拟南芥中鉴定出32个AtTPS基因,在小麦和葡萄中分别发现14个TaTPS和152个VvTPS基因。
对杜鹃花TPS基因家族进行的研究为杜鹃花香合成分子机制研究奠定基础。本研究采用生物信息学分析的方法,下载杜鹃花(Rhododendron simsii) 基因组序列及蛋白组序列,首次对杜鹃花中TPS基因家族成员进行鉴定,从基因结构及系统进化分析、染色体定位及保守基序等方面进行分析,结合云锦杜鹃转录组测定结果,分析云锦杜鹃不同生长发育时期花瓣中的表达水平,使用顶空固相微萃取和气相色谱-质谱联用技术,测定不同时期云锦杜鹃花瓣中萜类物质含量,本文实验的试供材料云锦杜鹃与诺娃杜鹃与鉴定TPS基因家族成员所采用基因组均为杜鹃花科、杜鹃属的常绿亚属灌木、落叶灌木植物,为研究TPS基因家族成员在杜鹃花香代谢调控中的作用奠定基础。
1 材料与方法
1.1 材料 试供材料为长势良好的9年生云锦杜鹃和15年生诺娃杜鹃,种植于浙江省宁波市四明山国家森林公园。随机选取5株长势基本一致的云锦杜鹃植株和5株长势基本一致的诺娃杜鹃植株,从中采集不同时期且无机械损伤的杜鹃花,根据花瓣开放程度分为4个时期。分别为花瓣紧闭的花芽期、花瓣未完全张开的花苞期、花瓣呈完全开放状态的盛开期和花瓣凋零出现黄色的衰败期。将样品用液氮迅速冷冻后置于–80 ℃超低温冰箱保存备用。
1.2 方法
1.2.1 RsTPS家族成员的鉴定 本研究从美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI) 上下载杜鹃花(Rhododendron simsii) 基因组序列和蛋白质序列。在Pfam (http://pfam.xfam.org/) 数据库中下载含有TPS基因结构域的种子文件PF01397和PF03936,利用HMMER3.1软件搜索杜鹃花TPS基因家族,得到65条候选序列;将得到的蛋白序列进一步通过NCBI Conserved Domain Database (CDD) (https://www.ncbi.nlm.nih.gov/cdd/)数据库进行结构域验证,经过手动确认后,除去非全长序列,得到候选的具有全长开放阅读框(open reading frame, ORF) 的杜鹃花TPS家族成员序列,最终得到47个RsTPS家族成员。利用在线软件ProtParam (https://web.expasy.org/protparam) 分析RsTPS家族成员的氨基酸数量、理论等电点、分子量大小和不稳定性指数等理化特性。利用WoLF PSORT (https://wolfpsort.hgc.jp/) 进行亚细胞定位预测分析。
1.2.2 RsTPS家族的系统进化树、保守基序以及基因结构分析 使用MEGA 7软件以默认参数将杜鹃花RsTPS蛋白序列进行多序列比对,再用邻接法构建系统发育树,bootstraps设置重复次数1 000次,其余参数为默认。使用在线软件MEME (http://meme-suite.org/tools/meme) 预测RsTPS家族蛋白序列保守结构域,使用TBtools软件展示保守基序结果。采用GSDS2.0在线网站(http://gsds.gao-lab.org/) 绘制杜鹃花47个RsTPS基因家族成员的基因结构,对其外显子数/编码区(coding sequence, CDS) 进行分析;利用TBtools软件将RsTPS系统进化树、保守结构域和基因结构结果按进化树顺序进行可视化。
1.2.3 RsTPS家族进化树分析与染色体定位分析 从在线网站Phytozome 13 (https://phytozome-next.jgi.doe.gov/) 下载模式植物拟南芥(Arabidopsis thaliana) 的AtTPS家族蛋白序列,使用MEGA 7软件以默认参数将杜鹃花和拟南芥的AtTPS蛋白序列进行多序列比对,再用邻接法构建系统发育树。从杜鹃基因组数据库中获取基因的位置信息,使用在线网站MG2C (http://mg2c.iask.in/mg2c_v2.1/) 绘制染色体定位示意图。
1.2.4 转录组数据分析TPS基因表达模式 将贮藏的样品用干冰保存,送至深圳华大基因股份有限公司进行转录组测序。将同一品种不同植株上采集的同一时期的样品混合建库测序,每组样品进行3次生物学重复。诺娃杜鹃与云锦杜鹃分别以NW和YJ表示,4个时期分别为花芽期A、花苞期B、盛开期C和衰败期D。使用单拷贝直系同源数据库BUSCO对组装的转录本进行质量评估,通过与保守基因进行比较,在一定程度上说明转录组组装的完整性。
1.2.5 qRT-PCR分析 使用Primer Premier 6软件设计TPS家族基因的特异性引物(表 1),用RNAprep Pure多糖多酚植物总RNA提取试剂盒(TIANGEN, China) 提取总RNA,使用NovoScript Plus All-in-one 1st Strand cDNA Synthesis SuperMix (gDNA Purge) 反转录试剂盒(Novoprotein, China) 合成cDNA。以EF1α为内参基因,使用TransStart Tip Green qPCR SuperMix试剂盒(北京全式金生物技术有限公司) 进行qRT-PCR反应,每个反应设3个重复。采用2−ΔΔCt法进行数据分析,使用SPSS 26.0进行单因素方差分析,用Origin软件绘图。
表 1 TPS家族基因表达分析的qRT-PCR引物
Table 1 qRT-PCR primers for expression analysis of TPS
| Gene name |
Forward primers (5'→3') |
Reverse primers (5'→3') |
| TPS1 |
TGGTTAGTCACGCCTTGGA |
CTTCTTGGTGAGTTGCTTGC |
| TPS4 |
TCGTTTGCCTTGGGACCTAT |
CTGCGGATGTCGTTGAGAAG |
| TPS9 |
CGACAGGGTTCTCCAAGTATG |
TAGCCTCAGCCACGGAATC |
| TPS10 |
TGCCGTTGAAAGGTGGGATA |
GGTGAGGGATGATGTGAATGC |
| TPS12 |
CAAGCTCATCCAATGATCCG |
GCCCAAATTCAGTGATTGCC |
| TPS13 |
GAACTAGGCACAGACATCAAGG |
CATCCAACCATCAAGGAGTAGG |
| EF1α |
CTCGATTGCCACACTTCCCA |
CATCTTCACCATCCCAGCGT |
1.2.6 不同时期杜鹃花瓣中萜类物质含量测定 本实验参考姚晨阳等[19]的方法,并作修改,采用顶空固相微萃取(head space-solid phase micro-extraction, HS-SPME) 结合气相色谱-质谱联用技术,测定不同时期云锦杜鹃及诺娃杜鹃花瓣中TPS对应产物相对含量。气相色谱-质谱联用仪品牌为Agilent,型号为78890B-5977B,色谱柱品牌为Agilent,型号为DB-Wax (30 m×250 μm×0.25 μm)。取样本(100±10) mg于20 mL顶空瓶中,加入10 μL 2-辛醇为内标,密封顶空瓶;每个样品进行6次生物学重复。
萃取条件:萃取温度:60 ℃;萃取时间:30 min;解析时间:4 min。
色谱条件:载气:氦气;流速:1 mL/min;升温程序:初始温度40 ℃,保持4 min,以5 ℃/min升至245 ℃,保持5 min;进样方式:不分流进样。
质谱条件:电离方式:EI源;电离能量:70 eV;离子源温度:230 ℃;接口温度:250 ℃;四级杆温度:150 ℃;全扫描模式,扫描范围20–400 m/z。
定性分析:通过化学工作站数据处理系统检索及核对NIST标准质谱图库并结合有关文献进行解析,当匹配度大于80 (最大值100) 的鉴定结果予以使用。
定量分析:以10 μL纯度≥99.5%的2-辛醇为标品,求得各挥发性成分相对于标准品的释放量(ng/g)=(各组分的峰面积/标准品的峰面积)×标准品体积(μL)×标准品浓度(μg/μL)× 1 000/样品量(g)。
1.2.7 TPS表达水平与萜类物质相关性分析 根据云锦杜鹃花瓣中萜类物质的含量变化和TPS成员的相对表达量,使用SPSS 26.0软件进行相关性分析后再用Origin软件绘制相关性热图,使萜类物质的释放与基因表达量的相关性结果可视化。
2 结果与分析
2.1 RsTPS家族成员信息 在杜鹃花基因组中鉴定获得47个RsTPS家族成员,根据其在染色体上的位置,依次命名为RsTPS1−RsTPS47 (表 2)。杜鹃花47个RsTPS家族成员编码蛋白的理化性质分析结果如表 2所示。RsTPS基因编码的蛋白质氨基酸为196−877个,预测的分子量范围为22.37− 101.69 kDa。理论等电点(isoelectric point, pI) 为4.37−8.2,4个成员(8.5%) 具有碱性(pI > 7),43个成员(91.5%) 被认为具有酸性(pI < 7)。不稳定性指数(instability index) 范围为29.32−50.82,具有10个不稳定性指数小于40的稳定蛋白,37个不稳定性指数大于40的不稳定蛋白。脂肪族指数(aliphatic index) 为74.04−106.45,平均亲水性为–0.441−0.182,具有39个平均疏水性为负值的亲水性蛋白。亚细胞定位预测分析结果表明,有24个RsTPS成员(51.0%) 主要存在于细胞质中,15个RsTPS成员(31.9%) 主要定位于叶绿体,少数成员主要定位于细胞核、内质网和细胞骨架。
表 2 杜鹃花TPS基因家族成员信息
Table 2 Members of RsTPS gene family in Rhododendron
| Gene name |
Protein ID
(NCBI) |
No. of AA |
Molecular weight |
pI |
Instability index |
Aliphatic index |
GRAVY |
Subcellular location |
| RsTPS1 |
KAF7150501.1 |
864 |
99.08 |
6.61 |
42.21 |
84.79 |
–0.325 |
Cytosol |
| RsTPS2 |
KAF7151762.1 |
835 |
51.63 |
6.19 |
39.99 |
85.51 |
–0.315 |
Cytosol |
| RsTPS3 |
KAF7151494.1 |
419 |
45.50 |
6.16 |
35.94 |
82.63 |
–0.325 |
Chloroplast |
| RsTPS4 |
KAF7150330.1 |
368 |
38.12 |
5.87 |
40.13 |
85.05 |
–0.278 |
Chloroplast |
| RsTPS5 |
KAF7151169.1 |
369 |
39.97 |
5.70 |
39.20 |
84.04 |
–0.324 |
Chloroplast |
| RsTPS6 |
KAF7148595.1 |
707 |
60.56 |
5.24 |
47.72 |
83.86 |
–0.320 |
Chloroplast |
| RsTPS7 |
KAF7144461.1 |
525 |
36.91 |
6.04 |
38.02 |
91.83 |
–0.258 |
Cytosol |
| RsTPS8 |
KAF7145602.1 |
480 |
52.52 |
5.78 |
45.51 |
89.83 |
–0.319 |
Cytosol |
| RsTPS9 |
KAF7146392.1 |
547 |
101.69 |
7.90 |
46.35 |
93.80 |
–0.293 |
Chloroplast |
| RsTPS10 |
KAF7145501.1 |
453 |
47.35 |
7.21 |
47.52 |
93.66 |
–0.255 |
Mitochondria and chloroplast |
| RsTPS11 |
KAF7146224.1 |
399 |
24.55 |
5.54 |
44.02 |
93.38 |
–0.331 |
Cytosol |
| RsTPS12 |
KAF7146676.1 |
331 |
95.09 |
8.20 |
31.28 |
98.43 |
–0.133 |
Cytosol |
| RsTPS13 |
KAF7144807.1 |
347 |
29.50 |
5.97 |
29.32 |
96.14 |
–0.216 |
Cytosol |
| RsTPS14 |
KAF7146169.1 |
533 |
28.98 |
5.59 |
49.40 |
87.47 |
–0.279 |
Cytosol |
| RsTPS15 |
KAF7145561.1 |
324 |
34.73 |
5.52 |
50.75 |
74.04 |
–0.441 |
Chloroplast |
| RsTPS16 |
KAF7145077.1 |
462 |
29.13 |
4.88 |
47.46 |
92.25 |
–0.242 |
Nucleus |
| RsTPS17 |
KAF7142879.1 |
877 |
24.86 |
7.33 |
40.65 |
83.07 |
–0.428 |
Cytosol |
| RsTPS18 |
KAF7141001.1 |
414 |
30.53 |
5.71 |
35.04 |
87.42 |
–0.263 |
Cytosol |
| RsTPS19 |
KAF7140427.1 |
210 |
42.92 |
4.73 |
43.54 |
92.43 |
0.069 |
Cytosol |
| RsTPS20 |
KAF7140868.1 |
256 |
26.04 |
4.37 |
35.81 |
90.74 |
–0.062 |
Chloroplast |
| RsTPS21 |
KAF7141198.1 |
245 |
25.07 |
5.15 |
41.68 |
91.14 |
–0.056 |
Cytosol |
| RsTPS22 |
KAF7142010.1 |
303 |
32.51 |
4.83 |
46.26 |
95.54 |
–0.110 |
Nucleus |
| RsTPS23 |
KAF7141456.1 |
251 |
48.03 |
5.02 |
50.82 |
106.45 |
0.182 |
Chloroplast |
| RsTPS24 |
KAF7141749.1 |
211 |
28.09 |
4.80 |
47.11 |
90.09 |
–0.131 |
Cytosol |
| RsTPS25 |
KAF7142095.1 |
265 |
29.27 |
5.21 |
36.81 |
95.66 |
–0.093 |
Cytosol |
| RsTPS26 |
KAF7140217.1 |
378 |
28.15 |
5.44 |
35.22 |
90.08 |
–0.245 |
Nucleus |
| RsTPS27 |
KAF7141331.1 |
224 |
35.36 |
4.93 |
44.60 |
96.70 |
0.070 |
Endoplasmic reticulum |
| RsTPS28 |
KAF7140772.1 |
218 |
34.46 |
4.87 |
47.17 |
101.51 |
–0.052 |
Cytosol |
| RsTPS29 |
KAF7139794.1 |
284 |
30.16 |
4.51 |
45.43 |
94.40 |
0.012 |
Cytosol |
| RsTPS30 |
KAF7141729.1 |
245 |
22.37 |
5.06 |
44.49 |
102.73 |
0.090 |
Chloroplast |
| RsTPS31 |
KAF7140902.1 |
259 |
72.28 |
4.69 |
45.29 |
99.38 |
0.137 |
Chloroplast |
| RsTPS32 |
KAF7141077.1 |
245 |
68.22 |
5.30 |
42.22 |
101.96 |
0.106 |
Chloroplast |
| RsTPS33 |
KAF7141507.1 |
315 |
50.89 |
5.76 |
41.41 |
93.75 |
–0.026 |
Cytosol |
| RsTPS34 |
KAF7141310.1 |
304 |
42.50 |
4.83 |
43.55 |
94.87 |
–0.015 |
Cytoskeleton |
| RsTPS35 |
KAF7129345.1 |
258 |
49.25 |
6.01 |
44.57 |
97.91 |
–0.033 |
Cytosol |
| RsTPS36 |
KAF7126774.1 |
196 |
46.26 |
4.93 |
47.72 |
97.45 |
0.058 |
Cytosol |
| RsTPS37 |
KAF7123307.1 |
621 |
71.25 |
5.53 |
45.34 |
86.81 |
–0.240 |
Chloroplast |
| RsTPS38 |
KAF7123219.1 |
586 |
63.99 |
5.13 |
42.31 |
89.32 |
–0.210 |
Chloroplast |
| RsTPS39 |
KAF7123140.1 |
437 |
55.27 |
4.98 |
43.2 |
88.99 |
–0.206 |
Cytosol |
| RsTPS40 |
KAF7123893.1 |
424 |
67.38 |
5.29 |
45.55 |
87.38 |
–0.229 |
Cytoskeleton |
| RsTPS41 |
KAF7125050.1 |
404 |
65.26 |
5.25 |
50.29 |
89.31 |
–0.182 |
Chloroplast |
| RsTPS42 |
KAF7120544.1 |
614 |
44.64 |
5.34 |
44.82 |
91.16 |
–0.305 |
Cytosol and Chloroplast |
| RsTPS43 |
KAF7119419.1 |
549 |
42.54 |
5.43 |
43.44 |
92.37 |
–0.364 |
Cytosol |
| RsTPS44 |
KAF7115943.1 |
484 |
81.54 |
5.57 |
45.74 |
93.29 |
–0.271 |
Cytosol |
| RsTPS45 |
KAF7115926.1 |
582 |
60.68 |
5.33 |
42.38 |
88.81 |
–0.322 |
Cytosol |
| RsTPS46 |
KAF7112783.1 |
560 |
55.19 |
5.05 |
42.56 |
86.52 |
–0.297 |
Cytosol |
| RsTPS47 |
KAF7112304.1 |
386 |
62.84 |
6.51 |
45.49 |
87.64 |
–0.201 |
Chloroplast |
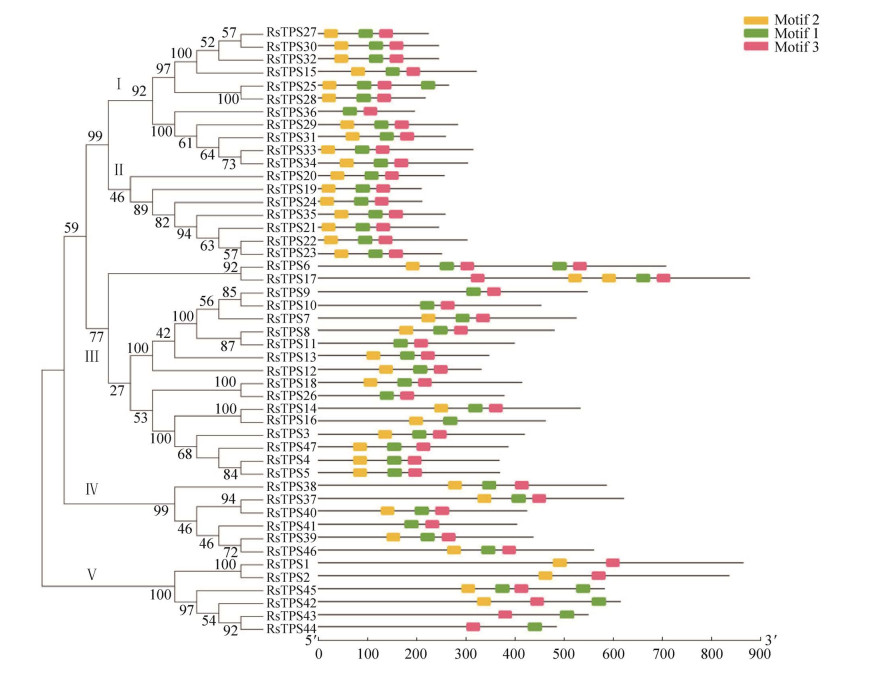
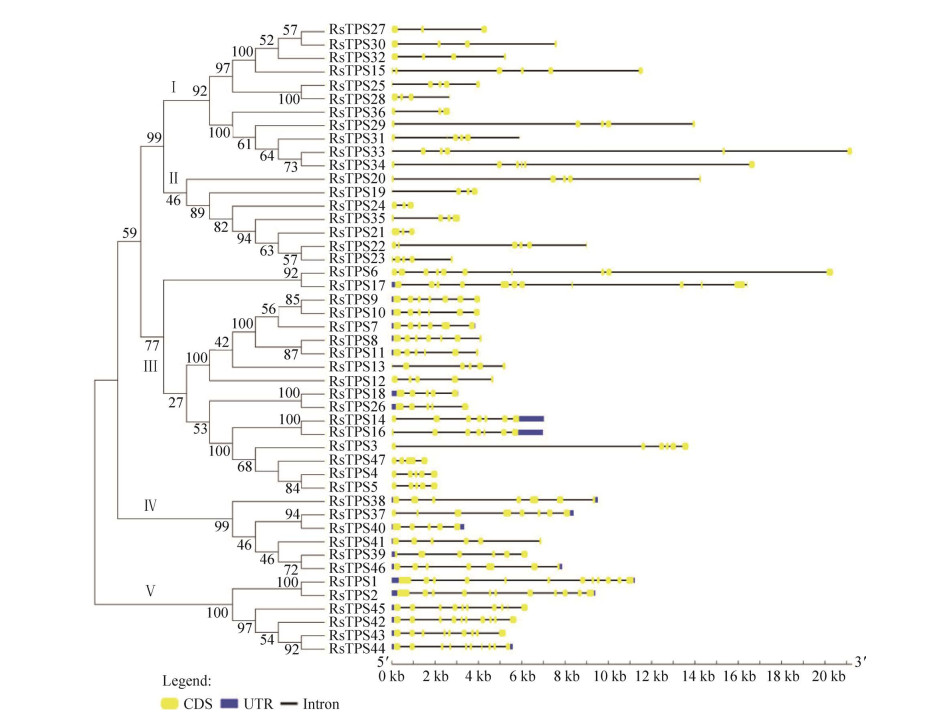
2.2 RsTPS家族的系统进化树、保守基序与基因结构分析 利用RsTPS蛋白氨基酸全长序列构建系统发育树,将47个RsTPS分为5个亚组,如图 1和2所示。保守基序分析表明,RsTPS家族成员中最保守的基序为motif3,存在于除RsTPS 16外的所有成员中;其次是motif1,有45个成员存在motif1,2个无motif1的成员均属于第Ⅴ亚组;有39个成员存在motif2,其中8个无motif2结构的成员中4个属于第Ⅲ亚组;motif1和motif3同时存在的家族成员有44个;motif1、motif2和motif3均存在的成员有36个。基因结构分析表明,RsTPS都具有外显子,不同亚组之间具有明显差异,外显子数介于3−12之间,同一亚组具有相似的基因结构。其中,第Ⅰ亚组和第Ⅱ亚组外显子数均在3−6范围内,第Ⅳ亚组外显子数在5−8之间,第Ⅴ亚组外显子数在10−12之间。
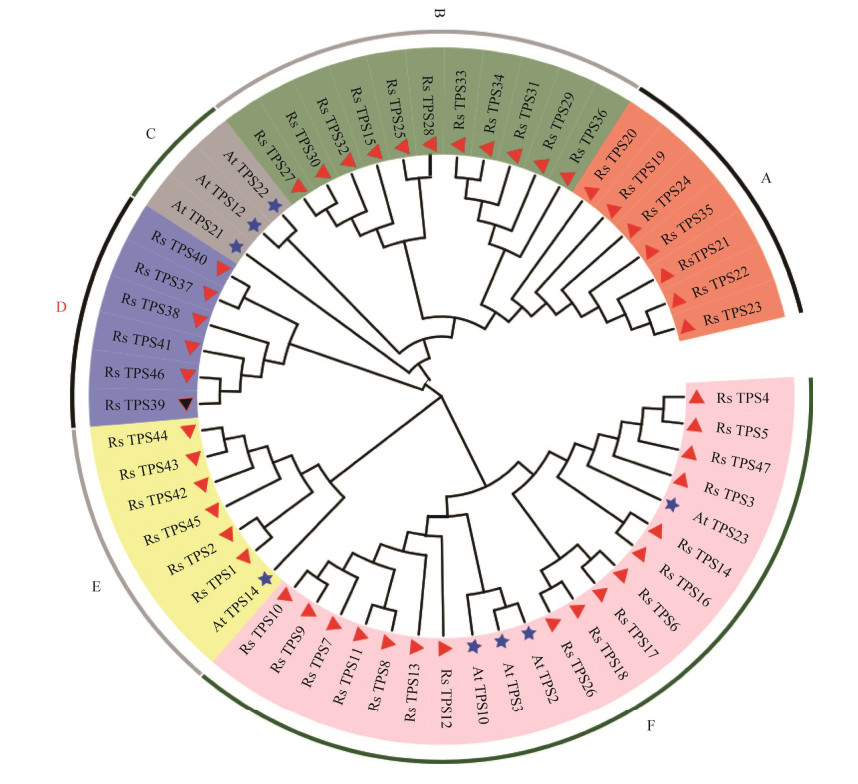
2.3 RsTPS家族的系统进化树和染色体定位分析 如图 3所示,利用杜鹃花和拟南芥AtTPS家族蛋白序列构建系统发育树,根据RsTPS家族的系统进化树、保守基序与基因结构分析结果和进化树聚类可将47个RsTPS和8个AtTPS分为A、B、C、D、E、F六个亚组,RsTPS15、RsTPS25、RsTPS27−34和RsTPS36基因结构相似,外显子数相近故分为一个亚组。F组RsTPS成员最多,有17个,其他各组RsTPS数都在6−11之间,C组成员全部来自于拟南芥,A、B、D组成员都属于杜鹃花,E组和F组拟南芥和杜鹃TPS均存在。如图 4所示,47个RsTPS家族成员分布在9条染色体上,RsTPS基因家族成员有5个分布在杜鹃的染色体Chr2上,10个分布在Chr4,17个分布在Chr6,Chr3、Chr5、Chr10和Chr11上分别分布1个RsTPS基因家族成员,Chr12上分布5个,Chr13上分布2个。
2.4 不同时期杜鹃花瓣TPS基因家族的表达水平 从云锦和诺娃杜鹃花转录组测序的数据库中筛选得到7条功能注释为TPS的基因。经软件对基因的表达量聚类,如图 5所示,这7个基因在8组样品中均显示表达差异。在不同品种中表达差异显著,TPS2只在诺娃杜鹃中高表达,TPS1、TPS4、TPS9、TPS10、TPS12和TPS13在云锦杜鹃中高表达。在同一品种不同时期中的表达也存在显著差异。TPS4和TPS9在云锦杜鹃花芽期表达量最高,其表达量随花期呈现逐渐降低的趋势。TPS4和TPS9在花芽期的表达量分别是花苞期、盛开期、衰败期的1.43倍、1.47倍、2.70倍和63.60倍、41.90倍、92.20倍。TPS1和TPS10、TPS12和TPS13在云锦杜鹃盛开期表达量最高,且呈现出先上升后下降的表达趋势。TPS1、TPS10、TPS12、TPS13盛开期的表达量依次是花芽期的84.65倍、163.10倍、57.87倍、5.37倍;花苞期的1.41倍、2.37倍、20.58倍、1.73倍;衰败期的3.10倍、2.17倍、2.06倍、10.17倍。
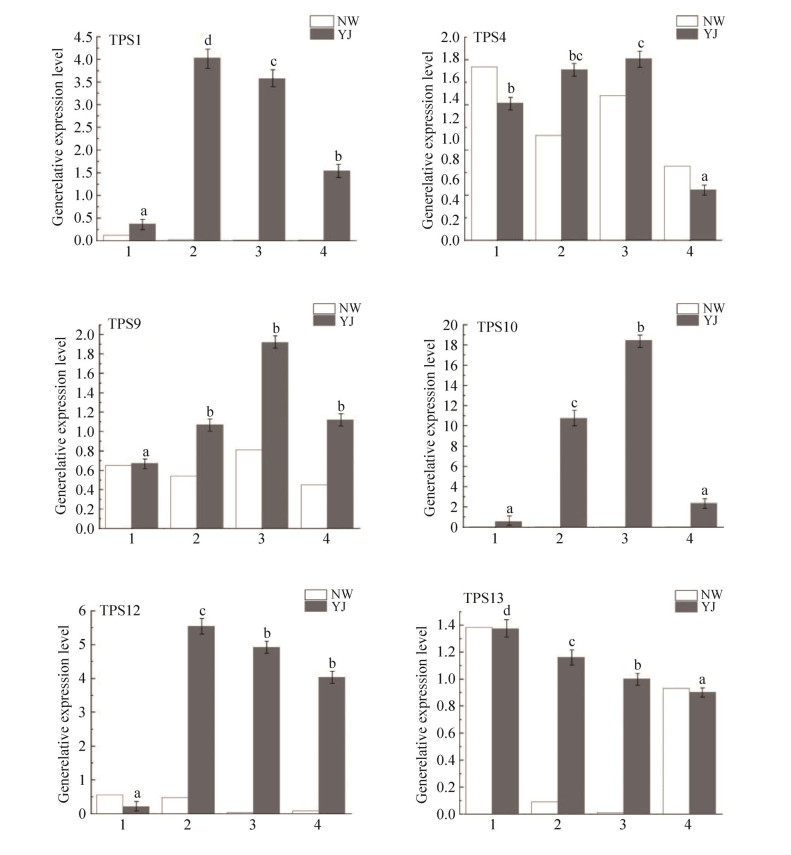
2.5 qRT-PCR分析 如图 6所示,这6个基因在8组样品中均显示表达差异。在不同品种中表达差异显著,TPS1、TPS4、TPS9、TPS10、TPS12和TPS13都在云锦杜鹃中高表达。在同一品种不同时期中的表达也存在显著差异。TPS1和TPS12在云锦杜鹃花苞期的表达量最高,呈现先上升后下降的表达趋势,分别是花芽期的10.89倍、26.38倍;盛开期的1.13倍、1.13倍、衰败期的2.62倍、1.22倍。TPS4、TPS9和TPS10在云锦杜鹃盛开期表达量最高,且呈现出先上升后下降的表达趋势。TPS4、TPS9和TPS10盛开期的表达量依次是花芽期的1.32倍、2.87倍和34.15倍;花苞期的1.06倍、1.79倍和1.72倍;衰败期的3.59倍、1.71倍和1.11倍。TPS13在云锦杜鹃花芽期表达量最高,其表达量随花期呈现逐渐降低的趋势。TPS13在花芽期的表达量分别是花苞期、盛开期和衰败期的1.18倍、1.37倍和1.52倍。
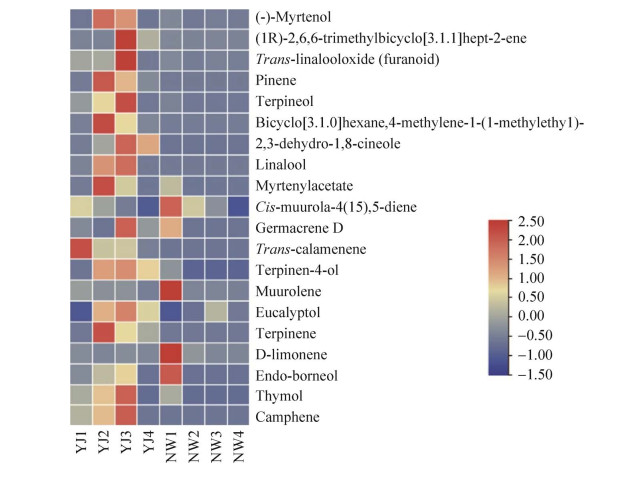
2.6 不同时期杜鹃花瓣中萜类物质含量测定分析 对云锦杜鹃和诺娃杜鹃4个时期花瓣挥发性成分进行鉴定分析,对其中萜类物质进行分析,利用软件绘制热图,使云锦杜鹃与诺娃杜鹃不同时期的萜类物质的释放结果可视化(图 7)。根据GC-MS检测出的20种含量最多的萜类物质分析,不同品种杜鹃花释放的萜类物质含量有较大差异,在同一品种杜鹃花的不同时期,萜类物质释放量也不同。在不同品种中,萜类物质在诺娃杜鹃中释放量较少,在云锦杜鹃中大量释放。在诺娃杜鹃中只检测出13种萜类物质,其中12种萜类物质在花芽期含量最多,含量呈逐渐下降趋势。在云锦杜鹃中,只有1种萜类物质在花芽期释放量最多,5种萜类物质在花苞期释放量最多,有12种萜类物质在盛开期释放量最多,2种萜类物质在衰败期释放量最多。在云锦杜鹃中,萜类物质含量呈现出先上升后下降的变化趋势。
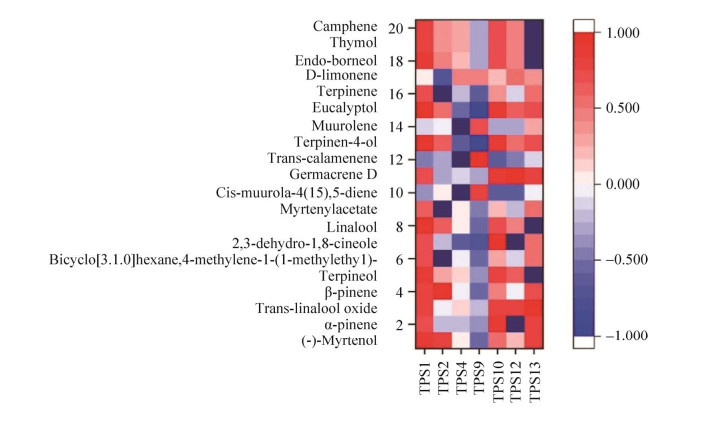
2.7 TPS表达量与萜类物质挥发量的相关性分析 转录组数据分析TPS表达量与萜类物质挥发量的相关性,如图 8所示,TPS1与16种(80%) 萜类物质呈正相关,TPS2、TPS4、TPS9、TPS10、TPS12和TPS13分别有9种(45%)、6种(30%)、4种(20%)、17种(85%)、11种(55%) 和13种(65%) 萜类物质呈正相关。TPS1与(–)-桃金娘醇、α-蒎烯、反式芳樟醇氧化物、β-蒎烯、芳樟醇、松油烯-4-醇、桉树醇、内龙脑、百里酚和莰烯呈显著性正相关;TPS9与顺式依兰油二烯-4, 5-二烯、反-菖蒲烯和衣兰油烯呈显著性正相关;TPS10与α-蒎烯、反式芳樟醇氧化物、松油醇、桉叶油醇、芳樟醇、大牛儿烯D、松油烯-4-醇和桉树醇呈显著性正相关;TPS12与反式芳樟醇氧化物和大牛儿烯D呈显著性正相关;TPS13与(–)-桃金娘醇、α-蒎烯和反式芳樟醇氧化物呈显著性正相关。TPS1、TPS9、TPS10、TPS12和TPS13表达量与云锦花瓣中萜类挥发性物质含量呈显著性正相关。
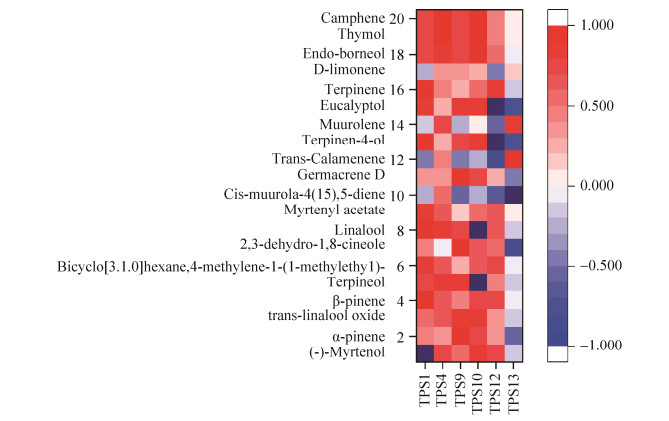
qRT-PCR分析TPS表达量与萜类物质挥发量的相关性,如图 9所示,TPS1与16种(80%) 萜类物质呈正相关,TPS4、TPS9、TPS10、TPS12和TPS13分别有19种(95%)、17种(85%)、15种(75%)、14种(70%) 和7种(35%) 萜类物质呈正相关。TPS1与其中12种物质呈显著性正相关,TPS4、TPS9、TPS10、TPS12和TPS13分别与其中7种、11种、11种、5种和2种萜类物质呈显著性正相关。
结合转录组与qRT-PCR数据,最终得到6个可能参与杜鹃花香调控的基因,分别为TPS1、TPS4、TPS9、TPS10、TPS12和TPS13。
3 讨论
3.1 RsTPS家族的鉴定和分析 本研究共在杜鹃花中鉴定出47个RsTPS基因家族成员,并对RsTPS基因的理化性质、系统进化树、基因结构、保守基序与染色体定位进行分析。所构建的进化树结果中RsTPS9、RsTPS10、RsTPS12和RsTPS13聚类在同一亚组中,但与RsTPS1聚类在不同亚组,表明RsTPS9、RsTPS10、RsTPS12和RsTPS13成员间蛋白序列相差较小,而与RsTPS1间蛋白序列差异较大。RsTPS家族成员编码的氨基酸个数在196−877个之间,分子质量为22.37− 101.69 kDa之间,与其他物种中TPS家族成员氨基酸个数及分子质量相近,推测TPS家族成员蛋白结构在不同物种中相对保守。保守基序分析表明,各亚组的RsTPS基因均含有一类或几类相同的保守基序,表明RsTPS基因在进化过程中相对保守。基因结构分析表明,同一亚组的RsTPS成员外显子数在同一范围内,说明RsTPS家族成员具有相似的基因结构。RsTPS基因家族存在不同分支上的差异、不同成员间基因结构的差异、预测的motif差异、等电点和不稳定性等理化参数的差异以及不同成员在杜鹃花瓣不同阶段的表达差异,预示了RsTPS家族成员存在功能差异,为进一步研究RsTPS家族提供理论依据。转录组数据表明,TPS1和TPS10、TPS12和TPS13在云锦杜鹃盛开期表达量最高,最高分别是衰败期的3.10倍、花苞期的2.37倍、花芽期的57.88倍、衰败期的10.17倍,且呈现出先上升后下降的表达趋势。TPS9在花芽期的表达量最高,最高是花苞期的63.60倍,呈现出先上升后下降的表达趋势。
3.2 TPS家族可能参与花香调控 TPS是通过催化复杂的碳正离子驱动的环化、重排和消除反应来生成不同种类萜类物质的关键酶,对TPS家族成员的进化差异和功能的继续研究将为萜类在调节与其他生物和环境的防御中的作用提供重要依据[20]。多位学者发现β-橙烯、芳樟醇、芳樟醇衍生物等是大多数品种花挥发物的主要成分[21],近年来已在多种植物中鉴定出TPS家族成员,如番茄、铁皮石斛和棉花等。Zeng等[21]发现两个不同品种的桂花‘Gecheng’和‘Liuye’在开花过程中‘Gecheng’的TPS1和TPS3的表达量是“Liuye”的100倍以上。TPS1表达量持续增加,而TPS3在盛开期的表达量最高,表达水平呈先上升后下降趋势,且它们的表达与花期单萜的产生一致,TPS基因的转录调控,特别是TPS1和TPS3,可能是调控芳樟醇或β-罗勒烯合成的最重要的机制,‘Gecheng’和‘Liuye’ TPS基因表达水平的差异表明TPS基因对桂花单萜的形成有重要贡献。Coppola等[22]利用RNA干扰技术,沉默了番茄植株中TPS9和TPS12基因的表达,并评估了这种转化对食草动物和拟寄生生物的影响,发现沉默TPS9和TPS12基因的植株产生的挥发性物质与对照组不同,降低了对蚜虫的吸引力,并导致了夜蛾幼虫的发育受损,从而提高了对外界的抵抗,提高存活率,进一步验证了TPS9和TPS12是调控挥发性物质产生的关键基因。Yu等[23]在铁皮石斛中共鉴定出34个DoTPS家族成员,其中DoTPS10可将GPP转化为芳樟醇;姜花的HcTPS6可催化GPP生成松萜、芳樟醇和香叶醇3种单萜类物质,HcTPS7催化GPP产生香桧烯[24];Huang等[25]于2021年发现PbTPS4、PbTPS5、PbTPS10可将GPP转化为芳樟醇,PbTPS3参与合成(β)-顺式/芳樟醇。Huang等[26]系统地分析了棉花挥发性物质,并对12种萜烯合成酶(GhTPS4–15)进行了表征。将过表达GhTPS12转化烟草植株,产生(3S)-芳樟醇,提高了棉花对棉铃虫和桃蚜两种害虫的驱避能力。Li等[27]发现AsTPS18和AsTPS19被注释为异戊二烯合成酶,它们通过MEP途径催化DMAPP生成异戊二烯。AsTPS21和AsTPS22被注释为共聚二磷酸合成酶,该酶参与二萜的生物合成。本研究中,在杜鹃花基因组中发现47个TPS家族成员,利用转录组数据对TPS家族成员表达水平进行分析,发现不同品种、不同时期TPS家族成员表达量不同。结合qRT-PCR测定结果和GC-MS测定结果,对基因表达水平和萜类物质含量进行相关性分析,TPS1与TPS4、TPS9、TPS10、TPS12和TPS13分别与12种、7种、11种、11种、5种和2种萜类物质呈显著性正相关。最终表明TPS1、TPS4、TPS9、TPS10、TPS12和TPS13可催化GPP生成多种萜类物质,参与花香调控。本文通过分析不同时期和不同品种杜鹃花瓣的TPS基因的表达模式,以及这些基因与萜类物质的相关性,从中鉴定出6个与多种萜类物质呈显著正相关的TPS基因,为后续研究杜鹃TPS家族基因调控花香提供了一定的理论依据。
 2022, Vol. 38
2022, Vol. 38