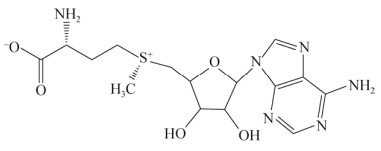
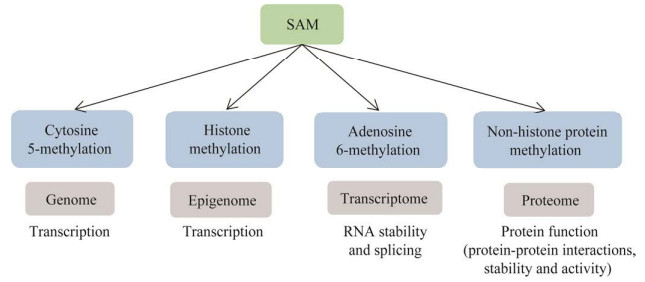
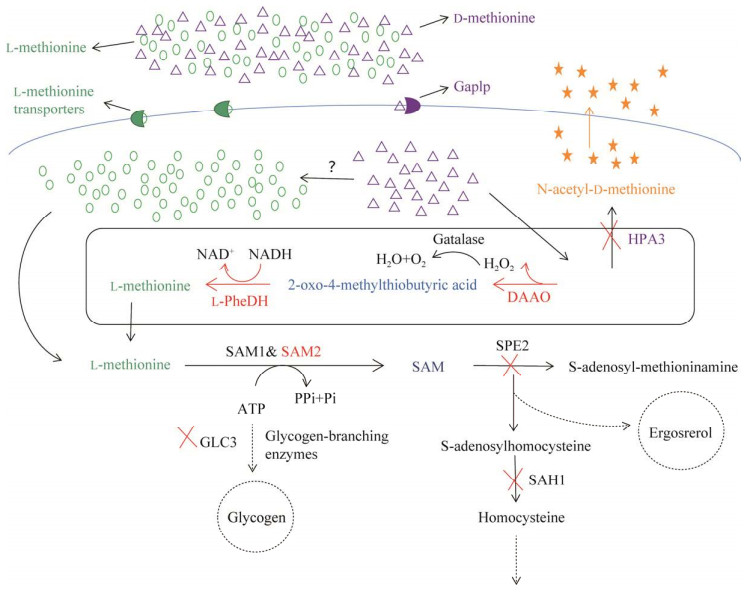
| [1] |
颜光波. ATP以及S-腺苷甲硫氨酸的多酶催化合成研究[D]. 武汉: 湖北大学硕士学位论文, 2021.
YAN GB. Studies on the multi-enzyme catalytic synthesis of ATP and S-adenosyl-l-homocysteine[D]. Wuhan: Master's Thesis of Hubei University, 2021 (in Chinese).
|
|
| [2] |
de la HG, CANTONI GL. The enzymatic synthesis of S-adenosyl-l-homocysteine from adenosine and homocysteine. Journal of Biological Chemistry, 1959, 234(3): 603-608. DOI:10.1016/S0021-9258(18)70253-6
|
|
| [3] |
ZHAO WJ, SHI F, HANG BJ, HUANG L, CAI J, XU ZN. The Improvement of SAM accumulation by integrating the endogenous methionine adenosyltransferase gene SAM2 in genome of the industrial Saccharomyces cerevisiae strain. Applied Biochemistry and Biotechnology, 2016, 178(6): 1263-1272. DOI:10.1007/s12010-015-1943-1
|
|
| [4] |
CHU J, QIAN JC, ZHUANG YP, ZHANG SL, LI YR. Progress in the research of S-adenosyl-l-methionine production. Applied Microbiology and Biotechnology, 2013, 97(1): 41-49. DOI:10.1007/s00253-012-4536-8
|
|
| [5] |
LIU Q, LIN Q, LI XY, ALI M, HE J. Construction and application of a "superplasmid" for enhanced production of antibiotics. Applied Microbiology Biotechnol, 2020, 104(4): 1647-1660. DOI:10.1007/s00253-019-10283-6
|
|
| [6] |
BIAN XY, SHI DM, XING KL, ZHOU HX, LU LL, YU DH, WU WZ. AMD1 upregulates hepatocellular carcinoma cells stemness by FTO mediated mRNA demethylation. Clinical and Translational Medicine, 2021, 11(3): e352.
|
|
| [7] |
GALIZIA I, OLDANI L, MACRITCHIE K, AMARI E, DOUGALL D, JONES TN, LAM RW, MASSEI GJ, YATHAM LN, YOUNG AH. S-adenosyl-l-methionine (SAMe) for depression in adults. The Cochrane Database of Systematic Reviews, 2016, 10(10): CD011286.
|
|
| [8] |
MAHMOOD N, ARAKELIAN A, CHEISHVILI D, SZYF M, RABBANI SA. S-adenosylmethionine in combination with decitabine shows enhanced anti-cancer effects in repressing breast cancer growth and metastasis. Journal of Cellular and Molecular Medicine, 2020, 24(18): 10322-10337. DOI:10.1111/jcmm.15642
|
|
| [9] |
SHOBAYASHI M, MUKAI N, IWASHITA K, HIRAGA Y, IEFUJI H. A new method for isolating S-adenosylmethionine (SAM)-accumulating yeast. Applied Microbiology and Biotechnology, 2006, 69(6): 704-710. DOI:10.1007/s00253-005-0009-7
|
|
| [10] |
刘艳辉. 酶法生产S-腺苷蛋氨酸[D]. 北京: 北京化工大学博士学位论文, 2016.
LIU YH. The enzymatic production of S-adenosyl-l- ethionine[D]. Beijing: Doctoral Dissertation of Beijing University of Chemical Technology, 2016 (in Chinese).
|
|
| [11] | |
|
| [12] | |
|
| [13] | |
|
| [14] |
WENG CY, MI ZY, LI MJ, QIN HB, HU ZC, LIU ZQ, ZHENG YG, WANG YS. Improvement of S-adenosyl-l-methionine production in Saccharomyces cerevisiae by atmospheric and room temperature plasma-ultraviolet compound mutagenesis and droplet microfluidic adaptive evolution. 3 Biotech, 2022, 12(9): 1-15.
|
|
| [15] |
RINSCHEN MM, IVANISEVIC J, GIERA M, SIUZDAK G. Identification of bioactive metabolites using activity metabolomics. Nature Reviews Molecular Cell Biology, 2019, 20(6): 353-367. DOI:10.1038/s41580-019-0108-4
|
|
| [16] |
LI XJ, EGERVARI G, WANG YG, BERGER SL, LU ZM. Regulation of chromatin and gene expression by metabolic enzymes and metabolites. Nature Reviews Molecular Cell Biology, 2018, 19(9): 563-578. DOI:10.1038/s41580-018-0029-7
|
|
| [17] |
RUDENKOA AY, MARIASINAA SS, SERGIEV PV, POLSHAKOV VI. Analogs of S-adenosyl-l-ethionine in studies of methyltransferases. Molecular Biology (Mosk), 2022, 56(2): 296-319.
|
|
| [18] |
KOTANDENIYA D, SEILER CL, FERNANDEZ J, PUJARI SS, CURWICK L, MURPHY K, WICKRAMARATNE S, YAN S, MURPHY D, SHAM YY, TRETYAKOVA NY. Can 5-methylcytosine analogues with extended alkyl side chains guide DNA methylation. Chemical Communications (Camb), 2018, 54(9): 1061-1064. DOI:10.1039/C7CC06867K
|
|
| [19] |
SHIMA H, MATSUMOTO M, ISHIGAMI Y, EBINA M, MUTO A, SATO Y, KUMAGAI S, OCHIAI K, SUZUKI T, IGARASHI K. S-adenosylmethionine synthesis is regulated by selective N 6-adenosine methylation and mRNA degradation involving METTL16 and YTHDC1. Cell Reports, 2017, 21(12): 3354-3363. DOI:10.1016/j.celrep.2017.11.092
|
|
| [20] |
ADAMS AM, KAPLAN NA, WEI ZY, BRINTON JD, MONNIER CS, ENACOPOL AL, RAMELOT TA, JONES JA. In vivo production of psilocybin in E. coli. Metabolic Engineering, 2019, 56: 111-119. DOI:10.1016/j.ymben.2019.09.009
|
|
| [21] |
KUNJAPUR AM, HYUN JC, PRATHER KLJ. Deregulation of S-adenosylmethionine biosynthesis and regeneration improves methylation in the E. coli de novo vanillin biosynthesis pathway. Microbial Cell Factories, 2016, 15(1): 1-17. DOI:10.1186/s12934-015-0402-6
|
|
| [22] |
LIU Q, LIN BX, TAO Y. Improved methylation in E. coli via an efficient methyl supply system driven by betaine. Metabolic Engineering, 2022, 72: 46-55. DOI:10.1016/j.ymben.2022.02.004
|
|
| [23] |
MARIASINA SS, CHANG CF, PETROVA OA, EFIMOV SV, KLOCHKOV VV, KECHKO OI, MITKEVICH VA, SERGIEV PV, DONTSOVA OA, POLSHAKOV VI. Williams-Beuren syndrome-related methyltransferase WBSCR27: cofactor binding and cleavage. The FEBS Journal, 2020, 28, 7(24): 5375-5393.
|
|
| [24] |
OUYANG Y, WU Q, LI JJ, SUN S, SUN SR. S-adenosylmethionine: a metabolite critical to the regulation of autophagy. Cell Proliferation, 2020, 53(11): e12891. DOI:10.1111/cpr.12891
|
|
| [25] | |
|
| [26] |
PIETROCOLA F, CASTOLDI F, KEPP O, CARMONA-GUTIERREZ D, MADEO F, KROEMER G. Spermidine reduces cancer-related mortality in humans. Autophagy, 2019, 15(2): 362-365. DOI:10.1080/15548627.2018.1539592
|
|
| [27] |
XU T, LI H, DAI Z, LAU GK, LI BY, ZHU WL, LIU XQ, LIU HF, CAI WW, HUANG SQ, WANG Q, ZHANG SJ. Spermidine and spermine delay brain aging by inducing autophagy in SAMP8 mice. Aging (Albany, NY.), 2020, 12(7): 6401-6414.
|
|
| [28] |
CAO XT, YANG MH, XIA Y, DOU J, CHEN K, WANG H, XI T, ZHOU CL. Strain improvement for enhanced production of S-adenosyl-l-methionine in Saccharomyces cerevisiae based on ethionine-resistance and SAM synthetase activity. Annals of Microbiology, 2012, 62(4): 1395-1402. DOI:10.1007/s13213-011-0389-0
|
|
| [29] |
SHIOZAKI S, SHIMIZU S, YAMADA H. Unusual intracellular accumulation of S-adenosyl-l-methionine by microorganisms. Agricultural and Biological Chemistry, 1984, 48(9): 2293-2300.
|
|
| [30] |
LI W, YE S, LUO KJ, GE FL, DU LJ, WU K, DING CW. Isolation and characterisation of Candida spmutants enriched in S-adenosylmethionine (SAM). Annals of Microbiology, 2007, 57(3): 383-387. DOI:10.1007/BF03175077
|
|
| [31] |
武晓乐. 重组毕赤酵母合成S-腺苷甲硫氨酸的发酵过程优化及转录谱分析[D]. 上海: 华东理工大学硕士学位论文, 2012.
WU XL. Fermentation optimization for S-adenosyl-l- ethionine production and transcriptome analysis in recombinant Pichia pastoris strains[D]. Shanghai: Master's Thesis of East China University of Science and Technology, 2012 (in Chinese).
|
|
| [32] |
万荣娥. S-腺苷甲硫氨酸高产菌株筛选及发酵工艺优化[D]. 武汉: 华中农业大学硕士学位论文, 2010.
WAN RE. Isolation of high-producers of S-adenosylmethionine and optimization of fermentation process[D]. Wuhan: Master's Thesis of Huazhong Agricultural University, 2010 (in Chinese).
|
|
| [33] |
LI GL, LI H, TAN YY, HAO N, YANG XL, CHEN KQ, OUYANG PK. Improved S-adenosyl-l-methionine production in Saccharomyces cerevisiae using tofu yellow serofluid. Journal of Biotechnology, 2020, 309: 100-106. DOI:10.1016/j.jbiotec.2020.01.004
|
|
| [34] |
王杰鹏, 韩晋军, 李晓楠, 刘沛溢, 谭天伟. S-腺苷-l-蛋氨酸高密度发酵工艺优化. 生物工程学报, 2009, 25(4): 533-536. WANG JP, HAN JJ, LI XN, LIU PY, TAN TW. Optimization of high-cell-density fermentation process for S-adenosyl-l-methionine production. Chinese Journal of Biotechnology, 2009, 25(4): 533-536 (in Chinese). DOI:10.3321/j.issn:1000-3061.2009.04.009
|
|
| [35] |
仝倩倩, 李亚亮, 王顺昌, 颜守保. 药源微生物遗传育种的研究进展. 辽宁大学学报(自然科学版), 2021, 48(4): 312-320. TONG QQ, LI YL, WANG SC, YAN SB. Research advances in the breeding of medicinal source microorganisms. Journal of Liaoning University (Natural Science Edition), 2021, 48(4): 312-320 (in Chinese).
|
|
| [36] |
邵娜, 卫功元, 葛晓光, 聂敏. 紫外线γ射线复合诱变筛选S-腺苷甲硫氨酸和谷胱甘肽联产发酵菌株. 辐射研究与辐射工艺学报, 2010, 28(2): 107-113. SHAO N, WEI GY, GE XG, NIE M. Complex mutagenesis of Candida utilis by UV and γ-rays for the co-production of S-adenosyl-l-methionine and glutathione. Journal of Radiation Research and Radiation Processing, 2010, 28(2): 107-113 (in Chinese).
|
|
| [37] |
HUANG Y, GOU X, HU H, XU Q, LU Y, CHENG J. Enhanced S-adenosyl-l-methionine production in Saccharomyces cerevisiae by spaceflight culture, overexpressing methionine adenosyltransferase and optimizing cultivation. Journal of Applied Microbiology, 2012, 112(4): 683-694. DOI:10.1111/j.1365-2672.2012.05251.x
|
|
| [38] |
王卫国, 张仟伟, 赵永亮, 李瑞静, 林强, 李晓丹, 陶宜辰, 王卫. 酿酒酵母的选育及其应用研究进展. 河南工业大学学报(自然科学版), 2015, 36(6): 104-112. WANG WG, ZHANG QW, ZHAO YL, LI RJ, LIN Q, LI XD, TAO YC, WANG W. Progress on the research of Saccharomyces cerevisiae's breeding and its applications. Journal of Henan University of Technology (Natural Science Edition), 2015, 36(6): 104-112 (in Chinese).
|
|
| [39] |
叶盛. S-腺苷甲硫氨酸高产菌株的筛选、诱变选育及其培养条件优化的优化[D]. 成都: 四川师范大学硕士学位论文, 2007.
YE S. Isolation of high S-adenosylmethionine producing strain by complex mutation and optimization of culture conditions[D]. Chengdu: Master's Thesis of Sichuan Normal University, 2007 (in Chinese).
|
|
| [40] |
CHEN HL, WANG ZL, CAI HB, ZHOU C. Progress in the microbial production of S-adenosyl-l-methionine. World Journal of Microbiol Biotechnology, 2016, 32(9): 1-8.
|
|
| [41] |
CHOI ES, PARK BS, LEE SW, OH MK. Increased production of S-adenosyl-l-methionine using recombinant Saccharomyces cerevisiae sake K6. Korean Journal of Chemical Engineering, 2009, 26(1): 156-159. DOI:10.1007/s11814-009-0025-x
|
|
| [42] |
YU P, ZHU PZ. Improving the production of S-adenosyl-l-methionine in Escherichia coli by overexpressing metk. Preparative Biochemistry and Biotechnology, 2017, 47(9): 867-873. DOI:10.1080/10826068.2017.1350976
|
|
| [43] |
ZHAO WJ, SHI F, HANG BJ, HUANG L, CAI J, XU ZN. The improvement of SAM accumulation by integrating the endogenous methionine adenosyltransferase gene SAM2 in genome of the industrial Saccharomyces cerevisiae strain. Applied Biochemistry and Biotechnology, 2016, 178(6): 1263-1272. DOI:10.1007/s12010-015-1943-1
|
|
| [44] |
HAN GQ, HU XQ, QIN TY, LI Y, WANG XY. Metabolic engineering of Corynebacterium glutamicum ATCC 13032 to produce S-adenosyl-l-methionine. Enzyme and Microbial Technology, 2016, 83: 14-21. DOI:10.1016/j.enzmictec.2015.11.001
|
|
| [45] |
CHEN HL, WANG Z, WANG ZL, DOU J, ZHOU CL. Improving methionine and ATP availability by MET6 and SAM2 co-expression combined with sodium citrate feeding enhanced SAM accumulation in Saccharomyces cerevisiae. World Journal of Microbiology and Biotechnology, 2016, 32(4): 1-10.
|
|
| [46] |
HU XQ, QUINN PJ, WANG Z, HAN GQ, WANG XY. Genetic modification and bioprocess optimization for S-adenosyl-l-methionine biosynthesis. Subcellular Biochemistry, 2012, 64: 327-341.
|
|
| [47] |
QIN XL, LU JJ, ZHANG Y, WU XL, QIAO XF, WANG ZP, CHU J, QIAN JC. Engineering Pichia pastoris to improve S-adenosyl-l-methionine production using systems metabolic strategies. Biotechnology and Bioengineering, 2020, 117(5): 1436-1445. DOI:10.1002/bit.27300
|
|
| [48] |
HE JY, DENG JJ, ZHENG YH, GU J. A synergistic effect on the production of S-adenosyl-l-methionine in Pichia pastoris by knocking in of S-adenosyl-l- ethionine synthase and knocking out of cystathionine-β synthase. Journal of Biotechnology, 2006, 126(4): 519-527. DOI:10.1016/j.jbiotec.2006.05.009
|
|
| [49] |
苏慧颖, 蒙世拯, 赵欣欣, 钱江潮, 储炬, 秦秀林. 阻断消耗途径提高毕赤酵母工程菌S-腺苷甲硫氨酸产量. 微生物学通报, 2023, 50(2): 441-453. SU HY, MENG SZ, ZHAO XX, QIAN JC, CHU J, QIN XL. Blocking consumption pathway increases production of S-adenosyl-l-methionine by Pichia pastoris. Microbiology China, 2023, 50(2): 441-453 (in Chinese).
|
|
| [50] |
JIANG C, RUAN LY, WEI XT, GUO AL. Enhancement of S-adenosylmethionine production by deleting thrB gene and overexpressing SAM2 gene in Bacillus amyloliquefaciens. Biotechnology Letters, 2022, 42(11): 2293-2298.
|
|
| [51] |
RUAN LY, LI L, ZOU D, JIANG C, WEN ZY, CHEN SW, DENG Y, WEI XT. Metabolic engineering of Bacillus amyloliquefaciens for enhanced production of S-adenosylmethionine by coupling of an engineered S-adenosylmethionine pathway and the tricarboxylic acid cycle. Biotechnology for Biofuels, 2019, 12(1): 1-12. DOI:10.1186/s13068-018-1346-y
|
|
| [52] |
CHEN HL, ZHU NQ, WANG Y, GAO XX, SONG YH, ZHENG J, PENG JP, ZHANG X. Increasing glycolysis by deletion of kcs1 and arg82 improved S-adenosyl-l-methionine production in Saccharomyces cerevisiae. AMB Express, 2021, 11(1): 1-16. DOI:10.1186/s13568-020-01157-6
|
|
| [53] |
MAN ZW, GUO J, ZHANG YY, CAI Z. Regulation of intracellular ATP supply and its application in industrial biotechnology. Critical Reviews in Biotechnology, 2020, 40(8): 1151-1162. DOI:10.1080/07388551.2020.1813071
|
|
| [54] | |
|
| [55] |
XU RY, WANG DH, WANG CL, ZHANG GC, WEI GY. Improved S-adenosylmethionine and glutathione biosynthesis by heterologous expression of an ATP6 gene in Candida utilis. Journal of Basic Microbiology, 2018, 58(10): 875-882. DOI:10.1002/jobm.201800151
|
|
| [56] |
CHEN YW, TAN TW. Enhanced S-adenosylmethionine production by increasing ATP levels in baker's yeast ( Saccharomyces cerevisiae). Journal of Agricultural and Food Chemistry, 2018, 66(20): 5200-5209. DOI:10.1021/acs.jafc.8b00819
|
|
| [57] |
CHEN YW, LIAO Y, KONG WZ, WANG SH. ATP dynamic regeneration strategy for enhancing co- roduction of glutathione and S-adenosylmethionine in Escherichia coli. Biotechnology Letters, 2020, 42(12): 2581-2587. DOI:10.1007/s10529-020-02989-9
|
|
| [58] |
江林林, 吴磊, 许海霞, 黄坚丽, 张永进, 徐期, 杨勇. ATP合成菌株的构建及用于联合生产S-腺苷甲硫氨酸. 生物技术通报, 2019, 35(6): 221-226. JIANG LL, WU L, XU HX, HUANG JL, ZHANG YJ, XU Q, YANG Y. Construction of ATP synthetic strain and its application in the production of S-adenosylmethionine. Biotechnology Bulletin, 2019, 35(6): 221-226 (in Chinese).
|
|
| [59] |
HU ZC, ZHENG CM, TAO YC, WANG SN, WANG YS, LIU ZQ, ZHENG YG. Improving ATP availability by sod1 deletion with a strategy of precursor feeding enhanced S-adenosyl-l-methionine accumulation in Saccharomyces cerevisiae. Enzyme and Microbial Technology, 2022, 164: 110189.
|
|
| [60] |
LIU W, TANG DD, SHI R, LIAN JZ, HUANG L, CAI J, XU ZN. Efficient production of S-adenosyl-l- ethionine from dl-methionine in metabolic engineered Saccharomyces cerevisiae. Biotechnology and Bioengineering, 2019, 116(12): 3312-3323.
|
|
| [61] |
DONG C, SCHULTZ JC, LIU W, LIAN JZ, HUANG L, XU ZN, ZHAO HM. Identification of novel metabolic engineering targets for S-adenosyl-l-methionine production in Saccharomyces cerevisiae via genome-scale engineering. Metabolic Engineering, 2021, 66: 319-327.
|
|
| [62] |
MASUMOTO H, MATSUYAMA S. The combination of NAD+-dependent deacetylase gene deletion and the interruption of gluconeogenesis causes increased glucose metabolism in budding yeast. PLoS One, 2018, 13(3): e0194942.
|
|
| [63] |
CHEREST H, EICHLER F, de ROBICHON- ZULMAJSTER H. Genetic and regulatory aspects of methionine biosynthesis in Saccharomyces cerevisiae. Journal of Bacteriology, 1969, 97(1): 328-336.
|
|
| [64] |
CHEN HL, CHAI XQ, WANG Y, LIU J, ZHOU GH, WEI PH, SONG YH, MA LM. The multiple effects of REG1 deletion and SNF1 overexpression improved the production of S-adenosyl-l-methionine in Saccharomyces cerevisiae. Microbial Cell Factories, 2022, 21(1): 1-15.
|
|
| [65] |
GUPTA D, BHATTACHARJEE O, MANDAL D, SEN MK, DEY D, DASGUPTA A, KAZI TA, GUPTA R, SINHAROY S, ACHARYA K, CHATTOPADHYAY D, RAVICHANDIRAN V, ROY S, GHOSH D. CRISPR-Cas9 system: a new-fangled dawn in gene editing. Life Sciences, 2019, 232: 116636.
|
|
| [66] | |
|
| [67] |
OYETUNDE T, LIU D, MARTIN HG, TANG YJ. Machine learning framework for assessment of microbial factory performance. PLoS One, 2019, 14(1): e0210558.
|
|
| [68] |
KANAI M, YASUDA N, MORIMOTO T, YOSHIDA S, NISHIBORI N, MIZUNUMA M, FUJII T, IEFUJI H. Breeding of a cordycepin-resistant and adenosine kinase-deficient sake yeast strain that accumulates high levels of S-adenosylmethionine. Bioscience, Biotechnology, and Biochemistry, 2019, 83(8): 1530-1537.
|
|
| [69] |
郭肖杰, 王立言, 张翀, 邢新会. 高通量自动化微生物微液滴进化培养与筛选技术及其装备化. 生物工程学报, 2021, 37(3): 991-1003. GUO XJ, WANG LY, ZHANG C, XING XH. Technology development and instrumentation of a high-throughput and automated microbial microdroplet culture system for microbial evolution and screening. Chinese Journal of Biotechnology, 2021, 37(3): 991-1003 (in Chinese).
|
|
| [70] |
CHOI KR, JANG WD, YANG D, CHO JS, PARK D, LEE SY. Systems metabolic engineering strategies: integrating systems and synthetic biology with metabolic engineering. Trends in Biotechnology, 2019, 37(8): 817-837.
|
|
| [71] |
江丽红, 董昌, 黄磊, 徐志南, 连佳长. 酿酒酵母代谢工程技术. 生物工程学报, 2021, 37(5): 1578-1602. JIANG LH, DONG C, HUANG L, XU ZN, LIAN JZ. Metabolic engineering tools for Saccharomyces cerevisiae. Chinese Journal of Biotechnology, 2021, 37(5): 1578-1602 (in Chinese).
|
|
| [72] |
HAYAKAWA K, MATSUDA F, SHIMIZU H. Metabolome analysis of Saccharomyces cerevisiae and optimization of culture medium for S-adenosyl-l- methionine production. AMB Express, 2016, 6(1): 1-8.
|
|
| [73] | |
|
| [74] |
UMEYAMA T, OKADA S, ITO T. Synthetic gene circuit-mediated monitoring of endogenous metabolites: identification of GAL11 as a novel multicopy enhancer of S-adenosylmethionine level in yeast. ACS Synthetic Biology, 2013, 2(8): 425-430.
|
|
| [75] |
CHEN YW, ZHENG HJ, YANG JJ, CAO YT, ZHOU HY. Development of a synthetic transcription factor-based sadenosylmethionine biosensor in Saccharomyces cerevisiae. Biotechnology Letters, 2022, 45(2): 255-262.
|
|
 2023, Vol. 39
2023, Vol. 39