双酶级联催化L-色氨酸合成靛蓝
罗诗琪
,
魏婉清
,
吴静
,
宋伟
,
胡贵鹏
,
刘立明
生物工程学报  2024, Vol. 40 2024, Vol. 40 Issue (8): 2444-2456 Issue (8): 2444-2456 |

靛蓝作为一种水溶性非偶氮类着色剂,是最常见、最古老的染料之一,广泛应用于纺织品染色[1-5]、食品、制药[6-13]、化学传感器[14-15]和半导体材料[16-17]等领域。目前全球对靛蓝的年需求量超过8万t,主要来源于:(1) 化学合成。苯基甘氨酸在氨基钠的存在下与NaOH和KOH融合,然后氧化生成靛蓝,收率为80%−83%;(2) 植物提取。从蓼科植物、龙葵科植物和菜豆科植物提取靛青或者靛苷[18]。化学工艺因大量使用有机溶剂和强碱,会造成严重的环境危害;而提取工艺因野生资源有限加上提取工艺复杂,限制了植物靛蓝的发展[19]。生物法因绿色、高效、环保等优点逐渐成为靛蓝生产的最有前途的方法[20-21]。能催化合成靛蓝的酶[22]有非血红素铁加氧酶[23]、含血红素加氧酶[24-25]和黄素依赖性单加氧酶(flavin-dependent monooxygenase, FMO)[26]。其中,非血红素铁加氧酶代表是萘双加氧酶(naphthalene dioxygenase, NDO),如来源于假单胞菌(Pseudomonas sp.) J26菌株的NDO在20 h内能够从2.5 mmol/L吲哚中生成138.1 μmol/L靛蓝[27]。含血红素加氧酶中能够羟基化吲哚的一个是依赖于氧化还原伴侣和昂贵辅因子的P450酶[28-30],如来源于巨大芽孢杆菌(Bacillus megaterium)的P450BM3与葡萄糖脱氢酶的共表达使5 mmol/L吲哚产生了2.9 mmol/L的靛蓝;另一个是依赖于H2O2作为氧供体和电子受体的非特异性过氧合酶(unspecific peroxygenases, UPO),能将一系列吲哚及其衍生物转化为相应的靛蓝或靛蓝衍生物[31]。黄素单加氧酶以NADPH为辅因子将色氨酸酶(tryptophanase)的N端和黄素单加氧酶的C端进行融合表达,能够将2 g/L的L-色氨酸完全转化为1.7 g/L靛蓝[32],这也是目前报道生物合成靛蓝产量最高的酶。尽管已有不同来源的酶能够催化吲哚生成靛蓝,但路径中关键限速酶活性低和中间体吲哚毒性问题限制着靛蓝的工业规模生产。
为此,本研究通过将来源于大肠杆菌(Escherichia coli)的色氨酸酶(tryptophanase, EcTnaA)和来源于Methylophaga aminisulfidivorans的黄素依赖性单加氧酶(flavin-dependent monooxygenase, MaFMO)整合到E. coli中,构建L-色氨酸生成靛蓝的双酶级联路径,减少中间体吲哚的大量积累。通过对限速酶MaFMO进行蛋白质工程改造以提高酶活并对发酵条件进行优化。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒和引物| Strains and plasmids | Relevant characteristics | Sources |
| Strains | ||
| Escherichia coli MG1655 (K12) | Used to extract EcTnaA | Lab store |
| E. coli BL21(DE3) | Expression host | Lab store |
| E. coli BL21(DE3)-EcTnaA | E. coli BL21(DE3) containing pET28a-EcTnaA | This study |
| E. coli BL21(DE3)-MaFMO | E. coli BL21(DE3) containing pET28a-MaFMO | This study |
| E. coli EM-IND01 (BL21-EcTnaA-MaFMO) | E. coli BL21(DE3) containing pET28a-EcTnaA-MaFMO | This study |
| E. coli EM-IND02 (BL21-EcTnaA-MaFMOD197E) | E. coli BL21(DE3) containing pET28a-EcTnaA-MaFMOD197E | This study |
| Plasmids | ||
| pET28a-EcTnaA | KanR, His tag, T7 promoter | This study |
| pET28a-EcTnaA | KanR, His tag, T7 promoter | This study |
| pET28a-EcTnaA-MaFMO | KanR, His tag, T7 promoter | This study |
| Primers | Primer sequence (5′→3′) | Size (bp) |
| EcTnaA-F | TAACTTTAAGAAGGAGATATACCATGGGCAGCAGCCATCATCATCATCAT | 50 |
| EcTnaA-R | CCAAGGGGTTATGCTAGTTATTGCTCAGCGGTGGCAGCAGCCAACTCAGC | 50 |
| MaFMO-F | GGATCCGCGACCCATTTGCTGTCCA | 25 |
| MaFMO-R | AAGCTTGCGGCCGCACTCGAGCACC | 25 |
| MaFMO-D197X-F | GCGCTGGAATTTAAANNKAAAACAGTATTATTAGTCGGCAGCTCG | 45 |
| MaFMO-D197X-R | AATAATACTGTTTTMNNTTTAAATTCCAGCGCATCGCGG | 39 |
分子生物学工具酶、SDS-PAGE Protein Marker、质粒提取试剂盒和胶回收试剂盒等分子生物学试剂购自TaKaRa公司;L-色氨酸、吲哚、靛蓝等标品购自阿拉丁生化科技股份有限公司;异丙基-β-d-硫代半乳糖(isopropyl β-d-thiogalactoside, IPTG)、硫酸卡那霉素(kanamycin, Kan)购自生工生物工程(上海)股份有限公司;供实验所用试剂盒、一步同源重组酶购自南京诺唯赞生物科技股份有限公司;PCR引物由苏州金唯智生物科技有限公司合成;其他试剂均购自国药集团化学试剂有限公司。5 L全自动发酵罐购自迪比尔生物工程(上海)有限公司。
1.1.3 培养基LB培养基(g/L):酵母粉5,蛋白胨10,氯化钠10 (固体培养基加入2%琼脂粉)。
发酵培养基(g/L):酵母粉5,蛋白胨10,氯化钠10,L-色氨酸1.5。
TB培养基(g/L):甘油4,胰蛋白胨12,酵母粉24,KH2PO4 2.31,K2HPO4 12.54,用于重组菌株全细胞培养。
1.2 方法 1.2.1 重组菌株构建表达质粒的构建采用标准分子克隆操作和吉布森组装法,其中一步同源重组法是根据目的片段与表达载体之间具有相同序列连接的方法。来源于Methylophaga aminisulfidivorans的FMO基因在天霖生物科技无锡有限公司进行密码子优化和合成。以基因组E. coli MG1655为模板,用引物对EcTnaA-F和EcTnaA-R扩增EcTnaA,并以引物对MaFMO-F和MaFMO-R剪切pET-28a-MaFMO质粒,最后将扩增得到的EcTnaA片段和剪切了的pET-28a-MaFMO质粒经同源重组连接获得重组质粒pET28a- EcTnaA-MaFMO,将构建完成的质粒导入E. coli BL21(DE3)后获得重组菌株EM-IND01。
1.2.2 培养方法平板活化:蘸取保藏于甘油管中的菌液,在LB固体平板上进行三区划线,37 ℃恒温培养12 h。
种子培养:挑取三区划线的单菌落移入装有含2 mmol/L硫酸卡那霉素的50 mL LB培养基的三角瓶中,在pH为7.0的条件下37 ℃、220 r/min培养8 h后再传代一次。
摇瓶发酵:在发酵培养基中加入2 mmol/L硫酸卡那霉素,以1%接种量将种子培养液接种于装有100 mL发酵培养基的250 mL三角瓶中,在pH为7.0的条件下37 ℃、220 r/min培养至OD600为2.5,后转为30 ℃培养45 h。
发酵罐发酵:采用5 L全自动搅拌发酵罐进行发酵,发酵初始装液量为2 L,加入2 mmol/L硫酸卡那霉素,并将种子液以1% (体积分数)接种量接种至发酵培养基中,发酵温度初始为37 ℃,培养至OD600为2.5后转为30 ℃发酵。维持初始转速220 r/min,通入100%无菌干燥空气进行发酵,过程中通过调整搅拌转速及通气速率维持溶解氧浓度为30%−40%,通过流加33.3%浓度的盐酸将pH维持在7.0−7.5之间,发酵至48 h结束。
全细胞培养方法:在TB培养基中加入2 mmol/L硫酸卡那霉素,以2%接种量将种子培养液接种于装有150 mL发酵培养基的500 mL三角瓶中,在37 ℃、220 r/min条件下培养至OD600为0.6−0.8,添加2 mmol/L IPTG诱导剂后转为16 ℃培养18 h。
1.2.3 酶活测定EcTnaA比酶活测定方法:1 mL反应体系包含1 mmol/L L-色氨酸、50 mmol/L磷酸钾缓冲液(pH 7.4)及10 μmol/L EcTnaA纯酶。在30 ℃下反应30 min后用HPLC检测吲哚的生成量,计算酶活。催化反应温度控制在30 ℃,每分钟生成1 μmol/L吲哚所需的酶量为1个酶活力单位。
MaFMO比酶活测定方法:1 mL反应体系包含1 mmol/L吲哚、50 mmol/L磷酸钾缓冲液(pH 9.0)、1 mmol/L NADPH及10 μmol/L MaFMO纯酶。在30 ℃下反应30 min后用HPLC检测靛蓝的生成量,计算酶活。催化反应温度控制在30 ℃,每分钟生成1 μmol/L的靛蓝所需酶量为1个酶活力单位。
1.2.4 动力学参数测定EcTnaA动力学参数测定方法:1 mL反应体系包含50 mmol/L磷酸钾缓冲液(pH 7.4)、10 μmol/L EcTnaA纯酶和浓度分别为0.5、1、2、5、7、10、15、20 mmol/L的L-色氨酸。在30 ℃下反应30 min后用HPLC检测吲哚的生成量。使用Origin 2021软件进行非线性曲线拟合。
MaFMO动力学参数测定方法:1 mL反应体系包含50 mmol/L磷酸钾缓冲液(pH 9.0)、10 μmol/L MaFMO纯酶、1 mmol/L NADPH和浓度分别为0.1、0.5、1.0、1.5、2.0、3.0、4.0、5.0 mmol/L的吲哚,在30 ℃下反应30 min后用HPLC检测靛蓝生成量。使用Origin 2021软件进行非线性曲线拟合。
1.2.5 分析检测方法细胞浓度检测:取细胞培养液,稀释至OD600在0.2−0.8之间,通过紫外分光光度计检测其在波长600 nm处的吸光度。
L-色氨酸浓度检测:具体方法参照已报道的研究[33]。
吲哚浓度检测:使用Agilent C18色谱柱(250 mm×4.6 mm, 5 μm, Agilent)。流动相配比为水与甲醇按3:7 (体积比)混合;紫外检测器检测波长为260 nm;进样量10 μL;流速0.5 mL/min;柱温35 ℃。
靛蓝浓度检测:使用Agilent C18色谱柱(250 mm×4.6 mm, 5 μm, Agilent)。流动相配比为水与甲醇按3:7 (体积比)混合;紫外检测器检测波长为620 nm;进样量10 μL;流速0.5 mL/min;柱温35 ℃。
2 结果与分析 2.1 L-色氨酸合成靛蓝的级联路径设计和体外构建从L-色氨酸出发合成靛蓝的生物催化路径如图 1所示。首先,色氨酸酶(TnaA, EC 4.1.99.1)将L-色氨酸分解产生吲哚、丙酮酸和氨;所生成的吲哚再经氧化酶(FMO, EC1.14.13.8)氧化生成3-羟基吲哚,接着3-羟基吲哚发生自发的烯醇酮式互变异构为3-吲哚酮,最后2分子的3-吲哚酮发生氧化缩合生成靛蓝并释放1分子水。
|
| 图 1 双酶级联催化L-色氨酸合成靛蓝路径 Fig. 1 Dual-enzyme cascade pathway for synthesis of indigo from L-tryptophan. |
| |
由于E. coli来源的TnaA具有优异的催化活性,因此我们选择其催化L-色氨酸分解产生吲哚、丙酮酸和氨[34]。能够催化吲哚氧化生成3-羟基吲哚的酶如表 3所示,其中来源于炭团菌(Hypoxylon sp.) EC38的非特异性过加氧酶(Hypoxylon sp. EC38 unspecific peroxygenases, HspUPO)转化率最高,为98%,但是以H2O2作为氧源的耐受性导致吲哚羟基化产物的收率仅为51 mg/L[35];而来源于Methylophaga aminisulfidivorans的黄素依赖性单加氧酶(MaFMO)对吲哚的羟基化产物转化率为85%,产量最高达到1 700 mg/L[32],因此选择MaFMO催化吲哚生成3-羟基吲哚。
| Enzymes | Organism | Oxygen source | Cofactor | kcat/Km (L/(mmol·min)) | Yield (mg/L) | Conversion (%) | References |
| HspUPOs | Hypoxylon sp. EC38 | H2O2 | − | 900.00 | 51.00 | 98.00 | [35] |
| FMO | Methylophaga aminisulfidivorans | O2 | NADPH/FAD | − | 1 700.00 | 85.00 | [32] |
| NgbA15C/H64D/F49Y | Human | H2O2 | − | 42.84 | 215.05 | 82.00 | [25] |
| P450 BM3F87A | Bacillus megaterium | H2O2 | NADPH | − | 760.60 | 68.50 | [24] |
| NDO | Comamonas sp. MQ | O2 | − | − | 205.00 | 68.30 | [23] |
| NbF43Y/H64D | Sperm whale | H2O2 | − | 6.60 | 47.21 | 54.00 | [36] |
| Indole oxygenase | Cupriavidus sp. SHE | O2 | NADH/FAD | − | 307.00 | 30.70 | [37] |
| FMOM185L/V402A | Corynebacterium glutamicum | O2 | NADPH/FAD | 36.36 | 800.00 | 40.00 | [38] |
| XiaI | Streptomyces sp. SCSIO 02999 | H2O2 | − | − | 26.00 | − | [39] |
| P450camF87W/Y96F | Pseudomonas putida | O2 | NADH | − | 20.00 | − | [40] |
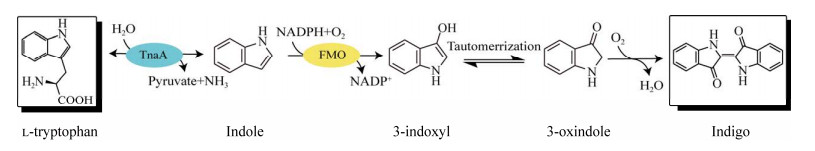
为了验证上述路径的可行性,将EcTnaA和MaFMO于E. coli BL21(DE3)中进行过表达,破碎细胞并纯化得到纯酶。将重组蛋白以不同的摩尔比添加在含有1 mmol/L L-色氨酸和1 mmol/L NADPH的1 mL反应体系中,在30 ℃摇床中反应30 min。最终生成的蓝色沉淀物质,经DMSO萃取后采用HPLC和液相色谱质谱联用(liquid chromotography-mass spectrometry, LC-MS)鉴定靛蓝的生成(图 2A),通过与靛蓝标准品的m/z对比,可以证明蓝色沉淀物质为目标产物靛蓝(图 2B)。当两种酶的摩尔比为1:1 (均为10 μmol/L)时,靛蓝的生产速率为3.01 mg/(L·min)。固定EcTnaA的摩尔添加量不变,将MaFMO的摩尔量增加1−3倍,生产速率相比于摩尔比为1:1时分别提高了1.07、1.25、1.34倍;固定MaFMO摩尔量不变,将EcTnaA摩尔量增加1−3倍,生产速率相比于1:1时分别提高了1.01、1.03、1.09倍(图 2C)。通过比较反应速率的提升倍数,可以看出MaFMO是级联反应的关键限速酶。
|
| 图 2 级联路径的体外构建与验证 Fig. 2 Construction and validation of cascade pathway in vitro. A: Analysis of the indigo with HPLC. B: Analysis of the products indigo with LC-MS. C: The effect of molar ratio of double enzyme addition on the synthesis rate of indigo. A:使用HPLC验证产物靛蓝的生成. B:使用LC-MS验证产物靛蓝的生成. C:双酶添加摩尔比对靛蓝生产速率的影响 |
| |
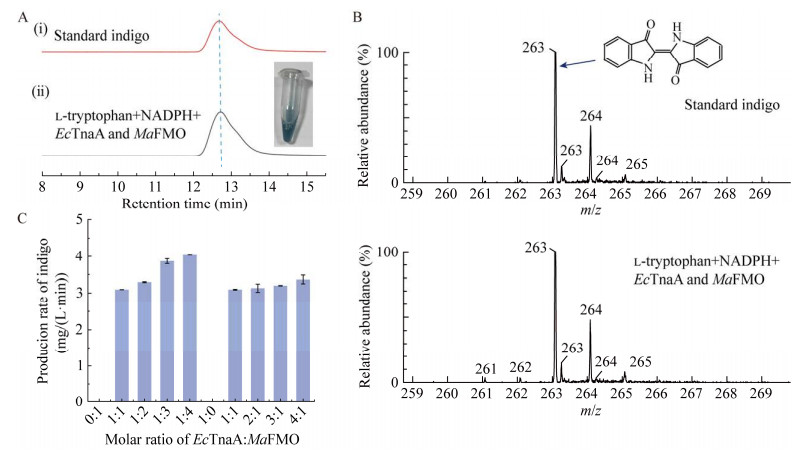
将EcTnaA和MaFMO基因克隆至pET-28a(+)质粒,然后转化到E. coli BL21(DE3)菌株中,得到菌株EM-IND01 (图 3A)。通过全细胞蛋白电泳考察重组大肠杆菌的蛋白表达情况,验证目标蛋白是否能够正常表达。SDS-PAGE胶染色后可清晰地观察到两条蛋白条带,与EcTnaA (53 kDa)和MaFMO (55 kDa)分子量大小相符(图 3B),说明EcTnaA和MaFMO在菌株EM-IND01中均实现了可溶性表达。
|
| 图 3 建高产靛蓝的菌株EM-IND01 Fig. 3 Constructing a high-yield indigo producing strain EM-IND01. A: Construction of the plasmid used for EM-IND01. B: The SDS-PAGE analysis of the EM-IND01 strain from cell-free extracts. C: Influence of different substrate concentrations on indigo production by the EM-IND01 strain. All the results are the average of three parallel experimental results. A:EM-IND01质粒的构建. B:来自菌株EM-IND01细胞提取物的SDS-PAGE分析图. C:不同L-色氨酸浓度对菌株EM-IND01转化生产靛蓝的影响. 所有结果均为3次平行实验结果的平均值 |
| |
为了验证菌株EM-IND01能否将L-色氨酸转化生成靛蓝,在100 mL LB培养基中研究不同L-色氨酸浓度的转化效率,结果如图 3C所示。当L-色氨酸浓度从0.5 g/L增加到2.0 g/L时,靛蓝产量从(114.10±12.49) mg/L增加至(328.59±9.68) mg/L,转化率从(70.54±8.00)%下降至(50.79±1.55)%;然而,当L-色氨酸浓度从2.0 g/L增加至3.0 g/L时,靛蓝产量从(328.59±9.68) mg/L降低至(201.99±3.92) mg/L,同时转化率进一步降至(20.81±0.42)%。其原因可能是中间体吲哚的积累,当L-色氨酸浓度为3.0 g/L时,吲哚积累量为(6.33±0.02) mg/L。此时,转化液中含有(258.23±20.90) mg/L的L-色氨酸,表明高底物浓度会抑制反应的进行。有研究表明吲哚浓度超过5 mmol/L就会抑制TnaA活性和L-色氨酸转运过程[41]。菌株EM-IND01中EcTnaA和MaFMO的比酶活分别为(1.61±0.19) U/g和(1.41±0.13) U/g,两者之间比值为1.14:1,EcTnaA和MaFMO的kcat/Km为(0.91±0.16) L/(mmol·s)和(0.82±0.22) L/(mmol·s),比值约为1.11:1 (表 4)。上述结果同样证明了MaFMO是L-色氨酸转化生成靛蓝的关键限速酶。
| Ezymes | Specific enzyme activity (U/g) | Km (mmol/L) | kcat (s–1) | kcat/Km (L/(mmol·s)) |
| EcTnaA | 1.61±0.19 | 0.83±0.12 | 0.67±0.02 | 0.91±0.16 |
| MaFMO | 1.41±0.13 | 0.84±0.04 | 0.69±0.21 | 0.82±0.22 |
| MaFMOD197E | 3.33±0.54 | 1.03±0.02 | 1.14±0.47 | 1.10±0.44 |
| U: The amount of enzyme required to produce 1 μmol product per minute. All the tests were performed twice. | ||||
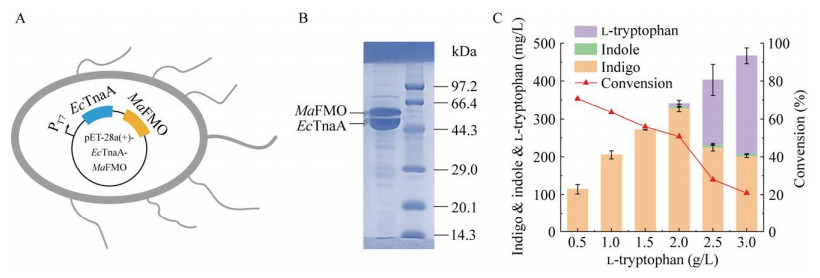
为了能够从大量的突变体文库中快速筛选出活性提高的MaFMO突变体,基于靛蓝在620 nm波长处具有吸收峰且存在显色反应构建了一个高通量筛选方法(图 4):具有MaFMO活性的菌株在LB固体平板上的菌落显蓝色。于是,将突变文库转化至BL21菌株中,涂布于LB固体平板,从平板上挑取已显色的菌落,接种于含有LB液体培养基的96孔板中于37 ℃培养24 h后,将96孔板离心,获得靛蓝沉淀,然后用等体积的DMSO溶解,于620 nm波长处进行初筛。最后,在100 mL LB培养基中添加1.5 g/L的L-色氨酸培养48 h,使用HPLC对阳性菌株进行复筛,就能够快速且准确地筛选出活性高于野生型的MaFMO突变体。
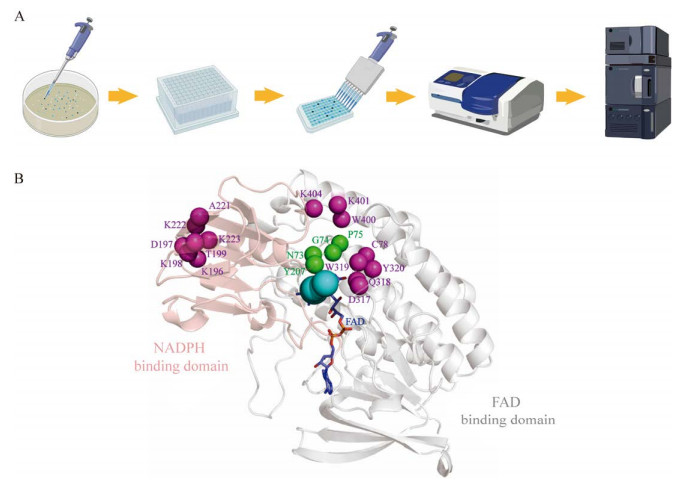
|
| 图 4 MaFMO的蛋白质工程改造 Fig. 4 Protein engineering modifications of MaFMO. A: Schematic diagram of high-throughput screening method for indigo. B: Display of mutation sites. The pink model represents the NADPH binding domain; the graymodel represents the FAD binding domain; the magenta spheres represent the remaining mutation sites identified through sequence alignment; the green spheres represent the amino acids Y207, N73, G74, P75, and the magenta spheres represent the amino acids C78, D317, Q318, W319, Y320, which form the active pocket; the light blue spheres represent the substrate channel; the dark blue sphere and stick model represent the cofactor FAD. A:靛蓝的高通量筛选方法示意图. B:突变位点展示图. 粉色模型表示NADPH结合域;灰色模型表示FAD结合域;玫红色球体表示通过序列比对后剩余的突变位点;绿色球体表示的Y207、N73、G74、P75氨基酸和玫红色球体表示的C78、D317、Q318、W319、Y320氨基酸构成活性口袋;浅蓝色球体表示底物通道;深蓝色球棍模型表示辅因子FAD |
| |
以野生型pET28a-EcTnaA-MaFMO质粒为模板,使用高通量筛选方法进行突变。通过序列比对,去除非保守氨基酸后对底物通道和底物结合腔周围5 Å范围内的氨基酸残基(W400、K401、K404、C78、D317、Q318、W319、Y320)以及NADPH结合域的氨基酸残基(K196、D197、K198、T199、A221、K222、K223)进行NNK定点饱和突变,最终获得一个有益突变体D197E。突变体MaFMOD197E的比酶活和kcat/Km值分别是野生型的2.36倍和1.34倍(表 4)。将菌株EM-IND01中的MaFMO替换成突变体MaFMOD197E,获得菌株EM-IND02。菌株EM-IND02能催化1.5 g/L L-色氨酸生成(540.44±3.44) mg/L靛蓝,转化率为(49.6±0.32)%;野生型产量为(495.83±3.89) mg/L,转化率为(45.49±0.36)%,突变体MaFMOD197E较野生型产量提高了44.63 mg/L,转化率提高了野生型的9.00%。
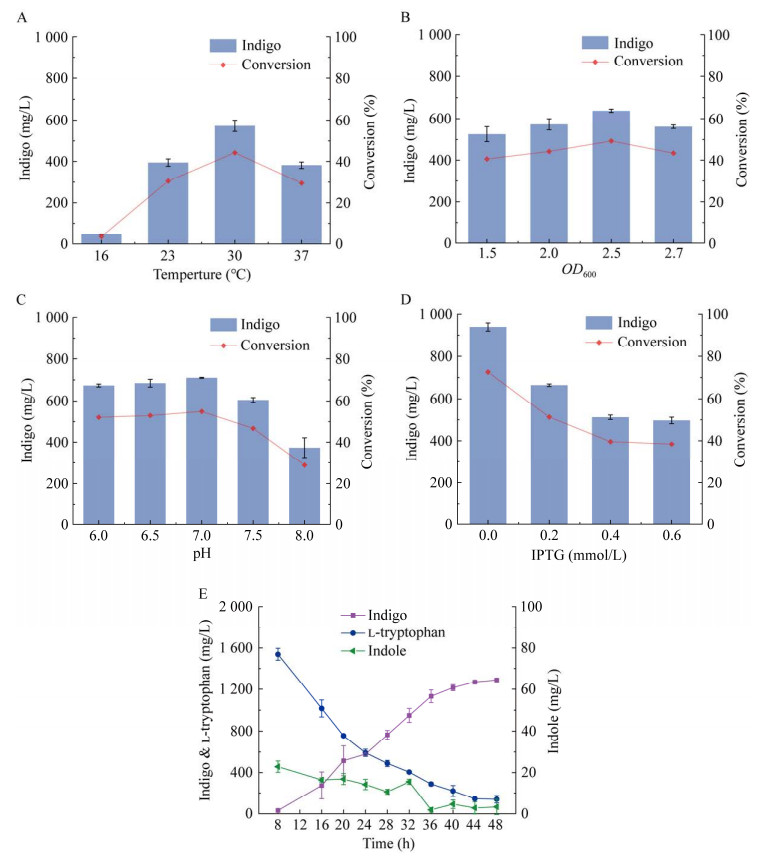
2.4 双酶级联一锅法生产靛蓝为了进一步提升靛蓝产量,在1.2.2摇瓶发酵的操作基础上对菌株EM-IND02的发酵温度(16、23、30、37 ℃)、种子液OD600 (1.5、2.0、2.5、2.7)、反应液pH (6.0、6.5、7.0、7.5、8.0)和诱导剂IPTG的浓度(0.0、0.2、0.4、0.6 mmol/L)进行分批优化。首先,当摇瓶发酵种子液OD600为2.0,反应液pH为7.5,添加0.2 mmol/L IPTG时,考察不同发酵温度对菌株EM-IND02生产靛蓝的影响,结果如图 5A所示,当反应温度为30 ℃时可以获得(572.77±26.67) mg/L靛蓝。在此基础上对种子液OD600进行优化,结果如图 5B所示,当种子液OD600为2.5时靛蓝产量进一步提高到(637.24±7.48) mg/L。然后对反应液pH进行优化,结果如图 5C所示,当反应液pH为7.0时靛蓝产量再次提高到(710.15±2.09) mg/L。最后对诱导剂IPTG浓度进行了优化,结果如图 5D所示,未添加IPTG时,靛蓝产量提升至(939.01±19.50) mg/L,此时转化率为72.60%。为了进一步考察菌株EM-IND02的工业化应用水平,在5 L发酵罐中进行实验。结果如图 5E所示,反应48 h后,靛蓝产量为(1 288.59±7.50) mg/L,产率为0.86 mg/mg色氨酸,生产强度为26.85 mg/(L·h)。
|
| 图 5 菌株EM-IND02转化生产靛蓝的条件优化 Fig. 5 Optimization of indigo production conditions with strain EM-IND02. A: Effect of temperature on indigo concentration. B: Effect of OD600 on indigo concentration. C: Effect of buffer pH on indigo concentration. D: Effect of IPTG concentration on indigo concentration. E: Conversion L-tryptophan production indigo experiments of EM-IND02 strain at 5 L fermentation vessel. A:不同反应温度对靛蓝产量的影响. B:不同OD600值对靛蓝产量的影响. C:不同初始pH对靛蓝产量的影响. D:不同IPTG浓度对靛蓝产量的影响. E:菌株EM-IND02在5 L发酵罐中转化L-色氨酸生产靛蓝 |
| |
在以L-色氨酸为底物合成靛蓝的级联路径中,限速酶的存在限制了产物靛蓝的高效合成。为了提升限速酶的活性,将限速酶进行蛋白质工程改造是一个有潜力的策略,如对Methylophaga sp.来源的mFMO的活性空腔进行饱和突变,得到突变体C78I的kcat虽然比野生型提升了1.5倍,但kcat/Km反而降低了24.70%,最终靛蓝产量仅为300.00 mg/L[26]。本研究对限速酶MaFMO进行了定点饱和突变,以进一步提高限速酶MaFMO活性。与野生型相比,有益突变体D197E的kcat提升了1.65倍,kcat/Km提高了1.34倍,最终靛蓝产量提高到(1 288.59±7.50) mg/L。
除了提升限速酶活性外,还可通过消除中间体吲哚的积累来增加靛蓝产量。已有文献报道超过5 mmol/L吲哚就会抑制TnaA活性和L-色氨酸转运过程[41],从而影响级联路径的合成效率。但若以吲哚为底物合成靛蓝,这会限制靛蓝的大批量合成。因此可通过调控酶表达水平来控制中间体的积累。本研究将EcTnaA和MaFMO构建到同一菌株中进行过表达,提高了EcTnaA的表达和稳定性,从而提高靛蓝产量。
目前,本研究主要集中在对限速酶的改造以及发酵条件的优化,从而提升反应效率。为了进一步提高整体路径的合成效率,在后续研究中将对路径中间体吲哚的积累进行优化,进而减少对路径酶EcTnaA的限制,提升整体路径的转化效率。此外,为了实现更低成本的生产,还可从葡萄糖出发,将葡萄糖发酵生成L-色氨酸,然后级联EcTnaA和MaFMO,实现以葡萄糖为底物大量合成靛蓝。
| [1] |
VÁZQUEZ-ORTEGA F, LAGUNES I, TRIGOS Á. Cosmetic dyes as potential photosensitizers of singlet oxygen generation[J]. Dyes and Pigments, 2020, 176: 108248. DOI:10.1016/j.dyepig.2020.108248
|
| [2] |
SADAUSKAS M, STATKEVIČIŪTĖ R, VAITEKŪNAS J, PETKEVIČIUS V, ČASAITĖ V, GASPARAVIČIŪTĖ R, MEŠKYS R. Enzymatic synthesis of novel water-soluble indigoid compounds[J]. Dyes and Pigments, 2020, 173: 107882. DOI:10.1016/j.dyepig.2019.107882
|
| [3] |
SAIKHAO L, SETTHAYANOND J, KARPKIRD T, BECHTOLD T, SUWANRUJI P. Green reducing agents for indigo dyeing on cotton fabrics[J]. Journal of Cleaner Production, 2018, 197: 106-113. DOI:10.1016/j.jclepro.2018.06.199
|
| [4] |
COSTA AFDS, de AMORIM JDP, ALMEIDA FCG, de LIMA ID, de PAIVA SC, ROCHA MAV, VINHAS GM, SARUBBO LA. Dyeing of bacterial cellulose films using plant-based natural dyes[J]. International Journal of Biological Macromolecules, 2019, 121: 580-587. DOI:10.1016/j.ijbiomac.2018.10.066
|
| [5] |
徐枫, 姜利利, 王永华, 王祥荣. 靛蓝染料染色技术研究进展[J]. 印染助剂, 2023, 40(5): 1-6. XU F, JIANG LL, WANG YH, WANG XR. Research progress of indigo dyeing technology[J]. Textile Auxiliaries, 2023, 40(5): 1-6 (in Chinese). DOI:10.3969/j.issn.1004-0439.2023.05.001 |
| [6] |
PULIT-PROCIAK J, CHWASTOWSKI J, KUCHARSKI A, BANACH M. Functionalization of textiles with silver and zinc oxide nanoparticles[J]. Applied Surface Science, 2016, 385: 543-553. DOI:10.1016/j.apsusc.2016.05.167
|
| [7] |
HEO BG, PARK YJ, PARK YS, BAE JH, CHO JY, PARK K, JASTRZEBSKI Z, GORINSTEIN S. Anticancer and antioxidant effects of extracts from different parts of indigo plant[J]. Industrial Crops and Products, 2014, 56: 9-16. DOI:10.1016/j.indcrop.2014.02.023
|
| [8] |
YOU WC, HSIEH CC, HUANG JT. Effect of extracts from indigowood root (Isatis indigotica Fort.) on immune responses in radiation-induced mucositis[J]. Journal of Alternative and Complementary Medicine, 2009, 15(7): 771-778. DOI:10.1089/acm.2008.0322
|
| [9] |
MARYAN AS, MONTAZER M, HARIFI T. Synthesis of nano silver on cellulosic denim fabric producing yellow colored garment with antibacterial properties[J]. Carbohydrate Polymers, 2015, 115: 568-574. DOI:10.1016/j.carbpol.2014.08.100
|
| [10] |
EISENBRAND G, HIPPE F, JAKOBS S, MUEHLBEYER S. Molecular mechanisms of indirubin and its derivatives: novel anticancer molecules with their origin in traditional Chinese phytomedicine[J]. Journal of Cancer Research and Clinical Oncology, 2004, 130(11): 627-635. DOI:10.1007/s00432-004-0579-2
|
| [11] |
LIN YK, LEU YL, YANG SH, CHEN HW, WANG CT, PANG JH S. Anti-psoriatic effects of indigo naturalis on the proliferation and differentiation of keratinocytes with indirubin as the active component[J]. Journal of Dermatological Science, 2009, 54(3): 168-174. DOI:10.1016/j.jdermsci.2009.02.007
|
| [12] |
HOESSEL R, LECLERC S, ENDICOTT JA, NOBEL MEM, LAWRIE A, TUNNAH P, LEOST M, DAMIENS E, MARIE D, MARKO D, NIEDERBERGER E, TANG WC, EISENBRAND G, MEIJER L. Indirubin, the active constituent of a Chinese antileukaemia medicine, inhibits cyclin-dependent kinases[J]. Nature Cell Biology, 1999, 1(1): 60-67. DOI:10.1038/9035
|
| [13] |
KAMELAMELA N, ZALESNE M, MORIMOTO J, ROBBAT A, WOLFE BE. Indigo- and indirubin-producing strains of Proteus and Psychrobacter are associated with purple rind defect in a surface-ripened cheese[J]. Food Microbiology, 2018, 76: 543-552. DOI:10.1016/j.fm.2018.07.011
|
| [14] |
PINHEIRO D, PINEIRO M, PINA J, BRANDÃO P, GALVÃO AM, SEIXAS de MELO JS. Tryptanthrin from indigo: synthesis, excited state deactivation routes and efficient singlet oxygen sensitization[J]. Dyes and Pigments, 2020, 175: 108125. DOI:10.1016/j.dyepig.2019.108125
|
| [15] |
KOLACZKOWSKI MA, LIU Y. Functional organic semiconductors based on bay-annulated indigo (BAI)[J]. Chemical Record, 2019, 19(6): 1062-1077. DOI:10.1002/tcr.201800159
|
| [16] |
KLIMOVICH IV, LESHANSKAYA LI, TROYANOV SI, ANOKHIN DV, NOVIKOV DV, PIRYAZEV AA, IVANOV DA, DREMOVA NN, TROSHIN PA. Design of indigo derivatives as environment-friendly organic semiconductors for sustainable organic electronics[J]. Journal of Materials Chemistry C, 2014, 2(36): 7621-7631. DOI:10.1039/C4TC00550C
|
| [17] |
NGAI JHL, LEUNG LM, SO SK, LEE HKH. Organic soluble indigoids derived from 3-hydroxybenzaldehyde for N-type organic field-effect transistor (OFET) applications[J]. Organic Electronics, 2016, 32: 258-266. DOI:10.1016/j.orgel.2016.02.035
|
| [18] |
LOPES HFS, TU ZH, SUMI H, YUMOTO I. Analysis of bacterial flora of indigo fermentation fluids utilizing composted indigo leaves (sukumo) and indigo extracted from plants (Ryukyu-ai and Indian indigo)[J]. Journal of Bioscience and Bioengineering, 2021, 132(3): 279-286. DOI:10.1016/j.jbiosc.2021.05.004
|
| [19] |
RAVELOMBOLA W, TROSTLE C, CASON J, ALE S, MANLEY A, PHAM H. Current status of the genetic and agronomic of industrial indigo Indigofera sp.[J]. Euphytica, 2023, 219(12): 128. DOI:10.1007/s10681-023-03256-4
|
| [20] |
CHANDEL N, SINGH BB, DUREJA C, YANG YH, BHATIA SK. Indigo production goes green: a review on opportunities and challenges of fermentative production[J]. World Journal of Microbiology and Biotechnology, 2024, 40(2): 62. DOI:10.1007/s11274-023-03871-2
|
| [21] |
李蓉蓉, 马文浩, 肖莹. 靛蓝生物合成酶的研究进展[J]. 生物资源, 2023, 45(4): 328-340. LI RR, MA WH, XIAO Y. Research advances in enzymes involved in indigo biosynthesis[J]. Biotic Resources, 2023, 45(4): 328-340 (in Chinese). |
| [22] |
FABARA AN, FRAAIJE MW. An overview of microbial indigo-forming enzymes[J]. Applied Microbiology and Biotechnology, 2020, 104(3): 925-933. DOI:10.1007/s00253-019-10292-5
|
| [23] |
ZHANG XW, QU YY, MA Q, ZHOU H, LI XL, KONG CL, ZHOU JT. Cloning and expression of naphthalene dioxygenase genes from Comamonas sp. MQ for indigoids production[J]. Process Biochemistry, 2013, 48(4): 581-587. DOI:10.1016/j.procbio.2013.02.008
|
| [24] |
LU Y, MEI LH. Co-expression of P450 BM3 and glucose dehydrogenase by recombinant Escherichia coli and its application in an NADPH-dependent indigo production system[J]. Journal of Industrial Microbiology & Biotechnology, 2007, 34(3): 247-253.
|
| [25] |
CHEN L, XU JK, LI LZ, GAO SQ, WEN GB, LIN YW. Design and engineering of neuroglobin to catalyze the synthesis of indigo and derivatives for textile dyeing[J]. Molecular Systems Design & Engineering, 2022, 7(3): 239-247.
|
| [26] |
LONČAR N, van BEEK HL, FRAAIJE MW. Structure-based redesign of a self-sufficient flavin-containing monooxygenase towards indigo production[J]. International Journal of Molecular Sciences, 2019, 20(24): 6148. DOI:10.3390/ijms20246148
|
| [27] |
MERCADAL JPR, ISAAC P, SIÑERIZ F, FERRERO MA. Indigo production by Pseudomonas sp. J26, a marine naphthalene-degrading strain[J]. Journal of Basic Microbiology, 2010, 50(3): 290-293. DOI:10.1002/jobm.200900276
|
| [28] |
LI YQ, LIN YW, WANG F, WANG JH, SHOJI O, XU JK. Construction of biocatalysts using the P450 scaffold for the synthesis of indigo from indole[J]. International Journal of Molecular Sciences, 2023, 24(3): 2395. DOI:10.3390/ijms24032395
|
| [29] |
KONG FH, CHEN J, QIN XQ, LIU CF, JIANG YP, MA L, XU HF, LI SY, CONG ZQ. Evolving a P450BM3 peroxygenase for the production of indigoid dyes from indoles[J]. ChemCatChem, 2022, 14: e202201151. DOI:10.1002/cctc.202201151
|
| [30] |
李妍清. 基于细胞色素P450构建吲哚合成靛蓝的生物催化剂[D]. 上海: 上海海洋大学硕士学位论文, 2023. LI YQ. Construction of biocatalysts using the P450 scaffold for the synthesis of indigo from indole[D]. Shanghai: Master's Thesis of Shanghai Ocean University, 2023 (in Chinese). |
| [31] |
ULLRICH R, PORAJ-KOBIELSKA M, HEROLD- MAJUMDAR OM, VIND J, HOFRICHTER M. Synthesis of indigo-dyes from indole derivatives by unspecific peroxygenases and their application for in-situ dyeing[J]. Catalysts, 2021, 11: 1495. DOI:10.3390/catal11121495
|
| [32] |
FABARA AN, FRAAIJE MW. Production of indigo through the use of a dual-function substrate and a bifunctional fusion enzyme[J]. Enzyme and Microbial Technology, 2020, 142: 109692. DOI:10.1016/j.enzmictec.2020.109692
|
| [33] |
丁爽, 陈修来, 高聪, 宋伟, 吴静, 魏婉清, 刘佳, 刘立明. 模块化工程改造大肠杆菌生产L-色氨酸[J]. 生物工程学报, 2023, 39(6): 2359-2374. DING S, CHEN XL, GAO C, SONG W, WU J, WEI WQ, LIU J, LIU LM. Modular engineering of Escherichia coli for high-level production of L-tryptophan[J]. Chinese Journal of Biotechnology, 2023, 39(6): 2359-2374 (in Chinese). |
| [34] |
LEE J, KIM J, SONG JE, SONG WS, KIM EJ, KIM YG, JEONG HJ, KIM HR, CHOI KY, KIM BG. Production of Tyrian purple indigoid dye from tryptophan in Escherichia coli[J]. Nature Chemical Biology, 2021, 17(1): 104-112. DOI:10.1038/s41589-020-00684-4
|
| [35] |
ROTILIO L, SWOBODA A, EBNER K, RINNOFNER C, GLIEDER A, KROUTIL W, MATTEVI A. Structural and biochemical studies enlighten the unspecific peroxygenase from Hypoxylon sp. EC38 as an efficient oxidative biocatalyst[J]. ACS Catalysis, 2021, 11(18): 11511-11525. DOI:10.1021/acscatal.1c03065
|
| [36] |
LIU C, XU JK, GAO SQ, HE B, WEI CW, WANG XJ, WANG ZH, LIN YW. Green and efficient biosynthesis of indigo from indole by engineered myoglobins[J]. RSC Advances, 2018, 8(58): 33325-33330. DOI:10.1039/C8RA07825D
|
| [37] |
DAI CX, MA Q, LI Y, ZHOU DD, YANG BY, QU YY. Application of an efficient indole oxygenase system from Cupriavidus sp. SHE for indigo production[J]. Bioprocess and Biosystems Engineering, 2019, 42(12): 1963-1971. DOI:10.1007/s00449-019-02189-4
|
| [38] |
YANG ZY, WANG RS, CHENG BY, RUAN V, YANG PJ, LIANG CH, SHEN CR. Key residues identified by random mutagenesis enhanced indole hydroxylation efficiency of the flavin-containing monooxygenase from Corynebacterium glutamicum[J]. Biochemical Engineering Journal, 2023, 199: 109064. DOI:10.1016/j.bej.2023.109064
|
| [39] |
YIN HF, CHEN HP, YAN M, LI ZK, YANG RD, LI YJ, WANG YF, GUAN JY, MAO HL, WANG Y, ZHANG YY. Efficient bioproduction of indigo and indirubin by optimizing a novel terpenoid cyclase XiaI in Escherichia coli[J]. ACS Omega, 2021, 6(31): 20569-20576. DOI:10.1021/acsomega.1c02679
|
| [40] |
MOURI T, KAMIYA N, GOTO M. New strategy to enhance catalytic performance of Escherichia coli whole cell biocatalysts harboring P450cam mutants[J]. Biochemical Engineering Journal, 2011, 53(2): 229-233. DOI:10.1016/j.bej.2010.09.016
|
| [41] |
LI G, YOUNG KD. Indole production by the tryptophanase TnaA in Escherichia coli is determined by the amount of exogenous tryptophan[J]. Microbiology, 2013, 159(Pt 2): 402-410.
|