代谢工程改造克雷伯氏菌生产1, 3-丙二醇
张少伦
,
高聪
,
李晓敏
,
刘佳
,
陈修来
,
刘立明
生物工程学报  2024, Vol. 40 2024, Vol. 40 Issue (8): 2386-2402 Issue (8): 2386-2402 |

1, 3-丙二醇(1, 3-propanediol, 1, 3-PDO)是一种重要的精细化工原料和医药中间体,主要用作聚酯和聚氨酯的合成单体。1, 3-丙二醇的生产方法主要分为化学合成法和生物合成法。化学合成法主要包括丙烯醛水解法和环氧乙烷羰基化法。(1) 丙烯醛水解法:以丙烯醛为原料,经水合制成3-羟基丙醛,然后催化加氢制得1, 3-丙二醇。由于丙烯醛自身的不稳定性会使其水合反应效率低,而且丙烯醛属剧毒易燃易爆品,难以储运和运输,同时反应过程中涉及贵金属催化,生产成本较高,该工艺已退出市场;(2) 环氧乙烷羰基化法:以乙烯为原料,在280 ℃下用银催化剂氧化成环氧乙烷,随后环氧乙烷在90 ℃、10 MPa条件下,外加催化剂作用生成1, 3-丙二醇。环氧乙烷羰基化法因为其技术难度大、设备投资高、催化剂体系复杂也已退出市场[1]。
利用微生物合成1, 3-丙二醇具有绿色可持续的优势,是目前主要的生产工艺,根据所使用底物的不同,可分为以葡萄糖为原料及以甘油为原料的2种工艺路线[2-5]:(1) 以葡萄糖为原料。使用葡萄糖为底物生产1, 3-丙二醇使用的底盘菌株多为大肠杆菌(Escherichia coli),由于大肠杆菌体内不存在直接将葡萄糖转化为1, 3-丙二醇的代谢路线,所以需要将酿酒酵母的dar1 (编码甘油-3-磷酸脱氢酶)和gpp2 (编码甘油-3-磷酸酶)基因转入大肠杆菌,使葡萄糖可以在大肠杆菌内转化为甘油,同时转入克雷伯氏菌来源的1, 3-丙二醇合成路径基因dhaB1、dhaB2、dhaB3、dhaBX和orfX使甘油转化为1, 3-丙二醇[6]。例如,杜邦公司在上述基础上,通过敲除glpK (编码甘油激酶)和gldA (编码甘油脱氢酶),阻止甘油进入碳代谢通路,并表达tpi (编码磷酸三糖异构酶)使甘油3-磷酸到磷酸二羟基丙酮的双向反应变为单向反应,构建的工程菌株在添加辅酶VB12条件下,1, 3-丙二醇产量为135 g/L,得率为0.50 g/g葡萄糖[7];北京化工大学谭天伟团队在大肠杆菌中设计了以葡萄糖为底物、天冬氨酸为前体的1, 3-丙二醇合成新路径,其1, 3-丙二醇产量在5 L发酵罐中为11 g/L,但无须添加辅酶VB12[8]。(2) 以甘油为原料。自然界中一些菌株,如肺炎克雷伯菌、丁酸梭菌、巴氏梭菌、弗氏柠檬酸杆菌、短乳杆菌,可以利用甘油为底物,天然合成1, 3-丙二醇。在厌氧或微需氧条件下,甘油首先通过VB12依赖性的甘油脱水酶转换为3-羟基丙醛(3-hydroxypropanal, 3-HPA),后者被NADH依赖性1, 3-PDO氧化还原酶还原为1, 3-丙二醇。目前,以甘油为原料的1, 3-丙二醇生产工艺大多采用克雷伯氏菌为底盘[9-12]。例如,清华大学刘德华教授团队通过对克雷伯氏菌的苹果酸酶基因进行过表达,增强了克雷伯氏菌三羧酸循环代谢通量,最终1, 3-丙二醇产量达106 g/L,得率为0.41 g/g甘油[13];华东理工大学宫衡教授团队通过在变栖克雷伯氏菌中增强基因tqsA (编码转运蛋白AI-2)的表达,工程菌株的1, 3-丙二醇产量达85 g/L,得率为0.50 g/g甘油[14]。以甘油作为底物用于生产1, 3-丙二醇比以葡萄糖为底物拥有更高的理论得率。但甘油作为生物柴油的主要副产物,在价格成本上受生物柴油产业影响波动较大,且相比于葡萄糖并无价格优势。因此如何提高甘油转化率及1, 3-丙二醇产量成为未来需要重点关注的研究方向。
本团队自无锡市蠡湖湖底淤泥中分离筛选获得了一株高产1, 3-丙二醇的克雷伯氏肺炎杆菌KP-wt。本研究以该菌株为研究对象,通过高通量筛选辅助的常压室温等离子(atmospheric room temperature plasma, ARTP)诱变技术并借助代谢工程策略,构建了一株重组克雷伯氏菌,该菌株在5 L发酵罐中1, 3-丙二醇产量为118 g/L。本研究为未来1, 3-丙二醇的工业化生产提供了技术支持。
1 材料与方法 1.1 材料 1.1.1 菌株、质粒和引物本研究所使用的重组质粒、工程菌株及引物分别见表 1、表 2和表 3。
| Plasmids | Relevant characteristics | Sources |
| pEM-mkate | PT5, KanR, f1 ori, mkate | Laboratory storage |
| pCasKP-Apr | PCat, AprR, Psc101 ori | Laboratory storage |
| pSGKP-Kan | PJ23119, KanR, f1 ori | Laboratory storage |
| pEM-Tac | PTac, KanR, f1 ori, mkate, test promtor PTac | This study |
| pEM-Trc | PTrc, KanR, f1 ori, mkate, test promtor PTrc | This study |
| pEM-KP | PKp, KanR, f1 ori, mkate, test promtor PKp | This study |
| pEM-Tac-RBS30 | pEM-Tac, RBS30, mkate, test promtor PTac and RBS30 | This study |
| pEM-Tac-RBS34 | pEM-Tac, RBS34, mkate, test promtor PTac and RBS34 | This study |
| pEM-Tac-RBS64 | pEM-Tac, RBS64, mkate, test promtor PTac and RBS64 | This study |
| pEM-Kp-RBS30 | pEM-Kp, RBS30, mkate, test promtor PKp and RBS30 | This study |
| pEM-Kp-RBS34 | pEM-Kp, RBS34, mkate, test promtor PKp and RBS34 | This study |
| pEM-Kp-RBS64 | pEM-Kp, RBS64, mkate, test promtor PKp and RBS64 | This study |
| pEM-T5-RBS30 | pEM-T5, RBS30, mkate, test promtor PT5 and RBS30 | This study |
| pEM-T5-RBS34 | pEM-T5, RBS34, mkate, test promtor PT5 and RBS34 | This study |
| pEM-T5-RBS64 | pEM-T5, RBS64, mkate, test promtor PT5 and RBS64 | This study |
| pSGKP-ldhA | pSGKP, ldhA-N20, Donor | This study |
| pSGKP-budA | pSGKP, budA-N20, Donor | This study |
| pSGKP-aldA | pSGKP, aldA-N20, Donor | This study |
| pSGKP-ackA | pSGKP, ackA-N20, Donor | This study |
| Strains | Relevant characteristics | Sources |
| KP-wt | Wild-type Klebsiella pneumoniae strain | Laboratory storage |
| KP-AR14 | After ARTP mutagenesis breeding from KP-wt | This study |
| KP-01 | KP-AR14ΔldhA | This study |
| KP-02 | KP-AR14ΔbudA | This study |
| KP-03 | KP-AR14ΔaldA | This study |
| KP-04 | KP-AR14ΔackA | This study |
| KP-05 | KP-AR14 H-dhaB | This study |
| KP-06 | KP-AR14 H-yqhD | This study |
| KP-07 | KP-AR14ΔldhAΔbudAΔaldA | This study |
| KP-FMME-1 | KP-07 H-dhaB-H-yqhd | This study |
| KP-FMME-2 | KP-07 H-dhaB-M-yqhd | This study |
| KP-FMME-3 | KP-07 H-dhaB-L-yqhd | This study |
| KP-FMME-4 | KP-07 M-dhaB-H-yqhd | This study |
| KP-FMME-5 | KP-07 M-dhaB-M-yqhd | This study |
| KP-FMME-6 | KP-07 M-dhaB-L-yqhd | This study |
| KP-FMME-7 | KP-07 L-dhaB-H-yqhd | This study |
| KP-FMME-8 | KP-07 L-dhaB-M-yqhd | This study |
| KP-FMME-9 | KP-07 L-dhaB-L-yqhd | This study |
| Primers | Sequences (5′→3′) | Size (bp) |
| Tac-pEM-F | TTGACAATTAATCATCGGCTCGTATAATGGATTCAATTGTGAGCGGATA | 49 |
| Tac-pEM-R | AGCCGATGATTAATTGTCAATTTCCTAGGCTACGCCGGACGCATCG | 46 |
| Trc-pEM-F | TTGACAATTAATCATCCGGCTCGTATAATGGATTCAATTGTGAGCGGATA | 50 |
| Trc-pEM-R | AGCCGATGATTAATTGTCAATTTCCTAGGCTACGCCGGACGCATCG | 46 |
| KP-pEM-F | CCTGAAGCGAGAAGGTATATTGATTCAATTGTGAGCGGATA | 41 |
| KP-pEM-R | AGCCGATGATTAATTGTCAATTTCCTAGGCTACGCCGGACG | 41 |
| ldhA-N20-F | TCCTAGGTATAATACTAGTTGGTCGCACTCCTCGGTATAGTTTTAGAGCTAGAAATAG | 58 |
| budA-N20-F | TCCTAGGTATAATACTAGTGCTGATCGCCTTCAGCAGTCGTTTTAGAGCTAGAAATAG | 58 |
| aldA-N20-F | TCCTAGGTATAATACTAGTCTGGGGGTGACCACCGGGATGTTTTAGAGCTAGAAATAG | 58 |
| ackA-N20-F | TCCTAGGTATAATACTAGTACACCGCGCTGATGGACGGCGTTTTAGAGCTAGAAATAG | 58 |
| N20-KZ-R | ACTAGTATTATACCTAGGACTGAGCTAGCTGTCA | 41 |
| UP-ldhA-F | CCGGTTTATTCCTTCCCAGC | 20 |
| UP-ldhA-R | GTACTTGAGATCTGAATCGGCAGCCTGCGAGTCA | 34 |
| Down-ldhA-F | ATTCAGATCTCAAGTACGAACAGCAGG | 27 |
| Down-ldhA-R | AAACAGTGGTTTCTCCATAAACACG | 25 |
| UP-budA-F | TGTTGCCGATTTCCTGGGGTAATTA | 25 |
| UP-budA-R | TCCATCTTATTGCCTCGCTTGAGGCAGCACAGGCCGATAG | 40 |
| Down-budA-F | AAGCGAGGCAATAAGATGGATTATA | 25 |
| Down-budA-R | CAGCCCGGGATGTACACATCGACCG | 25 |
| UP-aldA-F | GGCTGATAGCTGGTCATACT | 20 |
| UP-aldA-R | AACTTCGAAGCAGCTCCAGCAATGCTCTCCTGATAATGTT | 40 |
| Down-aldA-F | GCTGGAGCTGCTTCGAAGTT | 20 |
| Down-aldA-R | TCGCCAGCGTCAACTGGTTT | 20 |
| UP-ackA-F | TTTCAGCGTTCAGCGTTTTG | 20 |
| UP-ackA-R | TATCATAAAAAGGTACTTCCTTCCACATCGCCAGCCCTGA | 40 |
| Down-ackA-F | GGAAGTACCTTTTTATGATA | 20 |
| Down-ackA-R | CAGAGTCCCTGCATCGGCAG | 20 |
| Yqhd-UP-F | AGATGAGTATGACCTGACCC | 20 |
| Yqhd-UP-R | CAGCTGTCGCGACATTATACGAGCCGATGATTAATTGTCAATACAACTCGTCATTTTATTCTG | 63 |
| Yqhd-KZ-F | GGCTCGTATAATGTCGCGACAGCTGAAAGAGGAGAAACTGCAGATGAATAATTTCGACCTGCATACCCC | 69 |
| Yqhd-KZ-R | TTCGTACTTGAGATCTGAATTTACATCATGCCGCCCATAC | 40 |
| Yqhd-Down-F | ATAACTTCCCTGTGGGCATT | 20 |
| Yqhd-Down-R | AGCGAAAAGCGCTTCTTAAAC | 20 |
| dhaB-UP-F | CCGGTTTATTCCTTCCCAGC | 20 |
| dhaB-UP-R | GTACTTGAGATCTGAATCGGCAGCCTGCGAGTCA | 34 |
| dhaB-KZ-F dhaB-KZ-R |
TAATGTCGCGACAGCTGAAAGAGGAGAAACTGCAGATGAAGATCGTGTTAGTCCTTTACTTCGTACTTGAGATCTGAATTTATTATTTTTTATCGTGTT | 59 40 |
| dhaB-Down-F dhaB-Down-R |
ATTCAGATCTCAAGTACGAACAGCAGG AAACAGTGGTTTCTCCATAAACACG |
27 25 |
PCR扩增仪、凝胶成像仪、核酸胶电泳仪、电穿孔仪,Bio-Rad公司;恒温培养箱,上海跃进医疗器械有限公司;超声波清洗机,昆山市超声仪器有限公司;紫外可见分光光度计,岛津企业管理(中国)有限公司;M-100生物传感器,深圳市希尔曼科技有限公司;精密pH计,METTLER公司;高速离心机,Eppendorf公司;CHB-202恒温金属浴,Bioer Technology公司;5 L发酵罐,迪必尔生物工程(上海)有限公司。
Prime Star高保真酶、DNA marker购自宝生物工程(大连)有限公司;Taq DNA聚合酶购自生工生物工程(上海)股份有限公司;单片段一步同源重组酶、质粒提取试剂盒、胶回收试剂盒、细菌基因组提取试剂盒购自南京诺唯赞生物科技股份有限公司;PCR引物由苏州金唯智生物科技有限公司合成;其他试剂购自国药集团化学试剂有限公司。
1.1.3 培养基种子培养基(g/L):硫酸铵1,三水磷酸氢二钾3.2,磷酸二氢钾1.4,七水硫酸镁0.2,酵母粉1,甘油25,微量元素溶液1.2 mL。
发酵培养基(g/L):硫酸铵2,三水磷酸氢二钾0.85,磷酸二氢钾0.56,七水硫酸镁0.2,酵母粉1.5,甘油25,微量元素溶液2 mL。
微量元素溶液(g/L):四水硫酸锰0.08,氯化锌0.08,二水钼酸钠0.03,六水氯化钴0.35,硼酸0.06,五水硫酸铜0.03,六水氯化镍0.03,硫酸亚铁5,浓度为37%的盐酸4 mL。
以上培养基均在121 ℃条件下灭菌20 min,微量元素溶液过滤除菌。
1.2 方法 1.2.1 重组质粒的构建所有表达载体均通过一步同源重组法构建,根据目的片段与表达载体之间具有的同源臂序列相连接。以载体pEM为模板,用引物对Tac-pEM-F/Tac-pEM-R扩增PTac启动子片段。扩增产物均经琼脂糖凝胶回收后经过同源重组法连接,转化至大肠杆菌JM109中获得重组质粒pEM-Tac。其余重组质粒的构建方法均与此相同。
1.2.2 基因组整合菌株的构建基因组整合基因采用CRISPR-Cas9系统,具体方法参照文献[15-17]。以ldhA-N20-F/ N20-KZ-R为引物对,pSGKP-Kan为模板,进行全质粒PCR并胶回收,再经过同源重组连接并转化至E. coli JM109中获得重组质粒pSGKP-ldhA。以KP-wt为模板,用引物对UP-ldhA-F/UP-ldhA-R和Down-ldhA-F/Down- ldhA-R扩增基因ldhA的上下游600 bp同源臂;以扩增出的ldhA上下游同源臂胶回收产物为模板,用UP-ldhA-F/Down-ldhA-R引物对扩增ldhA表达框。通过制备重组菌株KP-wt的电转感受态,将片段ldhA与质粒pSGKP-ldhA按4:1的比例添加并电击转化导入菌株内,于30 ℃下培养24 h并对长出的单菌落进行验证。验证敲除成功后,通过在培养基中添加15%的蔗糖去除pSGKP质粒,37 ℃下培养18 h去除pCasKP质粒。其余整合菌株的构建方法均与此相同。
1.2.3 ARTP诱变将菌体在250 mL三角瓶中培养至OD650为0.8时收集1 mL菌体,使用无菌PBS重悬离心2次后将菌体重悬于100 μL无菌PBS中,吸取5 μL菌悬液滴在无菌金属载片中心后,置于ARTP育种机,使用He载气、100 W功率、2 mm辐照距离、10 standard liter per minute (SLM)气体流量分别辐照40、80、120和150 s。诱变结束后将载片置于装有1 mL种子培养基的1.5 mL离心管中,于超声波清洗机超声2 min洗脱菌体,将金属载片取出后离心收集菌体涂于100 g/L 1, 3-丙二醇浓度的无抗LB平板上,30 ℃培养24 h后进行筛选。
1.2.4 诱变菌株筛选方法以R值为指标[R值=诱变菌落直径(mm)/野生型菌落直径(mm)×菌落培养时间(h)],从含有100 g/L 1, 3-丙二醇的高渗固体筛选平板上,挑选具有较高R值的单菌落,置于无菌24孔板中扩大培养,然后转接400 μL至装有80 mL种子培养基的500 mL摇瓶中,37 ℃、200 r/min发酵培养24 h。1, 3-丙二醇产量较出发菌株高的突变菌株被重复验证3次。
1.3 菌体培养 1.3.1 种子培养取甘油管,将菌液三区划线接种至平板培养基上,在30 ℃培养箱中培养10−12 h。
一级种子液:使用接种环刮取半环三区划线后的菌体,然后接至装有50 mL种子培养基的250 mL摇瓶中,30 ℃、120 r/min培养5 h。
二级种子液:取400 μL一级种子液接入装有80 mL种子培养基的500 mL摇瓶中,30 ℃、220 r/min培养8−10 h。
1.3.2 摇瓶发酵移取400 μL种子液至装有80 mL发酵培养基的500 mL摇瓶中,37 ℃、200 r/min发酵24 h。每8 h测定甘油浓度进行补料,控制甘油浓度在40 g/L内。
1.3.3 5 L罐补料分批发酵发酵罐的装液量为3.5 L。按5%总体积的接种量将种子液接种至发酵罐。将发酵初始条件控制为:pH 6.3,转速300 r/min,通气量0.8 vvm,温度控制在37 ℃。当菌种开始产酸时流加30%氢氧化钠控制pH值为6.3。当初始甘油即将消耗完毕时,开始持续流加甘油至发酵结束,控制甘油浓度在40 g/L内。
1.4 分析方法细胞密度测定方法:采用比浊法,取0.1−3.0 mL发酵液,稀释0−20倍后振荡均匀,用紫外分光光度计于650 nm处测定吸光度(OD),发酵液的OD值为吸光度读数再乘以稀释倍数。
甘油测定方法:使用深圳希尔曼生物技术有限公司M-100生物传感器分析仪进行测定,取1.5 mL发酵液,12 000 r/min离心2 min,取上清液稀释10−100倍进行测定。
产物的测定:色谱柱为Bio-Rad Aminex HPX-87H柱,柱温65 ℃,流动相为0.005 mol/L H2SO4,流速为0.8 mL/min,检测器为Waters e2965型折光示差检测器,进样量为20 μL,取1.5 mL发酵液,12 000 r/min离心10 min,取上清液稀释100倍进行测定。
标准曲线的绘制:将分析纯的1, 3-丙二醇、乳酸(lactic acid, LAC)、乙酸(acetic acid, ACE)、乙醇(ethanol, EtOH)配制为0.3、0.6、0.9、1.2、1.5 g/L的稀释液,进行液相分析。根据得到的峰面积和相应的浓度作出标准曲线。
2 结果与分析 2.1 耐高渗底盘菌株的筛选为了研究出发菌株KP-wt的1, 3-丙二醇生产性能,在5 L发酵罐中对其进行了发酵试验,最终1, 3-丙二醇产量为75 g/L,得率为0.35 g/g甘油,生产强度为1.56 g/(h·L) (图 1A)。发酵过程中检测了发酵液渗透压,发现发酵液渗透压与产物浓度的积累呈正相关,最终达到1 550 mOmsmol/L (图 1B)。为了确定高渗透压对菌株生长和生产性能的影响,在不同1, 3-丙二醇浓度下测试了培养基渗透压与细胞生长的关系。结果表明,在发酵培养基中添加不同浓度的1, 3-丙二醇后,培养基渗透压随1, 3-丙二醇添加浓度呈正相关趋势(图 1C),添加60 g/L以上1, 3-丙二醇即可导致细胞生长受到明显抑制(图 1D)。综上可知,高产物浓度引起的高渗透压胁迫会抑制菌株KP-wt的生长和1, 3-丙二醇的合成,因此,提高菌株KP-wt对渗透压的耐受性,可能会提高菌株的1, 3-丙二醇产量。
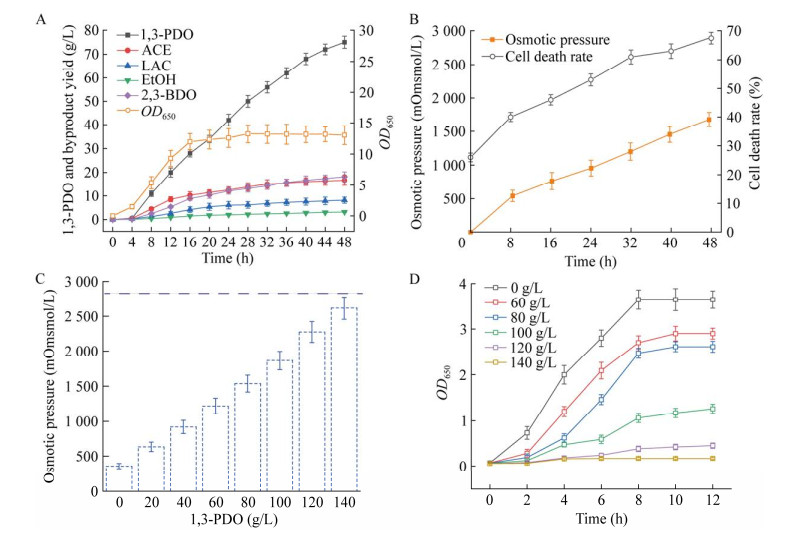
|
| 图 1 菌株KP-wt在5 L发酵罐中的发酵曲线变化 Fig. 1 Fermentation curve changes of strain KP-wt in a 5 L fermenter. A: 1, 3-PDO, 2, 3-butanediol (2, 3-BDO), LAC, ACE, EtOH and bacterial growth curves. B: Changes in osmotic pressure and cell death rate. C: Osmotic pressure changes with the addition of different concentrations of 1, 3-PDO in the fermentation medium. D: Effect of different concentrations of 1, 3-PDO on strain growth. A:1, 3-PDO、2, 3-丁二醇、乳酸、乙酸、乙醇和菌体生长曲线. B:渗透压和死亡率变化. C:发酵培养基中添加不同浓度1, 3-PDO渗透压变化曲线. D:不同浓度的1, 3-PDO对菌株生长的影响 |
| |
为了提高菌株对发酵液高渗透压的耐受性,本研究基于野生型KP-wt菌株进行了叠加ARTP诱变,将第一轮诱变获取的突变菌株以R > 48为评价标准进行筛选并进行下一轮诱变,最终经过3轮叠加诱变,挑取了其中R > 96的26株菌株进行摇瓶发酵测试(图 2A)。突变菌株KP-AR14在摇瓶中可生产5.6 g/L的1, 3-丙二醇,较出发菌株KP-wt提高了75%。最后,在摇瓶中对菌株KP-AR14进行了20代遗传稳定性测试,结果显示,菌株KP-AR14的1, 3-丙二醇产量较KP-wt稳定提升且不同代数之间变化幅度小,证明了菌株KP-AR14具有良好的遗传稳定性(表 4)。最终,选取了KP-AR14菌株进行5 L发酵罐测试,从细胞死亡率、细胞形态、菌体生长和生产性能4个方面与野生型菌株进行了比较。结果显示:菌株KP-AR14在发酵终点的死亡率(48%)较出发菌株KP-wt (68%)下降了29.4% (图 2B);在发酵过程中菌株KP-AR14的细胞表面破损较出发菌株KP-wt明显减少(图 2C);菌株KP-AR14的最大细胞生物量(dry cell weight, DCW) (5.92 g/L)较出发菌株KP-wt (5.45 g/L)提升了8.6%;菌株KP-AR14的1, 3-丙二醇产量、得率和生产强度分别为87 g/L、0.39 g/g甘油和1.81 g/(h·L),较KP-wt菌株分别提升了16.0%、11.4%和16.0% (图 2D)。上述结果表明,KP-AR14的渗透压耐受性与生长性能均得到提高,适合作为后续研究的底盘菌株。
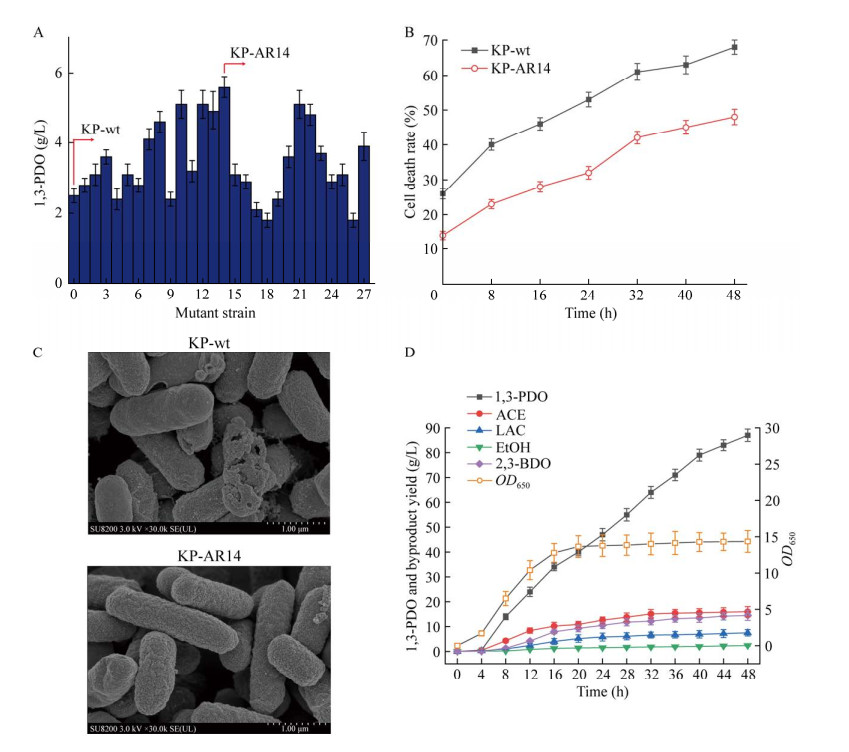
|
| 图 2 耐高渗1, 3-PDO生产菌株的筛选与表征 Fig. 2 Screening and characterization of high-osmolarity-tolerant 1, 3-PDO-producing strains. A: Production of 1, 3-PDO by mutant strains. B: Changes in mortality rate during fermentation. C: Morphological changes of bacterial cells after 48 h of fermentation. D: Fermentation curve of strain KP-AR14 in a 5 L fermenter. A:诱变菌株1, 3-PDO的生产情况. B:发酵过程死亡率变化. C:发酵48 h菌体形态变化情况. D:菌株KP-AR14在5 L发酵罐中的发酵曲线 |
| |
| Strains | Titer of 1, 3-PDO (g/L) | ||||
| 4th generation | 8th generation | 12th generation | 16th generation | 20th generation | |
| KP-wt | 3.2±0.1 | 2.8±0.4 | 3.3±0.3 | 3.5±0.2 | 2.9±0.3 |
| KP-AR14 | 5.6±0.2 | 5.4±0.3 | 5.8±0.4 | 5.5±0.1 | 5.3±0.3 |
克雷伯氏菌属于非模式菌株,缺乏表征明确的基因表达元件,如启动子和核糖体结合位点(ribosome binding site, RBS)序列。在进行分子改造前筛选出适宜其体内表达的不同强度的启动子及RBS,有助于理性调控基因的表达量。为了筛选出有效的启动子,根据大肠杆菌中的启动子表达强度分别选取了PTac、PTrc和PT5启动子,并在克雷伯氏菌基因组中筛选出了关键合成酶dhaT基因前300 bp序列作为潜在的启动子PKp进行扩增,将扩增片段连接至带有红色荧光蛋白的pEM表达载体中替换原有的启动子序列。分别将上述改造后的质粒转至KP-AR14菌中得到4株工程菌株,在摇瓶中用LB培养基培养至OD650约为0.6时添加IPTG诱导,分别在诱导3、6、9、12 h时取样测定荧光强度,结果如图 3A所示,PTac启动子在克雷伯氏菌中表达强度最高,分别是PKp和PT5启动子的1.4倍和4.4倍,而PTrc启动子在克雷伯氏菌中不表达。类似地,根据大肠杆菌中的RBS表达强度,选取了RBS64、RBS34和RBS30与PTac启动子进行组合表达,测试不同RBS的表达强度,实验操作和条件同上所述,结果如图 3B所示,RBS30在克雷伯氏菌中表达强度最高,分别是RBS34、RBS64的1.05倍和1.13倍。
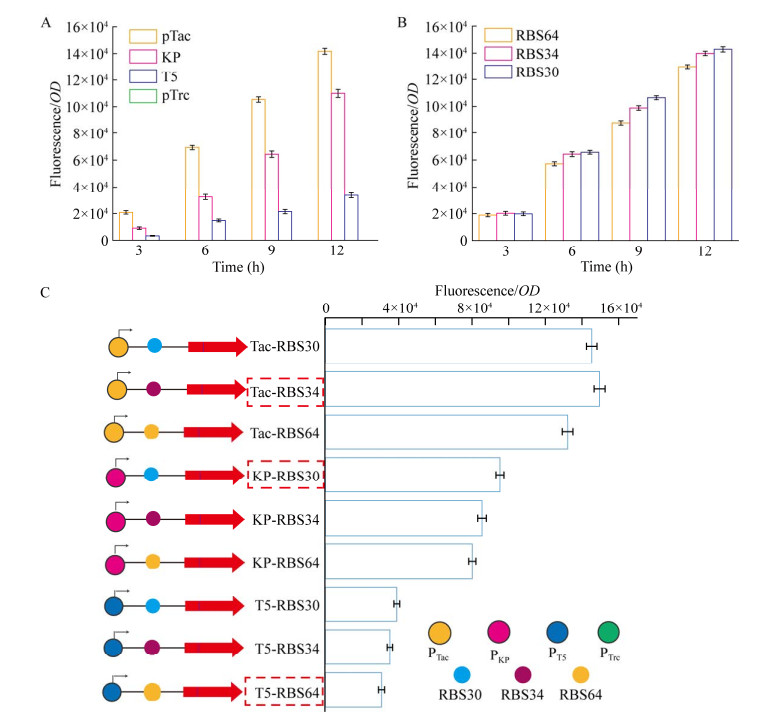
|
| 图 3 表达元件的筛选与评估 Fig. 3 Screening and evaluation of expression elements. A: Characterization of expression strength of different promoters. B: Characterization of expression strength of different ribosome binding sites (RBS). C: Screening results of expression elements. A:不同启动子表达强度表征结果. B:不同RBS表达强度表征结果. C:表达元件筛选结果 |
| |
为了筛选出不同表达强度的启动子及RBS序列,将PTac、PKp和PT5启动子与RBS64、RBS34和RBS30进行了交叉组合表达,实验操作和条件同上所述,结果如图 3C所示,PTac与RBS34组合(H)在克雷伯氏菌中的表达强度最高,是PKp与RBS30组合(M)和PT5与RBS64组合(L)的1.6倍和4.1倍。上述结果表明,在克雷伯氏菌中将不同表达的启动子及RBS组合能够有效地将基因表达量调控在高、中和低3个水平。
2.3 阻断冗余代谢支路提高1, 3-丙二醇产量尽管通过诱变筛选后得到的最优底盘菌株与出发菌株相比,菌株生产性能及耐受性均大幅提高,但是发酵过程中仍会产生18.2 g/L 2, 3-丁二醇(2, 3-butanediol, 2, 3-BDO)、6.4 g/L乳酸、15.3 g/L乙酸和1.8 g/L乙醇,这使得1, 3-丙二醇实际得率严重降低。通过分析代谢通路(图 4A)发现,可能是由于副产物2, 3-丁二醇、乳酸、乙酸及乙醇的合成路径与1, 3-丙二醇的合成路径都需要NADH作为辅因子,从而导致胞内NADH缺乏,严重影响了1, 3-丙二醇得率[18]。
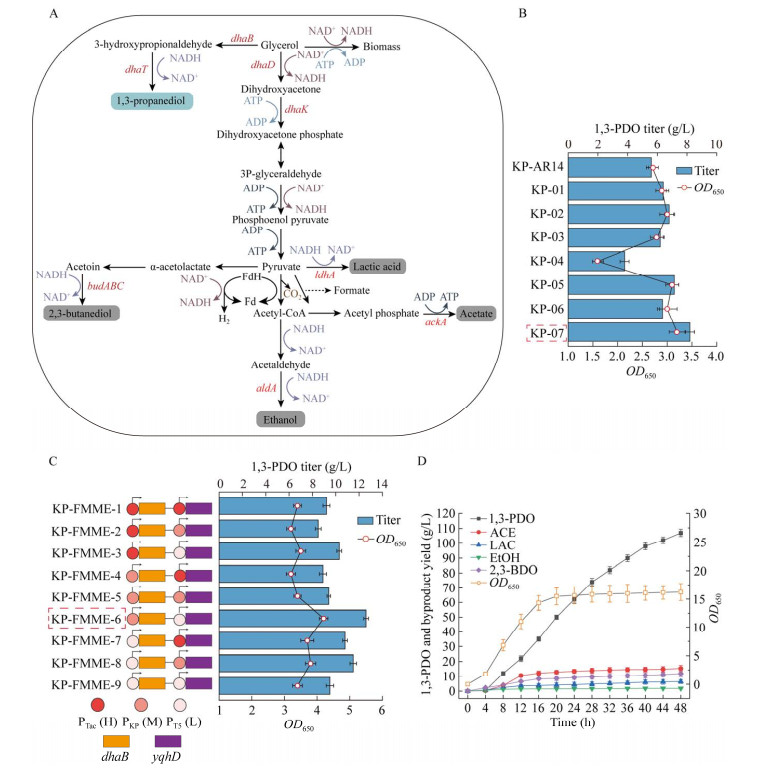
|
| 图 4 工程菌株构建与评估 Fig. 4 Construction and evaluation of engineered strains. A: 1, 3-PDO synthesis pathway in Klebsiella pneumoniae. B: Production of 1, 3-PDO by engineered strains. C: Cell growth and 1, 3-propanediol production of different strains with combination of promoters and RBS. D: Fermentation curve of strain KP-FMME-6 in a 5 L fermenter. A:克雷伯氏菌1, 3-PDO合成路径. B:工程菌株的1, 3-PDO生产情况. C:启动子RBS组合后高产菌株生长及生产情况. D:菌株KP-FMME-6在5 L发酵罐中的发酵曲线 |
| |
为了有效降低副产物积累,选择通过阻断冗余代谢支路将更多还原力用于1, 3-丙二醇生产。以KP-AR14菌株为底盘,在基因组中分别敲除了编码D-乳酸脱氢酶的ldhA基因、编码α-乙酰乳酸脱羧酶的budA基因、编码乙醛脱氢酶的aldA基因和编码乙酸激酶的ackA基因,构建菌株KP-02、KP-03和KP-04。
在摇瓶条件下:(1) 分别单独敲除ldhA、budA和aldA后,工程菌株的1, 3-丙二醇产量分别提高了14.3%、21.4%和10.7%,副产物乳酸、2, 3-丁二醇和乙醇在摇瓶中未检出;(2) 单独敲除ackA基因的菌株1, 3-丙二醇产量降低了32.1%,可能是因为敲除乙酸激酶后菌株生长受到影响,导致1, 3-丙二醇产量降低;(3) 同时敲除ldhA、budA和aldA的菌株KP-07的1, 3-丙二醇产量提高了46.4%,副产物乳酸、2, 3-丁二醇和乙酸在摇瓶中未检测到(图 4B)。上述结果表明,阻断冗余代谢支路关键基因能够有效地提高1, 3-丙二醇产量。
2.4 强化合成路径提高1, 3-丙二醇生产强度为了提高1, 3-丙二醇的合成效率,针对合成路径[19-20],以菌株KP-AR14为底盘,分别以PTac启动子和RBS34的组合过表达了本源的甘油脱水酶dhaB基因,并异源表达了来自大肠杆菌的1, 3-PDO氧化还原酶yqhD基因,构建工程菌株KP-05和KP-06。经摇瓶发酵测试,菌株KP-05和KP-06的1, 3-丙二醇产量分别提高了27.4%和13.9% (图 4B)。
为进一步提高1, 3-丙二醇的产量和得率,对阻断冗余代谢支路和强化合成路径中有效的基因靶点进行了启动子与RBS的组合优化,探究不同的表达强度对菌株生产的影响,从而构建了9株基因工程菌,分别命名为菌株KP-FMME-1至KP-FMME-9。在摇瓶条件下,菌株KP-FMME-6的1, 3-丙二醇生产能力最优,1, 3-丙二醇产量达到了12.6 g/L,较KP-AR14提升125.4%,菌株生长较KP-AR14提高18.5% (图 4C)。为了检测菌株KP-FMME-6的生产潜力,在5 L发酵罐上进行了补料分批发酵,发酵周期为48 h,其1, 3-丙二醇产量、得率及生产强度分别为107 g/L、0.41 g/g甘油和2.22 g/(h·L),与菌株KP-AR14相比分别提高了23.5%、7.6%和22.7%,菌株生长较KP-AR14相比提高13.2%。另外,菌株KP-FMME-6的2, 3-丁二醇(11.8 g/L)、乙酸(15.3 g/L)、乳酸(6.4 g/L)和乙醇(1.8 g/L)产量,分别较菌株KP-AR14降低了18.6%、4.3%、14.7%和25.0% (图 4D)。上述结果表明,阻断副产物代谢路径及强化1, 3-丙二醇合成路径能有效地提升菌体的生产能力。
2.5 优化发酵过程参数强化1, 3-丙二醇合成为了进一步提高菌株KP-FMME-6的1, 3-丙二醇生产潜力,分别对发酵过程中的pH、溶氧和补料速率进行了优化。
对于pH值优化,基于文献报道[21],设计了2种条件,即每间隔8 h分别将发酵体系的pH值在6.3和6.8之间切换和在6.8和7.3之间切换。发酵结果表明,每隔8 h在pH值6.3和6.8之间切换为最优条件,菌株的1, 3-丙二醇产量、得率及生产强度分别为112 g/L、0.42 g/g甘油和2.34 g/(h·L),副产物积累分别为2, 3-丁二醇(9.6 g/L)、乙酸(9.25 g/L)、乳酸(4.3 g/L)和乙醇(0.8 g/L) (图 5A、5B)。
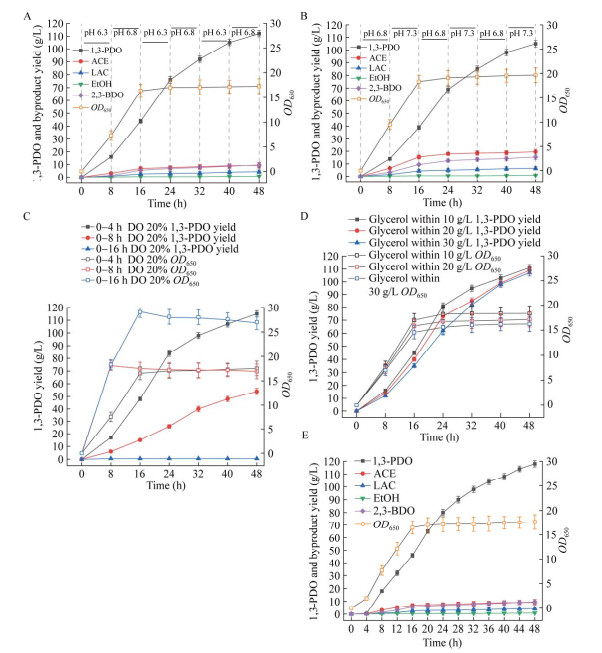
|
| 图 5 菌株KP-FMME-6的发酵工艺优化结果 Fig. 5 Fermentation process optimization results of strain KP-FMME-6. A: Fermentation curve with pH switching between 6.3 and 6.8. B: Fermentation curve with pH switching between 6.8 and 7.3. C: Fermentation curve of strain with optimized dissolved oxygen conditions. D: Fermentation curve of strain with optimized feeding rate. E: Fermentation curve of strain under optimal fermentation conditions in a 5 L fermenter. A:pH在6.3和6.8之间切换的发酵曲线. B:pH在6.8和7.3之间切换的发酵曲线. C:优化溶氧条件的菌株发酵曲线. D:优化补料速率的菌株发酵曲线. E:最优发酵工艺下菌株在5 L发酵罐中的发酵曲线 |
| |
对于溶氧条件优化,设计了3个条件:即分别在发酵0−4 h、0−8 h和0−16 h控制溶氧不低于20%。发酵结果表明,在发酵前4 h内将溶氧控制在20%,后续将转速通气降为与初始发酵条件一致时,菌株的生长及产能最优,1, 3-丙二醇产量、得率及生产强度分别为115 g/L、0.43 g/g甘油和2.4 g/(h·L) (图 5C)。
对于补料速率优化,设计了3个条件,即分别在菌体生长阶段(发酵0−16 h)控制发酵液甘油含量在10 g/L、20 g/L和30 g/L,菌体达到最大细胞密度(16 h)后控制甘油含量在30 g/L以内。发酵结果表明,在菌体生长阶段将甘油含量控制在10 g/L,菌体达到最大细胞密度后将甘油含量控制在30 g/L时,菌株的生长及产能最优,1, 3-丙二醇产量、得率及生产强度分别为111 g/L、0.44 g/g甘油和2.31 g/(h·L) (图 5D)。
综合上述最优发酵工艺条件,菌株KP-FMME-6在发酵48 h后,最大DCW为7.27 g/L,1, 3-丙二醇的产量、得率和生产强度分别达到了118 g/L、0.45 g/g甘油和2.46 g/(h·L),较菌株KP-wt分别提高了57.3%、20%和57.7%。菌株KP-FMME-6在发酵过程中仅积累了少量的副产物,如9.6 g/L 2, 3-丁二醇、9.25 g/L乙酸、0.8 g/L乙醇和4.3 g/L乳酸,较KP-wt分别降低了47.3%、39.5%、55.6%和32.8% (图 5E)。上述结果表明,通过优化发酵过程参数,可以提高菌株1, 3-丙二醇生产能力,同时减少副产物的积累。
3 讨论与结论本研究以实验室保藏的KP-wt作为底盘菌株,通过ARTP诱变、冗余代谢支路阻断以及合成路径强化,成功构建了一株工程菌株KP-FMME-6。该菌株在摇瓶中发酵1, 3-丙二醇产量可达到12.6 g/L,在5 L发酵罐体系中经分批补料发酵,1, 3-丙二醇的产量、得率和生产强度分别达到了118 g/L、45 g/g甘油和2.46 g/(h·L),具有良好的工业化潜力。
在1, 3-丙二醇发酵过程中,产物积累所造成的高渗透压会严重抑制菌体生长及产物合成。为了提高菌株1, 3-丙二醇合成效率,本研究通过ARTP叠加诱变筛选出一株能耐受100 g/L浓度1, 3-丙二醇的克雷伯氏菌KP-AR34,通过增强菌株的耐受性,有效减少了菌株在发酵过程中受到的产物抑制情况。此外,本研究首次在克雷伯氏菌中通过启动子和RBS筛选,得到了能够在克雷伯氏菌中将目标基因的表达量调控在高(H)、中(M)和低(L) 3个水平的表达元件。最后,本研究采用了阻断冗余代谢支路及强化合成通路的代谢工程策略,所构建的最优工程菌株KP-FMME-6的1, 3-丙二醇产量、得率和生产强度分别较野生型菌株分别提高了57.3%、20.2%和57.7%。
虽然本研究所构建的工程菌株1, 3-丙二醇产量已是目前文献报道中通过甘油生产1, 3-丙二醇的最高产量,但是现阶段的菌株的1, 3-丙二醇产量及转化率距离杜邦公司的生产指标还有一定差距。导致这一结果可能的原因有3个:(1) 1, 3-丙二醇氧化还原酶的酶活催化活性不足。本研究目前仅对合成路径进行了强化,尚未对合成酶进行酶工程改造[22-23];(2) 1, 3-丙二醇氧化还原酶是氧气敏感性,为保证酶活只能进行微需氧发酵,在微需氧环境中菌体生长受限[24];(3) 克雷伯氏菌中的冗余代谢支路较多,且较多代谢支路影响细胞生长,无法进行完全阻断,导致仍有较多代谢流进入副产物代谢路径中[25-26]。
为了进一步提高1, 3-丙二醇的产量及甘油转化率,后续的研究可以从以下3个方面进行探索:(1) 提高1, 3-丙二醇氧化还原酶的活性及特性。通过酶工程改造对关键合成酶进行蛋白质改造,从而提高酶的催化活性及氧敏感性,以提高1, 3-丙二醇的产量;(2) 强化胞内1, 3-丙二醇的外排能力。醇类产物在发酵过程中会因自身的特性,使发酵液中的渗透压随着产物浓度的积累而升高,导致菌体活力下降甚至死亡。1, 3-丙二醇的外排能力不足也是抑制产物高效合成的重要因素之一[27];(3) 反向代谢工程挖掘新靶点。通过组学分析可对诱变菌株进行全基因组分析,找到能使菌株耐受性增强的关键靶点,从而对工程菌株进行深度改造,将有助于进一步提升菌株的整体生产性能[28-33] (表 5)。
| Strains | Substrates | Titer (g/L) | Yield (g/g) | Productivity (g/(L·h)) | References |
| E. coli | Glucose | 135.0 | 0.50 | 3.50 | [7] |
| E. coli | Glucose | 11.2 | 0.22 | 0.10 | [8] |
| Corynebacterium glutamicum | Glucose, xylose | 110.0 | 0.42 | 2.30 | [5] |
| C. butyricum | Glycerol | 73.0 | 0.58 | 2.48 | [31] |
| C. butyricum | Glycerol | 60.2 | 0.51 | 1.72 | [12] |
| Vibrio natriegens | Glycerol | 56.2 | 0.50 | 2.36 | [3] |
| Lactobacillus reuteri | Glucose, glycerol | 93.2 | 0.81 (glycerol) | 1.29 | [32] |
| Clostridium sp. | Glycerol | 81.2 | 0.49 | 4.27 | [33] |
| K. pneumoniae | Glycerol | 106.0 | 0.43 | 2.15 | [13] |
| K. pneumoniae | Glucose | 62.0 | 0.54 | 0.94 | [6] |
| K. pneumoniae | Glycerol | 118.0 | 0.45 | 2.46 | This study |
| [1] |
CELIŃSKA E. Debottlenecking the 1, 3-propanediol pathway by metabolic engineering[J]. Biotechnology Advances, 2010, 28(4): 519-530.
|
| [2] |
ZHU FH, LIU DH, CHEN Z. Recent advances in biological production of 1, 3-propanediol: new routes and engineering strategies[J]. Green Chemistry, 2022, 24(4): 1390-1403.
|
| [3] |
ZHANG Y, LI ZH, LIU Y, CEN XC, LIU DH, CHEN Z. Systems metabolic engineering of Vibrio natriegens for the production of 1, 3-propanediol[J]. Metabolic Engineering, 2021, 65: 52-65.
|
| [4] |
LI ZH, WU ZY, CEN XC, LIU Y, ZHANG Y, LIU DH, CHEN Z. Efficient production of 1, 3-propanediol from diverse carbohydrates via a non-natural pathway using 3-hydroxypropionic acid as an intermediate[J]. ACS Synthetic Biology, 2021, 10(3): 478-486.
|
| [5] |
LI ZH, DONG YF, LIU Y, CEN XC, LIU DH, CHEN Z. Systems metabolic engineering of Corynebacterium glutamicum for high-level production of 1, 3-propanediol from glucose and xylose[J]. Metabolic Engineering, 2022, 70: 79-88.
|
| [6] |
LAMA SM, SEOL E, PARK S. Development of Klebsiella pneumoniae J2B as microbial cell factory for the production of 1, 3-propanediol from glucose[J]. Metabolic Engineering, 2020, 62: 116-125.
|
| [7] |
NAKAMURA CE, WHITED GM. Metabolic engineering for the microbial production of 1, 3-propanediol[J]. Current Opinion in Biotechnology, 2003, 14(5): 454-459.
|
| [8] |
LI MD, ZHANG Y, LI JC, TAN TW. Biosynthesis of 1, 3-propanediol via a new pathway from glucose in Escherichia coli[J]. ACS Synthetic Biology, 2023, 12(7): 2083-2093.
|
| [9] |
CHEN Z, LIU HJ, LIU DH. Metabolic pathway analysis of 1, 3-propanediol production with a genetically modified Klebsiella pneumoniae by overexpressing an endogenous NADPH-dependent alcohol dehydrogenase[J]. Biochemical Engineering Journal, 2011, 54(3): 151-157.
|
| [10] |
ZHU YT, WANG YX, GAO H, WANG HX, WAN ZJ, JIANG YJ, XIN FX, ZHANG WM, JIANG M. Current advances in microbial production of 1, 3-propanediol[J]. Biofuels, Bioproducts and Biorefining, 2021, 15(5): 1566-1583.
|
| [11] |
CHEN ZF, LIU JQ, HAN X, MA LN, XU P, TAO F. Developing a thermophilic cell factory for high-temperature production of 1, 3-propanediol via host-mining and metabolic engineering[J]. Chem Catalysis, 2023, 3(8): 100704.
|
| [12] |
YUN JH, ZABED HM, ZHANG YF, ZHANG GY, ZHAO M, QI XH. Improving tolerance and 1, 3-propanediol production of Clostridium butyricum using physical mutagenesis, adaptive evolution and genome shuffling[J]. Bioresource Technology, 2022, 363: 127967.
|
| [13] |
XU YZ, GUO NN, ZHENG ZM, OU XJ, LIU HJ, LIU DH. Metabolism in 1, 3-propanediol fed-batch fermentation by a D-lactate deficient mutant of Klebsiella pneumoniae[J]. Biotechnology and Bioengineering, 2009, 104(5): 965-972.
|
| [14] |
ZHONG ZH, LIU LF, ZHOU JJ, GAO LR, XU JJ, FU SL, GONG H. Influences of 3-hydroxypropionaldehyde and lactate on the production of 1, 3-propanediol by Klebsiella pneumoniae[J]. Bioresources & Bioprocessing, 2014. DOI:10.1186/s40643-014-0002-z
|
| [15] |
WANG Y, WANG SS, CHEN WZ, SONG LQ, ZHANG YF, SHEN Z, YU FY, LI M, JI QJ. CRISPR-Cas9 and CRISPR-assisted cytidine deaminase enable precise and efficient genome editing in Klebsiella pneumoniae[J]. Applied and Environmental Microbiology, 2018, 84(23): e01834-18.
|
| [16] |
WU ZW, ZHANG YF, YU HP, PAN D, WANG YJ, WANG YN, LI F, LIU C, NAN H, CHEN WZ, JI QJ. Programmed genome editing by a miniature CRISPR-Cas12f nuclease[J]. Nature Chemical Biology, 2021, 17: 1132-1138.
|
| [17] |
ZHANG YW, SUN XM, WANG QZ, XU JQ, DONG F, YANG SQ, YANG JW, ZHANG ZX, QIAN Y, CHEN J, ZHANG J, LIU YM, TAO RS, JIANG Y, YANG JJ, YANG S. Multicopy chromosomal integration using CRISPR-associated transposases[J]. ACS Synthetic Biology, 2020, 9(8): 1998-2008.
|
| [18] |
王学明, 潘静宇, 吴静, 陈修来, 高聪, 宋伟, 魏婉清, 刘佳, 刘立明. 调控大肠杆菌胞内ATP和NADH水平促进琥珀酸生产[J]. 生物工程学报, 2023, 39(8): 3236-3252. WANG XM, PAN JY, WU J, CHEN XL, GAO C, SONG W, WEI WQ, LIU J, LIU LM. Regulation of intracellular level of ATP and NADH in Escherichia coli to promote succinic acid production[J]. Chinese Journal of Biotechnology, 2023, 39(8): 3236-3252 (in Chinese). |
| [19] |
丁爽, 陈修来, 高聪, 宋伟, 吴静, 魏婉清, 刘佳, 刘立明. 模块化工程改造大肠杆菌生产L-色氨酸[J]. 生物工程学报, 2023, 39(6): 2359-2374. DING S, CHEN XL, GAO C, SONG W, WU J, WEI WQ, LIU J, LIU LM. Modular engineering of Escherichia coli for high-level production of L-tryptophan[J]. Chinese Journal of Biotechnology, 2023, 39(6): 2359-2374 (in Chinese). |
| [20] |
刘洁, 高聪, 陈修来, 郭亮, 宋伟, 吴静, 魏婉清, 刘佳, 刘立明. 代谢工程改造大肠杆菌合成己二酸[J]. 生物工程学报, 2023, 39(6): 2375-2389. LIU J, GAO C, CHEN XL, GUO L, SONG W, WU J, WEI WQ, LIU J, LIU LM. Metabolic engineering of Escherichia coli for adipic acid production[J]. Chinese Journal of Biotechnology, 2023, 39(6): 2375-2389 (in Chinese). |
| [21] |
YANG XG, CHOI HS, LEE JH, LEE SK, HAN SO, PARK C, KIM SW. Improved production of 1, 3-propanediol from biodiesel-derived crude glycerol by Klebsiella pneumoniae in fed-batch fermentation[J]. Chemical Engineering Journal, 2018, 349: 25-36.
|
| [22] |
WANG WJ, YU X, WEI YJ, LEDESMA-AMARO R, JI XJ. Reprogramming the metabolism of Klebsiella pneumoniae for efficient 1, 3-propanediol production[J]. Chemical Engineering Science, 2021, 236: 116539.
|
| [23] |
JIANG W, WANG SZ, WANG YP, FANG BS. Key enzymes catalyzing glycerol to 1, 3-propanediol[J]. Biotechnology for Biofuels, 2016, 9: 57.
|
| [24] |
CHEN Z, LIU DH. Toward glycerol biorefinery: metabolic engineering for the production of biofuels and chemicals from glycerol[J]. Biotechnology for Biofuels, 2016, 9: 205.
|
| [25] |
ZHANG YP, LI Y, DU CY, LIU M, CAO ZA. Inactivation of aldehyde dehydrogenase: a key factor for engineering 1, 3-propanediol production by Klebsiella pneumoniae[J]. Metabolic Engineering, 2006, 8(6): 578-586.
|
| [26] |
ZHANG YQ, JIA ZX, LIN J, XU DF, FU SL, GONG H. Deleting pck improves growth and suppresses by-product formation during 1, 3-propanediol fermentation by Klebsiella pneumoniae[J]. Journal of Applied Microbiology, 2017, 123(3): 678-687.
|
| [27] |
TENG Y, GUO C, XIE MM, FENG A, LU XY, ZONG H, ZHUGE B. Modification of substrate and product transport systems in Klebsiella pneumoniae to improve 1, 3-propanediol production[J]. FEMS Microbiology Letters, 2022, 369(1): fnac056.
|
| [28] |
LEE JH, JUNG HM, JUNG MY, OH MK. Effects of gltA and arcA mutations on biomass and 1, 3-propanediol production in Klebsiella pneumoniae[J]. Biotechnology and Bioprocess Engineering, 2019, 24(1): 95-102.
|
| [29] |
FOKUM E, ZABED HM, YUN J, ZHANG G, QI X. Recent technological and strategical developments in the biomanufacturing of 1, 3-propanediol from glycerol[J]. International Journal of Environmental Science and Technology, 2021, 18(8): 2467-2490.
|
| [30] |
WANG M, TIAN YJ, XU L, ZHANG FS, LU HG, LI MR, LI B. High osmotic stress increases OmpK36 expression through the regulation of KbvR to decrease the antimicrobial resistance of Klebsiella pneumoniae[J]. Microbiology Spectrum, 2022, 10(3): e0050722.
|
| [31] |
MARTINS FF, Da S S LIBERATO V, RIBEIRO CMS, COELHO MAZ, FERREIRA TF. Low-cost medium for 1, 3-propanediol production from crude glycerol by Clostridium butyricum[J]. Biofuels, Bioproducts and Biorefining, 2020, 14(5): 1125-1134.
|
| [32] |
JU JH, HEO SY, CHOI SW, KIM YM, KIM MS, KIM CH, OH BR. Effective bioconversion of 1, 3-propanediol from biodiesel-derived crude glycerol using organic acid resistance-enhanced Lactobacillus reuteri JH83[J]. Bioresource Technology, 2021, 337: 125361.
|
| [33] |
ZHANG CJ, SHARMA S, MA CW, ZENG AP. Strain evolution and novel downstream processing with integrated catalysis enable highly efficient coproduction of 1, 3-propanediol and organic acid esters from crude glycerol[J]. Biotechnology and Bioengineering, 2022, 119(6): 1450-1466.
|